È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Preparazione di microcapsule multifunzionali a base di seta caricate con plasmidi di DNA codificanti aptameri e ribointerruttori RNA
In questo articolo
Riepilogo
Il protocollo descrive la formazione di microcapsule robuste e biocompatibili cariche di DNA come biosensori in vitro multiplex in grado di tracciare diversi ligandi.
Abstract
Introduciamo un protocollo per la preparazione di microcapsule di fibroina di seta cariche di DNA tramite il metodo di assemblaggio Layer-by-Layer (LbL) su nuclei sferici sacrificali. Dopo l'adsorbimento di uno strato primario e plasmidi di DNA, la formazione di microcapsule robuste è stata facilitata inducendo fogli di β nella struttura secondaria della seta durante la disidratazione acuta di un singolo strato di seta. Quindi, la stratificazione è avvenuta tramite legami idrogeno multipli e interazioni idrofobiche. Dopo l'adsorbimento di gusci multistrato, le strutture del guscio centrale possono essere ulteriormente funzionalizzate con nanoparticelle d'oro (AuNP) e / o anticorpi (IgG) da utilizzare per il telerilevamento e / o la consegna mirata. La regolazione di diversi parametri chiave durante la deposizione sequenziale di macromolecole chiave su nuclei di silice come la presenza di un primer polimerico, la concentrazione di DNA e proteine della seta, nonché un numero di strati adsorbiti ha portato a microcapsule biocompatibili, cariche di DNA con permeabilità e carichi di DNA variabili. Dopo la dissoluzione dei nuclei di silice, il protocollo ha dimostrato la formazione di microcapsule cave e robuste con plasmidi di DNA immobilizzati sulla superficie interna della membrana della capsula. La creazione di una membrana biocompatibile selettivamente permeabile tra i plasmidi del DNA e l'ambiente esterno ha preservato il DNA durante la conservazione a lungo termine e ha svolto un ruolo importante nel miglioramento della risposta di uscita dai plasmidi spazialmente confinati. L'attività dei modelli di DNA e la loro accessibilità sono state testate durante le reazioni di trascrizione e traduzione in vitro (sistemi cell-free). I plasmidi del DNA che codificano aptameri e riboswitch di illuminazione dell'RNA sono stati attivati con successo con analiti corrispondenti, come è stato visualizzato durante la localizzazione di trascritti di RNA marcati con fluorescenza o proteina GFPa1 nelle membrane del guscio.
Introduzione
Il campo della biologia sintetica offre opportunità uniche per sviluppare capacità di rilevamento sfruttando meccanismi naturali evoluti dai microrganismi per monitorare il loro ambiente e le potenziali minacce. È importante sottolineare che questi meccanismi di rilevamento sono tipicamente legati a una risposta che protegge questi microrganismi dall'esposizione dannosa, regolando l'espressione genica per mitigare gli effetti negativi o prevenire l'assunzione di materiali tossici. Ci sono stati sforzi significativi per progettare questi microrganismi per creare sensori a cellule intere sfruttando queste risposte naturali ma reindirizzandoli a riconoscere nuovi bersagli e / o a produrre un segnale misurabile che può essere misurato a fini di quantificazione (tipicamente fluorescenza)1,2. Attualmente, le preoccupazioni relative all'uso di microrganismi geneticamente modificati (OGM), specialmente quando vengono rilasciati nell'ambiente o nel corpo umano, a causa della fuoriuscita di cellule intere o di parte del loro materiale genetico, anche se incapsulato in una matrice polimerica, suggeriscono che sono necessari modi alternativi per sfruttare questi approcci di rilevamento3.
Un approccio efficace per sfruttare i vantaggi del rilevamento basato su microrganismi senza preoccuparsi della diffusione di OGM è l'uso di sistemi di trascrizione/traduzione in vitro (IVTT). Da un punto di vista pratico, i sistemi IVTT consistono in una miscela contenente la maggior parte dei componenti cellulari in uno stato attivo che è stata "estratta" dalle cellule con diversi mezzi, tra cui sonicazione, battito di perline o altri4. Il prodotto finale di questo processo è una miscela di reazione biochimica già ottimizzata per eseguire la trascrizione e la traduzione che può essere utilizzata per testare diversi sensori in un formato "open vessel", senza i vincoli associati all'uso di cellule intere (diffusione di membrana, efficienza di trasformazione, tossicità cellulare, ecc.). È importante sottolineare che diversi componenti del sensore possono essere aggiunti quantitativamente e il loro effetto studiato con diverse tecniche ottiche e spettrometriche, come abbiamo dimostrato5. È stato notato che le prestazioni dei sistemi IVTT possono essere incoerenti; Tuttavia, studi recenti hanno mostrato approcci per standardizzare la loro preparazione e caratterizzazione, il che è di grande aiuto quando si studiano le loro prestazioni nella progettazione dei sensori6. Recentemente, sono stati dimostrati molti esempi di sistemi IVTT che utilizzano per creare saggi cartacei attraverso la liofilizzazione dei loro componenti in matrici cartacee, tra cui il rilevamento di ioni di metalli pesanti, farmaci, elementi di quorum sensing e altri 7,8,9. Uno spazio applicativo interessante per i sensori basati su IVTT è il loro utilizzo in applicazioni di rilevamento in diversi tipi di ambienti, tra cui suolo, acqua e corpo umano. Per distribuire questi sistemi IVTT in questi ambienti difficili, è necessario implementare un approccio di incapsulamento per contenere i componenti IVTT e proteggerli dal degrado.
Gli approcci di incapsulamento più comuni per i sistemi IVTT includono l'uso di capsule lipidiche, micelle, polimerisomi e altri microcontenitori strettamente chiusi10,11,12. Uno svantaggio di questo approccio è la necessità di incorporare meccanismi passivi o attivi per trasportare materiali dentro e fuori i contenitori per consentire la comunicazione con l'ambiente esterno e fornire capacità di rilevamento. Per superare alcuni di questi problemi, lo studio riporta un metodo che fornisce un approccio semplice ma efficace per incapsulare i materiali di codifica per diversi progetti di sensori da esprimere nei sistemi IVTT. Questo approccio si basa sull'utilizzo della deposizione Layer-by-Layer (LbL) di un biopolimero in presenza dei plasmidi di interesse per creare microcapsule cave ad alta porosità, che consente al materiale genetico protetto di interagire con i diversi componenti dell'IVTT di scelta. Lo studio ha dimostrato che i plasmidi incapsulati potrebbero dirigere la trascrizione e la traduzione quando attivati all'interno di questa matrice polimerica, come mostrato con la risposta di un aptamero codificato da plasmide e di un riboswitch ai loro bersagli corrispondenti. Inoltre, questo rivestimento LbL protegge i plasmidi per mesi senza particolari condizioni di conservazione.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Costruzione del vettore plasmide.
- Costruire un vettore plasmidico (pSALv-RS-GFPa1, 3.4 kb) mediante amplificazione della sequenza codificante di un riboswitch teofillina (ThyRS) accoppiato con GFPa1 dal vettore pJ201:23976-RS-GFPa1 (progettato e creato da DNA2.0) e inserimento nel vettore di espressione di E. coli , pSAL13. Utilizzare primer diretti (5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3') e inversi (5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') per amplificare la sequenza codificante di ThyRS accoppiato con GFPa1 ed eseguire una reazione PCR in volume di 50 μL utilizzando DNA polimerasi secondo il protocollo14 del produttore.
- Preparare un gel di agarosio all'1% da 0,5 g di agarosio, 50 ml di tampone TAE (40 mM Tris acetato, 1 mM EDTA, pH 8,0) e 3 μL di colorazione del DNA.
- Miscelare 5 μL di aliquota del prodotto amplificato con PCR con 5 μL di acqua priva di RNasi/DNasi e 2 μL di colorante caricante gel 6x e analizzare mediante elettroforesi su gel di agarosio. Caricare una scala del DNA (0,1-10,0 kb) come riferimento. Eseguire il gel a 120 V fino a quando la linea di colorante ha quasi raggiunto il fondo del gel.
- Visualizza i frammenti di DNA utilizzando un sistema di imaging transilluminatore UV per verificare la dimensione corretta del DNA15.
- Purificare il prodotto PCR utilizzando un kit di purificazione PCR secondo il protocollo del produttore16.
- Digerire il prodotto PCR e il vettore di espressione pSAL con enzimi di restrizione KpnI e BlpI in una reazione da 15 μL, contenente 10 μL di prodotto PCR o vettore plasmidico (concentrazione 20-50 ng/μL), 1,5 μL di tampone enzimatico 10x, 1 μL di ciascun enzima e 1,5 μL di acqua priva di RNasi/DNasi, a 37 °C per 2 ore.
- Aggiungere 3 μL di colorante caricante gel 6x alla miscela di reazione e separare i frammenti digeriti su un gel di agarosio all'1% come descritto nei passaggi 1.3-1.5.
- Purificare i frammenti di DNA utilizzando un kit di estrazione del gel secondo il protocollo del produttore16.
- Collegare il prodotto PCR digerito in un vettore plasmidico linearizzato digerito, pSAL, utilizzando T4 DNA ligasi e tampone ligasi integrato in una reazione da 10 μL contenente 3-20 fmol del vettore digerito, 9-60 fmol del prodotto PCR digerito, 2 μL di tampone ligasi, 1 μL (1 unità) di T4 DNA ligasi e acqua priva di DNasi/RNasi. Incubare la reazione di legatura a 25 °C per 3 ore.
NOTA: Assicurarsi che il contenuto totale di DNA nella miscela di reazione sia 0,01-0,1 μg. - Trasformare le cellule competenti di E. coli DH5α con 10 ng della miscela di reazione di legatura secondo il protocollo17 del produttore.
- Far crescere le cellule trasformate a 37 °C durante la notte su piastre di agar LB integrate con ampicillina (100 μg/mL).
- Prelevare 3-4 colonie batteriche dalla piastra e trasferire asetticamente ciascuna di esse in 5 ml di mezzi LB integrati con ampicillina (100 μg/mL). Coltivare le colture durante la notte a 37 °C in un'incubatrice a 225 giri/min.
- Pellettare le colture durante la notte mediante centrifugazione a 11 x g per 3 minuti a temperatura ambiente.
- Utilizzare un kit di purificazione per purificare i plasmidi secondo il protocollo del produttore16.
- Verificare le sequenze dei plasmidi purificati mediante sequenziamento del DNA. La mappa plasmidica e la sequenza del costrutto risultante (pSALv-RS-GFPa1) sono mostrate nella Figura 1.
2. Purificazione del DNA su larga scala.
- Trasformare il vettore plasmidico pSALv-RS-GFPa1 (3.4 kb) (che codifica per il riboswitch teofillina accoppiato con il gene reporter GFPa1) o pET28c-F30-2xBroccoli (5.4 kb) (codificante per l'aptamero di Broccoli) in cellule competenti per E. coli DH5α secondo il protocollo17 del produttore.
- Far crescere le cellule trasformate a 37 °C durante la notte su piastre di agar LB integrate con ampicillina (100 μg/mL) per cellule trasformate con pSALv-RS-GFPa1 o kanamicina (50 μg/mL) per cellule trasformate con broccoli pET28c-F30-2x.
- Prelevare 3-4 colonie batteriche dalla piastra e trasferire asetticamente ogni colonia in 5 ml di terreno LB integrato con antibiotico appropriato (100 μg/mL di ampicillina o 50 μg/mL di kanamicina). Coltivare le colture durante la notte a 37 °C in un'incubatrice a 225 giri/min.
- Utilizzare 3 ml di coltura overnight da inoculare in 150 mL di LB integrata con antibiotico appropriato (100 μg/mL di ampicillina o 50 μg/mL di kanamicina) e far crescere le colture per una notte a 37 °C in un incubatore agitatore a 225 giri/min.
- Pellettare le celle mediante centrifugazione a ≥3400 x g per 10 minuti a 4 °C.
- Utilizzare un kit di purificazione per purificare i plasmidi secondo il protocollo del produttore16.
- Eluire il DNA con 0,5 mL di acqua pura priva di DNasi/RNasi. Misurare la concentrazione di DNA e preparare 1 mL di soluzioni madre di DNA (100 ng/μL). Conservare le provette con DNA a 4 °C fino a ulteriore utilizzo.
3. Estrazione della fibroina di seta e preparazione dei materiali iniziali.
- Preparare una soluzione acquosa di proteina ricostituita di fibroina della seta (SF) dai bozzoli del baco da seta Bombyx mori secondo la procedura descritta in dettaglio altrove per rappresentare il 10% della soluzione di Silk-LiBr18.
- Determinare la concentrazione finale della soluzione acquosa di SF. Pipettare 0,5 mL di soluzione di seta su una capsula di Petri da 60 mm, lasciarla asciugare a 60 °C e misurare il peso della pellicola di seta asciutta. Dividere il peso secco per 0,5 ml per calcolare la percentuale di peso per volume.
- Diluire la soluzione concentrata di seta con acqua distillata priva di DNasi/RNasi aggiungendo lentamente acqua tramite pipetta sierologica per ottenere una concentrazione finale di 1 mg/ml. Conservare la soluzione a 4 °C per un uso futuro.
- Preparare fibroina di seta marcata con fluorescenza utilizzando un kit di etichettatura anticorpale. Utilizzare 1 mL di soluzione di fibroina di seta da 2 mg/ml per accoppiare i gruppi N-terminali α-amminico della proteina con un colorante derivato attivato con estere del NHS secondo il protocollo19 del produttore.
- Preparare 50 mL di soluzione acquosa di polietilenimmina (PEI) con una concentrazione di 6 mg/ml, regolare il pH a 4 con HCl (1 M). Filtrare la soluzione attraverso una membrana sterile da 0,2 μm. Lo stoccaggio è possibile in condizioni ambientali per mesi.
- Preparare i core SiO2 . Pipettare 300 μL di particelle di SiO 2 in una provetta da microcentrifuga da2 ml. Lavare le microparticelle due volte con 1 mL di acqua priva di DNasi/RNasi mediante centrifugazione a 0,2 x g per 1 minuto.
4. Eseguire la deposizione strato per strato di uno strato primo, plasmidi di DNA e strati di seta.
- Per depositare lo strato primario PEI sulle microparticelle di SiO2 , aggiungere 1 mL di soluzione PEI al pellet filato dal punto 3.6 e agitare la miscela in condizioni ambientali su un termomiscelatore a 800 giri / min per 15 minuti. Lavare le particelle quattro volte con 1 mL di acqua deionizzata priva di DNasi/RNasi mediante centrifugazione a 0,2 x g per 1 minuto.
- Per eseguire la deposizione dello strato di DNA, aggiungere 1 mL della soluzione acquosa di plasmidi di DNA dal punto 2.7 alle microparticelle innescate PEI e agitare delicatamente la miscela a 4 °C su un termomiscelatore a 800 giri / min per 15 minuti. Per preparare microcapsule con diversi carichi di DNA, regolare la concentrazione di plasmidi di DNA da 50-200 ng/μL utilizzando acqua distillata priva di DNasi/RNasi e utilizzare 1 mL di queste soluzioni per depositare il DNA. Raccogliere le microparticelle mediante centrifugazione a 0,2 x g per 1 minuto.
- Contrassegnare i tubi per plasmidi di DNA che codificano per il riboswitch teofillina accoppiato con GFPa1 come ThyRS-GFPa1 e i plasmidi del DNA che codificano per l'aptamero di Broccoli come BrocApt.
NOTA: Tenere le provette della microcentrifuga con il DNA sul ghiaccio. - Rimuovere con cautela il surnatante e lavare le microparticelle quattro volte con 1 mL di acqua distillata priva di DNasi/RNasi, scartando ogni volta il surnatante dopo centrifugazione a 0,2 x g per 1 minuto. Eseguire tutti gli esperimenti a temperatura ambiente (RT) se non diversamente specificato.
- Per eseguire la deposizione dello strato di fibroina di seta aggiungere 1 mL della soluzione acquosa ricostituita di SF dal punto 3.3 alle microparticelle adsorbite di DNA, vortice delicatamente e agitare la miscela a 10 °C sul termomiscelatore a 750 giri / min per 15 minuti. Raccogliere le microparticelle mediante centrifugazione a 0,2 x g per 1 minuto a 4 °C, rimuovere il surnatante e quindi lavarle una volta con 1 mL di acqua distillata priva di DNasi/RNasi. Ripetere la centrifugazione ed eliminare il surnatante.
NOTA: Durante l'esperimento, tenere la soluzione di seta sul ghiaccio per evitare gelificazioni indotte dalla temperatura. - Trattare gradualmente le particelle con metanolo per indurre la formazione di fogli di β nella struttura proteica della seta. In primo luogo, aggiungere 0,5 ml di acqua distillata DNasi / RNasi, vortice il tubo della microcentrifuga, quindi aggiungere 0,5 ml di metanolo al 100%. Agitare delicatamente le particelle sul termomiscelatore a 10 °C per 5 minuti. Raccogliere le particelle mediante centrifugazione a 0,2 x g per 1 min. Rimuovere il surnatante.
- Trattare le particelle con metanolo per favorire la formazione di fogli di β e garantire un forte assorbimento fisico dello strato di seta. Aggiungere 1 mL di metanolo al 100%. Agitare delicatamente le particelle sul termomiscelatore a 750 giri/min per 10 minuti a 10 °C.
- Raccogliere le particelle mediante centrifugazione a 0,2 x g per 1 minuto a 4 °C e lavarle due volte con 1 ml di acqua distillata priva di DNasi/RNasi ogni volta, scartando il surnatante e vorticando delicatamente prima della centrifugazione successiva.
- Ripetere i passaggi 4,5-4,8 20 volte per ottenere strutture di guscio multistrato di seta. Per l'ultima fase di deposizione, utilizzare seta etichettata con fluorescenza dal punto 3.4 (Silk-DyLight550, 1 mL).
- Eseguire l'ultima fase di lavaggio e mantenere le microparticelle in 1 mL di acqua distillata priva di DNasi/RNasi in condizioni ambientali.
NOTA: Per evitare l'aggregazione di particelle durante la deposizione di strati di seta, eseguire un'ispezione visiva della sospensione delle particelle e pipettarla su e giù utilizzando una punta della pipetta da 1 mL per promuovere una distribuzione omogenea delle particelle. - Calcolare il numero di copie del plasmide del DNA incapsulate in ogni microcapsula, NDNA utilizzando l'equazione 1:
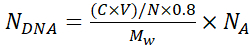 (1)
(1)
Dove N = 6,769 × 1011 - il numero di core SiO2 utilizzati per l'incapsulamento. Calcolarlo da una curva standard per le concentrazioni note di particelle di silice usando diluizioni seriali e assorbimento A 320 a λ =320 nm;
C- concentrazione iniziale di DNA utilizzato per l'adsorbimento
V- il volume di DNA utilizzato per l'adsorbimento
0.8- Efficienza di adsorbimento del DNA sui nuclei
Mw- Peso molecolare del plasmide del DNA
NA- Numero di Avogadro (6.022 × 1023)
5. Dissoluzione dei nuclei per ottenere microcapsule di seta.
- Preparare una soluzione di acido fluoridrico (HF) all'8%, pH 5,5, diluendo la soluzione madre (48%) con acqua distillata. Acquistare una provetta da centrifuga da 50 ml. Pipettare accuratamente 5 mL di HF e aggiungere 25 mL di acqua distillata per ottenere una soluzione HF all'8%.
ATTENZIONE: L'HF è un acido altamente corrosivo e può causare gravi ustioni ai tessuti. Estrema cautela deve essere presa durante la manipolazione e l'uso di HF per gli esperimenti. Aderire alla procedura operativa standard (SOP) per il corretto utilizzo e gestione dell'HF sviluppata dall'organizzazione per evitare incidenti indesiderati da fuoriuscita. Non utilizzare contenitori di vetro per diluire l'acido HF. Utilizzare la cappa chimica per eseguire questo passaggio del protocollo. - Sciogliere i nuclei di SiO2 aggiungendo 1,5 ml di soluzione HF all'8% alle microparticelle pellettate del guscio del nucleo dal punto 4.10. Vortice delicatamente e lasciare che i nuclei si dissolvano durante la notte in condizioni ambientali con un leggero scuotimento a 450 giri / min.
NOTA: Per evitare fuoriuscite di HF, utilizzare nastro adesivo per sigillare la provetta della microcentrifuga. Utilizzare una cappa chimica per eseguire questo passaggio del protocollo. - Preparare un bicchiere di vetro da 2 L riempito con 2 L di acqua deionizzata. Trasferire la soluzione di microcapsule ai dispositivi di dialisi (50 kDa MWCO) e dializzarle contro l'acqua deionizzata con un cambio ripetuto dell'acqua ogni 3 ore per i successivi 3 giorni.
NOTA: Raccogliere il surnatante durante i primi tre scambi di acqua e scartare la soluzione secondo il protocollo stabilito per i materiali di scarto pericolosi. - Utilizzare una pipetta da 1 mL per trasferire la sospensione dai dispositivi di dialisi in nuove provette da microcentrifuga da 2 mL per raccogliere le microcapsule.
NOTA: Conservare le soluzioni acquose delle microcapsule in condizioni ambientali per diversi anni.
6. Imaging di microcapsule di fibroina di seta mediante microscopio laser a scansione confocale (CLSM).
- Eseguire la colorazione del DNA utilizzando un colorante del DNA.
- Trasferire 300 μL di microcapsule di fibroina di seta cava in una provetta fresca da 1 mL di microcentrifuga. Aggiungere 500 μL di acqua distillata priva di RNasi/DNasi.
- Aggiungere 5 μL del colorante colorante del DNA, brevemente vortice, e incubare a RT per 2 ore al riparo dalla luce.
- Eseguire quattro fasi di lavaggio mediante centrifugazione a 0,1 x g per 20 minuti a 4 °C ogni volta, rimuovendo con cura 400 μL di surnatante e reintegrandolo con 400 μL di acqua distillata priva di RNasi/DNasi.
- Eseguire l'imaging di capsule di seta su sistemi confocali invertiti dotati di tre laser principali (405 nm, 488 nm, 561 nm) utilizzando l'obiettivo 100x oil-immersion (NA 1.49). Trasferire 100 μL del campione di capsula in un singolo pozzetto di vetrini a 8 pozzetti camerati, lasciare sedimentare le capsule per 20-30 minuti prima dell'imaging.
NOTA: I coloranti sono molto sensibili al fotosbiancamento. Proteggere i campioni coprendo i vetrini con un foglio di alluminio.
7. Stima della permeabilità di microcapsule cave mediante il metodo MWCO (molecular weight cut-off).
- Preparare 2 mL ciascuna di soluzioni di destrano fluoroforo marcate con FITC (20 μM, diH 2 O)di M w diverso (4 kDa, 20 kDa, 40 kDa, 70 kDa, 150 kDa, 250 kDa, 500 kDa e2MDa).
- Pipettare 100 μL della sospensione di capsule in un singolo pozzetto di un vetrino camerato. Analizzare separatamente ogni progetto di microcapsula (concentrazione di PEI, numero di carico di plasmidi di DNA, concentrazione di fibroina di seta e numero di strati).
- Ad ogni pozzetto, aggiungere 300 μL della soluzione specifica di fluoroforo partendodal M w più basso fino al più alto, in modo che ogni pozzetto corrisponda alla soluzione specifica di fluoroforo. Mescolare pipettando su e giù e lasciare incubare la miscela per 1 ora a RT fino a quando la diffusione delle soluzioni di fluorofori raggiunge l'equilibrio.
- Trasferire il vetrino su un microscopio a scansione laser confocale (CLSM) e visualizzare ogni pozzetto utilizzando un obiettivo ad immersione in olio 100x all'eccitazione λ = 488 nm.
- Identificare l'area di interesse regolando il piano focale per assicurarsi che le capsule appaiano sotto forma di cerchi del diametro maggiore. Ciò accade in genere quando si osservano i campioni più vicini al fondo del pozzo quando le capsule sedimentano a causa della gravità.
- Raccogliere diverse immagini di campioni di microcapsule spostando la diapositiva in direzione XY. Cattura immagini per rappresentare fino a 100-150 capsule per ogni campione.
- Utilizzare il software ImageJ per analizzare la permeabilità della membrana della capsula in ciascuna soluzione di fluoroforo Mw confrontando le intensità di fluorescenza all'interno e all'esterno delle capsule. Per questo, disegna una regione di interesse (ROI) sotto forma di un cerchio per delineare la circonferenza della capsula e fai clic su Analizza / Misura per misurare l'intensità della fluorescenza all'interno. Tabulare i dati in un foglio di calcolo. Eseguire questa operazione per ogni microcapsula per un totale di 200-300 capsule.
- Valutare l'intensità della fluorescenza esterna allo stesso modo delineando il ROI e misurando l'intensità lontano dalle capsule. Eseguire 3-5 misurazioni per l'analisi statistica.
- Per eseguire analisi statistiche, confrontare le intensità di fluorescenza all'interno e all'esterno delle capsule utilizzando il t-test accoppiato (p < 0,05).
- Utilizzare la tabella di conversione 2 per stimare la permeabilità delle microcapsule in base ai raggi idrodinamici per FITC-Dextran con M w variabile.
8. Attivazione in vitro di teofillina sintetica riboswitch in microcapsule di seta
- Preparare 1 mL di soluzione madre di teofillina (100 mM, DMSO). Preparare il sistema di estrazione di E. coli S30 per il DNA circolare scongelando i componenti sul ghiaccio per 40 minuti.
- Ottenere una provetta da microcentrifuga priva di DNasi/RNasi da 0,5 ml. Eseguire una reazione di trascrizione/traduzione in vitro , combinare i componenti cell-free con un campione di microcapsule nel seguente ordine (volume totale 50 μL): S30 premiscela senza amminoacidi (20 μL); Estratto di S30, circolare (15 μL); miscela completa di aminoacidi (5 μL); microcapsule cave contenenti plasmidi ThyRS-GFPa1 dal punto 4.10 (9 μL); e teofillina, 100 mM DMSO (1 μL).
NOTA: Dopo aver aggiunto tutti i componenti, ruotare brevemente la provetta e raccogliere il campione durante una breve centrifugazione a 0,2 × g per un paio di secondi. - Incubare il tubo a 30 °C per 4 ore e controllare la fluorescenza su un lettore di piastre utilizzando eccitazione a λ = 488 nm ed emissione per filtro GFP/FITC (510 nm ± 20 nm).
- Immagini delle capsule su qualsiasi sistema LCSM utilizzando laser a 488 nm e 561 nm. Ottieni immagini della migliore qualità utilizzando l'obiettivo ad immersione in olio 100x e 8 diapositive ben camerate.
9. Attivazione in vitro dell'aptamero dei broccoli in microcapsule di seta
- Preparare 1 mL di soluzione madre di colorante DFHBI-1T (30 μM, diH2O). Preparare il kit di reazione del sistema privo di cellule PURE (sintesi proteica con elementi ricombinanti) scongelando i componenti sul ghiaccio per 40 minuti.
- Ottenere una provetta da microcentrifuga priva di DNasi/RNasi da 0,5 ml. Eseguire la reazione di trascrizione in vitro combinando componenti di reazione cell-free con un campione di microcapsule nel seguente ordine (volume totale 50 μL): soluzione A (20 μL); soluzione B (15 μL); microcapsule cave contenenti plasmidi BrocApt dal punto 4.10 (14 μL); e colorante DFHBI-1T (1 μL).
NOTA: Dopo aver aggiunto tutti i componenti, ruotare brevemente la provetta e raccogliere il campione durante una breve centrifugazione a 0,2 × g per un paio di secondi. - Incubare il tubo a 37 °C per 6 ore e controllare la fluorescenza su un lettore di piastre usando eccitazione a λex = 470 nm ed emissione a λem = 510 nm ± 20 nm.
- Immagini delle capsule su qualsiasi sistema LCSM utilizzando laser a 488 nm e 561 nm. Ottieni immagini della migliore qualità utilizzando l'obiettivo ad immersione in olio 100x e le diapositive camerate in vetro con copertura a 8 pozzetti.
Access restricted. Please log in or start a trial to view this content.
Risultati
Qui, lo studio affronta la funzionalità dei modelli di DNA che codificano diversi design di sensori (due tipi di elementi di trascrizione/traduzione regolati dall'RNA) dopo l'incapsulamento in capsule proteiche della seta. Le microcapsule sono state preparate tramite un assemblaggio Layer-by-Layer (LbL) modellato dei componenti chiave: uno strato primo, plasmidi di DNA che codificano i progetti dei sensori e biopolimero di fibroina di seta (Figura 2). La deposizione di macromolecole in modo...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le microcapsule di idrogel selettivamente permeabili caricate con vari tipi di sensori codificati dal DNA possono essere preparate seguendo questo protocollo. Una delle caratteristiche distintive dell'approccio LbL è la capacità di adattare la complessità delle microcapsule durante l'assemblaggio bottom-up, che di solito inizia con l'adsorbimento di specie molecolari su modelli sacrificali. Regolando attentamente le concentrazioni dei componenti iniziali, le condizioni di pH e il numero di strati, è possibile prepara...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
I punti di vista e le opinioni qui presentati sono quelli degli autori e non rappresentano necessariamente i punti di vista del DoD o dei suoi componenti.
Riconoscimenti
Questo lavoro è stato supportato dalla sovvenzione LRIR 16RH3003J dell'Air Force Office of Scientific Research, nonché dal programma Synthetic Biology for Military Environments Applied Research for the Advancement of S&T Priorities (ARAP) dell'Ufficio del Sottosegretario alla Difesa degli Stati Uniti per la Ricerca e l'Ingegneria.
La sequenza del vettore plasmidico per ThyRS (pSALv-RS-GFPa1, 3.4 kb) è stata generosamente fornita dal Dr. J. Gallivan. I bozzoli del baco da seta di Bombyx mori sono stati generosamente donati dal Dr. D.L. Kaplan della Tufts University, MA.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
Riferimenti
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940(2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027(2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469(2014).
- Frey, B. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. Reischl, U. 13, Humana Press. Totowa, NJ. 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923(2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870(2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466(2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504(2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046(2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707(2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283(2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon