È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Combinazione di organoidi umani e tecnologia Organ-on-a-Chip per modellare la funzionalità specifica della regione intestinale
In questo articolo
Riepilogo
Gli organoidi intestinali derivati dalla biopsia e le tecnologie organ-on-a-chips sono combinati in una piattaforma microfisiologica per ricapitolare la funzionalità intestinale specifica della regione.
Abstract
La mucosa intestinale è una complessa barriera fisica e biochimica che svolge una miriade di funzioni importanti. Consente il trasporto, l'assorbimento e il metabolismo di nutrienti e xenobiotici, facilitando al contempo una relazione simbiotica con il microbiota e limitando l'invasione di microrganismi. L'interazione funzionale tra vari tipi di cellule e il loro ambiente fisico e biochimico è vitale per stabilire e mantenere l'omeostasi del tessuto intestinale. Modellare queste complesse interazioni e la fisiologia intestinale integrata in vitro è un obiettivo formidabile con il potenziale per trasformare il modo in cui nuovi bersagli terapeutici e farmaci candidati vengono scoperti e sviluppati.
Organoidi e tecnologie Organ-on-a-Chip sono stati recentemente combinati per generare chip intestinali rilevanti per l'uomo adatti allo studio degli aspetti funzionali della fisiologia intestinale e della fisiopatologia in vitro. Gli organoidi derivati dalle biopsie dell'intestino tenue (duodeno) e crasso vengono seminati nel compartimento superiore di un chip d'organo e quindi si espandono con successo come monostrati preservando le distinte caratteristiche cellulari, molecolari e funzionali di ciascuna regione intestinale. Le cellule endoteliali microvascolari specifiche del tessuto dell'intestino umano sono incorporate nel compartimento inferiore del chip dell'organo per ricreare l'interfaccia epitelio-endoteliale. Questa nuova piattaforma facilita l'esposizione luminale a sostanze nutritive, farmaci e microrganismi, consentendo studi sul trasporto intestinale, la permeabilità e le interazioni ospite-microbo.
Qui, viene fornito un protocollo dettagliato per la creazione di chip intestinali che rappresentano il duodeno umano (chip di duodeno) e il colon (chip del colon) e la loro successiva coltura sotto flusso continuo e deformazioni simili alla peristalsi. Dimostriamo metodi per valutare il metabolismo dei farmaci e l'induzione del CYP3A4 nel chip di duodeno utilizzando induttori e substrati prototipici. Infine, forniamo una procedura passo-passo per la modellazione in vitro dell'interruzione della barriera mediata dall'interferone gamma (IFNγ) (sindrome dell'intestino permeabile) in un chip del colon, compresi i metodi per valutare l'alterazione della permeabilità paracellulare, i cambiamenti nella secrezione di citochine e la profilazione trascrittomica delle cellule all'interno del chip.
Introduzione
L'intestino umano è un organo complesso e multitasking capace di auto-rigenerazione. È diviso nell'intestino tenue e crasso. La funzione primaria dell'intestino tenue è quella di digerire ulteriormente il cibo proveniente dallo stomaco, assorbire tutti i nutrienti e trasmettere il residuo all'intestino crasso, che recupera l'acqua e gli elettroliti. L'intestino tenue è ulteriormente diviso in più regioni anatomicamente distinte: il duodeno, il digiuno e l'ileo, ognuno dei quali è adattato per svolgere funzioni specifiche. Ad esempio, il duodeno aiuta a scomporre il chimo (contenuto dello stomaco) per consentire il corretto assorbimento dei nutrienti che coinvolgono proteine, carboidrati, vitamine e minerali nel digiuno. Questa parte prossimale dell'intestino tenue è anche il principale sito di assorbimento e metabolismo intestinale dei farmaci ed è caratterizzata dalla maggiore espressione di enzimi che metabolizzano i farmaci (ad esempio, CYP3A4) rispetto alla loro espressione nell'ileo e nel colon1. Oltre al suo ruolo principale nella digestione e nell'assorbimento dei nutrienti, l'intestino è anche una barriera efficace contro i contenuti luminali potenzialmente dannosi, come microrganismi patogeni, metaboliti microbici, antigeni alimentari e tossine 2,3. È interessante notare che il colon umano è abitato da un gran numero di microrganismi, di gran lunga superiore a quello delle cellule totali nel corpo umano, che forniscono molti benefici alla nutrizione, al metabolismo e all'immunità. Pertanto, il mantenimento dell'integrità della barriera mucosale formata dalle cellule epiteliali intestinali è fondamentale per consentire la relazione simbiotica tra il microbiota intestinale e le cellule ospiti separandole fisicamente per evitare un'inutile attivazione delle cellule immunitarie2. Inoltre, la morte cellulare intestinale programmata svolge un ruolo essenziale come meccanismo di autoprotezione che impedisce alle cellule infette di persistere o proliferare, diffondendo così potenziali agenti patogeni3, mentre il continuo auto-rinnovamento dell'epitelio intestinale ogni quattro-sette giorni compensa la perdita cellulare garantendo l'integrità della barriera e l'omeostasi dei tessuti. La compromissione delle funzioni intestinali descritte, tra cui l'assorbimento dei nutrienti, l'integrità della barriera o lo squilibrio nella morte delle cellule intestinali e nell'auto-rinnovamento, può provocare lo sviluppo di una serie di disturbi gastrointestinali, tra cui la malnutrizione e la malattia infiammatoria intestinale (IBD)2,3.
In precedenza, modelli animali e linee cellulari intestinali derivate dal cancro trasformate sono state utilizzate per studiare le funzioni fisiologiche e fisiopatologiche del tessuto intestinale umano. Tuttavia, le preoccupazioni sempre più importanti sulla traducibilità della ricerca sugli animali per l'uomo, causate dalla presenza di disparità significative tra le due specie, hanno evidenziato la necessità di metodi alternativi rilevanti per l'uomo4. Le linee cellulari intestinali comunemente usate in vitro includono cellule T84, Caco-2 e HT29. Mentre imitano alcuni aspetti della funzione di barriera intestinale e del trasporto di membrana, sono caratterizzati da un'espressione alterata degli enzimi che metabolizzano i farmaci5, i recettori di superficie e i trasportatori4. Inoltre, mancano di specificità del segmento intestinale e non riescono a ricapitolare la complessità dell'epitelio intestinale, con ogni modello contenente solo uno dei cinque tipi di cellule epiteliali presenti nell'intestino6.
Recentemente, colture organoidi intestinali umane stabilite da biopsie fresche dell'intestino tenue e del colon 7,8 o cellule staminali pluripotenti indotte (iPSC)9 sono state introdotte come modelli sperimentali alternativi con potenziale per integrare, ridurre e forse sostituire la sperimentazione animale in futuro. Mentre le iPSC possono essere ottenute in modo non invasivo, la creazione di organoidi da iPSC richiede l'uso di protocolli complessi e lunghi (con diverse fasi sperimentali) e genera colture simili al tessuto fetale umano. Al contrario, gli organoidi derivati dalla biopsia sono altamente scalabili, in quanto possono sfruttare la capacità di rinnovamento intrinseca del tessuto intestinale e possono essere trasmessi e propagati in vitro a tempo indeterminato. È importante sottolineare che gli organoidi derivati dalla biopsia mantengono le caratteristiche specifiche della malattia e della regione intestinale del tessuto primario da cui sono stati sviluppati ed emulano la diversità cellulare dell'epitelio intestinale. Gli organoidi possono essere utilizzati come avatar specifici del paziente in vitro per svelare la biologia e la patogenesi di vari disturbi gastrointestinali e migliorare la loro gestione terapeutica. Sebbene gli organoidi intestinali abbiano raggiunto un impressionante grado di funzionalità fisiologica, non riescono ancora a riprodurre la complessità degli organi nativi a causa della loro mancanza di componenti stromali critici, tra cui vasi sanguigni, tessuto connettivo, nervi periferici e cellule immunitarie, nonché stimolazione meccanica. I parametri meccanici, come il flusso, lo stress da taglio, l'allungamento e la pressione, sono noti per influenzare la morfogenesi tissutale e l'omeostasi in vivo e in precedenza hanno dimostrato di migliorare la maturazione delle cellule in vitro 10,11,12,13. Un ulteriore importante inconveniente dei sistemi organoidi è l'inaccessibilità del lume e, quindi, al lato apicale dell'epitelio. Ciò rappresenta una sfida per lo studio di vari meccanismi associati all'espressione polarizzata di trasportatori di ioni e farmaci, interazioni ospite-microbioma e test di tossicità farmaceutica. Infine, le colture organoidi soffrono di una notevole variabilità in termini di dimensioni, morfologia e funzione, a causa della natura stocastica del processo di auto-organizzazione in vitro e delle scelte del destino cellulare. Pertanto, per realizzare il pieno potenziale degli organoidi intestinali nella modellazione delle malattie, nello screening farmacologico e nella medicina rigenerativa, è necessario esplorare nuove strategie che riducano la variabilità nello sviluppo degli organoidi, migliorino l'accesso al compartimento luminale e incorporino le interazioni cellula-cellula mancanti.
La tecnologia Organ-on-a-Chip ha introdotto molte tecniche per l'incorporazione di forze meccaniche e flusso di fluidi in colture cellulari intestinali in vitro. Tuttavia, poiché la maggior parte degli studi proof-of-concept iniziali hanno utilizzato linee cellulari derivate dal cancro che non mostravano sufficiente diversità cellulare, la rilevanza di questi sistemi è stata messa in discussione. Recentemente, abbiamo combinato sinergicamente organoidi intestinali e tecnologia organ-on-a-chip per incorporare le migliori caratteristiche di ciascun approccio in un unico sistema in vitro 14,15,16. Il chip intestinale risultante ricapitola l'architettura multicellulare dell'epitelio intestinale, la presenza dell'interfaccia del tessuto epiteliale-endoteliale e le forze meccaniche del flusso e dell'allungamento del fluido, consentendo l'emulazione delle funzioni a livello di organo in vitro. Inoltre, l'uso di organoidi derivati da tessuti primari (che possono essere campionati da diverse regioni dell'intestino umano) come materiale di partenza aumenta la versatilità di questo modello, poiché i chip che rappresentano duodeno umano, digiuno, ileo e colon possono essere stabiliti seguendo procedure di semina e coltura simili. È importante sottolineare che Intestine-Chips consente la valutazione in tempo reale di: l'integrità della barriera intestinale; attività del bordo del pennello e degli enzimi del metabolismo dei farmaci; produzione di mucine; secrezione di citochine; e l'interazione delle cellule intestinali con microrganismi patogeni e commensali, come dimostrato nei rapporti precedentemente pubblicati. In particolare, quando i chip intestinali sono stati stabiliti utilizzando organoidi generati dal tessuto di individui diversi, questi modelli hanno catturato la variabilità inter-donatore attesa nelle risposte funzionali a vari farmaci e trattamenti. Complessivamente, la fusione di organoidi con la tecnologia Organ-on-a-Chip apre la porta a modelli più avanzati, personalizzati e rilevanti in vivo che potrebbero migliorare la rilevanza fisiologica e l'accuratezza dei risultati in vitro, nonché la loro estrapolazione per gli esseri umani. Qui, viene presentato un protocollo dettagliato per stabilire il chip intestinale e la sua applicazione negli studi delle funzioni fisiologiche dei due segmenti intestinali: duodeno e colon. In primo luogo, vengono descritti i metodi per valutare l'attività dell'enzima CYP3A4 che metabolizza i farmaci nel chip del duodeno, nonché la sua induzione da parte di composti prototipici come la rifampicina e la vitamina D3. In secondo luogo, i passaggi necessari per modellare "intestino permeabile" nel chip del colon sono delineati nel protocollo, con l'interruzione della barriera epiteliale eseguita utilizzando citochine caratteristiche implicate nella patogenesi dell'IBD. In breve, gli organoidi derivati da biopsie umane vengono propagati in vitro, sottoposti a digestione enzimatica e introdotti nel canale superiore del chip. In presenza di perfusione continua con mezzi arricchiti da fattori di crescita, si sviluppano in un monostrato epiteliale confluente con architettura 3D e una superficie cellulare apicale facilmente accessibile. Il compartimento inferiore del chip "vascolare" è seminato con cellule endoteliali microvascolari isolate dall'intestino tenue o crasso. L'epitelio e l'endotelio sono separati da una membrana porosa estensibile, che facilita le interazioni paracrine tra i due tessuti e, se sottoposti a deformazioni cicliche, emula i movimenti simili alla peristalsi dell'intestino umano. La co-coltura viene mantenuta nelle condizioni di flusso dinamico generate dalla perfusione luminale e vascolare con mezzi di coltura cellulare appropriati. Infine, descriviamo numerosi tipi di saggi e analisi degli endpoint che possono essere eseguiti direttamente su chip o da effluenti di colture cellulari campionati.
Protocollo
NOTA: Tutte le colture cellulari devono essere gestite utilizzando una tecnica asettica adeguata.
Gli organoidi intestinali umani impiegati in questo studio sono stati ottenuti dalla Johns Hopkins University e tutti i metodi sono stati eseguiti in conformità con le linee guida e i regolamenti approvati. Tutti i protocolli sperimentali sono stati approvati dal Johns Hopkins University Institutional Review Board (IRB #NA 00038329).
1. Preparazione dei reagenti di coltura cellulare
- Preparare il mezzo di crescita organoide umano seguendo le istruzioni del produttore (Tabella dei materiali) e integrarlo con 100 μg/mL di primocina.
- Preparare il mezzo di crescita delle cellule endoteliali microvascolari umane seguendo le istruzioni del produttore (Tabella dei materiali) e integrare con 1:20 vol/vol FBS e 50 μg/ml invece di 100 μg/ml di primocina.
- Risospesso Y-27632 (inibitore della rho-chinasi) in BSA sterile allo 0,1% in DPBS per preparare una soluzione madre da 10 mM. Aliquotare in microtubi sterili e conservare a -20 °C per un massimo di 6 mesi.
- Sospendere CHIR99021 (inibitore GSK-3) in dimetilsolfossido sterile (DMSO) per preparare una soluzione madre da 5 mM. Aliquotare in microtubi sterili e conservare a -20 °C per un massimo di 6 mesi.
- Preparare la soluzione di digestione organoide umana mescolando una soluzione di dissociazione di organoidi 1:1 vol/vol (Table of Materials) con soluzione DPBS (Table of Materials) e integrandola con una soluzione madre da 10 μM di Y-27632. Questo reagente deve essere reso fresco per ogni uso.
2. Coltura di cellule endoteliali microvascolari intestinali umane (HIMECs)
- Iniziare la coltura HIMEC (Table of Materials) 7 giorni prima di seminarla sul chip. Solo gli HIMEC al passaggio 1-6 possono essere utilizzati per la semina di trucioli.
- Aggiungere 5 mL della soluzione del fattore di attacco (Tabella dei materiali) a un matraccio T150; incubare a 37 °C per 1 minuto ed eliminare la soluzione.
- Aggiungere al matraccio 19 mL di terreno di crescita cellulare endoteliale preriscaldato a temperatura ambiente (cfr. punto 1.2).
- Scongelare un flaconcino congelato di HIMEC (2 milioni di cellule/flaconcino) a bagnomaria a 37°C.
- Trasferire il contenuto del flaconcino nel matraccio e scuotere delicatamente il matraccio per distribuire uniformemente le cellule. Posizionare il matraccio in un incubatore a 37 °C durante la notte per consentire alle cellule endoteliali di aderire.
- Sostituire il mezzo con 20 mL di terreno di crescita cellulare endoteliale preriscaldato a temperatura ambiente il giorno successivo e successivamente ogni 3 giorni. Le colture dovrebbero raggiungere il 90% della confluenza il giorno della raccolta delle cellule per gli esperimenti sui chip.
3. Microfabbricazione e preparazione del chip
- Ottenere chip da un fornitore commerciale (Tabella dei materiali). Disimballare i chip e posizionarli nella base del chip collocata in una capsula di Petri quadrata (Table of Materials). Etichettare il lato anteriore di ciascun supporto chip con le condizioni sperimentali (Figura 1A).
NOTA: Mentre il protocollo presentato si basa sull'uso di uno specifico chip e strumentazione disponibile in commercio, ci sono diversi dispositivi microfluidici offerti attraverso diversi fornitori, che possono offrire vantaggi alternativi ma non hanno la capacità di incorporare stretch. Inoltre, la microfabbricazione degli Organs-on-Chips può essere eseguita "in casa" seguendo le procedure descritte in Huh et al.21.
4. Attivazione e rivestimento ECM della membrana
- Preparazione della soluzione di attivazione
- Portare i reagenti ER-1 ed ER-2 (Tabella dei materiali) a temperatura ambiente per equilibrare prima dell'uso. ER-1 è sensibile alla luce e deve essere maneggiato al buio.
- Ricostituire ER-1 in 10 mL di reagente ER-2 per ottenere una concentrazione finale di 0,5 mg/ml e confermare che l'ER-1 è completamente disciolto.
- Attivazione di Surface
- Spingere delicatamente 50 μL della soluzione ER-1 attraverso entrambi i canali del chip (Figura 1A).
- Rimuovere qualsiasi soluzione ER-1 in eccesso dalla superficie del chip utilizzando un aspiratore.
- Ispezionare i chip per assicurarsi che tutti i chip abbiano ricevuto la soluzione ER-1. In caso di bolle d'aria, introdurre più soluzione ER-1 fino a quando le bolle non sono completamente rimosse.
- Incubare i trucioli all'interno della camera della lampada UV (Table of Materials) per 15 min. Verificare che la spia UV sia impostata sull'impostazione Coerente.
- Riportare i chip attivati ER-1 nell'armadio di biosicurezza (BSC). Aspirare la soluzione ER-1 da entrambi i canali. Lavare qualsiasi residuo della soluzione ER-1 con 200 μL di ER-2.
- Spingere 200 μL di soluzione salina sterile tamponata con fosfato (DBPS) di Dulbecco attraverso i canali (Tabella dei materiali) e lasciare DBPS nei canali fino al passaggio successivo.
- Preparazione della matrice extracellulare (ECM) e del rivestimento della membrana
- Preparazione di soluzioni stock di componenti ECM
- Ricostituire il collagene IV (Table of Materials) e la fibronectina (Table of Materials) utilizzando acqua sterile di coltura cellulare (Table of Materials) ad una concentrazione finale di 1 mg/mL. Preparare le aliquote e conservarle a -20°C fino all'uso.
- Preparazione della soluzione di lavoro ECM per il rivestimento delle membrane
- Preparare 1,5 ml di ogni soluzione ECM per il canale superiore e inferiore per ogni 12 chip da rivestire. La soluzione ECM deve essere sempre preparata appena prima dell'uso.
- Per il canale superiore, mescolare collagene IV e matrice di membrana basale solubilizzata (BMM) (Table of Materials) in DBPS freddo sterile a 200 μg / mL e 100 μg / mL, rispettivamente. Per il canale inferiore, mescolare collagene IV e fibronectina in DPBS freddo sterile a 200 μg/mL e 30 μg/mL, rispettivamente.
- Rivestimento ECM dei trucioli
- Aspirare il DBPS da entrambi i canali dei chip e sostituirlo con la soluzione di lavoro ECM appropriata per ciascun canale (Figura 1A).
- Ispezionare ogni chip per assicurarsi che abbia ricevuto la soluzione di rivestimento. In caso di bolle d'aria, spingere attraverso più soluzione di rivestimento fino a quando tutte le bolle sono completamente rimosse.
- Aggiungere DPBS sterile alla base del chip e posizionare la capsula di Petri contenente i trucioli in un incubatore a 37 °C. Incubare durante la notte per consentire alle proteine ECM di formare legami ionici con la membrana PDMS attivata. Se lo si desidera, le cellule possono essere seminate in qualsiasi momento tra 2 ore e 1 giorno dopo il rivestimento dei trucioli. In alternativa, i trucioli rivestiti possono essere conservati a 4 °C durante la notte, seguiti dall'incubazione notturna a 37 °C e dalla semina dei trucioli.
- Preparazione di soluzioni stock di componenti ECM
5. Semina di cellule endoteliali microvascolari intestinali (HIMEC) nel canale inferiore del chip
NOTA: gli HIMEC dell'intestino tenue e del colon vengono seminati rispettivamente nel canale inferiore del duodeno e del chip del colon.
- Preparare i chip
- Trasferire i chip rivestiti in ECM al BSC. Aspirare delicatamente il rivestimento ECM da entrambi i canali dei trucioli, quindi lavare entrambi i canali con 200 μL di terreno di crescita organoide e mezzo di crescita cellulare endoteliale, rispettivamente.
- Conservare i trucioli lavati in un incubatore a 37°C fino a procedere con la semina delle cellule endoteliali.
- Raccogli le cellule endoteliali
- Portare il pallone di coltura HIMEC al BSC e lavare con DPBS sterile.
- Aggiungere 3 mL di soluzione di dissociazione al matraccio e metterlo nell'incubatore a 37°C per 2 minuti per consentire il completo distacco cellulare.
- Raccogliere le cellule in un tubo conico da 15 ml e aggiungere mezzi di crescita cellulare endoteliale fino a raggiungere i 10 ml. Campione 15-20 μL della sospensione cellulare per il conteggio cellulare. Centrifuga a 150 x g per 5 min.
- Aspirare accuratamente il surnatante e ri-sospendere gli HIMEC ad una densità di 8-10 x 106 cellule/mL nei mezzi di crescita cellulare endoteliali.
- Semi cellule endoteliali nel canale inferiore del chip
- Portare la capsula di Petri contenente i chip al BSC e rimuovere delicatamente tutti i supporti dal canale inferiore utilizzando una pipetta da 1.000 μL.
- Introdurre 10-15 μL della sospensione HIMEC nel canale inferiore del chip. Ispezionare il chip subito dopo la semina per garantire una densità di semina ottimale (80%-90% della copertura) e una distribuzione cellulare omogenea all'interno del canale (Figura 1D). Se la densità di semina è superiore o inferiore al previsto o irregolare, lavare il canale 2x con 200 μL di mezzo di crescita cellulare endoteliale e ripetere la procedura di semina.
- Invertire la capsula di Petri immediatamente dopo aver seminato ogni lotto di sei trucioli per consentire l'attaccamento della cellula endoteliale al lato inferiore della membrana PDMS. Posizionare le piastre di Petri nell'incubatore a 37°C per 30 minuti a 1 ora, o fino a quando gli HIMEC nel canale inferiore non si sono attaccati alla membrana (Figura 1D).
- Lavare le patatine
- Spingere delicatamente 200 μL di mezzo di crescita cellulare endoteliale attraverso l'ingresso del canale inferiore per rimuovere eventuali cellule non attaccate e reintegrare i nutrienti nel mezzo.
- Lavare via le cellule che non si sono attaccate nel canale superiore con 200 μL di terreno di crescita organoide integrato con 5 μM CHIR99021 e 10 μM Y-27632.
- Posizionare i trucioli nell'incubatrice a 37°C fino a procedere alla semina delle cellule epiteliali.
6. Semina di frammenti organoidi nel canale superiore del chip
NOTA: Gli organoidi isolati da biopsie di varie regioni intestinali possono essere coltivati nel chip7 dell'intestino. Seguire le procedure descritte in Fujii et al. per l'isolamento delle cripte intestinali umane e la creazione di colture organoidi22. Qui, gli organoidi duodenali e del colon vengono utilizzati per generare rispettivamente i chip del duodeno e del colon. Data l'elevata variabilità da lotto a lotto e da donatore a donatore nella formazione e nella crescita degli organoidi, si suggerisce di eseguire una valutazione pilota della densità cellulare nella coltura in sospensione organoide (formato a piastra a 24 pozzetti) per ottenere la densità di semina ottimale di 8 milioni di cellule / ml.
- Valutazione pilota del numero di cellule/pozzetto di coltura organoidi statica.
NOTA: La seguente procedura consiste esclusivamente nel determinare il numero di pozzetti di coltura di sospensione organoide necessari per la semina del truciolo. Le singole celle risultanti non devono essere utilizzate per la semina del chip.- Aspirare accuratamente il surnatante da tre pozzetti della piastra di coltura organoide. Aggiungere 500 μL di soluzione di dissociazione BMM ghiacciata (Table of Materials) a ciascun pozzetto e utilizzare un raschietto di plastica per staccare il BMM solubilizzato dalla plastica.
- Raccogliere la sospensione organoide in un tubo conico sterile a bassa legante le proteine utilizzando una pipetta ghiacciata da 1.000 μL. Incubare su ghiaccio per 60 minuti, mescolando ogni 15 minuti agitando delicatamente il tubo. Centrifugare a 300 x g per 5 min a 4°C.
- Aspirare il surnatante e aggiungere 2 ml di tripsina. Incubare a 37°C per 30 minuti per digerire gli organoidi a livello unicellulare e aggiungere 10 ml di terreno di crescita organoide completo per fermare la reazione enzimatica. Centrifugare a 300 x g per 5 min a 4°C.
- Risospesso il pellet cellulare in 1 mL di terreno di crescita organoide completo e contare il numero totale di cellule utilizzando un ematocitometro secondo metodi standard.
- Preparazione di frammenti organoidi e loro semina nel chip
NOTA: le seguenti procedure possono essere utilizzate per seminare organoidi duodenali o del colon nel canale superiore del chip. La creazione di un monostrato epiteliale con una citoarchitettura 3D rilevante in vivo è subordinata alla presenza del flusso15,23 e al successo della semina di frammenti organoidi.- Aspirare accuratamente il mezzo dal numero di pozzetti della coltura organoide statica, che è sufficiente, come determinato nella fase 6.1, per raggiungere una densità di semina finale di 8 milioni di cellule/ml. Aggiungere 500 μL di soluzione di dissociazione BMM ghiacciata a ciascun pozzetto.
- Staccare il BMM solubilizzato dalla superficie dei pozzetti utilizzando un raschietto di plastica o una pipetta da 1.000 μL. Raccogliere la sospensione in un tubo conico a bassa legante le proteine da 15 ml.
- Conservare la sospensione su ghiaccio per 60 minuti, mescolando ogni 15 minuti agitando delicatamente il tubo. Procedere alla centrifugazione a 300 x g per 5 min a 4°C. Un pellet organoide ben definito dovrebbe essere visibile dopo la centrifugazione. Se si osserva uno strato di gel trasparente sopra il pellet cellulare (residui di BMM solubilizzato) (Figura 1B), procedere con i seguenti passaggi.
- Mescolare il surnatante e il pellet cellulare e aggiungere un volume uguale di soluzione di dissociazione BMM nel tubo. Mescolare delicatamente e conservare sul ghiaccio per altri 10 minuti e procedere alla centrifugazione a 300 x g per 5 minuti a 4°C.
- Se necessario, ripetere il passaggio 6.2.3.1 fino a quando non sono presenti residui di BMM solubilizzati.
- Scartare il surnatante e risospesciare il pellet organoide con una soluzione di digestione organoide (vedere passaggio 1.5). Utilizzare un volume sufficiente di soluzione di digestione organoide per garantire la completa immersione del pellet. 2 mL di soluzione sono appropriati per circa 2.400-12.000 organoidi di medie dimensioni (Figura 1D, Post-Semina). Incubare a 37°C per 1-3 min.
- Aggiungere Advanced DMEM/F-12 al tubo per fermare la reazione enzimatica. Utilizzare quattro volte il volume della soluzione di digestione utilizzata. Centrifugare a 300 x g per 5 min a 4°C.
- Aspirare il surnatante e risospendere il pellet di frammenti organoidi in un mezzo di crescita organoide completo integrato con 5 μM CHIR99021 e 10 μM Y-27632 per ottenere 8 milioni di cellule / ml. Preparare aliquote da 360 μL della sospensione in tubi sterili a bassa legante proteica da 1,5 mL per evitare la riduzione della densità cellulare a causa dell'attaccamento alle pareti.
- Rimuovere il mezzo dal canale superiore dei trucioli rivestiti. Aggiungere 30 μL di sospensione cellulare nel canale superiore di ciascun chip. Incubare i trucioli durante la notte in un incubatore a 37°C per consentire ai frammenti organoidi di aderire alla membrana rivestita in ECM (Figura 1D).
7. Coltura dinamica del chip intestinale - iniziazione e mantenimento del flusso e dei movimenti simili alla peristalsi
- Preparazione e degasaggio dei supporti
- Per mantenere costante il flusso laminare attraverso il chip, consentire alla temperatura del fluido di equilibrarsi a temperatura ambiente e quindi sottoporla a filtrazione sottovuoto per 10 minuti utilizzando una pompa per vuoto e tubi conici con filtro PVDF (degasaggio dei fluidi).
- Priming di moduli portatili
NOTA: i moduli portatili sono serbatoi che fungono da interfaccia tra i chip e il modulo di coltura consentendo il campionamento ripetuto e il dosaggio dei supporti.- Aprire i moduli portatili, nel BSC, e posizionarli sui vassoi dei moduli di coltura, che sono contenitori specializzati per l'allineamento dei moduli portatili.
- Aggiungere 3 mL di terreno di crescita organoide completo pre-bilanciato integrato con 5 μM CHIR99021 e 10 μM Y-27632 al serbatoio di ingresso superiore e 3 mL di terreno di crescita cellulare endoteliale preequilibrato nel serbatoio di ingresso inferiore (Figura 1C). Aggiungere 300 μL dello stesso mezzo nei rispettivi serbatoi di uscita.
- Portare i vassoi nell'incubatore a 37°C e farli scorrere nel modulo di coltura (Figura 1C). Utilizzare i controlli sullo schermo del modulo di coltura per eseguire il "Ciclo Prime" (1 minuto di durata). Quando la barra di stato indica "Pronto", il ciclo Prime è completato. Ripeti il ciclo Prime per assicurarti che si siano formate goccioline sufficienti per collegare correttamente i chip.
- Assicurarsi che tutti i moduli portatili siano innescati e abbiano goccioline di supporti visibili.
- Introduzione del flusso
NOTA: Il flusso viene in genere avviato 24 ore dopo la semina di frammenti organoidi per consentire alle cellule epiteliali intestinali di attaccarsi saldamente alla membrana.- Lavare entrambi i canali dei trucioli seminati con 200 μL del rispettivo mezzo per rimuovere eventuali bolle o detriti cellulari. Lasciare piccole goccioline di supporto sulle porte dopo il lavaggio. Far scorrere il supporto del chip nel modulo portatile (Figura 1C).
- Posizionare i moduli portatili sui vassoi e quindi nel modulo di coltura. Utilizzare i controlli sullo schermo del modulo di coltura per programmare le condizioni di coltura del chip dell'organo appropriate (portata e allungamento).
NOTA: per le condizioni standard di coltura del duodeno e del colon, impostare la portata rispettivamente su 30 μL/h15 e 60 μL/h16 , sia per i canali superiore che per quelli inferiori. Tuttavia, le portate per ciascun canale sono controllate in modo indipendente e possono essere impostate tra 0-1.000 μL/h. Le pompe a siringa o peristaltica possono essere utilizzate, al posto dei moduli di coltura qui presentati, per introdurre il flusso laminare nei chip microfluidici. Tuttavia, la creazione di connessioni fluidiche affidabili tra chip e pompe utilizzando tubi e connettori potrebbe essere tecnicamente impegnativa quando è richiesta la perfusione simultanea di più chip. - Avviare il "Ciclo regolatore" (lungo 2 ore) che pressurizza il terreno di coltura in modulo portatile e chip per impedire la nucleazione delle bolle d'aria. Le condizioni programmate riprenderanno dopo il completamento del Ciclo regolatore.
- Cambiamento dei media
- Preparare i fluidi freschi per entrambi i canali e ricostituirli ogni 48 ore aggiungendo 2 ml di terreno fresco ai serbatoi di ingresso (Figura 1C).
- Metti in pausa i moduli di coltura e porta i vassoi in BSC. Aspirare i mezzi in tutti i serbatoi e ricostituire con mezzi freschi. Riportare i vassoi e riavviare il flusso.
- Introduzione dello stretching
NOTA: Lasciare che le cellule crescano fino al 100% di confluenza prima dell'applicazione del ceppo ciclico. Lo stretching viene in genere introdotto 3 giorni dopo la semina o 48 ore dopo l'inizio della coltura fluidica. Il ceppo ciclico al 2% alla frequenza di 0,2 o 0,15 Hz, per il duodeno11 e il chip intestinaledel colon 24 , rispettivamente, viene applicato per le 24 ore iniziali. Viene quindi ulteriormente aumentato al 10% per la durata residua della coltura di chip intestinali per assomigliare molto al ceppo ciclico sperimentato dalle cellule epiteliali intestinali in vivo (Figura 1D)25. Il modulo di coltura può supportare l'applicazione di deformazioni cicliche del 2%-12% e una frequenza fino a 0,4 Hz.- Per introdurre l'allungamento alla cultura fluidica in corso, metti in pausa il modulo della cultura. Utilizzando i controlli sullo schermo, modificare le impostazioni di allungamento impostandole su 2% di allungamento, frequenza 0,2 o 0,15 Hz e riavviare il modulo di coltura.
- Dopo 24 ore, ripetere il passaggio 7.5.1 per applicare una frequenza di allungamento del 10%, 0,2 o 0,15 Hz.
8. Induzione del CYP450 utilizzando induttori CYP prototipici nel chip di duodeno
NOTA: Il test di induzione del citocromo P450 (CYP450) consente di valutare se il composto in esame aumenta i livelli di mRNA e/o l'attività catalitica di specifici enzimi CYP450. Qui, descriviamo il protocollo per la valutazione dell'induzione del CYP3A4 da parte dello standard industriale e del regolatore raccomandato in vitro induttori CYP, rifampicina (RIF) e 1,2-diidrossi vitamina D3 (VD3). Il metodo presentato può essere utilizzato per identificare il potenziale di vari composti in esame per indurre diverse isoforme di CYP450 nel tessuto intestinale umano. Per ciascuna isoforma enzimatica da valutare dovranno essere selezionati set specifici di primer e substrato di sonda.
- Esposizione agli induttori del CYP
- Preparare soluzioni stock di 20 mM RIF, 100 mM VD3 e 200 mM Testosterone (Tabella dei materiali) utilizzando DMSO sterile.
ATTENZIONE: Il testosterone è una sostanza controllata dalla Tabella III. Seguire le procedure normative durante la manipolazione. - Preparare il mezzo di dosaggio con induttori CYP diluendo le soluzioni stock in mezzo di crescita organoide completo e mezzo di crescita cellulare endoteliale per ottenere 20 μM RIF, 100 nmol/L di VD3. Preparare il controllo del veicolo diluendo DMSO nei rispettivi mezzi per ottenere lo 0,1% di DMSO.
- Metti in pausa il modulo di coltura e porta i vassoi in BSC. Sostituire il fluido in tutti i serbatoi di ingresso con 2 mL di mezzi di dosaggio con induttori o controllo del veicolo. Riportare i moduli portatili al modulo di coltura e riavviare il flusso a 30 μL/h.
- Dopo 24 ore, sostituire il supporto con una soluzione induttrice e ripetere i passaggi 8.1.2-8.1.3. Le soluzioni induttrici dovrebbero essere preparate fresche ogni giorno e reintegrate ogni 24 ore nel corso dell'esperimento, che in genere è lungo 48-72 ore.
- Preparare soluzioni stock di 20 mM RIF, 100 mM VD3 e 200 mM Testosterone (Tabella dei materiali) utilizzando DMSO sterile.
- Incubazione con un substrato prototipico (Testosterone)
- Il giorno della raccolta, preparare la soluzione di substrato della sonda di Testosterone insieme ai corrispondenti induttori in DMEM avanzato / F12 per ottenere una concentrazione finale di 200 μM. Omettere l'aggiunta del siero al mezzo in quanto ciò potrebbe interferire con l'analisi LCMS.
- Portare i vassoi a BSC e aspirare il mezzo di dosaggio da tutti i serbatoi. Lavare e sostituire i serbatoi di ingresso superiore e inferiore rispettivamente con il mezzo caldo Advanced DMEM/F12 e il mezzo di crescita cellulare endoteliale.
- Rimuovere il mezzo di lavaggio dai serbatoi e sostituirlo con 1 mL di soluzione di substrato della sonda, preparata al punto 8.2.1. Perfondere i trucioli ad una portata elevata di 1.000 μL/h per 5 minuti e aspirare sia i serbatoi di uscita superiore che quelli inferiori. Riportare i trucioli al modulo di coltura e incubare per 1 ora sotto il flusso costante di 300 μL/h.
- Analisi dei dati
- Raccolta di campioni per l'analisi LCMS
- Aliquota 200 μL di soluzione di arresto contenente acetonitrile con acido formico allo 0,1% in tubi premarcati da 1,5 mL e metterli su ghiaccio. La composizione della soluzione di arresto LCMS può variare in base al substrato che deve essere analizzato.
- Dopo 1 ora di trattamento è completo, interrompere il flusso e riportare i vassoi a BSC. Raccogliere 100 μL di effluente dal serbatoio di uscita superiore e aggiungerlo al tubo corrispondente contenente la soluzione di arresto (Figura 2A). Posizionare immediatamente i tubi sul ghiaccio secco. Conservare i campioni a -80°C prima di procedere all'analisi.
- Estrarre e analizzare i campioni utilizzando tecniche HPLC o LC-MS/MS, monitorando la formazione del metabolita-6β-idrossitestosterone (6β-OH-T). L'attività enzimatica del CYP può essere espressa come proteina pmol/min/mg dove pmol si riferisce alla quantità di metabolita (6β-OH-T) formatosi durante la reazione. Il contenuto proteico totale per chip (proteina; mg) è determinato eseguendo un saggio di Bradford descritto nel punto 8.3.2.

L'attività di induzione della piega è calcolata da: attività CYP (indotta) / attività CYP (veicolo). - Se si raccolgono campioni per l'analisi dell'mRNA, vedere il passaggio 8.3.3.
- Lisi delle cellule per l'analisi dell'espressione proteica
NOTA: L'estrazione delle proteine dalle cellule su chip viene eseguita utilizzando un tampone di lisi proteica, integrato con inibitori della proteasi e della fosfatasi (Tabella dei materiali). La proteina estratta viene quindi quantificata utilizzando il test di Bradford e utilizzata nell'analisi a valle.- Staccare i chip dai moduli portatili e metterli in una capsula di Petri. Lavare entrambi i canali con 200 μL di DPBS sterile.
- Bloccare l'uscita del canale inferiore con una punta della pipetta del filtro da 200 μL. Perfondere 50 μL della soluzione di dissociazione attraverso il canale inferiore e incubare per 2 minuti a temperatura ambiente.
- Ispezionare per confermare il completo distacco delle cellule endoteliali. Pipettare su e giù 1-2 volte e rimuovere la soluzione di dissociazione dal canale. Ripetere il lavaggio.
- Bloccare l'uscita del canale superiore con una punta della pipetta filtrante da 200 μL. Perfondere 75 μL di tampone di lisi proteica attraverso il canale superiore. Lasciare la punta della pipetta inserita, per bloccare l'ingresso del canale superiore e incubare per 5 minuti a temperatura ambiente. Pipettare su e giù 5-10 volte e raccogliere i lisati cellulari in un tubo pre-marcato da 1,5 ml.
- Ripetere il passaggio 8.3.2.4. fino a quando non si osserva il completo distacco delle cellule. Raccogliere i lisati cellulari nello stesso tubo da 1,5 ml e conservarli a -80 °C fino all'analisi.
- Estrarre la frazione proteica secondo metodi standard e quantificare la proteina totale utilizzando il saggio Bradford (Table of Materials).
- Lisi delle cellule per l'analisi dell'espressione genica
NOTA: la lisi cellulare su chip per l'estrazione dell'RNA può essere ottenuta utilizzando un tampone di lisi dell'RNA, integrato con 0,1% di 2-mercaptoetanolo (Tabella dei materiali). In alternativa, è possibile utilizzare un tampone di lisi dell'RNA a base di fenolo se sono richieste alte rese di RNA di alta qualità adatto per l'analisi di NGS e microarray.- Seguire i passaggi da 8.3.2.1 a 8.3.2.2 per preparare il chip intestinale per la lisi.
- Bloccare l'uscita del canale superiore utilizzando una punta della pipetta filtrante da 200 μL. Perfondere 150 μL di tampone di lisi dell'RNA attraverso il canale superiore. Lasciare la punta della pipetta inserita per bloccare l'ingresso del canale superiore. Incubare per 2 minuti a temperatura ambiente. Per la lisi utilizzando un tampone di lisi dell'RNA a base di fenoli, utilizzare 350 μL del reagente.
- Pipettare su e giù 5-10 volte e raccogliere i lisati cellulari in un tubo pre-marcato da 1,5 ml. Ripetere il passaggio con altri 150 μL di tampone di lisi dell'RNA e raccogliere. Conservare i lisati cellulari a -80 °C fino all'analisi. In caso di successiva analisi di sequenziamento dell'RNA, procedere con l'analisi entro 1 mese dopo la lisi.
- Estrarre l'RNA totale dai lisati cellulari utilizzando un kit di purificazione dell'RNA.
- Trascrivere inversamente in cDNA utilizzando un kit di trascrittasi inversa ed eseguire la PCR in tempo reale utilizzando un ciclo PCR in tempo reale e i primer e il buffer appropriati. Quantificare i risultati utilizzando il metodo 2-ΔΔCt .
- Raccolta di campioni per l'analisi LCMS
9. Interruzione della barriera epiteliale utilizzando citochine proinfiammatorie nel chip del colon
NOTA: Questo protocollo descrive l'interruzione della barriera epiteliale intestinale da parte della citochina interferone gamma (IFNγ)26,27,28,29. La citochina viene dosata nel canale inferiore del chip del colon data l'espressione basolaterale del recettore IFNγ sulle cellule epiteliali intestinali. Lo stimolo proinfiammatorio viene introdotto nel chip il giorno 5 della cultura, non appenal'app P è stata stabilizzata al di sotto di 0,5 x 10-6 cm/s. Un regime di dosaggio simile può essere utilizzato per altre citochine proinfiammatorie e agenti di interferenza.
- Stimolazione con IFNγ
- Preparare una soluzione madre da 100 μg/mL di IFNγ (Table of Materials) in acqua sterile di coltura cellulare. Conservare la soluzione madre a -80°C nel corso dell'esperimento. Per ogni esperimento, utilizzare sempre una scorta fresca di IFNγ ed evitare più di tre cicli di congelamento e scongelamento.
- Preparare la soluzione dosatrice IFNγ diluendo la soluzione madre nel mezzo di crescita cellulare endoteliale degassato per ottenere una concentrazione finale di 10-100 ng/mL.
- Portare i vassoi a BSC e rimuovere il mezzo dai serbatoi di ingresso del canale inferiore e sostituirlo con 3 ml del mezzo contenente IFNγ. Aggiornare il mezzo di dosaggio IFNγ ogni giorno.
- Posizionare i vassoi nel modulo di coltura e perfondere i trucioli ad una portata elevata di 1.000 μL/h per 5 minuti per iniziare il trattamento IFNγ. Riportare la portata a 60 μL/h e continuare la coltura fluidica.
- Analisi dei dati
- Valutazione della funzione di barriera epiteliale. La permeabilità apparente epiteliale (Papp), o funzione barriera, può essere misurata in vari punti temporali del post-trattamento di coltura con IFNγ, nei mezzi di scarico dei serbatoi di ingresso e di uscita di entrambi i canali. Il tracciante fluorescente deve essere aggiunto al mezzo di crescita organoide del canale superiore 4 ore prima della valutazionedell'app P. Tipicamente, 3 kDa Dextran Cascade Blue viene utilizzato come tracciante fluorescente e viene aggiunto nel mezzo 24 ore prima.
- Metti in pausa il modulo di coltura e porta i vassoi al BSC.
- Etichettare e preparare una piastra a parete nera da 96 pozzetti con 100 μL di DPBS per pozzetto. Utilizzando una pipetta multicanale da 200 μL, raccogliere e aggiungere 50 μL di effluente da tutti i serbatoi ai rispettivi pozzi (Figura 2B).
- Per preparare la curva standard, diluire il mezzo contenente 100 μg/mL di 3 kDa destrano Cascade Blue 1:3 in DPBS. Successivamente eseguire diluizioni seriali utilizzando una triplice diluizione del mezzo di crescita cellulare endoteliale in DPBS.
- Leggere la fluorescenza a 375 nm di eccitazione e 420 nm di emissione utilizzando un lettore di piastre. Utilizzare i valori OD misurati per calcolare la permeabilità apparente (app P) come segue:
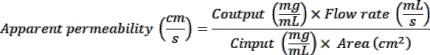
- Colorazione immunofluorescenza delle cellule su chip
- Lavare utilizzando 200 μL di DPBS per ogni canale, tre volte.
- Perfondere 200 μL della soluzione fissativa (4% PFA in DPBS) in ciascun canale. Incubare per 15 minuti a temperatura ambiente.
- Ripetere la fase di lavaggio 9.2.2.1. In questa fase, i trucioli lavati possono essere conservati fino a 7 giorni a 4°C.
- Permeabilizzare le cellule utilizzando 200 μL di soluzione di permeabilizzazione (0,1% Triton-X 100 in 10% di siero d'asino normale (NDS) in DPBS) per ciascun canale. Incubare per 30 minuti a temperatura ambiente. Ripetere la fase di lavaggio 9.2.2.1.
- Bloccare le celle sul chip utilizzando 200 μL di soluzione di blocco (10% NDS in DPBS) per ciascun canale. Incubare per 1 ora a temperatura ambiente.
- Diluire gli anticorpi primari in soluzione anticorpale (5% NDS in DPBS) come segue: anti-Zonula Occludens 1 (ZO-1) (1:100, epithelial tight junction marker), anti-Occludin (1:100, epithelial tight junction marker), anti-Claudin 4 (1:100, epithelial tight junction marker), anti-E-cadherin (1:100, epithelial adherens junctions marker (Table of Materials). Perfondere 200 μL della soluzione anticorpale primaria attraverso ciascun canale e incubare durante la notte a 4 °C. Ripetere la fase di lavaggio 9.2.2.1.
- Diluire gli anticorpi secondari in soluzione anticorpale (1:300, 5% NDS in DPBS). Perfondere 200 μL di questa soluzione attraverso ciascun canale e incubare per 2 ore a temperatura ambiente. Ripetere la fase di lavaggio 9.2.2.1. Se lo si desidera, la falloidina, un marcatore citoscheletrico, può essere aggiunta alla soluzione anticorpale secondaria.
- Preparare una soluzione da 50 μg/mL di 4′,6-diamidino-2-fenilindolo (DAPI) in DPBS e utilizzarla per perfondere 200 μL attraverso ciascun canale. Incubare a temperatura ambiente per 15 min. Ripetere la fase di lavaggio. A questo punto, i trucioli macchiati possono essere conservati fino a 14 giorni a 4°C.
- Valutazione della scissione della caspasi 3
- Isolare e quantificare la quantità totale di proteine delle cellule epiteliali come descritto nella fase 8.3.2. Omettere la fase di lavaggio 8.3.2.1. Diluire i campioni con DPBS ad una concentrazione finale di 400 μg/mL.
- Quantificare i livelli di caspasi 3 totale e scissa utilizzando il kit di rilevamento della caspasi 3 seguendo il protocollo del produttore.
- Valutazione della secrezione di citochine proinfiammatorie
- Utilizzare gli effluenti raccolti dall'uscita di entrambi i canali del chip del colon per quantificare le citochine proinfiammatorie in fase acuta secrete, seguendo i protocolli forniti dal produttore. Eseguire una diluizione 5 volte per V-PLEX Vascular Injury Panel 2 Human Kit e una doppia diluizione per V-PLEX Human Proinflammatory Panel II.
- Valutazione della funzione di barriera epiteliale. La permeabilità apparente epiteliale (Papp), o funzione barriera, può essere misurata in vari punti temporali del post-trattamento di coltura con IFNγ, nei mezzi di scarico dei serbatoi di ingresso e di uscita di entrambi i canali. Il tracciante fluorescente deve essere aggiunto al mezzo di crescita organoide del canale superiore 4 ore prima della valutazionedell'app P. Tipicamente, 3 kDa Dextran Cascade Blue viene utilizzato come tracciante fluorescente e viene aggiunto nel mezzo 24 ore prima.
Risultati
La Figura 1D riassume la cronologia della coltura del chip intestinale e illustra le cellule endoteliali intestinali e gli organoidi prima e dopo la semina sul chip. Inoltre, dimostra le distinte differenze morfologiche tra il duodeno e i chip del colon, evidenziate dalla presenza delle formazioni villi-like nel chip del duodeno e rappresentative dell'architettura del piccolo intestino.
La Figura 3A,B mostra le rispos...
Discussione
La combinazione della tecnologia organ-on-a-chip e degli organoidi intestinali promette una modellazione accurata della fisiologia intestinale umana e della fisiopatologia. Qui, forniamo un protocollo passo-passo semplice e robusto (delineato nella Figura 1) per la creazione del chip intestinale contenente epitelio intestinale o del colon derivato dalla biopsia e cellule endoteliali microvascolari intestinali co-coltivate in un dispositivo microfluidico. Questa simulazione basata su chip del...
Divulgazioni
Gauri Kulkarni, Athanasia Apostolou, Lorna Ewart, Carolina Lucchesi e Magdalena Kasendra sono dipendenti attuali o precedenti di Emulate Inc. e possono detenere azioni proprie. Emulate Inc. è la società che produce i dispositivi a chip d'organo e ha pubblicato brevetti relativi al lavoro dichiarato in questo articolo.
Riconoscimenti
Ringraziamo il professor Mark Donowitz per aver fornito gli organoidi derivati dalla biopsia intestinale e Brett Clair per aver progettato le illustrazioni scientifiche del chip, del portatile e del modulo di coltura. Tutte le altre illustrazioni scientifiche sono state generate utilizzando il BioRender.
Materiali
| Name | Company | Catalog Number | Comments |
| small intestine Human Intestinal Microvascular Endothelial Cells | AlphaBioRegen | ALHE15 | 0.5 cells M/ml ; cryopreserved |
| colon Human Intestinal Microvascular Endothelial Cells | AlphaBioRegen | ALHE16 | 0.5 cells M/ml ; cryopreserved |
| Biopsy-derived Human Duodenal Organoids | John Hopkin's University | - | The organoids were provided by Professor Mark Donowitz (Institutional Review Board Number: NA_00038329). |
| Biopsy-derived Human Colonic Organoids | John Hopkin's University | - | The organoids were provided by Professor Mark Donowitz (Institutional Review Board Number: NA_00038329). |
| Zoë CM-1™ Culture Module | Emulate Inc. | - | Culture module |
| Orb-HM1™ Hub Module | Emulate Inc. | - | 5% CO2, vacuum stretch, and power supply |
| Chip-S1™ Stretchable Chip | Emulate Inc. | - | Organ-Chip |
| Pod™ Portable Modules | Emulate Inc. | - | Portable module |
| UV Light Box | Emulate Inc. | - | - |
| Chip Cradle | Emulate Inc. | - | 1 per square culture dish |
| Steriflip®-HV Filters | EMD Millipore | SE1M003M00 | 0.45 μm PVDF filter |
| Square Cell Culture Dish (120 x 120 mm) | VWR | 82051-068 | - |
| Handheld vacuum aspirator | Corning | 4930 | - |
| Aspirating pipettes | Corning / Falcon | 357558 | 2 mL, polystyrene, individually wrapped |
| Aspirating tips | - | Sterile (autoclaved) | |
| Serological pipettes | - | 2 mL, 5 mL, 10 mL, and 25 mL low endotoxin, sterile | |
| Pipette | P20, P200, P1000 and standard multichannel | ||
| Pipette tips | P20, P200, and P1000. | ||
| Conical tubes (Protein LoBind® Tubes) | Eppendorf | 0030122216; 0030122240 | 15 mL, 50 mL tubes |
| Eppendorf Tubes® lo-bind | Eppendorf | 022431081 | 1.5 mL tubes |
| 96 wells black walled plate | - | - | for epithelial permeability analysis |
| Microscope (with camera) | - | - | For bright-field imaging |
| Water bath (or beads) | - | - | Set to 37°C |
| Vacuum set-up | - | - | Minimum pressure: -70 kPa |
| Cell scrapers | Biotium | 220033 | |
| T75 flasks | BD Falcon | 353136 | Cell culture flask |
| Emulate Reagent-1 (ER-1) | Emulate Inc. | - | Chip coating solution |
| Emulate Reagent-2 (ER-2) | Emulate Inc. | - | Chip coating solution |
| Dulbecco’s PBS (DPBS) | Corning | 21-031-CV | 1X |
| Cell Culture Grade Water | Corning | MT25055CV | |
| Trypan blue | Sigma | 93595 | For cell counting |
| TryplE Express | ThermoFisher Scientific | 12604013 | Organoids dissociation and endothelium cells detachment solution |
| Advanced DMEM/F12 | ThermoFisher Scientific | 12634028 | Medium |
| IntestiCult™ Organoid Growth Medium (Human) | Stem Cell technologies | 06010 | Organoid Growth Medium |
| Endothelial Cell Growth Medium MV 2 | Promocell | C-22121 | Endothelial medium |
| Fetal bovine serum (FBS) | Sigma | F4135 | Serum |
| Primocin™ | InvivoGen | ANT-PM-1 | antimicrobial agent |
| Attachment Factor™ | Cell Systems | 4Z0-210 | coating solution for flask |
| Matrigel - Growth Factor Reduced | Corning | 356231 | Solubilized basement membrane matrix |
| Collagen IV | Sigma | C5533 | ECM component |
| Fibronectin | Corning | 356008 | ECM component |
| Y-27632 | Stem Cell technologies | 72304 | organoid media supplement |
| CHIR99021 | Reprocell | 04-0004-10 | organoid media supplement |
| Cell Recovery Solution | Corning | 354253 | Basement mebrane matrix dissociationsolution |
| Bovine Serum Albumin (BSA) | Sigma | A9576 | 30%, Sterile |
| Cell Culture Grade Water | Corning | MT25055CV | Sterile, Water |
| DMSO | Sigma | D2650 | solvent |
| 3KDa Dextran Cascade Blue | Invitrogen | D7132 | 10 mg powder |
| Rifampicin (RIF) | Sigma | Cat# R3501 | CYP inducer |
| Testosterone hydrate | Sigma | T1500 | CYP substrate |
| 1,25-dihyroxy Vitamin D3 (VD3) | Sigma | Cat# D1530 | CYP inducer |
| Acetonitrile with 0.1% (v/v) Formic acid | Sigma | 159002 | LCMS stop solution |
| IFNγ | Peprotech | 300-02 | |
| 4% Paraformaldehyde (PFA) | EMS | 157-4 | Fixative |
| Triton-X 100 | Sigma | T8787 | |
| Normal Donkey Serum (NDS) | Sigma | 566460 | |
| anti-Occludin | ThermoFisher Scientific | 33-1500 | tight junctions marker |
| anti-Claudin 4 | ThermoFisher Scientific | 36-4800 | tight junctions marker |
| anti-E-cadherin | Abcam | ab1416 | epithelial adherens junctions marker |
| anti-VE-cadherin | Abcam | ab33168 | endothelial adherent junctions marker |
| anti- Zonula Occludens 1 (ZO-1) | Thermo Fischer | 339194 | tight junctions marker |
| DAPI | ThermoFisher Scientific | 62248 | nuclear stain |
| 2-mercaptoethanol | Sigma | M6250 | |
| PureLink RNA Mini Kit | Invitrogen | 12183020 | RNA lysis, isolation and purification kit |
| SuperScript™ IV VILO™ Master Mix | Invitrogen | 11756050 | reverse transcriptase kit |
| TaqMan™ Fast Advanced Master Mix | Applied Biosystems | 4444557 | qPCR reagent |
| QuantStudio™ 5 Real-Time PCR System | Applied Biosystems | A28573 | Real-time PCR cycler |
| 18S primer | ThermoFisher Scientific | Hs99999901_s1 | Eukaryotic 18S rRNA |
| CYP3A4 primer | ThermoFisher Scientific | Hs00604506_m1 | Cytochrome family 3 subfamily A member 4 |
| Pierce™ Coomassie Plus (Bradford) Assay Kit | ThermoFisher Scientific | 23236 | Protein quantification kit |
| MSD Tris lysis buffer | Meso Scale Diagnostics | R60TX-3 | Protein lysis buffer |
| Cleaved/Total Caspase-3 Whole Cell Lysate Kit | Meso Scale Diagnostics | K15140D | Caspase 3 detection kit |
| V-PLEX Vascular Injury Panel 2 Human Kit | Meso Scale Diagnostics | K15198D | |
| V-PLEX Human Proinflammatory Panel II (4-Plex) | Meso Scale Diagnostics | K15053D | |
| Zeiss LSM 880 | Zeiss | - | Confocal microscope |
| Zeiss LD plan-Neofluar 20x/0.40 Korr M27 | Zeiss | - | 20X long-distance objective lenses |
| Zeiss AXIOvert.A1 | Zeiss | - | Brightfield microscope |
| Zeiss LD A-Plan 10X/0.25 Ph1 | Zeiss | - | 10X objective lenses |
Riferimenti
- Fritz, A., et al. Expression of clinically relevant drug-metabolizing enzymes along the human intestine and their correlation to drug transporters and nuclear receptors: An intra-subject analysis. Basic and Clinical Pharmacology and Toxicology. 124 (3), 245-255 (2019).
- Okumura, R., Takeda, K. Maintenance of intestinal homeostasis by mucosal barriers. Inflammation and Regeneration. 38 (1), 1-8 (2018).
- Delgado, M. E., Grabinger, T., Brunner, T. Cell death at the intestinal epithelial front line. FEBS Journal. 283 (14), 2701-2719 (2016).
- Mestas, J., Hughes, C. C. W. Of mice and not men: Differences between mouse and human immunology. The Journal of Immunology. 172 (5), 2731-2738 (2004).
- Sun, H., Chow, E. C. Y., Liu, S., Du, Y., Pang, K. S. The Caco-2 cell monolayer: Usefulness and limitations. Expert Opinion on Drug Metabolism and Toxicology. 4 (4), 395-411 (2008).
- Yu, H., et al. The contributions of human mini-intestines to the study of intestinal physiology and pathophysiology. Annual Review of Physiology. 79, 291-312 (2017).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett’s epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470 (7332), 105-110 (2011).
- Durel, J. F., Nerurkar, N. L. Mechanobiology of vertebrate gut morphogenesis. Current Opinion in Genetics and Development. 63, 45-52 (2020).
- Gayer, C. P., Basson, M. D. The effects of mechanical forces on intestinal physiology and pathology. Cellular Signalling. 21 (8), 1237-1244 (2009).
- Xu, Y., et al. Mechanical stimulation activates Piezo1 to promote mucin2 expression in goblet cells. Journal of Gastroenterology and Hepatology (Australia). 36 (11), 3127-3139 (2021).
- Navabi, N., McGuckin, M. A., Lindén, S. K. Gastrointestinal cell lines form polarized epithelia with an adherent mucus layer when cultured in semi-wet interfaces with mechanical stimulation. PLoS One. 8 (7), 68761 (2013).
- Kasendra, M., et al. Development of a primary human Small Intestine-on-a-Chip using biopsy-derived organoids. Scientific Reports. 8 (1), 1-14 (2018).
- Kasendra, M., et al. Duodenum intestine-chip for preclinical drug assessment in a human relevant model. eLife. 9, 50135 (2020).
- Apostolou, A., et al. A novel microphysiological colon platform to decipher mechanisms driving human intestinal permeability. Cellular and Molecular Gastroenterology and Hepatology. 12 (5), 1719-1741 (2021).
- Jalili-Firoozinezhad, S., et al. Author correction: A complex human gut microbiome cultured in an anaerobic intestine-on-a-chip. Nature Biomedical Engineering. 3 (7), 583 (2019).
- Yin, J., et al. Fluid shear stress enhances differentiation of jejunal human enteroids in Intestine-Chip. American Journal of Physiology-Gastrointestinal and Liver Physiology. 320 (3), 258-271 (2021).
- Sontheimer-Phelps, A., et al. Human colon-on-a-chip enables continuous in vitro analysis of colon mucus layer accumulation and physiology. Cellular and Molecular Gastroenterology and Hepatology. 9 (3), 507-526 (2020).
- Tovaglieri, A., et al. Species-specific enhancement of enterohemorrhagic E. coli pathogenesis mediated by microbiome metabolites. Microbiome. 7 (1), 43 (2019).
- Huh, D., et al. Microfabrication of human organs-on-chips. Nature Protocols. 8 (11), 2135-2157 (2013).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab on a Chip. 12 (12), 2165-2174 (2012).
- Sarna, S. K. Colonic motility: From bench side to bedside. Morgan & Claypool Life Sciences. , (2010).
- Basson, M. D. Paradigms for mechanical signal transduction in the intestinal epithelium - category: Molecular, cell, and developmental biology. Digestion. 68 (4), 217-225 (2003).
- Wang, F., et al. Interferon-gamma and tumor necrosis factor-alpha synergize to induce intestinal epithelial barrier dysfunction by up-regulating myosin light chain kinase expression. The American Journal of Pathology. 166 (2), 409-419 (2005).
- Nava, P., et al. Interferon-γ regulates intestinal epithelial homeostasis through converging β-Catenin signaling pathways. Immunity. 32 (3), 392-402 (2010).
- Madara, J. L., Stafford, J. Interferon-γ directly affects barrier function of cultured intestinal epithelial monolayers. Journal of Clinical Investigation. 83 (2), 724-727 (1989).
- Bruewer, M., et al. Proinflammatory cytokines disrupt epithelial barrier function by apoptosis-independent mechanisms. The Journal of Immunology. 171 (11), 6164-6172 (2003).
- Jones, S. C., et al. Adhesion molecules in inflammatory bowel disease. Gut. 36 (5), 724-730 (1995).
- Uhlar, C. M., Whitehead, A. S. Serum amyloid A, the major vertebrate acute-phase reactant. European Journal of Biochemistry. 265 (2), 501-523 (1999).
- Pérez-González, C., Ceada, G., Matejčić, M., Trepat, X. Digesting the mechanobiology of the intestinal epithelium. Current Opinion in Genetics & Development. 72, 82-90 (2022).
- In, J., et al. Enterohemorrhagic escherichia coli reduces mucus and intermicrovillar bridges in human stem cell-derived colonoids. Cellular and Molecular Gastroenterology and Hepatology. 2 (1), 48-62 (2016).
- Grassart, A., et al. Bioengineered human organ-on-chip reveals intestinal microenvironment and mechanical forces impacting shigella infection. Cell Host and Microbe. 26 (3), 435-444 (2019).
- Kerns, S. J., et al. Human immunocompetent organ-on-chip platforms allow safety profiling of tumor-targeted t-cell bispecific antibodies. eLife. 10, 67106 (2021).
- Bhatia, S. N., Ingber, D. E. Microfluidic organs-on-chips. Nature Biotechnology. 32 (8), 760-772 (2014).
- Ingber, D. E. Reverse engineering human pathophysiology with organs-on-chips. Cell. 164 (6), 1105-1109 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon