È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento dei vettori di terapia genica di prossima generazione attraverso l'ingegneria, il codice a barre e lo screening delle varianti del capside del virus adeno-associato (AAV)
In questo articolo
Riepilogo
Generazione della libreria di visualizzazione dei peptidi AAV e successiva validazione attraverso il codice a barre di candidati con nuove proprietà per la creazione di AAV di nuova generazione.
Abstract
I vettori di rilascio genico derivati dal virus adeno-associato (AAV) sono uno degli strumenti più promettenti per il trattamento delle malattie genetiche, evidenziato da dati clinici incoraggianti e dall'approvazione di diverse terapie geniche AAV. Due ragioni principali per il successo dei vettori AAV sono (i) il precedente isolamento di vari sierotipi virali naturali con proprietà distinte e (ii) la successiva creazione di potenti tecnologie per la loro ingegneria molecolare e il loro riutilizzo ad alto rendimento. A rafforzare ulteriormente il potenziale di queste tecniche sono state recentemente implementate strategie per la codifica a barre di capsidi AAV selezionati a livello di DNA e RNA, consentendo la loro stratificazione completa e parallela in vivo in tutti i principali organi e tipi di cellule in un singolo animale. Qui, presentiamo una pipeline di base che comprende questo insieme di strade complementari, utilizzando la visualizzazione del peptide AAV per rappresentare il variegato arsenale di tecnologie di ingegneria del capside disponibili. Di conseguenza, descriviamo prima i passaggi cruciali per la generazione di una libreria di visualizzazione del peptide AAV per la selezione in vivo di candidati con proprietà desiderate, seguita da una dimostrazione di come codificare a barre le varianti di capside più interessanti per lo screening secondario in vivo. Successivamente, esemplificamo la metodologia per la creazione di librerie per il sequenziamento di nuova generazione (NGS), tra cui l'amplificazione dei codici a barre e la legatura degli adattatori, prima di concludere con una panoramica dei passaggi più critici durante l'analisi dei dati NGS. Poiché i protocolli qui riportati sono versatili e adattabili, i ricercatori possono facilmente sfruttarli per arricchire le varianti ottimali di capside AAV nel loro modello di malattia preferito e per applicazioni di terapia genica.
Introduzione
La terapia di trasferimento genico è l'introduzione di materiale genetico nelle cellule per riparare, sostituire o alterare il materiale genetico cellulare per prevenire, trattare, curare o migliorare la malattia. Il trasferimento genico, sia in vivo che ex vivo, si basa su diversi sistemi di veicola, non virali e virali. I virus si sono evoluti naturalmente per trasdurre in modo efficiente le loro cellule bersaglio e possono essere utilizzati come vettori di consegna. Tra i diversi tipi di vettori virali impiegati nella terapia genica, i virus adeno-associati sono stati sempre più utilizzati, a causa della loro mancanza di patogenicità, sicurezza, bassa immunogenicità e, soprattutto, la loro capacità di sostenere l'espressione a lungo termine e non integrante 1,2,3. La terapia genica AAV ha prodotto risultati considerevoli negli ultimi dieci anni; tre terapie sono state approvate dall'Agenzia europea per i medicinali e dalla Food and Drug Administration statunitense per l'uso nell'uomo 3,4. Diversi studi clinici sono anche in corso per trattare una varietà di malattie, come l'emofilia, le malattie muscolari, cardiache e neurologiche, come esaminato altrove3. Nonostante decenni di progressi, il campo della terapia genica ha subito una serie di battute d'arresto negli ultimi anni4, soprattutto decessi negli studi clinici5 che sono stati sospesi a causa di tossicità dose-limitanti, in particolare per i tessuti che sono massicci, come i muscoli, o difficili da raggiungere, come il cervello6.
I vettori AAV attualmente utilizzati negli studi clinici appartengono ai sierotipi naturali con poche eccezioni1. L'ingegneria AAV offre l'opportunità di sviluppare vettori con specificità ed efficienza superiori per organi o cellule. Negli ultimi due decenni, diversi approcci sono stati applicati con successo, come la visualizzazione del peptide, il loop-swap, il rimescolamento del DNA del capside, la PCR soggetta a errori e la progettazione mirata, per generare singole varianti AAV o librerie di esse con proprietà diverse7. Questi sono poi sottoposti a più cicli di evoluzione diretta per selezionare le varianti al loro interno con le proprietà desiderate, come esaminato altrove 1,3. Di tutte le strategie di evoluzione del capside, le librerie AAV di visualizzazione dei peptidi sono state le più utilizzate, a causa di alcune proprietà uniche: sono relativamente facili da generare e possono raggiungere un'elevata diversità e un sequenziamento ad alto rendimento, che consente di seguire la loro evoluzione.
Le prime librerie AAV di successo per l'inserimento di peptidi sono state descritte quasi 20 anni fa. In uno dei primi, Perabo et al.8 hanno costruito una libreria di capsidi AAV2 modificati, in cui un pool di oligonucleotidi generati casualmente è stato inserito in un plasmide in una posizione corrispondente all'amminoacido 587 della proteina del capside VP1, nel triplice asse che sporge dal capside. Utilizzando la co-infezione da adenovirus, la libreria AAV è stata evoluta attraverso più cicli di selezione e le varianti finali re-mirate hanno dimostrato di essere in grado di trasdurre linee cellulari refrattarie all'AAV2 8 parentale. Poco dopo, Müller et al.9 introdussero il sistema in due fasi per la produzione di biblioteche, un miglioramento significativo del protocollo. Inizialmente, la libreria plasmidica, insieme a un plasmide aiutante adenovirale, viene utilizzata per produrre una libreria AAV che contiene capsidi chimerici. Questa libreria navetta AAV viene utilizzata per infettare le cellule a bassa molteplicità di infezione (MOI), con l'obiettivo di introdurre un genoma virale per cellula. La co-infezione con adenovirus garantisce la produzione di AAV con un genoma e un capside 9corrispondenti. Circa un decennio dopo, Dalkara10 ha utilizzato l'evoluzione diretta in vivo per creare la variante 7m8. Questa variante ha un'inserzione di 10 aminoacidi (LALGETTRPA), tre dei quali agiscono come linker, e colpisce efficacemente la retina esterna dopo l'iniezione intravitreale10. Questo capside ingegnerizzato è una storia di successo eccezionale, in quanto è uno dei pochi capsidi ingegnerizzati ad arrivare alla clinica finora11.
Il campo ha registrato un secondo impulso con l'introduzione di tecniche di sequenziamento di nuova generazione (NGS). Due pubblicazioni di Adachi et al.12 nel 2014 e di Marsic et al.13 nel 2015, hanno mostrato la potenza di NGS per tracciare la distribuzione di librerie di capsidi AAV con codice a barre con elevata precisione. Alcuni anni dopo, l'NGS delle regioni con codice a barre è stato adattato alla regione di inserzione del peptide per seguire l'evoluzione del capside. Körbelin et al.14 hanno eseguito uno screening guidato da NGS per identificare un capside basato su AAV2 mirato ai polmoni. L'analisi NGS ha aiutato a calcolare tre punteggi di valutazione: il punteggio di arricchimento tra i turni di selezione, il punteggio di specificità generale per determinare la specificità del tessuto e infine il punteggio combinato14. Il laboratorio Gradinaru15 ha pubblicato nello stesso anno il sistema di evoluzione mirata AAV basato sulla ricombinazione Cre, che facilita una selezione specifica del tipo di cellula. In questo sistema, la libreria capside trasporta uno switch Cre-invertibile, poiché il segnale polyA è affiancato da due siti loxP. La libreria AAV viene quindi iniettata nei topi Cre, dove il segnale polyA viene invertito solo nelle cellule Cre+, fornendo il modello per il legame di un primer PCR inverso con il primer forward all'interno del gene del capside. Questo salvataggio PCR altamente specifico ha permesso l'identificazione dell'AAV-PHP. Variante B che può attraversare la barriera emato-encefalica15. Questo sistema è stato ulteriormente evoluto in M-CREATE (Multiplexed-CREATE), in cui NGS e generazione di librerie sintetiche sono stati integrati nella pipeline16.
Una versione migliorata basata sull'RNA di questo sistema dal laboratorio Maguire17, iTransduce, consente la selezione a livello del DNA dei capsidi che trasducono funzionalmente le cellule ed esprimono i loro genomi. Il genoma virale della libreria di visualizzazione del peptide comprende un gene Cre sotto il controllo di un promotore ubiquitario e il gene del capside sotto il controllo del promotore p41. La libreria viene iniettata in topi che hanno una cassetta loxP-STOP-loxP a monte di tdTomato. Le cellule trasdotte con varianti AAV che esprimono il genoma virale e quindi Cre esprimono tdTomato e, in combinazione con marcatori cellulari, possono essere selezionatee selezionate 17. Allo stesso modo, Nonnenmacher et al.18 e Tabebordbar et al.19 hanno posto la libreria genica del capside sotto il controllo di promotori tessuto-specifici. Dopo l'iniezione in diversi modelli animali, l'RNA virale è stato utilizzato per isolare le varianti del capside.
Un approccio alternativo consiste nell'utilizzare il codice a barre per contrassegnare le librerie di capside. Il laboratorio Björklund20 ha utilizzato questo approccio alle librerie di capsidi di inserzione di peptidi con codice a barre e ha sviluppato l'evoluzione razionale del vettore AAV con codice a barre (BRAVE). In un plasmide, la cassetta Rep2Cap viene clonata accanto a un transgene con codice a barre che esprime una proteina fluorescente gialla (YFP) con ripetizioni terminali invertite (ITR). Utilizzando siti loxP tra la fine del tappo e l'inizio del codice a barre, una ricombinazione Cre in vitro genera un frammento abbastanza piccolo per NGS, consentendo così l'associazione dell'inserimento del peptide con il codice a barre univoco (look-up table, LUT). La produzione di AAV viene eseguita utilizzando la libreria plasmidica e i codici a barre espressi nell'mRNA vengono sottoposti a screening dopo l'applicazione in vivo , sempre con NGS20. Quando le librerie del capside comprendono varianti dell'intero gene del capside (cioè librerie mescolate), è necessario utilizzare il sequenziamento a lunga lettura. Diversi gruppi hanno utilizzato codici a barre per etichettare queste diverse librerie, il che consente a NGS una maggiore profondità di lettura. Il laboratorio Kay21 ha etichettato librerie mescolate di capside molto diverse con codici a barre a valle del segnale polyA del cappuccio . In una prima fase, è stata generata una libreria di plasmidi con codice a barre e la libreria del gene del capside mescolato è stata clonata in essa. Quindi è stata utilizzata una combinazione di MiSeq (lettura breve, profondità di lettura più elevata) e PacBio (lettura lunga, profondità di lettura inferiore) NGS e sequenziamento Sanger per generare la loro LUT21. Nel 2019, Ogden e colleghi del laboratorio Church22 hanno delineato l'idoneità del capside AAV2 per più funzioni utilizzando librerie che avevano mutazioni a singolo punto, inserzioni e delezioni in ogni posizione, che alla fine hanno permesso la progettazione guidata dalla macchina. Per la generazione della libreria, frammenti più piccoli del gene del capside sono stati sintetizzati, etichettati con un codice a barre, sequenziati di nuova generazione e quindi clonati nel gene del capside completo. I dati NGS sono stati utilizzati per generare una LUT. La libreria è stata quindi vagliata utilizzando solo i codici a barre e il sequenziamento a lettura breve, che a sua volta consente una profondità di lettura più elevata22.
Le librerie con codice a barre sono state utilizzate prevalentemente per vagliare un pool di varianti note, naturali e ingegnerizzate a seguito di diversi cicli di selezione delle librerie di capsidi o indipendentemente da uno studio sull'evoluzione del capside. Il vantaggio di tali librerie è l'opportunità di esaminare più capsidi, riducendo al contempo il numero di animali e minimizzando le variazioni tra gli animali. I primi studi che hanno introdotto questa tecnologia nel campo AAV sono stati pubblicati quasi un decennio fa. Il laboratorio Nakai 12 ha etichettato 191 mutanti doppi dell'alanina che coprono gli amminoacidi da 356 a 736 sul VP1 da AAV9 con una coppia di codici a barre a12 nucleotidi. Utilizzando NGS, la libreria è stata esaminata in vivo per il legame con il galattosio e altre proprietà12. Marsic e colleghi hanno delineato la biodistribuzione delle varianti AAV utilizzando anche un'analisi a doppia barra 1 anno dopo13. Uno studio più recente su primati non umani ha confrontato la biodistribuzione nel sistema nervoso centrale di 29 capsidi utilizzando diverse vie di consegna23. Il nostro laboratorio ha recentemente pubblicato schermi di librerie AAV con codice a barre di 183 varianti che includevano AAV naturali e ingegnerizzati. Questi screening a livello di DNA e RNA hanno portato all'identificazione di una variante AAV altamente miotropica24 nei topi e in altri che mostrano un'elevata specificità di tipo cellulare nel cervello del topo25.
Qui, descriviamo la metodologia utilizzata in questo lavoro e la espandiamo per includere lo screening delle librerie di visualizzazione dei peptidi AAV. Ciò comprende la generazione di librerie di visualizzazione dei peptidi AAV2, un metodo di PCR digitale a goccia (dd-PCR) per la quantificazione e, infine, una pipeline NGS per analizzare le varianti AAV, basata in parte sul lavoro di Weinmann e colleghi24. Infine, viene fornita una descrizione della generazione di librerie AAV con codice a barre e della pipeline NGS utilizzata nella stessa pubblicazione.
Protocollo
1. Preparazione della libreria di visualizzazione del peptide casuale 7-mer AAV2
NOTA: Per la preparazione di una libreria di visualizzazione di peptidi casuali AAV2, sintetizzare gli oligonucleotidi degenerati come DNA a singolo filamento, convertirlo in DNA a doppio filamento, digerire, ligate al plasmide accettore ed elettromerato.
- Progettazione di oligonucleotidi degenerati
- Ordinare gli oligonucleotidi degeneri ed evitare la distorsione del codone. Nell'oligonucleotide 5' CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3', X01 corrisponde a 20 codoni, ciascuno dei quali codifica per uno dei 20 amminoacidi. Il W può essere A o T, producendo i codoni AGA o AGT, che codificano gli amminoacidi arginina (R) o serina (S).
- Ordinare il primer di amplificazione: 5' CTCGTCAGCCGCCTGG 3' (vedere la Figura 1 per i dettagli). Questo produce il seguente inserto proteico: R/S G X7. La diversità teorica è calcolata come segue: 1 x 2 x 207 = 2,56 x 109 varianti uniche.
NOTA: Va notato che questa diversità potrebbe essere limitata dall'efficienza della trasformazione.
- Sintesi del secondo filamento
- Risospendere entrambi gli oligonucleotidi (oligonucleotidi degenerati e primer di amplificazione) ad una concentrazione finale di 100 μM con tampone TE.
- Per la reazione PCR, impostare una reazione da 50 μL con 1 μL di ciascun primer, 10 μL del tampone, 1,5 μL di DMSO, 0,5 μL di dNTPs (10 mM), 0,5 μL di polimerasi II ad alta fedeltà e 35,5 μL di acqua priva di nucleasi.
- Trasferire la reazione su un termociclatore ed eseguire una fase di pre-incubazione per 10 s a 98 °C, seguita da tre cicli di 10 s a 98 °C, 30 s a 59 °C e 10 s a 72 °C, quindi 5 minuti a 72 °C e una fase di raffreddamento finale.
- Purificare la reazione utilizzando un kit di rimozione dei nucleotidi ed eluire in 100 μL di acqua priva di nucleasi.
- Confermare l'efficienza della sintesi del secondo filamento mediante analisi su un bioanalizzatore (vedere Figura 2). Analizzare le dimensioni e la purezza dell'inserto a doppio filamento caricando 1 μL della reazione a un chip microfluidico da un kit di reagenti DNA 1000 secondo le istruzioni del produttore. Questo kit è ottimizzato per misurare le dimensioni e la concentrazione di frammenti di DNA a doppio filamento da 25-1.000 bps.
- Risospendere entrambi gli oligonucleotidi (oligonucleotidi degenerati e primer di amplificazione) ad una concentrazione finale di 100 μM con tampone TE.
- Digestione dell'inserto e del vettore plasmidico
- Digerire 85 μL dell'inserto purificato con 10 μL di tampone 10x e 5 μL di enzima BglI in un volume finale di reazione di 100 μL (vedere Figura 1 per i dettagli). Incubare a 37 °C durante la notte. Purificare utilizzando un kit di rimozione dei nucleotidi, eluire in 50 μL di acqua priva di nucleasi e quantificare utilizzando il tipo "Oligo DNA" in uno spettrofotometro.
- Digerire 10 μg di un plasmide AAV (pRep2Cap2_PIS)26 competente per la replicazione (genoma virale affiancato da ITR) con 20 μL di tampone 10x e 10 μL di enzima SfiI in un volume di reazione finale di 200 μL (vedere Figura 1 per i dettagli). Incubare a 50 °C durante la notte. Purificare il vettore su un gel di agarosio all'1% utilizzando il kit di estrazione del gel seguito da un'ulteriore fase di purificazione utilizzando un kit di purificazione del DNA. Quantificare la concentrazione in uno spettrofotometro.
- Legatura dell'inserto al vettore
- Ligate 955 ng di vettore plasmidico con 45 ng di inserto con 2 μL di tampone e 2 μL di ligasi in una reazione di legatura da 20 μL. Incubare a 16 °C durante la notte, seguito da 10 minuti a 70 °C per inattivare termicamente la ligasi.
- Trasformazione, calcolo della complessità e preparazione della libreria di plasmidi
- Purificare la reazione con un kit di purificazione del DNA seguendo le istruzioni del produttore. Eluire la reazione in circa l'80% del volume iniziale di acqua priva di nucleasi e immagazzinarla sul ghiaccio per la successiva trasformazione.
- Trasformare le cellule elettrocompetenti: scongelare una fiala di cellule elettrocompetenti sul ghiaccio per 10 minuti. Quindi aggiungere 1-2 μL della reazione di legatura purificata a 30 μL (un flaconcino) di cellule elettrocompetenti e mescolare picchiettando delicatamente. Quindi, pipettare accuratamente la miscela cellula/DNA in una cuvetta di elettroporazione a gap da 1 mm pre-raffreddata senza introdurre bolle d'aria.
- Elettropore utilizzando le seguenti impostazioni: 1800 V, 600 Ω e 10 μF. Entro 10 s dall'impulso di elettroporazione, aggiungere 970 μL di mezzi di recupero preriscaldati (forniti con le celle elettrocompetenti) alla cuvetta e miscelare mediante pipettaggio. Infine, trasferire le cellule in una provetta da microcentrifuga e incubare per 1 ora a 37 °C a 250 giri/min. Per ottenere la diversità desiderata, eseguire 10-100 reazioni e, dopo l'incubazione, raggruppare tutte le reazioni in un provetto.
- Calcolare la diversità diluendo 10 μL delle trasformazioni aggregate 10-, 100- o 1.000 volte in PBS e distribuire 100 μL su piastre di agar nutrienti contenenti l'antibiotico appropriato (75 mg / ml di ampicillina). Incubare le piastre di agar per una notte a 37 °C e poi contare le colonie sulle piastre di agar.
- Calcola la diversità teorica come segue:
Diversità massima teorica = 10 x fattore di diluizione x numero di colonie x numero di reazioni di elettroporazione.
NOTA: Per confermare la qualità della libreria, sequenziare almeno 20 colonie mediante sequenziamento Sanger. La maggior parte dei cloni dovrebbe contenere un inserto e tutti dovrebbero essere unici. - Inoculare 400-1.000 mL di terreno LB contenente l'antibiotico appropriato con il resto delle trasformazioni aggregate e incubare per una notte a 37 °C, 180 giri/min.
- Preparazione della libreria di plasmidi
- Dalla coltura notturna, preparare uno stock di glicerolo (mescolare volumi uguali di coltura batterica e soluzione di glicerolo al 50% in acqua priva di nucleasi e congelare a -80 °C) e purificare la libreria plasmidica utilizzando un maxi kit plasmide.
- Produzione di libreria virale AAV
- Preparare la libreria virale come descritto in precedenza27. Transfettare la libreria plasmidica (pRep2Cap2_PI, inserto peptidico) insieme a un plasmide adeno-helper alle cellule HEK293T utilizzando un reagente di trasfezione come la polietilenimina (PEI).
- Raccogliere le cellule dopo 3 giorni e sottoporle a tre cicli di congelamento-disgelo. Purificare il lisato virale utilizzando l'ultracentrifugazione a gradiente di cloruro di cesio, seguita dallo scambio tampone a PBS e infine concentrare le particelle virali.
- Titolazione vettoriale AAV mediante dd-PCR
- Diluire in serie 2 μL del materiale madre vettoriale AAV in 198 μL di acqua priva di nucleasi per ottenere una diluizione finale 1:106 . Miscelare accuratamente ogni volta utilizzando una pipetta da 200 μL. Aggiungere un controllo senza modello (NTC) come controllo negativo.
NOTA: Ulteriori diluizioni inferiori o superiori possono essere analizzate (1:105-1:107). - Preparare un mix 20x primer-sonda. Aggiungere 3,6 μL di ciascuno dei primer da 100 μM (avanti e indietro, Rep2 e ITR), 1 μL ciascuna delle sonde dd-PCR da 100 μM (Rep2 e ITR) e 3,6 μL di acqua priva di nucleasi a una provetta da centrifuga da 1,5 ml.
NOTA: La libreria AAV viene misurata utilizzando un set di sonda primer mirato al transgene (Rep2) rilevato con una sonda marcata con FAM e un set di sonda primer mirato a ITR rilevato con una sonda marcata HEX. - Preparare una reazione PCR da 22 μL aggiungendo 5,5 μL di campione, 1,1 μL di miscela primer-sonda 20x, 11 μL di supermiscela dd-PCR per sonde (senza dUTP) e 4,4 μL di acqua priva di nucleasi. Ciò produce concentrazioni di 900 nM e 250 nM rispettivamente per i primer e la sonda.
- Generare le goccioline utilizzando un generatore di gocce, trasferire la reazione su una piastra a 96 pozzetti, posizionare la piastra in un termociclatore ed eseguire una fase di denaturazione per 10 minuti a 94 °C, seguita da 40 cicli di 30 s a 94 °C e 1 min a 58 °C. Quindi, inattivare a caldo la polimerasi per 10 minuti a 98 °C e aggiungere una fase di raffreddamento finale. Leggere le reazioni in un lettore di gocce e procedere all'analisi28.
- Aprire il file di lastra dd-PCR salvato utilizzando il software di analisi. Utilizzare lo strumento soglia nella scheda Ampiezza 1D (ampiezza della fluorescenza rispetto al numero dell'evento) per separare le goccioline negative e positive per ciascun canale, utilizzando l'NTC come guida ed esportare i dati in un file CSV.
- Per calcolare la concentrazione del vettore, calcolare prima il fattore di correzione CF usando la formula:
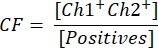
La CF determina la proporzione di goccioline positive per il transgene [Positives] che sono positive sia per il transgene che per l'ITR [Ch1+ Ch2+], per garantire la rilevazione di particelle vettoriali funzionali. La concentrazione vettoriale finale c può ora essere calcolata usando la seguente equazione: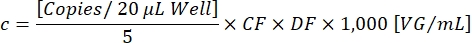
DF è il fattore di diluizione (1:105-1:107 come determinato in precedenza). Le copie per reazione a 20 μL/pozzetto corrispondono a 5 μL del campione diluito. Il fattore 1.000 corregge la scala in VG/mL (genoma virale/mL). Un risultato esemplare della titolazione è dimostrato nella Tabella 1 e nella Figura 3.
- Diluire in serie 2 μL del materiale madre vettoriale AAV in 198 μL di acqua priva di nucleasi per ottenere una diluizione finale 1:106 . Miscelare accuratamente ogni volta utilizzando una pipetta da 200 μL. Aggiungere un controllo senza modello (NTC) come controllo negativo.
- Analisi della libreria virale AAV mediante NGS
- Amplificare il frammento di inserzione del peptide a 96 nucleotidi impostando una reazione PCR da 20 μL utilizzando un kit di correzione di bozze della polimerasi (2x; vedere Figura 4). Aggiungere 1 μL di AAV contenente 1 x 108 vg, 0,5 μL di ciascuno dei 100 μM di primer (NGS_forward e NGS_reverse) e 10 μL della miscela enzimatica alla reazione. Regolare il volume finale a 20 μL con acqua priva di nucleasi.
- Trasferire la reazione a un termociclatore ed eseguire una fase di denaturazione per 3 minuti a 98 °C, seguita da 30-35 cicli di 10 s a 98 °C, 10 s a 59 °C e 20 s a 72 °C, seguiti da 5 minuti a 72 °C e una fase di raffreddamento finale.
- Purificare i campioni utilizzando un kit di purificazione PCR. Quantificare la concentrazione in uno spettrofotometro ed eseguire un gel di agarosio al 3% per verificare la purezza e la dimensione del frammento.
- Elaborare i frammenti PCR utilizzando il sistema di librerie per il kit di campioni a bassa complessità secondo le istruzioni del produttore per la preparazione di una libreria NGS. Eseguire la reazione di riparazione finale con 30 ng di frammento di PCR, seguita da legatura dell'adattatore e amplificazione PCR per 10 cicli. Utilizzare il kit di purificazione PCR per la purificazione delle reazioni.
- Elaborare i prodotti finali su un Bioanalyzer per verificarne le dimensioni e la purezza, utilizzando un kit di reagenti del DNA secondo le istruzioni del produttore.
- Quantificare gli ampliconi utilizzando un fluorometro e raggrupparli. Quantificare nuovamente la libreria NGS aggregata finale su un fluorometro (secondo le istruzioni del produttore) e verificare la qualità su un bioanalizzatore.
- Sequenziare le librerie NGS in modalità single-end (SE), utilizzando un kit ad alto output di 75 cicli, con una lunghezza di lettura di 84 e un indice 1 di 8.
NOTA: La sequenza degli esempi in questo articolo è stata eseguita presso la struttura GeneCore di EMBL Heidelberg (http://www.genecore.embl.de/). - Analizza i dati di sequenziamento NGS con Python 3 e biopython. I file sono disponibili all'indirizzo https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (in alternativa su https://doi.org/10.5281/zenodo.7032215). L'analisi NGS è composta da due fasi.
- Nella prima fase, cercare nei file di sequenza le sequenze che soddisfano determinati criteri (presenza di sequenze di riconoscimento che fiancheggiano il sito di inserimento) (vedere Figura 4, passaggio 1.9.8.5.). Questo viene fatto utilizzando uno script (Script # 1) e un file di configurazione che fornisce le informazioni necessarie. Una volta identificata la sequenza corretta, il programma estrae e memorizza la sequenza nel file di output, che è un file txt con lo stesso nome del file di sequenziamento.
- Il secondo passo è l'analisi dei file di output. Le sequenze nella libreria iniziano con uno qualsiasi dei sei nucleotidi (AGWggc, W = A / T) nell'inserto di nove aminoacidi. Sulla base di questa sequenza iniziale, il peptide viene tradotto. Questo genera i file di output che contengono le varianti peptidiche (PV).
- Preparare due cartelle: Script e Dati. Nella cartella Dati, copiare i file compressi gzip risultanti dalla sequenziazione. Nella cartella Script, copiare i seguenti file, file Python: Script#1_DetectionExtraction_JoVE_Py3.py; File Python: Script#2_PV_extraction_and_ranking_Py3.py; File di configurazione: Barcode_Script_JoVE.conf; e file della tabella di ricerca (LUT): Zuordnung.txt.
- Prima di eseguire gli script, modificare i seguenti file nella cartella script. Apri il file "Zuordnung.txt" e aggiungi due colonne separate da tabulazioni, i nomi dei file gzip (colonna 1) e il nome finale desiderato (colonna 2; valori separati da tabulazioni).
Nota : i file txt di esempio si trovano nella cartella GitHub "PV_analysis_script". I file forniti nella cartella GitHub vengono preparati per l'analisi di tre dati di esempio dalla libreria precedente: xaa.txt.gz, xab.txt.gz e xac.txt.gz. Vengono forniti anche i file di output. - Modificare le seguenti variabili nel file di configurazione "Barcode_Script_JoVE.conf":
my_dir = "~/Data/"
filename_sample_file = "~/Script/Zuordnung.txt"
Le variabili specifiche della sequenza: BCV_size = 27, BCVleft = TCCAGGGCCAG, BCVright = GCCCAGG, BCVloc = 30, BCVmargin = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC e BCVloc_revcomp = 41 (vedere la Figura 4 per i dettagli). - Utilizzare il comando seguente per chiamare il rilevamento e l'estrazione della sequenza di varianti:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
NOTA: L'output sono file txt con le sequenze di DNA estratte e il loro numero di letture. L'intestazione di questo file contiene dati statistici (cioè il numero totale di letture e le letture estratte). Questi dati vengono trasferiti ai file successivi. Questi dati txt sono i file di input per Script#2, in cui le sequenze di DNA vengono tradotte, classificate e analizzate. - Eseguire l'estrazione e l'analisi FV utilizzando il seguente comando:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Analizza i file di output di testo di Script # 2. I file di output di Script#2 sono denominati utilizzando la seconda colonna della LUT in "Zuordnung.txt" con estensioni basate sul tipo di analisi.
NOTA: assicurarsi che i tre file di output contengano dati statistici nelle prime righe ("# di letture PV valide", "# di letture PV non valide" e "# di letture PV univoche"), una prima colonna con l'indice di ogni sequenza di DNA dai file txt di input (output di Script # 1) e le seguenti colonne: (1) "... analyzed_all.csv": "Campione:" (sequenza del DNA), "#" (numero di letture), "Frw o Rev" (lettura diretta o inversa) e "PVs" (sequenza peptidica tradotta). Le sequenze non valide hanno "NA" e "not valid" nelle ultime due colonne. (2) "... analyzed_validSeq.csv": uguale al file precedente, filtrato per sequenze valide. (3) "... analyzed_PV.csv": "PVs" (sequenza peptidica tradotta), "#" (numero di letture) e "count" (i conteggi frw e rev nei file precedenti vengono uniti e il conteggio viene dato 1 o 2). - Visualizza i file di output utilizzando il software disponibile in base alle esigenze dell'utente.
2. Selezione della libreria di visualizzazione del peptide casuale 7-mer AAV2
- Utilizzare la libreria AAV dopo la quantificazione e il controllo di qualità (sezione 1) per l'evoluzione diretta in un modello di scelta per selezionare iterativamente i candidati con le proprietà desiderate (vedi Figura 5)16,18,21.
NOTA: Questi candidati vengono quindi utilizzati per la generazione di una libreria di codici a barre come descritto di seguito nella sezione 3.
3. Preparazione e analisi della libreria di capsidi AAV con codice a barre
NOTA: A seguito dell'identificazione di una serie di capsidi AAV potenzialmente specifici ed efficienti nella schermata di visualizzazione del peptide, verificare la funzionalità delle sequenze peptidiche identificate e confrontarle con una serie di varianti di capside AAV di riferimento comunemente usate o ben descritte. A tale scopo, la sequenza capside viene inserita in un costrutto helper Rep/Cap senza ITR.
- Produzione di librerie AAV con codice a barre
- Eseguire la produzione di AAV ricombinante per ciascuna variante di capside utilizzando il sistema a tre plasmidi, come descritto in precedenza24.
NOTA: Per distinguere le diverse varianti di capside, il plasmide transgene reporter affiancato da ITR contiene un codice a barre univoco di 15 nucleotidi di lunghezza. Il codice a barre si trova al 3' UTR (regione non tradotta) tra la proteina fluorescente gialla potenziata (EYFP) e il segnale polyA (vedi Figura 6A). L'espressione di EYFP è guidata da un forte promotore ubiquitario del citomegalovirus (CMV) che fornisce livelli sufficienti di trascritti di RNA. - Progettare codici a barre di 15 nucleotidi di lunghezza con omopolimeri di meno di tre nucleotidi, contenuto di GC di <65%29 e una distanza di Hamming superiore a quattro nucleotidi24.
- Produrre ciascun capside separatamente in combinazione con un plasmide transgene che trasporta un codice a barre univoco. In questo modo, ogni variante di capside è contrassegnata con un codice a barre distinto che ne consente il tracciamento specifico (vedere la Figura 6B).
- Eseguire la produzione di AAV ricombinante per ciascuna variante di capside utilizzando il sistema a tre plasmidi, come descritto in precedenza24.
- Titolazione vettoriale AAV mediante dd-PCR
- Eseguire la titolazione AAV come descritto in precedenza nella sezione 1.8, sostituendo la coppia di primer Rep2 con la coppia di primer YFP.
- Quantificare le singole produzioni AAV e raggruppare quantità uguali di ciascuna produzione per generare la libreria finale di codici a barre.
- Quantificare nuovamente la libreria finale per verificare la concentrazione e la qualità finali (vedere Figura 7).
- Libreria AAV con codice a barre in vivo applicazione
- Applicare sistematicamente la libreria AAV con codice a barre al sistema modello scelto (ad esempio sistematicamente nei topi24).
- Raccogliere tessuti ON- e OFF-target (cioè fegato, polmone, cuore, diaframma, muscolatura liscia, duodeno, pancreas, colon, bicipiti, ovaie, stomaco, orecchio interno, rene, aorta addominale, aorta toracica, cervello, grasso bruno e bianco e milza) o tipi di cellule in base all'esperimento. Congelarli a -80 °C, estrarre il DNA/RNA e applicare l'analisi di quantificazione NGS, come descritto nella sezione successiva.
- Estrazione DNA/RNA
- Estrarre il DNA e l'RNA dai tessuti di interesse utilizzando il Mini Kit DNA/RNA.
- Posizionare un piccolo pezzo del tessuto di interesse (1 mm3, circa 5 mg) in una provetta di reazione da 2 ml.
- Aggiungere 350 μL di tampone di lisi miscelato con β-mercaptoetanolo (1%) e sfere di acciaio da 5 mm al tessuto (maneggiare campioni con β-mercaptoetanolo sotto una cappa aspirante).
- Omogeneizzare il tessuto in un tissueLyser per 45 s a 40 Hz.
- Aggiungere 10 μL di proteinasi K (10 mg/ml) e incubare per 15 minuti a 55 °C agitando a 400 giri/min.
- Centrifugare a 20.000 x g per 3 minuti a temperatura ambiente, raccogliere il surnatante e procedere con il protocollo del produttore del Kit DNA/RNA.
- Dividere la fase di lavaggio in due fasi con 350 μL di tampone di lavaggio in ogni fase. Tra queste fasi di lavaggio, digerire il DNA residuo sulla colonna con DNasi I. Aggiungere 80 μL della soluzione di DNasi I, preparata secondo le istruzioni del produttore, sulla colonna e incubare a temperatura ambiente per 15 minuti.
- Eluire RNA/DNA dalla colonna con acqua priva di nucleasi. Conservare l'RNA isolato a -80 °C e il gDNA a -20 °C.
- Sintesi del cDNA
- Sottoporre i campioni di RNA ad un altro ciclo di trattamento con DNasi I di 15-30 minuti (per la completa rimozione del DNA contaminante dai campioni di RNA) prima della reazione di trascrizione inversa. Aggiungere 1 μL della soluzione di DNasi I, 4 μL di tampone (fornito con il kit) e acqua priva di nucleasi ad un volume finale di 40 μL a 212 ng di RNA. Incubare per 30 minuti a temperatura ambiente e inattivare a 70 °C per 10 min.
- Sintetizzare il cDNA, utilizzando 150 ng di RNA utilizzando un kit secondo le istruzioni del produttore. Includere controlli senza trascrittasi inversa, per garantire l'assenza di DNA virale contaminante dal campione. Il cDNA è immagazzinato a -20 °C.
NOTA: La quantità di RNA di input per una trascrizione inversa ottimale può variare a seconda del tipo di tessuto e dell'efficienza di trasduzione attesa nel rispettivo tessuto.
- Analisi della libreria virale AAV (in-vivo) di NGS
- Per ottenere un'elevata profondità di sequenziamento a basso costo, eseguire NGS tramite sequenziamento Illumina come descritto in precedenza (sezione 1.9). Amplificare la sequenza di codici a barre, quindi collegare gli adattatori di sequenziamento all'amplicone.
- A causa della lunghezza di lettura ridotta e della legatura degli adattatori di sequenziamento su entrambi i lati dell'amplicone, durante la progettazione, verificare che l'amplicone sia sufficientemente piccolo da garantire la presenza della sequenza di codici a barre all'interno della lettura NGS. Per il sequenziamento dei codici a barre all'interno dei genomi virali e delle trascrizioni virali, l'amplicone PCR è progettato per essere lungo 113 bp (vedi Figura 8).
- Amplifica la regione con codice a barre con i primer BC-seq in avanti e BC-seq al contrario. Preparare la seguente reazione PCR: 0,5 μL di DNA polimerasi ad alta fedeltà, 10 μL di tampone 5x, 0,25 μL di ciascun primer da 100 μM (BC-seq fw/BC-seq rv) e 1 μl di 10 mM dNTPs. Utilizzare 25 ng di cDNA o DNA/reazione come modello e regolare il volume finale a 50 μL con acqua priva di nucleasi.
- Preparare il master-mix PCR sotto un cappuccio PCR pulito per evitare la contaminazione. Utilizzare le seguenti condizioni di ciclismo: 30 s a 98 °C, seguiti da 40 cicli a 98 °C per 10 s e 72 °C per 20 s e un ultimo passo di 5 minuti a 72 °C.
- Includere controlli PCR per confermare l'assenza di DNA contaminante nel master-mix PCR. Per i campioni di cDNA, includere i controlli senza trascrittasi inversa. Infine, includere un esempio con la libreria di input AAV. Queste informazioni verranno utilizzate per generare il file Normalization_Variant.txt utilizzato nell'analisi.
- Verificare la dimensione del frammento PCR di ciascun campione mediante elettroforesi su gel prima della purificazione PCR. Quest'ultimo è ottenuto utilizzando perle magnetiche disponibili in commercio o sistemi di purificazione del DNA basati su colonne (vedi Tabella dei materiali).
- Preparare la libreria NGS utilizzando il sistema di librerie per campioni a bassa complessità secondo le istruzioni del produttore, come descritto in precedenza nella sezione 1.9.
- Determinare la concentrazione di DNA tramite il kit HS dsDNA e analizzare la qualità della libreria come descritto in precedenza (sezione 1.9.6), seguito dal pooling. Quantificare la libreria aggregata su un fluorometro e valutare la qualità su un bioanalizzatore.
- Eseguire il sequenziamento NGS come discusso nella sezione 1.9.7.
- Quantificare mediante qPCR il numero di copie del transgene (genomi virali) e del gene housekeeping per valutare la distribuzione della libreria aggregata tra tessuti o organi sul DNA.
- Impostare una reazione qPCR da 30 μL come segue, per determinare il numero di copie di EYFP (transgene) e GAPDH (gliceraldeide-3-fosfato deidrogenasi, gene housekeeping):
- Preparare una miscela primer/sonda 60x per EYFP (1,5 μM YFP_fw, 1,5 μM YFP_rv e 0,6 μM YFP_probe; vedere Tabella dei materiali). Utilizzare la miscela primer/sonda GAPDH (vedere la tabella dei materiali) per determinare il numero di copie del gene della governante. Imposta la reazione sul ghiaccio.
- Preparare una miscela master PCR (15 μL, vedere Tabella dei materiali) e aggiungere 60x miscela primer/sonda (0,5 μL) per tutti i campioni e gli standard (per calcolare il numero di copie per gli standard, utilizzare il seguente link: http://cels.uri.edu/gsc/cndna.html). Imposta la reazione sul ghiaccio.
- Trasferire 15,5 μL della miscela principale in una piastra da 96 pozzetti e aggiungere 14,5 μL di campione (75 ng di concentrazione totale di DNA) o standard al rispettivo pozzetto. Sigillare brevemente la piastra a 96 pozzetti con un foglio, un vortice e una rotazione.
- Trasferire 10 μL di ciascun campione in una piastra da 384 pozzetti in duplicati. Sigillare la piastra con un foglio e centrifugare a 800 x g per 5 minuti a 4 °C.
- Incubare la miscela di reazione in un termociclatore utilizzando una temperatura iniziale di 50 °C per 2 minuti, seguita da una fase iniziale di attivazione di 10 minuti a 95 °C. Eseguire 40 cicli di denaturazione a 95 °C per 15 s e ricottura/estensione a 60 °C per 1 min24.
- Per ottenere il numero di genomi diploidi (dg), utilizzare il numero di copia GAPDH e dividere per due. Quindi, prendi il valore del numero di copia dell'EYFP e dividi per il numero di dg, ottenendo genomi vettoriali per genoma diploide (vg / dg). Utilizzare questo valore per generare il file di Normalization_Organ.txt per l'analisi bioinformatica.
- Eseguire l'analisi dei dati di sequenziamento NGS come Weinmann et al.24, usando codice personalizzato in Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Il flusso di lavoro comprende il rilevamento di sequenze di codici a barre guidate da sequenze di fiancheggiamento, la loro lunghezza e posizione (Script # 1_BarcodeDetection.py), nonché l'analisi dell'arricchimento e della distribuzione del codice a barre sul set di tessuti (Script # 2_BarcodeAnalysis.py).
- Rileva i codici a barre e assegnali alle varianti AAV. Posizionare i dati di sequenziamento come file fastq archiviati in una directory (ad esempio, "Data_to_analyze"). Il file di dati di sequenziazione per la libreria di input è incluso in questa directory e utilizzato solo per calcolare le proporzioni del capside nella libreria di input.
- Prima di eseguire lo script, creare due file di testo delimitati da tabulazioni: il file delle varianti del capside (vedere il file di esempio "Varianti.txt") con le sequenze di codici a barre assegnate ai nomi delle varianti del capside AAV e il file di contaminazione (vedere "Contaminazioni.txt") con sequenze di codici a barre che provengono da una possibile contaminazione (altri codici a barre disponibili in laboratorio, che contribuiscono alla contaminazione).
- Infine, modificare il file di configurazione "Barcode_Script.conf" per includere le seguenti informazioni: percorso della cartella con i dati di sequenziamento (ad esempio, "Data_to_analyze"), sequenza delle regioni laterali dei codici a barre, loro posizione e dimensione della finestra per il rilevamento dei codici a barre (simile a 1.9.8.5, vedere Figura 8).
- Utilizzare il seguente comando per chiamare il rilevamento dei codici a barre con i percorsi forniti a Script#1_BarcodeDetection.py e ai file di configurazione:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
NOTA: l'output dell'esecuzione di Script#1_BarcodeDetection.py è costituito da file di testo con conteggi di lettura per variante di capside e dal numero totale di letture recuperate dai dati non elaborati. - Valutare la distribuzione dei capsidi AAV con codice a barre tra tessuti o organi, eseguendo Script#2_BarcodeAnalysis.py insieme ai seguenti file txt:
- Nel file "Zuordnung.txt", assegnare il nome a ciascun file txt ottenuto dall'esecuzione di rilevamento del codice a barre a un nome di tessuto/organo: nomi di file txt nella prima colonna e nomi di tessuti/organi corrispondenti in assegnazione delimitata da tabulazioni.
NOTA: per un esempio, controllare nella cartella "Esempio" (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Da notare che il nome del tessuto/organo può includere caratteri che definiscono la misurazione del cDNA o del gDNA e il numero di replica biologica (M1, M2, ecc.). - Creare un file di testo "organi.txt" con l'elenco dei nomi per gli organi ON e OFF-target, che corrispondono ai nomi indicati nel file di assegnazione "Zuordnung.txt" (vedere la cartella "Esempio": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- Creare file di testo delimitati da tabulazioni "Normalization_Organ.txt" e "Normalization_Variant.txt" con valori normalizzati per tutte le varianti di capside e tutti gli organi/tessuti. Nella prima colonna del file "Normalization_Organ.txt", scrivere i nomi indicati per ciascun organo (come nel file di assegnazione "Zuordnung.txt") e nella seconda colonna i valori di normalizzazione per i tessuti corrispondenti, generati nella sezione 3.6.11.
- Riempire la prima colonna del file "Normalization_Variant.txt" con l'elenco dei nomi dei capside e la seconda colonna con i valori normalizzati dei conteggi di lettura per ogni capside nella libreria in pool (la normalizzazione può essere calcolata in base al file di output txt per la libreria di input risultante dal primo script).
- Modificare il file di configurazione specificando i percorsi completi di tutti i file aggiuntivi sopra menzionati. Execute Script#2_BarcodeAnalysis.py come:
>python3 /script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
NOTA: lo script di analisi dei codici a barre produce diversi file: file di testo con valori di concentrazione relativa (RC) della distribuzione del capside all'interno di diversi tessuti in base a più passaggi di normalizzazione descritti in precedenza e il file di foglio di calcolo che combina i dati del file di testo nei dati della matrice uniti. Quest'ultimo può essere utilizzato per l'analisi e la visualizzazione dei cluster. - Visualizza i dati ed esegui l'analisi dei cluster dei dati della matrice al fine di distinguere le proprietà del capside e valutare le loro somiglianze in base ai profili RC tra i tessuti. Utilizzare lo script aggiuntivo PCA_heatmap_plot. R inserito nel repository:
>Rscript --vanilla ~/PCA. R ~/concentrazione relativa.xls
NOTA: lo script accetta i file relativeconcentration.xls come input e genera due grafici di mappa di calore del cluster gerarchico e analisi dei componenti principali (PCA). - Per modificare i grafici (assi della mappa di calore, componenti principali della PCA) o i parametri png (colore, dimensione, etichettatura), aprire lo script R e seguire le istruzioni fornite nelle sezioni commentate.
- Nel file "Zuordnung.txt", assegnare il nome a ciascun file txt ottenuto dall'esecuzione di rilevamento del codice a barre a un nome di tessuto/organo: nomi di file txt nella prima colonna e nomi di tessuti/organi corrispondenti in assegnazione delimitata da tabulazioni.
Risultati
Generazione di una libreria di visualizzazione del peptide AAV2. Come primo passo verso la selezione di AAV ingegnerizzati, viene descritta la generazione di una libreria di plasmidi. L'inserto peptidico viene prodotto utilizzando primer degenerati. Ridurre la combinazione di codoni in quelli da 64 a 20 ha il vantaggio di eliminare i codoni di stop e facilitare l'analisi NGS, riducendo la diversità della libreria sul DNA ma non il livello proteico. L'inserto oligonucleotidico viene acquistato come DNA ...
Discussione
In questo protocollo, vengono delineati i passaggi necessari per l'ingegneria del capside AAV di visualizzazione del peptide e per lo screening della libreria AAV con codice a barre, nonché per l'analisi bioinformatica della composizione della libreria e delle prestazioni del capside. Questo protocollo si concentra sui passaggi che facilitano l'analisi bioinformatica di questi tipi di librerie, perché la maggior parte dei laboratori di virologia sono in ritardo nelle capacità di programmazione per abbinare la loro com...
Divulgazioni
D.G. è co-fondatore di AaviGen GmbH. D.G. e K.R. sono inventori di una domanda di brevetto pendente relativa alla generazione di varianti di capside AAV che eludono il sistema immunitario. Il resto degli autori non ha nulla da rivelare.
Riconoscimenti
D.G. apprezza molto il sostegno della Fondazione tedesca per la ricerca (DFG) attraverso i centri di ricerca collaborativa DFG SFB1129 (Projektnummer 240245660) e TRR179 (Projektnummer 272983813), nonché del Centro tedesco per la ricerca sulle infezioni (DZIF, BMBF; TTU-HIV 04.819).
Materiali
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
Riferimenti
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon