È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Produzione e ottimizzazione di LTE, un sistema di espressione proteica libera derivato da Leishmania tarantolae per la produzione di proteine ricombinanti
In questo articolo
Riepilogo
L'estratto traslazionale di Leishmania (LTE) è un sistema di espressione proteica eucariotico privo di cellule derivato dal parassita unicellulare Leishmania tarentolae. Questo protocollo ottimizzato rende l'LTE semplice ed economico da produrre. È adatto per varie applicazioni incentrate sull'espressione multiparallela e sullo studio di proteine eucariotiche complesse e delle loro interazioni.
Abstract
Questo protocollo delinea la produzione e l'ottimizzazione di un sistema di espressione proteica eucariotica (CFPS) derivato dal flagellato unicellulare Leishmania tarentolae, denominato Leishmania Translational Extract o LTE. Sebbene questo organismo si sia originariamente evoluto come parassita dei gechi, può essere coltivato facilmente ed economicamente in fiasche o bioreattori. A differenza della Leishmania major, non è patogena per l'uomo e non richiede particolari precauzioni di laboratorio. Un altro vantaggio dell'utilizzo di Leishmania per la CFPS è che l'aggiunta di un singolo oligonucleotide antisenso alla CFPS, mirata a una sequenza leader di splicing conservata sull'estremità 5' di tutti gli RNA codificanti proteine, può sopprimere l'espressione proteica endogena. Forniamo procedure per la disgregazione cellulare e il trattamento del lisato, che sono state semplificate e migliorate rispetto alle versioni precedenti. Queste procedure iniziano con semplici colture in fiaschetta. Inoltre, spieghiamo come introdurre informazioni genetiche utilizzando vettori contenenti siti di inizio della traduzione specie-indipendenti (SITS) e come eseguire l'ottimizzazione dei lotti e il controllo di qualità per garantire una qualità costante dell'espressione proteica.
Introduzione
Negli anni '60, i sistemi di espressione proteica libera da cellule hanno svolto un ruolo fondamentale nella scoperta del codice genetico1. Tuttavia, i sistemi di espressione proteica liberi da cellule procariotiche, basati principalmente su E. coli, attualmente dominano sia le applicazioni di laboratorio che quelle commerciali. Sebbene i sistemi basati su E. coli offrano vantaggi quali economicità, scalabilità e alte rese di espressione, devono affrontare sfide quando producono proteine multidominio nelle loro forme attive e facilitano l'assemblaggio di complessi proteici 2,3. Al giorno d'oggi, le forme comunemente usate di sintesi proteica eucariotica libera da cellule (CFPS) includono l'estratto di germe di grano (WGE), il lisato di reticolociti di coniglio (RRL) e il lisato di cellule di insetto (ICL)4,5,6. Questo lavoro introduce un sistema alternativo privo di cellule eucariotiche che è sia semplice che scalabile, basato sul parassita flagellato unicellulare Leishmania tarentolae.
Leishmania tarentolae può essere coltivata facilmente in fiasche utilizzando terreni economici e può anche essere ingrandita in bioreattori per ottenere una maggiore densità cellulare. La presenza di mRNA endogeni nel lisato cellulare, che potrebbero altrimenti competere con i messaggi introdotti, può essere neutralizzata utilizzando oligonucleotidi antisenso che hanno come bersaglio la sequenza7 conservata dello splicing dell'mRNA di Leishmania. A differenza del suo parente stretto Leishmania major, che causa malattie umane, L. tarentolae infetta il geco moresco (Tarentolae mauritanica), rendendolo adatto alla coltivazione in ambienti di laboratorio PC2 senza la necessità di particolari precauzioni. È stato precedentemente utilizzato come organismo transgenico per l'espressione proteica in vivo8.
Per facilitare il priming del modello nei sistemi privi di cellule, sono state progettate sequenze universali basate su strutture polimeriche di RNA che migliorano l'inizio traduzionale9. Queste sequenze di traduzione specie-indipendenti (SITS) sono applicabili sia ai sistemi procariotici che eucariotici e sono adatte per introdurre informazioni genetiche nell'LTE. Sebbene questo protocollo non fornisca una spiegazione dettagliata della costruzione del vettore per l'espressione proteica libera da cellule LTE, l'ottimizzazione e il controllo di qualità richiedono vettori adeguati contenenti fusioni di fluorofori delle proteine desiderate di interesse a valle del sito SITS. A tale scopo, sono stati depositati appropriati vettori LTE nel repository genico Addgene, come il vettore pCellFree_G03, che codifica una fusione N-terminale di eGFP con la proteina desiderata di interesse utilizzando i siti di clonazione Gateway.
L'LTE ha dimostrato il suo valore in un'ampia gamma di applicazioni che richiedono l'espressione di proteine, tra cui l'analisi dell'autoassemblaggio delle proteine10,16, la produzione di proteine di membrana integrali umane17, la ricerca su candidati farmaci antivirali18, lo sviluppo di enzimi biotecnologicamente utili19, la prototipazione di biosensori proteici20,21 e lo studio di farmaci biologici da anchilostomi22. L'LTE è stato anche determinante nella mappatura delle reti di interazione proteina-proteina nei campi della virologia e delle strutture cellulari21,32. L'LTE è stato confrontato per funzionare in modo simile ad altri sistemi privi di cellule eucariotiche nell'esprimere proteine a lunghezza intera, monodisperse e non aggregate33, il tutto offrendo una produzione più economica e scalabile.
Questo protocollo fornisce tecniche per coltivare e distruggere l'organismo ospite, preparare il lisato e integrare una soluzione di alimentazione (FS) per l'espressione proteica accoppiata di trascrizione/traduzione. Inoltre, include un protocollo per l'ottimizzazione dei lotti di produzione. Nella versione iniziale del sistema cell-free Leishmania, è stata osservata una variazione indesiderata da lotto a lotto nei livelli di espressione, nella frazione di proteine a lunghezza intera e nella presenza di aggregati proteici, portando allo smaltimento dei lotti34. Per risolvere questo problema sono stati apportati successivi miglioramenti al protocollo25. L'attuale protocollo si basa su questi miglioramenti, consentendo di ottimizzare i singoli lotti per il picco di espressione e dimensione delle proteine. Raggiunge questo obiettivo controllando attentamente il carico del disgregatore cellulare (misurato come densità ottica a 600 nm; OD600nm) e normalizzando l'output di lisato risultante utilizzando l'assorbanza a 280 nm (Abs280nm). Inoltre, incorpora un metodo per integrare parzialmente il lisato con rNTP e magnesio durante la produzione, con successiva ottimizzazione di questi componenti della soluzione alimentare durante le espressioni di prova. Sebbene questa ottimizzazione sia presentata come un'opzione nel protocollo, è fortemente raccomandata dagli autori.
Protocollo
Questo protocollo include ricette dettagliate e passaggi dei terreni che coinvolgono la coltura, la centrifugazione, la misurazione della fluorescenza GFP utilizzando un lettore di piastre multimodale, la misurazione della coltura OD600 nm e la valutazione del lisato Abs280 nm. Copre anche la configurazione e l'imaging dei gel proteici SDS-PAGE. I materiali richiesti o suggeriti per questo protocollo sono elencati nel foglio di calcolo dei materiali. È importante notare che le tipiche risorse di laboratorio come componenti di terreni, centrifughe, provette, spettrofotometri e configurazioni di elettroforesi su gel possono probabilmente essere utilizzate in modo intercambiabile, se non diversamente specificato. La Figura 1 fornisce un riepilogo del processo di produzione LTE.
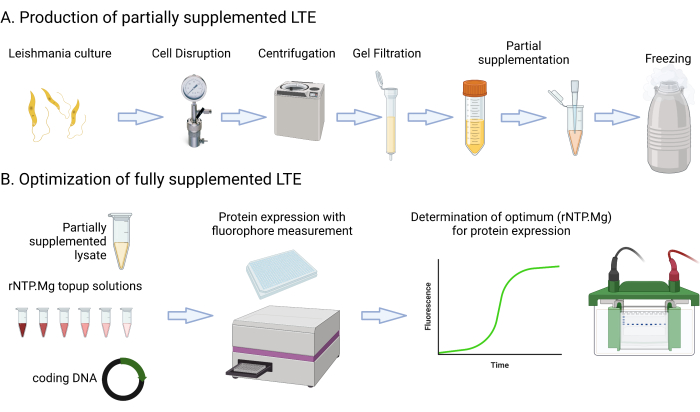
Figura 1: Panoramica del protocollo di produzione LTE. Questo cartone animato fornisce un breve riassunto del protocollo di produzione LTE. Clicca qui per visualizzare una versione più grande di questa figura.
1. Crescita delle colture di Leishmania tarentolae
- Preparare almeno 3 L di terreno di coltura TBGG (Bactotriptone 12 g/L, Estratto di lievito 24 g/L, Glicerolo 8 mL/L, Glucosio 1 g/L, KH2PO4 2,3 g/L, K2HPO4 2,5 g/L, vedere la Tabella dei materiali). Sterilizzare il terreno utilizzando un filtro da 0,22 μm sotto vuoto o una configurazione simile.
- Conservare il terreno a temperatura ambiente (RT), con le aggiunte finali (Hemin, antibiotici) aggiunte appena prima dell'inoculazione con L. tarentolae. L'emina (0,25% v/v in 50% trietanolammina) viene aggiunta allo 0,2% v/v, la penicillina (10.000 unità/mL) più la streptomicina (10.000 μg/mL) si mescolano allo 0,5% v/v.
NOTA: Il punto di partenza di questo protocollo è una coltura mantenuta di 2 x 10 mL di L. tarentolae wild type. Le colture di mantenimento vengono coltivate a 27 °C in fiasche standard da 50 mL per colture tissutali con bassa agitazione (75 giri/min). Tali colture da 10 mL possono essere mantenute indefinitamente con ~1/20 diluizioni in sterili (TBGG + emina, penicillina, streptomicina) ogni 2-3 giorni. Si consiglia una cabina di biosicurezza standard in un laboratorio PC2; tuttavia, le contaminazioni batteriche tendono ad essere prevenute dagli antibiotici aggiunti, mentre le contaminazioni fungine sono generalmente superate da L. tarentolae. - Nell'arco di due giorni, espandere le colture di mantenimento di L. tarentolae a 200 ml (giorno 1) e poi a 2 litri (giorno 2) tramite diluizioni 1:10 con un volume crescente di TBGG + emina/antibiotici ogni giorno. Eseguire entrambe le diluizioni in palloni di vetro deflettori da 5 L sterilizzati in autoclave (riempiti fino a un massimo di 1 L). La seconda diluizione deve avvenire nel pomeriggio tra le 15 e le 18, con l'intenzione di iniziare la produzione di lisato il giorno successivo tra le 8 e le 11.
NOTA: questo protocollo utilizza il volume iniziale minimo per la produzione di LTE (2 colture da 1 L). È anche possibile espandere la coltura fino a 10 L per la produzione di LTE incorporando un'ulteriore fase di espansione (ad esempio, Giorno 1: 100 mL; Giorno 2: 1 L; Giorno 3: 10 L). Sebbene questo protocollo utilizzi palloni deflettori (vedi Tabella dei materiali) per coltivare L. tarentolae, opzionalmente, possono essere utilizzati bioreattori convenzionali progettati per la crescita batterica con giranti Rushton, a condizione che la velocità di agitazione sia mantenuta al di sotto di 100 giri/min. Il miglioramento dell'aerazione e del controllo del pH nei bioreattori generalmente prolunga la crescita in fase logaritmica delle colture di L. tarentolae , consentendo di utilizzare un raccolto più elevato di600 nm di 10 da utilizzare nella fase 1.4. - Registrare l'OD600nm della coltura in triplicato tramite una diluizione 1:10 in TBGG direttamente nella cuvetta dello spettrofotometro. Un intervallo di partenza adatto per la produzione di lisato è OD600nm = 4.0-8.0.
- Fornire un tempo di incubazione aggiuntivo se OD600nm < 4.0. Una coltura con OD600nm > 8 è utilizzabile e si tradurrà in un volume maggiore di lisato di espressione cell-free, ma con una qualità inferiore a causa dell'inizio della fase di crescita tardiva. Posizionare i fiasche di coltura sul ghiaccio, in attesa dei passaggi successivi.
NOTA: La misurazione accurata della coltura finale OD600nm è fondamentale, in quanto viene utilizzata per calcolare il volume finale delle cellule concentrate prima dell'interruzione. Questo calcolo sostituisce un metodo di pesatura dei pellet utilizzato nelle versioni precedenti della produzione LTE per calibrare la concentrazione delle celle prima dell'interruzione34, al fine di semplificare il protocollo. Assicurarsi di diluire in TBGG per la misurazione OD600nm , altrimenti lo shock osmotico modifica la forma della cella, causando errori di misurazione. Pipettare la diluizione 1:10 per la misurazione OD600nm (direttamente nella cuvetta) pochi secondi prima di eseguire la lettura spettrofotometrica, poiché le cellule di L. tarentolae si depositano rapidamente con un caratteristico aspetto torbido. Se il volume finale della coltura di espressione è considerato approssimativo, si raccomanda anche di pesare i palloni al momento del raccolto (con tara vuota adeguata) per ottenere un volume stimato migliore (a 1 g = 1 mL). Il diametro esterno massimodi 600 nm possibile dalla crescita di L. tarentolae in fiasche deflette è 15-20, sebbene ciò non sia appropriato per la produzione di lisato a causa del raggiungimento della fase stazionaria.
2. Concentrazione delle colture di L. tarentolae
- Le cellule della Leishmania devono essere lavate e concentrate circa 60 volte prima della disgregazione. Calcolare il volume target per la concentrazione cellulare in base a OD600nm = 300 per il concentrato finale. L'equazione è V = volume del raccolto (mL) x (raccolto OD600nm/300). Ad esempio, utilizzando una coltura da 2 L con un raccolto OD600 = 5, il volume target è 33 mL.
NOTA: L'obiettivo OD600nm di 300 può essere modificato; la precedente produzione di LTE ha utilizzato valori compresi tra 150 e 350. Concentrazioni più elevate di cellule che vanno in disgregazione tenderanno a produrre reazioni finali di espressione libera da cellule con rese proteiche più elevate, ma con una maggiore tendenza delle proteine vulnerabili ad aggregarsi. OD600nm = 300 rappresenta un obiettivo predefinito adatto per la produzione LTE. - Trasferire i fermenti raccolti in apposite bottiglie da centrifuga e centrifugarli a 2500 x g per 10 minuti a 4 °C. Decantare con cura il surnatante nei rifiuti di coltura.
NOTA: È importante ridurre al minimo la perdita di cellule nel surnatante scartato, poiché influisce sul calcolo del carico di interruzione. Nei precedenti protocolli di produzione LTE, la concentrazione di cellule di L. tarentolae per l'interruzione è stata calibrata facendo girare il concentrato di cellule in una provetta per microcentrifuga di prova e misurando il peso del pellet rispetto al peso totale34. Questo protocollo semplificato utilizza invece un target teorico di OD600 nm per il concentrato, basato sul OD misurato del raccolto di600 nm, e presuppone una bassa perdita cellulare durante la concentrazione e il lavaggio delle cellule. - Lavare il pellet cellulare in tampone SEB (45 mM HEPES-KOH pH 7,6, 250 mM di saccarosio, 100 mM di KOAc, 3 mM di Mg(OAc)2, tenuto in ghiaccio) tre volte, centrifugando ogni volta a 2500 x g per 10 minuti a 4 °C. Per il primo lavaggio, risospendere ogni 1 litro di coltura pellettata in 100 ml di tampone SEB, quindi combinarli in un unico pallone da centrifuga. Per il secondo lavaggio, utilizzare anche 100 ml di SEB per ogni 1 litro di coltura originale.
NOTA: Per la risospensione finale del pellet, aggiungere il tampone SEB al 50% del volume di risospensione target finale (passaggio 2.1). Ciò consente di rabboccare con cura il concentrato raggruppato fino al volume target finale nella fase 2.4. Ogni risospensione deve essere il più delicata possibile per evitare la lisi prematura di L. tarentolae, ad esempio, facendo roteare delicatamente il SEB aggiunto attorno al pellet decantato o pipettando il SEB sul pellet che aderisce alla parete della provetta della centrifuga. Potrebbe essere più conveniente trasferire i surnatanti in provette da centrifuga più piccole per la fase finale. - Versare il concentrato risospeso in un cilindro volumetrico di vetro lavato adatto, quindi rabboccare il volume fino al volume target (passaggio 2.1) utilizzando un ulteriore SEB freddo e mescolare delicatamente.
3. Lisi del concentrato di L. tarentolae
- Trasferire il concentrato di cellule nel dispositivo di cavitazione dell'azoto (vedi Tabella dei materiali) preraffreddato a 4 °C, pressurizzarlo a 70 bar di azoto e incubare per 45 minuti su ghiaccio.
NOTA: Sebbene gli interferenti per cavitazione da azoto non siano comuni articoli di laboratorio, sono consigliati per la produzione di LTE. Sono stati provati metodi alternativi come il congelamento-scongelamento cellulare e gli interferenti di tipo French-press; Tuttavia, l'attività di espressione proteica era del <50% rispetto all'utilizzo del metodo di cavitazione dell'azoto. Il dispositivo di cavitazione dell'azoto deve essere pulito accuratamente prima dell'uso e tra una corsa e l'altra, in modo simile a tutti i recipienti riutilizzati che entrano in contatto con il lisato cellulare da questa fase in poi (come il pallone ricevente). Un regime di pulizia adeguato prevede il lavaggio con detergenti da laboratorio seguito da un accurato risciacquo con acqua deionizzata. - Aprire lo sfiato del dispositivo di cavitazione dell'azoto ed espellere il lisato risultante in un contenitore adeguatamente robusto, come un pallone ricevitore a vuoto su ghiaccio. Inclinare il pallone del ricevitore per assicurarsi che tutto il lisato risultante si depositi e possa essere pipettato in una nuova provetta da centrifuga o in un recipiente simile.
ATTENZIONE: Gli interferenti per cavitazione con azoto si basano sulla brusca transizione del concentrato di celle da 70 bar di azoto alla pressione ambiente, ottenuta attraverso un forte flusso di prima liquido e poi di azoto attraverso la valvola di uscita del dispositivo. Lo sfiato deve essere effettuato con adeguati dispositivi di protezione individuale (DPI) in una cappa di sicurezza chimica. C'è il rischio di rompere il recipiente di destinazione e di perdere il lisato, motivo per cui utilizziamo un robusto ricevitore del vuoto invece di un pallone generico. Se la valvola di uscita del dispositivo è un tubo, evitare di posizionare il tubo direttamente all'interno del ricevitore per evitare un eccessivo accumulo di pressione nel punto di sfiato.
4. Centrifugazione del lisato cellulare
- Trasferire il lisato in provette da centrifuga con forza g adeguate e centrifugare a 10.000 x g per 15 minuti a 4 °C. Rimuovere il surnatante in provette da centrifuga fresche e simili.
- Centrifugare il lisato a 30.000 x g per 15 minuti a 4 °C, quindi rimuovere il surnatante finale in una nuova provetta da centrifuga o in un contenitore simile posto su ghiaccio. Stimare il volume totale.
5. Filtrazione su gel del lisato cellulare
NOTA: La filtrazione su gel viene utilizzata per rimuovere il saccarosio incluso nel tampone SEB. Mentre il saccarosio aiuta a stabilizzare il macchinario cellulare durante la disgregazione cellulare, diminuisce la resa se trattenuto nelle reazioni di espressione proteica.
- Installare un numero sufficiente di colonne di filtrazione su gel alimentate a gravità PD-10 (vedere la Tabella dei materiali) in un formato rack che consenta loro di gocciolare in un vassoio di raccolta o in un contenitore simile sottostante, assicurando che possano filtrare l'intero volume del lisato a 2,5 ml per colonna. Pre-equilibrare le colonne facendo passare in anticipo 10 mL di tampone EB a 4 °C (45 mM HEPES-KOH pH 7,6, 100 mM KOAc, 3 mM Mg(OAc)2).
NOTA: Tutte le fasi da questo punto in poi traggono vantaggio dall'essere condotte in una cella frigorifera a 4 °C. Tuttavia, è adatto anche per conservare tutti i lisati e i reagenti su una vaschetta del ghiaccio da banco. Un'eccezione è la fase di filtrazione su gel, in cui gli autori posizionano un rack di colonne all'interno di un frigorifero a 4 °C durante il rebuffering. Nelle versioni originali di questo protocollo, le nuove colonne di filtrazione su gel venivano "bloccate" inizialmente tamponando il lisato e scartando il primo output. Sebbene ciò non sia ritenuto necessario, le colonne devono essere lavate con tampone EB e conservate a 4 °C tra i lotti di lisato. Il primo lisato di output può avere un'attività di espressione proteica inferiore rispetto agli output successivi a causa di una certa ritenzione di fondo dei componenti del lisato sulla nuova colonna. - Aggiungere 2,5 ml di lisato a ciascuna colonna e attendere che passi nella colonna. Aggiungere altri 0,5 mL di EB per depositare il lisato nella colonna mentre si scarta l'eluato.
- Eluire il lisato filtrato in gel aggiungendo altri 2,5 ml di EB a ciascuna colonna, raccogliendo l'output posizionando un vassoio fresco e pulito o un altro recipiente sotto le colonne.
6. Integrazione di lisato cellulare
- Utilizzare lo spettrofotometro nanodrop (vedere la tabella dei materiali) per misurare l'Abs280nm del lisato filtrato in gel. Se supera i 60, diluirlo per raggiungere Abs280nm = 60 utilizzando un ulteriore tampone EB a 4 °C.
NOTA: Mentre il controllo dell'input della densità cellulare nella rottura utilizzando OD600nm determina approssimativamente la forza di uscita del lisato, la normalizzazione dell'Abs280nm dopo la rottura e l'elaborazione del lisato migliora ulteriormente la coerenza del batch delle prestazioni del lisato. Il lisato Abs280nm può essere regolato su e giù, con conseguenze per la resa e l'aggregazione dell'espressione proteica (vedi sezione Discussione). Se il lisato non integrato indica un Abs280nm < 60, potrebbe essere necessario includere più biomassa Leishmania nella fase di disgregazione, cioè aumentare il carico del disgregatore cellulare aOD 600nm > 300 nel passaggio 2.1. - Aggiungere 5x Feeding Solution (5x FS, Tabella 1) al lisato in un rapporto 2:5 e mescolare accuratamente a vortice. Aliquotarlo in contenitori idonei (ad es. provette per microfuge da 1,5 mL) e congelarlo in azoto liquido. Se si seguono i passaggi facoltativi 7.1-7.3 per l'ottimizzazione delle espressioni LTE riportati di seguito, utilizzare il rNTP.Mg ridotto 5x FS della Tabella 1 invece del 5x FS predefinito. Includere 5 aliquote da 100 μl per il congelamento da utilizzare negli esperimenti di ottimizzazione.
NOTA: Il congelamento con il 5x FS predefinito in un rapporto 2:5 crea LTE integrato pronto per l'espressione, utilizzato a 7 μL/10 μL di espressione (quindi, il 5x FS diventa 1x FS nella reazione finale). Tuttavia, gli autori raccomandano di seguire gli ulteriori passaggi opzionali in cui 0,6 volte la quantità predefinita di rNTP e magnesio è fornita nel 5x FS. Questo è seguito da una fase di ottimizzazione in cui viene aggiunta una miscela equimolare dei due (denominata rNTP.Mg) per completare le reazioni di prova a un valore ottimizzato. Il parziale 5x FS contiene anche un oligonucleotide che interrompe l'espressione endogena dell'mRNA (vedi sezione Introduzione). La sequenza dell'oligonucleotide è CAATAAAGTACAGAAACTGATACTTATATAGCGTT.
7. Controllo qualità e ottimizzazione dell'LTE integrato finale
NOTA: I passaggi minimi necessari per determinare l'appropriata aggiunta di rNTP.Mg al rNTP ridotto e al lisato integrato con magnesio prevedono l'espressione di eGFP o di un fluoroforo simile (ad esempio, sfGFP) senza un partner di fusione. Concentrazioni crescenti di rNTP.Mg vengono aggiunte alle reazioni per determinare il punto in cui il livello di espressione (misurato come eGFP RFU tramite un lettore di piastre multimodale) viene ottimizzato. Le terminazioni premature di eGFP, che non sono fluorescenti, diventano evidenti diminuendo l'RFU di eGFP a concentrazioni di rNTP.Mg troppo elevate. Tuttavia, i malfunzionamenti a corto prodotto di LTE si verificano più frequentemente nelle proteine espresse più grandi (>50 kDa). Pertanto, è possibile eseguire questa ottimizzazione utilizzando un modello più grande di eGFP, soprattutto se è disponibile in un vettore di espressione adatto, fornendo una fusione di fluorofori che si desidera venga prodotta da LTE per una particolare applicazione o studio (vedere la sezione Risultati rappresentativi).
- Scongelare un'aliquota da 100 μL e impostare sei reazioni di espressione da 10 μL, ciascuna composta da 7 μL di lisato parzialmente integrato dalla fase 6.2, 1 μL di soluzione di rabbocco come da Tabella 2 e 2 μL di acqua ultrapura contenente una quantità sufficiente di DNA per raggiungere una concentrazione finale di 50 ng/μL nella reazione.
- Incubare le reazioni per 2 ore a 25 °C e monitorare l'aumento della fluorescenza della GFP utilizzando un lettore di piastre multimodale.
NOTA: I valori di configurazione adatti per GFP sono l'eccitazione a 485 nm (larghezza di banda 5 nm), l'emissione a 516 nm (larghezza di banda 5 nm), con un intervallo di lettura di 1 min per 2 ore. - Classificare i valori dell'espressione finale per determinare la concentrazione rNTP.Mg corrispondente alla RFU eGFP più alta. Se sono disponibili dati cinetici, un eccesso di rNTP.Mg sarà indicato anche da un aumento bifasico di eGFP RFU durante il periodo di espressione di 2 ore (vedere la sezione Risultati rappresentativi).
- Una volta determinata la concentrazione di rNTP.Mg top-up ottimizzata, aggiungerla a tutte le ulteriori espressioni proteiche utilizzando il lotto di LTE che è stato parzialmente integrato nelle fasi precedenti.
NOTA: Se l'aliquotazione al punto 6.2 viene eseguita con attenzione con volumi fissi, il rabbocco può essere aggiunto successivamente a ciascuna aliquota senza scongelamento, ad esempio con le aliquote poste su ghiaccio secco. Queste aliquote sono ora completamente integrate, poiché il rabbocco di rNTP.Mg corretto verrà miscelato attraverso ciascuna di esse quando vengono scongelate e miscelate per l'uso.
Risultati
Lo scopo dell'espressione proteica cell-free è quello di produrre proteine a lunghezza intera in una forma attiva ripiegata adatta a un'ampia gamma di applicazioni. L'LTE (Leishmania tarentolae extract) è stato precedentemente confrontato con altri sistemi di espressione liberi da cellule procariotiche ed eucariotiche, dimostrando un'elevata capacità di evitare il troncamento e l'aggregazione quando funziona in modo ottimale, in particolare rispetto all'espressione libera da ...
Discussione
I protocolli per la creazione di LTE sono stati pubblicati nell'ultimo decennio7 e sono stati sottoposti ad aggiornamenti periodici25,34. Tuttavia, i neofiti della tecnica incontrano spesso una curva di apprendimento ripida, con conseguenti ritardi nel raggiungimento di un'espressione proteica di alta qualità e ad alto rendimento. Sfide simili sono state segnalate da altri gruppi di ricerca che lavorano c...
Divulgazioni
Non sono presenti interessi finanziari concorrenti.
Riconoscimenti
Gli autori desiderano ringraziare i numerosi membri del laboratorio Alexandrov che hanno contribuito allo sviluppo dei sistemi LTE negli ultimi 10 anni, in particolare Sergey Mureev che ha aperto la strada al sistema e ha sviluppato il sito di ingresso del ribosoma SITS. La Figura 1 è stata creata da Biorender.com e riprodotta su licenza.
Materiali
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
Riferimenti
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon