È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microbiota dei giardini delle formiche di Attine: visualizzazione di un paesaggio microbico mediante microscopia elettronica a scansione
In questo articolo
Riepilogo
Proponiamo un protocollo ottimizzato di microscopia elettronica a scansione per la visualizzazione di campioni altamente eterogenei e delicati contenenti biomassa vegetale e fungina, insieme a microbiota e biofilm. Questo protocollo consente di descrivere le dimensioni spaziali dell'organizzazione del microbiota.
Abstract
Negli ecosistemi su macroscala, come le foreste pluviali o le barriere coralline, la localizzazione spaziale degli organismi è la base della nostra comprensione dell'ecologia delle comunità. Nel mondo microbico, allo stesso modo, gli ecosistemi su microscala sono ben lungi dall'essere una miscela casuale e omogenea di organismi e habitat. L'accesso alla distribuzione spaziale dei microbi è fondamentale per comprendere il funzionamento e l'ecologia del microbiota, poiché le specie conviventi hanno maggiori probabilità di interagire e influenzare la fisiologia dell'altra.
Un ecosistema microbico interregno è al centro delle colonie di formiche che coltivano funghi, che coltivano funghi basidiomiceti come risorsa nutrizionale. Le formiche attine si nutrono di diversi substrati (per lo più a base vegetale), metabolizzati dal fungo coltivato mentre formano una struttura spugnosa, un "giardino microbico" che funge da intestino esterno. Il giardino è una rete intrecciata di ife fungine che crescono metabolizzando il substrato, aprendo nicchie per l'insediamento di un microbiota caratteristico e adattato. Si ritiene che il microbiota contribuisca alla degradazione del substrato e alla crescita fungina, anche se la sua organizzazione spaziale deve ancora essere determinata.
Qui, descriviamo come impieghiamo la microscopia elettronica a scansione (SEM) per indagare, con dettagli senza precedenti, il microbiota e l'organizzazione spaziale del biofilm in diversi sistemi di fungicoltura di formiche che crescono funghi. L'imaging SEM ha fornito una descrizione della struttura spaziale e dell'organizzazione del microbiota. Il SEM ha rivelato che il microbiota si assembla comunemente in biofilm, una struttura diffusa dei paesaggi microbici nella fungicoltura. Presentiamo i protocolli impiegati per fissare, disidratare, asciugare, spruzzare il rivestimento e visualizzare una comunità così complessa. Questi protocolli sono stati ottimizzati per trattare campioni delicati ed eterogenei, comprendenti biomassa vegetale e fungina, nonché il microbiota e il biofilm.
Introduzione
Gli ecosistemi sono composti da organismi interconnessi da processi in una specifica posizione geografica (ad esempio, l'ambiente). Gli organismi interagiscono con il loro ambiente nel tempo, da cui emergono modelli spaziali complessi ed eterogenei. La modellazione spaziale determina la diversità e la stabilità ecologica e, in ultima analisi, il funzionamento dell'ecosistema 1,2,3,4. Negli ecosistemi su macroscala, come le zone umide, le savane, le barriere coralline e gli ecosistemi aridi, i modelli spaziali sono correlati al flusso e alla concentrazione delle risorse. Consentendo l'ottimizzazione delle risorse, l'eterogeneità spaziale e la modellizzazione si ottengono ecosistemi più resilienti rispetto a quelli omogenei2. La localizzazione spaziale degli organismi, essendo alla base dell'ecologia di comunità, viene tradotta anche nel mondo microbico.
Gli ecosistemi microbici, lontani da organismi mescolati casualmente e in modo omogeneo in tutti i microhabitat, mostrano modelli spaziali che definiscono gran parte del loro funzionamento 5,6,7. Dalle colonne di Winogradsky al microbiota associato all'ambiente e all'ospite, questi ecosistemi sono organizzati in modo eterogeneo nello spazio, con disposizioni spaziali che suscitano diverse risposte fenotipiche. Le specie conviventi hanno maggiori probabilità di interagire e influenzare la fisiologia dell'altra. Così, l'organizzazione spaziale della comunità, più che la sua composizione di per sé, delimita le proprietà dell'ecosistema e le nicchie ecologiche 5,7,8. Illustrando questi concetti, i cambiamenti nel pattern spaziale sembrano correlati alla progressione patologica delle placche dentali, della carie, delle malattie gengivali 9,10, delle malattie infiammatorie intestinali11, delle infezioni polmonari da fibrosi cistica, delle infezioni croniche delle ferite12,13, del cancro del colon-retto e degli adenomi14.
Nell'ambito della biogeografia microbica (lo studio della distribuzione e della modellizzazione della biodiversità nello spazio e nel tempo su microscala), la conoscenza degli ecosistemi microbici è enormemente avvantaggiata dalla comprensione dei loro modelli spaziali 6,13,15,16,17. Abbiamo esaminato i modelli spaziali di un ecosistema microbico costruito da insetti, che si trova al centro delle carismatiche colonie di formiche attine (Hymenoptera: Formicidae: Myrmicinae: Attini: Attina) che crescono funghi. Lì risiede un "giardino microbico", incentrato su un fungo basidiomicete della tribù Leucocoprinae (Basidiomycota: Agaricaceae) o della famiglia Pterulaceae (Basidiomycota: Agaricales)18,19,20,21,22. Il giardino è una struttura spugnosa che emerge da una rete intrecciata di ife che cresce metabolizzando il substrato per lo più vegetale incorporato dalle formiche (Figura 1). Questi possono includere, secondo i generi attini: parti secche di piante, escrementi e carcasse di insetti, foglie appena tagliate, semi e parti floreali23,24. Analogamente a un intestino erbivoro esterno, l'orto converte enzimaticamente e chimicamente i polimeri recalcitranti in risorse nutritive labili, fornendo alle formiche aminoacidi essenziali, lipidi e zuccheri solubili 21,25,26,27,28.
Le analisi ultrastrutturali, enzimatiche e trascrittomiche effettuate per giardini dei generi Atta e Acromyrmex suggeriscono che questi ambienti strutturano un continuum di degradazione del substrato e macchie nutrizionali 26,29,30,31,32. Le parti giovani del giardino tendono ad essere più scure a causa del substrato appena incorporato dopo essere stato frammentato. Questi substrati aggiunti di recente sono spesso colonizzati dai bordi, che sono stati tagliati dalle formiche operaie e inoculati con grumi miceliali. Irradiandosi dai bordi tagliati, le ife fungine si diffondono sul substrato 29,32,33. L'abbondanza ife aumenta con il progredire della degradazione del substrato, con conseguente comparsa di regioni biancastre e metabolicamente attive 30,31,32. Le regioni più vecchie, con substrato più degradato e un abbondante microbiota29,32, tendono a presentare toni brunastri e maggiore umidità. I lavoratori rimuovono i frammenti di questa regione, separandoli in cumuli di rifiuti, dove prendono anche substrati che danneggiano il simbionte fungino 34,35,36. I cumuli di rifiuti, anche se fisicamente staccati dal giardino, sono un punto di continua degradazione del substrato e di ciclo dei nutrienti da parte dell'abbondante microbiota abitante 29,32,37,38,39.
Un microbiota composto principalmente da Enterobacter, Klebsiella, Pantoea, Pseudomonas e Serratia, abita anche il giardino, apparentemente condiviso da diversi sistemi di fungicoltura attina. Codificando vie metaboliche che potrebbero integrare il metabolismo fungino, il microbiota partecipa potenzialmente alle risposte fisiologiche del giardino 40,41,42,43,44. Non solo i dati metagenomici indicavano che il microbiota era presente 41,42, ma anche l'analisi al microscopio elettronico a scansione (SEM) della fungicoltura delle formiche tagliafoglie ha mostrato per lo più batteri a forma di bastoncino sul substrato vegetale32. Sebbene i batteri (compresi i ceppi cellulolitici) fossero isolati dall'intero giardino, venivano visualizzati solo nelle parti più vecchie del giardino e nei cumuli di rifiuti, così come nel pellet iniziale trasportato dalle regine fondatrici29,32. Era anche incerto se il microbiota potesse formare biofilm in vivo (cioè in giardino e nei rifiuti), come suggerito dalla loro capacità metabolica42 e osservato in vitro44.
Qui, abbiamo impiegato il SEM per comprendere ulteriormente l'organizzazione spaziale del microbiota nelle regioni del giardino, dettagliando le interazioni fisiche microbiota-substrato e microbiota-ife. Fornendo immagini con una maggiore profondità focale, il SEM consente osservazioni di strutture microscopiche tridimensionali ad alta risoluzione, consentendo un'analisi approfondita dei modelli spaziali del microbiota del giardino. Descriviamo in dettaglio i passaggi per fissare, disidratare, asciugare, polverizzare il pelo e visualizzare campioni a base fungina così eterogenei e delicati. Rimuovendo la fase di postfissazione utilizzando tetrossido di osmio (OsO4) e riducendo il tempo di disidratazione, abbiamo semplificato i protocolli 32,33,45 per la preparazione di campioni di giardino e rifiuti per l'analisi SEM. Questo protocollo adattato preserva i modelli strutturali ifali, nonché l'organizzazione spaziale del microbiota e del biofilm, e potrebbe essere applicato ad altri delicati ecosistemi microbici e biofilm.
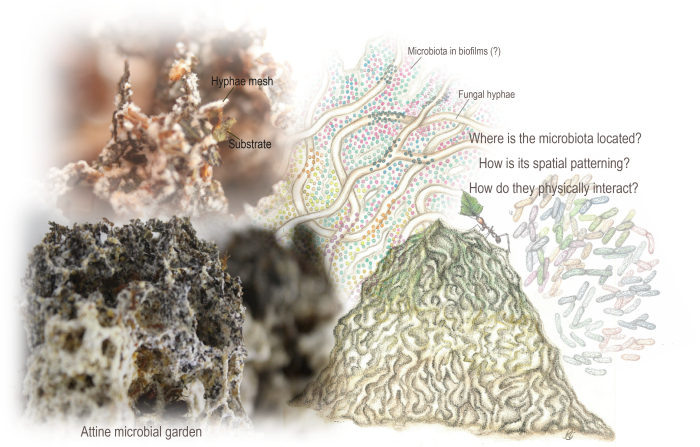
Figura 1: Giardini microbici di Attine. Il giardino è una struttura spugnosa risultante da una rete intrecciata di ife che cresce metabolizzando il substrato per lo più vegetale incorporato dalle formiche. Abita il giardino anche il microbiota, che codifica le vie metaboliche che potrebbero integrare il metabolismo fungino. I dati metagenomici e le precedenti analisi di microscopia elettronica a scansione indicavano la sua presenza, anche se avevamo scarsa conoscenza della sua organizzazione spaziale e delle interazioni fisiche con il substrato e le ife fungine. Abbiamo impiegato il SEM per svelare l'organizzazione spaziale e il pattern del microbiota e del biofilm. Illustrazioni di Mariana Barcoto (giardino e microbiota adattati da Barcoto e Rodrigues 94) e foto di Mariana Barcoto ed Enzo Sorrentino. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Campionamento delle colonie in campo
NOTA: Quando si raccolgono colonie di formiche, certificare che tutte le autorizzazioni richieste dalla legislazione locale siano state ottenute prima della raccolta. Nel nostro caso, il permesso di raccolta #74585 è stato rilasciato dall'Instituto Chico Mendes de Conservação e Biodiversidade (ICMBio). Quando i campioni provengono da una colonia di laboratorio, vai alla sezione 2.
- Individua e contrassegna la colonia. Scavare una trincea che circonda l'area del nido fino a quando la camera del giardino non è esposta (Figura 2A).
NOTA: Alcune specie di attine possono costruire le loro colonie sotto la lettiera di foglie o all'interno di tronchi marci. In questi casi, girare con cura la lettiera o rompere con cura i tronchi per raccogliere i campioni. Per informazioni dettagliate sulla localizzazione, la raccolta e il mantenimento di colonie viventi di diverse specie di formiche attine, vedi Sosa-Calvo et al.46. - Aprire lateralmente la camera del giardino per evitare che il terreno cada sulla superficie del giardino. Raccogli con cura i campioni del giardino usando una pinza entomologica, un cucchiaio o una schiumarola da cucina, a seconda delle dimensioni del giardino.
NOTA: Assicurarsi di sterilizzare gli strumenti prima di raccoglierli. Quando raccogli i giardini delle formiche che tagliano le foglie, indossa guanti di stoffa spessa per prevenire (o almeno attenuare) i morsi delle operaie. Per altre specie di attine, i guanti in tessuto sono opzionali. - Trasferisci i campioni del giardino in un recipiente di plastica pulito contenente uno strato di intonaco per bilanciare l'umidità del giardino. Dopo aver trasferito il giardino e le formiche operaie, chiudere ermeticamente il recipiente per evitare l'essiccazione del campione. Conservare i campioni da giardino a 23-25 °C fino alla lavorazione.
- Chiudere la trincea con il terreno precedentemente rimosso.
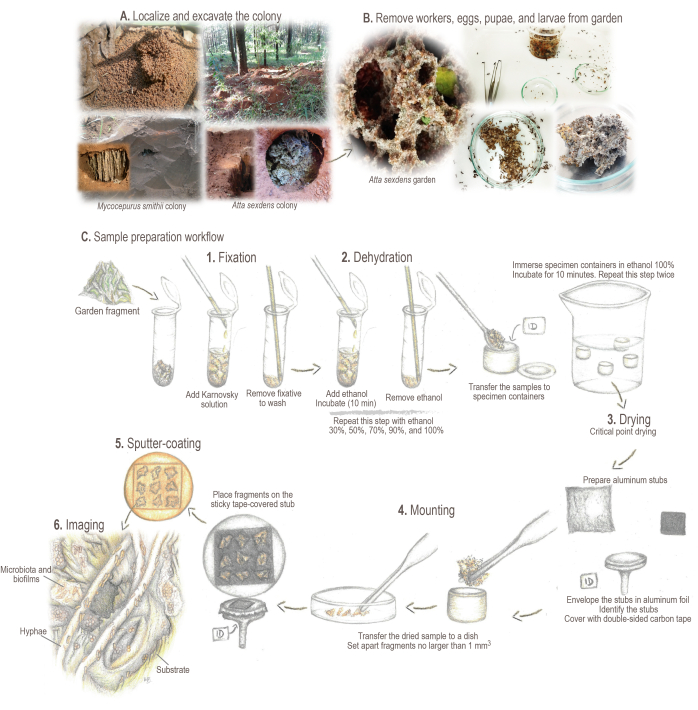
Figura 2: Protocollo di preparazione del campione. (A) Campionamento delle colonie di campo. (B) Elaborazione dei campioni. (C) Brevi fondamenti e flusso di lavoro per la preparazione del campione: 1. Fissaggio: per rafforzare e preservare la struttura del campione. 2. Disidratazione: il contenuto d'acqua dei campioni viene sostituito con etanolo. 3. Essiccazione del punto critico: la CO2 liquida sostituisce l'etanolo ed evapora. 4. Montaggio: campione visualizzato per l'analisi. 5. Rivestimento sputtering con oro: impedisce la carica del campione. 6. Imaging. Illustrazioni e foto di Mariana Barcoto. Clicca qui per visualizzare una versione più grande di questa figura.
2. Reagenti
NOTA: Tenere presente che le seguenti soluzioni devono essere preparate in anticipo.
- Preparare un tampone di cacodilato di sodio 0,2 M. Per questo, sciogliere 42,8 g di cacodilato di sodio in 800 ml di acqua distillata, mescolando fino alla dissoluzione e stabilizzando il pH a 7,2 (se necessario, regolare il pH con acido cloridrico). Portare il volume della soluzione a 1 L aggiungendo acqua distillata. Conservare la soluzione a 4 °C (per ~1 mese).
ATTENZIONE: La glutaraldeide e la paraformaldeide sono tossiche e devono essere maneggiate all'interno di una cappa aspirante. Indossare sempre guanti in nitrile e occhiali protettivi durante la manipolazione di tali reagenti. - Prepara il fissativo di Karnovsky (modificato da Karnovsky)47. Per questo, aggiungere 10 mL di soluzione acquosa di glutaraldeide al 25% e 10 mL di soluzione acquosa di paraformaldeide al 20% a 25 mL di tampone cacodilato di sodio 0,2 M e mescolare. Aggiungere 1 mL di cloruro di calcio 0,1 M (CaCl2) e portare il volume a 100 mL aggiungendo acqua distillata. Conservare la soluzione a 4 °C (per un massimo di 1 mese).
ATTENZIONE: Il fissativo di Karnovsky è dannoso se inalato e può causare irritazione alla pelle e agli occhi. Pertanto, evitare di respirare i vapori, utilizzando solo all'aperto o in un'area ben ventilata. Indossare sempre guanti in nitrile e occhiali protettivi quando si maneggia la soluzione. - Utilizzando etanolo assoluto (grado analitico), preparare soluzioni di etanolo al 30%, 50%, 70% e 90% in acqua distillata; È inoltre necessario il 100% di etanolo.
3. Fissazione del campione
NOTA: I fissativi induriscono e conservano i campioni, mantenendo inalterate le caratteristiche morfologiche. Le aldeidi (come la paraformaldeide e la glutaraldeide) sono fissativi non coagulanti di tipo reticolante, che inducono legami incrociati all'interno e tra le proteine e gli acidi nucleici48.
- Rimuovere le operaie, le uova, le pupe e le larve dai campioni del giardino utilizzando una pinza entomologica (Figura 2B). Mettere da parte i frammenti di giardino di dimensioni non superiori a 5 mm3. Aggiungere i frammenti in una provetta da 2 mL (Figura 2C).
- Alle provette contenenti i campioni, utilizzare una pipetta di vetro Pasteur per aggiungere ~1 mL di soluzione fissativa Karnovsky (assicurarsi che il campione sia completamente coperto). Mescolare agitando delicatamente per aiutare il campione ad immergersi e incubare a 4 °C per almeno 24 ore prima di continuare il trattamento del campione (Figura 2C.1).
NOTA: Quando le seguenti fasi di disidratazione non vengono eseguite immediatamente dopo la fissazione, il protocollo può essere messo in pausa in questa fase e i campioni possono essere conservati per ~1 anno a 4 °C.
Si consiglia di utilizzare una pipetta in vetro Pasteur, poiché è composta da un materiale inerte ed è più facile da pulire per essere riutilizzata in seguito. Il materiale da giardino è altamente idrofobo, tendente a galleggiare sulla superficie della soluzione fissativa. Di solito ci vogliono fino a 5 minuti per essere completamente immersi nel fissativo. Assicurati che il fissativo copra i frammenti del giardino poiché il suo volume tende a ridursi man mano che entra nei pori del campione. Abbiamo verificato empiricamente che dopo che il campione è stato immerso e si è bagnato, i suoi componenti (in particolare, il delicato micelio fungino) sono diventati suscettibili di frantumarsi quando mescolati ulteriormente. Pertanto, si consiglia di evitare il più possibile di agitare i campioni.
4. Disidratazione del campione
NOTA: La serie di lavaggio dell'etanolo scambia gradualmente l'acqua nei campioni con etanolo. È importante iniziare con una soluzione di etanolo a bassa concentrazione (vedi sotto) per evitare di danneggiare eccessivamente o far collassare campioni così delicati49.
- Rimuovere completamente la soluzione fissativa di Karnovsky utilizzando una pipetta di vetro, facendo attenzione a non distruggere il campione (Figura 2C.1).
NOTA: Scartare il fissativo di Karnovsky in un recipiente adeguatamente etichettato per la gestione dei residui chimici tossici. - Subito dopo aver rimosso il fissativo, aggiungere 1 mL di etanolo al 30%, facendo attenzione a non disturbare il campione, e incubare per 10 minuti a temperatura ambiente (Figura 2C.2).
NOTA: I campioni devono essere sempre immersi nella soluzione. Assicurarsi di sostituire rapidamente le soluzioni durante la serie di lavaggio graduale con etanolo. Poiché il giardino perde il suo aspetto poroso e si aggrega sul fondo della provetta, 1 mL di etanolo tende ad essere sufficiente per coprire il campione (quando il campione non è più grande di 5 mm3). Tuttavia, se i campioni non sono completamente coperti, aggiungere etanolo fino a coprire completamente il campione. - Rimuovere completamente l'etanolo al 30% con una pipetta di vetro, facendo attenzione a non disturbare il campione. Scartare correttamente l'etanolo al 30%.
NOTA: Per l'intera serie di lavaggio graduale con etanolo, gettare l'etanolo in un recipiente adeguatamente etichettato per la gestione dei residui chimici tossici. - Aggiungere 1 mL di etanolo al 50% e incubare per 10 minuti a temperatura ambiente. Rimuovere completamente l'etanolo al 50% con una pipetta di vetro, facendo attenzione a non disturbare il campione. Scartare correttamente l'etanolo al 50%.
- Aggiungere 1 mL di etanolo al 70% e incubare per 10 minuti a temperatura ambiente. Rimuovere completamente l'etanolo al 70% con una pipetta di vetro, facendo attenzione a non danneggiare il campione. Scartare correttamente l'etanolo al 70%.
NOTA: Nella fase di lavaggio con etanolo al 70%, l'utente può mettere in pausa il protocollo, se necessario, poiché le provette del campione possono essere conservate durante la notte a 4 °C, quando il materiale non verrà immediatamente lavorato. - Aggiungere 1 mL di etanolo al 90% e incubare per 10 minuti a temperatura ambiente. Rimuovere completamente l'etanolo al 90% con una pipetta di vetro, facendo attenzione a non disturbare il campione. Scartare correttamente l'etanolo al 90%.
- Aggiungere 1 mL di etanolo al 100% e incubare per 10 minuti a temperatura ambiente. Rimuovere completamente l'etanolo al 100% con una pipetta di vetro, facendo attenzione a non disturbare il campione. Scartare correttamente l'etanolo al 100%.
- Utilizzando una pinza e/o una spatola, trasferire con cura i campioni in contenitori per campioni per l'essiccatore a punto critico (CPD), contenenti etichette di identificazione del campione (realizzate in precedenza con carta e matita). Per evitare l'essiccazione dei campioni, questo trasferimento viene effettuato con il contenitore posto in una capsula di Petri ricoperta di etanolo al 100%.
- Mettere i coperchi sui contenitori e immergerli in un becher di vetro graduato contenente etanolo al 100% sufficiente per immergere i contenitori. Coprire il bicchiere di vetro e incubare per 10 minuti a temperatura ambiente; Quindi scartare correttamente l'etanolo al 100%.
- Trasferire i contenitori dei campioni in un altro becher di vetro graduato contenente una quantità sufficiente di etanolo al 100% per immergere i contenitori. Coprire il bicchiere di vetro e incubare per 10 minuti a temperatura ambiente; Quindi, trasferire i contenitori dei campioni nell'essiccatore del punto critico.
NOTA: Dopo la serie di lavaggi, le pipette e i becher di vetro devono essere abbondantemente risciacquati con acqua distillata e l'acqua residua deve essere gettata in un recipiente adeguatamente etichettato per la gestione dei residui chimici tossici. Dopo il risciacquo, gli oggetti in vetro possono essere lavati con un detergente neutro, risciacquati con acqua di rubinetto e asciugati all'aria.
5. Essiccazione del punto critico
NOTA: Un essiccatore a punti critici sostituisce l'etanolo nei campioni con anidride carbonica liquida (CO2), che evapora dal campione a temperature e pressioni più elevate. Si prega di seguire le istruzioni del produttore per tali procedure.
- Accendere l'apparecchiatura.
- Aprire la camera, posizionare i contenitori dei campioni all'interno e aggiungere etanolo al 100% fino a coprire i contenitori. Chiudere la camera.
- Attivare l'opzione Freddo e attendere che la temperatura raggiunga i 10 °C.
- Aprire la valvola della bombola di CO2 e attivare l'opzione Agitatore .
- Attiva l'opzione CO2 in , controllando sempre la camera per verificare quanta CO2 l'ha già riempita. Quando la camera è quasi piena, disattivare l'opzione CO2 in e attivare l'opzione Scambio , mantenendola attivata fino a quando non c'è abbastanza CO2 solo per coprire i contenitori. Assicurarsi che i contenitori siano sempre coperti con CO2 (ad esempio, disattivare l'opzione Sostituisci prima che tutto il CO2 abbia lasciato la camera). Ripetere il passaggio 5.5 6 volte.
- Attivare la CO2 un'ultima volta e riempire la camera fino a coprire i contenitori.
- Attiva l'opzione Calore e disattiva l'opzione Agitatore . Chiudere la valvola della bombola di CO2.
- Attendere che la temperatura salga a 35 °C; quindi, attiva l'opzione Gas out .
NOTA: A circa 30 °C, la camera raggiunge pressioni di 70-80 bar, raggiungendo il punto critico, dove il liquido scompare. - Quando la pressione della camera raggiunge 1 bar, tutto il contenuto di gas è stato rimosso. Aprire la camera e rimuovere i contenitori.
- Spegnere l'apparecchiatura.
6. Montaggio
- Preparare i portacampioni SEM (ad esempio, tronchetti di alluminio; Figura 2C.4).
- Avvolgere i tronchetti con un foglio di alluminio, coprendo solo la parte superiore, per facilitare la pulizia dei tronchetti dopo l'analisi.
- Identificare i mozziconi scrivendo il codice/numero di campione sul fondo del supporto, garantendo l'identificazione di ciò che è posto sopra.
- Coprire la parte superiore con nastro biadesivo in carbonio. Posizionare i tronchetti in un portacampioni.
- Aprire il coperchio del contenitore del campione e trasferire con cura il campione essiccato in una capsula di Petri di vetro utilizzando una pinza e una spatola.
NOTA: I giardini essiccati a punto critico tendono ad agglomerarsi formando campioni altamente impacchettati che devono essere accuratamente separati come frammenti di dimensioni non superiori a 1 mm3. - Posiziona con cura i frammenti sulla superficie adesiva del mozzicone ricoperto di nastro adesivo. Una volta che il frammento di giardino tocca il nastro, è molto difficile (ri)spostarlo, quindi fai attenzione a non posizionarlo in luoghi o posizioni indesiderate. Aggiungi fino a nove frammenti per stub.
- Ripetere i passaggi 6.2 e 6.3 per ciascun campione.
7. Rivestimento sputtering con oro
NOTA: Il rivestimento del campione è necessario per impedirne la carica. Seguire le istruzioni del produttore per regolare impostazioni come la pressione del gas di funzionamento (0,5 × 10-1 mm Hg di pressione del gas in questo protocollo), il tempo di sputtering (220 s), lo spessore dello strato d'oro (~120 Å), la corrente (50 mA) e la tensione di alimentazione. Lo sputtering tende a seguire un flusso di lavoro comune, anche se le apparecchiature di produttori diversi possono funzionare in modo leggermente diverso.
- Aprire il braccio di mira incernierato e posizionare i tronchetti sul tavolo del campione.
- Chiudere il braccio di mira incernierato e controllare se il paraschegge della camera a vuoto in vetro è incorporato correttamente.
- Aprire la valvola della bombola di argon e accendere l'interruttore di alimentazione principale.
- Seguire l'aumento del vuoto sul display dell'apparecchiatura fino a raggiungere il segno di 0.5 × 10-1 mm Hg sul display; quindi, attiva il risciacquo. Ripetere l'operazione 5 volte.
- Accendere il sistema di circolazione dell'acqua, attivare l'opzione HV On e aprire il coperchio della pellicola d'oro. Conferma che il colore del plasma sia rosato. Per seguire questo protocollo, impostare 220 s di sputtering con una tensione di 50 mA, che depositerà uno strato d'oro di ~120 Å (12 nm).
- HV On si spegne automaticamente. Spegnere il sistema di circolazione dell'acqua e chiudere il coperchio della pellicola d'oro.
- Spegnere l'interruttore di alimentazione principale, consentendo all'aria di entrare nella camera a vuoto. Per campioni di giardino e rifiuti, ripetere i passaggi 7.1-7.7 3x.
NOTA: Se l'imaging non avviene immediatamente dopo la preparazione del campione, conservare i tronchetti in un contenitore ermetico riempito con uno strato di silice per evitare di reidratare i campioni.
8. Imaging
NOTA: Seguire le istruzioni del produttore per regolare le impostazioni SEM, come il diametro dell'apertura dell'obiettivo, la tensione di esercizio, l'allineamento del sistema a fascio di elettroni, l'allineamento assiale e gli stigmatori.
- Posizionare i mozziconi nel supporto del campione, prendendo appunti sulla posizione di ciascun campione.
NOTA: Indossare guanti quando si inserisce o si rimuove il supporto del campione e mantenerlo il più pulito possibile. - Avviare il software operativo dal desktop.
- Selezionare le impostazioni dello strumento. Visualizzazione di campioni da giardino con diametro dell'apertura dell'obiettivo di 30 μm (cioè nel secondo stadio), funzionamento in alto vuoto, rilevamento di segnali da elettroni secondari (SED), tensione di accelerazione di 20 kV, distanza di lavoro di 15 - 20 mm, corrente della sonda di 40,0 (in modalità ad alta corrente) e variazione dell'ingrandimento in base al campione.
- Seguendo le istruzioni di navigazione, premere l'icona Vent e attendere che la camera del campione venga ventilata. Una barra di avanzamento indica lo stato del vuoto.
- Quando la pressione atmosferica è stata raggiunta, aprire la camera del campione e inserire con cautela il supporto del campione.
- Chiudere delicatamente lo sportello della camera e premere l'icona Evac per evacuare lo strumento, seguendo lo stato del vuoto attraverso la barra di avanzamento. Il sistema di navigazione mostrerà la posizione del tavolino in movimento e fornirà un grafico del supporto quando il trasloco è completato. Premere l'icona Fotocamera per registrare una foto del supporto per ottenere una vista dall'alto che aiuterà a navigare tra i campioni durante l'imaging.
- Premi l'icona Osservazione per accendere il cannone elettronico e attendi che si formi l'immagine. Utilizzare l'interfaccia utente manuale (controllo manuale) per spostare manualmente l'asse Z all'altezza corretta (in base all'altezza del campione e alla distanza di lavoro determinata).
NOTA: Per prolungare la durata del filamento di tungsteno, mettere in pausa la funzione di osservazione ogni volta che non si esegue l'imaging attivo. - Utilizzare le opzioni mostrate sullo schermo o il controllo manuale per modificare la velocità raster e la posizione dell'immagine. Sposta il tavolino per ottenere una visione completa del campione, utilizzando la funzione RDC per concentrarti su aree specifiche. Quando si osserva una struttura target (o interessante), regolare di conseguenza l'ingrandimento, la messa a fuoco, la luminosità, il contrasto e la stigmatizzazione . Correggere la stigmatizzazione utilizzando l'interfaccia utente manuale per spostare il tavolino nelle direzioni X e Y.
- Regola l'ingrandimento tra 100x e 700x per visualizzare gli aspetti generali del giardino (come la densità ifale, il substrato e i modelli di colonizzazione).
- Regola l'ingrandimento tra 700x e 1.500x per visualizzare i modelli spaziali del microbiota.
- Regola l'ingrandimento tra 1.500x e 3.000x per osservare le interazioni fisiche tra microbiota e biofilm.
- Regola l'ingrandimento tra 3.000x e 4.000x per concentrarti su specifici cluster microbici.
- Per salvare un'immagine, utilizzare la funzione Blocca , fare clic sull'icona Foto e impostare il percorso del file. Come standard, analizzare almeno tre frammenti di giardino, riprendendo ciascuno di essi in tutti gli intervalli di ingrandimento menzionati al punto 8.8. Ciò si traduce in almeno 12 immagini per campione, anche se un numero maggiore di immagini può supportare descrizioni più dettagliate. Suggeriamo che 15-25 immagini per campione, che vanno tutti gli ingrandimenti nel passaggio 8.8, tendano a fornire dettagli precisi per la descrizione del campione.
NOTA: Il rastering e il salvataggio di un'immagine richiedono alcuni secondi, durante i quali è necessario evitare qualsiasi vibrazione sulla tavola mobile SEM. - Dopo aver terminato l'imaging, premere l'opzione Sfiato per sfiatare la camera, seguendo lo stato del vuoto attraverso la barra di avanzamento. Rimuovere con cautela il supporto, chiudere delicatamente lo sportello della camera e premere Evac per evacuare la camera.
- Non appena l'evacuazione è completata, uscire dal software operativo.
- Regola la luminosità e il contrasto per migliorare la visualizzazione utilizzando un editor di immagini.
Risultati
Qui, abbiamo presentato un protocollo semplificato per visualizzare i componenti del giardino di attine e i campioni di scarto, come ife fungine, substrato, microbiota e biofilm. Il SEM ha migliorato la nostra comprensione di come il giardino e i rifiuti impalcano i modelli strutturali del microbiota (Figura 3). Nei giardini attini, le ife fungine sono strutture simili a rami che coprono porzioni della superficie del substrato. Poiché le ife fungine tendono...
Discussione
Il SEM utilizza un fascio di elettroni per scansionare il campione, generando un'immagine ingrandita di esso in modo tale da poter visualizzare microstrutture tridimensionali ad alta risoluzione. Poiché il SEM funziona in alto vuoto, è necessaria la rimozione di fino a / più del 99% di acqua dai campioni. All'interno della camera a vuoto SEM, i campioni parzialmente idratati possono disidratarsi e collassare, oltre a disperdere elettroni. Per l'imaging ad alta risoluzione nel SEM, la ...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare la Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) per il sostegno finanziario (Grant #2019/03746-0). MOB ringrazia per la borsa di dottorato ricevuta da FAPESP (processo 2021/08013-0) e Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - Brasile (CAPES) - Codice finanziario 001. AR ringrazia anche il Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) per una borsa di ricerca (#305269/2018). Gli autori ringraziano Marcia Regina de Moura Aouada e Antonio Teruyoshi Yabuki per l'aiuto nei test pilota per la preparazione del campione, Renato Barbosa Salaroli per l'assistenza tecnica e Enzo Sorrentino per l'aiuto nello shooting fotografico. Questo studio è stato condotto nell'ambito dell'autorizzazione all'accesso al patrimonio genetico # SISGen AA39A6D.
Materiali
| Name | Company | Catalog Number | Comments |
| 2 mL tube | Axygen | MCT-200-C-BRA | To fix and dehydrate samples |
| Calcium chloride anhydrous | Merck | C4901 | CaCl2 anhydrous to prepare Karnovsky’s fixative |
| Critical point dryer | Leica | EM CPD 300 | For critical point drying |
| Double Sided Carbon Conductive Tape, 12 mm (W) X 5 M (L) | Electron Microscopy Sciences | 77819-12 | For mounting samples |
| Entomological forceps | No specific supplier | To manipulate garden samples | |
| Ethyl alcohol (=ethanol), pure (≥99.5%) | Sigma-Aldrich | 459836 | For dehydration |
| Forceps | No specific supplier | To manipulate garden samples | |
| Glass beaker | No specific supplier | For dehydration | |
| Glass Petri dish | No specific supplier | To manipulate garden samples | |
| Glass pipette | No specific supplier | To fix and dehydrate samples | |
| Glutaraldehyde (Aqueous Glutaraldehyde EM Grade 25%) | Electron Microscopy Sciences | 16220 | To prepare Karnovsky’s fixative |
| Gold target | Ted Pella, Inc. | 8071 | To sputter coat with gold |
| Hydrochloric acid | Sigma-Aldrich | 320331 | For adjusting solutions pH |
| Image editor | Photoshop | any version | To adjust images |
| Paraformaldehyde (Paraformaldehyde 20% Aqueous Solution EM Grade) | Electron Microscopy Sciences | 15713 | To prepare Karnovsky’s fixative |
| Propilene recipient | No specific supplier | For maintaining alive ant colonies | |
| Scanning Electron Microscope | JEOL | IT300 SEM | For sample imaging |
| Sodium cacodylate trihydrate | Sigma-Aldrich | C0250 | For preparing sodium cacodylate buffer |
| Spatula | No specific supplier | To manipulate garden samples | |
| Specimen containers with 15 mm dia. x 10 mm high | Ted Pella, Inc. | 4591 | For critical point drying |
| Sputter coater | Baltec | SCD 050 | To coat with gold |
| Stub (Aluminium mount, flat end pin) 12.7 mm x 8 mm | Electron Microscopy Sciences | 75520 | For mounting samples |
Riferimenti
- Turner, M. G. Landscape ecology: the effect of pattern on process. Annu Rev Ecol Evol Syst. 20 (1), 171-197 (1989).
- Rietkerk, M., Van de Koppel, J. Regular pattern formation in real ecosystems. Trends Ecol Evol. 23 (3), 169-175 (2008).
- Schmitz, O. J. Spatial dynamics and ecosystem functioning. PLOS Biol. 8 (5), e1000378 (2010).
- Pringle, R. M., Doak, D. F., Brody, A. K., Jocqué, R., Palmer, T. M. Spatial pattern enhances ecosystem functioning in an African savanna. PLOS Biol. 8 (5), e1000377 (2010).
- Wimpenny, J. W. Spatial order in microbial ecosystems. Biol Rev. 56 (3), 295-342 (1981).
- Martiny, J. B. H., et al. Microbial biogeography: putting microorganisms on the map. Nat Rev. Microbiol. 4 (2), 102-112 (2006).
- McCallum, G., Tropini, C. The gut microbiota and its biogeography. Nat Rev Microbiol. 22 (2), 105-118 (2024).
- Lamont, R. J., Hajishengallis, G., Koo, H. Social networking at the microbiome-host interface. Infec Immun. 91 (9), e00124-e00223 (2023).
- Welch, J. L. M., Rossetti, B. J., Rieken, C. W., Dewhirst, F. E., Borisy, G. G. Biogeography of a human oral microbiome at the micron scale. Proc Natl Acad Sci USA. 113 (6), E791-E800 (2016).
- Kim, D., et al. Spatial mapping of polymicrobial communities reveals a precise biogeography associated with human dental caries. Proc Natl Acad Sci USA. 117 (22), 12375-12386 (2020).
- Swidsinski, A., Weber, J., Loening-Baucke, V., Hale, L. P., Lochs, H. Spatial organization and composition of the mucosal flora in patients with inflammatory bowel disease. J Clin Microbiol. 43 (7), 3380-3389 (2005).
- Ibberson, C. B., Barraza, J. P., Holmes, A. L., Cao, P., Whiteley, M. Precise spatial structure impacts antimicrobial susceptibility of S. aureus in polymicrobial wound infections. Proc Natl Acad Sci USA. 119 (51), e2212340119 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Dejea, C. M., et al. Microbiota organization is a distinct feature of proximal colorectal cancers. Proc Natl Acad Sci USA. 111 (51), 18321-18326 (2014).
- Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., Martiny, J. B. Beyond biogeographic patterns: processes shaping the microbial landscape. Nat Rev Microbiol. 10 (7), 497-506 (2012).
- Adade, E. E., Al Lakhen, K., Lemus, A. A., Valm, A. M. Recent progress in analyzing the spatial structure of the human microbiome: Distinguishing biogeography and architecture in the oral and gut communities. Curr Opin Endocr. 18, 275-283 (2021).
- Mony, C., Bohannan, B. J., Leibold, M. A., Peay, K., Vandenkoornhuyse, P. Microbial landscape ecology: Highlights on the invisible corridors. Front Ecol Evol. 9, 753213 (2021).
- Hölldobler, B., Wilson, E. O. . The Ants. , (1990).
- Chapela, I. H., Rehner, S. A., Schultz, T. R., Mueller, U. G. Evolutionary history of the symbiosis between fungus-growing ants and their fungi. Science. 266 (5191), 1691-1694 (1994).
- Mueller, U. G., Rehner, S. A., Schultz, T. R. The evolution of agriculture in ants. Science. 281 (5385), 2034-2038 (1998).
- Mueller, U. G., Gerardo, N. M., Aanen, D. K., Six, D. L., Schultz, T. R. The evolution of agriculture in insects. Annu Rev Ecol Evol Syst. 36, 563-595 (2005).
- Dentinger, B. T., Lodge, D. J., Munkacsi, A. B., Desjardin, D. E., McLaughlin, D. J. Phylogenetic placement of an unusual coral mushroom challenges the classic hypothesis of strict coevolution in the Apterostigma pilosum group ant-fungus mutualism. Evolution. 63 (8), 2172-2178 (2009).
- Schultz, T. R., Brady, S. G. Major evolutionary transitions in ant agriculture. Proc Natl Acad Sci USA. 105, 5435-5440 (2008).
- de Fine Licht, H. H., Boomsma, J. J. Forage collection, substrate preparation, and diet composition in fungus-growing ants. Ecol Entomol. 35 (3), 259-269 (2010).
- Martin, M. M. The biochemical basis of the fungus-attine ant symbiosis: A complex symbiosis is based upon integration of the carbon and nitrogen metabolisms of the two organisms. Science. 169 (3940), 16-20 (1970).
- Grell, M. N., et al. The fungal symbiont of Acromyrmex leaf-cutting ants expresses the full spectrum of genes to degrade cellulose and other plant cell wall polysaccharides. BMC Genomics. 14, 928 (2013).
- Lange, L., Grell, M. N. The prominent role of fungi and fungal enzymes in the ant-fungus biomass conversion symbiosis. Appl Microbiol Biotechnol. 98, 4839-4851 (2014).
- Huang, E. L., et al. The fungus gardens of leaf-cutter ants undergo a distinct physiological transition during biomass degradation. Environ Microbiol Rep. 6 (4), 389-395 (2014).
- Craven, S. E., Dix, M. W., Michaels, G. E. Attine fungus gardens contain yeasts. Science. 169 (3941), 184-186 (1970).
- De Fine Licht, H. H., Boomsma, J. J., Tunlid, A. Symbiotic adaptations in the fungal cultivar of leaf-cutting ants. Nat Commun. 5 (1), 5675 (2014).
- Aylward, F. O., et al. Enrichment and broad representation of plant biomass-degrading enzymes in the specialized hyphal swellings of Leucoagaricus gongylophorus, the fungal symbiont of leaf-cutter ants. PLoS One. 10 (8), e0134752 (2015).
- Moreira-Soto, R. D., Sanchez, E., Currie, C. R., Pinto-Tomás, A. A. Ultrastructural and microbial analyses of cellulose degradation in leaf-cutter ant colonies. Microbiology. 163 (11), 1578-1589 (2017).
- Erthal Jr, M., Silva, C. P., Cooper, R. M., Samuels, R. I. Hydrolytic enzymes of leaf-cutting ant fungi. Comp Biochem. 152 (1), 54-59 (2009).
- North, R. D., Jackson, C. W., Howse, P. E. Communication between the fungus garden and workers of the leaf-cutting ant, Atta sexdens rubropilosa, regarding choice of substrate for the fungus. Physiol Entomol. 24 (2), 127-133 (1999).
- Herz, H., Hölldobler, B., Roces, F. Delayed rejection in a leaf-cutting ant after foraging on plants unsuitable for the symbiotic fungus. Behav Ecol. 19 (3), 575-582 (2008).
- Schiøtt, M., De Fine Licht, H. H., Lange, L., Boomsma, J. J. Towards a molecular understanding of symbiont function: identification of a fungal gene for the degradation of xylan in the fungus gardens of leaf-cutting ants. BMC Microbiol. 8, 40 (2008).
- Bot, A. N., Currie, C. R., Hart, A. G., Boomsma, J. J. Waste management in leaf-cutting ants. Ethol Ecol Evol. 13 (3), 225-237 (2001).
- Scott, J. J., et al. Microbial community structure of leaf-cutter ant fungus gardens and refuse dumps. PLOS One. 5 (3), e9922 (2010).
- Lewin, G. R., et al. Cellulose-enriched microbial communities from leaf-cutter ant (Atta colombica) refuse dumps vary in taxonomic composition and degradation ability. PLOS One. 11 (3), e0151840 (2016).
- Suen, G., et al. An insect herbivore microbiome with high plant biomass-degrading capacity. PLOS Genet. 6 (9), e1001129 (2010).
- Aylward, F. O., et al. Convergent bacterial microbiotas in the fungal agricultural systems of insects. MBio. 5 (6), e02077 (2014).
- Barcoto, M. O., et al. Fungus-growing insects host a distinctive microbiota apparently adapted to the fungiculture environment. Sci Rep. 10 (1), 12384 (2020).
- Francoeur, C. B., et al. Bacteria contribute to plant secondary compound degradation in a generalist herbivore system. mBio. 11, e02146-e02220 (2020).
- Martiarena, M. J. S., Deveau, A., Montoya, Q. V., Flórez, L. V., Rodrigues, A. The hyphosphere of leaf-cutting ant cultivars is enriched with helper bacteria. Microb Ecol. 86 (3), 1773-1788 (2023).
- Leal-Dutra, C. A., et al. Evidence that the domesticated fungus Leucoagaricus gongylophorus recycles its cytoplasmic contents as nutritional rewards to feed its leafcutter ant farmers. IMA Fungus. 14 (1), 19 (2023).
- Sosa-Calvo, J., Jesovnik, A., Okonski, E., Schultz, T. R. Locating, collecting, and maintaining colonies of fungus-farming ants (Hymenoptera: Myrmicinae: Attini). Sociobiology. 62 (2), 300-320 (2015).
- Karnovsky, M. J. A formaldehyde glutaraldehyde fixative of high osmolality for use in electron microscopy. J Cell Biol. 27, 137-138 (1965).
- Eltoum, I., Fredenburgh, J., Myers, R. B., Grizzle, W. E. Introduction to the theory and practice of fixation of tissues. J Histotechnol. 24 (3), 173-190 (2001).
- Gusnard, D., Kirschner, R. H. Cell and organelle shrinkage during preparation for scanning electron microscopy: effects of fixation, dehydration and critical point drying. J Microsc. 110 (1), 51-57 (1977).
- Surman, S. B., et al. Comparison of microscope techniques for the examination of biofilms. J Microbiol Methods. 25 (1), 57-70 (1996).
- Augustin, J. O., et al. Yet more "weeds" in the garden: fungal novelties from nests of leaf-cutting ants. PLOS One. 8 (12), e82265 (2013).
- Montoya, Q. V., Martiarena, M. J. S., Polezel, D. A., Rodrigues, A. More pieces to a huge puzzle: Two new Escovopsis species from fungus gardens of attine ants. MycoKeys. 46, 97 (2019).
- Varanda-Haifig, S. S., et al. Nature of the interactions between hypocrealean fungi and the mutualistic fungus of leaf-cutter ants. Antonie van Leeuwenhoek. 110, 593-605 (2017).
- Custodio, B. C., Rodrigues, A. Escovopsis kreiselii specialization to its native hosts in the fungiculture of the lower attine ant Mycetophylax morschi. Antonie van Leeuwenhoek. 112, 305-317 (2019).
- Schröttner, H., Schmied, M., Scherer, S. Comparison of 3D surface reconstruction data from certified depth standards obtained by SEM and an Infinite Focus Measurement Machine (IFM). Microchim Acta. 155, 279-284 (2006).
- Zhou, W., Apkarian, R., Wang, Z. L., Joy, D., Zhou, W., Wang, Z. L. Fundamentals of scanning electron microscopy (SEM). Scanning microscopy for nanotechnology: Techniques and applications. , 1-40 (2007).
- Kannan, M., Raja, K., Subramanian, K. S., Kannan, M. Scanning electron microscopy: Principle, components and applications. A Textbook on Fundamentals and Applications of Nanotechnology. , 81-92 (2018).
- Ruffolo, J. J. Critical point drying of protozoan cells and other biological specimens for scanning electron microscopy: apparatus and methods of specimen preparation. Trans Am Microsc. 93 (1), 124-131 (1974).
- Echlin, P. Recent advances in specimen coating techniques for electron microscopy. Scanning Electron Microscopy 1981/1. , 79-90 (1981).
- Ris, H. The cytoplasmic filament system in critical point-dried whole mounts and plastic-embedded sections. J Cell Biol. 100 (5), 1474-1487 (1985).
- Bray, D. F., Bagu, J., Koegler, P. Comparison of hexamethyldisilazane (HMDS), Peldri II, and critical-point drying methods for scanning electron microscopy of biological specimens. Microsc Res Tech. 26 (6), 489-495 (1993).
- Bergmans, L., Moisiadis, P., Van Meerbeek, B., Quirynen, M., Lambrechts, P. Microscopic observation of bacteria: review highlighting the use of environmental SEM. Int Endod J. 38 (11), 775-788 (2005).
- Kirk, S. E., Skepper, J. N., Donald, A. M. Application of environmental scanning electron microscopy to determine biological surface structure. J Microsc. 233 (2), 205-224 (2009).
- Kemmenoe, B. H., Bullock, G. R. Structure analysis of sputter-coated and ion-beam sputter-coated films: a comparative study. J Microsc. 132 (2), 153-163 (1983).
- Kinden, D. A., Brown, M. F. Technique for scanning electron microscopy of fungal structures within plant cells. Phytopathology. 65, 74-76 (1975).
- Masaphy, S., Levanon, D., Tchelet, R., Henis, Y. Scanning electron microscope studies of interactions between Agaricus bisporus (Lang) Sing hyphae and bacteria in casing soil. Appl Environ Microbiol. 53 (5), 1132-1137 (1987).
- Massicotte, H. B., Melville, L. H., Peterson, R. L. Scanning electron microscopy of ectomycorrhizae potential and limitations. Scanning Microsc. 1 (3), 58 (1987).
- Visen, A., Singh, P. N., Chakraborty, B., Singh, A., Bisht, T. S. Scanning electron microscopy indicates Pseudomonad strains facilitate AMF mycorrhization in litchi (Litchi chinensis Sonn.) air-layers and improving survivability, growth and leaf nutrient status. Curr Res Microb Sci. 2, 100063 (2021).
- Fleeman, R. M., Mikesh, M., Davies, B. W. Investigating Klebsiella pneumoniae biofilm preservation for scanning electron microscopy. Access Microbiol. 5 (2), 000470-000473 (2023).
- Wells, M., Mikesh, M., Gordon, V. Structure-preserving fixation allows scanning electron microscopy to reveal biofilm microstructure and interactions with immune cells. J Microsc. 293 (1), 59-68 (2024).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nat Rev Microbiol. 14 (9), 589-600 (2016).
- Madsen, J. S., et al. Coexistence facilitates interspecific biofilm formation in complex microbial communities. Environ Microbiol. 18 (8), 2565-2574 (2016).
- Madsen, J. S., Sørensen, S. J., Burmølle, M. Bacterial social interactions and the emergence of community-intrinsic properties. Curr Opin Microbiol. 42, 104-109 (2018).
- Flemming, H. C., Wuertz, S. Bacteria and archaea on Earth and their abundance in biofilms. Nat Rev Microbiol. 17 (4), 247-260 (2019).
- Bringhurst, B., Allert, M., Greenwold, M., Kellner, K., Seal, J. N. Environments and hosts structure the bacterial microbiomes of fungus-gardening ants and their symbiotic fungus gardens. Microb Ecol. 86 (2), 1374-1392 (2023).
- Bringhurst, B., Greenwold, M., Kellner, K., Seal, J. N. Symbiosis, dysbiosis and the impact of horizontal exchange on bacterial microbiomes in higher fungus-gardening ants. Sci Rep. 14 (1), 3231 (2024).
- Vargas, S., et al. Body-plan reorganization in a sponge correlates with microbiome change. Mol Biol Evol. 40 (6), msad138 (2023).
- Apprill, A., et al. Humpback whale populations share a core skin bacterial community: towards a health index for marine mammals. PLOS One. 9 (3), e90785 (2014).
- Fraune, S., et al. Bacteria-bacteria interactions within the microbiota of the ancestral metazoan Hydra contribute to fungal resistance. ISME J. 9 (7), 1543-1556 (2015).
- Gowen, R., Gamal, A., Di Martino, L., McCormick, T. S., Ghannoum, M. A. Modulating the microbiome for Crohn's disease treatment. Gastroenterology. 164 (5), 828-840 (2023).
- Moya, P., et al. Molecular phylogeny and ultrastructure of the lichen microalga Asterochloris mediterranea sp. nov. from Mediterranean and Canary Islands ecosystems. Int J Syst Evol Microbiol. 65 (6), 1838-1854 (2015).
- Zettler, E. R., Mincer, T. J., Amaral-Zettler, L. A. Life in the "plastisphere": microbial communities on plastic marine debris. Environ Sci Technol. 47 (13), 7137-7146 (2013).
- Porter, K. R., Kallman, F. The properties and effects of osmium tetroxide as a tissue fixative with special reference to its use for electron microscopy. Exp Cell Res. 4 (1), 127-141 (1953).
- Ligon, J. J., Abraham, J. L., Boyd, A. S. Traumatic osmium tetroxide inoculation. J Am Acad Dermatol. 45 (6), 949-952 (2001).
- Friedova, N., et al. Osmium absorption after osmium tetroxide skin and eye exposure. Basic Clin Pharmacol Toxicol. 127 (5), 429-433 (2020).
- Fratesi, S. E., Lynch, F. L., Kirkland, B. L., Brown, L. R. Effects of SEM preparation techniques on the appearance of bacteria and biofilms in the Carter Sandstone. J Sediment. 74 (6), 858-867 (2004).
- Dassanayake, R. P., et al. Identification of a reliable fixative solution to preserve the complex architecture of bacterial biofilms for scanning electron microscopy evaluation. PLOS One. 15 (5), e0233973 (2020).
- Desiro, A., et al. Detection of a novel intracellular microbiome hosted in arbuscular mycorrhizal fungi. ISME J. 8 (2), 257-270 (2014).
- Morales, D. P., et al. Advances and challenges in fluorescence in situ hybridization for visualizing fungal endobacteria. Front Microbiol. 13, 892227 (2022).
- Shi, H., et al. Highly multiplexed spatial mapping of microbial communities. Nature. 588 (7839), 676-681 (2020).
- Valm, A. M., Welch, J. L. M., Borisy, G. G. CLASI-FISH: principles of combinatorial labeling and spectral imaging. Syst Appl Microbiol. 35 (8), 496-502 (2012).
- Fernandez-Brime, S., Muggia, L., Maier, S., Grube, M., Wedin, M. Bacterial communities in an optional lichen symbiosis are determined by substrate, not algal photobionts. FEMS Microbiol Ecol. 95 (3), fiz012 (2019).
- Schluter, S., Eickhorst, T., Mueller, C. W. Correlative imaging reveals holistic view of soil microenvironments. Environ Sci Technol. 53 (2), 829-837 (2018).
- Barcoto, M. O., Rodrigues, A. Lessons from insect fungiculture: from microbial ecology to plastics degradation. Front Microbiol. 13, 812143 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon