このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
のテロメアG -オーバーハング構造の検討トリパノソーマ
要約
テロメアは染色体の安定化に不可欠であり、テロメアG -オーバーハング構造はテロメラーゼを介したテロメア維持に不可欠です。我々は最近ではテロメアG -オーバーハング構造を検出するための2つの方法を採用しているトリパノソーマ、。
要約
テロメアG -オーバーハング構造は酵母、脊椎動物、およびトリパノソーマを含む多くの真核生物で同定されている。それは 、de novoテロメアDNA合成のためのテロメラーゼの基質として機能し、テロメアの維持のために重要である。T.トリパノソーマは牛で、人間とナガナ病における睡眠病を引き起こす原虫寄生虫です。一度感染哺乳動物宿主、T.トリパノソーマの細胞は、定期的に宿主の免疫攻撃を回避するために、その表面抗原を切り替えます。我々は最近、T.ことが実証されているトリパノソーマテロメアの構造は、T.ために重要な表面抗原の遺伝子発現の調節に重要な役割を果たしているトリパノソーマの病因。しかし、T.トリパノソーマテロメア構造が広範囲にこの特殊な構造の解析のための方法の制限のために研究されていない。我々は正常にネイティブゲル内ハイブリダイゼーションとテロメアG -オーバーハング構造とT.の塩基テロメア端末の決定のためのアダプターのライゲーション法の検討のためのライゲーションを介したプライマー伸長法を採用しているトリパノソーマの細胞。ここで、我々は詳細にプロトコルを説明し、それぞれ異なる長所と限界を比較します。
プロトコル
1。 T.を検出ネイティブゲル内ハイブリダイゼーション3を使用してトリパノソーマテロメアG -オーバーハング
原理(図1):ネイティブ条件下では、末端標識(CCCTAA)4(テルチ)オリゴプローブは唯一の一本鎖テロメアG -オーバーハング領域にハイブリダイズすることができる。ハイブリダイゼーションの強度は、オーバーハングの長さに比例します。変性および中和した後、同じプローブは全体テロメア領域にハイブリダイズすることができます。ハイブリダイゼーションの強度は、全体のテロメアのDNAの量を表し、ローディングコントロールとして使用することができます。この実験のための2つの標準コントロールには、T.、任意の信号が得られないはず末端標識(TTAGGG)4(TELG)オリゴプローブを使用して、ハイブリダイゼーションですトリパノソーマの細胞にはテロメアC -オーバーハング構造を持っていない、そしてそのようなネイティブTelcのハイブリダイゼーションシグナルを排除すべきエキソIまたはエキソハイブリダイゼーションの前にT、として3' -特異的一本鎖エキソヌクレアーゼとゲノムDNAを扱うために。
ステップ1。パルスフィールドゲル電気泳動(PFGE)のためにDNAのプラグを準備する
無傷の染色体はPFGEで区切られます。そのため、ゲノムDNAは、DNAのプラグとして調製される。
- 血流の形の細胞の100mLの(150 -2000000細胞/ mLの時)またはprocyclic細胞(107細胞/ mLの時)の20mLで起動します。
- Lバッファー(0.01 Mトリス•CL pHは7.6、0.02 M塩化ナトリウム、0.1 M EDTA液pH8.0)、50でそれを暖かく保つ℃で、低融点(LMP)アガロース1.6%溶かす
- 1.5 krpmの遠心分離によって細胞を回収、4℃で10分間。 500万細胞/ mlの最終濃度までLバッファーで上清と再懸細胞ペレットを削除します。
- 10分間42から50℃で細胞懸濁液をインキュベートする。
- 細胞の1ボリュームにLMPアガロースの1ボリュームを追加します。よく混ぜが、すぐに。
- 使い捨てプラグモールド(BioRad社)の各ウェルに細胞懸濁液のアリコートを85μL。
- 5〜10分間後、またはプラグが固化されるまでは、5mLのLバッファー/ 1%サルコシルと15 mLのコニカルチューブに移すプラグ。プロテイナーゼKパウダー250 mg / mLのプロテイナーゼKまたはピンチを100μLを加え、よく混ぜ、50℃でインキュベート48時間C。
- バッファを破棄し、二回Lの緩衝液5 mLでプラグを洗浄し、別の48時間プロテイナーゼK消化を繰り返します。再び新鮮なLバッファーでプラグを洗う。店舗は4でプラグ· LのバッファでC。これらのプラグは2週間以内にご使用ください。
ステップ2。別無傷T.パルスフィールドゲル電気泳動を用いたトリパノソーマ染色体
- アガロースゲルを準備する
- 0.5 × TBEバッファーとCHEF DRII(BioRad社)単位のゲル実行室への転送の2-3 Lを準備します。実行中の温度を設定し、ポンプを起動して、冷却ユニットを起動します。
- 0.5 × TBE緩衝液中で1.2%アガロースゲル100mLを準備する。クールアガロースはゲルトレイを設定中。
- サンプル順序を決める。 DNAの標準を含めることを忘れないでください。櫛を乾燥させ、平らな面に置きます。適切な歯の上にそれぞれのDNAのプラグインを置きます。プラグが歯に付着されるように吸引することによりDNAのプラグの周りに過剰なバッファを削除します。
- コームを挿入せずにアガロースゲルを注ぐ。それは40-50 ° C(数分)まで冷却させ、徐々にゲルに付属のプラグが付いている櫛を挿入します。プラグは櫛をオフにスライドさせていないことを確認してください。ゲルが固化してみましょう。徐々にゲルからコームを抜き取る。
- パルスフィールドゲル電気泳動
120時間で12時700 S - 1500のパルス、2.5 V / cmの、℃:CHEF DRIIのユニットでゲルを実行します。
ステップ3。アガロースゲルを染色し、汚れを除く
その後、0.5 × TBEの中で穏やかに振盪しながら1時間、エチジウムブロマイドなしで1時間、室温で0.5 × TBEで1μgの/ mlのエチジウムブロマイドでゲルを染色する。ゲルの写真を撮る。
ステップ4。アガロースゲルを乾燥させる
3ミリメートルワットマン濾紙の二つの層にアガロースゲルを置き、ラップでゲルをカバー。室温でゲルを乾燥させる(ゲルが50以上に加熱してはならない° Cは、DNAサンプルの任意の変性を避けるため)。それぞれ、乾燥の2および4時間後に乾いたものと湿ったワットマンのペーパーを交換してください。ゲルが完全に乾くまで乾燥してください。これは、乾燥機に応じて、一晩かかることがあります。
ステップ5。オリゴプローブをラベル
10 ×ポリヌクレオチドキナーゼ(PNK)バッファ、5μLの1μLを50 ng /μLのテルチまたはTELGオリゴの1μLを混合γ[32P] ATP、のddH 2 O 2μL、およびT4 PNKの1μL。 37℃で1時間インキュベートする。反応混合物にTNESバッファー(10mMトリスClを、pHが8.0、100mMのNaCl、10mMのEDTA、pH8.0の、1%SDS)を90μLを加える。
oligoesにラベルを付ける一方で、G - 25カラムを準備:3 ccシリンジにいくつかのグラスウールを入れて、ピペットの先端でしっかりとそれを詰め。セファデックスG - 25ファイン(autoclで注射器を埋める3mLのマークに)TEでaved。それは重量で解決しましょう。標識プローブを精製するために:列の上部にプローブをロードする。 TNESバッファー700μLで洗浄し、そしてTNESバッファ600μLで溶出。また、ミニクイックスピンを用いて標識プローブを精製™カラム(ロシュダイアグノスティックス)。
ステップ6:ハイブリダイゼーション
- 簡単に言えば蒸留水で乾燥ゲルを濡らし、ゲルに付着したあらゆるワットマン濾紙を洗い流す
- ハイブリダイゼーションバッグにゲルを置き、(0.25MのNa 2 HPO 4 pH7.2、1mMのEDTA、7%SDS、1%BSA、0.22μmフィルターを通して濾過)hybridiztionの緩衝液20 mLを加え。 ° C穏やかな振盪し、少なくとも1時間50℃の水浴中でインキュベートする。
- プレハイブリダイゼーションバッファーを除去する。 5分間標識プローブを沸騰させ、新鮮なハイブリダイゼーションバッファー25 mLにそれを追加し、0.22μmのシリンジフィルターでミックスをろ過滅菌し、ハイブリダイゼーションバッグに直接ハイブリダイゼーションミックスを追加。暖かい水と浅い容器に袋と場所を密閉する。水浴中に全体の容器を置きます。穏やかなロッキングと℃で一晩50でインキュベートする。
- ハイブリダイゼーションバッグに小さな開口部をカット。可能な限りハイブリダイゼーションミックスを注ぎ、50 mLコニカルチューブに保存してください。 -20℃で凍結プローブを保持するハイブリダイゼーションバッグからゲルを取り出して、容器に収納してください。
このステップでエヴリシングが熱くなっていると大規模な掃除を(ベンチ、SCREEN、はさみ、ETCニーズいたしません。手袋の二重重ね着してください! - 50℃で30分のための4 × SSC中でゲルを洗浄してください新鮮な洗浄バッファーで交換して、二回繰り返す。
- 50℃で4 × SSC/0.1%SDSでゲルを洗浄℃に
- 3ミリメートルワットマンの紙の上にゲルを入れ、軽くゲルを乾燥させる。
- ラップでゲルをラップして〜3日間phosphorimagerにさらす
- phosphorimagerをスキャン
- 室温で30分間変性バッファー(1.5 MのNaCl、0.5MのNaOH)にゲルをインキュベート
- 中和バッファーでゲルを洗浄する(3 MのNaCl、0.5 Mトリス•/ ClのpHは7.0)を室温で30分間
- 3分間蒸留水ですすぎ
- 1時間プレハイブリダイズする55 ° C
- 同じプローブを含有する55℃で一晩ハイブリダイゼーションミックスを用いてハイブリダイゼーションを繰り返します。
- 55を除いて、上記のように洗う℃を
- ゲルをラップして〜2時間phosphorimagerにさらす。
- phosphorimagerをスキャンします。変性後に得られたものと変性する前に得られるハイブリダイゼーションシグナルを正規化する。
2。アダプターライゲーション6により塩基テロメアターミナルを決定する
原理(図2Aおよび2B):5 G -オーバーハングの終わり'末端標識されたユニークなオリゴヌクレオチドは、3に連結されている"。これは、特定の補完的なガイドのオリゴにアニーリングすることによって達成される。テロメアG -オーバーハング末端配列に互換性のある唯一のユニーク/ガイドオリゴアダプターのベアリングの配列を連結されます。 T.用トリパノソーマ 、テロメアは、TTAGGG反復配列の異なる6つのヌクレオチドのいずれかで終了することができます。したがって、6種類のガイドoligoesは、(TG1 - TG6、図2A)が使用されます。ライゲーション後、DNAを制限酵素で消化し、アガロースゲルで分離。
- ユニークなオリゴのキナーゼ処理。
10倍PNKバッファの3μLを10ピコモル/μLユニークなオリゴの6μLを混合し、5μLγ[32 P] ATP、のddH 2 Oの14μL、およびT4 PNKの2μL(10 U)。 37℃で1時間混合物をインキュベートする。 - ヌクレオチドホット法人を削除します。
非法人のホットATPを除去し、溶出バッファー60μL(終濃度は〜1ピコモル/μLとなる)で末端標識オリゴを溶出するQIAクイックヌクレオチド除去キットを使用してください。 - ガイドオリゴへのユニークなオリゴを(アダプタを作成する)アニール。
各ガイドオリゴと1μLアニーリング緩衝液(200mMのNaCl、100mMトリス- HCl pH8.0)を2μLにユニークなオリゴを精製の10μLを追加。 85℃のヒートブロックにチューブを入れ、ヒートブロックをオフにしてください。 RTに冷却管とヒートブロックをしてみましょう。これは数時間かかります。もし急いで、トランスファーチューブの65〜5分間、それぞれの後° C、その後37℃、次いで室温にそれが冷えてから。 - トータルゲノムDNAにアダプターをライゲーションする。
インタクトなゲノムDNAの2.5μLに、5 ×リガーゼバッファーの4μL、アニールされたオリゴヌクレオチド、のddH 2 O 2.5μL、およびT4 DNAリガーゼの1μLの10μLを加える。 16℃で一晩。 - 制限エンドヌクレアーゼ消化
ライゲーション産物20μLの10 × NEB緩衝液4の3μL、ALUIとMboI各1.5μL、とのddH 2 Oを4μLを加える37℃で一晩混合物をインキュベートする。 - 電気泳動。
各サンプルに10 ×オレンジG色素(50%グリセロール、0.5%オレンジG)の3μLを加える。は0.2μg/ mlのエチジウムブロマイドを0.5 × TBEで20 × 20センチメートル0.7%アガロースゲルにDNAサンプルをロードします。その後、30分間30Vで動作しますCより高い電圧が1000 V時間の合計に120V未満のontinue。 DNAマーカーの隣に定規を持つゲルの写真を撮る。 - 乾燥ゲルと公開。
DE81ろ紙および3 mmワットマン濾紙の二つの層の層の上にアガロースゲルを入れ、ラップでゲルをカバー。室温で乾燥したゲル。一晩phosphorimagerに乾燥ゲルを公開。
3。ライゲーション媒介プライマー伸長6
原理(図2Aおよび2C):ユニークなオリゴヌクレオチドは、アダプタにその相補的な配列によって決定される特定の末端配列、(ガイド)オリゴと3'G -オーバーハングに連結される。精製した後、G-overhang/uniqueオリゴにアニール残るというラベルの付いたガイドのオリゴは、鎖置換および5' - 3'エキソヌクレアーゼ活性を欠くT4 DNAポリメラーゼを使用して、SS - DSの接合に拡張プライマーです。プライマー伸長産物は、したがって、G -オーバーハングの長さを与える。
- ガイドとユニークなoligoesのキナーゼ処理
- 10 × PNKバッファ、20μLの5 ×リガーゼバッファー(10 mM ATPを含む)、T4 PNKの3μL(30 U)、とのddH 2 Oの47μLを10μLを10ピコモル/μLユニークなオリゴの20μLを混合。 37℃で60分間混合物をインキュベートする。
- 10 × PNKバッファ、2μLの1μLを10ピコモル/μLガイドオリゴの1μLを混合γ[32 P] ATP、T4 PNKの1μL(10 U)、とのddH 2 Oの5μL 37℃で60分間混合物をインキュベートする。
- ガイドオリゴとユニークなオリゴをアニーリングし
1.5mlのエッペンドルフチューブで末端標識ガイドオリゴ(オリゴを導くためにユニークなの2:1の比率がすべてのガイドオリゴはアニールされることが保証されるのリン酸化ユニークなオリゴおよび10μL(10ピコモル)を10μL(20ピコモル)を追加)。 oligoesをアニールするためのプロトコルIIで説明したのと同じ手順に従ってください。 - テロメアの末端にアニールしたガイド/ユニークなオリゴアダプターをライゲーション
DNAのライゲーション用プロトコールIIに記載されて同じ手順に従ってください。 - 沈殿物の酢酸ナトリウムの1 / 10量とDNAと-80℃の氷冷エタノールの2.5容℃で30分間。 15分で12 krpmの、4℃でDNAペレットをスピンダウンします。エタノールの70%でDNAを洗浄してのddH 2 O10μlにパレットを溶かす
- プライマー伸長
1 mg / mLのBSA、25mMのdATPを、dCTPの1μL、及びdTTP各、T4 DNAポリメラーゼの1μL(3 U)、1μL、10 × T4 DNAポリメラーゼ緩衝液2μLを追加するたびに10μLのDNAサンプルへとのddH 2 O 3μL
ポリメラーゼ、緩衝液、等と各DNAサンプルにマスターミックスのアリコートを適切な量のマスターミックスを作る。 30℃で30分間インキュベートします。その後、反応にすぐに0.5M EDTAを1.2μLを加える。 - 電気泳動
10%アクリルアミド/ 7 M urea/1xTBEゲルに拡張製品を実行します。各サンプルの4μLをロードする。ロードの前に10分間のサンプルを煮沸してください。青色の色素がゲルの底に達するまで、1 × TBEで800Vで動作します。 65℃でゲルを乾燥させ室温で30分間、30分間、2時間phosphorimagerにさらす。
10%ポリアクリルアミドゲル100mLを準備するには、10倍TBEの10 mLを加え、蒸留水30mLに尿素41gのと40%アクリルアミド溶液(アクリルアミド/ビスアクリルアミド29:1)21 mLを溶かす。ガラスプレートを組み立てる。右のゲルを注ぐ前に、10%APSとTEMEDを100μLの1 mLを加える。ゲルを注入し、気泡を避けるようにしてください。
4。代表的な結果:
T.を検出ネイティブゲル内ハイブリダイゼーションを用いたトリパノソーマテロメアG -オーバーハング構造
そのままT. PFGEで分離されたトリパノソーマ染色体は、図3Aおよび3B(左のパネル)に示されている。T.はトリパノソーマの細胞は、通常、ミニ染色体の約100枚、すべて同じようなサイズで(50〜150キロバイト)を含んでおり、ゲル上の同じ位置(MC)に移行します。この事実のために、テルチオリゴプローブとのハイブリダイゼーション後、テロメアG -オーバーハングの信号は、ミニ染色体(図3A、中央のパネル)で最も顕著です。 TELGオリゴプローブとのハイブリダイゼーションは、通常、ハイブリダイゼーションシグナルが得られない(図3B、中央のパネル)ので、T.トリパノソーマの細胞はテロメアC -オーバーハング構造を持っていない。変性および中和した後、テルチまたはTELGオリゴプローブのいずれかとのハイブリダイゼーションは、すべての染色体末端(図3A&3B、右パネル)のテロメアシグナルを明らかにする必要があります。
アダプターライゲーションにより塩基テロメア端末を決定する
各レーンでのDNAの同量をロードすると、このアッセイのために不可欠であり、これはゲルのエチジウムブロマイド染色によって示されています。例を図4A、左側のパネルに表示されます。
テロメアG -オーバーハングガイドオリゴと互換性のあるシーケンスで終了するときにこのプロトコルでは、末端標識ユニークなオリゴとガイドオリゴアダプタは、染色体末端に連結されています。 Siのユニークなオリゴの末端標識であるNCEは、ライゲーション産物は、放射性になると強いシグナルを与える。図4A、右側のパネルに示すように、レーン1はunique/TG1アダプタ大量のテロメア末端に連結されていることを示す、強力な信号を与える。 TG1は"5'CCCTAA3の終了シーケンスを持っています。したがって、テロメアG -オーバーハングは5'TTAGGG3"でこのアダプタの端に連結。ライゲーション産物の有意な量は、TTAGGGで終わるテロメアはTで優勢であることを示し、他のガイドのoligoesを使用して観察されないトリパノソーマの細胞。
3'オーバーハング特定のヌクレアーゼであるエキソIまたはエキソT、でゲノムDNAを扱う、ライゲーションは、実際にテロメアに起因することを示す、ライゲーション産物の損失(図4A、右側のパネル、レーン2及び3)の結果、G -オーバーハング構造
ライゲーション媒介プライマー伸長
結紮することなく、末端標識ガイドオリゴは、22ヌクレオチドの長です。ローディング終了標識ガイドオリゴは、ネガティブコントロールとサイズマーカーとして役立つ。 (図4B、レーン11、アスタリスクは、末端標識ガイドオリゴを示します)。我々は通常、さらに可能性が最も高い残留非変性のアダプタであるこのネガティブコントロール(図4B、レーン11、オープンの三角形)、に〜44ヌクレオチドのバンドを観察。染色体末端にライゲーションした後、拡張された製品のサイズは、G -オーバーハング構造の長さを反映している。ほとんどのT. 5'TTAGGG3のトリパノソーマテロメアG -突出末端の終わり"(図4A)。連結されたTG1ガイドオリゴから拡張製品はガイドオリゴ自体(図4B、レーン10)よりもはるかに長くはない、しかし、彼らはのライゲーションを可能にするしかし、これらのG -オーバーハングは、(のみ〜10ヌクレオチドの長)非常に短いように見えるTG1アダプタ(図4A、右側のパネル、レーン1)。 TG4アダプタのわずかな量がテロメアの両端(図4A、右側のパネル、レーン6)に連結したものの、結紮TG4から拡張製品は、はるかに長い(図4B、レーン7、矢頭)であり、〜という長い製品と55 NT。したがって、我々はT.でテロメアの2種類のG -オーバーハングを観察トリパノソーマの細胞。しかし5'TTAGGG3"における支配的なテロメアG -突出末端のエンドは、長いだけ〜10 ntのです。 5'GGGTTA3の少数テロメアG -突出末端の終わり"が、長い40 NT件まですることができます。 G -オーバーハングの両方の種類のエキソT(またはエキソI)治療(、図4B、レーン1、矢印図4A、右側のパネル、レーン2、3、7)に敏感です。

図1。ネイティブゲル内のハイブリダイゼーションの原理。左は、ネイティブの条件の下、末端標識Telcのオリゴプローブは一本鎖G -リッチなテロメアオーバーハングとハイブリダイズすることができる。ハイブリダイゼーションの強度はテロメアG -オーバーハングの長さを反映している。右、変性後、Telcのオリゴプローブは、すべてのテロメアDNAとハイブリダイズする。このハイブリダイゼーションの強度は、テロメアDNAの総量を表し、ローディングコントロールとして使用され、そして最後のG -オーバーハングのレベルは、変性後に得られたことにより、ネイティブのハイブリダイゼーションシグナルを割って算出されます。
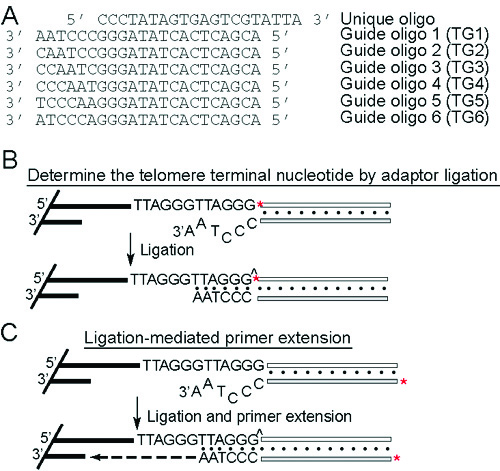
図2。アダプターのライゲーションとライゲーション媒介プライマー伸長の原理。()ユニークと6つのガイドoligoesのシーケンス。 (B)アダプターライゲーションにより塩基テロメア端末を決定します。ユニークなオリゴは、末端標識(赤色のアスタリスクが付いている)、ガイドのoligoesにアニーリングし、テロメア末端に連結した。ユニーク/ガイドアダプタは、端末のテロメアのシーケンスアダプターが連結されるとの互換性がある3'オーバーハングを負うときのみ。 (C)ライゲーション媒介プライマー伸長では、ガイドオリゴは末端標識(赤色のアスタリスクが付いている)と染色体の末端に連結する前にユニークなオリゴにアニール。 G -オーバーハングと互換性の3'オーバーハングをもつ唯一のユニークな/ガイドアダプターをライゲーションされます。 ^連結されたphophordiesterの結合を示す。 unligatedオリゴのペアを除去した後、プライマー伸長は、鎖置換および5' - 3'エキソヌクレアーゼ活性を欠くT4 DNAポリメラーゼで実施される。拡張ガイドオリゴの最終的な長さは、G -オーバーハングの長さを反映している。
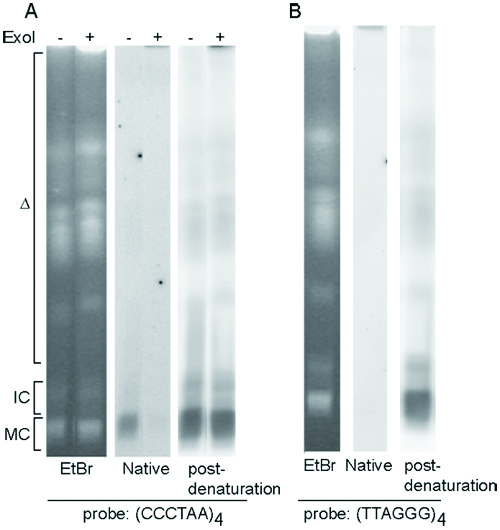
図3。T.ハイブリダイゼーション。()Telcのオリゴプローブとのハイブリダイゼーション、ゲルゲル内でネイティブによって分析トリパノソーマテロメアG -オーバーハング構造 。 TELGオリゴプローブと(B)ゲル内ハイブリダイゼーション。の両方(A)と(B)左、Ethediumブロマイド染色PFG。中央の、ネイティブのハイブリダイゼーションの結果。右、後の変性のハイブリダイゼーションの結果。野生型T.トリパノソーマの細胞が使用されていました。私は、ゲノムDNAを扱う無傷またはエキソはPFGEにより分離した。オープン三角形はメガベースの染色体の略で、IC、中間サイズの染色体、MC、ミニ染色体。
"/>
図4。T.ライゲーション媒介プライマー伸長によって分析トリパノソーマテロメアG -オーバーハング構造。()ほとんどのT.トリパノソーマテロメアG -オーバーハングは5'TTAGGG3"で終了する。左、Ethediumブロマイド染色したDNAゲル。 DNAのほぼ等しい量は、各レーンにロードされます。右、乾燥後に同じゲルの露光結果。そのまま、エキソI -治療、またはエキソT -処理されたDNAが示すように6種類のガイド/ユニークなオリゴアダプタ末端標識に連結した。 (B)T.トリパノソーマのテロメアは短くG -オーバーハングを持っている。無傷またはエキソT処理したゲノムDNAは、T4 DNAポリメラーゼを使用して、プライマー伸長が続く、6種類のガイド/ユニークなオリゴアダプターに連結した。末端ラベルされたTG4オリゴは、ネガティブコントロール(レーン11)としてロードされました。オリゴ自体は、22ヌクレオチド(*)で動作しますが、も〜44 NT(Δ)で暗いのシグナルを与えた。唯一unique/TG4アダプタが大幅に長くガイドオリゴ自体(矢頭、レーン7)より伸長生成物を得た。
ディスカッション
トリパノソーマはヒトの睡眠病を引き起こす。この病気は、放置すれば、必然的に致命的である。T.は哺乳動物宿主のトリパノソーマの細胞は、定期的に宿主の免疫攻撃7を回避するために抗原変異を起こす。それゆえにT.を排除することは非常に困難です感染かつてトリパノソーマの細胞が確立されます。我々は最近、テロメアがTの発現の調?...
開示事項
謝辞
我々は、科学的な議論や洞察力の提案のために博士キャロリンの価格を感謝したいと思います。この作品は、NIHの助成金AI066095(:畢博リチウムPI)によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| SeaKem LE Agarose | Lonza Inc. | 50004 | |
| Agarose Type VII (low melting agarose) | Sigma-Aldrich | A4018 | |
| Proteinase K, recombinant, PCR grade | Roche Group | 03 115801001 | |
| Exo I | New England Biolabs | M0293 | |
| Exo T | New England Biolabs | M0265 | |
| T7 exonuclease | New England Biolabs | M0263 | |
| T4 DNA polymerase | New England Biolabs | M0203 | |
| QIAquick nucleotide removal kit | Qiagen | 28304 |
参考文献
- Munoz-Jordan, J. L., Cross, G. A., de Lange, T., Griffith, J. D. t-loops at trypanosome telomeres. EMBO J. 20, 579-588 (2001).
- Li, B., Espinal, A., Cross, G. A. M. Trypanosome telomeres are protected by a homologue of mammalian TRF2. Mol Cell Biol. 25, 5011-5021 (2005).
- Wellinger, R. J., Wolf, A. J., Zakian, V. A. Saccharomyces telomeres acquire single-strand TG1-3 tails late in S phase. Cell. 72, 51-60 (1993).
- McElligott, R., Wellinger, R. J. The terminal DNA structure of mammalian chromosomes. EMBO J. 16, 3705-3714 (1997).
- Harrington, L. A., Greider, C. W. Telomerase primer specificity and chromosome healing. Nature. 353, 451-454 (1991).
- Jacob, N. K., Skopp, R., Price, C. M. G-overhang dynamics at Tetrahymena telomeres. Embo J. 20, 4299-4308 (2001).
- Barry, J. D., McCulloch, R. Antigenic variation in trypanosomes: enhanced phenotypic variation in a eukaryotic parasite. Adv Parasitol. 49, 1-70 (2001).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137, 99-109 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved