Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
L'examen des télomères G-structure en porte à faux Trypanosoma brucei
Dans cet article
Résumé
Les télomères sont essentiels pour la stabilité des chromosomes et le G-télomères surplombent la structure est essentielle pour la télomérase médiée par le maintien des télomères. Nous avons récemment adopté deux méthodes pour détecter les télomères G-faux structure dans Trypanosoma brucei, Qui sont natifs en gel d'hybridation et la ligature médiée par extension d'amorce, qui seront décrits.
Résumé
Le G-télomères surplombent structure a été identifiée chez les eucaryotes, y compris de nombreuses levures, les vertébrés, et Trypanosoma brucei. Elle sert de substrat pour la télomérase pour la synthèse de novo d'ADN des télomères et est donc important pour le maintien des télomères. T. brucei est un parasite protozoaire qui provoque la maladie du sommeil chez les humains et le nagana chez les bovins. Une fois infecté hôte mammifère, T. cellulaire brucei commutateurs régulièrement ses antigènes de surface pour échapper à l'attaque immunitaire de l'hôte. Nous avons récemment démontré que le T. brucei télomères structure joue un rôle essentiel dans la régulation de l'expression du gène antigène de surface, ce qui est essentiel pour T. pathogenèse brucei. Toutefois, T. brucei télomères structure n'a pas été étudiée en raison de la limitation des méthodes d'analyse de cette structure spécialisée. Nous avons maintenant adopté avec succès le natif de gel-hybridation et ligation médiée par des méthodes d'extension d'amorces pour l'examen des télomères G-faux structure et une méthode de ligature adaptateur pour la détermination de la borne télomères nucléotides dans T. cellules brucei. Ici, nous allons décrire les protocoles en détail et comparer leurs avantages et leurs limites.
Protocole
1. Détecter T. brucei télomères G-faux à l'aide maternelle hybridation in-gel 3
Principe (Figure 1): Sous la condition indigène, une fin marquée (CCCTAA) 4 (TELC) oligo sonde ne peut s'hybrider avec le télomère simple brin G-faux région. L'intensité d'hybridation est proportionnel à la longueur du surplomb. Après dénaturation et de neutralisation, la même sonde sera capable de s'hybrider à la région des télomères ensemble. L'intensité d'hybridation représente le montant total des télomères de l'ADN et peut être utilisé comme un contrôle du chargement. Deux contrôles standard pour cette expérience sont d'hybridation utilisant marqués aux extrémités (TTAGGG) 4 (Telg) oligo sonde, qui ne doit pas céder tout signal que T. cellulaire brucei n'a pas télomères C-faux la structure, et de traiter l'ADN génomique avec 3'-spécifiques simple brin exonucléases telles que Exo Exo I ou T avant l'hybridation, ce qui devrait éliminer le signal d'hybridation natif TELC.
Etape 1. Préparer les bouchons d'ADN pour électrophorèse en champ pulsé (PFGE)
Chromosomes intacts seront séparés par électrophorèse en champ pulsé. Par conséquent, l'ADN génomique est préparé comme les bouchons d'ADN.
- Commencez avec 100 ml de cellules forme sanguine (1,5 -2.000.000 cellules / ml) ou 20 ml de cellules procycliques (à 10 millions de cellules / ml).
- Dissoudre 1,6% à faible point de fusion (LMP) agarose dans du tampon L (0,01 M Tris • CL pH 7,6, 0,02 M de NaCl, 0,1 M EDTA pH 8,0) et le garder au chaud à 50 ° C.
- Récolte des cellules par centrifugation à 1,5 kRPM, 4 ° C pendant 10 min. Retirer le surnageant et remettre le culot cellulaire avec un tampon de L à une concentration finale de 5 millions de cellules / mL.
- Incuber suspension cellulaire à 42-50 ° C pendant 10 min.
- Ajouter 1 volume de agarose LMP pour 1 volume de cellules. Mélangez bien, mais rapidement.
- Aliquoter 85 uL de suspension cellulaire dans chaque puits d'un moule branchez jetables (BioRad).
- Après 5-10 min ou jusqu'à ce que les fiches sont solidifiées, les bouchons transférer dans un tube 15 ml conique avec 5 tampons L mL / Sarkosyl 1%. Ajouter 100 uL de 250 mg / ml de protéinase K ou une pincée de poudre de protéinase K, bien mélanger et laisser incuber à 50 ° C pendant 48 heures.
- Jeter le tampon, se laver les bouchons avec 5 ml de tampon L à deux reprises et répétez la protéinase K de digestion pour un autre 48 heures. Lavez les bouchons avec un tampon de l fraîche encore. Magasin de bouchons à 4 ° C dans un tampon de L. Ces bouchons devraient être utilisés dans les 2 semaines.
Etape 2. Séparée intacte T. chromosomes brucei utilisant électrophorèse en champ pulsé
- Préparer un gel d'agarose
- Préparer 2-3 l de 0,5 x tampon TBE et le transfert de la chambre de gel de fonctionnement d'un CHEF DRII (BioRad) l'unité. Réglez la température de fonctionnement, démarrer la pompe, puis démarrer l'unité de refroidissement.
- Préparer 100 ml d'un gel d'agarose 1,2% en 0,5 x tampon TBE. Refroidir agarose lors de la configuration du plateau de gel.
- Décidez de l'ordre de l'échantillon. N'oubliez pas d'inclure la norme ADN. Sécher le peigne et le poser sur une surface plane. Placez chaque plug ADN sur la dent appropriée. Retirer du tampon autour de la prise excessive de l'ADN par l'aspiration de sorte que le connecteur se coller à la dent.
- Verser le gel d'agarose sans insérer le peigne. Laissez refroidir à 40-50 ° C (quelques minutes), insérez lentement le peigne avec les bouchons attachés dans le gel. Assurez-vous que les bouchons ne pas glisser le peigne. Laissez le gel se solidifier. Retirer du gel peigne lentement.
- Électrophorèse en champ pulsé
Exécuter un gel dans une unité CHEF DRII: 700 s-1500 s impulsions, 2,5 V / cm, à 12 ° C pendant 120 heures.
Étape 3. Taches et Décolorer le gel d'agarose
Tache le gel avec 1 pg / ml de bromure d'éthidium dans 0,5 x TBE à température ambiante pendant 1 heure puis à 0,5 x TBE, sans bromure d'éthidium pour 1 heure avec doux balancement. Prenez une photo du gel.
Étape 4. Sécher le gel d'agarose
Placer le gel d'agarose sur deux couches de 3 mm papier Whatman et couvrir le gel avec une pellicule de plastique. Sécher le gel à température ambiante (le gel ne doit pas être chauffé à plus de 50 ° C pour éviter toute dénaturation des échantillons d'ADN). Remplacer le papier Whatman mouillés avec ceux sec après 2 et 4 heures de séchage, respectivement. Gardez le séchage jusqu'à ce que le gel est complètement sec. Cela peut prendre du jour au lendemain en fonction de la sécheuse.
Étape 5. Marquer les sondes oligo
Mélanger 1 uL de 50 ng / uL TELC ou oligo Telg avec 1 ul de 10 x kinase (PNK) tampons polynucléotide, 5 pi de γ [32P] ATP, 2 pl de DDH 2 O et 1 pl de PNK T4. Incuber à 37 ° C pendant 1 heure. Ajouter 90 ul de tampon TNES (10 mM Tris Cl, pH 8,0, NaCl 100 mM, EDTA 10 mM, pH 8,0, SDS 1%) au mélange réactionnel.
Bien que l'étiquetage de la oligoes, préparer une colonne G-25: mettre un peu de laine de verre dans une seringue de 3 cc et le farcir avec un bas serré pointe de la pipette. Remplir la seringue avec de Sephadex G-25 fine (autoclAved en TE) pour la marque de 3 mL. Laisser reposer en poids. Pour purifier la sonde marquée: la charge de la sonde sur le haut de la colonne. Laver avec 700 uL de tampon TNES, et éluer avec 600 uL de tampon TNES. Alternativement, purifier la sonde marquée en utilisant un mini-Spin ™ rapide colonne (Roche Applied Science).
Étape 6: Hybridation
- Brièvement mouiller le gel séché dans de l'eau distillée et laver tout papier Whatman coincé sur le gel
- Placer le gel dans un sac d'hybridation et ajouter 20 ml de tampon hybridiztion (0,25 M Na 2 HPO 4 pH 7,2, 1 mM EDTA, 7% SDS, 1% de BSA, filtrée à travers du filtre de 0,22 um). Incuber dans un bain-marie à 50 ° C avec doux balancement pendant au moins 1 heure.
- Retirer le tampon de pré-hybridation. Faire bouillir la sonde marquée pendant 5 min et ajoutez 25 ml de tampon d'hybridation douce, filtre à stériliser le mélange avec un filtre de 0,22 um seringue et ajouter le mélange d'hybridation directement dans le sac d'hybridation. Scellez le sac et le placer dans un récipient peu profond avec de l'eau tiède. Placez le récipient entier dans l'eau du bain. Incuber à 50 ° C pendant la nuit avec des doux balancement.
- Couper une petite ouverture dans le sac de l'hybridation. Verser autant que possible le mélange d'hybridation et de l'enregistrer dans un tube conique de 50 ml. Maintenir la sonde congelé à -20 ° C. Retirer le gel du sac d'hybridation et le mettre dans un récipient.
À cette étape TOUT SERA CHAUD ET BESOINS un nettoyage en profondeur (banc, ECRAN, ciseaux, ETC. S'assurer de porter deux épaisseurs de gants!! - Laver le gel dans 4 x SSC pendant 30 min à 50 ° C. Remplacez-le par un tampon de lavage fraîche et répéter deux fois.
- Laver le gel en 4 x SSC/0.1% SDS à 50 ° C.
- Mettre le gel sur un morceau de papier Whatman 3 mm et légèrement sécher le gel.
- Envelopper le gel avec une pellicule plastique et pour les exposer à phosphorimager ~ 3 jours
- Analyser le phosphorimager
- Incuber le gel dans un tampon de dénaturation (1,5 M de NaCl, NaOH 0,5 M) à température ambiante pendant 30 min
- Laver le gel dans un tampon de neutralisation (3 M de NaCl, 0,5 M Tris • pH / Cl 7,0) à température ambiante pendant 30 min
- Rincer à l'eau distillée pendant 3 min
- Pré-hybridation à 55 ° C pendant 1 heure
- Répétez l'hybridation avec la sonde contenant la même mélange d'hybridation à 55 ° C pendant la nuit.
- Laver comme décrit ci-dessus, sauf à 55 ° C.
- Envelopper le gel et exposer à phosphorimager pour ~ 2 heures.
- Analyser le phosphorimager. Normaliser le signal d'hybridation obtenus avant dénaturation à celle obtenue après dénaturation.
2. Déterminer le terminal des télomères par 6 nucléotides ligature adaptateur
Principe (Figure 2A et 2B): Une extrémité 5 'oligonucléotide marqué unique est ligaturé à l'extrémité 3' du G-faux. Ceci est accompli par un recuit d'oligo guide spécifique complémentaire. Seule l'unique guide / séquences oligo portant adaptateur compatible avec les séquences télomériques borne G-faux sera ligaturé. Pour T. brucei, les télomères peut se terminer dans l'un des six nucléotides différents des répétitions TTAGGG. Ainsi six oligoes Guide différentes sont utilisées (TG1-TG6, figure 2A). Après ligature, l'ADN est digéré par les endonucléases de restriction et résolus sur un gel d'agarose.
- Le traitement de l'oligo Kinase unique.
Mélangez 6 ul de 10 pmol / uL oligo unique avec 3 pi de tampon PNK 10x, 5 pi de γ [32 P] ATP, 14 uL de DDH 2 O, et 2 uL (10 U) de PNK T4. Incuber le mélange à 37 ° C pendant 1 heure. - Retirez non constituée en société nucléotide chaud.
Utilisez QIAquick retrait nucléotide pour supprimer la personnalité morale chaude ATP et éluer les oligo fin étiquetés avec 60 uL de tampon d'élution (concentration finale serait ~ 1 pmole / uL). - Recuit oligo unique pour guider oligo (pour créer l'adaptateur).
Ajouter 10 ul d'oligo purifiée unique de 2 pi de chaque oligo guide et 1 pi de tampon de recuit (200 mM NaCl, 100 mM Tris-HCl pH 8,0). Mettez les tubes dans un bloc de 85 ° C chaleur et d'éteindre le bloc thermique. Laissez les tubes et bloquer la chaleur refroidir à température ambiante. Cela va prendre quelques heures. Si, dans un tube de transfert pressé, après 5 minutes chacune à 65 ° C, puis 37 ° C, puis laisser refroidir à température ambiante. - Adaptateurs Ligate au total ADN génomique.
Pour 2,5 uL d'ADN génomique intact, ajoutez 4 pi de 5 x Ligase tampon, 10 uL d'oligos recuit, 2,5 pi de ddH 2 O et 1 ul d'ADN ligase T4. Incuber à 16 ° C pendant la nuit. - Digestion par des endonucléases de restriction
À 20 ul du produit de ligature ajouter 3 ul de 10 tampon NEB 4 x, 1,5 ul de AluI et Mbol chacune, et 4 pl de DDH 2 O. Incuber le mélange à 37 ° C pendant la nuit. - L'électrophorèse.
Ajouter 3 pi de 10 x orange colorant G (glycérol 50%, 0,5% d'Orange G) pour chaque échantillon. Chargez les échantillons d'ADN sur un 20x20 cm à 0,7% en gel d'agarose TBE 0,5 x avec 0,2 pg / ml de bromure d'éthidium. Run à 30V pendant 30 min, puis continuer avec une tension plus élevée, mais moins de 120 pour un total de 1000 v hr. Prenez une photo du gel avec une règle à côté du marqueur d'ADN. - Gel sec et exposer.
Mettez le gel d'agarose sur une couche de papier filtre DE81 et deux couches de 3 mm papier Whatman et couvrir le gel avec une pellicule de plastique. Gel sec à température ambiante. Exposer le gel séché au phosphorimager nuit.
3. Extension d'amorce Ligation Mediated 6
Principe (Figure 2A et 2C): Un oligonucléotide unique est ligaturé à l'extrémité 3 'du G-faux avec séquence terminal spécifique, déterminé par ses séquences complémentaires dans l'adaptateur (guide) oligo. Après purification, les oligo Guide étiquetés qui reste recuit pour les oligo G-overhang/unique n'est amorce étendue à la jonction ss-DS en utilisant la T4 ADN polymérase, qui manque de déplacement de brin et les activités d'exonucléase 5'-3 '. Les produits d'extension d'amorces donc donner la longueur de la G-faux.
- Le traitement de l'Kinase oligoes guide et uniques
- Mélanger 20 ul de 10 pmol / uL oligo unique avec 10 uL de tampon de 10 x PNK, 20 pl de tampon ligase 5 x (qui contient 10 mM ATP), 3 pi (30 U) de PNK T4, et 47 uL de DDH 2 O . Incuber le mélange à 37 ° C pendant 60 minutes.
- Mélanger 1 ul de 10 pmol / guide uL oligo avec 1 ul de tampon de 10 x PNK, 2 pl de γ [32 P] ATP, 1 pi (10 U) de PNK T4, et 5 pi de DDH 2 O. Incuber le mélange à 37 ° C pendant 60 minutes.
- Recuit Guide oligo et unique oligo
Ajouter 10 uL (20 pmoles) de l'oligo phosphorylée uniques et 10 uL (10 pmoles) de l'extrémité marquée oligo guider dans un tube Eppendorf de 1,5 ml (le ratio de 2:1 de unique pour guider oligo assure que tous les oligo guide est recuit ). Suivez la même procédure décrite dans le Protocole II pour le recuit oligoes. - Ligaturer le guide recuit / adaptateur oligo uniques à des fins télomères
Suivez la même procédure décrite dans le Protocole II à la ligature d'ADN. - L'ADN précipité avec 1 / 10 volume d'acétate de sodium et 2,5 volume d'éthanol glacé à -80 ° C pendant 30 min. Isoler culot d'ADN à 12 kRPM, 4 ° C pendant 15 min. Laver l'ADN avec 70% d'éthanol et de dissoudre palette dans 10 uL de DDH 2 O.
- Extension d'amorce
Pour chaque échantillon d'ADN 10 uL ajouter 2 uL de 10 x ADN polymérase T4 de tampon, 1 pl de 1 mg / ml de BSA, 1 pl de 25 mM, dATP dCTP et dTTP chacun, 1 ul (3 U) de l'ADN polymérase T4, et 3 pl de DDH 2 O.
Faites un mélange maître avec la polymérase, tampon, etc et la quantité aliquote appropriée du mélange maître à chaque échantillon d'ADN. Incuber à 30 ° C pendant 30 min. Ensuite, ajoutez 1,2 uL d'EDTA 0,5 M immédiatement à la réaction. - L'électrophorèse
Exécuter des produits d'extension sur un acrylamide à 10% / 7 M urea/1xTBE gel. Charge 4 ul de chaque échantillon. Faire bouillir les échantillons pendant 10 minutes avant le chargement. Run à 800V en 1x TBE jusqu'à ce que le colorant bleu atteint le bas du gel. Sécher le gel à 65 ° C pendant 30 min puis à TA pendant 30 min et exposer à phosphorimager pendant 2 heures.
Pour préparer 100 ml de gel de polyacrylamide 10%, dissoudre 41 g d'urée et 21 ml de solution d'acrylamide à 40% (acrylamide / bis-acrylamide 29:1) dans 30 ml d'eau distillée, ajouter 10 ml de TBE 10x. Assemblez les plaques de verre. Juste avant de verser le gel, ajouter 1 mL de l'APS de 10% et 100 pi de TEMED. Verser le gel et essayer d'éviter les bulles.
4. Les résultats représentatifs:
Détecter T. brucei télomères G-faux structure à l'aide maternelle dans une hybridation-gel
Intact T. brucei chromosomes séparés par PFGE sont présentés dans la figure 3A et 3B (panneaux de gauche). T. brucei cellules contiennent normalement ~ 100 exemplaires de minichromosomes, tous avec la même taille (50-150 kb) et migrer vers la même position sur le gel (MC). De ce fait, après hybridation avec la sonde oligo TELC, le G-télomères faux signal est le plus important sur les minichromosomes (figure 3A, le panneau du milieu). L'hybridation avec la sonde oligo Telg n'a normalement pas donné de signal d'hybridation (figure 3B, panneau du milieu), parce que T. brucei cellules n'ont pas de télomères C-faux structure. Après dénaturation et de neutralisation, l'hybridation avec soit TELC ou Telg oligo sonde devrait révéler les signaux sur tous les télomères extrémités des chromosomes (figure 3A et 3B, les panneaux de droite).
Déterminer le terminal télomères nucléotide par ligature adaptateur
Chargement quantité égale de l'ADN dans chaque couloir est essentielle pour ce test, et cela est démontré par coloration au bromure d'éthidium du gel. Un exemple est montré dans la figure 4A, le panneau gauche.
Dans ce protocole, la fin marqués uniques oligo et guider les adaptateurs ne sont que des oligo ligaturé à l'extrémité des chromosomes lors de la G-télomères surplombe terminant par des séquences qui sont compatibles avec les oligo guide. Si nce les oligo unique est étiqueté fin, le produit de ligature sera radioactifs et de donner un signal fort. Comme le montre la figure 4A, le panneau de droite, la piste 1 donne un signal fort, indiquant qu'une grande quantité de unique/TG1 adaptateur a été ligaturé à la fin des télomères. TG1 a une séquence de fin d'5'CCCTAA3 ». D'où le G-télomères surplombe ligaturé à cette fin l'adaptateur dans 5'TTAGGG3 ». Aucune quantité importante de produits de ligation est observée à l'aide oligoes autre guide, indiquant que les télomères se terminant en TTAGGG sont prédominants dans T. cellules brucei.
Traiter de l'ADN génomique avec Exo Exo I ou T, qui sont faux nucléases spécifiques 3 ', a entraîné la perte des produits ligature (figure 4A, le panneau de droite, lignes 2 & 3), indiquant que la ligature effet résulte de la G-télomères structure de faux
Extension d'amorce Ligation Mediated
Sans ligature, la fin marqués Guide oligo est de 22 nt longtemps. Chargement fin étiquetés Guide oligo servirait comme un contrôle négatif et un marqueur de taille. (Figure 4B, la piste 11, astérisque indique la fin marqués Guide oligo). Normalement, nous observons également une bande de ~ 44 nt dans ce contrôle négatif (figure 4B, la piste 11, le triangle ouvert), qui sont les plus susceptibles de résidus non dénaturé adaptateurs. Après ligature de la fin des chromosomes, la taille du produit étendu reflète la longueur du G-faux structure. La plupart des T. brucei G-télomères surplombe fin en 5'TTAGGG3 '(figure 4A). Cependant, ces G-surplombs semblent être très courte (seulement ~ 10 nt de long), en tant que produits s'étend de la ligature des oligo TG1 guide ne sont pas beaucoup plus longtemps que les oligo guide lui-même (figure 4B, Lane 10), mais ils permettent la ligature des TG1 adaptateur (figure 4A, le panneau de droite, piste 1). Bien que seulement une petite quantité de TG4 adaptateur a été ligaturés aux extrémités des télomères (figure 4A, le panneau de droite, piste 6), les produits étendue de TG4 ligaturé sont beaucoup plus longs (figure 4B, piste 7, tête de flèche), avec la plus longue étant de produit ~ 55 nt. Par conséquent, nous avons observé deux types de G-télomères faux dans T. cellules brucei. Le G-télomères prédominante surplombe fin en 5'TTAGGG3 », mais sont seulement ~ 10 nt longtemps. Peu de G-télomères surplombe fin en 5'GGGTTA3 », mais peut être jusqu'à 40 nt longtemps. Les deux types de G-faux sont sensibles à l'Exo T (ou Exo I) traitement (figure 4A, le panneau de droite, la piste 2, 3 et 7; figure 4B, piste 1, flèches).

Figure 1. Principe de maternelle dans le gel d'hybridation. Gauche, sous la condition indigène, la fin marqués TELC oligo sonde ne peut s'hybrider avec le simple-brin riche en G-faux des télomères. L'intensité de l'hybridation reflète la longueur du télomère G-faux. A droite, après dénaturation, la sonde sera TELC oligo s'hybrider avec l'ADN des télomères tous. L'intensité de cette hybridation représente la quantité totale d'ADN des télomères et est utilisé comme un contrôle du chargement, et la finale du G-faux niveau est calculé en divisant le signal d'hybridation native par celle obtenue après dénaturation.
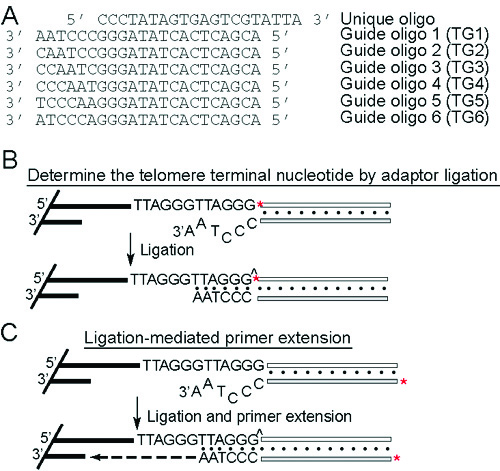
Figure 2. Principe de la ligature adaptateur et extension d'amorce ligature médiée. (A) Séquences de l'oligoes guide unique et six. (B) Déterminer le terminal télomères nucléotide par ligature adaptateur. Les oligo unique est fin marqués (marqués d'un astérisque rouge), recuit à l'oligoes guide et ligaturé à la fin des télomères. Ce n'est que lorsque l'adaptateur unique / guide porte surplomb 3 'qui est compatible avec la séquence des télomères terminal de l'adaptateur être ligaturés. (C) En extension de l'amorce ligature médiation, les oligo guide est fin marqués (marqués d'un astérisque rouge) et recuit à l'oligo unique avant ligaturés aux extrémités des chromosomes. Seuls l'adaptateur uniques / guide portant surplomb compatible avec le G-faux 3 'sera ligaturé. ^ Indique le lien phophordiester ligaturé. Après élimination de la paire oligo non ligaturés, extension de l'amorce sera réalisée avec l'ADN polymérase T4, qui manque de déplacement de brin et les activités d'exonucléase 5'-3 '. La longueur finale de l'étendue d'oligo guide reflète donc la longueur de la G-faux.
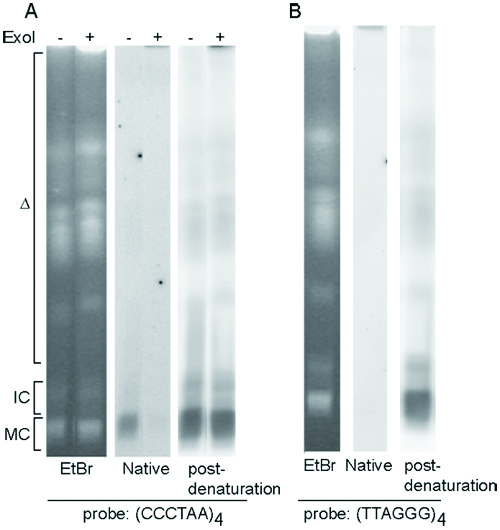
Figure 3. T. brucei G-télomères surplombent la structure analysée par natif dans le gel d'hybridation. (A)-gel hybridation avec TELC oligo sonde. (B) En-gel hybridation avec Telg oligo sonde. Dans les deux (A) et (B) à gauche, PFG Ethedium teinté bromure. Orient, l'hybridation résulte natale. A droite, l'hybridation résulte de post-dénaturation. Wild-type T. cellules brucei ont été utilisés. Intacte ou Exo j'ai traité l'ADN génomique ont été séparés par électrophorèse en champ pulsé. Triangle ouvert est synonyme de chromosomes mégabase; IC, intermédiaire chromosomes de taille; MC, minichromosomes.
"/>
Figure 4. T. brucei G-télomères surplombent la structure analysée par ligature médiée par extension d'amorce. (A) La plupart des T. brucei G-télomères surplombe fin en 5'TTAGGG3 ». Gauche, le gel de l'ADN au bromure Ethedium tachés. Montant approximativement égal de l'ADN est chargé dans chaque ruelle. A droite, résultat l'exposition du même gel après séchage. Intact, Exo I-traités, ou traités Exo T-ADN a été ligaturé à six différents marqués aux extrémités uniques / guide adaptateurs oligo comme indiqué. (B) T. télomères courts sont brucei G-surplombs. Intacte ou Exo ADN génomique T-traités a été ligaturé à six différents uniques / guide adaptateurs oligo suivie par extension d'amorce en utilisant l'ADN polymérase T4. Fin marqué TG4 oligo a été chargé comme un contrôle négatif (piste 11). Les oligo se tourne à 22 nt (*), mais aussi donné un signal plus faible à ~ 44 nt (Δ). Seuls unique/TG4 adaptateur donné des produits d'extension significativement plus longtemps que les oligo guide lui-même (flèche têtes, piste 7).
Discussion
Trypanosoma brucei provoque la maladie du sommeil chez les humains. Cette maladie, si elle n'est pas traitée, est inévitablement mortelle. T. cellules brucei chez des hôtes mammifères subissent une variation antigénique régulièrement afin d'échapper à l'attaque immunitaire de l'hôte 7. C'est pourquoi il est très difficile d'éliminer T. cellules brucei fois l'infection est établie. Nous avons récemment démontré que les té...
Déclarations de divulgation
Remerciements
Nous tenons à remercier le Dr Carolyn Prix pour les discussions scientifiques et suggestions judicieuses. Ce travail est soutenu par le NIH octroi AI066095 (PI: Bibo Li).
matériels
| Name | Company | Catalog Number | Comments |
| SeaKem LE Agarose | Lonza Inc. | 50004 | |
| Agarose Type VII (low melting agarose) | Sigma-Aldrich | A4018 | |
| Proteinase K, recombinant, PCR grade | Roche Group | 03 115801001 | |
| Exo I | New England Biolabs | M0293 | |
| Exo T | New England Biolabs | M0265 | |
| T7 exonuclease | New England Biolabs | M0263 | |
| T4 DNA polymerase | New England Biolabs | M0203 | |
| QIAquick nucleotide removal kit | Qiagen | 28304 |
Références
- Munoz-Jordan, J. L., Cross, G. A., de Lange, T., Griffith, J. D. t-loops at trypanosome telomeres. EMBO J. 20, 579-588 (2001).
- Li, B., Espinal, A., Cross, G. A. M. Trypanosome telomeres are protected by a homologue of mammalian TRF2. Mol Cell Biol. 25, 5011-5021 (2005).
- Wellinger, R. J., Wolf, A. J., Zakian, V. A. Saccharomyces telomeres acquire single-strand TG1-3 tails late in S phase. Cell. 72, 51-60 (1993).
- McElligott, R., Wellinger, R. J. The terminal DNA structure of mammalian chromosomes. EMBO J. 16, 3705-3714 (1997).
- Harrington, L. A., Greider, C. W. Telomerase primer specificity and chromosome healing. Nature. 353, 451-454 (1991).
- Jacob, N. K., Skopp, R., Price, C. M. G-overhang dynamics at Tetrahymena telomeres. Embo J. 20, 4299-4308 (2001).
- Barry, J. D., McCulloch, R. Antigenic variation in trypanosomes: enhanced phenotypic variation in a eukaryotic parasite. Adv Parasitol. 49, 1-70 (2001).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137, 99-109 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon