Method Article
PAR -クリップ - RNA結合タンパク質のトランスクリプトーム全体の結合部位を同定する方法
要約
RNA転写物は、トランス作用RNA結合タンパク質(RBPs)の多数によって媒介される大規模な転写後調節の対象となります。ここでは正確にとトランスクリプトーム全体の規模でRBPsのRNA結合部位を同定するために一般化の方法を提示する。
要約
RNA転写産物は、RNA結合タンパク質(RBPs)とマイクロRNAを含む、多くの場合依存的細胞型において発現されるリボ核タンパク質複合体(miRNPs)の何百と相互作用することによって転写後の遺伝子発現制御に供される。これらのRNA結合因子の相互作用は、個々の転写産物の規制をどのように影響するかを理解するために、in vivoでのタンパク質- RNA相互作用での高解像度の地図は1が必要です。
遺伝的、生化学的および計算的アプローチの組み合わせは一般的にRNA - RBPまたはRNA - RNP相互作用を識別するために適用されます。免疫精製RBPsに関連付けられているRNAのマイクロアレイプロファイリング(RIP -チップ)2は、トランスクリプトームレベルでの目標を定義しますが、そのアプリケーションは、速度論的に安定な相互作用の特性に制限されており、のみまれに3,4 RBP認識の要素(RREを識別することができます)長い標的RNA中。より直接的なRBPのターゲットサイトの情報は、架橋RNAセグメントの分離とcDNAのシークエンシング(CLIP)10に続いて免疫沈降7月9日 の in vivoのUV架橋5,6 で結合することによって得られる。 CLIPはRBPs 11月17日の数のターゲットを識別するために使用されました。しかし、CLIPはUV 254 nmのRNA -タンパク質架橋の効率が低いことによって制限されており、架橋の位置は、それが困難な背景の非架橋からUV -架橋標的RNAセグメントを分離すること、塩基配列架橋フラグメント内に容易に識別可能ではないRNAはまた、サンプル中に存在するフラグメント。
我々は、高解像度と携帯RBPsとmiRNPsのトランスクリプトーム全体の結合部位で決定するために強力な細胞系架橋アプローチを開発している我々の用語PAR -クリップ(光活性化-リボヌクレオシド-強化された架橋と免疫)(概要は図1Aを参照してくださいメソッドの)。方法は、生きた細胞によって新生RNA転写物に、例えば4 - チオウリジン(4 - SU)と6 -チオグアノシン(6 - SG)のような光反応性リボヌクレオシド類縁体の取り込みに依存しています。 365nmの紫外光による細胞の照射は、相互作用RBPsに光反応性ヌクレオシド標識細胞のRNAの効率的な架橋を誘導する。興味のRBPの免疫沈降は、架橋と共免疫沈降RNAの分離が続いている。単離されたRNAは、cDNAライブラリーに変換し、深いSolexaの技術を用いて配列決定される。 PAR -クリップが作成したcDNAライブラリーの一つの特徴は、架橋の正確な位置を配列決定したcDNA内に存在する変異によって識別できることです。突然変異をアデノシンにグアノシンに6 - SGの結果を使用して、一方シチジンへの移行4 - SUを使用して、架橋のシーケンスはチミジン、。架橋配列における変異の存在は、豊富な細胞のRNAから由来する配列の背景からそれらを分離することが可能になります。
多様なRNA結合タンパク質の数に方式のアプリケーションは、ハーフナーらに報告されている。18
プロトコル
以下のプロトコルは、FLAG /ドキシサイクリンによる誘導時にHAタグIGF2BP1を発現するHEK293細胞に対するPAR -クリップの手順を説明します。我々は、免疫沈降のために抗FLAG抗体を使用します。
免疫沈降のための効率的な抗体が利用できる場合は、PAR -クリップは、内因性の検出可能なレベルを表す任意のセルラインで動作する、興味のRNA結合タンパク質(RBP)をタグなし。
拡大するセル
- 増殖培地中でFlpIn-HEK293/TO/FLAG/HA-IGF2BP1セルを展開します。我々は出発点として100〜400 × 10 6個の細胞(約10〜40 15cmの細胞培養プレート)の間に使用することをお勧めします。約80%コンフルエントにそれらを成長する。
- 架橋前の14時間は、直接細胞培養培地に100μMの最終濃度(1 M 4 -チオウリジン原液の1:1000 V / V)に)4 -チオウリジンを追加し、b)の FLAG / HAの発現を誘導するドキシサイクリン(10 mg / mlのドキシサイクリンの原液の1:10,000 V / V)の1μg/ mlの添加によりIGF2BP1をタグ付け。注:代わりに、6 - チオグアノシン100μMを使用することができます4チオウリジンの。
UVクロスリンク
- プレートあたり10 mlの氷冷したPBSで細胞を1回洗浄し、完全にPBSを取り除く。
- Stratalinker 2400(ストラタジーン社製)または同様の装置は0.15 J / cm 2で 365nmの紫外光で発見された氷と照射するとトレイの上に置きますがプレート。
- 50 mlの遠心チューブにプレート、転送ごとに1 mlのPBSでラバーポリスマンで細胞をこすり落とすと4℃で5分間、500 × gで遠心分離によって収集し、上清を捨てる。 100 × 10 6 HEK293細胞は、(10 15 cmのプレート)約が得られます。ウェット細胞ペレットを1ml。
- (オプション)を使用すると、細胞溶解と直接続行する場合を除き、衝撃は-80℃で液体窒素とストア内の細胞ペレットを凍結℃に細胞ペレットは、少なくとも12ヶ月間保存することができます。
細胞溶解とRNaseT1ダイジェスト
- 1X NP40溶解バッファーの3倍量の架橋の細胞の細胞ペレットを取るし、10分間氷上でインキュベートする。
- 4℃で15分間、13,000 × gで遠心分離により透明な細胞ライセート℃の
- 0.2μmのメンブレンシリンジフィルター(ポールアクロディスクまたは同等品)を介して濾過することによって、さらにライセートをクリアします。
- 22℃15分間水浴中で1 U /μlとインキュベートの最終濃度にRNase T1を(Fermentas、10,000 U /μl)を添加℃に次に進む前に、氷上で5分間、その後涼しい反応。
免疫沈降と架橋標的RNA断片の回収
磁気分離装置を使用する
乾燥から磁気ビーズを防ぐために試料調製を通じて、以下のガイドラインに従ってください。
- 1 2分間マグネチックスタンドでビーズを含むチューブを置きます。
- チューブは、マグネット分離装置にある間チューブにバッファを追加します。
- チューブにキャップを、磁気分離装置から削除し、そしてビーズを懸濁します。あなたの指でチューブをフリックでビーズを再懸濁したり、5に設定したボルテックスを使用することができます。
- チューブのキャップに残っている可能性のあるビーズを収集するためにスピンダウンする。
- 必要に応じて繰り返し、手順1〜4を繰り返します。
磁性ビーズの調製
- 1.5mlマイクロチューブにダイナプロテインG磁性粒子(Invitrogen社製)1ml当たり細胞ライセート(それは約あるべき典型的な実験のため。ビーズの40 50μL)の10μlを移してください。クエン酸 - リン酸緩衝液1mlで二回ビーズを洗浄してください。
- ビーズ懸濁液を元のボリュームに対する相対的なクエン酸 - リン酸緩衝液の倍のボリュームに再懸濁します。
- 抗FLAG M2モノクローナル抗体(Sigma社製)1ml当たり、室温で40分間回転する車輪のサスペンションとインキュベート0.25μgのを追加。
- 未結合抗体を除去するクエン酸 - リン酸緩衝液1mlに二回ビーズを洗浄する。
- ビーズ懸濁液を元のボリュームに対する相対的なクエン酸 - リン酸緩衝液の二倍量でビーズを再懸濁します。
免疫沈降(IP)、第二のRNase T1の消化、および脱リン酸化
- 部分的なリボヌクレアーゼT1処理した細胞溶解液のmlあたり、新たに調製した抗体結合磁性ビーズ20μlを追加し、4℃で1時間回転するホイールに15 mlの遠心チューブでインキュベート℃の
- 15と50 ml遠心チューブ(Invitrogen社)のために磁性粒子コレクタに磁気ビーズを収集し、1.5mlマイクロチューブに移す。
- IP洗浄緩衝液1ml中にビーズを3回洗浄する。
- 100 U /μlの最終濃度にRNaseT1を(Fermentas、10,000 U /μl)を添加し、22℃15分間水浴中でビーズ懸濁液をインキュベート℃に氷の上に続いてクール5分間。
- 高塩分洗浄緩衝液1ml中にビーズを3回洗浄する。
- 脱リン酸化緩衝液の1ボリュームでビーズを再懸濁し
- 0.5 U /μlの最終濃度にウシ腸アルカリホスファターゼ(CIAP、NEB)を追加し、37℃で10分間懸濁液をインキュベート℃に
- ホスファターゼの洗浄緩衝液1mlに二回ビーズを洗浄
- DTTのないポリヌクレオチドキナーゼ(PNK)バッファー(酵素反応に必要なDTT濃度は、磁気ビーズを傷つけないように十分な高さ)で二回ビーズを洗浄する。
- PNKバッファの1つのオリジナルビーズのボリュームでビーズを再懸濁し
免疫沈降したタンパク質に架橋したRNAセグメントの放射性標識
- ビーズ懸濁液は、上記に一つのオリジナルビーズのボリュームで1 U /μlに0.5μCiの/μlおよびT4 PNK(NEB)の最終濃度にΥ- 32 P - ATPを加える。 37℃で30分間懸濁液をインキュベート℃に
- 100μMの最終濃度を取得し、37℃でさらに5分間インキュベートする非放射性ATPを追加℃に
- DTTなしPNKバッファー800μLと磁気ビーズを5回洗浄する。
- SDS - PAGEローディングバッファー70μlでビーズを再懸濁します。
ゲルスライスからSDS - PAGEおよび架橋RNA -タンパク質複合体の電気溶出
- 95℃のヒートブロックで5分間放射性標識、懸濁液をインキュベート° C変性させ、架橋RNAと渦との免疫沈降RBPを解放する。
- セパレータ上に磁気ビーズを外し、1.5 mlのマイクロ遠心チューブに上清を移す。
- 40の上清をウェルあたりのNOVEXビス - トリス4から12パーセント(Invitrogen社)のプレキャストポリアクリルアミドゲルをロードし、200で55分間ゲルを実行V.
- ゲルのチャンバーを分解し、それがワンプレートに取り付けられたまま、ゲルを解体。 phosphorimagerのプリントアウトにゲルの配置を容易にするために、我々は、ゲルの四隅の三時に非対称的つの小さな放射性ゲル片を移植することをお勧めします。放射性ゲル片は、以前は放射性標識合成オリゴヌクレオチドを精製するために使用されたゲルから回収することができる。汚染を避けるために、プラスチックフィルム(例:サランラップ)でゲルをラップします。
- 1時間のブランクphosphorimager画面にゲルを公開し、phosphorimagerに視覚化する。
- 向きを移植されたゲル片を使用してphosphorimagerのプリントアウトの上にゲルを合わせます。 RBPの予想サイズ(IGF2BP1、約75kDaの)に対応するバンドを切り出し、D -チューブダイアライザーミディチューブに移すと800μlの1 × SDS泳動バッファーを加える。
- 2時間、100Vで1 × SDSランニングバッファーで架橋したRNA - RBP複合体をElectroeluteゲルから切り出し、製造業者の指示にしたがって800μlのSDSランニングバッファーでD -チューブダイアライザーミディ(Novagen社)にelectroeluted。
プロテイナーゼK消化
- は1.2 mg / mlの最終濃度にプロテイナーゼK(ロシュ)を加えて、electroeluateを基準にして2倍のプロテイナーゼK等量のバッファーを追加。 55℃で30分間インキュベートする
- 酸性フェノール/クロロホルム/ IAA抽出(25:24:1、pH4.0)をクロロホルム抽出が続くことによりRNAを回復する。グリコーゲンを1μl(10 mg / mlストック)を追加するには、エタノールの3倍量を添加することによりRNAを沈殿させる。 10.5μlの水でペレットを溶解する。
cDNAライブラリーの準備と深いシーケンシング
もともと小さな規制RNAを19のクローニングのために説明した標準的なcDNAライブラリーの準備のプロトコルを介して回収したRNAを運ぶ。回収されたRNAの10.5μlを用いて20μlのスケールで説明したように、最初のステップ、3'アダプターのライゲーションは、実施した。 Solexaシーケンシングアダプター説明した設定を使用してください。回収されたRNAの量に応じて、挿入せずに5' -アダプタ- 3' -アダプター製品は、追加のPCRのバンドのようなcDNAの増幅後に検出されることがあります。このようなケースでは、消費税3%NuSieveから予想されたサイズの長いPCR産物は、低融点アガロースゲル、Solexaの技術を使用してGelEluteキット(キアゲン)とシーケンスを使用してゲル片からのPCR産物を溶出させる。 One Solexaシーケンシングの実行は通常、6と1000万シーケンスの間にaffords RNA結合タンパク質の結合部位のトランスクリプトームを幅広くカバーするために十分であることを読み取ります。
バイオインフォマティクスの解析
シーケンスの慎重なバイオインフォマティクス解析には、RBPが持つようなRNAの認識の要素として検討RBPのRNA結合部位、に有意義な洞察を得るために行われるニーズ、優先結合領域を読み込み(エクソン対イントロン、順序対非翻訳をコーディングシーケンス)。読み出しシーケンスは、ゲノムやESTデータベースに対して整列する必要があります。我々は通常、読み取りmappiを使用してくださいまで1つのミスマッチにあるユニークなゲノムのngは、シーケンスのクラスタを構築するために挿入または欠失は、さらに分析することができます読み込みます。 6 - SGを使用して遷移する4 - SUおよびGを使用するときにクラスタ化されたシーケンスの特性変異の頻度は、Cの遷移に、Tを読み込み、正常に架橋シーケンスを示すものである。我々の経験では4 - SUで標識した未架橋のRNAは、約20%のバックグラウンドの突然変異率を示しています。この率は約に増加しています。架橋時に50から80パーセント。
バイオインフォマティクス解析の詳細な説明は、ハーフナーらによって出版物の補足材料で見つけることができます。18
オプションの手順
トータルRNAへの4 - チオウリジンの取り込みレベルの決定
安定的に収穫前に100μM4SU 16時間を添加した培地で生育した後、興味のRBPを発現する細胞株から全RNAを分離する。コントロールとして、収穫の細胞は4SU添加することなく成長して。製造業者の指示に従って洗浄細胞ペレットにTrizol試薬(シグマ)の3倍量を添加することによりトータルRNAを分離します。さらに、製造業者のプロトコールに従ってキアゲンしたRNeasyを使用してトータルRNAを精製した。 RNAの単離と解析中に4SUの酸化を防止するために、洗浄バッファーとその後の酵素のステップに0.1 mMジチオスレイトール(DTT)を追加します。ダイジェストとHPLC分析のための単一のヌクレオシドに脱リン酸化されたトータルRNAは、20日までに説明した。簡単に言うと、30μlの体積で、37℃で16時間トータルRNAを精製したの40μgをしたインキュベートします0.4 U細菌アルカリホスファターゼ(生化学ワーシントン)および0.09 Uのヘビの毒を持つCホスホジエステラーゼ(ワーシントン生化学)。参照標準として、合成4SU標識RNA、(我々は標準的にCGUACGCGGAAUACUUCGA(4SU)Uを使用)し、また、酵素消化を完了するために件名を使用する。スペルコディスカバリーC18(結合相のシリカ5μm粒子、250 × 4.6 mm)の逆相カラム(ベルフォンテ、ペンシルバニア、米国)のHPLCによるリボヌクレオシドの結果として得られる混合物を分離します。 HPLCのバッファは3%アセトニトリル()と水で90%アセトニトリル(B)に、0.1M TEAAです。 15分、20分の場合は0〜10%B、30分、10〜100%Bの0%B:アイソクラティック勾配を使用してください。 HPLCカラムを洗浄するために実行するまでの間に適用される洗浄5分100%Bを適用します。
代表的な結果
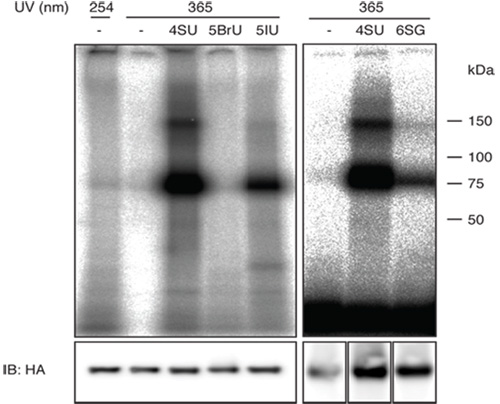
図1(右パネル)は、FLAG / 4 - SUと6 - SGでIGF2BP1 HA -タグを発現する細胞株を使用して実行PAR -クリップの代表的な結果を示しています。 IGF2BP1 6 - SGの架橋効率が4 - SUのための架橋効率よりも低いことに注意してください。低架橋効率が豊富な細胞のRNAのフラグメントに由来する配列の高いバックグラウンドになります、そのためには効率の低い光反応性ヌクレオシドを使用する際に、実験のスケールアップを検討する必要があります。
図1の左側のパネルには、潜在的に従来のUV 254nmの架橋に比べてPAR -クリップを使用することができる別の光反応性ウリジンアナログを使用しての比較を示しています。
正しい長さの放射性バンドの強度は、あなたにPAR -クリップ実験が働いていると、小さなRNAシークエンシングプロトコール(ステップバイステップの小さなのcDNAライブラリーの調製のための記述を通して運ぶために十分なRNAを単離しているかどうか良いアイデアを与えるRNAのシーケンシング)が19で見つけることができます。 6 - SGを使用して遷移する4 - SUおよびGを使用してCに遷移するために配列読み取り、Tの特徴的な変異の頻度は、成功した架橋シーケンスを示すものである。我々の経験では4 - SUで標識した未架橋のRNAは、約20%のバックグラウンドの突然変異率を示しています。この率は約に増加しています。架橋時に50から80パーセント。
開示事項
謝辞
我々は有用な議論のためTuschl研究室のメンバーに感謝。 MHは、ドイツ学術交流会(DAAD)によってサポートされています。この作品は、スイス国立基金助成MZへ#3100A0 - 114001によってサポートされていました; TTは、HHMIの研究者でもある、と彼の研究室での作業は、NIHの助成金GM073047とMH08442とスター財団によってサポートされていました。
資料
バッファーと試薬
成長培地HEK293細胞
- DMEM
- 10%FBS
- 2mMのL -グルタミン
- 100 U / mlペニシリン
- 100 U / mlのストレプトマイシン
- 100μg/ mlのハイグロマイシン
- 15μg/ mlのブラストサイジン
4チオウリジンストック溶液(1 M)
- 260.27 mgを4チオウリジン
- 1ミリリットルDMSO
Doxycyclin株式(10 mg / ml)を
- 10 mgのdoxycyclin
- 1ミリリットルDMSO
1X NP40溶解バッファー
DTTとプロテアーゼ阻害剤なしで5倍のバッファーの株式を準備します。実験の前に直接DTTとプロテアーゼ阻害剤を追加。
- 50mMのHEPES、pH7.5の
- 150mMのKClを
- 2mMのEDTA
- 1mMのNaFを
- 0.5%(v / v)のNP40
- 0.5mMのDTT
- 完全なEDTA -フリーのプロテアーゼ阻害剤カクテル(Roche社)
クエン酸 - リン酸緩衝液
- 4.7グラム/ lのクエン酸
- 9.2グラム/ LのNa 2 HPO 4
- pH5.0で
IP -洗浄緩衝液
- 50mMのHEPES - KOH、pH7.5の
- 300mMのKClを
- 0.05%(v / v)のNP40
- 0.5mMのDTT(実験の前に直接追加する)
- 完全なEDTA -フリーのプロテアーゼ阻害剤カクテル(Roche)(直接実験の前に追加)
高塩洗浄緩衝液
- 50mMのHEPES - KOH、pH7.5の
- 500mMのKClを
- 0.05%(v / v)のNP40
- 0.5mMのDTT(実験の前に直接追加する)
- 完全なEDTA -フリーのプロテアーゼ阻害剤カクテル(Roche)(実験の前に直接追加する)
脱リン酸化緩衝液
- 50mMトリス- HCl、pHを7.9
- 100mMのNaCl
- 10mMのMgCl 2
- 1mMのDTT
ホスファターゼの洗浄バッファー
- 50mMトリス- HCl、pH7.5の
- 20 mMのEGTA
- 0.5%(v / v)のNP40
DTTのないポリヌクレオチドキナーゼ(PNK)バッファ
- 50mMトリス- HCl、pH7.5の
- 50mMのNaCl
- 10mMのMgCl 2
PNKバッファ
- 50mMトリス- HCl、pH7.5の
- 50mMのNaCl
- 10mMのMgCl 2
- 5mMのDTT
SDS PAGEローディングバッファー
- 10%グリセロール(v / v)の
- 50mMトリス- HCl、pH 6.8の
- 2mMのEDTA
- 2%SDS(w / v)の
- 100mMのDTT
- 0.1パーセントブロモフェノールブルー
2倍のプロテイナーゼKのバッファ
- 100mMトリス- HCl、pH7.5の
- 150mMのNaCl
- 12.5 mMのEDTA
- 2%(w / v)のSDS
参考文献
- Keene, J. D. RNA regulons: coordination of post-transcriptional events. Nat. Rev. Genet. 8 (7), 533-533 (2007).
- Tenenbaum, S. A. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Nat. Acad. Sci. 97 (26), 14085-14085 (2000).
- Gerber, A. P. Genome-wide identification of mRNAs associated with the translational regulator PUMILIO in Drosophila melanogaster. Proc. Nat. Acad. Sci. 103 (12), 4487-4487 (2006).
- Lopez de Silanes, I. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Nat. Acad. Sci. 101 (9), 2987-2987 (2004).
- Greenberg, J. R. Ultraviolet light-induced crosslinking of mRNA to proteins. Nucl. Acids Res. 6 (2), 715-715 (1979).
- Wagenmakers, A. J. Cross-linking of mRNA to proteins by irradiation of intact cells with ultraviolet light. Eur. J. Biochem. 112 (2), 323-323 (1980).
- Mayrand, S. Structure of nuclear ribonucleoprotein: identification of proteins in contact with poly(A)+ heterogeneous nuclear RNA in living HeLa cells. The Journal of Cell Biology. 90 (2), 380-380 (1981).
- Dreyfuss, G. Characterization of heterogeneous nuclear RNA-protein complexes in vivo with monoclonal antibodies. Mol. Cell. Biol. 4 (6), 1104-11 (1984).
- Adam, S. A., Dreyfuss, G. Adenovirus proteins associated with mRNA and hnRNA in infected HeLa cells. J. Virol. 61 (10), 3276-3276 (1987).
- Ule, J. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1212 (2003).
- Licatalosi, D. D. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-464 (2008).
- Yeo, G. W. An RNA code for the FOX2 splicing regulator revealed by mapping RNA-protein interactions in stem cells. Nat. Struct. Mol. Biol. 16 (2), 130-130 (2009).
- Sanford, J. R. Splicing factor SFRS1 recognizes a functionally diverse landscape of RNA transcripts. Genome Res. 19 (3), 381-381 (2009).
- Granneman, S. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proc. Nat. Acad. Sci. , (2009).
- Guil, S., Caceres, J. F. The multifunctional RNA-binding protein hnRNP A1 is required for processing of miR-18a. Nat. Struct. Mol. Biol. 14 (7), 591-591 (2007).
- Chi, S. W. Argonaute HITS-CLIP decodes microRNA-mRNA interaction maps. Nature. 460 (7254), 479-479 (2009).
- Zisoulis, D. G. Comprehensive discovery of endogenous Argonaute binding sites in Caenorhabditis elegans. Nat. Struct. Mol. Biol. , (2010).
- Hafner, M. Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell. , (2010).
- Hafner, M. Identification of microRNAs and other small regulatory RNAs using cDNA library sequencing. Methods. 44 (1), 3-3 (2008).
- Andrus, A., Kuimelis, R. G. Base composition analysis of nucleosides using HPLC. Current Protocols in Nucleic Acid Chemistry. Chapter 10 (Unit 10.6), (2001).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved