このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
メタゲノムライブラリからセルラーゼ活性をBiominingためのハイスループットスクリーニング
要約
このプロトコルは、大腸菌で発現メタゲノムライブラリーからのセルロース分解活性のためのハイスループットスクリーニングを説明します。画面には、ソリューションベースと高度に自動化され、吸光度測定など最終的な読み出しと384ウェルマイクロプレートでのワンポット化学を使用しています。
要約
セルロース、地球上の有機炭素の最も豊富な源は、バイオ燃料生産の1の増加を重視した幅広い産業用途を持っています。セルロースを変更したり、劣化させる化学的方法は、通常、強酸と高温を必要とする。このように、酵素法は、生物変換プロセスで顕著になっている。細菌および真菌分離株からのアクティブなセルラーゼの識別がやや効果があった一方で、自然界における微生物の大半は、実験室培養をレジスト。また、メタゲノム、スクリーニングの手法として知られている環境のゲノムは、新たな生物変換酵素の検索で栽培ギャップを埋めることに大きな期待を持っている。土壌2、水牛ルーメン3とコンゴレッド色素(Teatherとウッド5の方法に基づいて)で染色されたカルボキシメチルセルロース(CMC)寒天プレートを使用してシロアリ後肢腸4など様々なメタゲノムスクリーニングのアプローチが成功した環境からの新規セルラーゼを回復した。しかし、CMCメソッドは、スループットに制限され、定量的ではなく、ノイズ比6に低信号が現れます。他の方法は、7,8の報告が、それぞれが大規模な挿入のゲノムライブラリーのハイスループットスクリーニングのための望ましくない寒天プレートベースのアッセイを、使用されています。ここでは、ソリューションベースの発色ジニトロフェノール(DNP)- cellobioside基板9を使用して、セルラーゼ活性のための画面を提示する。私たちの図書館は、コピー数の誘導10を介して分析感度を向上させるフォスミドPCC1コピー制御にクローニングした。方法は、吸光度測定として提供される最終的な読み出しと384ウェルマイクロプレートでのワンポット化学を使用しています。この読み出しは、付属のスタッキングシステムを持つ液体ハンドラとプレートリーダーを使用して一日あたりの100倍、384ウェルプレートまでのスループットと、定量的な敏感と自動化です。
プロトコル
このプロトコルを開始する前に、384ウェルプレートフォーマットで保存されているメタゲノムライブラリーが必要になります。私たちの研究では、ファージT1 -耐性TransforMax EPI300 - T1 R E.との組み合わせでPCC1コピー制御フォスミドベクターを使用ライブラリのホストおよび-80 ° C 11時に私たちのプレート格納されているとして大腸菌細胞。
1。メタゲノムライブラリーのプレートの複製
- 37あなたのライブラリが含まれている霜取りプレート℃で約20分、またはまで、全てのウェルが解凍されています。
- 15分間ロボットを殺菌するqPix2にUV光殺菌機能を使用してください。
- 500mLに試薬瓶に100ug/mLで12.5ug/mLとアラビノースの最終濃度でクロラムフェニコールを含むLB培養液を準備します。各プレートは、約20mlの使用に加え、デッドボリュームを可能にするために追加の50mLのを行います。
- 滅菌チューブを介してマニホールドに接続されたメディアのボトルを持つあたりは、メーカーの指示としてqFill3を設定します。メディアの適切な量のためにそれをプログラムし、各ウェルに45uLボリュームを埋めるように設定します。
- メディアが多様体の各端子からの表示されるまで、ロボットのパージ機能を使用してチューブとマニホールドから空気をパージします。
- qFill3を使用してLB培地でプレートの希望番号を記入してください。各プレートには、埋めるために約20秒かかります。
- qPix2ロボットの適切な領域にライブラリのプレートと、新鮮なプレートをロードします。リアお風呂で2%Micro90、途中風呂に蒸留水をオートクレーブ、フロントバスで80%エタノール、適切な試薬で洗浄槽を埋める。
- qPix2の"複製"プログラムを使用してください。ソフトウェアでは、ソースプレートとデスティネーションプレートの適切な頭部、数と種類を選択します。また、各槽内で通常のレプリケーション、6サイクルの間にきれいに頭をセットアップするだけで十分です。マシンの能力は、一度に10枚であり、そしてそれは、384ピンのヘッド(または96ピンの頭を持つ約50分)で、それらすべてを複製するために約15分かかります。
注:"ソースを混ぜ"または"宛先を混ぜ"するオプションがあります。それは、問題が以前にそれらを使用して私たちのロボットで発生しているとして、これらのオプションを使用しないことをお勧めします。 - プレートが複製されると、湿度のボックスで24時間、37℃でプレートを成長させる。我々はアラビノースの添加によりフォスミドの高いコピー数を誘導されているため、クローンは、非誘導のクローンよりも遅く成長する傾向がある。 -80℃にライブラリのプレートを返す
注:湿度のボックスのインキュベーションは、メディアの蒸発を保証するには、すべてのウェルのために均一な環境を維持し、プレートの端にある井戸では発生しません。 - まず水(〜50ml)にしてから、80%エタノール(〜50ml)でそれをパージすることによってqFill3ロボットを清掃してください。コンポーネントを分解し、アルミ箔に包まれたボトル、蓋、とチューブを洗浄し、オートクレーブ。
2。 E.の成長の測定大腸菌のクローン
- 37℃のインキュベータからプレートを取り外します。最大25枚の最大、RapidStakで提供される雑誌の荷台の上に取り出して、ふたを取っておく、と場所のプレート。
注:スタックの一番下にあるプレートが読み出される最初のものになる。ソフトウェアは、これを記録しないように、プレートの順番を追跡することを確認してください。 - 迅速なStakから1つのマガジンを削除する、それが空であることを確認し、そして読まれるプレートのスタックにプッシュする。ハンドルでそれをつかみ、そしてプレートはすべてのマガジンにロードする必要があります。 RapidStakにマガジンをロードする。
注:雑誌にはプレートのA1ウェルが行く必要がありますどの角示す隅にある小さなネジを持っている。マガジンは、正しい向きでのみRapidStakにマウントされます。 - マシンに接続されたコンピュータ上でSkanIt REプログラムを開きます。 "新しいセッション"を選択し、適当な名前。プレートの種類として"コーニングフラットボトム384ウェルプレート"を選択します。
- "プレートのレイアウト"領域で、"ウィザード"ボタンを選択して、お皿に384の未知数を追加するには、それを選択します。
- "プロトコル"領域で、"まあループ"オプションを選択して、384ウェルのために入力してください。 "まあループ"のアイコンが画面の左側に流れ、ツリーに表示されます。アイコンを選択し、"測光"を追加する]をクリックします。 600nmの時に読み込むように設定します。
- 一意の名前を使用してプロトコルを保存し、SkanIt REのプログラムを閉じます。
- コンピュータ上で、PolaraRSプログラムを開きます。
- メインページにRapidStakデバイスに接続されている測定器の一覧表があるでしょう。 VarioSkanは、この表の左側に配置する必要があります。 VarioSkan見出しの下に、2つのリンクが存在します。"RunSession"と"インキュベート"。 "RunSession"オプションをクリックします。
- 新しいウィンドウ、アッセイウィンドウは、中央のフローチャートで表示されます。 "RunSession"の項目を(それが唯一の項目の存在するはず)を選択します。画面の右側のドロップダウンメニューで表示されます。このメニューで実行して保存されてSkanItのREの名前を見つける。
注:このメニューには合理的な順序付けはないようである。それは、アルファベット順ではない、または日付によって追加、またはコンピュータ上のプロトコルが保存されている場所に関連する。これは苦痛であるあなた自身を見つける前に、すべてのプロトコルを、目を通す必要があることを意味します。 - あなたのプロトコルを選択したら、右上隅の"このアッセイを実行する"をクリックします。
- ボックスは、ソースとして使用するマガジンを示すためにあなたを求める、ポップアップ表示されます、とどの雑誌は空です。画面上のボックスの下部にはフロントマガジンに対応しています。 [オプション:それはそれに要する時間の量の見積もりを決定するためにロードされたプレートの番号を入力するよう求めた。この見積もりは、通常の方法オフになっています。]
- RapidStakとVarioSkan両方の電源がオンになっていることを確認し、"OK"を押します。
- RapidStakは、自動的に板の連続測定を可能にする、プレートリーダーにプレートをロードします。 25プレートを読み取るには、約50分かかります。
注:これはマシンの適切な配置を確保するためにVarioSkanに最初のプレートの負荷を監視することをお勧めします。 RapidStakはプレートリーダーを正しくロードされない場合は、PolaraRSウィンドウの右上隅にある"一時停止このアッセイ"ボタンを使用してください。 - VarioSkanはすべてのプレートを読んで完了すると、フルマガジンを削除し、雑誌の荷台の上に置きます。プラットフォームの外側の長方形を持ち上げ、そして雑誌では真ん中の山に立っているプレートを残し、オフにスライドするはずです。適切なプレートにふたを取り付けます。
3。各プレートにアッセイミックスの加算
- pH5.5で50mmの酢酸カリウム緩衝液中で溶解ミックスの既成の10倍の株式(10%トリトンX - 100、100mMのRIS、10mMのEDTA)を用いてアッセイミックスを調製する。基板が完全に溶解することを確認し、DMSOに75mg/mL DNP - cellobioside基板までの株式を準備します。アッセイミックスに0.1mg/mLの最終濃度にDNP - cellobioside原液を追加。各プレートには、溶液約20mlのを使用し、加えてデッドボリュームを可能にするために追加の50mLのを行います。
注:DNP - Cellobiosideの溶解度を増加させるので、DMSOに溶解し、水に容易に溶解しない。最終的なアッセイ溶液中のDMSOの存在は、観察可能な効果はありません。 - 滅菌チューブを介してマニホールドに接続されたメディアのボトルを持つあたりは、メーカーの指示としてqFill3を設定します。メディアの適切な量のためにそれをプログラムし、各ウェルに45uLボリュームを埋めるように設定します。
- メディアが多様体の各端子からの表示されるまで、ロボットのパージ機能を使用してチューブとマニホールドから空気をパージします。
- qFill3を使用して、各プレートにアッセイミックスを追加。各プレートには、埋めるために約20秒かかります。
- アッセイミックスが追加されると、37℃でプレートをインキュベート° Cを湿度のボックスに12〜16時間のために。
注意:プレートのインキュベーションを超えるアッセイミックスが追加されるとは、内部の井戸のために見られるよりも高い吸光度をもたらす、プレートの外側のウェル中で蒸発の原因となります。これは、湿度チャンバ内でプレートをインキュベートすることによって相殺することができます。
4。アッセイしたクローンの読書吸光度
- 37℃のインキュベータからプレートを取り外します。最大25枚の最大、RapidStakで提供される雑誌の荷台の上に取り出して、ふたを取っておく、と場所のプレート。
注:スタックの一番下にあるプレートが読み出される最初のものになる。ソフトウェアは、これを記録しないように、プレートの順番を追跡することを確認してください。 - 迅速なStakから1つのマガジンを削除する、それが空であることを確認し、そして読まれるプレートのスタックにプッシュする。ハンドルでそれをつかみ、そしてプレートはすべてのマガジンにロードする必要があります。 RapidStakにマガジンをロードする。
注:雑誌にはプレートのA1ウェルが行く必要がありますどの角示す隅にある小さなネジを持っている。マガジンは、正しい向きでのみRapidStakにマウントされます。 - マシンに接続されたコンピュータ上でSkanIt REプログラムを開きます。 "新しいセッション"を選択し、適当な名前。プレートの種類として"コーニングフラットボトム384ウェルプレート"を選択します。
- "プレートのレイアウト"領域で、"ウィザード"ボタンを選択して、お皿に384の未知数を追加するには、それを選択します。
- "プロトコル"領域で、"まあループ"オプションを選択して、384ウェルのために入力してください。 "まあループ"のアイコンが画面の左側に流れ、ツリーに表示されます。アイコンを選択し、"測光"を追加する]をクリックします。 400nmの時に読み込むように設定します。
- 一意の名前を使用してプロトコルを保存し、SkanIt REのプログラムを閉じます。
- コンピュータ上で、PolaraRSプログラムを開きます。
- メインページにRapidStakデバイスに接続されている測定器の一覧表があるでしょう。 VarioSkanは、この表の左側に配置する必要があります。 VarioSkan見出しの下に、2つのリンクが存在します。"RunSession"と"IncubateはRunSession"オプション"。をクリックして"。
- 新しいウィンドウ、アッセイウィンドウは、中央のフローチャートで表示されます。 "RunSession"の項目を(それが唯一の項目の存在するはず)を選択します。画面の右側にあるドロップダウンメニューが表示されます。このメニューで実行して保存されてSkanItのREの名前を見つける。
- あなたのプロトコルを選択したら、右上隅の"このアッセイを実行する"をクリックします。
- ボックスは、ソースとして使用するマガジンを示すためにあなたを求める、ポップアップ表示されます、とどの雑誌は空です。画面上のボックスの下部にはフロントマガジンに対応しています。 [オプション:それはそれに要する時間の量の見積もりを決定するためにロードされたプレートの番号を入力するよう求めた。この見積もりは、通常の方法オフになっています。]
- RapidStakとVarioSkan両方の電源がオンになっていることを確認し、"OK"を押します
- RapidStakは、自動的に板の連続測定を可能にする、プレートリーダーにプレートをロードします。
注:これはマシンの適切な配置を確保するためにVarioSkanに最初のプレートの負荷を監視することをお勧めします。 RapidStakはプレートリーダーを正しくロードされない場合は、PolaraRSウィンドウの右上隅にある"一時停止このアッセイ"ボタンを使用してください。 - VarioSkanはすべてのプレートを読んで完了すると、フルマガジンを削除し、雑誌の荷台の上に置きます。プラットフォームの外側の長方形を持ち上げ、そして雑誌では真ん中の山に立っているプレートを残し、オフにスライドするはずです。プレートは、ラボの手続きに従って廃棄することができます。
- 吸光度、オープンSkanItのREのソフトウェアを、エクスポートして、"既存のファイルを開く"を選択する。
- あなたのセッションを保存したディレクトリを選択するには、リストを展開すると、タイトルの見出しの下にプレートの数に応じて、"コンテナN"に"コンテナ1"というラベルのオプションになる"+"アイコンを押してください。最初に分析するプレートを選択します。
- ページの上部にあるメニューバーで、"データ処理>レポート/エクスポート"を選択します。
- 輸出のために必要なオプションを選択します。
注:私は一般的に"プレートのレイアウト"と輸出のための"光度"のデータを選択します。 - "[レポートの表示"をクリックし、"ファイル>保存"。目的のディレクトリを選択します。出力形式はMicrosoft Excelのスプレッドシートです。
5。代表的な結果
陽性クローンを含む単一の384ウェルプレートから吸光度の例を図2に示されています。ポジティブクローンは、セルラーゼ活性を発現していないものより吸光度の顕著な増加を示しています。アッセイ時間の違い、ウェルプレート上の場所、またはDNPの濃度は、(非溶解DNPを除外することによって導入されるかもしれない)の絶対吸光度に影響を与える可能性があります。このようなプレートの平均値または列の平均以上の吸光度の差として相対的な吸光度測定値は、、セルラーゼ陽性クローンを同定するのより堅牢な方法です。
ライブラリのプレートからポジティブクローンの同定に続いて、それは二次スクリーニングのための新たなプレートにすべての陽性クローンを複製することをお勧めします。これはよく場所やプレートの変動に起因する影響を排除し、陽性クローンの間でより多くの直接比較が可能になります。

E.にクローン化されたメタゲノムライブラリーのためのハイスループットアッセイの図1。フローチャート大腸菌と-80℃で保存
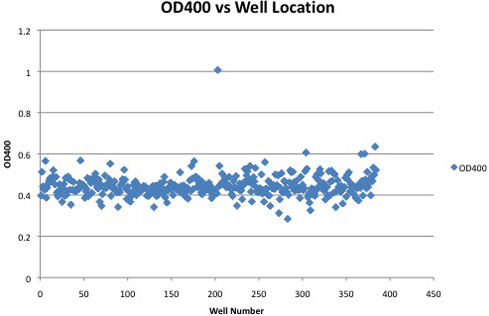
陽性クローンを含むもの384ウェルプレートから図2。吸光度から。セルラーゼ陽性クローンが負のクローン以上の有意な増加吸光度によって識別することができます。
ディスカッション
E.で表される大規模な挿入のゲノムDNAのメタゲノムライブラリーからのセルロース分解活性の迅速な検出のためのハイスループットスクリーニング大腸菌は、このプロトコルで説明されています。このメソッドは、一般的に文献で使用されるCMC /コンゴーレッドアッセイよりも優れています。それは解決策に基づいており、定量分析を可能にするプレートリーダーからの吸光度?...
開示事項
謝辞
著者らは、DNP - Cellobioside基板を提供するための博士スティーブウィザースと弘明陳に感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| qPix2 | Genetix | With 384-pin gridding head | |
| qFill3 | Genetix | With 384-well manifold | |
| Varioskan | Thermo Fisher Scientific, Inc. | ||
| RapidStak | Thermo Fisher Scientific, Inc. | Connected to Varioskan | |
| Micro90 Detergent | Cole-Parmer | 18100-00 | Diluted to 2% in water |
| Ethanol | Major Lab Supplier | Diluted to 80% in water | |
| Chloramphenicol | Sigma-Aldrich | C0378 | 12.5mg/mL in ethanol |
| LB broth, Miller | Fisher Scientific | BP1426-2 | 25g/L, autoclaved |
| 384-well flat bottom plates | Corning | 3680 | |
| L-(+)-Arabinose | Sigma-Aldrich | A3256 | 100mg/mL in water |
| Potassium Acetate | Fisher Scientific | P171 | 50mM in water, autoclaved, adjusted to pH 5.5 with HCl |
| Triton X-100 | Fisher Scientific | BP151 | |
| Trizma hydrochloride | Sigma-Aldrich | T3253 | In TE buffer solution, 100mM |
| EDTA disodium salt | Sigma-Aldrich | E5134 | In TE buffer solution, 10mM |
| 2,4-dinitrophenyl cellobioside | Provided by Dr. Steve Withers, UBC | ||
| Dimethyl Sulfoxide | Sigma-Aldrich | D8418 |
参考文献
- Rubin, E. M. Genomics of cellulosic biofuels. Nature. 454, 841-845 (2008).
- Voget, S., Steele, H. L., Streit, W. R. Characterization of a metagenome derived halotolerant cellulase. J. Biotechnol. 126, 26-36 (2006).
- Duan, C. J. Isolation and partial characterization of novel genes encoding acidic cellulases from metagenomes of buffalo rumens. J. Appl. Microbiol. 107, 245-256 (2009).
- Zhang, Y. H. P. Outlook for cellulase improvement: Screening and selection strategies. Biotech. Advances. 24, 452-481 (2006).
- Teather, R. M., Wood, P. J. Use of congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl. And Environ. Microbiol. 43, 777-780 (1982).
- Sharrock, K. R. Cellulase assay methods: a review. J. Biochem and Biophys Methods. 17, 81-106 (1988).
- Kasana, R. C., Salwan, R., Dhar, H., Dutt, S., Gulati, A. A rapid and easy method for detection of microbial cellulases on agar plates using Gram's Iodine. Curr. Microbiology. 57, 503-507 (2008).
- Huang, J. S., Tang, J. Sensitive assay for cellulase and dextranase. Anal. Biochem. 73, 369-377 (1976).
- Tull, D., Withers, S. G. Mechanisms of cellulases and xylanases: A detailed kinetic study of the exo-beta-1,4-glycanase from Cellulomonas fimi. Biochemistry. 33, 6363-6370 (1994).
- Martinez, A., Bradley, A. S., Waldbauer, J. R., Summons, R. E., DeLong, E. F. Proteorhodopsin photosystem gene expression enables photophosphorylation in a heterologous host. PNAS. 104, 5590-5595 (2007).
- Taupp, M., Lee, S., Hawley, A., Yang, J., Hallam, S. J. Large Insert Environmental Genomic Library Production. J Vis Exp. , (2009).
- Martinez, A., Tyson, G. W., DeLong, E. F. Widespread known and novel phosphonate utilization pathways in marine bacteria revealed by functional screening and metagenomic analyses. Environ Microbiol. 12, 222-238 (2010).
- Wild, J., Hradecna, Z., Szybalski, W. Conditionally amplifiable BACs: Switching from single-copy to high-copy vectors and genomic clones. Genome Research. 12, 1434-1444 (2002).
- Johnson, E. A. Sacchirification of complex cellulosic substrates by the cellulase system from Clostridium thermocellum. Appl Environ Microbiology. 43, 1125-1132 (1982).
- Stutzenberger, F. J. Cellulase Production by Thermomonospora curvata isolated from municipal solid waste compost. Appl Environ Microbiol. 22, 147-152 (1971).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved