Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Экран с высоким Пропускная Biomining Целлюлаза активность метагеномных из библиотеки
В этой статье
Резюме
Этот протокол описывает экран высокого пропускную способность для целлюлолитических деятельности от метагеномных библиотеки выражается в кишечной палочки. Экран урегулирования, основанного и высокой степенью автоматизации, и использует одну-пот в 384 химии и планшеты с окончательным считывания как измерения абсорбции.
Аннотация
Целлюлоза, самый богатый источник органического углерода на планете, имеет широкий круг промышленных применений с увеличением акцента на производство биотоплива 1. Химические методы для изменения или деградации целлюлозы как правило, требуют сильных кислот и высоких температур. Таким образом, ферментативные методы стали видные в биоконверсии процесса. В то время как идентификация активных целлюлаз от бактериальных и грибковых изолятов был несколько эффективных, подавляющее большинство микробов в природе противостоять лаборатории культивирования. Экологические геномной, также известный как метагеномных, скрининг подходы большие надежды в преодолении разрыва в выращивание поиск новых ферментов биоконверсии. Метагеномных подходов скрининга успешно восстановлен роман целлюлозы из сред, столь же разнообразны, как почвы 2, буйволов рубца 3 и термитов задних кишки 4 с использованием карбоксиметилцеллюлозы (КМЦ) агаром окрашивают конго красного красителя (на основе метода Teather и деревообрабатывающей 5). Тем не менее, CMC метод ограничен по пропускной способности, не количественные и проявляется низким отношением сигнал-шум 6. Другие методы были зарегистрированы 7,8, но каждое использование агар пластины основе анализа, что является нежелательным для высокопроизводительного скрининга больших библиотек геномной вставки. Здесь мы представляем на базе решений экран для целлюлазы активности с использованием хромогенного динитрофенол (DNP)-cellobioside подложки 9. Наша библиотека была клонирована в pCC1 управления копированием fosmid увеличить чувствительность анализа по количеству копий индукции 10. Метод использует одно-пот химии в 384-луночных микропланшетов с окончательным считывания предоставляется в качестве измерения абсорбции. Данное показание является количественным, чувствительные и автоматизированные с пропускной способностью до 384-100X также пластин в день, используя жидкий проводник и ридер с прикрепленными укладки системы.
протокол
Перед началом этого протокола, вам понадобится ваш метагеномных библиотеке хранятся в формате 384 и пластины. В нашем исследовании мы использовали pCC1 управления копированием fosmid вектор в сочетании с фагом Т1-стойкие TransforMax EPI300-T1 R E. кишечной клетки, как хозяин библиотеки и хранятся наши тарелки при -80 ° C 11.
1. Репликация метагеномных Плиты библиотека
- Размораживание чашки, содержащие библиотеки при 37 ° С в течение приблизительно 20 минут, или пока все лунки размораживают.
- Использование ультрафиолета стерилизации функцию qPix2 стерилизовать робота в течение 15 минут.
- Подготовка LB бульоне с хлорамфениколом при конечной концентрации 12.5ug/mL и арабинозы в 100ug/mL в 500 мл бутылки реагента. Каждая пластина будет использовать около 20 мл, а также сделать дополнительные 50 мл, чтобы обеспечить мертвый объем.
- Настройка qFill3 также инструкции на производителя с бутылкой СМИ прилагаются к многообразию с помощью стерильных трубок. Программа его на соответствующее количество средств массовой информации, и поставил его, чтобы заполнить 45uL объем в каждую лунку.
- Чистки воздуха из трубки и многообразием использования чистки особенность робота до средств массовой информации видно из каждого контактный многообразия.
- Заполнить необходимое количество пластин с Л. Б. массовой информации, использующих qFill3. Каждая пластина занимает примерно 20 секунд, чтобы заполнить.
- Загрузите библиотеку пластин и свежие пластин в соответствующих областях робот qPix2. Заполните очистки ванн с соответствующими реагентами, 2% Micro90 в задней ванна, автоклавного дистиллированной водой в середине ванны, 80% этанола в передней ванну.
- Используйте "Репликация" программы qPix2. В программное обеспечение, выберите соответствующую головы, количество и типы исходных пластин и назначения плит. Также настроен голову для очистки межзубных репликаций, как правило, 6 циклов в каждой ванне вполне достаточно. Мощность машины составляет 10 пластин в то время, и это занимает примерно 15 минут, чтобы повторить их всех с 384 булавочную головку (или приблизительно 50 минут с 96 булавочную головку).
Примечание: Существует возможность "Движение Источник" или "Движение назначения". Рекомендуется не использовать эти варианты в качестве проблемы, ранее произошло с нашим роботом, используя их. - После пластин реплицируются, растут пластин при 37 ° С в течение 24 часов в поле влажности. Потому что мы вызывающие большое количество копию fosmid с добавлением арабинозы, клоны, как правило, растут медленнее, чем не-индуцированных клонов. Вернуться библиотеки пластин до -80 ° C.
Примечание: Инкубационный в поле влажности обеспечивает испарение СМИ не происходит в скважинах на краю пластины, сохраняя единую среду для всех скважин. - Чистая qFill3 робот сначала очищать его с водой (~ 50 мл), а затем 80% этанола (~ 50 мл). Разберите компоненты, а чистый и автоклава бутылки, крышки, и трубки, завернутые в алюминиевую фольгу.
2. Измерение роста E. кишечной клонов
- Удалить пластин от 37 ° C инкубатора. Удалите и отложите в сторону крышки, и место пластины на платформу журнал загрузки обеспечены RapidStak, до максимум 25 пластин.
Примечание: пластины на нижней части стека будет первым, кто будет читать. Убедитесь в том, чтобы отслеживать порядок пластин, а программное обеспечение не зафиксировать это. - Удалите один журнал из Быстрое Stak, убедитесь, что он пуст, и толкать ее в стек пластин для чтения. Захватите его за ручку, и все плиты должны быть загружены в журнале. Нагрузка магазин в RapidStak.
Примечание: Журнал винтик в углу, обозначающее какой угол A1 скважин плиты должны идти. Журнал будет только смонтировать в RapidStak в одной ориентации. - Откройте программу SkanIt RE на компьютере, подключенном к машинам. Выберите "New Session" и назовите его соответствующим образом. Выберите "Corning плоским дном 384-луночный планшет», как пластины типа.
- В "Пластина Макет" выберите "Мастер" кнопку, и выбрать его, чтобы добавить 384 неизвестных вашей тарелке.
- В "Протокол" выберите "Ну Loop" вариант, и ввести для 384 скважин. "Ну Loop" появится в потоке дерево на левой стороне экрана. Выберите значок и нажмите кнопку добавить "Фотометрическое измерение". Установите его читать на 600 нм.
- Сохраните протокол с уникальным именем, и закрыть программу SkanIt RE.
- На компьютере откройте PolaraRS программы.
- На главной странице будет таблица с перечнем документов, подключенного к устройству RapidStak. VarioSkan должен быть на левой стороне этой таблицы. Под заголовком VarioSkan, там будет две ссылки, "RunSession" и "Инкубируйте". Нажмите "RunSession" вариант.
- Новое окно, анализ окна, появится с блок-схему в середине. Выберите "RunSession" пункт (он должен быть только настоящего пункта). С правой стороны экрана выпадающего менюпоявится. Найдите имя сохраненного SkanIt RE работать в этом меню.
Примечание: Там, кажется, никакого рационального заказа в это меню. Это не алфавитный или по дате добавления, или связанные с ним, где на компьютере протокол будет сохранен. Это означает, что вы должны просмотреть все протоколы, прежде чем нашел свой собственный, который является боль. - Как только вы выбрали протокол, в правом верхнем углу нажмите "Запустить анализ".
- Появится окно с запросом для обозначения которого журнал для использования в качестве источника, и который журнал пуст. Нижнем окне на экране соответствует передней журнала. [Дополнительно: Он попросит Вас ввести количество пластин загружены, чтобы определить оценку количества времени это займет. Эта оценка, как правило, далеко.]
- Убедитесь, что оба RapidStak и VarioSkan включены, и нажмите кнопку "OK".
- RapidStak будет автоматически загружать ваши тарелки в ридер, обеспечивая непрерывное измерение пластин. Чтобы прочитать 25 пластин занимает около 50 минут.
Примечание: Рекомендуется соблюдать первой загрузки пластины в VarioSkan для обеспечения надлежащего выравнивания машины. Если RapidStak не загружает ридер неправильно, то воспользуйтесь "Пауза этого анализа" кнопку в правом верхнем углу окна PolaraRS. - После VarioSkan осуществляется чтение всех пластин, удалите полный журнал и поместите его на платформу загрузки журнала. Поднимите внешний прямоугольник платформы, и журнал должен скользить, оставляя пластин стоя в кучу в центре. Замените крышки к соответствующему пластин.
3. Добавление Пробирной Mix на каждой пластине
- Подготовка анализа смеси использованием готовых 10x запас лизис смеси (10% Тритон Х-100, 100 мм RIS, 10 мМ ЭДТА) в 50 мМ калий ацетатного буфера при рН 5,5. Подготовить запас до 75mg/mL DNP-cellobioside субстрата в ДМСО, убедившись, что субстрат полного растворения. Добавить DNP-cellobioside маточного раствора для анализа смеси до конечной концентрации 0.1mg/mL. Каждая пластина будет использовать около 20 мл раствора, а также внести дополнительные 50 мл, чтобы обеспечить мертвый объем.
Примечание: DNP-Cellobioside не хорошо растворимы в воде, поэтому растворение в ДМСО увеличивает растворимость. Наличие ДМСО в окончательном решении анализ не имеет наблюдаемых эффектов. - Настройка qFill3 также инструкции на производителя с бутылкой СМИ прилагаются к многообразию с помощью стерильных трубок. Программа его на соответствующее количество средств массовой информации, и поставил его, чтобы заполнить 45uL объем в каждую лунку.
- Чистки воздуха из трубки и многообразием использования чистки особенность робота до средств массовой информации видно из каждого контактный многообразия.
- Добавить анализа смеси для каждой пластины с использованием qFill3. Каждая пластина занимает примерно 20 секунд, чтобы заполнить.
- После анализа смесь добавляется, инкубировать пластин при 37 ° С влажность поле в течение 12-16 часов.
Примечание: В течение инкубации пластин раз анализ смеси добавляется вызовет испарение во внешнем скважин пластины, уступая выше, чем считывание абсорбции видели внутри скважины. Это могут быть компенсированы путем инкубирования пластин в камере влажности.
4. Чтение поглощения анализировали клоны
- Удалить пластин от 37 ° C инкубатора. Удалите и отложите в сторону крышки, и место пластины на платформу журнал загрузки обеспечены RapidStak, до максимум 25 пластин.
Примечание: пластины на нижней части стека будет первым, кто будет читать. Убедитесь в том, чтобы отслеживать порядок пластин, а программное обеспечение не зафиксировать это. - Удалите один журнал из Быстрое Stak, убедитесь, что он пуст, и толкать ее в стек пластин для чтения. Захватите его за ручку, и все плиты должны быть загружены в журнале. Нагрузка магазин в RapidStak.
Примечание: Журнал винтик в углу, обозначающее какой угол A1 скважин плиты должны идти. Журнал будет только смонтировать в RapidStak в одной ориентации. - Откройте программу SkanIt RE на компьютере, подключенном к машинам. Выберите "New Session" и назовите его соответствующим образом. Выберите "Corning плоским дном 384-луночный планшет», как пластины типа.
- В "Пластина Макет" выберите "Мастер" кнопку, и выбрать его, чтобы добавить 384 неизвестных вашей тарелке.
- В "Протокол" выберите "Ну Loop" вариант, и ввести для 384 скважин. "Ну Loop" появится в потоке дерево на левой стороне экрана. Выберите значок и нажмите кнопку добавить "Фотометрическое измерение". Установите его читать на 400 нм.
- Сохраните протокол с уникальным именем, и закрыть программу SkanIt RE.
- На компьютере откройте PolaraRS программы.
- На главной странице будет таблица с перечнем документов, подключенного к устройству RapidStak. VarioSkan должен быть на левой стороне этой таблицы. Под заголовком VarioSkan, там будет две ссылки, "RunSession" и "Incubatе ". Нажмите" RunSession "вариант.
- Новое окно, анализ окна, появится с блок-схему в середине. Выберите "RunSession" пункт (он должен быть только настоящего пункта). С правой стороны экрана выпадающего меню. Найдите имя сохраненного SkanIt RE работать в этом меню.
- Как только вы выбрали протокол, в правом верхнем углу нажмите "Запустить анализ".
- Появится окно с запросом для обозначения которого журнал для использования в качестве источника, и который журнал пуст. Нижнем окне на экране соответствует передней журнала. [Дополнительно: Он попросит Вас ввести количество пластин загружены, чтобы определить оценку количества времени это займет. Эта оценка, как правило, далеко.]
- Убедитесь, что оба RapidStak и VarioSkan включены, и нажмите кнопку "OK"
- RapidStak будет автоматически загружать ваши тарелки в ридер, обеспечивая непрерывное измерение пластин.
Примечание: Рекомендуется соблюдать первой загрузки пластины в VarioSkan для обеспечения надлежащего выравнивания машины. Если RapidStak не загружает ридер неправильно, то воспользуйтесь "Пауза этого анализа" кнопку в правом верхнем углу окна PolaraRS. - После VarioSkan осуществляется чтение всех пластин, удалите полный журнал и поместите его на платформу загрузки журнала. Поднимите внешний прямоугольник платформы, и журнал должен скользить, оставляя пластин стоя в кучу в центре. Пластины могут быть утилизированы в соответствии с вашими процедурами лаборатории.
- Для экспорта считывание абсорбции, открытые RE SkanIt программного обеспечения, и выберите "Открыть существующий файл".
- Выберите каталог, в котором вы сохранили вашу сессию, нажмите значок "+", чтобы раскрыть список и под заглавие будут варианты с надписью "Контейнер 1" до "Контейнер N" в зависимости от количества пластин. Выберите первый пластины для анализа.
- В строке меню в верхней части страницы, выберите "Обработка данных> Отчет / Экспорт"
- Выбрать варианты желаемого для экспорта.
Примечание: Я обычно выбирает "Пластина Layout" и "Фотометрические" данные для экспорта. - Нажмите кнопку "Просмотреть отчет", а затем "Файл> Сохранить". Выберите нужный каталог. Выходной формат электронных таблиц Microsoft Excel
5. Представитель Результаты
Примером считывание абсорбции из одного 384-луночного планшета содержащие положительные клон показано на рисунке 2. Положительные клоны показывают заметное увеличение абсорбции за тех, кто не выразил целлюлазы деятельности. Различия в анализе времени, а место на пластинке, или ДНП концентрации (может быть введена за счет фильтрации не-растворенных DNP) может повлиять на показания абсолютного поглощения. Относительные показания оптической плотности, таких как различия в абсорбции выше средней пластины или столбца среднем, более надежный метод идентификации целлюлазы положительных клонов.
После выявления положительных клонов из библиотеки пластины, рекомендуется, чтобы повторить все положительные клоны в новой пластинкой для вторичной проверки. Это позволяет устранить эффекты, возникающие от расположения скважины или пластину вариации и позволяет более прямое сравнение между положительными клонов.

Рисунок 1. Блок-схема высокой пропускной способностью для анализа метагеномных библиотеки клонирован в E. палочки и хранили при -80 ° C.
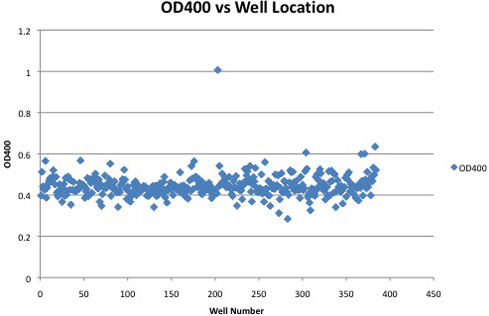
Рисунок 2. Поглощение показания от одного 384-луночного планшета содержащих положительный клон. Целлюлаза положительных клонов можно определить по значительно увеличили поглощения над негативными клонов.
Обсуждение
Высокая пропускная способность экрана для быстрого обнаружения целлюлолитических деятельности от большой библиотеки геномной ДНК вставки метагеномных выражается в Е. кишечной описано в данном протоколе. Этот метод является улучшением по сравнению с CMC / конго красный анализ шир...
Раскрытие информации
Благодарности
Авторы хотели бы поблагодарить д-р Стив Холка и Гонг-Ming Chen за предоставление DNP-Cellobioside подложки.
Материалы
| Name | Company | Catalog Number | Comments |
| qPix2 | Genetix | With 384-pin gridding head | |
| qFill3 | Genetix | With 384-well manifold | |
| Varioskan | Thermo Fisher Scientific, Inc. | ||
| RapidStak | Thermo Fisher Scientific, Inc. | Connected to Varioskan | |
| Micro90 Detergent | Cole-Parmer | 18100-00 | Diluted to 2% in water |
| Ethanol | Major Lab Supplier | Diluted to 80% in water | |
| Chloramphenicol | Sigma-Aldrich | C0378 | 12.5mg/mL in ethanol |
| LB broth, Miller | Fisher Scientific | BP1426-2 | 25g/L, autoclaved |
| 384-well flat bottom plates | Corning | 3680 | |
| L-(+)-Arabinose | Sigma-Aldrich | A3256 | 100mg/mL in water |
| Potassium Acetate | Fisher Scientific | P171 | 50mM in water, autoclaved, adjusted to pH 5.5 with HCl |
| Triton X-100 | Fisher Scientific | BP151 | |
| Trizma hydrochloride | Sigma-Aldrich | T3253 | In TE buffer solution, 100mM |
| EDTA disodium salt | Sigma-Aldrich | E5134 | In TE buffer solution, 10mM |
| 2,4-dinitrophenyl cellobioside | Provided by Dr. Steve Withers, UBC | ||
| Dimethyl Sulfoxide | Sigma-Aldrich | D8418 |
Ссылки
- Rubin, E. M. Genomics of cellulosic biofuels. Nature. 454, 841-845 (2008).
- Voget, S., Steele, H. L., Streit, W. R. Characterization of a metagenome derived halotolerant cellulase. J. Biotechnol. 126, 26-36 (2006).
- Duan, C. J. Isolation and partial characterization of novel genes encoding acidic cellulases from metagenomes of buffalo rumens. J. Appl. Microbiol. 107, 245-256 (2009).
- Zhang, Y. H. P. Outlook for cellulase improvement: Screening and selection strategies. Biotech. Advances. 24, 452-481 (2006).
- Teather, R. M., Wood, P. J. Use of congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl. And Environ. Microbiol. 43, 777-780 (1982).
- Sharrock, K. R. Cellulase assay methods: a review. J. Biochem and Biophys Methods. 17, 81-106 (1988).
- Kasana, R. C., Salwan, R., Dhar, H., Dutt, S., Gulati, A. A rapid and easy method for detection of microbial cellulases on agar plates using Gram's Iodine. Curr. Microbiology. 57, 503-507 (2008).
- Huang, J. S., Tang, J. Sensitive assay for cellulase and dextranase. Anal. Biochem. 73, 369-377 (1976).
- Tull, D., Withers, S. G. Mechanisms of cellulases and xylanases: A detailed kinetic study of the exo-beta-1,4-glycanase from Cellulomonas fimi. Biochemistry. 33, 6363-6370 (1994).
- Martinez, A., Bradley, A. S., Waldbauer, J. R., Summons, R. E., DeLong, E. F. Proteorhodopsin photosystem gene expression enables photophosphorylation in a heterologous host. PNAS. 104, 5590-5595 (2007).
- Taupp, M., Lee, S., Hawley, A., Yang, J., Hallam, S. J. Large Insert Environmental Genomic Library Production. J Vis Exp. , (2009).
- Martinez, A., Tyson, G. W., DeLong, E. F. Widespread known and novel phosphonate utilization pathways in marine bacteria revealed by functional screening and metagenomic analyses. Environ Microbiol. 12, 222-238 (2010).
- Wild, J., Hradecna, Z., Szybalski, W. Conditionally amplifiable BACs: Switching from single-copy to high-copy vectors and genomic clones. Genome Research. 12, 1434-1444 (2002).
- Johnson, E. A. Sacchirification of complex cellulosic substrates by the cellulase system from Clostridium thermocellum. Appl Environ Microbiology. 43, 1125-1132 (1982).
- Stutzenberger, F. J. Cellulase Production by Thermomonospora curvata isolated from municipal solid waste compost. Appl Environ Microbiol. 22, 147-152 (1971).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены