Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein High-Throughput-Screen für Biomining Cellulase Aktivität von Metagenombanken
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen hohen Durchsatz Bildschirm für cellulolytische Aktivität von einem metagenomische Bibliothek in Escherichia coli exprimiert. Der Bildschirm ist Lösung und hoch automatisiert und nutzt Ein-Topf-Chemie in 384-Well-Mikroplatten mit dem letzten Auslesen als Extinktionsmessung.
Zusammenfassung
Cellulose, die ergiebigste Quelle von organischem Kohlenstoff auf der Erde, verfügt über weit reichende industrielle Anwendungen mit zunehmendem Schwerpunkt auf die Produktion von Biokraftstoffen 1. Chemische Methoden zu ändern oder zu degradieren Zellulose erfordern in der Regel starke Säuren und hohen Temperaturen. Als solche haben enzymatischen Methoden werden in der biologischen Umwandlung Prozess im Vordergrund. Während die Identifizierung von aktiven Cellulasen aus Bakterien und Pilzen isoliert wurde etwas effektiver, widerstehen die überwiegende Mehrheit der Mikroben in der Natur Labor Anbau. Environmental Genomics, auch als Metagenom, Screening-Ansätze bekannt sind äußerst viel versprechend bei der Überbrückung der Anbau Lücke in der Suche nach neuen Biokonversion Enzyme. Metagenomische Screening-Methoden erfolgreich neuartige Cellulasen aus Umgebungen wieder wie als Böden 2, Büffel Pansen 3 und die Termiten Enddarm 4 mit Carboxymethylcellulose (CMC)-Agarplatten mit Kongorot-Farbstoff (basierend auf der Methode der Teather und Holz 5) gefärbt vielfältig. Doch die CMC-Methode in den Durchsatz begrenzt ist, ist nicht quantitativ und manifestiert ein niedriges Signal-Rausch-Verhältnis 6. Andere Methoden wurden 7,8 berichtet, aber jeder Verwendung einer Agar-Platte-basierter Assay, was unerwünscht für das Hochdurchsatz-Screening von großen einfügen genomische Bibliotheken ist. Hier präsentieren wir eine Lösung basierende Bildschirm für Cellulase-Aktivität unter Verwendung eines chromogenen Dinitrophenol (DNP)-cellobiosid Substrat 9. Unsere Bibliothek wurde in den pCC1 Kopierschutz Fosmid zu Assaysensitivität durch Kopienzahl Induktion 10 erhöhen geklont. Das Verfahren nutzt Ein-Topf-Chemie in 384-Well-Mikroplatten mit der endgültigen Anzeige als Extinktionsmessung zur Verfügung gestellt. Diese Anzeige ist quantitativ, empfindlich und automatisiert mit einem Durchsatz von bis zu 100x 384-Well-Platten pro Tag mit einem Liquid Handler und Platten-Lesegerät mit angeschlossenem Stapelsystem.
Protokoll
Bevor dieses Protokoll, müssen Sie Ihre metagenomische Bibliothek in einer 384-Well-Platte-Format gespeichert. In unserer Studie haben wir die pCC1 Kopierschutz Fosmid Vektor in Kombination mit dem Phagen T1-resistente TransforMax EPI300-T1 R E. coli-Zellen, da die Bibliothek Host gespeichert und unsere Platten bei -80 ° C 11.
1. Die Replikation der metagenomische Bibliothek Plates
- Defrost die Platten mit Ihrer Bibliothek bei 37 ° C für ca. 20 Minuten, oder bis alle Brunnen sind aufgetaut.
- Verwenden Sie das UV-Licht sterilisiert Funktion auf dem qPix2, um den Roboter für 15 Minuten zu sterilisieren.
- Bereiten LB-Medium mit Chloramphenicol in einer Endkonzentration von 12.5ug/mL und Arabinose bei 100ug/mL in einem 500 ml Reagenzflasche. Jede Platte verwenden ca. 20 ml, plus einen zusätzlichen 50 mL für Totvolumen zu ermöglichen.
- Richten Sie die qFill3 gemäß den Anweisungen des Herstellers mit den Medien Flasche an den Verteiler über die sterile Schlauch befestigt. Programm ist für die entsprechende Menge an Medien, und setzen Sie ihn auf eine 45uL Volumen in jede Vertiefung zu füllen.
- Spülen Sie die Luft aus dem Schlauch und Verteiler mit der Säuberung Funktion des Roboters bis Medien zu sehen ist, die aus jedem Pin des Verteilers.
- Füllen Sie die gewünschte Anzahl von Platten mit LB-Medium mit dem qFill3. Jede Platte dauert ca. 20 Sekunden zu füllen.
- Laden Sie die Bibliothek Platten und die frische Platten in die entsprechenden Bereiche der qPix2 Roboter. Füllen Sie die Reinigungsbäder mit den entsprechenden Reagenzien, 2% Micro90 im hinteren Bad, autoklaviert destilliertem Wasser in der Mitte Bad, 80% Ethanol in den vorderen Bad.
- Verwenden Sie die "Replizieren"-Programm der qPix2. In der Software, wählen Sie die entsprechende Kopf, Anzahl und Art der Quelle Platten-und Ziel-Platten. Auch bis der Kopf zwischen den Replikationen, in der Regel 6 Zyklen in jedem Bad sauber eingestellt ist genug. Kapazität der Maschine ist 10 Platten in einer Zeit, und es dauert etwa 15 Minuten, um sie alle mit einem 384 Pin Kopf (oder ca. 50 Minuten mit einem 96 pin Kopf) zu replizieren.
Hinweis: Es gibt eine Option, "Stir Source" oder "Stir Destination". Es wird empfohlen, nicht zu verwenden, diese Optionen als Probleme vorher mit unserem Roboter mit ihnen aufgetreten sind. - Sobald die Platten repliziert werden, wachsen die Platten bei 37 ° C für 24 Stunden in einer feuchten Box. Weil wir Induktion einer hohen Kopienzahl Fosmid werden unter Zusatz von Arabinose, neigen die Klone wachsen langsamer als nicht-induzierten Klone. Return der Bibliothek Platten bis -80 ° C.
Hinweis: Die Inkubation in einer feuchten Box sorgt für Verdunstung von Medien nicht in den Brunnen am Rand der Platten auftreten, halten eine einheitliche Umgebung für alle Vertiefungen. - Reinigen Sie die qFill3 Roboter, indem Sie zuerst Spülen mit Wasser (~ 50ml) und dann 80% Ethanol (~ 50ml). Zerlegen der Komponenten und sauber und Autoklaven die Flasche, Deckel und Schlauch in Aluminiumfolie eingewickelt.
2. Messung des Wachstums von E. coli-Klone
- Entfernen Platten von 37 ° C Inkubator. Nehmen und beiseite stellen den Deckeln verschließen und Platten auf das Magazin Ladefläche mit dem RapidStak vorgesehen, bis zu einem Maximum von 25 Platten.
Hinweis: Die Platte an der Unterseite des Stapels wird der erste, der gelesen werden kann. Achten Sie darauf, den Überblick über die Reihenfolge der Platten zu halten, da die Software nicht aufzeichnen tut dies. - Nehmen Sie ein Magazin aus dem Rapid Stak, sicherzustellen, dass sie leer ist, und schieben Sie sie auf den Stapel der Platten zu lesen. Anmeldung durch den Griff, und die Platten sollten alle in das Magazin geladen werden. Laden Sie das Magazin in den RapidStak.
Hinweis: Das Magazin hat eine kleine Schraube in der Ecke bezeichnet die Ecke der A1 Vertiefungen der Platten gehen sollte. Das Magazin wird nur in der RapidStak Montage in eine Richtung. - Öffnen Sie die SkanIt RE-Programm auf den Computer angeschlossen, um die Maschinen. Wählen Sie "New Session" und nennen Sie sie entsprechend. Wählen Sie "Corning Flachboden 384-Well-Platte", wie die Platte geben.
- In der "Plate Layout"-Bereich, wählen Sie den "Wizard"-Schaltfläche, und wählen Sie es zu 384 Unbekannten auf Ihren Teller hinzuzufügen.
- In dem "Protokoll"-Bereich, wählen Sie die "Well-Loop"-Option, und geben Sie für 384 Wells. Ein "Gut Loop"-Symbol wird in den Fluss-Baum auf der linken Seite des Bildschirms angezeigt. Wählen Sie das Symbol und klicken Sie auf "Photometrische Messung". Setzen Sie ihn auf bei 600nm gelesen.
- Speichern Sie Ihre Protokoll mit einem eindeutigen Namen, und schließen Sie die SkanIt RE-Programm.
- Auf dem Computer, öffnen Sie die PolaraRS Programm.
- Auf der Hauptseite gibt es tabellarische Auflistung der Instrumente mit dem RapidStak Gerät sein. Die VarioSkan sollte auf der linken Seite dieser Tabelle. Unter der Überschrift VarioSkan, wird es zwei Links werden; "RunSession" und "Inkubation". Klicken Sie auf die "RunSession"-Option.
- Ein neues Fenster, das Assay-Fenster wird mit einem Flussdiagramm in der Mitte angezeigt. Wählen Sie den "RunSession" item (es sollte das einzige Element vorhanden sein). Auf der rechten Seite des Bildschirms ein Drop-Down-Menüwird angezeigt. Suchen Sie den Namen der gespeicherten SkanIt RE in diesem Menü ausgeführt werden.
Hinweis: Es scheint keine rationalen Ordnung, um dieses Menü werden. Es ist nicht alphabetisch oder nach Datum hinzugefügt oder im Zusammenhang mit dem auf dem Computer das Protokoll gespeichert wird. Das heißt, Sie müssen über alle Protokolle vor der Suche nach Ihrem eigenen, die Schmerzen zu suchen. - Sobald Sie Ihr Protokoll ausgewählt, in der oberen rechten Ecke klicken Sie auf "Run diesem Assay".
- Ein Feld wird angezeigt, in dem Sie bezeichnen die Zeitschrift als Quelle zu verwenden, und die Zeitschrift ist leer. Das untere Feld auf dem Bildschirm entspricht dem vorderen Magazin. [Optional: Es fordert Sie auf, die Anzahl der Platten geladen werden, um eine Schätzung für die Zeit, es dauert bestimmt geben. Diese Schätzung wird in der Regel weg.]
- Stellen Sie sicher, sowohl die RapidStak und die VarioSkan eingeschaltet sind, und drücken Sie "OK".
- Die RapidStak wird automatisch geladen Ihre Platten in die Platten-Lesegerät, so dass für die kontinuierliche Messung der Platten. So lesen 25 Stück dauert etwa 50 Minuten.
Hinweis: Es wird empfohlen, die erste Platte Laden in den VarioSkan beobachten, um die richtige Ausrichtung der Maschinen zu gewährleisten. Wenn die RapidStak nicht geladen wird die Platte Leser richtig, verwenden Sie die "Pause diesen Test"-Taste in der oberen rechten Ecke des PolaraRS Fenster. - Sobald die VarioSkan erfolgt das Lesen der Platten, entfernen Sie den vollen Magazin und legen Sie es auf das Magazin Ladefläche. Heben Sie die äußere Rechteck der Plattform, und das Magazin sollte abrutschen, so dass die Platten stehen in einem Haufen in der Mitte. Ersetzen Sie den Deckel auf den entsprechenden Platten.
3. Neben der Assay Mix zu jeder Platte
- Bereiten Assay-Mix mit vorgefertigten 10fach Lyse-Mix (10% Triton X-100, 100 mM ris, 10 mM EDTA) in 50 mM Kalium-Acetat-Puffer bei pH 5,5. Bereiten Sie einen Vorrat von bis zu 75mg/ml DNP-cellobiosid Substrat in DMSO, so dass das Substrat vollständig gelöst ist. Add DNP-cellobiosid Stammlösung zu Assay-Mix, um eine endgültige Konzentration von 0.1mg/mL. Jede Platte verwenden ca. 20 mL der Lösung, plus einen zusätzlichen 50 mL für Totvolumen zu ermöglichen.
Hinweis: DNP-cellobiosid ist nicht leicht löslich in Wasser, so löst in DMSO erhöht die Löslichkeit. Die Anwesenheit von DMSO in der letzten Test-Lösung hat keine beobachtbaren Effekte. - Richten Sie die qFill3 gemäß den Anweisungen des Herstellers mit den Medien Flasche an den Verteiler über die sterile Schlauch befestigt. Programm ist für die entsprechende Menge an Medien, und setzen Sie ihn auf eine 45uL Volumen in jede Vertiefung zu füllen.
- Spülen Sie die Luft aus dem Schlauch und Verteiler mit der Säuberung Funktion des Roboters bis Medien zu sehen ist, die aus jedem Pin des Verteilers.
- Add-Assay-Mix zu jeder Platte mit dem qFill3. Jede Platte dauert ca. 20 Sekunden zu füllen.
- Sobald Assay Mix zugegeben, inkubieren Sie die Platten bei 37 ° C in einer feuchten Box für 12-16 Stunden.
Hinweis: Over-Inkubation von Platten, wenn der Assay-Mix hinzugefügt wird Verdunstung in den äußeren Vertiefungen der Platte verursachen, was zu höheren Absorptionswerte als für die Innen-Brunnen zu sehen. Dies kann durch Inkubation Platten in eine feuchte Kammer ausgeglichen werden.
4. Messung der Absorption von Assay-Clones
- Entfernen Platten von 37 ° C Inkubator. Nehmen und beiseite stellen den Deckeln verschließen und Platten auf das Magazin Ladefläche mit dem RapidStak vorgesehen, bis zu einem Maximum von 25 Platten.
Hinweis: Die Platte an der Unterseite des Stapels wird der erste, der gelesen werden kann. Achten Sie darauf, den Überblick über die Reihenfolge der Platten zu halten, da die Software nicht aufzeichnen tut dies. - Nehmen Sie ein Magazin aus dem Rapid Stak, sicherzustellen, dass sie leer ist, und schieben Sie sie auf den Stapel der Platten zu lesen. Anmeldung durch den Griff, und die Platten sollten alle in das Magazin geladen werden. Laden Sie das Magazin in den RapidStak.
Hinweis: Das Magazin hat eine kleine Schraube in der Ecke bezeichnet die Ecke der A1 Vertiefungen der Platten gehen sollte. Das Magazin wird nur in der RapidStak Montage in eine Richtung. - Öffnen Sie die SkanIt RE-Programm auf den Computer angeschlossen, um die Maschinen. Wählen Sie "New Session" und nennen Sie sie entsprechend. Wählen Sie "Corning Flachboden 384-Well-Platte", wie die Platte geben.
- In der "Plate Layout"-Bereich, wählen Sie den "Wizard"-Schaltfläche, und wählen Sie es zu 384 Unbekannten auf Ihren Teller hinzuzufügen.
- In dem "Protokoll"-Bereich, wählen Sie die "Well-Loop"-Option, und geben Sie für 384 Wells. Ein "Gut Loop"-Symbol wird in den Fluss-Baum auf der linken Seite des Bildschirms angezeigt. Wählen Sie das Symbol und klicken Sie auf "Photometrische Messung". Setzen Sie ihn auf bei 400 nm gelesen.
- Speichern Sie Ihre Protokoll mit einem eindeutigen Namen, und schließen Sie die SkanIt RE-Programm.
- Auf dem Computer, öffnen Sie die PolaraRS Programm.
- Auf der Hauptseite gibt es tabellarische Auflistung der Instrumente mit dem RapidStak Gerät sein. Die VarioSkan sollte auf der linken Seite dieser Tabelle. Unter der Überschrift VarioSkan, wird es zwei Links werden; "RunSession" und "INCUBATe ". Klicken Sie auf die" RunSession "-Option.
- Ein neues Fenster, das Assay-Fenster wird mit einem Flussdiagramm in der Mitte angezeigt. Wählen Sie den "RunSession" item (es sollte das einzige Element vorhanden sein). Auf der rechten Seite des Bildschirms ein Drop-Down-Menü erscheint. Suchen Sie den Namen der gespeicherten SkanIt RE in diesem Menü ausgeführt werden.
- Sobald Sie Ihr Protokoll ausgewählt, in der oberen rechten Ecke klicken Sie auf "Run diesem Assay".
- Ein Feld wird angezeigt, in dem Sie bezeichnen die Zeitschrift als Quelle zu verwenden, und die Zeitschrift ist leer. Das untere Feld auf dem Bildschirm entspricht dem vorderen Magazin. [Optional: Es fordert Sie auf, die Anzahl der Platten geladen werden, um eine Schätzung für die Zeit, es dauert bestimmt geben. Diese Schätzung wird in der Regel weg.]
- Stellen Sie sicher, sowohl die RapidStak und die VarioSkan eingeschaltet sind, und drücken Sie "OK"
- Die RapidStak wird automatisch geladen Ihre Platten in die Platten-Lesegerät, so dass für die kontinuierliche Messung der Platten.
Hinweis: Es wird empfohlen, die erste Platte Laden in den VarioSkan beobachten, um die richtige Ausrichtung der Maschinen zu gewährleisten. Wenn die RapidStak nicht geladen wird die Platte Leser richtig, verwenden Sie die "Pause diesen Test"-Taste in der oberen rechten Ecke des PolaraRS Fenster. - Sobald die VarioSkan erfolgt das Lesen der Platten, entfernen Sie den vollen Magazin und legen Sie es auf das Magazin Ladefläche. Heben Sie die äußere Rechteck der Plattform, und das Magazin sollte abrutschen, so dass die Platten stehen in einem Haufen in der Mitte. Die Platten können nach Ihrem Labor entsorgt werden.
- Um die Absorption Lesungen, offene SkanIt RE-Software, Export und wählen Sie "Öffnen einer vorhandenen Datei".
- Wählen Sie das Verzeichnis, in dem Sie Ihre Sitzung, auf den "+"-Symbol, um die Liste zu erweitern und unter dem Titel Überschrift wird Möglichkeiten der Beschriftung "Container 1" bis "Container N" in Abhängigkeit von der Anzahl der Platten sein. Wählen Sie die erste Platte analysiert werden.
- In der Menüleiste am oberen Rand der Seite, wählen Sie "Data Processing> Report / Export"
- Wählen Sie die Optionen für den Export gewünscht.
Hinweis: ich normalerweise wählen "Plate Layout" und "Photometrische" Daten für den Export. - Klicken Sie auf "View Report" und dann "Datei> Speichern". Wählen Sie das gewünschte Verzeichnis. Das Ausgabeformat ist eine Microsoft Excel-Tabelle
5. Repräsentative Ergebnisse
Ein Beispiel Absorptionswerte aus einer 384-Well-Platte mit einem positiven Klon ist in Abbildung 2 dargestellt. Positive Klone zeigen eine deutliche Zunahme der Absorption über diejenigen, die nicht zum Ausdruck Cellulase-Aktivität. Unterschiede in der Assay-Zeit, auch Standort auf der Platte oder DNP-Konzentration (kann durch das Herausfiltern von nicht gelösten DNP eingeführt werden) können sich auf absolute Absorptionswerte. Relative Absorptionswerte, wie der Unterschied in der Absorption oberhalb der Platte durchschnittliche oder Spalte Durchschnitt sind eine robuste Methode zur Identifizierung von Cellulase positive Klone.
Nach der Identifizierung der positiven Klone aus der Bibliothek Platten, ist es empfehlenswert, alle positiven Klone in eine neue Platte für sekundäre Screening replizieren. Dadurch entfällt Effekte aus gut Standort oder Platte Variation und sorgt für mehr direkte Vergleich zwischen positiven Klone.

Abbildung 1. Ablaufschema des High-Throughput-Assay für eine metagenomische Bibliothek in E. geklont coli und bei -80 ° C.
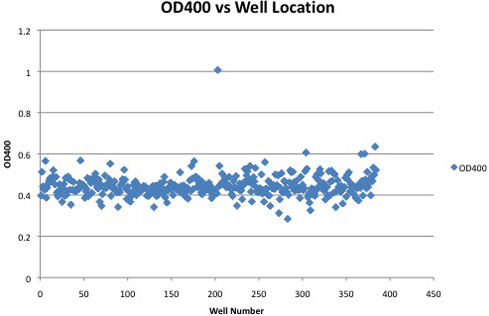
Abbildung 2. Absorptionswerte von einer 384-Well-Platte mit einem positiven Klon. Cellulase positive Klone können durch eine deutlich erhöhte Absorption über die negativen Klone identifiziert werden.
Diskussion
Ein hoher Durchsatz-Bildschirm für den schnellen Nachweis von cellulolytische Aktivität aus einer großen einfügen genomischer DNA metagenomische Bibliothek in E. ausgedrückt coli ist in diesem Protokoll beschrieben. Diese Methode ist eine Verbesserung gegenüber dem CMC / Congo Red Assay häufig in der Literatur verwendet. Es ist Lösung, und ermöglicht die Ein-Topf-Chemie-Screening in 384-Well-Platten, mit der endgültigen Ausgabe als Absorptionswerte von einem Platten-Lesegerät ermöglicht fü...
Offenlegungen
Danksagungen
Die Autoren bedanken sich bei Dr. Steve Withers und Hong-Ming Chen für die Bereitstellung von DNP-cellobiosid Substrat danken.
Materialien
| Name | Company | Catalog Number | Comments |
| qPix2 | Genetix | With 384-pin gridding head | |
| qFill3 | Genetix | With 384-well manifold | |
| Varioskan | Thermo Fisher Scientific, Inc. | ||
| RapidStak | Thermo Fisher Scientific, Inc. | Connected to Varioskan | |
| Micro90 Detergent | Cole-Parmer | 18100-00 | Diluted to 2% in water |
| Ethanol | Major Lab Supplier | Diluted to 80% in water | |
| Chloramphenicol | Sigma-Aldrich | C0378 | 12.5mg/mL in ethanol |
| LB broth, Miller | Fisher Scientific | BP1426-2 | 25g/L, autoclaved |
| 384-well flat bottom plates | Corning | 3680 | |
| L-(+)-Arabinose | Sigma-Aldrich | A3256 | 100mg/mL in water |
| Potassium Acetate | Fisher Scientific | P171 | 50mM in water, autoclaved, adjusted to pH 5.5 with HCl |
| Triton X-100 | Fisher Scientific | BP151 | |
| Trizma hydrochloride | Sigma-Aldrich | T3253 | In TE buffer solution, 100mM |
| EDTA disodium salt | Sigma-Aldrich | E5134 | In TE buffer solution, 10mM |
| 2,4-dinitrophenyl cellobioside | Provided by Dr. Steve Withers, UBC | ||
| Dimethyl Sulfoxide | Sigma-Aldrich | D8418 |
Referenzen
- Rubin, E. M. Genomics of cellulosic biofuels. Nature. 454, 841-845 (2008).
- Voget, S., Steele, H. L., Streit, W. R. Characterization of a metagenome derived halotolerant cellulase. J. Biotechnol. 126, 26-36 (2006).
- Duan, C. J. Isolation and partial characterization of novel genes encoding acidic cellulases from metagenomes of buffalo rumens. J. Appl. Microbiol. 107, 245-256 (2009).
- Zhang, Y. H. P. Outlook for cellulase improvement: Screening and selection strategies. Biotech. Advances. 24, 452-481 (2006).
- Teather, R. M., Wood, P. J. Use of congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl. And Environ. Microbiol. 43, 777-780 (1982).
- Sharrock, K. R. Cellulase assay methods: a review. J. Biochem and Biophys Methods. 17, 81-106 (1988).
- Kasana, R. C., Salwan, R., Dhar, H., Dutt, S., Gulati, A. A rapid and easy method for detection of microbial cellulases on agar plates using Gram's Iodine. Curr. Microbiology. 57, 503-507 (2008).
- Huang, J. S., Tang, J. Sensitive assay for cellulase and dextranase. Anal. Biochem. 73, 369-377 (1976).
- Tull, D., Withers, S. G. Mechanisms of cellulases and xylanases: A detailed kinetic study of the exo-beta-1,4-glycanase from Cellulomonas fimi. Biochemistry. 33, 6363-6370 (1994).
- Martinez, A., Bradley, A. S., Waldbauer, J. R., Summons, R. E., DeLong, E. F. Proteorhodopsin photosystem gene expression enables photophosphorylation in a heterologous host. PNAS. 104, 5590-5595 (2007).
- Taupp, M., Lee, S., Hawley, A., Yang, J., Hallam, S. J. Large Insert Environmental Genomic Library Production. J Vis Exp. , (2009).
- Martinez, A., Tyson, G. W., DeLong, E. F. Widespread known and novel phosphonate utilization pathways in marine bacteria revealed by functional screening and metagenomic analyses. Environ Microbiol. 12, 222-238 (2010).
- Wild, J., Hradecna, Z., Szybalski, W. Conditionally amplifiable BACs: Switching from single-copy to high-copy vectors and genomic clones. Genome Research. 12, 1434-1444 (2002).
- Johnson, E. A. Sacchirification of complex cellulosic substrates by the cellulase system from Clostridium thermocellum. Appl Environ Microbiology. 43, 1125-1132 (1982).
- Stutzenberger, F. J. Cellulase Production by Thermomonospora curvata isolated from municipal solid waste compost. Appl Environ Microbiol. 22, 147-152 (1971).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten