このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
に関与する宿主因子のハイスループットスクリーニングのための顕微鏡ベースのアッセイ
* これらの著者は同等に貢献しました
要約
Two assays for microscopy-based high-throughput screening of host factors involved in Brucella infection are described. The entry assay detects host factors required for Brucella entry and the endpoint assay those required for intracellular replication. While applicable for alternative approaches, siRNA screening in HeLa cells is used to illustrate the protocols.
要約
ブルセラ種は、それらの天然の宿主としての動物に感染する通性細胞内病原体です。ヒトへの送信は、最も一般的に感染した動物との直接接触により、または汚染された食品の摂取によって引き起こされ、重度の慢性感染症につながることができます。
ブルセラ菌は、プロと非専門的な食細胞に侵入し、小胞体(ER)由来の液胞内で複製することができます。 ER-ような区画内のブルセラ宿主細胞への侵入、リソソーム分解の回避、および複製に必要な宿主因子はほとんどわかっていません。ここでは、HeLa細胞におけるブルセラエントリおよび複製に関与する宿主因子を同定するために2つのアッセイを説明します。代替的なスクリーニング方法が適用できるがプロトコルは、RNA干渉の使用を記載しています。アッセイは、蛍光標識された細菌の検出自動使用して蛍光標識された宿主細胞に基づいています広視野顕微鏡。蛍光画像は、単一の細胞ベースの感染スコアリングを可能CellProfilerで標準化された画像解析パイプラインを用いて分析します。
エンドポイントアッセイでは、細胞内複製は感染の2日後に測定されます。これは、増殖が細菌侵入後12時間の周りに開始され、その複製ニッチへのトラフィックへの細菌を許可します。正常に細胞内ニッチを確立したブルセラ菌は、このように強く、宿主細胞内で増殖しています。細胞内細菌が大きく、個々の細胞外または細胞内の非複製の細菌数を上回るので、構成的にGFPを発現する株を使用することができます。強力なGFP信号は、感染した細胞を同定するために使用されます。
対照的に、エントリアッセイのために、細胞内および細胞外の細菌を区別することが重要です。ここでは、テトラサイクリン誘導性GFPのためのひずみエンコードが使用されます。誘導ゲンタマイシンによる細胞外細菌の同時不活性化とGFPの唯一の細胞内細菌は、GFPを発現することができることで、細胞内およびGFP信号に基づいて、細胞外細菌との区別を可能にします。細胞内増殖が開始される前に、これは、単一の細胞内細菌のロバストな検出を可能にします。
概要
ブルセラ種は、αプロテオバクテリアのクラスに属するグラム陰性、通性細胞内病原体です。彼らはそのような流行地域における深刻な経済的損失をもたらしウシ、ヤギ、またはヒツジなどのそれらの自然宿主における中絶や不妊の原因となります。ブルセラ症は、世界的に毎年1万人以上の新しいヒトへの感染を引き起こす最も重要な人獣共通感染症の一つです。人間への送信は、最も一般的に感染した動物との直接接触により、または低温殺菌牛乳のような汚染された食品の摂取によって引き起こされます。熱性疾患の症状は、ブルセラ症の診断に困難を引き起こす、非特異的です。未治療の場合、患者は、関節炎、心内膜炎、及び神経障害2などのより重篤な症状と慢性感染症を発症することができます。
細胞レベルではブルセラ菌は食細胞および非貪食細胞に侵入することができ、細胞内内で複製しますブルセラ含有液胞(BCV)として知られているコンパートメント。細菌の内部移行は、RAC、ロー、およびCdc42の3の直接的な活性化によって、アクチン細胞骨格の再編成が必要です。真核生物宿主細胞の内部では、BCVは、エンドサイトーシス経路に沿って入稿するとリソソームとの相互作用にもかかわらず、細菌が分解4を避けるために管理します。水疱性ATPアーゼによってBCVの酸性化は、細菌IV型分泌系(T4SS)5の発現を誘導するために必要とされます。 ブルセラ菌は 、その複製ニッチを確立するT4SSによって分泌された細菌のエフェクターは、細胞内ニッチ7の確立の欠陥にT4SS 6の削除や水疱性ATPアーゼリードの阻害するので、必須であると考えられています。細菌は彼らまで人身売買のフェーズ中に複製されません ER由来液胞コンパートメント8に達します。細胞内増殖が発生すると、BCVはCLに見出されますこのようなカルネキシンおよびグルコース-6-ホスファターゼ6のようなERマーカーと大証の関連。
ブルセラ菌は、ER-ような区画内に、細胞に入り、リソソーム分解を回避し、最終的に複製される分子メカニズムはほとんどわかっていません。感染の異なる段階に関与する宿主因子は、主にターゲットにアプローチまたはショウジョウバエ細胞9で行われる小規模な低分子干渉RNA(siRNA)は、画面によって同定されています。これらは、 ブルセラ感染の間の個々の宿主因子の寄与に光を当てるしているが、我々は、プロセス全体の包括的な理解程遠いです。
ここでは、自動化された広視野蛍光顕微鏡および自動画像解析と組み合わせて、大規模なRNA干渉(RNAi)スクリーニングを用いてヒト宿主因子の同定を可能にするプロトコルが提供されます。逆HeLa細胞のsiRNAトランスフェクションはdescribように行われますエド以前10,11のマイナーな修正を加えました。エンドポイントアッセイは、出力および隣接細胞の感染を除くブルセラ細胞内のライフサイクルの大部分を覆っています。さらに、エンドポイントアッセイで同定されたヒットを特徴付けるために、感染の初期段階に関与する因子を同定するための修正されたプロトコルが使用されます。
構成的にGFPを発現ブルセラ流産株は、細菌を二日間細胞を感染させて、エンドポイントアッセイのために使用されます。この間、細菌は細胞、ER由来の複製ニッチへのトラフィックを入力し、核周囲の空間に複製します。 GFP信号のハイレベルは、その後、確実に複製する細菌が含まれている個々の細胞を検出するために使用することができます。
ハイスループットアッセイにおけるブルセラエントリを研究するために、細胞内および細胞外の細菌を区別できることが重要です。この方法は、ここに提示circumv細胞内および細胞外の細菌のエント差動抗体染色。これは、DsRedのの構成的発現と組み合わせたテトラサイクリン誘導性GFPを発現ブルセラ株に基づいています。構成のDsRedマーカーの存在は、試料中に存在するすべての細菌の同定を可能にします。 GFP発現は、ゲンタマイシン(GM)によって細胞外の細菌の不活性化と同時に非毒性テトラサイクリンアナログアンヒドロテトラサイクリン(ATC)の添加によって誘導されます。細胞不透過性抗生物質Gmは細胞外細菌を死滅させる一方で、ATCは、宿主細胞に入り、細胞内細菌に選択的にGFPの発現を誘導することができます。このデュアルレポーターは、広視野顕微鏡を用いて細胞外細菌(のみのDsRed信号)からの単一細胞内細菌(GFPとDsRedの信号)の堅牢な分離を可能にします。細胞内ブルセラによって検出可能なGFP発現を達成するために、我々は、ATCによる誘導の4時間はRELIになることを見出しましたできる信号。選択的細胞内細菌でGFPを発現するための同様の誘導方式は、細胞内の赤痢菌 12を研究するために以前に使用されてきました。
プロトコル
注意:ライブウシ流産菌の株を持つすべての作業は、すべての必要な規制や安全上の注意事項を考慮しバイオセーフティーレベル3(BSL3)実験室の内部で実行する必要があります。
1.スクリーニングプレートの調製および細菌および細胞の培養
- ウシ流産菌スターター培養物の調製
- ストリークブルセラ 50μg/ mlのカナマイシン(TSA /キロ)を含むトリプシン大豆寒天(TSA)プレート上で-80℃のミルクストックから2308(B.流産 ) 流産 。 37℃で3〜4日間プレートをインキュベート。エントリーアッセイのために2308 pJC43(apHT :: GFP) アボルタス歪みブルセラエンドポイントアッセイのための13とひずみウシ流産菌 pAC042.08(apht :: DsRedを、のtetO ::にtetR-GFP)を使用します。
注: ウシ流産菌の円滑なリポポリサッカライド(LPS)が重要な毒性決定因子であるが、大まかな変異体は、比較的高い周波数14で発生する可能性があります。 WEは、このように、この実験で始まる前に、培養株の状態をテストすることをおすすめします。 - フルプレートをカバーする4 TSA /キロプレート上で画線細菌と37℃で2〜3日間インキュベートします。
- 10ミリリットル10パーセント、オートクレーブ処理スキムミルクでTSA /キロプレートから使い捨てプラスチックループ、転送および再懸濁菌を使用しました。細菌はよくすべてのスターター培養物における一貫性のある細菌の濃度を確実にするために再懸濁されていることを確認します。
- 2ミリリットルに細菌懸濁液のアリコート250μlを-80℃でキャップチューブ、凍結をねじ込みます。
- 50μg/ mlのカナマイシン(TSB /キロ)を含む50 mlのトリプシン大豆ブロス(TSB)とキャップボトルスクリュースターター培養物のアリコートを解凍し、250ミリリットルを分離するために25μlの、50μlの、および細菌のスターター培養物100μlを移します。パラフィルムでボトルを密封します。
- 100 rpmで37℃でオービタルシェーカー上で一晩ボトルをインキュベートします。
- 600 nにおける光学密度を測定M(OD 600)を一晩培養した後、OD 600 = 0.8~1.1に達するのに必要なスターター培養物の容積を決定します。
- ストリークブルセラ 50μg/ mlのカナマイシン(TSA /キロ)を含むトリプシン大豆寒天(TSA)プレート上で-80℃のミルクストックから2308(B.流産 ) 流産 。 37℃で3〜4日間プレートをインキュベート。エントリーアッセイのために2308 pJC43(apHT :: GFP) アボルタス歪みブルセラエンドポイントアッセイのための13とひずみウシ流産菌 pAC042.08(apht :: DsRedを、のtetO ::にtetR-GFP)を使用します。
- HeLa細胞の調製
- ダルベッコでHeLa細胞の成長を、5%CO 2、80%コンフルエントに細胞を増殖させると、37℃湿ったインキュベーター中で、10%ウシ胎児血清(FCS)(DMEM / 10%FCS)を補充したイーグル培地(DMEM)を改変しました。
- siRNAでスクリーニングプレートを準備します。
- 0.32μMの最終濃度になるようにRNaseフリー水でsiRNAを希釈します。 5μlを採取/ウェルに黒色の透明ウェル平底384ウェルプレート。
- 標準コントロールの列1、2、23、および24を使用してください。陰性対照のために、siRNAは(唯一のトランスフェクション試薬)ことなく、非標的とするsiRNA(スクランブル)およびモック井戸を使用しています。トランスフェクションコントロールの場合、細胞死を誘導するsiRNA(KIF11)、ならびにブルセラ感染症( 例えば 、ARPC3に関与する既知の宿主因子のポジティブコントロールを使用アクチンの重合に関与ARP2 / 3複合体)の構成要素。残りのウェルに市販のsiRNAライブラリーまたはカスタムsiRNAを転送します。
- 剥離可能なアルミ箔でシールプレート。 -20℃で保存プレート。
注意:プレートを、製造業者の推奨に応じ年、少なくとも6ヶ月、最大-20℃で保存することができます。
- 0.32μMの最終濃度になるようにRNaseフリー水でsiRNAを希釈します。 5μlを採取/ウェルに黒色の透明ウェル平底384ウェルプレート。
2.リバースsiRNAのトランスフェクション
- 室温で20分間、300×gで遠心分離することにより黒色384ウェルプレートを解凍。
注:これは、蓋または井戸の側に付着する可能性のあるすべての液体が停止します。 - 室温でFCSを含まないDMEM中で200:トランスフェクション試薬1を希釈することによりトランスフェクション培地を準備します。もはや使用前に20分よりトランスフェクション培地を準備していません。慎重に使用する前に、溶液を混合。
- 25μlのトランスフェクション培地をよく試薬ディスペンサーを使用して、それぞれに追加しますそして、プレートを前後に移動させることにより、溶液を混合。
- siRNAの/トランスフェクション試薬複合体の形成を可能にするために、室温で1時間プレートをインキュベートします。
- 一方で、PBS中の2.5ミリリットル0.05%トリプシン-EDTA(トリプシン)で1回75cm 2のフラスコのサブコンフルエント細胞を洗浄することにより、HeLa細胞を準備します。
- 1.5ミリリットル新鮮なトリプシンを加え、細胞は切り上げるまで2-3分間37℃にフラスコを転送します。
- 10ミリリットル予め温めておいたDMEM / 16%FCSで細胞を再懸濁します。
- 自動細胞カウンターを用いて細胞を計数し、DMEM / 16%FCS中10,000細胞/ mlの細胞懸濁液を調製します。
- よく500細胞/その結果試薬ディスペンサーを使用して、各ウェルに50μlの細胞懸濁液を追加します。
- 細胞の均一な分布を達成するために、プレートを前後に移動します。細胞が落ち着くために可能にするために5〜10分間室温でプレートを残します。
- パラフィルムでプレートをシールし、予め温めておいたアルミニウムの72時間のためにそれをインキュベート5%CO 2で37℃、湿度の高いインキュベーターでプレート。
注:予熱したアルミ板は、全体の384ウェルプレート全体に均等な温度分布を可能にします。
3.感染と固定
- 感染前にある日、Bの1.1.7で決定された量を接種250ミリリットルで50ミリリットルTSB /キロ媒体における流産のスターター培養物は、キャップボトルをねじ込みます。パラフィルムでボトルを密封し、OD 600 = 0.8から1.1に37℃、オービタルシェーカー上で100rpmで一晩細菌を成長させます。
- 細菌培養物のOD 600を測定し、感染の所望の多重度(MOI)を達成するために、DMEM / 10%FCS中で細菌培養物を希釈することによって、感染培地を調製。
注:HeLa細胞における5〜10%の感染率を得るために、10,000 MOIが使用されます。 MOI滴定曲線は、最適な感染率を得るために、各細胞型のために行われるべきです。 - 384ウェルプレートのトランスフェクション培地を交換感染培地50μlの自動プレート洗浄機を使用して。
- 400×gで384ウェルプレートを遠心し、20分間、4°C。
注:これは、感染プロセスを同期するのに役立ちます。 - パラフィルムでプレートをシールし、4時間、5%CO 2で37℃、湿度の高いインキュベーター中で予熱したアルミ板上にそれをインキュベートします。
- (200μlの/ウェルオーバーフロー洗浄を使用)、自動プレート洗浄機を使用して、細胞外細菌を不活性化するためには100μg/ mlのGMを含むDMEM / 10%FCSで細胞を洗浄。
注意:オーバーフロー洗浄時には、自動プレート洗浄機を同時に分配し、培地を吸引。 - エントリーアッセイのために、自動プレート洗浄機を使用して、100μg/ mlのGMと100ng / mlのATCを含有するDMEM / 10%FCS(200μlの/ウェルオーバーフロー洗浄を使用)を用いて細胞をもう一度洗浄します。
- パラフィルムでプレートをシールし、別の5%CO 2で37℃、湿度の高いインキュベーター中で予熱したアルミ板に戻しますそれぞれのエンドポイントアッセイおよびエントリーアッセイ、40時間または4時間、。
- 固定のため、(200μL/ウェルオーバーフロー洗浄を使用)、自動プレート洗浄機を使用して、PBSでウェルを洗浄します。
- pH7.4の0.2MのHEPES中の50μlの3.7%パラホルムアルデヒド(PFA)、自動プレート洗浄機を使用してとの交流PBS。
注意:注意:PFAは、皮膚に接触すると飲み込む、吸入による毒性があります。 - 室温で20分間インキュベートします。
- 自動プレート洗浄機を用いて50μlのPBSで固定媒体を交換します。注:サンプルは現在、必要に応じてBSL3から取り出しの準備ができています。
注意:BSL3施設から任意のサンプルの除去手順のリスク評価と検証の対象であり、生物学的安全性のために適用される規制に依存します。
4.染色
- 50μlのPBSで細胞を2回洗浄します。
- 10分間、50μlの0.1%トリトンX-100 PBS中で細胞を透過性。
- W灰細胞をPBSで3回。 1μg/ mLのDAPI(4 '、6-ジアミジノ-2-フェニルインドール)を含有するPBSを20μlを加え、室温で30分間インキュベートします。
- PBSで細胞を3回洗浄し、アルミホイルで光から染色を保護します。
5.イメージング
- 画像取得のための自動化された広視野顕微鏡を設定します。
注:MetaXpressソフトウェアを使用して分子デバイスImageXpress顕微鏡の設定は以下の通りです。適切なハードウェアの仕様およびイメージングソフトウェアで代替2D広視野顕微鏡装置を使用することができる(詳細は材料/機器の表を参照のこと)。- 10X対物レンズ、カメラビニング= 1、ゲイン= 1を選択します。
- 384ウェルプレートに対応するプレートのフォーマットを選択します。
- ウェル当たり9サイトを取得します。
- 「レーザーベースフォーカスを有効にする」と「プレートとウェル底に焦点を当てる "を使用します。
- 初期よく用として「最初だけでなく "設定サイトのオートフォーカス用のサンプルと、「すべてのサイト」を見つけます。
- 手動Z-フォーカスオフセット用を設定するには、DAPIチャネルを使用してください。
- 手動で他の全てのチャネルのためのZ-オフセットDAPIからを選択します。
- 手動で低露出オーバーと広いダイナミックレンジを確保するために、すべてのチャンネルの露出時間を補正します。
- 自動露出機能を用いて、代表的な部位の画像を取得します。プレート全体に3-5サイトにイメージにこの露光時間を使用し、最も明るいピクセルはすべてサイト上で最大の明るさの〜80%に達するまで手動で露光時間を調整します。
- 画像のエンドポイントアッセイのためのDAPIやGFPのチャンネルを使用してフルプレート。エントリーアッセイのために加えて、RFPのチャンネルを選択します。
6.自動画像解析
注:CellProfiler 2 15は、携帯セグメントおよび細菌のオブジェクトに画像解析ソフトウェアとして採用され、measurを自動実行します特定されたオブジェクト内のements。ソフトウェアは、自動的に特定の画像解析タスクを実行するために、すべての画像に連続してモジュールを実行するパイプラインに結合することができる個々のモジュールの画像解析アルゴリズムを提供します。プロトコルに従うには、CellProfiler 2.1.1以降のバージョンをインストールします。そして、提供パイプラインをロードして、モジュール内の必要なパラメータを調整するには、以下の手順に従ってください。すべてのパイプラインの個々のモジュールの説明は補足ファイルで見つけることができます。
注:2つの別個のパイプラインは、各アッセイのために使用されます。第1のパイプラインは、分析の前に画像を補正するために第2のパイプラインによって使用されるシェーディングモデルを計算します。シェーディング補正は、顕微鏡からの不均一な光路の影響を低減するために画像に適用されます。第1のパイプラインでのシェーディングモデルを計算することは時間のかかるプロセスであるが、結果は、より高精度になります。
- エンドポイントアッセイ
- シェーディングモデルを計算します
- メニュー「ファイル」に移動し、「インポート」→「ファイルからパイプラインを...」を選択し、提供パイプライン「BrucellaShadingCorrectionPart1-Ver007」を選択します。レガシーパイプラインを変換するかどうかを尋ねたところ、答えは「変換しないでください」。
- 「出力」→「ビューの出力設定」に移動し、その結果、シェーディングモデル用画像と入力フォルダと出力フォルダを選択します。
- モジュールでは(1)「LoadImages」は、画像の名前と一致するように、「これらの画像に共通していることをテキスト "の名前を調整します。
- モジュールのいずれも前のモジュールの隣にすべて目のシンボルをオフにして、完全な分析を実行する彼らの画面を表示しないことを確認します。注:パイプラインの処理中にこれが大幅に必要な計算資源を減少させます。
- プレスシェーディングモデルの計算を開始するために「画像を分析します」。
- 実行して画像解析
- メニュー「ファイル」に移動し、「インポート」→「ファイルからパイプラインを...」を選択し、提供パイプライン「BrucellaEndpointWithShadingCorrectionPart2-Ver007」を選択します。レガシーパイプラインを変換するかどうかを尋ねたところ、答えは「変換しないでください」。
- 「出力」→「ビューの出力設定」に移動し、結果として得られるスプレッドシート用の画像と入力フォルダと出力フォルダを選択します。
- モジュールでは(1)「LoadImages」、画像の名前と一致するように、「これらの画像に共通していることをテキスト」の名前を調整します。
- モジュール(2)「LoadSingleImage」を、2シェーディングモデルの場所とファイル名を選択することで、6.1.1の下で計算されたシェーディングモデルをロードします。
- モジュール(4) - (5)「ImageMath」パラメータを調整し、顕微鏡カメラのビット深度に一致する」ことにより、第1の画像を掛け」。 8および16ビット用画像は、12ビットの画像はパラメータを設定するために、「1.0」への "16.0"パラメータを設定します。
- モジュール(9)「ImageMath」パラメータを調整し、核を抑制することなく、DAPI染色でブルセラ信号を抑制します値(0.25で開始)」により、第2の画像を掛け」。
- 「スタートテストモード」をクリックし、モジュールの対応するアイコンをクリックして一時停止シンボルとモジュール(9)の表示機能を有効にしてください。
注:活性化した場合、目のシンボルが開いている目が表示されている間、一時停止シンボルは、黄色で表示されます。 - アクティブなポーズ記号で(9)モジュールまでの分析を実行するには、「ファイル名を指定して実行」をクリックします。モジュール、押して "ステップ"の値をテストするには。
- 新しくオープンした画像を右クリックし、「正規化ログ」に「画像のコントラスト」に設定します。繰り返しバックモジュールへのステップ(9)、パラメータを調整する」ことにより、第二の画像を掛け」、およびトン以上のステップ同時にオーバー減算を示す黒い部分が結果画像に最小化されている間ブルセラ信号が十分に抑制されるまで、彼は、再びモジュール。
- 「スタートテストモード」をクリックし、モジュールの対応するアイコンをクリックして一時停止シンボルとモジュール(9)の表示機能を有効にしてください。
- モジュールでは(10)「IdentifyPrimaryObjects」、パラメータを設定し、「しきい値の下限 "十分に高い(ゼロで始まる)核をセグメント化され、背景は無視されるようになっています。
- モジュールの上にモジュール(10)、ステップのために良い値を特定し、ステップ6.1.2.6.1及び6.1.2.6.2で説明したように入力画像を表示します。
- 入力画像を右クリックし、ヒストグラムを表示します。
注:ヒストグラムのピークは、一般的な背景を示しています。 - 出力画像に表示されているように核の良好なセグメンテーションが達成されるまで、バックグラウンド強度から出発してパラメータの値を大きくします。
注:背景強度よりも高いしきい値が空のサイトのために特に重要である設定。
- モジュール内の(15)4;核内ではっきりと見えるブルセラ持つ細胞が維持され、他のすべてが離れてフィルタリングされるようにFilterObjectsは、「最小値」、パラメータを調整し、「このステップでは、核の外にブルセラを無視します 。
- 良い値は、モジュールの上にステップを識別し、ステップ6.1.2.6.1及び6.1.2.6.2で説明したように、出力画像を表示します。感染しているすべてのセルを識別する、ゼロから開始し、核ではっきりと見えるブルセラ持つ細胞のみが保持されるまで、小さなステップによって値を増やします。
- モジュール(16)「FilterObjects」では、perinucleusではっきりと見えるブルセラ持つ細胞が保持されるように、パラメータ「最小値」を調整し、他のすべてのセルを離れてフィルタリングされます。前のモジュールと同様のアプローチを使用してください。
- モジュール(17)「FilterObjects」で、パラメータ「最小値」を調整するようにはVorではっきりと見えるブルセラ持つ細胞onoi細胞体が保持され、そして他のすべてのセルを離れてフィルタリングされます。前のモジュールと同様のアプローチを使用してください。
注:ボロノイ電池本体は、隣接するボロノイ細胞体との重複がないと25ピクセル核の半径方向の拡張です。 - テストモードを終了し、モジュールのいずれも前のモジュールの隣にすべて目のシンボルをオフにして、完全な分析を実行する彼らの画面を表示しないことを確認してください。
注:パイプラインの処理中にこれが大幅に必要な計算資源を減少させます。 - 押して分析を開始するには「画像を分析します」。
- 解析が終了すると、分析がプレート全体に確実に働いていたことを確実にするために、得られたPNG画像だけでなく、CSVシートを検査します。
- シェーディングモデルを計算します
- エントリーアッセイ
- シェーディングモデルを計算します
- 「インポート」→「ファイルからパイプラインを...」を選択し、メニューの「ファイル」に移動し、提供を選択Dパイプライン」BrucellaShadingCorrectionPart1-Ver007」。レガシーパイプラインを変換するかどうかを尋ねたところ、答えは「変換しないでください」。
- 「出力」→「ビューの出力設定」に移動し、その結果、シェーディングモデル用画像と入力フォルダと出力フォルダを選択します。
- モジュールでは(1)「LoadImages」、画像の名前と一致するように、「これらの画像に共通していることをテキスト」の名前を調整します。
- モジュールのいずれも前のモジュールの隣にすべて目のシンボルをオフにして、完全な分析を実行する彼らの画面を表示しないことを確認します。
注:パイプラインの処理中にこれが大幅に必要な計算資源を減少させます。 - プレスシェーディングモデルの計算を開始するために「画像を分析します」。
- 実行して画像解析
- メニュー「ファイル」に移動し、「インポート」→「ファイルからパイプラインを...」を選択し、の提供しを選択エド・パイプライン」BrucellaEntryWithShadingCorrectionPart2-Ver007」。レガシーパイプラインを変換するかどうかを尋ねたところ、答えは「変換しないでください」。
- 「出力」→「ビューの出力設定」に移動し、結果として得られるスプレッドシート用の画像と入力フォルダと出力フォルダを選択します。
- モジュールでは(1)「LoadImages」、画像の名前と一致するように、「これらの画像に共通していることをテキスト」の名前を調整します。
- モジュール(2)「LoadSingleImage」を、2シェーディングモデルの場所とファイル名を選択することで、6.2.1の下で計算されたシェーディングモデルをロードします。
- モジュール(4) - (5)「ImageMath」パラメータを調整し、顕微鏡カメラのビット深度に一致する」ことにより、第1の画像を掛け」。 8および16ビットの画像は12ビット画像のためのパラメータを「1.0」を、設定するために「16.0」パラメータを設定します。
- モジュール(6)「IdentifyPrimaryObjects "、パラメータ"ローを設定WERは、核のみがセグメント化され、背景は無視されていることを十分に高い「閾値に結合しました。
- モジュール(6)、モジュールの上のステップに良い値を特定し、ステップ6.1.2.6.1及び6.1.2.6.2で説明したように入力画像を表示します。
- 入力画像を右クリックし、ヒストグラムを表示します。
注:ヒストグラムのピークは、一般的な背景を示しています。 - 出力画像に表示されているように核の良好なセグメンテーションが達成されるまで、バックグラウンド強度から出発してパラメータの値を大きくします。
注:背景の上にしきい値を設定すると、空のサイトのために重要です。
- モジュール(8)「IdentifyPrimaryObjects」、「しきい値の下限」のみのブルセラがセグメント化され、背景が無視されていることを十分に高いパラメータを設定します。
- モジュール(8)、モジュールの上のステップに良い値を特定し、ステップ6.1.2.6.1及び6.1.2.6.2で説明したように入力画像を表示します。
- 入力画像を右クリックし、ヒストグラムを表示します。
注:ヒストグラムのピークは、一般的な背景を示しています。 - 出力画像に表示されているようにブルセラの良いセグメンテーションが達成されるまで、バックグラウンド強度から出発してパラメータの値を大きくします。
注:背景の上にしきい値を設定すると、空のサイトのために重要です。
- モジュール(11)「FilterObjects」で、パラメータ「最小値」という背景とアーティファクトが離れてフィルタリングされ、唯一のブルセラコロニーが保持されるように調整します。
- 良い値は、モジュールの上にステップを識別し、ステップ6.1.2.6.1及び6.1.2.6.2で説明したように、出力画像を表示します。 6.2.2.7で選択した値から始めて、唯一のブルセラオブジェクトが保持されるまで、段階的にそれを増加させます。
- テストモードを終了し、前uncheによって完全な分析を実行するモジュールのどれもが自分の画面を表示しないことを確認してくださいモジュールに次のすべての目のシンボルを弄ぶ天使。
注:パイプラインの処理中にこれが大幅に必要な計算資源を減少させます。 - 押して分析を開始するには「画像を分析します」。
- 解析が終了すると、分析がプレート全体によく働いていることを確実にするために、得られたPNG画像だけでなく、CSVシートを検査します。
- シェーディングモデルを計算します
7.感染得点
注:細胞生存率に大きな影響を与えるsiRNAは慎重に考慮しなければならない、これは偽陽性の発見を促進することができるからです。改変された細胞数は、実際のMOIに影響を及ぼし、必須遺伝子の標的化は、病原体感染に多面的効果を有することができます。 siRNAによる不完全な枯渇が必須遺伝子の研究を可能にしますが、そのようなターゲットは宿主因子としての役割を裏付けるための代替方法( 例えば、製薬干渉)によって検証されなければなりません感染の間。
- エンドポイントアッセイ
- ステップ6.1.2.12で生成されたCSVシートを開き、感染細胞(CellProfiler読み出し:「Count_InfectedCells」)の数を割ることによっても感染率あたりの計算のセルの合計数(「CountNuclei」CellProfiler読み出し)によって。ウェルあたり複数のサイトが撮像されている場合は、最初に感染した細胞のあたりウェル数および細胞のあたりウェル総数を構築するために、各ウェルのためのすべてのサイトをまとめています。
- プレートは十分な非ヒットのsiRNAが含まれている場合はプレート間のばらつきを考慮するために、Zスコアリングの正規化を実行します。完全なライブラリーがスクリーニングされる場合、プレートあたりの非ヒットsiRNAの十分な数を前提としています。
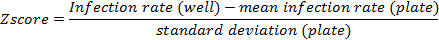
- エントリーアッセイ
- ステップ6.2.2.10で生成されたCSVシートを開き、感染した細胞の数を割ることによっても感染率あたりの計算(CellProfiler読み出し: "Count_InfectedCeLLS」)細胞(CellProfiler読み出しの合計数で: "。CountNuclei」)ウェル当たりの複数の部位が撮影されている場合は、最初に感染した細胞のあたりのウェル数と当たりウェルを構築するために、各ウェルのためのすべてのサイトをまとめます細胞の総数。
- 感染した細胞の細菌負荷を定量化するために、感染細胞あたりの細胞内細菌によって占められる平均面積を推定します。このサイトで感染した細胞の数によって、この目的を達成するために、細胞(「AreaOccupied_AreaOccupied_TrueIntPathogenInCells」CellProfiler読み出し)と重複するすべての細胞内細菌の積分面積を分割します。アーティファクトを説明するために、ウェル読み出し、この読み出しのすべてのサイトの中央値を使用します。
注:このアッセイはブルセラ感染に関与する多数の遺伝子を含むフォローアップアッセイとして使用されている場合、Zスコア正規化を適用しません。その代わりに、プレート間のばらつきを考慮するための基準として模擬井戸を使用して正規化を行います。
結果
図1Aは、自動的に、エンドポイントアッセイで感染細胞を同定するために使用される画像解析の例を示しています。 DAPIで染色されたHeLa細胞の核は、核の周囲の8画素幅の周囲核を同定し、そして25ピクセル核の伸長によってボロノイセル本体を算出しました。細菌は主に核周囲空間に増殖するので、細胞のこの領域におけるGFP強度は、感染と非感染細胞?...
ディスカッション
Bacterial pathogens have evolved numerous strategies to manipulate eukaryotic host cells to their benefit. Pathogens causing acute infections often show rapid proliferation which is accompanied by significant alarming of the immune system and loss of viability of infected cells. In contrast, Brucella and other pathogens that cause chronic infections manage to establish long-lasting interactions within host cells. Therefore, bacteria need to fine tune host cell functions to their benefit without disrupting cellul...
開示事項
The authors declare no conflicts of interest.
謝辞
This work was supported by grants 51RT 0_126008 and 51RTP0_151029 for the Research and Technology Development (RTD) project InfectX and TargetInfectX, respectively, in the frame of SystemsX.ch, the Swiss Initiative for Systems Biology. We acknowledge grant 310030B_149886 from the Swiss National Science Foundation (SNSF). Work of S.H.L and A.C. was supported by the International PhD Program "Fellowships for Excellence" of the Biozentrum. Simone Muntwiler is acknowledged for technical assistance. We would like to thank Dirk Bumann for providing pNF106 and Jean Celli for pJC43 and pJC44.
資料
| Name | Company | Catalog Number | Comments |
| Tryptic Soy Agar (TSA) | BD | 236950 | |
| Tryptic Soy Broth (TSB) | Fluka | 22092 | |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | |
| Skim milk | |||
| 250 ml screw cap bottle | Corning | 8396 | |
| DMEM | Sigma-Aldrich | D5796 | |
| Fetal Calf Serum (FCS) | Gibco | 10270 | Heat-inactivated |
| Trypsin-EDTA (0.5%) | Gibco | 15400-054 | 10x stock solution, dilute 1:10 in PBS |
| Scepte 2.0 Cell Counter | Merck Milipore | PHCC20060 | Alternative cell counting devices can be used |
| Greiner CELLSTAR 384-well plate | Sigma-Aldrich | M2062 | |
| Peelable aluminum foil | Costar | 6570 | |
| Reagent dispenser: "Multidrop 384 Reagent Dispenser" | Thermo Scientific | 5840150 | Alternative reagent dispenser can be used. |
| Transfection reagent: "Lipofectamine RNAiMAX | Invitrogen | 13778-150 | |
| Automated plate washer: "Plate washer ELx50-16" | BioTek | ELX5016 | This plate washer contains a 16-channel manifold suitable for 384-well plates. It fits into a biosafety cabinet and has a lid covering the plate during washing which reduces the risk of aerosol production. Alternative plate washers with similar features could be used. |
| Gentamicin | Sigma-Aldrich | G1397 | |
| Anhydrotetracycline hydrochloride | Sigma-Aldrich | 37919 | 100 μg/ml solution in 100% ethanol is kept at -20 °C protected from light in aluminum-foil |
| PBS | Gibco | 20012 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Dissolve in 0.2 M HEPES buffer, pH 7.4. Store at -20 °C and thaw freshly the day before use. Caution, PFA is toxic by inhalation, in contact with skin and if swallowed. |
| HEPES | Sigma-Aldrich | H3375 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| DAPI | Roche | 10236276001 | |
| Scrambled siRNA | Dharmacon | D-001810-10 | |
| Kif11 siRNA | Dharmacon | L-003317-00 | |
| ARPC3 siRNA | Dharmacon | L-005284-00 | |
| Brucella abortus 2308 | |||
| pJC43 (apHT::GFP) | Celli et al.12 | ||
| pAC042.08 (apht::dsRed, tetO::tetR-GFP) | Construction: pJC44 4 was digested with EcoRI followed by generation of blunt ends with Klenov enzyme and subsequent digestion with SalI. TetR-GFP was amplified from pNF106 using primer prAC090 and prAC092. Following digestion with SalI, the TetR-GFP product was ligated to the digested pJC44 vector. | ||
| Primer prAC090 | Sigma-Aldrich | TTT TTG AAT TCT GGC AAT TCC GAC GTC TAA GAA ACC | |
| Primer prAC092 | Sigma-Aldrich | TTT TTG TCG ACT TTG TCC TAC TCA GGA GAG CGT TC | |
| HeLa CCL-2 | ATCC | CCL-2 | |
| ImageXpress Micro | Molecular Devices | IXM IMAGING MSCOPE | Automated cellular imaging microscope equipped with a precision motorized Z-stage. Alternative systems for automated microscopy and alternative components for hard- and software specified below can be employed. |
| High-Speed Laser Auto-Focus | Molecular Devices | 1-2300-1037 | |
| CFI Super Fluor 10X objective | Nikon | MRF00100 | N.A 0.50, W.D 1.20 mm, DIC Prism: 10X, Spring loaded |
| Photometrics CoolSNAP HQ Monochrome CCD Camera | Molecular Devices | 1-2300-1060 | 1,392 x 1,040 imaging pixels, 6.45 x 6.45 µm pixels, 12 bits digitization |
| MetaXpress software | Molecular Devices | 9500-0100 | |
| LUI-Spectra-X-7 | Lumencor | SPECTRA X V-XXX-YZ | Light engine. The following light sources are used: violet (DAPI), cyan (GFP), green/yellow (RFP) |
| Single Band Exciter for DAPI | Semrock | FF01-377/50-25 | |
| Single Band Emitter for DAPI | Semrock | FF02-447/60-25 | |
| Single Band Dichroic for DAPI | Semrock | FF409-Di03-25x36 | |
| Single Band Exciter for GFP | Semrock | FF02-472/30-25 | |
| Single Band Emitter for GFP | Semrock | FF01-520/35-25 | |
| Single Band Dichroic for GFP | Semrock | FF495-Di03-25x36 | |
| Single Band Exciter for RFP | Semrock | FF01-562/40-25 | |
| Single Band Emitter for RFP | Semrock | FF01-624/40-25 | |
| Single Band Dichroic for RFP | Semrock | FF593-Di03-25x36 |
参考文献
- Pappas, G., Papadimitriou, P., Akritidis, N., Christou, L., Tsianos, E. V. The new global map of human brucellosis. Lancet Infect Dis. 6, 91-99 (2006).
- Atluri, V. L., Xavier, M. N., de Jong, M. F., den Hartigh, A. B., Tsolis, R. E. Interactions of the human pathogenic Brucella species with their hosts. Annu Rev Microbiol. 65, 523-541 (2011).
- Guzman-Verri, C., et al. GTPases of the Rho subfamily are required for Brucella abortus internalization in nonprofessional phagocytes: direct activation of Cdc42. J Biol Chem. 276, 44435-44443 (2001).
- Starr, T., Ng, T. W., Wehrly, T. D., Knodler, L. A., Celli, J. Brucella intracellular replication requires trafficking through the late endosomal/lysosomal compartment. Traffic. 9, 678-694 (2008).
- Boschiroli, M. L., et al. The Brucella suis virB operon is induced intracellularly in macrophages. Proc Natl Acad Sci U S A. 99, 1544-1549 (2002).
- Celli, J., et al. Brucella evades macrophage killing via VirB-dependent sustained interactions with the endoplasmic reticulum. J Exp Med. 198, 545-556 (2003).
- Porte, F., Liautard, J. P., Kohler, S. Early acidification of phagosomes containing Brucella suis is essential for intracellular survival in murine macrophages. Infect Immun. 67, 4041-4047 (1999).
- Anderson, T. D., Cheville, N. F. Ultrastructural morphometric analysis of Brucella abortus-infected trophoblasts in experimental placentitis. Bacterial replication occurs in rough endoplasmic reticulum. Am J Pathol. 124, 226-237 (1986).
- Qin, Q. M., et al. RNAi screen of endoplasmic reticulum-associated host factors reveals a role for IRE1alpha in supporting Brucella replication. PLoS Pathog. 4, e1000110 (2008).
- Ramo, P., et al. Simultaneous analysis of large-scale RNAi screens for pathogen entry. BMC genomics. 15, 1162 (2014).
- Kuhbacher, A., Gouin, E., Cossart, P., Pizarro-Cerda, J. Imaging InlC secretion to investigate cellular infection by the bacterial pathogen Listeria monocytogenes. J Vis Exp. , e51043 (2013).
- Kentner, D., et al. Shigella reroutes host cell central metabolism to obtain high-flux nutrient supply for vigorous intracellular growth. Proc Natl Acad Sci U S A. 111, 9929-9934 (2014).
- Celli, J., Salcedo, S. P., Gorvel, J. P. Brucella coopts the small GTPase Sar1 for intracellular replication. Proc Natl Acad Sci U S A. 102, 1673-1678 (2005).
- von Bargen, K., Gorvel, J. P., Salcedo, S. P. Internal affairs: investigating the Brucella intracellular lifestyle. FEMS microbiology reviews. 36, 533-562 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27, 1179-1180 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved