Method Article
点突然変異修復 CRISPR/Cas9 とひと細胞での SsODN によって触媒の機能として敷地内変異原性を調べるための標準方法論
要約
このプロトコルは哺乳類細胞における点突然変異の修復システムを編集 CRISPR/Cas9 ベースの遺伝子のワークフローについて説明します。ここで、ターゲット ・ サイトで塩基形成測定の詳細な後続実験的戦略と遺伝子編集へのコンビナトリアル アプローチを使用-本質的に、オンサイトの突然変異を分析します。
要約
組合せ遺伝子編集 CRISPR/Cas9 と一本鎖のオリゴヌクレオチドを使用して単一ベース点突然変異は、ヒトの遺伝性疾患のさまざまな担当が多いの補正のための効果的な戦略です。老舗のセルベースのモデル システムを使用して、HCT116 細胞に統合されたシングル コピー変異 eGFP 遺伝子の点突然変異はこの組合せのアプローチを使用して修復されました。修正と裸眼の細胞を分析オクターリピート ターゲット サイトを囲む DNA シーケンスで裸眼の細胞で作成するとき遺伝子編集の精度と遺伝的病変の開発を明らかにします。ここでは、ターゲット ・ サイトで塩基形成を測定する詳細な実験的戦略と相まって、点突然変異の遺伝子編集するこの組合せアプローチを分析するために使用する特定の方法論を説明します。このプロトコルには、基礎的なアプローチおよび人間療法編集 CRISPR/Cas9 ベース遺伝子の開発を目指した調査のワークフローについて説明します。この仕事の結論はその敷地内の変異は点突然変異修復のプロセス中に CRISPR/Cas9 活動の結果として起こる。この作品は、臨床実施に向けどのようなアプローチの重要な側面をする必要があります突然変異の程度を識別する標準化された方法の場所に置きます。
概要
Mandecki (1986 年) による研究の先駆的な細菌1、ワルダーながら変身オリゴヌクレオチドとワルダー (1986) 酵母2で似たような研究を実施を使用してプラスミッド DNA の永続的な変更を示した。その後まもなく、シャーマンと同僚は、一本鎖のオリゴヌクレオチドが遺伝子3の継承を変更する酵母細胞に導入された論文のシリーズを公開しました。建物は、微生物細胞の精液の仕事、Kmiec と同僚は、哺乳類細胞4シングル基本修理を監督独自の単剤オリゴヌクレオチドを開発し始めた。この概念は、軸受 RNA 分子が DNA 塩基で構成されるよりターゲット サイトにしっかりとバインドことを示した生化学的データに基づいていた。キメラ オリゴヌクレオチド5,6,7を使用して遺伝子編集成功の多数の報告があるものの、レベルを編集残った非常に可変実験間および異なるターゲット/セル組み合わせ。
8ゲノム内のサイトにランダムに統合する可能性は低いですので、ドナー二本鎖 DNA にドナーの単一座礁させた DNA が勧めします。しかし、外因導入一本鎖オリゴヌクレオチドは細胞の核酸分解酵素によって急速に劣化しやすいです。オリゴヌクレオチドのテルミニを劣化から守るためにいくつかの方法が採用されています。任意のシーケンスを含むエキソヌクレアーゼ保護、一本鎖 DNA が 0.1 - 1 編集効率を得るのに十分だった % 範囲6。改善し、表現型リードアウトと相まってより一貫性のあるトランスフェクション技術をより一貫性のある複数の研究グループ8,9,10、11、によって編集の許可 12,13。
過去 10 年間で、標的細胞に遺伝子を編集するのに向いているように多大な努力がずっとあります。1 つの戦略では、細胞周期14,,1516変調方式を採用しています。培養細胞にオリゴヌクレオチド 0.1% と 1% の増加の 3-5 倍時の編集遺伝子の頻度間マウス導入一本鎖オリゴヌクレオチド (ssODNs) で正常な反応条件下で周波数を編集中S 段階を移行する際に細胞に導入されました。別の一連の実験の Brachman と Kmiec (2005 年)、編集 (3-5%) 正確な遺伝子の比較的高レベルが 2'3 ' dideoxycytosine (ddC) されたオリゴヌクレオチド17 添加前に 24 h のための細胞培養と得られたことの実証で.ddC は、遺伝子の可能性がある細胞の複製に ssODNs によって編集を含む ODNs のアクティブなレプリケーション11,18の地域への取り込みを示唆、DNA 複製フォークの動きの速度を低減します。一緒に取られて、ドナー DNA が遺伝子の編集、操作の真の機構の一部として成長している複製フォークに組み込まれている概念に導かれたこれらの研究二本鎖切断が存在するかどうかまたはない19の独立しました。したがって、点突然変異の補正や染色体内の DNA のセグメントの置き換えに DNA のペアリングと一本鎖同化経路を介して発生します。
成長している小さな分子エージェントと細胞のランダムな二本鎖 DNA 切断を誘発するセルがダメージ20,21を修復しようと DNA 複製イベントが停止している環境を作成します。複製フォークのこの一時的な減速によりターゲット ユーザー補助15を向上させる単一座礁させた編集オリゴヌクレオチド、クロマチン構造のより効率的な浸透します。VP16、ブレオマイシンなどの薬では二本鎖 DNA の作成は中断またはカンプトテシン2223,,は、遺伝子レベルの約 10 倍、編集を刺激するために示されている 6-8% まで。ただし、ランダムな二本鎖 DNA 切断は治療上の設定では望ましくありません。
遺伝子プログラマブル核酸オリゴヌクレオチドと編集は細胞分裂24,25.中の刺激もリベラ トーレスと同僚が活動をターゲットの同じ増加を示した同期 HCT 116 細胞における相同性監督修理を遂行する核酸ベースの配布を使用するとときに、転写活性化因子のようなエフェクター核酸分解酵素 (TALENs)複製セル人口の26,27に導入両方一本鎖オリゴヌクレオチドと用いられました。最近では、Bialk および同僚示した一本鎖オリゴヌクレオチドと単一基本変異の修復との配列が定期的に空間の短い回文繰り返し Cas9 をクラスター化する (CRISPR/Cas9) 分子起こる高い治療効果といつセル人口は S 相28を走査します。CRISPR 分子ターゲットの胸の谷間で指定された DNA シーケンス内の領域を識別するために RNA と機能で構成されます。Cas9 は、その機能は、修飾アミノ酸交換反応で二本鎖 DNA を切断する酵素です。したがって、CRISPR ゲノムのターゲット上の複合体の位置、Cas9 ヌクレアーゼは、二本鎖切断を実行します。林ら(2014 年) はまた細胞周期遺伝子の高頻度を達成するための重要性を示した編集、変更された CRISPR/Cas9 ribonulceoprotien (RNP) 複合体を介した配信システムを使用してプライマリ新生児線維芽細胞、ヒトの胚性幹細胞と他のセルの行では、24が。したがって、遺伝子編集と単剤遺伝子を編集するための細胞周期の進行の間に確立された関係は組合せ遺伝子プログラマブル核酸とドナー DNA テンプレートを使用して、編集に適用されます。遺伝子編集へのコンビナトリアル アプローチは、さまざまなドナーの DNA のテンプレートを持つパートナーをまとめている、フィールドのほとんどの労働者は二本鎖切断の機能を提供するために CRISPR/Cas9 を使用します。この選択は、使用の容易さのこの特定の遺伝学的ツールと柔軟性を利用できます遺伝子の機能を無効にするまたは特定のサイトに DNA の外国作品を紹介する時に前提です。ノックアウトの世代は技術的に、遺伝子の交換、遺伝子のコピーを「正しい」または通常の病気サイトへの取り込みに精度と行う必要が比較されるより容易。研究者の数を識別する、特定の薬と突然変異の基盤の補正と高周波数29で適切な位置に通常遺伝シーケンスの挿入を有効にする試薬の使用を勉強します。
最近では、リベラ トーレスら30使用組合せ遺伝子編集、専用 CRISPR/Cas9 システムと修復する一本鎖オリゴヌクレオチド ドナーの DNA テンプレートによって提供される遺伝情報の二本鎖切断活性を利用して、点突然変異で、強化された緑色蛍光タンパク質 (eGFP) 遺伝子の単一コピーは HCT116 細胞に統合。著者は、ターゲット ・ サイトを取り巻く胸の谷間の特異性を評価するこの特徴モデルのセルラインを利用しました。データでは、特に修正の点突然変異が含まれていないセルにターゲット ・ サイトでの不均一性が存在することを明らかにします。本稿では、我々 は詳細し、焦点を当てる CRISPR/Cas9 遺伝子の編集によって作成された敷地内変異の不均一性を調べるこれらの労働者によって使用される方法に応じて。
プロトコル
次のプロトコルは、哺乳動物細胞での作業; 滅菌法/細胞培養の知識を期待します
。1 細胞と培養条件
- HCT 116 細胞の培養のための培地を作る 500 mL: マッコイ ' s 5 a 変更 10% 牛胎児血清 (FBS)、2 mM L-グルタミン (これは完全な 1% ペニシリンと培。媒体).
注: は、めっき前に T-75 または T-175 フラスコの 116 19 セル HCT を育てます。90% 合流各 T-75 フラスコ 10 6 セル、大まかに 5 つの 10 cm プレート x 8.4 になります、各 T-175 10 6 セル、約 15 の 10 cm プレート x 18.4 になります 。
2。フラスコからセルを収穫
- 媒体を離れて吸い出しなさい、ダルベッコで洗う ' s Phosphate-Buffered 生理食塩水吸引と magniesium (PBS) (10 mL T-75 または T-175 25 mL) のカルシウムなし 。
- 追加トリプシン 2 mL ピペット (2 mL T-175 または T-75 1 mL) を使用してフラスコに滴下し。セルをデタッチするように 5 分間の 37 ° C および 5% の CO 2 でインキュベーターでフラスコを配置します 。
- のすべてのセルが外れているし、フラスコ (8 mL T-175 または 4 mL T-75) の表面全体に分散させることによって完全培地をいやすようにフラスコをタップします 。
- 上下の細胞塊を破るし、15 mL の円錐管にセルを転送するために複数回ピペット
- セルを回転する前にダウン、15 mL の円錐形から 10 μ L を取るし、トリパン ブルーのセルをカウントする 10 μ L とそれを組み合わせます。125 g x と 16 で 5 分間回してセルをペレット ° C
3。セルをカウント
- セルの転送 10 μ L 混合トリパン ブルー、検定します。(それぞれのグリッドは、16 の正方形を含んでいる) 外側の周り 4 グリッドをカウントします。
- 16 角の正方形の各セットの平均細胞数を取ます 。
- 10,000 を掛けます (10 4).
- トリパン ブルー色素添加から希釈を補正するための細胞を採取するために使用中の容量を乗算します
。 注: 次の細胞を再懸濁しますにボリュームを計算するための方程式の形式:
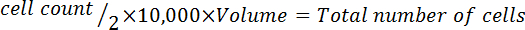
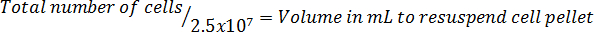
4。めっきセル
- 同期する細胞の各 10 cm プレート、5 mL の完全培と 6 μ M の aphidicholin (200-証拠エタノールで 2.5 mM の在庫の 12 μ L) を追加します 。
- 転送 100 μ L 再懸濁細胞のペレット、2.5 x 10 の 6 セルの各 10 cm プレートとミックスにゆっくり旋回します 。
- 16-24 h G1/S のボーダーでセルを同期するための 37 ° C および 5% の CO 2 で板をインキュベートします 。
5。アフィディコリン同期から細胞
- 、
- ターゲット、前に 4 h 培地を吸引、PBS で洗浄、吸引、PBS、完全培地 5 mL を追加
- 配置 37 ° C で 4 時間 5% CO 2 インキュベーターに
6。RNA 錯
- 張研究室に変異 eGFP 遺伝子の塩基配列を入力してください ' s オンライン発電機 25 (http:// crispr.mit.edu/)、ターゲット サイトに近接してバインド シーケンス CRISPR ガイドを選択。商業ソースから CRISPR ガイドのシーケンスを取得します 。
- は、CRISPR RNA (crRNA)、トランス活性化 crRNA (tracrRNA)、および 20 の ° C の Cas9 蛋白質しメーカーの提案に従って利用します。
- 45 μ m 追加 6.75 μ L に等モル濃度の crRNA の 200 μ M 在庫と 1.5 mL 遠心分離機に tracrRNA の 200 μ M 在庫の 6.75 μ L の RNA ミックス チューブ。30 μ L の最終巻に TE バッファーの 16.50 μ L を追加します 。
- 熱ブロックまたは PCR 機械で 5 分 95 ° C で熱
。 注意: ホット! - 室温に冷却することができます
。 注: PCR 機械を使用している場合 0.2 の ° C の冷却を設定/s の - は、各サンプルの次の手順を実行します。
- CrRNA:tracrRNA 5 μ L の最終巻に TE バッファー (10 mM トリス、pH 8.0、0.1 ミリメートルの EDTA; pH 8.0) 2.78 μ で複雑な希釈 2.22 μ 。
- Cas9 蛋白質の低血清培地の 3.33 μ L で 60 μ M の在庫からの希釈 1.67 の μ L 5 μ L の最終巻にします 。
- Cas9 蛋白質複合体の RNA の 5 μ L のミックス 5 μ L
7。ターゲットのセルを収穫
- 吸引中、PBS、5 mL で洗い、PBS を吸引し、各 10 cm プレートに予め温めておいたトリプシンの 1 つの mL を追加。5 分の 37 ° C、5% CO 2 でインキュベーターでプレートを入れて
- すべてのセルは外れているし、プレートの表面全体に分散させることによって 4 ml の完全培地の癒すように 10 cm プレートをタップします 。
- 細胞塊を破るし、15 mL の円錐管にセルを転送する複数回上下ピペット 。
- セルを回転する前にダウン、15 mL の円錐管から 10 μ L を取るし、トリパン ブルーのセルをカウントする 10 μ L とそれを組み合わせます。125 x g と部屋の温度で 5 分間回してセルをペレットします 。
- 培地を吸引し、5 mL の PBS で洗浄します。室温で 5 分間 125 x g で回してセルをペレットします 。
8。セルをカウント
- セルの転送 10 μ L 混合トリパン ブルー、検定します。(それぞれのグリッドは、16 の正方形を含んでいる) 外側の周り 4 グリッドをカウントします。
- 16 角の正方形の各セットの平均細胞数を取ます 。
- 10,000 を掛けます (10 4).
- トリパン ブルー色素添加から希釈を補正するための細胞を採取するために使用中の容量を乗算します
。 注: 次は、細胞を再懸濁しますにボリュームを計算するための方程式の形式です。マッコイの無血清細胞の必要な数を再停止 ' s 5 a 改変中です
。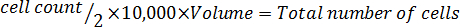
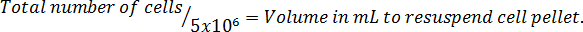
9 サンプルをターゲット
- 100 を転送。細胞懸濁液 (5 x 10 の 5 セル) 8.1 のステップから 4 mm のギャップにある各エレクトロポレーションのキュベットに μ。5 x 10 の 5 セル密度のセルの手順 6.7 に 100 μ L から複雑な RNP の 10 μ L を追加します。ODN を追加 (2 μ M) 各サンプルにします
。 注: 肯定的な制御のエレクトロポレーション マシンにラックを取る eGFP 発現プラスミド- 1 μ g/μ L での 1 μ L を追加, 軽くフリック、各サンプルと商工会議所にそれらを置きます。Electroporate 250 V、LV。2 パルス、1 s;13 ms;単極性パルスします 。
- ラックをフードに転送します。含む 2 mL の完全培地の各サンプルを転送私n 6 ウェル プレート。補正レベルをチェックする前に 72 時間のための 37 ° C および 5% の CO 2 をインキュベートします 。
10。遺伝子編集細胞のトランスフェクション効率分析
- 培地を吸引し、2 ml の PBS のセルを洗浄しています。PBS を吸引し、6 ウェル プレートの各ウェルに予め温めておいたトリプシンの 500 μ L を追加します。5 分の 37 ° C、5% CO 2 でインキュベーターでプレートを入れて
- すべてのセルは外れているし、井戸の表面全体に分散することで 1 ml の完全培地の癒すようにプレートをタップします 。
- 1.5 mL 遠心管、室温で 5 分間 5,000 × g でペレットに細胞を通過します 。
- は、培地を吸引します。FACS バッファー (0.5 %bsa、2 ミリメートルの EDTA、PBS で 2 μ G/ml propidium ヨウ化) を 500 μ l 添加の細胞ペレットを再停止します 。
- は、フローサイトメトリーによる細胞蛍光 (eGFP +) を測定します。
- リベラ トーレス ら 30 の説明に従って各サンプルの生きているセルの合計数以上合計ライブ eGFP 陽性細胞の割合として補正効率を計算します 。
11。DNA シーケンス解析
- Electroporate 同期されリリースされた HCT 116-19 5 x 10 の 5 の濃度で細胞 細胞/100 μ L, 100 の pmols と 72NT で複雑な RNP 2.0 μ M で ODN 。
- 6 ウェル プレートにセルを転送し 72 h の回復
- を並べ替えるセル個別に +/-、eGFP の 488 nm (100 mw) レーザーを用いた FACS ソーターを使用して 96 ウェル プレートにリベラ トーレス ら 30 で説明したようします
。 注: しないすべての井戸は成長する正常にします 。
- セル以上 6 週間を展開し、収穫の手順 7 で説明した 。
- 市販 DNA の隔離を使用して携帯電話の gDNA を切り分け、成長している井戸からキット (材料の表 を参照してください)、PCR によって基本のターゲットを囲む領域を増幅 (718 bp; 前方プライマー 5 '-ATGGTGAGCAAGGGCGAGGA-3 ' 逆プライマー 5 と ' - ACTTGTACAGCTCGTCCATGC - 3 ').
- サンプルを実行 DNA シーケンス解析します 。
結果
我々 は哺乳類セルの遺伝子編集 HCT116 細胞の単一コピーとして統合された eGFP 遺伝子内に埋め込まれた点突然変異の補正に依存する勉強するのにモデル システムを使用しました。これは単一コピーの遺伝子であることに注意することが重要です。したがって、DNA 変化または突然変異のより複雑なビューを作成できます。図 1 aには、対象となるベース、終止コドン赤で強調表示されているタグの三塁変異 eGFP 遺伝子の塩基配列が表示されます。部分的に eGFP 遺伝子 (72NT) の非転写鎖に相補的な G から C への基盤交換を誘発するように設計、72 ベース オリゴヌクレオチドことも示しています。さらに、図 1 aに 2 C で、5' から 3' 方向、示された protospacer シーケンスとして指定の CRISPR に描かれています。この遺伝子編集の反作用を遂行する Cas9 遺伝子から成る哺乳類発現ベクターを使用する代わりに CRISPR (cr) RNA および浄化された Cas9 蛋白質 (図 1 b) に結合された tracr (tr) RNA から成るリボ核タンパク質 (RNP) を使用し、適切な特定ガイド RNA シーケンス。
特別に設計された RNP 粒子 HCT116 細胞へ遺伝子編集、電気穿孔法による一本鎖オリゴヌクレオチド配信時 eGFP の単一の基本突然変異の修復によって立証される、フローを使用して培養 72 時間後を観察cytometer。機能的な修復は、この蛍光のため並べ替えによって全体の人口からも分離できるターゲット細胞の蛍光グリーンの出現によって観測可能なオブジェクトです。図 2に示すとおり、RNP と 72NT の増加の協調レベルと段階的な用量反応がみれます。分子の比率、RNP の picomoles、マイクロモル濃度の図に表示されている 72NT Cas9 と特定ガイド RNA transfected 式からの導入に依存する遺伝子編集の反作用で使用された最適な投与量に基づいていますベクトル。図 2にはめ込み RNP 粒子の不在で遂行遺伝子編集の反応が表示されます。ここでは、単剤遺伝子編集反応で 72NT のオリゴヌクレオチドの 10 倍の高濃度を使用する場合、標的細胞の約 1% が修正されます。
ターゲット サイト、いわゆる敷地内の突然変異誘発効果で遺伝的異質性が存在するかを決定するために-遺伝子個々 の細胞の活動を編集の結果を確認することにしました。しばらくの間、本来遺伝的足跡や病変の全体的な人口を調べることを確認拡大クローン細胞のゲノムを調べるよりもはるかに困難です。図 2、RNP 複合の 100 pmol と 72NT のオリゴヌクレオチドの 2.0 µmol を使用してこの時間を示した実験を繰り返しました。として、上記 HCT 116 細胞 aphidicholine と 24 h の同期, G1/S の国境で逮捕された.4 h 後、セルがリリースされた、遺伝子編集ツールは、電気穿孔法によって導入されました。72 h facs や 96 ウェルのプレート (実験のプロセスを図 3に示します) に個別に並べ替えられます後、セルを行った。緑の蛍光性を表示するセルは、クローン性増殖の 96 ウェル プレートの個々 の井戸にフローサイトメトリーによる分類されました。重要なは、eGFP 発現に欠けているセルがまた分離・同じ条件下での拡張のための同様の方法で並べ替えられます。
成長の 14 日後個々 のクローンのほとんどは DNA の隔離を有効にするのに十分に拡大していた。など、eGFP 陽性検体の 16 個のクローンを選択した、ターゲット サイトを取り巻く遺伝的整合性が DNA の配列によって分析しました。周囲の人口内の対立遺伝子の DNA 配列情報は、サンガー シーケンス、野生型対立遺伝子 (図 4 a) のシーケンスを比較するシーケンスの可視化ソフトウェアを使用して組み立てを使用して生成されました。RNP 複合体のカットのサイトは、バーの (2 C crRNA) 緑の上にある黒い矢印で示されます。図 4 aに示すように、すべて 16 eGFP 陽性細胞はターゲット ・ サイトで予測されたヌクレオチド交換を含めます。変換後の C 残基が赤で強調表示されます、その正確な変更を反映してピークのプロフィールは eGFP 陽性シーケンスの下に提供されます。同様に、15 非緑クローン分離、十分に DNA の抽出を可能にする拡大し、シーケンスをターゲット ・ サイトでの不均一性の行った。予想通り、サンプルの約半分で、DNA の基盤交換認められなかった。これは、図 4 bに示すように、ターゲット ・ サイトで G 残基の保守に反映されます。これらの実験で検討したクローン展開の残りの部分には、蛍光グリーンの不足のためしたがって会計欠失変異体の異種集団が表示されます。削除サイズ 19 拠点に 1 つのベースからであった。EGFP 陽性細胞の 15 サンプルを行っただけ、その他の種類またはオクターリピートのフォーム可能性がある CRISPR/Cas9 活動が残した遺伝的病変のタイプのかなりの代表であると考えています中に、注意することが重要です。対象となる人口に存在する可能性があります。
一緒に取られて、図 4に表示される結果はターゲット システム eGFP で表現型読み出しを確認してください。C ヌクレオチドに G の変換により、修正 HCT116 細胞に蛍光グリーンの出現です。この実験のため分離されたクローンまたは以前実験27,28ターゲット サイトを取り巻く塩基置換は観察されていません。データはまた細胞遺伝子の点突然変異修復を介して編集を経るに失敗したまま裸眼がない不変のターゲット サイトを取り巻く遺伝的異質性の範囲で、いくつかのケースでことを示します。
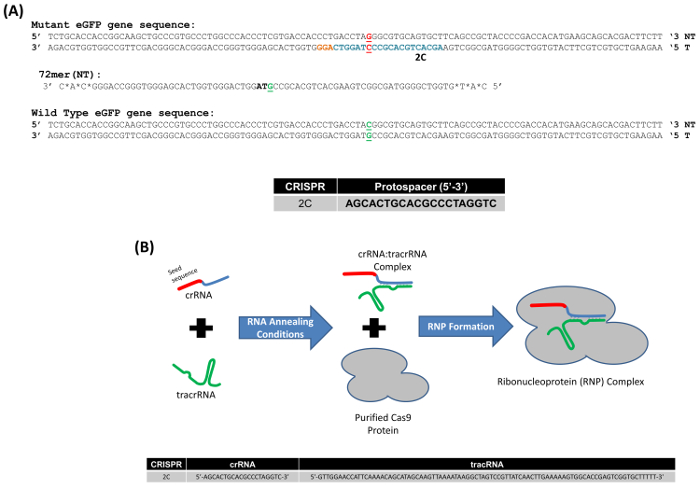
図 1。(変異 eGFP 遺伝子の遺伝子編集 A) モデル システム。野生型と変異 eGFP 遺伝子のシーケンスの中心部にあるターゲットのコドンの適切なセグメントは、それぞれ緑と赤で表示されます。Exchange 向けヌクレオチドは太字、下線します。2 C CRISPR protospacer シーケンス表現する青い強調された拠点し、オレンジの拠点は PAM サイトをハイライトします。これらの実験で使用されるオリゴヌクレオチドは 3 つのターミナル ベースで変更ホスホロチオエート結合を軸受長さ 72 拠点です。72 mer 対象非転写 (NT) 鎖 (72NT) とします。(B) CRISPR/Cas9リボ核蛋白質のアセンブリの反応。crRNA は、tracrRNA (緑) 2 C protospacer シーケンスおよび相互作用ドメイン (青) に対応するターゲット特異性 (20 拠点、赤いセクション) を提供します。crRNA と tracrRNA は、モル濃度でアニールされます。RNP 組み立て完了する Cas9 蛋白質 (グレー) が追加されます。ガイド Rna (gRNAs) は直接、Cas9 エンドヌクレアーゼをアクティブ化し、ターゲット DNA を切断します。2 C 種シーケンスと tracrRNA シーケンス図の下のセクションを示しています。(名)ら、リベラ トーレスからこの図が変更されました。(2017).この図の拡大版を表示するのにはここをクリックしてください。
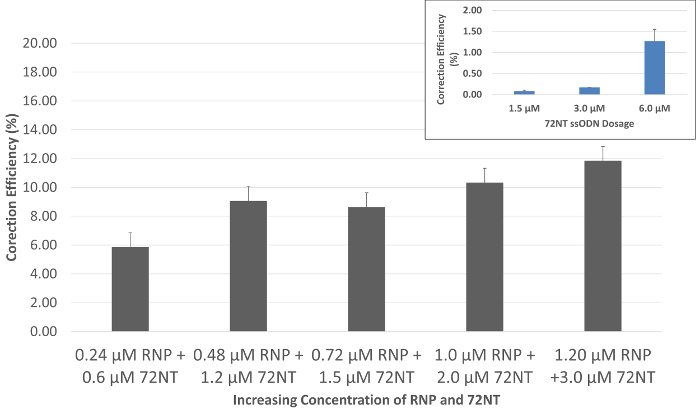
図 2。編集遺伝子は用量依存、RNP と、ssODN によって指示される。同期し、リリース HCT 116 19 セル CRISPR/Cas9 RNP の 24-120 pmol と 0.6 3.0 electroporated をした 72mer の μ M。72 時間の回復期間後遺伝子編集活動は、流れの cytometer を用いて測定しました。遺伝子編集は eGFP 陽性細胞人口中の生菌数の合計数で割った値の数によって決定されます補正効率 (%) として表示されます。エラーバーは標準エラーの基本的な計算を使用して 3 つの独立した実験で生成されたデータ ポイントの 3 つのセットから生成されます。嵌め込む式: 単剤遺伝子編集。同一条件の下で複雑な RNP の不在で一本鎖オリゴヌクレオチド (72NT) 監督遺伝子編集活動は増加する集中の機能として提示されます。(名)ら、リベラ トーレスからこの図が変更されました。(2017).この図の拡大版を表示するのにはここをクリックしてください。

図 3。単一細胞のクローンを分離するための実験的戦略。出展 eGFP 発現細胞として肯定的なクローン性増殖の個々 の井戸に単一セルとして流れの cytometer を使用して並べ替えられスコア化しました。EGFP 発現に欠けているセルが分離されたと同様の方法で並べ替えられ、同じ条件の下で拡大しました。当時の DNA 分離、eGFP 遺伝子を増幅され、サンガーを受けるターゲット サイトを取り巻く遺伝子編集活動を分析するためのシーケンス。(名)ら、リベラ トーレスからこの図が変更されました。(2017).この図の拡大版を表示するのにはここをクリックしてください。
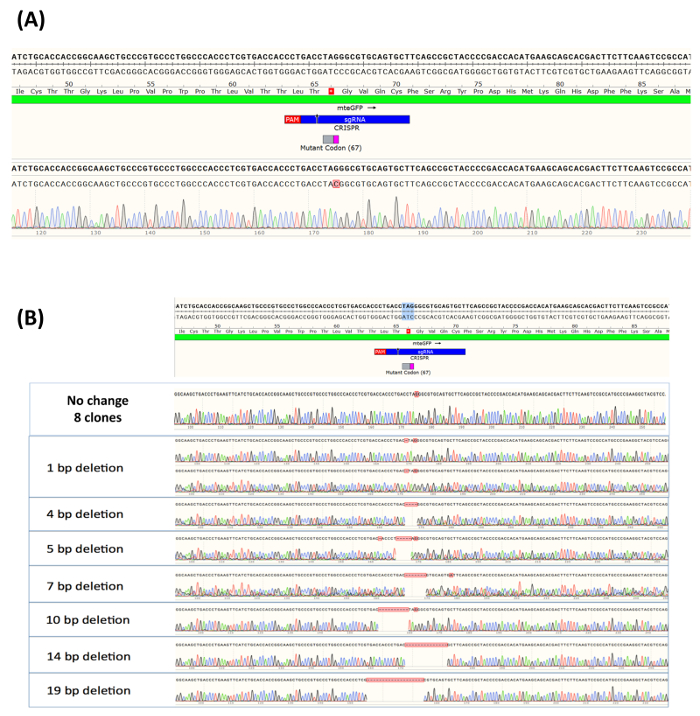
図 4。(EGFP 陽性細胞の A) 対立遺伝子解析のクローン個体として拡張します。クローン分離し、拡張 eGFP 陽性検体 (16 クローン) それぞれから対象となるベースと DNA を取り巻くサイトで分析、収穫、精製、増幅、シーケンスします。対立の分析は、サンガー シーケンス、シーケンスの可視化ソフトウェアを使用して組み立て、図の上部に示されている野生型対立遺伝子のシーケンスと比較しての使用を行った。RNP 複合体のカットのサイトは、バーの (2 C crRNA) 緑の上にある小さな黒い矢印として示されます。(B)EGFP 陰性細胞の対立遺伝子解析のクローン個体として拡張します。裸眼の人口に由来するクローンから拡大した 15 の個々 のサンプルがランダムに選択し、ターゲットのヌクレオチドを取り巻く現場塩基形成を分析します。として、上記対立遺伝子分析を行ったサンガー シーケンスとシーケンスの可視化ソフトウェアを使用して組み立てを使用しています。もう一度、RNP のカットのサイトに合わせて、図の上部に野生型対立遺伝子のシーケンスが表示されます。(名)ら、リベラ トーレスからこの図が変更されました。(2017).この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
遺伝子の編集は、CRISPR/Cas9 システムの出現のために主に主流の科学訓練として浮上しています。細菌細胞における養子免疫の促進、この顕著な経路は、ひと染色体のゲノム変化を可能にする分子ツールとして用途が変更されています。CRISPR/Cas9 の自然な機能は、断片化30,31につながる二重鎖 DNA 切断を導入することによってウイルスの DNA を無効にすることです。この活動は、外因性 DNA を侵略の破壊の結果し、同じウイルスの粒子によってその後の感染を抑制する原核生物の免疫力に 。驚いたことに、このシステムの現象が肺、それ以前の感染イベントによって作成された特定の CRISPR gRNA を再アクティブ化できます。
効率と触媒 CRISPR/Cas9 による遺伝子破壊の精度は、目的が機能している遺伝子32,33を無効にするのには多くの真核生物の遺伝的システムで使用されています。その基底のレベルに減少したとき、プロセスは 2 つの基本的な手順ので構成されます。最初は二本鎖切断、2 番目は非相同末端結合 (NHEJ) ノックアウトを完了するための固有のプロセスに依存しています。ほとんどの場合、鈍終了の作成には二本鎖切断の結果上の空の染色体セグメントと NHEJ に関与する酵素染色体の壊れ目に反応し、壊れた端を結合する行為。このプロセスは時々 切断サイトで個々 のヌクレオチドの損失を含むことができます。さらにいくつかの基地の損失は、フレーム シフト変異を引き起こすことができます、機能発現が破棄されます。CRISPR/Cas9 NHEJ の自然の過程で遺伝子のノックアウトを作成するの使用をもたらしました真核生物の遺伝学;ターゲット ・ サイトで DNA の損失が予測可能と予想されます。
遺伝子ノックアウトとは異なり、研究者数はリダイレクトし、ホモロジー監督修復のプロセスに向かって CRISPR/Cas9 活動を転用しようまいりました。研究の目的は、遺伝子の訂正です。この戦略に CRISPR/Cas9 は、先天異常を修復する、または健全な遺伝子に突然変異を挿入する遺伝情報を提供するドナーの DNA のテンプレートと結合されます。相同性監督の修理を触媒に CRISPR/Cas9 の驚くべき能力の強調表示の初期の報告は、高精度なファッション24,34でプロセスが発生したこと (直接または間接的に) に示されます。当研究室は相同性監督の修理または遺伝子補正機構と調節回路15,17,18 周囲を定義する試みで一本鎖オリゴヌクレオチドを使用して勉強されています。 ,23。これは最も基本的な遺伝的変異の多くの遺伝性疾患を担当する知られているので、単一の点突然変異を編集遺伝子に主に注力します。遺伝子編集の基礎知識はオクターリピートの形成に遺伝子ノックアウト結果に CRISPR/Cas9 関数以来、これらの反応に CRISPR/Cas9 活動の精度を質問に私たちを率いてください。明らかに還元主義的方法で敷地内変異を研究する非選択可能まだ臨床的に関連する遺伝子ではなく、明確に定義されたモデルのシステムを使いました。遺伝子型と表現型のレベルでどの遺伝子に補正を測定できる簡易ターゲット遺伝子を使用して、ターゲット ・ サイトで発生する不均一性を信頼性と堅牢なファッションで識別できることを推論しました。
我々 のデータは、編集システム確立された遺伝子、変異 eGFP から成る遺伝子 HCT116 細胞に統合できる遺伝的病変と敷地内の突然変異のプロセスの生成についての基本情報を確認します。タンデムで働く CRISPR/Cas9 と一本鎖オリゴヌクレオチド ドナー DNA 分子は eGFP 遺伝子の点変異の正確な修復に します。ドナー DNA が突然変異体の基本は、我々 が正確な30と呼ばれるプロセスの修理のためのレプリケーション テンプレートとして機能する分子経路ポイント突然変異の修復のための新しいモデルを提案する.対象となる人口は、修正された表現型を出展しない細胞異種を含むと広く DNA オクターリピート周囲のターゲット ・ サイトに至るまでのさまざまなが表示されます。約半分のクローンは、裸眼の人口から分離された、ターゲット ・ サイトで削除の突然変異が観察されました。一本鎖オリゴヌクレオチドとして単剤遺伝子編集ツールは、ターゲット ・ サイトでオクターリピートを引き起こすことができる、我々 は、その CRISPR/Cas9 アクティビティを締結前のレポートを示していますないのでこれらの突然変異の役割です。
本稿では、ターゲット ・ サイトで遺伝的多様性は、信頼性と堅牢なファッションで測ることができるように詳細な方法論を提供します。注目の膨大な量は、分析に支払われているし、オフサイトの突然変異のマッピング、それはターゲット ・ サイトで作成された異種の突然変異は遺伝子の臨床分野での編集の成否に大きな影響を持つことがあります。その他のテクノロジまたは変更された Cas9 タンパク質先天性哺乳類セル35の相同性向け修理の精度を向上させる必要があります。これらの技術の架け橋、染色体の端を一緒に持って、NHEJ の破壊的な操作を回避する補助オリゴヌクレオチドの使用が含まれます。遺伝子編集活動の結果として、ターゲット ・ サイトでの不均一性の程度を定義することは、治療的介入に設計された任意のプロトコルの重要な部分をする必要があります。
開示事項
開示することは何も著者。
謝辞
著者の謝辞があります。
資料
| Name | Company | Catalog Number | Comments |
| McCoy’s 5A Modified medium | ATCC | 30-2007 | |
| Fetal Bovine Serum (FBS) | ATCC | 30-2020 | |
| L-glutamine | ATCC | 30-2214 | |
| Penicillin-Streptomycin Solution | ATCC | 30-2300 | |
| Dulbecco's Phosphate Buffered Saline | ATCC | 30-2200 | No Calcium or magnesium |
| Trypsin EDTA Solution | ATCC | 30-2101 | |
| Aphidicolin 1mg | Thermo Fisher | AC611970010 | |
| Alt-R CRISPR crRNA, 10 nmol | IDT | No catalog number since its made to your specific gene target. | |
| CRISPR-Cas9 tracrRNA, 20 nmol | IDT | 1072533 | |
| S.p. Cas9 Nuclease 3NLS, 500 µg | IDT | 1074182 | |
| IDTE Buffer | IDT | 11-01-03-01 | |
| Zeus Electroporation Cuvettes 0.4 Cm | VWR | 10497-474 | |
| Gene Pulser Xcell Electroporation Systems | BioRad | 1652660 | |
| Bovine serum albumin lyophilized powder, crystallized, ≥98.0% (GE) | Sigma | 05470-1G | |
| EDTA (0.5 M), pH 8.0 | ThermoFisher Scientific | AM9260G | |
| Propidium Iodide - 1.0 mg/mL Solution in Water | ThermoFisher Scientific | P3566 | |
| DNeasy Blood & Tissue Kit (250) | Qiagen | 69581 | |
| 6-well Standard Line Multiwell Cell Culture Plates | VWR | 10062-892 | |
| Petri Dishes | Fisher | 12-565-90 | |
| Conical Tubes (15 mL) (racked) | Thermo Fisher | AM12500 | |
| Trypan Blue Solution, 0.4% | Thermo Fisher | 15250061 | |
| Reduced Serum Medium | Thermo Fisher | 31985062 |
参考文献
- Mandecki, W. Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis. Proc Natl Acad Sci. 83 (19), 7177-7181 (1986).
- Walder, R. Y., Walder, J. A. Oligodeoxynucleotide-directed mutagenesis using the yeast transformation system. Gene. 42 (2), 133-139 (1986).
- Moerschell, R. P., Tsunasawa, S., Sherman, F. Transformation of yeast with synthetic oligonucleotides. Proc Natl Acad Sci. 85 (2), 524-528 (1988).
- Yoon, K., Cole-Strauss, A., Kmiec, E. B. Targeted gene correction of episomal DNA in mammalian cells mediated by a chimeric RNADNA oligonucleotide. Genetics. 93, 2071-2076 (1996).
- Beetham, P. R., Kipp, P. B., Sawycky, X. L., Arntzen, C. J., May, G. D. A tool for functional plant genomics: chimeric RNA/DNA oligonucleotides cause in vivo gene-specific mutations. Proc Natl Acad Sci U S A. 96 (15), 8774-8778 (1999).
- Bertoni, C., Morris, G. E., Rando, T. A. Strand bias in oligonucleotide-mediated dystrophin gene editing. Hum Mol Gen. 14 (2), 221-233 (2004).
- Alexeev, V., Yoon, K. Stable and inheritable changes in genotype and phenotype of albino melanocytes induced by an RNA-DNA oligonucleotide. Nat Biotechnol. 16 (13), 1343-1346 (1998).
- Zorin, B., Hegemann, P., Sizova, I. Nuclear-gene targeting by using single-stranded DNA avoids illegitimate DNA integration in Chlamydomonas reinhardtii. Eukaryot Cell. 4 (7), 1264-1272 (2005).
- Brachman, E. E., Kmiec, E. B. DNA replication and transcription direct a DNA strand bias in the process of targeted gene repair in mammalian cells. J Cell Sci. 117 (Pt 17), 3867-3874 (2004).
- Pierce, E. A., et al. Oligonucleotide-directed single-base DNA alterations in mouse embryonic stem cells. Gene Ther. 10 (1), 24-33 (2003).
- Radecke, S., Radecke, F., Peter, I., Schwarz, K. Physical incorporation of a single-stranded oligodeoxynucleotide during targeted repair of a human chromosomal locus. J Gene Med. 8 (2), 217-228 (2006).
- Bertoni, C., Rustagi, A., Rando, T. A. Enhanced gene repair mediated by methyl-CpG-modified single-stranded oligonucleotides. Nucleic Acids Res. 37 (22), 7468-7482 (2009).
- Andrieu-Soler, C., et al. Stable transmission of targeted gene modification using single-stranded oligonucleotides with flanking LNAs. Nucleic Acids Res. 33 (12), 3733-3742 (2005).
- Olsen, P. A., Randol, M., Krauss, S. Implications of cell cycle progression on functional sequence correction by short single-stranded DNA oligonucleotides. Gene Ther. 12 (6), 546-551 (2005).
- Engstrom, J. U., Kmiec, E. B. DNA replication, cell cycle progression and the targeted gene repair reaction. Cell Cycle. 7 (10), 1402-1414 (2008).
- Aarts, M., te Riele, H. Parameters of oligonucleotide-mediated gene modification in mouse ES cells. J Cell Mol Med. 14 (6b), 1657-1667 (2010).
- Brachman, E. E., Kmiec, E. B. Gene repair in mammalian cells is stimulated by the elongation of S phase and transient stalling of replication forks. DNA Repair. 4 (4), 445-457 (2005).
- Engstrom, J. U., Suzuki, T., Kmiec, E. B. Regulation of targeted gene repair by intrinsic cellular processes. BioEssays. 31 (2), 159-168 (2009).
- Parekh-Olmedo, H., Ferrara, L., Brachman, E., Kmiec, E. B. Gene therapy progress and prospects: targeted gene repair. Gene Ther. 12 (8), 639-646 (2005).
- Olsen, P. A., Randol, M., Luna, L., Brown, T., Krauss, S. Genomic sequence correction by single-stranded DNA oligonucleotides: role of DNA synthesis and chemical modifications of the oligonucleotide ends. J Gene Med. 7 (12), 1534-1544 (2005).
- Wang, Z., Zhou, Z. -. J., Liu, D. -. P., Huang, J. -. D. Double-stranded break can be repaired by single-stranded oligonucleotides via the ATM/ATR pathway in mammalian cells. Oligonucleotides. 18 (1), 21-32 (2008).
- Ferrara, L., Kmiec, E. B. Camptothecin enhances the frequency of oligonucleotide-directed gene repair in mammalian cells by inducing DNA damage and activating homologous recombination. Nucleic Acids Res. 32 (17), 5239-5248 (2004).
- Ferrara, L., Parekh-Olmedo, H., Kmiec, E. B. Enhanced oligonucleotide-directed gene targeting in mammalian cells following treatment with DNA damaging agents. Exp Cell Res. 300 (1), 170-179 (2004).
- Lin, S., Staahl, B., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. eLife. 3, 04766 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnol. 31 (9), 827-832 (2013).
- Rivera-Torres, N., et al. The position of DNA cleavage by TALENs and cell synchronization influences the frequency of gene editing directed by single-stranded oligonucleotides. PLoS One. 9 (5), (2014).
- Strouse, B., Bialk, P., Niamat, R. a., Rivera-Torres, N., Kmiec, E. B. Combinatorial gene editing in mammalian cells using ssODNs and TALENs. Sci Rep. 4 (ii), 3791 (2014).
- Bialk, P., Rivera-Torres, N., Strouse, B., Kmiec, E. B. Regulation of Gene Editing Activity Directed by Single-Stranded Oligonucleotides and CRISPR/Cas9 Systems. PloS One. 10 (6), e0129308 (2015).
- Yu, C., et al. Small Molecules Enhance CRISPR Genome Editing in Pluripotent Stem Cells. Cell Stem Cell. 16 (2), 142-147 (2015).
- Rivera-Torres, N., Banas, K., Bialk, P., Bloh, K. M., Kmiec, E. B. Insertional Mutagenesis by CRISPR/Cas9 Ribonucleoprotein Gene Editing in Cells Targeted for Point Mutation Repair Directed by Short Single-Stranded DNA Oligonucleotides. PLoS One. 12 (1), e0169350 (2017).
- Mali, P., et al. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nat Biotechnol. 31 (9), 833-838 (2013).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnol. 34 (3), 339-344 (2016).
- Slaymaker, I. M., Gao, L., Zetsche, B., Scott, D. A., Yan, W. X., Zhang, F. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved