Method Article
バイオセーフティレベル3(BSL-3)条件下での病原体感染と単一細胞シーケンシングに対する消化管オルガノイドの適応
要約
このプロトコルは、単一細胞RNAシーケンシング(scRNAEq)技術を用いて、単細胞レベルで宿主/病原体相互作用を特徴付けるために、彼らの補助的またはバソラターレ側からヒト腸器官に感染する方法を説明する。
要約
ヒト腸器官は、消化管の病原体感染を研究するのに最適な細胞モデルを構成する。これらのオルガノイドは、消化管のすべてのセクション(胃、十二指腸、十二指腸、結腸、直腸)から誘導され、分化時に、各個々のセクションに自然に見られる細胞タイプのほとんどを含むことができる。例えば、腸管オルガノイドは、栄養吸収エンテロサイト、分泌細胞(ゴブレット、パネス、および腸内分泌)、幹細胞、ならびにすべての系統特異的分化中間体(初期または未熟な細胞タイプ)を含む。消化管由来のオルガノイドを使用して感染症を研究する最大の利点は、腸内病原体が標的とする細胞型を正確に特定し、胃腸管の異なる切片とその特定の細胞型が同様に病原体の課題に対応するかどうかに対処する可能性である。過去数年間、胃腸モデルは、他の組織からのオルガノイドと同様に、ウイルス性の栄養と病因のメカニズムを研究するために採用されてきました。しかし、高病原性ウイルスを使用する際にオルガノイドを使用することの利点をすべて利用することは技術的な課題であり、厳格なバイオセーフティの考慮事項が必要です。さらに、オルガノイドは3次元で成長することが多いため、細胞のバソラショナル側はオルガノイドの外側に向き、その補助側はオルガノイドの内側(内腔)に向いています。この組織は、多くの腸内感染が摂取後に細胞の有端/明るい側から開始するので、腸内病原体にとって課題となっています。以下の原稿は、ヒト腸器官を腸内病原体に感染させる準備をするための包括的なプロトコルを提供する。この方法は、オルガノイドの調製と、バイオセーフティレベル3(BSL-3)の封じ込め条件下でこの作業を行うために必要な考慮事項を詳述する。
概要
ヒト腸ウイルスに対する細胞型特異的な栄養刺激性と細胞型特異的免疫応答の研究は、ヒトの原発性細胞モデルの欠如により歴史的に困難であった。この制限は、オルガノイド1の開発に伴って部分的に根絶されました。胃腸管の場合、胃および腸のオルガノイドモデルは、ヒトおよび他のいくつかの種(例えば、ネズミ、ウシ、ネコ、コウモリ)2、3、4、5、6のために開発されている。腸器官は、ヒト腸上皮の構造アーキテクチャを再現し、陰窩および絨毛様構造、機能的な腸系統を含み、これまで知られていなかった細胞系統を同定するためにも使用されてきた。腸のオルガノイドを成長させるために2つの異なるアプローチを使用することができます。まず、クリプトを含む腸幹細胞は組織切除または生検から分離され、特定の培養条件(例えば、Wnt3A、R-spondin、Noggin、EGF)で増殖し、次いで幹細胞をほとんどの腸細胞系統(例えば、腸球、パネチ細胞、ゴブレット細胞、腸内腸切分)に分化する。この方法は、消化管のすべてのセクション(例えば、胃、十二指腸、精腸、腸管)からオルガノイドの分離を可能にする。第2の方法は、ヒトが誘導する多能性幹細胞または胚性幹細胞に依存し、その後、腸上皮細胞8に段階的なプロセスで分化される。これらの誘導幹細胞ベースのオルガノイドは、患者由来のオルガノイドと比較して、本質的により胚性であるとしばしば説明される。これらのオルガノイドモデルはすべて腸管を形成するために必要な発達的手掛かりを解明するために重要であったが、感染症研究におけるその使用はまだ初期段階にある。
腸管ウイルスは、ピコルナウイルス(例えば、EV-71)、レオウイルス(例えば、ロタウイルス)、カリシウイルス(例えば、ノロウイルス)9のような胃腸管を介して感染するすべてのウイルスをカバーする広い用語である。腸ウイルスは汚染された食物および水の摂取を通じて感染性のライフサイクルを開始し、開発途上国の人々は、未処理の廃棄物の環境への排出および感染の発症後の医療不足によるリスクが高いままにする。病原体の種類によっては、腸内膜の漏れによる胃腸炎、嘔吐、水漏れにつながる可能性があります。ヒトノロウイルスは、非常に流行し、感染性の高い腸病原体であり、世界中で6億人以上の感染と1500万人の入院につながる。オルガノイドは、ヒトノロウイルスの感染および複製をサポートするため、ノロウイルス研究の鍵となってきましたが、以前は標準的な細胞培養モデル12では培養できませんでした。
過去20年間で、コロナウイルスは主要なヒト病原体13として出現した。このファミリーには、高病原性MERS、SARS-CoV-1、SARS-CoV-2が含まれており、これらのウイルスの研究を行う際に厳格な安全レベルの封じ込めが必要です。興味深いことに、これらの3つの病原体はすべて、主に誘発された呼吸器症状と苦痛のために認識されているが、これらのウイルスは気道だけでなく他の器官にも感染しないことが明らかである。呼吸困難以外の感染患者であるSARS-CoV-2に誘発される重要な病理は、胃腸症状14の存在である。SARS-CoV-2感染患者の一部は、非常に軽度から重度の下痢に至るまで、このような症状を示しています。さらに、SARS-CoV-2ゲノムは、感染した患者15の便および胃腸管生検で検出することができる。重要なことに、胃腸症状の存在は、MERSおよびSARS-CoV-1感染患者においても観察されたSARS-CoV-2に限定されない。SARS-CoV-2が消化管苦痛を誘発し、消化管内のSARS-CoV-2のトロピズムを正確に同定する方法を理解するために、ヒト腸器官は重要なツールであり、現在この病原体16,17に対する細胞型特異的応答を解明するために利用されている。
細胞集団の転写プロファイリング(バルクRNAシーケンシング)は、オルガノイドと同様に不死化細胞株の両方の病原体感染を評価する際に標準的な習慣であった。これにより、病原体(サイトカインのアップレギュレーションなど)に対応して世界的な変化を判断することができますが、バルクRNAseqは、集団内の特定の細胞が他の細胞よりも感染しやすい理由を判断することを可能にしません。単細胞RNAシーケンシング(scRNAeq)は、細胞系分別転写プログラムを解明する強力なツールとなっており、これらのプログラムがウイルス感染をサポートまたは抑制する方法を決定するために使用することができる18,19。scRNAeqの最初の説明は2009年であり、マウスブラストミア20に見られる異なる細胞の転写プロファイルを評価するために使用された。これらのテクノロジは現在拡張され、複数の異なるプラットフォームを通じて実装できます。この技術の初期のバージョンでは、蛍光活性化細胞選別機(FACS)を使用してシーケンシング用の個々の細胞を分離し、96-または384ウェルプレートに制限されることが多く、これにより、サンプル21あたり300個の細胞を分析する。これらの方法は、ビーズを含むバーコードを含む個々の液滴に単一の細胞をカプセル化するためにマイクロ流体デバイスを使用する単一細胞シーケンシングプラットフォームによって進められている。この技術により、サンプル条件ごとに最大10,000個の細胞をキャプチャできます。
オルガノイド技術とscRNAEqを組み合わせることで、腸内病原体が細胞型特異的な方法で胃腸管にどのような影響を与えるかを研究することができます。ただし、技術的およびバイオセーフティに関する考慮事項を考慮する必要があります。まず第一に、古典的なオルガノイド培養方法(3次元(3D)オルガノイドは、細胞外マトリックス(ECM)に埋め込まれた)、表皮細胞のバソラショナル側をオルガノイドの外側に露出させる。腸内病原体が汚染された食物/水の摂取によって感染を開始するにつれて、感染は最も頻繁にこれらの3D腸器官ではアクセスできない細胞の有端側から開始される。したがって、オルガノイドは、2D播種を介して、直接細胞の補助側を露出、またはマイクロインジェクション22、23を介して、病原体感染のために有端側をアクセス可能にする準備が必要である。第二に、感染した生体試料のscRNAeqを行うためには、それらの感染性を考慮することが重要である。その後のRNAEqに対する単細胞分離の前に細胞を固定し、病原体を不活性化する方法が提案されているが、これらの方法はしばしばシーケンシング品質18の低下を招く。以下のプロトコルは、感染側(経腸対バソララル感染)を考慮した腸内ウイルスに腸内オルガノイドを感染させるいくつかのアプローチを説明する(図1)。さらに、このプロトコルには、scRNAEqの高病原性ウイルスに感染したオルガノイドから単一細胞を解離および分離するワークフローが含まれる。このプロトコルは、エアロゾルの発生や汚染の可能性を避けるために、バイオセーフティレベル3(BSL-3)の封じ込め条件下で作業する際に実装する必要がある重要なステップを強調します。
プロトコル
ヒト組織は、次のプロトコルのために大学病院ハイデルベルクから大腸切除または回腸生検から受け取った。この研究は、ヘルシンキ宣言に従って、すべての被験者からのインフォームド書面による同意を得て、ハイデルベルク大学病院の勧告の下で行われました.すべてのサンプルを匿名で受信し、維持しました。プロトコルS-443/2017の下で大学病院ハイデルベルクの倫理委員会によって承認されました.
1. 腸および大腸オルガノイドの維持及び通過
注意:ヒトの腸器官は、ヒト組織または人工多能性/胚性幹細胞に由来するため、倫理的承認が必要です。国別の規制に従う必要があります。ヒト材料は一般に試験されないので、BSL-2材料と考えられることが多い。実験が行われる国では、適切な封じ込め条件を確認する必要があります。
- 先に説明した方法2を用いて、単離組織および/または生検から腸および大腸オルガノイドを準備する。さらに、患者由来の材料またはiPSCからオルガノイドを調製する方法についての技術的な詳細は、LeesらとMaheらら24,25に見つけることができます。
- オルガノイド培養が確立されたら、以下に記載の分割ルーチンに従って、腸内病原体での感染を行うオルガノイドを準備する。
- 24ウェル非組織培養処理プレートで50μLの100%ECM溶液に20〜100オルガノイドをシード。500 μL の成長培地を加える (表 1) ウェルあたり.
- 250 μLの古いメディアを取り除き、各井戸に250 μLの新しい成長媒体を追加することで、2日ごとにメディアを交換します。メディアを交換する前に、メディアを室温まで温めます。
注意:冷たい媒体はオルガノイドを含むECM溶液を液化する。 - 死んだ細胞の蓄積により内部が暗くなり始めると、オルガノイドを週に1回通過させる。この例は 図 2を参照してください。
- 分割の日には、-20°CからECM溶液を取り除き、氷の上で解凍します。
- 37°Cで分割した後にオルガノイドを播種するために使用される新しい空の細胞培養プレートを置きます(これは最低1時間を暖かくする必要があり、一晩温めることができる)。培養培地を室温まで温めます。
- P1000ピペットで成長培地を取り出し、500 μLの冷たい1xリン酸緩衝生理食塩水(PBS)を各ウェルに3分間加えて、ECM溶液を部分的に液化し、プレートから解離します。
- ECMソリューションの完全な中断を確実にするため、P1000ピペット(450 μLでセットアップ)を使用してください。ピペットを10回上下してPBS、ECM溶液、オルガノイドを再中断し、再中断されたオルガノイドを15 mLの円錐形チューブに移し、氷の上に置きます。
- 同じ円錐形のチューブに同じオルガノイドの複数のウェルを収集します。
- 複数のオルガノイド(異なるドナー、異なるセクション、異なる前処理など)が同時に分割される場合は、それらを異なる円錐形のチューブに集めます。収集しながら、氷の上に円錐管を維持します。
- 4 °Cで5分間500 x g でサンプルを回転させます。 慎重にピペットでPBSを除去し、下部にオルガノイドペレットを維持します。
- オルガノイドのペレットが非常に緩く、PBSをあまりにも速く除去するときに簡単に吸引することができるので、ピペットを取り付けた真空廃棄物システムを使用しないでください。
- 円錐管に0.05%トリプシンの1 mLを加え、P1000ピペットで10回上下にピペットを入れ、オルガノイドを再中断します。オルガノイドを含む円錐管を37°Cで3分間インキュベートする。
- 10%のウシ胎児血清(FBS)と1%ペニシリン/ストレプトマイシンを含むDMEM/F12培地を2mL加えて消化を止め、P1000ピペットで10回上下してオルガノイドを破壊し直します。
- 4 °Cで5分間500 x g でサンプルを回転させます。 遠心分離機から円錐形のチューブを取り出し、氷の上に保管します。
- 5 mL の使い捨てピペットで慎重にメディア/トリプシンを取り出し、下部にオルガノイドペレットを維持します。約500μLの培地を残し、P1000ピペットで取り外します。
- すべての円錐形チューブから培地/トリプシンを取り除いたら、37°Cインキュベーターから24ウェル非細胞培養処理プレートを取り出し、細胞培養フードの下に置きます。
- 個々のドナーオルガノイドの成長習慣に応じて、1:3〜1:5比で分割オルガノイドを持つ円錐チューブに100%ECM溶液(氷上に保持)を加えます。(例えば、ECM溶液の50 μLドロップに20〜100オルガノイドを含む井戸が1つ通過した場合、オルガノイドペレットを150-250 μLの氷冷ECM溶液に再懸濁させる。
- 24ウェル非細胞培養処理プレートのウェル当たりECM溶液/オルガノイドミックスの種子50μLを37°Cに予め温めた。
注:ECM溶液は、それが暖かいことになったら非常に迅速に重合します。使用中は常にECM溶液を冷たく保ち(氷の上に保存)してください。オルガノイドが再懸濁されたら、すぐに新しいプレートに種をまきます。初心者には、シードのための余分な時間を許可するために-20°Cでピペットのヒントの箱を保存することをお勧めします。 - 24ウェルプレートを37°Cで10〜15分間インキュベートし、ECM溶液を重合させます。
- 重合後、500μLの成長培地(表1)26を各ウェルに加え、37°Cでオルガノイドをインキュベートする。 オルガノイドを顕微鏡で毎日チェックしてください。ステップ 1.4 のように、2 日ごとにメディアを変更します。
2. 2次元(2D)でのアガノイドの準備
注:次のプロトコルは、腸管の器官を細胞培養プレートの単層として播種し、その上端から腸上皮細胞に感染する方法を説明します。シーケンシング実験には48ウェルプレート、8ウェルガラス底チャンバースライドを使用して、免疫蛍光アプローチを使用して感染を制御します。
- セクション 1 で説明されているようにオルガノイドを成長および維持します。
- オルガノイドを2Dで播種する前に、48ウェルプレートと8ウェルガラス底チャンバースライドに200 μLの200 μLの人間のコラーゲンを水に37°Cで1時間コーティングします。
注:腸オルガノイドは、48ウェルプレートと8ウェルガラス底チャンバースライドで最もよく播種されています。経験は、彼らが96ウェルプレートでうまく感染しないことを示しています。トランスウェルインサートは、2Dでの有端およびバソララル感染の両方を可能にするためにも使用できます。トランスウェルを使用する場合は、感染前にトランス上皮電気抵抗(TEER)を監視し、コンフルエント単層を確認します。通常、腸のオルガノイドは450-600オーム/cm2のTEERとのきつい障壁を有する。 - 48ウェルプレートの各ウェルまたは8ウェルガラス底チャンバースライドの1つの井戸に100〜150オルガノイドをシード。
- オルガノイドの数を推定するには、セクション1で調製した24ウェルプレートの50μLのECM溶液滴に存在するオルガノイドの数を数える。平均して、1~2個のウェルが必要で、約15,000~30,000個の細胞が必要となります。
- ECM溶液を破壊し、オルガノイドをトリプシン化するには、ステップ1.8-1.17に従ってください。
- 48ウェルプレートと8ウェルガラス底チャンバースライドから人間のコラーゲンを取り除きます。
- 250 μL の成長培地/ウェルで円錐形チューブ内のオルガノイドペレットを再懸濁し、コラーゲンコーティングされたウェルに混合物を追加します。プレートを37°Cインキュベーターに入れる。
注:実験を行う場合、複数の条件が比較されます(モック と 感染した+/- 対象の治療)。2Dシードオルガノイドの各ウェル間の変動を最小限に抑えるために、同じ円錐形のチューブに必要なオルガノイドの総数を収集します。24ウェルプレートの最大12の井戸からオルガノイドを収集すると、トリプシンの1 mLと中和媒体の2 mLを使用することができます。12~24ウェルを使用する場合は、これを2mLのトリプシンと4mLの中和培地に増やします。 - 48時間後、インキュベーターからプレートを取り出し、細胞培養フードに入れる。成長培地を取り外し、分化培地のウェル当たり250μLに交換する(表1)。このメディア変更を 48 時間後に繰り返します。
- 48時間後、RNAを抽出し、250ngのRNAでcDNAを作り、幹細胞(OLFM4および/またはSMOC2)、ゴブレット細胞(MUC2)、腸内分泌細胞(CHGA)、腸腸細胞(SIおよび図34)を用いてSYBRグリーンベースのqPCRを行うことによって分化(分化培地への切り替え後4日間)を確認する。
注:成長培地から分化培地に切り替えると、オルガノイドは幹細胞マーカー(OLFM4および/またはSMOC2)の発現を減少させ、ゴブレット細胞(MUC2)、腸内分泌細胞(CHGA)、および腸球(SIおよび/またはCYP3A4)マーカー(SIおよび/またはCYP3A4)マーカーをqPCRによって増加させます。成長媒体 と分化 媒体における細胞型マーカーの発現レベルを比較するために、余分な井戸を用意し、成長媒体で維持します。 - 分化の確認に際して、オルガノイドは、有端な感染症の準備ができています。オルガノイドを、選択した病原体に必要な生体安全レベルの封じ込めに移動します。
注意:BSL-2またはBSL-3封じ込めの下で病原体を取り扱う場合は、研究所の現地の規制と標準的な操作手順を参照してください。 - 2Dで成長した腸内オルガノイドを補助側から感染させるには、P1000ピペットで培地を取り出し、分化培地で実験に必要な感染の多重度で希釈された病原体を添加します(病原体/培地ミックスの最小体積はウェルあたり50μL、最大体積はウェルあたり250μLです)。
- 細胞培養インキュベーターに位置するロッカーテーブルまたは15〜20分ごとにプレートを手動で揺らすことで、37°Cで2時間感染を進行させます。選択の病原体に基づいて、この時間を最適化します。
- 2時間後、P1000ピペットでメディアを取り外し、新鮮な分化メディアのウェルあたり250 μLに交換します。細胞を37°Cで単細胞シーケンシングの時点までインキュベートする。
- 単一セルのシーケンス処理のためにセルを準備するには、セクション 4 に進みます。
3. アペラルおよびバソララル感染のための3次元(3D)でのオルガノイドの調製
- 24ウェルでセクション1に記載されているようにオルガノイドを成長させ、非細胞培養処理プレート。
- 過越後2日間、インキュベーターからプレートを取り出し、細胞培養フードに入れる。
- P1000ピペットで成長培地を取り出し、室温に予め温めた500 μL/ウェルの分化培地に交換し、プレートを37°Cインキュベーターに入れてください。
- 48時間後、新鮮な分化メディア(500 μL/well)に交換してください。
注:オルガノイドは、感染前の合計4日間、分化培地に保持されています。 - ステップ 2.9 で説明した分化を確認します。
- 分化が起こったことを確認すると、オルガノイドは感染の準備ができています。
- 氷の上でECMを解凍します。分化媒体を室温まで温め、37°Cで24ウェルプレートを温めます。
- 感染を行うために、ECM溶液からオルガノイドを取り除く。
注: ウイルスがセルよりも ECM ソリューションに固執することを好むので、できるだけ多くの ECM ソリューションを削除します。オルガノイドの周りにECM溶液が多く残っている場合、感染性は深刻な影響を受けます。 - 500 μLのコールド1x PBSを加え、3分間インキュベートしてECMを中断します。P1000を使用して、10倍上下にピペットを使用します。チューブ内のオルガノイドと500 x gの遠心分離機を5分間組み合わせます。P1000でPBSを取り外します。
注:各感染状態間の変動を最小限に抑えるために、24ウェルプレートのいくつかのウェルから来るオルガノイドを同じ円錐形のチューブに組み合わせます。例えば、感染に8つの条件が必要な場合、24ウェルプレート(それぞれ〜100個のオルガノイドを含む)の8つのウェルが組み合わされ、その後、針の破壊に続いて新しい24ウェルプレートの8つの井戸に分けられる(ステップ3.12を参照)。 - 分化培地の1 mLでオルガノイドを再中断する。有端およびバソラテラ感染の場合、ステップ3.10.1に従い、バソラテラ感染の場合はステップ3.10.2に従うだけです。
- 27G針を1mLの注射器に取り付け、ペレットを6回再中断してオルガノイドの破壊を可能にし、感染が補助側側とバソラテラ側の両方で起こるようにします。ステップ 3.11 に進みます。
注:オルガノイドは、針の破壊の前に観察された場合、暗い中心を持つ古典的な嚢胞に見えるタコの芽のオルガノイドでなければなりません。混乱に続いて、これらのオルガノイドは暗いインテリアを少なくして小さく見えます。感染を行う場合、常に最低2つの条件(模擬および感染)があり、24ウェルプレートの最低2つの井戸が最初に針ベースの破壊のために15 mL円錐形管に組み合わされます(少なくとも1mLが再懸濁に使用される理由を説明します)。2つ以上の条件を実行する場合は、1つの15 mL円錐形チューブに24ウェルプレートの10個までのウェルを組み合わせ、針の破砕のために1mLの分化培地で再中断します。中断後、n + 1当たり500 μLの分化メディアを追加します(たとえば、10ウェルを使用する場合は、合計5.5 mLを持つ4.5 mLでトップアップ)。10以上の条件が必要な場合は、いくつかの円錐形のチューブを使用してください。オルガノイドは、シリンジのこのボリュームでより良い破壊されるように1 mLの注射器をお勧めします。注射器の代わりに、平坦化されたP1000チップを採用する。 - オルガノイドを井戸当たり500μLの分化培地で再懸濁する。オルガノイドを破壊しないように注意し、補助側がアクセスできないように注意してください。ステップ 3.11 に進みます。
- 27G針を1mLの注射器に取り付け、ペレットを6回再中断してオルガノイドの破壊を可能にし、感染が補助側側とバソラテラ側の両方で起こるようにします。ステップ 3.11 に進みます。
- P1000ピペットを使用して24ウェル細胞培養プレートにウェルあたり500 μLのオルガノイド懸濁液を移し、選択した病原体に必要な生体安全レベルの封じ込めにプレートを移動します。
注意:BSL-2またはBSL-3封じ込めの下で病原体を取り扱う場合は、現地の規制および研究所の標準的な操作手順を参照してください。常に、BSL-3外のオルガノイドの針の破壊を行い、針による潜在的な事故を避けてください。現地の規制は、BSL-3での針の使用を妨げる可能性があります。 - 分化培地で希釈した病原体を添加して、実験に必要な感染の多重度に達する。合計体積500μLを超えないようにしてください(24ウェルプレートに合計1mLを持つ場合)。
- 細胞培養インキュベーターに位置するロッカーテーブルまたは15〜20分ごとにプレートを手動で揺らすことで、37°Cで2時間(この時間は選択した病原体に適応する必要があります)のために感染を進行させます。
- 2 h インキュベーションに続いて、オルガノイドを 1 つの 1.5 mL チューブに 1 つの条件に集め、500 x g で 4 °C で 5 分間回転します。
- P1000ピペットで病原体を含む培地を取り出し、氷の上に保管してください。オルガノイドペレットをPBSで一度洗います。
- オルガノイドを100%ECM溶液(以前は氷上で解凍した)の50μLで再懸濁し、プレートを24ウェル非細胞培養処理プレートに37°Cに予熱し、37°Cで24ウェルプレートを10〜15分間インキュベートしてECM溶液を重合させます。
- 重合後、各ウェルに室温で500μLの分化培地を加え、単一細胞シーケンシングのために収穫するまで37°Cでインキュベートします。
注:収穫が48時間以上のシード処理を行う必要がある場合は、古いメディアを取り外して500μLの新しい分化メディアに交換して、2日ごとに交換してください。しかし、経験からは、オルガノイドは分化培地下で成長するため、48時間を超える長くは生き残れないことを示しています。
4. バイオセーフティレベル3(BSL-3)条件における単細胞溶液の調製とゲルビーズ-インエマルジョン(GE)の調製
注:以下の手順を完了するには、単一セルシーケンシング装置(材料表)と100 μL反応を処理できるPCRマシンがBSL-3施設内に存在する必要があります。オルガノイドは、病原体および進入経路に応じてセクション2-3に記載されているように、セクション1に記載されているように成長し、感染する。感染後の事前決定された時間に、オルガノイドが収穫される。腸オルガノイドの収穫に用いられる方法を以下に記載する。
- 感染したオルガノイドを収穫する前に、-80°Cから単細胞ゲルビーズ(材料表)を取り除き、室温(少なくとも30分前)まで温めます。
- さらに、RT試薬、還元剤BおよびRT酵素C(すべて−20°Cで保存)を室温まで平衡化する。製造元の説明に記載されているとおりに、テンプレート スイッチの oligo を再び中断します。
注:以下の手順はすべて、考慮された病原体に対する確立された標準的な操作手順に従って、地域の生物安全規制に関して実行する必要があります。BSL-3の研究の経験則として、細胞培養フードから出る全ての血管(プレート、チューブなど)の外装が適切に汚染されていることを確認する必要があります。実験を行う人の手/手袋についても同様です。 - 2Dに播種したオルガノイド(セクション2)に対して感染を行うため、ステップ4.4に進みます。3Dオルガノイド(セクション3)に感染を行うため、セクション4.5に進みます。
- 2Dオルガノイドの感染の場合は、細胞培養プレートをボンネットに持ち込み、P1000ピペットで分化培地を取り除きます。
- 各ウェルに室温で1x PBSの250 μLを加えます。
- P1000ピペットでPBSを取り出し、48ウェルプレートの各ウェルに250 μLの室温解離酵素(例えば、TrypLE Express)を加えます。手袋を交換し、プレートを洗浄し、37°Cインキュベーターに解離酵素を入れなさい。
- 顕微鏡観察による細胞解離を5分ごとに観察する。
注:2Dで培養した腸器官を単一細胞に解約するのに約15分かかります。一部の病原体は細胞性が高く、オルガノイドが他の病原体よりもはるかに速く解体を引き起こすので、この時間は、最初に播種され、感染したオルガノイドの数に依存するので、調整する必要があります。 - 明らかな単一細胞懸濁液の確認後、プレートを細胞培養フードに戻し、48ウェルプレートのウェルあたり10%FBSを含むDMEM/F12培地250μLを添加して消化を停止します。
- P1000ピペットを使用して細胞を15 mLの円錐形チューブに集めます。
- 手袋を交換し、チューブを洗浄し、4°Cで5分間500 x g でサンプルを回転させます。
- P1000で慎重に培地/解離酵素を除去し、細胞ペレットを底部に保持します。0.1%BSAを含むPBSの最小量の単一細胞を再懸濁します。腸のオルガノイドの場合は、48ウェルプレートの各ウェルに対して250 μLで再懸濁します。
- フィルターを使用して FACS チューブにセル懸濁液を入れ、大きな塊を取り除き、細胞を含む FACS チューブを氷の上に置きます。
- ステップ 4.6 に進み、マスターミックスのセルカウントと準備を続行します。
- 3Dオルガノイドの感染の場合は、インキュベーターからプレートを取り出し、細胞培養フードに入れる。
- P1000ピペットを用いて、24ウェルプレートの各ウェルから分化培地を取り出します。各ウェルに500 μLの氷冷1x PBSを加え、氷上で3分間インキュベートします。
- ECMソリューションの完全な中断を確実にするため、P1000ピペット(450 μLに設定)を使用します。ピペットは、PBS、ECM溶液およびオルガノイドを再中断するために10回上下する。再中断されたオルガノイドを15 mLの円錐形のチューブに移し、氷の上に置きます。独自の15 mL円錐管で各感染状態を収集します。
注:15 mL円錐管を使用すると、50 mLの円錐管または1.5 mLチューブよりも、より明確で明確な細胞ペレットが得られます。 - 手袋を交換し、細胞培養フードからチューブを取り外します。チューブの外側を清掃します。
- 4 °Cで5分間500 x g でサンプルを回転させます。
- チューブを細胞培養フードに戻し、P1000ピペットでPBSを取り除き、チューブの底部からオルガノイドペレットを再懸濁させないようにします。
- ペレットを解離酵素の1 mL(例えば、トリプルエクスプレス)で再懸濁する。手袋を交換し、チューブを洗浄し、37°Cでサンプルをインキュベートします。
- 30分間10分ごとに、チューブを細胞培養フードに戻し、P1000ピペットを10回ピペットで上下にピペット化してオルガノイドを再中断します。
注:通常、腸のオルガノイドは、3Dで感染したときに単一の細胞懸濁液を形成するために約30分を必要とします。 - オルガノイドが単一細胞に解離される時期を決定するには、p10ピペットを使用してオルガノイド懸濁液を10μLとします。
- 使い捨てプラスチックセルカウンタースライドにサスペンションを置きます。サンプル入力ポートをクリアテープでシールします。
- 手袋を交換し、セルカウンターの外側を清掃してください。明視野顕微鏡を使用して、単一の細胞懸濁液が作られるかどうかを判断します。
- 単一細胞懸濁液の確認後、P1000ピペットで10%FBSとピペットを含むDMEM/F12培地の1mLを1mL加えて消化を停止します。
- 手袋を交換し、チューブを洗浄し、4°Cで5分間500 x g でサンプルを回転させます。
- P1000ピペットで慎重に培地/解離酵素を除去し、チューブの底部から細胞ペレットを再懸濁させないようにします。
- 0.1% BSA を含む PBS の最小量の単一セルを再中断します。腸オルガノイドの場合は250μLで再懸濁する。
- フィルターを使用して FACS チューブにセル懸濁液を入れ、大きな塊を取り除き、細胞を含む FACS チューブを氷の上に置きます。
- ステップ 4.6 に進み、マスターミックスのセルカウントと準備を続行します。
- 使い捨てプラスチックセル計数チャンバーに10 μLのセルサスペンションを加えて、μL当たりの細胞数を決定します。
- サンプルを透明テープで密封してから、細胞培養フードからサンプルを取り出す前に、この段階では、細胞懸濁液は依然として感染性である。
- 手袋を交換し、細胞計数チャンバーを清掃し、明視野顕微鏡を使用してセル数を数えます。
- 細胞培養フードの内部で、実験のサンプル数に応じて、1.5 mLチューブにRT試薬、テンプレートスイッチオリゴ、還元剤B、RT酵素Cのマスターミックスを準備します。各サンプルについて、アリコート33.4 μLのマスターがPCRチューブに混合し、氷の上に保存します。
- メーカーの指示に記載されているとおり、ターゲットセル番号に従って、マスターミックスに細胞と水を追加します。
注:細胞の50%〜60%は通常回収されます(すなわち、チップに10,000個のセルをロードすると、5,000-6,000個の細胞が分析に使用されます)。したがって、常に 10,000 個のセルを読み込みます。細胞密度がこれを許可しない場合は、細胞を遠心分離し、より低い体積で再中断します。チップが詰まってしまうので、チップを過負荷にしないように注意してください。また、細胞の解体が適切でない場合、ビーズごとに複数の細胞を取得するリスクが高くなり、下流のバイオインフォマティクス処理で除去する必要があります。 - 手袋を交換し、単一細胞コントローラを細胞培養フードに移動し、チップが密封されていないガスケットで覆われているだけであるようにチップを準備します。
注:感染した細胞懸濁液や潜在的なエアロゾルへの暴露を防ぐために、マシンはフードの内側にする必要があります。 - チップホルダーにシングルセルチップを追加し、未使用のレーンを50%グリセロールで満たします。
- メーカーの指示に従って、サンプルに使用されるレーンにマスターミックス、ビーズ、仕切り油を追加します。
- チップをガスケットで覆い、チップをコントローラにロードして、プログラムを起動します。
注:8車線のうち6車線のみをロードすることをお勧めします。不適切なエマルジョンの問題は、多くの場合、レーン1と8で発生します。同社は、すべての車線が独立しており、これは起こるべきではないと述べています。ただし、可能であれば、これら 2 つのレーンを避け、8 つのサンプルが必要な場合は 2 つのチップを実行します。 - プログラムが完了したら、チップとガスケットを取り外します。
- マルチチャンネルピペットを使用し、100 μLのエマルジョンをクリーンなPCRチューブに移します。完全なエマルジョンが発生したことを示す均一な白色を各ウェルに設定します。
- 手袋を交換し、チューブを洗浄し、PCRチューブを100 μLの反応をサポートできるPCRマシンに移します。プログラムを実行する: 53 °C 45分間;85°C 5分間
- 完了時に、4°Cでサンプルを保管してください。 この時点で、製造者の指示に従って反応を処理するか、4°Cで3日間、または-20°Cで1週間保存してください。
- 85°Cで5分後、ほとんどのエンベロープウイルスは不活性化され、通常の操作手順に従ってBSL-3からcDNAを取り除き、BSL-1の実験室でライブラリ調製を行います。
注:各細胞タイプの分化の程度は各実験によって若干異なるので、この実験は3つの生物学的複製(例えば、3つの異なる日)として行う必要があります。結果として得られるシーケンスライブラリは、1 回のシーケンス実行で異なるインデックスを作成し、一緒にシーケンス処理できます。
結果
単細胞シーケンシング用オルガノイドの調製
単一細胞のシーケンシング結果は、良質の細胞を使用することに大きく依存しています。オルガノイドの品質を確保するために、オルガノイドを適切に維持し、分割する準備ができている時期を決定するために、毎日観察する必要があります(図2)。オルガノイドを分割するタイミングはドナー依存です。一部のドナーはより迅速に成長し、5日ごとに分割する必要がありますが、他のドナーは遅く、10日ごとに分割する必要があります。平均して、オルガノイドは、中心が暗くなるときに週に1回分割されます(図2B)。オルガノイドが大きくなりすぎて、中央に死んだ細胞が多すぎると、オルガノイドは死んでしまいます。
オルガノイドは、Wnt3Aの量が多いメディアで維持されます。これは幹細胞ニッチをサポートし、成長し、増殖し続けるためにオルガノイドを促進します。これらの成長条件下では、オルガノイドには大量の幹細胞とトランジット増幅細胞が含まれ、成熟した腸球、ゴブレット細胞、腸内分泌細胞などの分化された細胞集団が少ない。しかし、ヒトの腸内で見られる細胞の複雑さを模倣するには、細胞分化を推進し、これらの細胞の多くを産生することが重要である。これは、メディア条件を変更し、Wnt3Aを削除し、R-SpondinとNogginを減らすことによって達成されます(表1)。通常、腸球、ゴブレット細胞、および腸内分泌細胞に対する細胞分化は、4日間分化培地を必要とする(図3)。良い差別化を得ることが重要です。そうでない場合は、病原体のトロピズムと細胞型特異的応答の評価が困難になります。
BSL-3病原体の不活性化の確認
完全な不活性化は、選択した病原体と確認し、BSL-3からcDNAを除去しても安全であることを検証する必要があります。SARS-CoV-2の場合、ウイルスの完全な不活性化は、SARS-CoV-2の100 μLを取り、85°Cで5分間PCRマシンでインキュベートすることによって検証されました。 その後、ウイルスをナイーブVero細胞に戻し、ウイルス感染を24時間、48時間、および72時間の感染後の免疫蛍光およびプラークアッセイによって非熱処理ウイルスと比較し、すべての粒子が感染しなくなったことを確認した。これらの結果は、地元の規制機関に送信され、その承認に応じて、単一細胞実験とcDNA処理が行われました。
単一セルのシーケンスの結果
SARS-CoV-2がヒト結腸およびイレナムオルガノイドに感染する方法を評価するために、単一細胞シーケンシングを行った。オルガノイドは上述のように調製し、SARS-CoV-2による有端感染を可能にする2D形式で感染した。感染細胞は、感染後12時間および24時間で採取し、上記のように単細胞シーケンシング用に処理した。単一細胞シーケンシングデータの解析により、ヒト腸上皮細胞の亜集団(未熟腸球2)のみがSARS-CoV-2の感染を支持していることを判断することができた(図4)。さらに、集団内のすべての細胞が感染したわけではないので、感染した細胞と非感染した傍観者細胞の両方を分析した(図5)。これらの結果は、SARS-CoV-2が感染細胞に伝播する炎症促進シグナルを誘導し、非感染した傍観者細胞がインターフェロン媒介免疫応答を示したことを示した。さらに、scRNA-Seqは、感染した細胞がウイルス媒介経路の閉塞によるインターフェロンを感知できないことを示した(図5)。バルクRNAシーケンシングを使用する場合、この情報を得ることができませんでした。

図1:腸内病原体に感染するためにヒト腸器官を準備する3つの異なる方法を描いた概略図。 有限性感染症は、腸内オルガノイドを2Dで播種することによって達成することができる。3Dオルガノイドを破壊することによって、有端およびバソララル感染を行うことができる。最後に、バソラテラのみの感染は、無傷の3D腸器官に感染することによって行うことができる。これらの各メソッドを使用して、単一セルのシーケンスのサンプルを生成できます。 この図の大きなバージョンを表示するには、ここをクリックしてください。
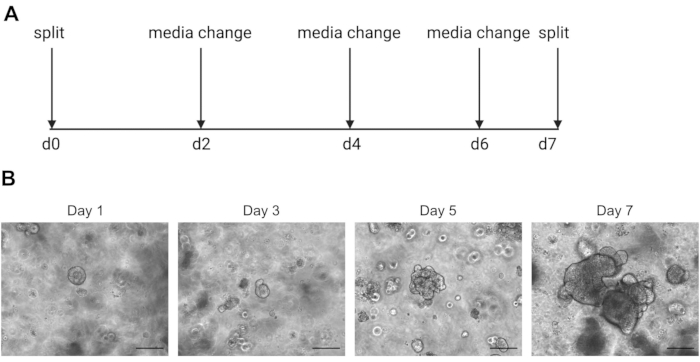
図2:オルガノイド維持回路図と代表的な明視野画像( A)ヒト腸器官の維持及び通過のための概略図(B) 1 日、3 日目、5 日目、7 日目のスプリット後の明視野画像を表します。7日目までに、オルガノイドは死んだ細胞の蓄積のために大きくて暗くなり、分割する準備ができています。スケールバーは25 μmを示します。 この図の大きなバージョンを表示するには、ここをクリックしてください。
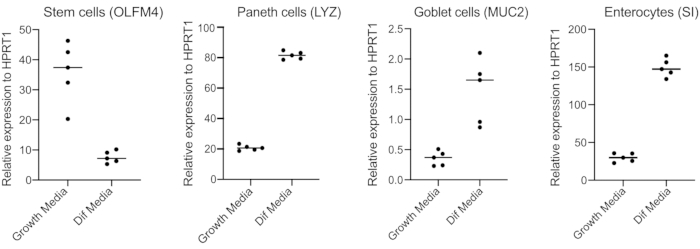
図3:ヒト腸器官の代表qPCRは分化培地への切り替え後4日後に行った。 腸管オルガノイドは、成長培地で維持されたか、または4日間分化培地に切り替えた。RNAを採取し、幹細胞(OLFM4)、パネス細胞(LYZ)、ゴブレット細胞(MUC2)、および腸球(SI)のマーカーに対してqPCRを行った。N = 5. この図の大きなバージョンを表示するには、ここをクリックしてください。
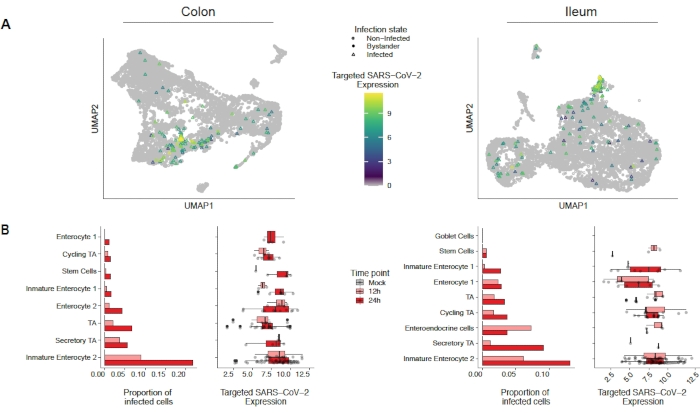
図4: SARS-CoV-2に感染した細胞集団の同定ヒト結腸およびイリウム由来オルガノイドは、SARS-CoV-2に感染した。12時間および24時間後に、感染後の細胞を採取し、単細胞RNAシーケンシングを行い、SARS-CoV-2感染を支持した細胞集団を同定した。ウイルス感染は時間の経過とともに増加し、主に未熟な腸球2に感染することが判明した。この図は、Trianaら19.この図の大きなバージョンを表示するには、ここをクリックしてください。
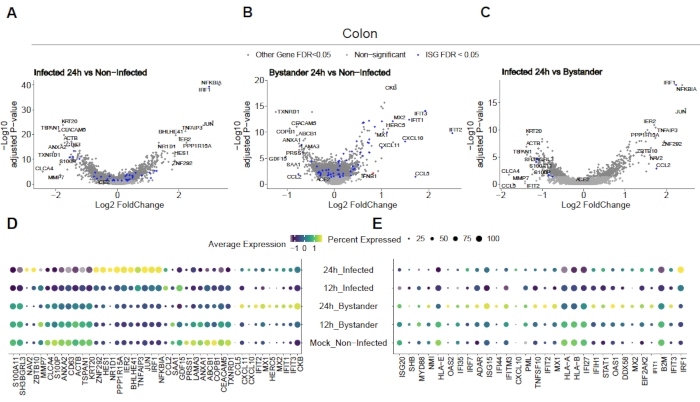
図5: 本来の自然免疫応答の決定ヒト結腸由来オルガノイドはSARS-CoV-2に感染した。12および24時間後に、感染後の細胞を採取し、単一細胞RNAシーケンシングを行い、ウイルス感染細胞および非感染性の傍観者細胞の両方における本質的な自然免疫応答を決定した。SARS-CoV-2感染細胞は強い炎症反応を示し、非感染した傍観者細胞はインターフェロン媒介応答を示した。トリアナら19.この図の大きなバージョンを表示するには、ここをクリックしてください。
| 成長メディア | |
| 化合物 | 最終濃度 |
| 広告 DMEM/F12 | グルタマックス (1X) |
| +グルタマックス | ヘペス 1 mM |
| +ヘペス | ペン 10 U/mL |
| +P/S | ストレップ 10 μg/mL |
| L-WRN | ボリュームで50% |
| B27 | 01:50 |
| N-アセチルシステイン | 1 mM |
| EGF | 50 ng/mL |
| A83-01 | 500 nM |
| IGF-1 | 100 ng/mL |
| FGF 基本 | 50 ng/mL |
| ガストリン | 10mM |
| 差別化メディア | |
| 化合物 | 最終濃度 |
| 広告 DMEM/F12 | グルタマックス (1x) |
| +グルタマックス | ヘペス 1 mM |
| +ヘペス | ペン 10 U/mL |
| +P/S | ストレップ 10 μg/mL |
| B27 | 01:50 |
| N-アセチルシステイン | 1 mM |
| R-スポンディン | ボリュームで5% |
| 頭 | 50 ng/mL |
| EGF | 50 ng/mL |
| ガストリン | 10mM |
| A83-01 | 500 nM |
表1:成長および分化媒体のためのメディア構成
ディスカッション
腸内病原体は、腸管の内腔に面した上端側から腸上皮細胞に感染することによって、そのライフサイクルを開始することが最も多い。オルガノイドは、腸上皮の細胞の複雑さと組織を再現するための良いモデルであるとよく認識されているが、その組織は三次元、閉じた構造として、その頭膜を病原体にアクセスできないようにする。このプロトコルは、彼らの補助側、そのそば側側、またはBLS-3病原体との両方から腸器官に感染する方法を説明した。これらのプロトコルは、以下に示すいくつかの重要なステップに従うことによって、BSL-2またはBLS-3封じ込めまたは他のオルガノイドモデルの下で任意の腸病原体を研究するために容易に適応することができる。上記の方法は、ドイツの規制に従って単細胞液滴の単離および調製を目的としている。免責事項として、このプロトコルはBSL-3条件下で作業する必要があるバイオセーフティの取り扱い措置(標準的な操作手順)を記述していません。また、他の国では規制が異なる可能性があり、すべての現地規制が尊重されるように地方自治体に連絡する必要があることを主張することも重要です。
有端感染のためにオルガノイドを2次元で播種する上で重要なステップの1つは、古典的な3次元オルガノイドとして成長したときと同様に細胞が分化することを制御することです。腸内病原体に応じて、トロピズムは非常にまれな細胞または高度に分化する必要がある細胞に制限される可能性があります。この場合、完全に分化しなかった2次元オルガノイドを使用すると、この腸内病原体は腸内オルガノイドに感染することができないという誤った結論を生じる可能性があります。可能であれば、このプロトコルの3つの構成を使用して感染を行うことが示唆されている:有端な感染症のための2Dオルガノイド(セクション2)、有端およびバソラテラ感染のための開いた3Dオルガノイド(セクション3)、およびバソラテラ感染のための完全な3Dオルガノイド(セクション3)。このアプローチは、病原体の進入経路(有端 対 バソラテラ)を識別するのに役立ち、同様のレベルの分化が達成されたことを制御します。2Dアプリカル感染の代替手段は、3Dオルガノイドを使用するが、病原体を補助側に直接送達するマイクロインジェクションである(詳細についてはBartfeldら27 を参照)。この方法は、病原体が適切に配置され、オルガノイドがそのまま残っていることを確認するために熟練した注射器を必要とします。マイクロインジェクションは、BSL-2封じ込めで一般的に使用され、BSL-3封じ込めには適さない場合があります。
2Dシードオルガノイドでの感染実験を行う場合の追加の重要な考慮事項は、最終的な細胞密度です。ステップ2.3で述べたように、100-150オルガノイドは48ウェルプレートの1つの井戸または8ウェルガラス底チャンバースライドの1つの井戸に播種されます。オルガノイドラインとオルガノイドを扱う人に応じて、これらのオルガノイドの大きさは有意に異なる場合があります。これは48ウェル版または8-wellガラス底の部屋のスライドの非常に異なった細胞密度をもたらすかもしれない。腸内ウイルスに応じて、一部のウイルスはよりまばらな細胞を好むが、他のウイルスはコンフルエント細胞に感染することもできる。異なる細胞合流のための感染性のこのような違いの分子起源は明らかではない。したがって、選択した腸病原体に最適な細胞密度を見つけることを目的としたパイロット実験は、さらに下流特性を実行する前に行われるべきである。
FACSの選別は、単細胞液滴エマルジョンを行う前に行われることが多い。このステップは、多くの場合、生細胞と単一細胞からダブレットから死んだ細胞を分離するために使用されます。BSL-3病原体を扱う場合、施設には適切なFACSソーターが装備されている必要があり、これはしばしばそうではありません。また、オルガノイド内のすべての細胞が同じサイズを持つわけではないため、多くの場合、ダブレットまたはより大きな細胞を区別することが困難であり、特定の細胞タイプに対して否定的に選択される危険性があります。また、各サンプルの5,000から10,000の間でソートに必要な時間が個々の細胞の転写プロファイルの大幅な変更をもたらす可能性があるかどうかについては、まだ現場で議論があります。単細胞シーケンシングと互換性のある細胞固定法(例えば、メタノールおよびRNAassist)が記載されているが、これがシーケンシング18の品質の低下につながることが観察された。最後に、細胞死マーカーを使用して細胞を選別することもバイアスにつながる可能性があると考えられています。クリルの先端に、クリの先端に、クリを流して放出される最も分化した細胞は、クリル軸を介した細胞の指向性増殖と分化を考えると、これらの細胞は、しばしば細胞死経路の異なるマーカー(例えば、アポトーシス、 壊死、およびネクロトーシス)に陽性である。しかし、マウス腸のロタウイルス感染を見ると、絨毛の先端が最も感染した領域28である。したがって、死のマーカーに陽性に見える細胞をフィルタリングすると、生理学的感染を表す可能性のある感染細胞の否定的な選択が生じる。現在、単一細胞シーケンシングの前にオルガノイドを選別し、固定するための良い解決策はありません。適切な代替プロトコルを見つけるためにさらなる研究が必要であるため、ライブを使用して、並べ替えられていないセルをお勧めします。
単一細胞シーケンシングは、細胞応答の評価方法に革命をもたらしています。この技術は、基底状態と病原体感染の両方で細胞系に特異的な応答を同定することを可能にする。このメソッドは、以前は一括読み出しによって制限されていた多くのフィールドでドアを開いています。この方法は非常に強力ですが、制限があります。重要な制限は、シーケンシングの下流で必要とされる広範なバイオインフォマティクス分析です。これは、組織を分析し、現在注釈がないセルタイプを割り当てる場合に特に重要です。熟練したバイオインフォマティクスを有することは、全ての単細胞研究を支援するために必要である。
このプロトコルは、ヒト腸器官を播種し、処理し、腸内病原体に感染し、scRNAeqを行う方法を記述する。オルガノイドモデルシステムは、ほとんどの臓器のために開発されているとして、他の臓器にこのアプローチを適応させることが可能になりました。肺と肝オルガノイドは腸内オルガノイドと同様に組織化されており、同様のアプローチを用いてこれらのオルガノイドに転置することができる。重要な制御は、2次元で成長するか、開いたひび割れ時に、これらのオルガノイドが3Dオルガノイドと同様の分化を達成することを検証することです。分化された状態を定義する特定の特徴と遺伝子は、各臓器モデルに固有です。腎臓および血管オルガノイドのような他のオルガノイドモデルは、大きな密な構造、単一細胞にこれらの構造を連続的に解体するための追加の方法が必要になる。
開示事項
著者らは、競合する財政的利益を宣言しない。
謝辞
メーガン・スタニファーとスティーヴ・ブーラントは、ドイツ・フォルシュングスゲコインシャフト(DFG)の研究助成金によって支えられました(プロジェクト番号240245660、 278001972、415089553、そしてスティーブ・ブーラントへの272983813とメーガン・スタニファーへの416072091、バーデン・ヴュルテンベルク州、ブンデスミニウム・フュル・ビルドゥン・ウント・フォルシュンBMBFからMSと01KI20198Aおよび(NUM-COVID 19、オルガノ・ストラット01KX202)がSBに作成された。
資料
| Name | Company | Catalog Number | Comments |
| Recombinant mouse noggin | Peprotech | Cat#250-38 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | Cat# G9145 | |
| 0.05% Trypsin-EDTA | Thermo Fischer Scientific | Cat#25300054 | |
| 24-well non-cell culture treated plate | Corning | Cat#3738 | |
| 8-well glass bottom chamber slide | iBIDI | Cart#80827 | |
| A83-01 | Tocris | Cat#2939 | |
| Advanced DMEM/F12 | Thermo Fischer Scientific | Cat# 12634010 | |
| B27 | Thermo Fischer Scientific | Cat#17504-044 | |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | Cat#1000202 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | Cat #1000127 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Single Cell 3′ Kit v3.1 | 10X Genomics | Cat#1000268 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Collagen from human placenta | Sigma Aldrich | Cat#C5533-5MG | |
| CYP34A forward | Eurofins | GATGGCTCTCATCCCAGACTT | Primers used to check for differentiation |
| CYP3A4 reverse | Eurofins | AGTCCATGTGAATGGGTTCC | Primers used to check for differentiation |
| DMEM/F12 | Thermo Fischer Scientific | Cat#11320074 | |
| EDTA | Sigma Aldrich | Car#E9884 | |
| Fast Read 102 counting slides | Biosigma | Cat# BVS100 | |
| Fetal Bovein Serum (FBS) | Capricorn | Cat#FBS-12A | |
| GlutaMAX | Thermo Fischer Scientific | Cat# 35050061 | |
| HEPES | Thermo Fischer Scientific | Cat3 15630080 | |
| L-WRN cells | ATCC | CRL-3276 | This cell line is used to make the conditioned media containg Wnt 3A, R-Spondin and Noggin. The protocol for the production of the conditioned media can be found on the manufacterures site. |
| MatriGel. GFR, LDEV free | Corning | Cat#354230 | |
| MUC-2 forward | Eurofins | TGTAGGCATCGCTCTTCTCA | Primers used to check for differentiation |
| MUC-2 reverse | Eurofins | GACACCATCTACCTCACCCG | Primers used to check for differentiation |
| N-acetyl-cysteine | Sigma Aldrich | Cat# A9165 | |
| OLMF4 forward | Eurofins | ACCTTTCCCGTGGACAGAGT | Primers used to check for differentiation |
| OLMF4 reverse | Eurofins | TGGACATATTCCCTCACTTTGGA | Primers used to check for differentiation |
| Penicillin/Streptomycin | Thermo Fischer Scientific | Cat#15140122 | |
| Recombinant human FGF basic | Peprotech | Cat#100-18B | |
| Recombinant human IGF-1 | BioLegend | Cat#590904 | |
| Recombinant mouse EGF | Thermo Fischer Scientific | Cat# PMG8043 | |
| SI forward | Eurofins | AATCCTTTTGGCATCCAGATT | Primers used to check for differentiation |
| SI reverse | Eurofins | GCAGCCAAGAATCCCAAT | Primers used to check for differentiation |
| TrypLE Express | Thermo Fischer Scientific | Cat#12605036 | |
| Y-27632 | Caymann Chemicals | Cat#10005583 |
参考文献
- Kretzschmar, K., Clevers, H. Organoids: Modeling development and the stem cell niche in a dish. Developmental Cell. 38, 590-600 (2016).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141, 1762-1772 (2011).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Tekes, G., Ehmann, R., Boulant, S., Stanifer, M. L. Development of feline ileum- and colon-derived organoids and their potential use to support feline coronavirus infection. Cells. 9, (2020).
- Derricott, H., et al. Developing a 3D intestinal epithelium model for livestock species. Cell and Tissue Research. 375, 409-424 (2019).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nature Medicine. 26, 1077-1083 (2020).
- Sato, T., Clevers, H. Growing self-organizing mini-guts from a single intestinal stem cell: mechanism and applications. Science. 340, 1190-1194 (2013).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470, 105-109 (2011).
- de Graaf, M., van Beek, J., Koopmans, M. P. Human norovirus transmission and evolution in a changing world. Nature Reviews Microbiology. 14, 421-433 (2016).
- Leshem, E., et al. Real-world effectiveness of pentavalent rotavirus vaccine among bedouin and jewish children in southern Israel. Clinical Infectious Diseases : An Official Publication of the Infectious Diseases Society Of America. 62, 155-160 (2016).
- Karst, S. M. The influence of commensal bacteria on infection with enteric viruses. Nature Reviews Microbiology. 14, 197-204 (2016).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science. 353, 1387-1393 (2016).
- de Wit, E., van Doremalen, N., Falzarano, D., Munster, V. J. SARS and MERS: recent insights into emerging coronaviruses. Nature Reviews Microbiology. 14, 523-534 (2016).
- Scaldaferri, F., et al. The thrilling journey of SARS-CoV-2 into the intestine: From pathogenesis to future clinical implications. Inflammatory Bowel Diseases. 26, 1306-1314 (2020).
- Kipkorir, V., Cheruiyot, I., Ngure, B., Misiani, M., Munguti, J. Prolonged SARS-CoV-2 RNA detection in anal/rectal swabs and stool specimens in COVID-19 patients after negative conversion in nasopharyngeal RT-PCR test. Journal of Medical Virology. 92, 2328-2331 (2020).
- Lamers, M. M., et al. SARS-CoV-2 productively infects human gut enterocytes. Science. 369, 50-54 (2020).
- Stanifer, M. L., et al. Critical role of type III interferon in controlling SARS-CoV-2 Infection in human intestinal epithelial cells. Cell Reports. 32, 107863 (2020).
- Triana, S., et al. Single-cell transcriptomics reveals immune response of intestinal cell types to viral infection. bioRxiv. , (2020).
- Triana, S., et al. Single-cell analyses reveal SARS-CoV-2 interference with intrinsic immune response in the human gut. Molecular Systems Biology. 17, 10232 (2021).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50, 1-14 (2018).
- García-Rodríguez, I., Sridhar, A., Pajkrt, D., Wolthers, K. C. Put some guts into it: Intestinal organoid models to study viral infection. Viruses. 12, 1288 (2020).
- Stanifer, M. L., et al. Asymmetric distribution of TLR3 leads to a polarized immune response in human intestinal epithelial cells. Nature Microbiology. 5, 181-191 (2020).
- Lees, E. A., et al. Using human induced pluripotent stem cell-derived intestinal organoids to study and modify epithelial cell protection against salmonella and other pathogens. Journal of Visualized Experiments: JoVE. (147), e59478 (2019).
- Mahe, M. M., Sundaram, N., Watson, C. L., Shroyer, N. F., Helmrath, M. A. Establishment of human epithelial enteroids and colonoids from whole tissue and biopsy. Journal of Visualized Experiments: JoVE. (97), e52483 (2015).
- Fujii, M., et al. Human Intestinal organoids maintain self-renewal capacity and cellular diversity in niche-inspired culture condition. Cell Stem Cell. 23 (6), 787-793 (2018).
- Bartfeld, S., Clevers, H. Organoids as model for infectious diseases: Culture of human and murine stomach organoids and microinjection of Helicobacter Pylori. Journal of Visualized Experiments: JoVE. (105), e53359 (2015).
- Hernandez, P. P., et al. Interferon-lambda and interleukin 22 act synergistically for the induction of interferon-stimulated genes and control of rotavirus infection. Nature Immunology. 16, 698-707 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved