Method Article
Anpassung gastrointestinaler Organoide für Pathogeninfektionen und Einzelzellsequenzierung unter Biosicherheitsstufe 3 (BSL-3)
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt, wie menschliche Darmorganoide entweder von ihrer apikalen oder basolateralen Seite infiziert werden können, um Wirt/Pathogen-Interaktionen auf Einzelzellebene mit hilfe der Einzelzell-RNA-Sequenzierungstechnologie (scRNAseq) zu charakterisieren.
Zusammenfassung
Menschliche Darmorganoide stellen das beste zelluläre Modell dar, um pathogene Infektionen des Magen-Darm-Traktes zu untersuchen. Diese Organoide können aus allen Abschnitten des GI-Traktes (Magen, Jejunum, Zwölffingerdarm, Ileum, Dickdarm, Rektum) abgeleitet werden und enthalten bei der Differenzierung die meisten Zelltypen, die in jedem einzelnen Abschnitt natürlich vorkommen. Zum Beispiel enthalten Darmorganoide nährstoffabsorbierende Enterozyten, sekretorische Zellen (Kelch, Paneth und enteroendokrine), Stammzellen sowie alle linienspezifischen Differenzierungszwischenprodukte (z. B. frühe oder unreife Zelltypen). Der größte Vorteil bei der Verwendung von Organoiden aus dem Magen-Darm-Trakt zur Untersuchung von Infektionskrankheiten ist die Möglichkeit, genau zu identifizieren, auf welchen Zelltyp der enterische Erreger abzielt, und zu untersuchen, ob die verschiedenen Abschnitte des Magen-Darm-Trakts und ihre spezifischen Zelltypen in ähnlicher Weise auf pathogene Herausforderungen reagieren. In den letzten Jahren wurden gastrointestinale Modelle sowie Organoide aus anderen Geweben eingesetzt, um den viralen Tropismus und die Mechanismen der Pathogenese zu untersuchen. Die Nutzung aller Vorteile der Verwendung von Organoiden beim Einsatz hochpathogener Viren stellt jedoch eine technische Herausforderung dar und erfordert strenge Überlegungen zur biologischen Sicherheit. Da Organoide oft dreidimensional gezüchtet werden, ist die basolaterale Seite der Zellen der Außenseite des Organoids zugewandt, während ihre apikale Seite der Innenseite (Lumen) der Organoide zugewandt ist. Diese Organisation stellt eine Herausforderung für enterische Pathogene dar, da viele enterische Infektionen nach der Einnahme von der apikalen / luminalen Seite der Zellen ausgehen. Das folgende Manuskript wird ein umfassendes Protokoll zur Vorbereitung menschlicher Darmorganoide auf die Infektion mit enterischen Pathogenen liefern, indem die Infektionsseite (apikal vs. basolateral) berücksichtigt wird, um eine Einzelzell-RNA-Sequenzierung durchzuführen, um zelltypspezifische Wirt/Pathogen-Interaktionen zu charakterisieren. Diese Methode beschreibt die Herstellung der Organoide sowie die Überlegungen, die erforderlich sind, um diese Arbeit unter den Eindämmungsbedingungen der Biosicherheitsstufe 3 (BSL-3) durchzuführen.
Einleitung
Die Untersuchung des zelltypspezifischen Tropismus und der zelltypspezifischen Immunantwort auf menschliche enterische Viren war aufgrund des Mangels an primären menschlichen Zellmodellen historisch eine Herausforderung. Diese Einschränkung wurde nun mit der Entwicklung von Organoiden teilweise beseitigt1. Im Falle des Magen-Darm-Traktes wurden Magen- und Darmorganoidmodelle für Menschen und mehrere andere Spezies (z. B.Murine, Rind, Katze, Fledermaus)entwickelt 2,3,4,5,6. Darmorganoide reproduzieren die strukturelle Architektur des menschlichen Darmepithels und enthalten krypten- und zottenartige Strukturen, funktionelle Darmlinien und wurden sogar verwendet, um bisher unbekannte Zelllinien zu identifizieren. Zwei verschiedene Ansätze können verwendet werden, um Darmorganoide zu züchten. Zuerst werden intestinale Stammzellen, die Krypten enthalten, aus Geweberesektionen oder Biopsien isoliert und unter bestimmten Kulturbedingungen (z. B. Wnt3A, R-Spondin, Noggin und EGF) gezüchtet, um sich auszudehnen, und dann die Stammzellen zu den meisten Darmzelllinien (z. B.Enterozyten, Paneth-Zellen, Kelchzellen, enteroendokrine Zellen)differenzieren 7. Diese Methode ermöglicht die Isolierung von Organoiden aus allen Abschnitten des Magen-Darm-Traktes(z. B.Magen, Zwölffingerdarm, Jejunum, Ileum und Dickdarm). Die zweite Methode stützt sich auf humaninduzierte pluripotente oder embryonale Stammzellen, die dann in einem schrittweisen Prozess zu Darmepithelzellen differenziert werden8. Diese induzierten Stammzell-basierten Organoide werden oft als embryonaler im Vergleich zu von Patienten abgeleiteten Organoiden beschrieben. Während all diese Organoidmodelle entscheidend für die Entschlüsselung von Entwicklungshinweisen waren, die zur Bildung des Darmtraktes benötigt werden, steckt ihre Verwendung in der Erforschung von Infektionskrankheiten noch in den Kinderschuhen.
Enterisches Virus ist ein weit gefasster Begriff, der alle Viren umfasst, die sich über den Magen-Darm-Trakt infizieren, wie Picornaviren (z.B.EV-71), Reoviren (z. B. Rotaviren) und Caliciviren (z. B. Norovirus)9. Enterische Viren beginnen ihren infektiösen Lebenszyklus durch die Aufnahme kontaminierter Lebensmittel und Wasser, wodurch Menschen in Entwicklungsländern aufgrund der Einleitung unbehandelter Abfälle in die Umwelt und mangelnder medizinischer Versorgung nach Beginn der Infektion einem hohen Risiko ausgesetzt sind10. Abhängig von der Art des Erregers kann die Infektion aufgrund des Auslaufens der Darmschleimhaut zu Gastroenteritis, Erbrechen und / oder wässrigem Durchfall führen. Humane Noroviren sind ein weit verbreiteter und hochinfektiöser enterischer Erreger, der weltweit zu über 600 Millionen Infektionen und 15 Millionen Krankenhausaufenthalten führt11. Organoide waren der Schlüssel zur Norovirusforschung, da sie die Infektion und Replikation des humanen Norovirus unterstützen, das zuvor in Standard-Zellkulturmodellen nicht kultiviert werden konnte12.
In den letzten zwei Jahrzehnten haben sich Coronaviren als wichtige menschliche Krankheitserreger herauskristallisiert13. Zu dieser Familie gehören die hochpathogenen MERS, SARS-CoV-1 und SARS-CoV-2, die bei der Erforschung dieser Viren strenge Sicherheitsstufen erfordern. Interessanterweise sind zwar alle drei dieser Erreger meist für ihre induzierten Atemwegssymptome und ihre Belastung bekannt, aber es ist jetzt offensichtlich, dass diese Viren nicht nur die Atemwege, sondern auch andere Organe infizieren. Eine wichtige Pathologie, die bei SARS-CoV-2-infizierten Patienten neben der Atemnot induziert wird, ist das Vorhandensein von gastrointestinalen Symptomen14. Ein Teil der SARS-CoV-2-infizierten Patienten zeigt solche Symptome, die von sehr leichtem bis schwerem Durchfall reichen. Darüber hinaus können SARS-CoV-2-Genome in Stuhl- und Magen-Darm-Traktbiopsien infizierter Patienten nachgewiesen werden15. Wichtig ist, dass das Vorhandensein von gastrointestinalen Symptomen nicht auf SARS-CoV-2 beschränkt ist, da sie auch bei MERS- und SARS-CoV-1-infizierten Patienten beobachtet wurden. Um zu verstehen, wie SARS-CoV-2 Magen-Darm-Beschwerden induziert und den Tropismus von SARS-CoV-2 im Magen-Darm-Trakt genau zu identifizieren, waren menschliche Darmorganoide ein Schlüsselwerkzeug und werden nun genutzt, um zelltypspezifische Reaktionen auf diesen Erreger zu entschlüsseln16,17.
Transkriptionelles Profiling einer Zellpopulation (Bulk-RNA-Sequenzierung) war Standardpraxis bei der Bewertung von Pathogeninfektionen sowohl von immortalisierten Zelllinien als auch von Organoiden. Während dies uns erlaubt, globale Veränderungen als Reaktion auf Krankheitserreger zu bestimmen (z. B. Hochregulierung von Zytokinen), erlaubt uns Bulk-RNAseq nicht zu bestimmen, warum bestimmte Zellen in einer Population anfälliger für Infektionen sind als andere. Die Einzelzell-RNA-Sequenzierung (scRNAseq) ist zu einem leistungsfähigen Werkzeug geworden, um zelllinienspezifische Transkriptionsprogramme zu entwirren, und kann verwendet werden, um zu bestimmen, wie diese Programme eine Virusinfektion unterstützen oder unterdrücken18,19. Die Erstbeschreibung von scRNAseq erfolgte 2009 und wurde verwendet, um die Transkriptionsprofile der verschiedenen Zellen zu bewerten, die in einem Mausblastomer20gefunden wurden. Diese Technologien wurden nun erweitert und können über mehrere verschiedene Plattformen implementiert werden. Frühe Versionen dieser Technologie verwendeten fluoreszenzaktivierten Zellsortierer (FACS), um einzelne Zellen für die Sequenzierung zu trennen, die oft auf 96- oder 384-Well-Platten beschränkt war, wodurch 300 einzelne Zellen pro Probe analysiert werdenkonnten 21. Diese Methoden wurden nun von den Einzelzell-Sequenzierungsplattformen weiterentwickelt, die ein mikrofluidisches Gerät verwenden, um einzelne Zellen in einzelne Tröpfchen mit Barcode-enthaltenden Kügelchen zu verkapseln. Diese Technologie ermöglicht es, bis zu 10.000 Zellen pro Probenzustand zu erfassen.
Durch die Kombination der Organoid-Technologie mit scRNAseq können wir untersuchen, wie enterische Pathogene den Magen-Darm-Trakt zelltypspezifisch beeinflussen. Es müssen jedoch mehrere technische und biologische Sicherheitsüberlegungen berücksichtigt werden. In erster Linie belichten klassische Organoidkulturmethoden (3-dimensionale (3D) Organoide, eingebettet in eine extrazelluläre Matrix (ECM)) die basolaterale Seite der Epithelzellen nach außen zum Organoid. Da enterische Erreger ihre Infektion durch die Einnahme von kontaminierter Nahrung / Wasser initiieren, beginnt die Infektion am häufigsten von der apikalen Seite der Zellen, die in diesen 3D-Darmorganoiden nicht zugänglich ist. Daher müssen Organoide so vorbereitet werden, dass sie die apikale Seite für eine Pathogeninfektion entweder durch 2D-Seeding, wodurch die apikale Seite der Zellen direkt freigelegt wird, oder durch Mikroinjektion22,23zugänglich machen. Zweitens, um scRNAseq von infizierten biologischen Proben durchzuführen, ist es wichtig, ihre infektiöse Natur zu berücksichtigen. Während Methoden zur Fixierung von Zellen und zur Inaktivierung von Krankheitserregern vor der Einzelzellisolierung für nachfolgende RNAseq vorgeschlagen wurden, führen diese Methoden häufig zu einer Abnahme der Sequenzierungsqualität18. Das folgende Protokoll beschreibt mehrere Ansätze zur Infektion von Darmorganoiden mit enterischen Viren unter Berücksichtigung der Infektionsseite (apikale vs. basolaterale Infektion) (Abbildung 1). Darüber hinaus wird das Protokoll einen Workflow zur Dissoziierung und Isolierung einzelner Zellen von Organoiden, die mit hochpathogenen Viren für scRNAseq infiziert sind, enthalten. Das Protokoll wird die wichtigsten Schritte hervorheben, die bei der Arbeit unter den Eindämmungsbedingungen der Biosicherheitsstufe 3 (BSL-3) umgesetzt werden müssen, um die Erzeugung von Aerosolen und potenzielle Kontaminationen zu vermeiden.
Protokoll
Menschliches Gewebe wurde aus Kolonresektions- oder Ileumbiopsien des Universitätsklinikums Heidelberg für das folgende Protokoll erhalten. Diese Studie wurde auf Empfehlung des Universitätsklinikums Heidelberg mit informierter schriftlicher Zustimmung aller Probanden gemäß der Deklaration von Helsinki durchgeführt. Alle Proben wurden anonymisiert empfangen und gepflegt. Das Protokoll wurde von der Ethikkommission des Universitätsklinikums Heidelberg unter dem Protokoll S-443/2017 genehmigt.
1. Erhaltung und Weitergabe von Darm- und Dickdarmorganoiden
VORSICHT: Menschliche Darmorganoide werden aus menschlichem Gewebe oder aus induzierten pluripotenten/embryonalen Stammzellen gewonnen, und als solche ist eine ethische Genehmigung erforderlich. Länderspezifische Vorschriften müssen befolgt werden. Menschliches Material wird im Allgemeinen nicht getestet und wird daher oft als BSL-2-Material betrachtet. In dem Land, in dem das Experiment stattfindet, müssen die richtigen Eindämmungsbedingungen bestätigt werden.
- Darm- und Dickdarmorganoide aus isolierten Geweben und/oder Biopsien unter Verwendung der zuvor beschriebenen Methoden2 herstellen. Darüber hinaus finden sich weitere technische Details zur Herstellung von Organoiden aus patientenabgeleitetem Material oder aus iPS-Zellen in Lees et al. und Mahe et al.24,25.
- Sobald Organoidkulturen etabliert sind, befolgen Sie die unten beschriebene Spaltroutine, um Organoide auf Infektionen mit enterischen Krankheitserregern vorzubereiten.
- Säen Sie 20-100 Organoide in 50 μL 100% ECM-Lösung in 24-Well-Nicht-Gewebekultur-behandelten Platten. Fügen Sie 500 μL Wachstumsmedien (Tabelle 1) pro Vertiefung hinzu.
- Wechseln Sie die Medien alle 2 Tage, indem Sie 250 μL der alten Medien entfernen und 250 μL der frischen Wachstumsmedien zu jeder Vertiefung hinzufügen. Erwärmen Sie das Medium auf Raumtemperatur, bevor Sie das Medium wechseln.
VORSICHT: Kalte Medien verflüssigen die ECM-Lösung, die Organoide enthält. - Passieren Sie die Organoide einmal pro Woche, wenn das Innere aufgrund der Ansammlung von toten Zellen dunkel zu werden beginnt. Ein Beispiel dafür finden Sie in Abbildung 2.
- Am Tag der Spaltung die ECM-Lösung von -20 °C entfernen und auf Eis auftauen.
- Legen Sie die neue leere Zellkulturplatte, die zur Aussaat der Organoide verwendet wird, nach der Spaltung bei 37 ° C auf (dies erfordert mindestens 1 h, um warm zu sein und könnte über Nacht erwärmt werden). Erwärmen Sie die Kulturmedien auf Raumtemperatur.
- Entfernen Sie das Wachstumsmedium mit einer P1000-Pipette und geben Sie 500 μL kalte 1x phosphatgepufferte Kochsalzlösung (PBS) für 3 min in jede Vertiefung, um die ECM-Lösung teilweise zu verflüssigen und von der Platte zu dissoziieren.
- Um eine vollständige Unterbrechung der ECM-Lösung zu gewährleisten, verwenden Sie eine P1000-Pipette (auf 450 μL eingestellt). 10-mal nach oben und unten pipettieren, um PBS, ECM-Lösung und Organoide wieder aufzunehmen, die resuspendierten Organoide in ein 15 ml konisches Rohr zu übertragen und auf Eis zu legen.
- Sammeln Sie mehrere Vertiefungen der gleichen Organoide in demselben konischen Rohr.
- Wenn mehrere verschiedene Organoide (verschiedene Spender, verschiedene Abschnitte, unterschiedliche Vorbehandlung usw.) gleichzeitig gespalten werden, sammeln Sie sie in verschiedenen konischen Röhrchen. Halten Sie während des Sammelns die konischen Röhren auf Eis.
- Die Proben bei 500 x g für 5 min bei 4 °C drehen. Entfernen Sie das PBS vorsichtig mit einer Pipette, um das Organoidpellet am Boden zu erhalten.
- Vermeiden Sie die Verwendung eines Vakuumabfallsystems mit einer eingebauten Pipette, da das Pellet von Organoiden sehr locker ist und leicht abgesaugt werden kann, wenn das PBS zu schnell entfernt wird.
- 1 ml 0,05% Trypsin in das konische Röhrchen geben und Organoide resuspendieren, indem sie 10 Mal mit einer P1000-Pipette auf und ab pipettieren. Das konische Röhrchen, das Organoide enthält, bei 37 °C für 3 min inkubieren.
- Fügen Sie 2 ml DMEM / F12-Medien hinzu, die 10% fötales Rinderserum (FBS) und 1% Penicillin / Streptomycin enthalten, um die Verdauung zu stoppen, und resuspend, um die Organoide zu stören, indem Sie 10-mal mit einer P1000-Pipette auf und ab pipettieren.
- Die Proben bei 500 x g für 5 min bei 4 °C drehen. Konische Röhrchen aus der Zentrifuge entfernen und auf Eis lagern.
- Entfernen Sie Medien / Trypsin vorsichtig mit einer 5 ml Einwegpipette, um das Organoidpellet am Boden zu erhalten. Lassen Sie etwa 500 μL Medien stehen und entfernen Sie dieses mit einer P1000-Pipette.
- Sobald Das Medium/Trypsin aus allen konischen Röhrchen entfernt wurde, entfernen Sie die mit 24 Well-Nicht-Zellkultur behandelte Platte aus dem 37 °C-Inkubator und legen Sie sie unter die Zellkulturhaube.
- Geben Sie 100% ECM-Lösung (auf Eis gehalten) in das konische Rohr mit den gespaltenen Organoiden im Verhältnis 1: 3 zu 1: 5, abhängig von den Wachstumsgewohnheiten der einzelnen Spenderorganoide. (Zum Beispiel, wenn eine Vertiefung mit 20-100 Organoiden in einem 50 μL Tropfen ECM-Lösung durchgelassen wurde, resuspendieren Sie das Organoidpellet in 150-250 μL eiskalte ECM-Lösung.)
- Samen 50 μL ECM-Lösung/Organoid-Mischung pro Vertiefung der 24-Well-Nicht-Zellkultur-behandelten Platte, die auf 37 °C vorgewärmt wurde.
HINWEIS: ECM-Lösung polymerisiert sehr schnell, sobald sie sich erwärmt hat. Halten Sie die ECM-Lösung während des Gebrauchs immer kalt (auf Eis gelagert). Wenn Organoide resuspendiert wurden, samen Sie sofort auf einer neuen Platte. Für Anfänger wird empfohlen, eine Schachtel Mit Pipettenspitzen bei -20 ° C aufzubewahren, um zusätzliche Zeit für die Aussaat zu haben. - Inkubieren Sie die 24-Well-Platte bei 37 °C für 10-15 min, damit die ECM-Lösung polymerisieren kann.
- Nach der Polymerisation werden zu jeder Vertiefung 500 μL Wachstumsmedien (Tabelle 1)26 gegeben und die Organoide bei 37 °C inkubiert. Überprüfen Sie die Organoide täglich durch Mikroskopie. Wechseln Sie die Medien alle 2 Tage wie in Schritt 1.4.
2. Vorbereitung von Organoiden in zwei Dimensionen (2D) für apikale Infektionen
HINWEIS: Das folgende Protokoll beschreibt, wie Darmorganoide als Monoschicht von Zellen in einer Zellkulturplatte ausgesät werden, um Darmepithelzellen von ihrer apikalen Seite aus zu infizieren. Verwenden Sie die 48-Well-Platte für die Sequenzierungsexperimente und den 8-Well-Glasbodenkammer-Objektträger, um die Infektion mit Immunfluoreszenzansätzen zu kontrollieren.
- Anbau und Erhaltung von Organoiden wie in Abschnitt 1 beschrieben.
- Bevor Sie Organoide in 2D aussäen, beschichten Sie die 48-Well-Platten und den 8-Well-Glasbodenkammer-Objektträger mit 200 μL 2,5% menschlichem Kollagen in Wasser pro Well für 1 h bei 37 ° C.
HINWEIS: Darmorganoide werden am besten in 48-Well-Platten und in 8-Well-Glasbodenkammer-Objektträgern ausgesät. Die Erfahrung hat gezeigt, dass sie sich in 96-Well-Platten nicht gut infizieren. Transwell-Einsätze können auch verwendet werden, um sowohl eine apikale als auch eine basolaterale Infektion in 2D zu ermöglichen. Wenn Transwells verwendet werden, überwachen Sie den transepithelialen elektrischen Widerstand (TEER) vor der Infektion, um eine konfluente Monoschicht zu bestätigen. Normalerweise haben Darmorganoide eine enge Barriere mit einem TEER von 450-600Ohm/cm2. - Säen Sie 100-150 Organoide in jede Vertiefung einer 48-Well-Platte oder für eine Vertiefung eines 8-Well-Glasbodenkammer-Objektträgers.
- Um die Anzahl der Organoide zu schätzen, zählen Sie die Anzahl der Organoide, die in den 50 μL ECM-Lösungstropfen der in Abschnitt 1 hergestellten 24-Well-Platte vorhanden sind. Im Durchschnitt werden 1-2 Bohrlöcher benötigt, die ungefähr 15.000-30.000 Zellen ergeben.
- Um die ECM-Lösung zu unterbrechen und die Organoide zu versuchen, führen Sie die Schritte 1.8-1.17 aus.
- Entfernen Sie menschliches Kollagen von den 48-Well-Platten und dem 8-Well-Glasbodenkammer-Objektträger.
- Resuspendieren Sie das Organoidpellet im konischen Röhrchen in 250 μL Wachstumsmedium / Well und geben Sie die Mischung in die kollagenbeschichteten Vertiefungen. Legen Sie die Platte in einen 37 °C Inkubator.
HINWEIS: Bei der Durchführung der Experimente werden mehrere Bedingungen verglichen (Mock vs. infizierte +/- Behandlung von Interesse). Um die Variabilität zwischen jeder Vertiefung von 2D-ausgesäten Organoiden zu minimieren, sammeln Sie die Gesamtzahl der notwendigen Organoide im selben konischen Rohr. Das Sammeln von Organoiden aus bis zu 12 Vertiefungen einer 24-Well-Platte kann 1 ml Trypsin und 2 ml neutralisierende Medien verwenden. Wenn Sie 12-24 Vertiefungen verwenden, erhöhen Sie dies auf 2 ml Trypsin und 4 ml neutralisierende Medien. - Nach 48 h entfernen Sie die Platte aus dem Inkubator und legen Sie sie in die Zellkulturhaube. Entfernen Sie das Wachstumsmedium und ersetzen Sie es durch 250 μL pro Vertiefung Differenzierungsmedium (Tabelle 1). Wiederholen Sie diesen Medienwechsel 48 h später.
- Bestätigen Sie nach 48 h die Differenzierung (vier Tage nach dem Wechsel zu Differenzierungsmedien), indem Sie RNA extrahieren, cDNA mit 250 ng RNA herstellen und eine SYBR green based qPCR unter Verwendung von Primern für Stammzellen (OLFM4 und / oder SMOC2), Kelchzellen (MUC2), enteroendokrine Zellen (CHGA) und Enterozyten (SI und / oder CYP3A4) durchführen (Abbildung 3).
HINWEIS: Beim Wechsel von Wachstumsmedien zu Differenzierungsmedien verringern Organoide ihre Expression von Stammzellmarkern (OLFM4 und/oder SMOC2) und erhöhen die Expression von Becherzellen (MUC2), enteroendokrinen Zellen (CHGA) und Enterozyten (SI und/oder CYP3A4) Markern durch qPCR. Bereiten Sie ein extra gut vor und pflegen Sie es mit Wachstumsmedien, um das Expressionsniveau jedes zelltypspezifischen Markers in Wachstums- und Differenzierungsmedien zu vergleichen. - Bei Bestätigung der Differenzierung sind die Organoide bereit für eine apikale Infektion. Verschieben Sie die Organoide in die Biosicherheitsstufe, die für den Erreger der Wahl erforderlich ist.
VORSICHT: Beachten Sie die lokalen Vorschriften und Standardarbeitsanweisungen des Instituts beim Umgang mit Krankheitserregern unter BSL-2- oder BSL-3-Containment. - Um die in 2D gezüchteten Darmorganoide von ihrer apikalen Seite zu infizieren, entfernen Sie die Medien mit einer P1000-Pipette und fügen Sie pathogen hinzu, der bei der für das Experiment erforderlichen Vielzahl von Infektionen in Differenzierungsmedien verdünnt ist (das Mindestvolumen des Pathogen/Medien-Mix beträgt 50 μL pro Vertiefung und das maximale Volumen beträgt 250 μL pro Vertiefung).
- Lassen Sie die Infektion für 2 h bei 37 °C entweder mit einem Wipptisch im Zellkultur-Inkubator oder durch manuelles Schaukeln der Platte alle 15-20 min ablaufen. Optimieren Sie diese Zeit basierend auf dem Erreger Ihrer Wahl.
- Entfernen Sie das Medium nach 2 h mit einer P1000-Pipette und ersetzen Sie es durch 250 μL pro Vertiefung frischer Differenzierungsmedien. Inkubieren Sie Zellen bei 37 °C bis zum Zeitpunkt der Einzelzellsequenzierung.
- Um Zellen für die Einzelzellsequenzierung vorzubereiten, fahren Sie mit Abschnitt 4 fort.
3. Herstellung von Organoiden in drei Dimensionen (3D) für apikale und basolaterale Infektionen
- Züchten Sie Organoide wie in Abschnitt 1 beschrieben in einer mit 24 Well, nicht mit Zellkultur behandelten Platte.
- Entfernen Sie die Platte zwei Tage nach dem Passieren aus dem Inkubator und legen Sie sie in eine Zellkulturhaube.
- Entfernen Sie das Wachstumsmedium mit einer P1000-Pipette, ersetzen Sie es durch 500 μL/Well differenziertes Differenzierungsmedium, das auf Raumtemperatur vorgewärmt wurde, und legen Sie die Platte in den 37 °C-Inkubator.
- Nach 48 h durch frisches Differenzierungsmedium (500 μL/Well) ersetzen.
HINWEIS: Organoide werden insgesamt 4 Tage vor der Infektion in Differenzierungsmedien gehalten. - Bestätigen Sie die Differenzierung wie in Schritt 2.9 beschrieben.
- Nach Bestätigung, dass eine Differenzierung stattgefunden hat, sind Organoide bereit für eine Infektion.
- ECM auf Eis auftauen. Erwärmen Sie die Differenzierungsmedien auf Raumtemperatur und erwärmen Sie eine 24-Well-Platte bei 37 °C.
- Um Infektionen durchzuführen, entfernen Sie die Organoide aus der ECM-Lösung.
HINWEIS: Entfernen Sie so viel ECM-Lösung wie möglich, da Viren es vorziehen, sich an die ECM-Lösung und nicht an Zellen zu halten. Wenn zu viel ECM-Lösung um die Organoide herum verbleibt, wird die Infektiosität stark beeinträchtigt. - Unterbrechen Sie das ECM, indem Sie 500 μL kalte 1x PBS hinzufügen und 3 Minuten lang inkubieren. Verwenden Sie einen P1000, um 10x nach oben und unten zu pipettieren. Organoide in einem Röhrchen kombinieren und 5 min mit 500 x g zentrifugieren. Entfernen Sie PBS mit einem P1000.
HINWEIS: Um die Variabilität zwischen den einzelnen Infektionszuständen zu minimieren, kombinieren Sie die Organoide, die aus mehreren Vertiefungen einer 24-Well-Platte stammen, in demselben konischen Rohr. Wenn beispielsweise acht Bedingungen für eine Infektion erforderlich sind, werden acht Vertiefungen einer 24-Well-Platte (die jeweils ~ 100 Organoide enthält) kombiniert und anschließend nach einer Nadelzerstörung in acht Vertiefungen einer neuen 24-Well-Platte aufgeteilt (siehe Schritt 3.12). - Resuspendieren Sie die Organoide in 1 ml Differenzierungsmedium. Für apikale und basolaterale Infektionen folgen Sie Schritt 3.10.1 und für basolaterale Infektionen nur Schritt 3.10.2.
- Befestigen Sie eine 27 G-Nadel an einer 1-ml-Spritze und resuspen sie das Pellet sechsmal, um eine Störung der Organoide und eine Infektion sowohl an der apikalen als auch an der basolateralen Seite zu ermöglichen. Fahren Sie mit Schritt 3.11 fort.
HINWEIS: Die Organoide sollten die klassischen zystenartigen und oktopusknospenförmigen Organoide mit einem dunklen Zentrum sein, wenn sie vor der Nadelstörung beobachtet werden. Nach der Störung erscheinen diese Organoide kleiner mit weniger bis gar keinem dunklen Inneren. Bei der Durchführung einer Infektion gibt es immer mindestens zwei Bedingungen (Mock und Infektion), mindestens zwei Vertiefungen einer 24-Well-Platte werden zunächst zu einem konischen 15-ml-Röhrchen für nadelbasierte Störungen kombiniert (was erklärt, warum mindestens 1 ml für die Resuspension verwendet wird). Wenn sie mehr als zwei Bedingungen erfüllen, kombinieren Sie bis zu 10 Vertiefungen einer 24-Well-Platte in einem einzigen konischen 15-ml-Rohr und resuspendieren Sie in 1 ml Differenzierungsmedien für Nadelunterbrechungen. Fügen Sie nach der Unterbrechung 500 μL Differenzierungsmedien pro n + 1 hinzu (z. B. wenn Sie 10 Vertiefungen verwenden, dann füllen Sie 4,5 ml auf, um insgesamt 5,5 ml zu erhalten). Wenn mehr als 10 Bedingungen erforderlich sind, verwenden Sie mehrere konische Rohre. Eine 1-ml-Spritze wird empfohlen, da die Organoide mit diesem Volumen der Spritze besser gestört werden. Eine Alternative zu einer Spritze ist die Verwendung einer gut abgeflachten P1000-Spitze. - Resuspendieren Sie die Organoide in 500 μL Differenzierungsmedien pro Vertiefung. Achten Sie darauf, die Organoide nicht zu stören und stellen Sie sicher, dass die apikale Seite nicht zugänglich ist. Fahren Sie mit Schritt 3.11 fort.
- Befestigen Sie eine 27 G-Nadel an einer 1-ml-Spritze und resuspen sie das Pellet sechsmal, um eine Störung der Organoide und eine Infektion sowohl an der apikalen als auch an der basolateralen Seite zu ermöglichen. Fahren Sie mit Schritt 3.11 fort.
- Übertragen Sie 500 μL Organoidsuspensionen pro Vertiefung mit einer P1000-Pipette auf eine Zellkulturplatte mit 24 Well und bringen Sie die Platte in die für den Erreger der Wahl erforderliche Sicherheitsstufe.
VORSICHT: Beachten Sie die lokalen Vorschriften und die Standardarbeitsanweisungen des Instituts beim Umgang mit Krankheitserregern unter BSL-2- oder BSL-3-Eindämmung. Führen Sie immer die Nadelzerstörung der Organoide außerhalb des BSL-3 durch, um mögliche Unfälle mit der Nadel zu vermeiden. Lokale Vorschriften können auch die Verwendung von Nadeln im BSL-3 verhindern. - Fügen Sie den in Differenzierungsmedien verdünnten Erreger hinzu, um die für das Experiment erforderliche Vielzahl von Infektionen zu erreichen. Überschreiten Sie nicht ein Gesamtvolumen von 500 μL (um insgesamt 1 ml in der 24-Well-Platte zu haben).
- Lassen Sie die Infektion 2 h (dieses Mal muss an den Erreger der Wahl angepasst werden) bei 37 °C entweder mit einem Wipptisch im Zellkultur-Inkubator oder durch manuelles Schaukeln der Platte alle 15-20 Min.
- Nach der 2-stündigen Inkubation werden die Organoide in einem 1,5-ml-Röhrchen pro Zustand gesammelt und bei 500 x g für 5 min bei 4 °C gedreht.
- Entfernen Sie das erregerhaltige Medium mit einer P1000-Pipette und lagern Sie es auf Eis. Waschen Sie das Organoid-Pellet einmal mit PBS.
- Resuspendieren Sie die Organoide in 50 μL 100% ECM-Lösung (die zuvor auf Eis aufgetaut wurde), platten Sie sie in eine 24-Well-Nicht-Zellkultur-behandelte Platte, die auf 37 ° C vorgewärmt wurde, und inkubieren Sie die 24-Well-Platte bei 37 ° C für 10-15 min, damit die ECM-Lösung polymerisieren kann.
- Nach der Polymerisation 500 μL Differenzierungsmedien bei Raumtemperatur in jede Vertiefung geben und bei 37 °C inkubieren, bis sie für die Einzelzellsequenzierung geerntet werden.
HINWEIS: Wenn die Ernte mehr als 48 h nach der Aussaat erfolgen muss, wechseln Sie das Medium alle 2 Tage, indem Sie das alte Medium entfernen und durch 500 μL frische Differenzierungsmedien ersetzen. Die Erfahrung hat jedoch gezeigt, dass Organoide, da sie unter Differenzierungsmedien gezüchtet werden, nicht viel länger als 48 h überleben werden.
4. Herstellung von Einzelzelllösung und Herstellung von Gelperlen-in-Emulsion (GEMs) unter Bedingungen der Biosicherheitsstufe 3 (BSL-3)
HINWEIS: Die Durchführung der folgenden Schritte erfordert, dass die Einzelzellsequenzierungsausrüstung (Materialtabelle) und eine PCR-Maschine, die 100 μL-Reaktionen verarbeiten kann, in einer BSL-3-Anlage vorhanden sind. Organoide werden wie in Abschnitt 1 beschrieben gezüchtet und je nach Erreger und Eintrittsweg wie in den Abschnitten 2-3 beschrieben infiziert. Zu einem vorbestimmten Zeitpunkt nach der Infektion werden Organoide geerntet. Im Folgenden wird die Methode zur Entnahme von Darmorganoiden beschrieben.
- Vor der Entnahme infizierter Organoide die Einzelzellgelperlen (Table of Materials) von -80 °C entfernen und auf Raumtemperatur erwärmen (mindestens 30 min vorher).
- Zusätzlich wird das RT-Reagenz, das Reduktionsmittel B und das RT-Enzym C (alle bei -20 °C gelagert) auf Raumtemperatur ausgeglichen. Resuspendieren Sie das Template-Schalter-Oligo wie in der Anleitung des Herstellers beschrieben.
HINWEIS: Alle folgenden Schritte müssen in Bezug auf die lokalen Biosicherheitsvorschriften gemäß dem festgelegten Standardarbeitsverfahren für den betrachteten Erreger durchgeführt werden. Als Faustregel für BSL-3-Arbeiten muss sichergestellt werden, dass die Außenseite aller Gefäße (Platten, Röhrchen usw.), die aus einer Zellkulturhaube austreten, ordnungsgemäß desinfiziert werden. Gleiches gilt für die Hände/Handschuhe der Person, die die Experimente durchführt. - Um Infektionen an Organoiden durchzuführen, die in 2D ausgesät wurden (Abschnitt 2), fahren Sie mit Schritt 4.4 fort. Um Infektionen an 3D-Organoiden durchzuführen (Abschnitt 3), fahren Sie mit Abschnitt 4.5 fort.
- Bei Infektionen von 2D-Organoiden die Zellkulturplatte in die Haube bringen und das Differenzierungsmedium mit einer P1000-Pipette entfernen.
- Fügen Sie 250 μL 1x PBS bei Raumtemperatur zu jeder Vertiefung hinzu.
- Entfernen Sie das PBS mit einer P1000-Pipette und geben Sie 250 μL Dissoziationsenzym bei Raumtemperatur (z. B. TrypLE Express) zu jeder Vertiefung einer 48-Well-Platte hinzu. Wechseln Sie die Handschuhe, reinigen Sie die Platte und legen Sie die Platte mit dem Dissoziationsenzym in den 37 °C-Inkubator.
- Beobachten Sie die Zelldissoziation durch Mikroskopie alle 5 Minuten.
HINWEIS: Ungefähr dauert es 15 Minuten, um intestinale Organoide, die in 2D kultiviert wurden, in einzelne Zellen zu dissoziieren. Diese Zeit muss angepasst werden, da sie davon abhängt, wie viele Organoide ursprünglich ausgesät und infiziert wurden, sowie vom Erreger, da einige Krankheitserreger zytopathischer sind und dazu führen, dass Organoide viel schneller dissoziieren als andere. - Nach Bestätigung einer scheinbaren Einzelzellsuspension bringen Sie die Platte zurück in die Zellkulturhaube und stoppen Sie den Aufschluss durch Zugabe von 250 μL DMEM/F12-Medien, die 10% FBS pro Vertiefung einer 48-Well-Platte enthalten.
- Sammeln Sie die Zellen in einem 15 ml konischen Rohr mit einer P1000 Pipette.
- Wechseln Sie die Handschuhe, reinigen Sie das Röhrchen und drehen Sie die Proben bei 500 x g für 5 min bei 4 °C.
- Entfernen Sie vorsichtig das Medien- / Dissoziationsenzym mit einem P1000, um das Zellpellet am Boden zu erhalten. Resuspend einzelne Zellen in einem minimalen Volumen von PBS mit 0,1% BSA. Für Darmorganoide resuspend in 250 μL für jede Vertiefung der 48-Well-Platte.
- Geben Sie die Zellsuspensionen in ein FACS-Röhrchen mit einem Filter, um große Klumpen zu entfernen, und legen Sie das FACS-Röhrchen, das Zellen enthält, auf Eis.
- Fahren Sie mit Schritt 4.6 fort, um mit der Zellzählung und der Vorbereitung des Master-Mixes fortzufahren.
- Bei Infektionen mit 3D-Organoiden entfernen Sie die Platte aus dem Inkubator und legen Sie sie in die Zellkulturhaube.
- Entfernen Sie das Differenzierungsmedium aus jeder Vertiefung der 24-Well-Platte mit einer P1000-Pipette. Fügen Sie 500 μL eiskaltes 1x PBS zu jedem Bohrloch hinzu und inkubieren Sie für 3 min auf Eis.
- Um eine vollständige Unterbrechung der ECM-Lösung zu gewährleisten, verwenden Sie eine P1000-Pipette (eingestellt auf 450 μL). 10-mal nach oben und unten pipettieren, um pbS, ECM-Lösung und Organoide wieder aufzunehmen; die resuspendierten Organoide in ein 15 ml großes konisches Rohr geben und auf Eis legen. Sammeln Sie jeden Infektionszustand in einem eigenen 15 ml konischen Rohr.
HINWEIS: Die Verwendung eines konischen 15-ml-Rohrs ergibt ein besseres, deutlicheres Zellpellet als ein 50-ml-konisches Rohr oder ein 1,5-ml-Rohr. - Wechseln Sie die Handschuhe und entfernen Sie die Tube von der Zellkulturhaube. Reinigen Sie die Außenseite des Röhrchens.
- Die Proben bei 500 x g für 5 min bei 4 °C drehen.
- Bewegen Sie das Röhrchen zurück in die Zellkulturhaube und entfernen Sie PBS mit einer P1000-Pipette, um zu vermeiden, dass das Organoid-Pellet vom Boden des Röhrchens wiederverwendet wird.
- Resuspendieren Sie das Pellet in 1 ml Dissoziationsenzym (z. B. TrypLE Express). Wechseln Sie die Handschuhe, reinigen Sie das Röhrchen und inkubieren Sie die Proben bei 37 °C.
- Bewegen Sie das Röhrchen alle 10 Minuten für 30 Minuten zurück in die Zellkulturhaube und resuspenieren Sie die Organoide, indem Sie mit einer P1000-Pipette 10 Mal auf und ab pipettieren.
HINWEIS: Normalerweise benötigen Darmorganoide etwa 30 Minuten, um eine Einzelzellsuspension zu bilden, wenn sie in 3D infiziert werden. - Um festzustellen, wann Organoide zu einzelnen Zellen dissoziiert werden, nehmen Sie 10 μL der Organoidsuspension mit einer p10-Pipette.
- Legen Sie die Suspension in einen Einweg-Kunststoffzellen-Gegenträger. Versiegeln Sie den Probeneingang mit durchsichtigem Klebeband.
- Wechseln Sie die Handschuhe und reinigen Sie die Außenseite des Zellenzählers. Verwenden Sie ein Hellfeldmikroskop, um festzustellen, ob eine einzelne Zellsuspension hergestellt wird.
- Nach Bestätigung einer Einzelzellsuspension stoppen Sie den Aufschluss durch Zugabe von 1 ml DMEM/F12-Medien mit 10% FBS und Pipette 10 Mal mit einer P1000-Pipette.
- Wechseln Sie die Handschuhe, reinigen Sie das Röhrchen und drehen Sie die Proben bei 500 x g für 5 min bei 4 °C.
- Entfernen Sie das Medien-/Dissoziationsenzym vorsichtig mit einer P1000-Pipette, um zu vermeiden, dass das Zellpellet vom Boden des Röhrchens wiederverwendet wird.
- Resuspendieren Sie die einzelnen Zellen in einem minimalen Volumen von PBS, das 0,1% BSA enthält. Für intestinale Organoide resuspend in 250 μL.
- Geben Sie die Zellsuspensionen in ein FACS-Röhrchen mit einem Filter, um große Klumpen zu entfernen, und legen Sie das FACS-Röhrchen, das Zellen enthält, auf Eis.
- Fahren Sie mit Schritt 4.6 fort, um mit der Zellzählung und der Vorbereitung des Master-Mixes fortzufahren.
- Bestimmen Sie die Anzahl der Zellen pro μL, indem Sie 10 μL der Zellsuspension in eine Einweg-Plastikzellzählkammer geben.
- Verschließen Sie den Probeneingangsanschluss mit durchsichtigem Klebeband, bevor Sie die Probe aus der Zellkulturhaube entnehmen, da die Zellsuspension in diesem Stadium noch infektiös ist.
- Wechseln Sie die Handschuhe, reinigen Sie die Zellzählkammer und zählen Sie die Zellzahl mit einem Hellfeldmikroskop.
- Bereiten Sie in der Zellkulturhaube eine Mastermischung aus RT-Reagenz, Template Switch Oligo, Reducing Agent B und RT-Enzym C in einem 1,5-ml-Röhrchen gemäß den Anweisungen des Herstellers vor, abhängig von der Anzahl der Proben im Experiment. Für jede Probe aliquot 33,4 μL Mastermischung in ein PCR-Röhrchen geben und auf Eis lagern.
- Fügen Sie die Zellen und das Wasser gemäß der Zielzellnummer, wie in den Anweisungen des Herstellers beschrieben, in die Mastermischung ein.
HINWEIS: 50%-60% der Zellen werden normalerweise zurückgewonnen (d.h. beim Laden von 10.000 Zellen auf den Chip werden 5.000-6.000 Zellen für die Analyse verwendet). Laden Sie daher immer 10.000 Zellen ein. Wenn die Zelldichte dies nicht zulässt, zentrifugieren Sie die Zellen und resuspendieren Sie in einem niedrigeren Volumen. Achten Sie darauf, den Chip nicht zu überladen, da dies zu einer Verstopfung des Chips führt. Wenn Zellen nicht richtig dissoziiert werden, besteht außerdem ein erhöhtes Risiko, mehrere Zellen pro Perle zu erhalten, die in der nachgelagerten bioinformatischen Verarbeitung entfernt werden müssen. - Wechseln Sie die Handschuhe, bewegen Sie den Einzelzellen-Controller in die Zellkulturhaube und bereiten Sie den Chip vor, da der Chip nur mit einer Dichtung bedeckt ist, die nicht versiegelt ist.
HINWEIS: Das Gerät muss sich in der Haube befinden, um eine Exposition gegenüber infizierten Zellsuspensionen und potenziellen Aerosolen zu vermeiden. - Fügen Sie den Einzelzellchip zum Chiphalter hinzu und füllen Sie die unbenutzten Bahnen mit 50% Glycerin.
- Fügen Sie die Mastermischung, die Perlen und das Partitionierungsöl gemäß den Anweisungen des Herstellers zu den für Proben verwendeten Bahnen hinzu.
- Decken Sie den Chip mit der Dichtung ab, laden Sie den Chip in den Controller und starten Sie das Programm.
HINWEIS: Es wird empfohlen, nur sechs der acht Fahrspuren zu laden. Probleme mit unsachgemäßen Emulsionen treten häufig in den Bahnen eins und acht auf. Das Unternehmen sagt, dass alle Fahrspuren unabhängig sind, und dies sollte nicht passieren; Wenn möglich, vermeiden Sie jedoch diese beiden Bahnen und lassen Sie zwei Chips laufen, wenn acht Proben benötigt werden. - Entfernen Sie nach Abschluss des Programms den Chip und die Dichtung.
- Verwenden Sie eine Mehrkanalpipette und übertragen Sie 100 μL der Emulsionen in ein sauberes PCR-Röhrchen. Stellen Sie sicher, dass jede Vertiefung eine gleichmäßige weiße Farbe aufweist, die anzeigt, dass eine vollständige Emulsion aufgetreten ist.
- Wechseln Sie die Handschuhe, reinigen Sie die Röhrchen und übertragen Sie das PCR-Röhrchen auf ein PCR-Gerät, das 100-μL-Reaktionen unterstützen kann. Führen Sie das Programm aus: 53 °C für 45 min; 85 °C für 5 Min.
- Nach Fertigstellung lagern Sie die Proben bei 4 °C. An dieser Stelle die Reaktion gemäß den Anweisungen des Herstellers verarbeiten oder bei 4 °C für 3 Tage oder -20 °C für 1 Woche lagern.
- Nach 5 min bei 85 °C werden die meisten behüllten Viren inaktiviert, die cDNA gemäß dem normalen Betriebsverfahren aus dem BSL-3 entfernt und die Bibliotheksvorbereitungen in einem BSL-1-Labor durchgeführt.
HINWEIS: Dieses Experiment muss als drei biologische Replikate (z. B. an drei verschiedenen Tagen) durchgeführt werden, da das Ausmaß der Differenzierung jedes Zelltyps zwischen jedem Experiment leicht variieren kann. Die resultierenden Sequenzierungsbibliotheken könnten in einem Sequenzierungslauf unterschiedlich indiziert und sequenziert werden.
Ergebnisse
Herstellung von Organoiden für die Einzelzellsequenzierung
Einzelzellsequenzierungsergebnisse hängen stark von der Verwendung von Zellen guter Qualität ab. Um sicherzustellen, dass Organoide von guter Qualität sind, sollten sie ordnungsgemäß gewartet und täglich beobachtet werden, um festzustellen, wann sie bereit sind, gespalten zu werden (Abbildung 2). Der Zeitpunkt der Spaltung von Organoiden ist spenderabhängig; Einige Spender wachsen schneller und müssen alle 5 Tage aufgeteilt werden, während andere langsamer sind und alle 10 Tage aufgeteilt werden müssen. Im Durchschnitt werden Organoide einmal pro Woche gespalten, wenn die Zentren dunkel werden (Abbildung 2B). Wenn Organoide zu groß werden und zu viele tote Zellen im Zentrum ansammeln, stirbt das Organoid ab.
Organoide werden in einem Medium gehalten, das hohe Mengen an Wnt3A enthält. Dies unterstützt die Stammzellnische und fördert das weitere Wachstum und die Vermehrung der Organoide. Unter diesen Wachstumsbedingungen enthalten die Organoide hohe Mengen an Stammzellen und transitverstärkenden Zellen und eine geringere Menge an differenzierten Zellpopulationen wie reife Enterozyten, Kelchzellen und enteroendokrine Zellen. Um jedoch die zelluläre Komplexität im menschlichen Darm nachzuahmen, ist es wichtig, die Zelldifferenzierung voranzutreiben und mehr dieser Zellen zu produzieren. Dies wird erreicht, indem die Medienbedingungen geändert und Wnt3A entfernt und R-Spondin und Noggin reduziert werden (Tabelle 1). Normalerweise erfordert die zelluläre Differenzierung zu Enterozyten, Kelchzellen und enteroendokrinen Zellen 4 Tage Differenzierungsmedien (Abbildung 3). Es ist der Schlüssel zu einer guten Differenzierung; Andernfalls wird die Bewertung des Pathogentropismus und der zelltypspezifischen Reaktionen schwierig.
Bestätigung der Inaktivierung des BSL-3-Erregers
Die vollständige Inaktivierung muss mit dem Erreger der Wahl bestätigt und validiert werden, dass es sicher ist, die cDNA aus dem BSL-3 zu entfernen. Für SARS-CoV-2 wurde die vollständige Inaktivierung des Virus validiert, indem 100 μL SARS-CoV-2 entnommen und in einer PCR-Maschine für 5 min bei 85 °C inkubiert wurden. Das Virus wurde dann wieder zu naiven Vero-Zellen hinzugefügt, und die Virusinfektion wurde mit einem nicht wärmebehandelten Virus durch Immunfluoreszenz- und Plaque-Assays nach 24 h, 48 h und 72 h nach der Infektion verglichen, um sicherzustellen, dass alle Partikel nicht mehr infektiös waren. Diese Ergebnisse wurden an die örtliche Zulassungsbehörde geschickt, und nach ihrer Genehmigung wurde das Einzelzellexperiment und die cDNA-Verarbeitung durchgeführt.
Ergebnisse der Einzelzellsequenzierung
Um zu bewerten, wie SARS-CoV-2 menschliche Dickdarm- und Ileum-Organoide infiziert, wurde eine Einzelzellsequenzierung durchgeführt. Organoide wurden wie oben beschrieben hergestellt und in einem 2D-Format infiziert, um eine apikale Infektion durch SARS-CoV-2 zu ermöglichen. Infizierte Zellen wurden 12 h und 24 h nach der Infektion geerntet und für die Einzelzellsequenzierung wie oben beschrieben verarbeitet. Die Analyse der Einzelzellsequenzierungsdaten ermöglichte es uns festzustellen, dass nur eine Subpopulation (unreifer Enterozyt 2) menschlicher Darmepithelzellen die Infektion von SARS-CoV-2 unterstützte (Abbildung 4). Da nicht alle Zellen in einer Population infiziert waren, wurden sowohl infizierte Zellen als auch nicht infizierte Umstehende analysiert (Abbildung 5). Diese Ergebnisse zeigten, dass SARS-CoV-2 eine entzündungsfördernde Signalkaskade in infizierten Zellen induzierte, während nicht infizierte Umstehende eine Interferon-vermittelte Immunantwort zeigten. Zusätzlich zeigte scRNA-Seq, dass infizierte Zellen aufgrund einer virusvermittelten Blockade des Signalwegs nicht in der Lage waren, Interferone wahrzunehmen (Abbildung 5). Es war nicht möglich, diese Informationen bei der Verwendung von Bulk-RNA-Sequenzierung zu erhalten.

Abbildung 1: Schematische Darstellung der drei verschiedenen Methoden zur Vorbereitung menschlicher Darmorganoide auf eine Infektion mit enterischen Krankheitserregern. Eine apikale Infektion kann durch die Aussaat von Darmorganoiden in 2D erreicht werden. Eine apikale und basolaterale Infektion kann durch Störung des 3D-Organoids durchgeführt werden. Schließlich kann eine basolaterale Infektion durchgeführt werden, indem intakte 3D-Darmorganoide infiziert werden. Jede dieser Methoden kann verwendet werden, um Proben für die Einzelzellsequenzierung zu generieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
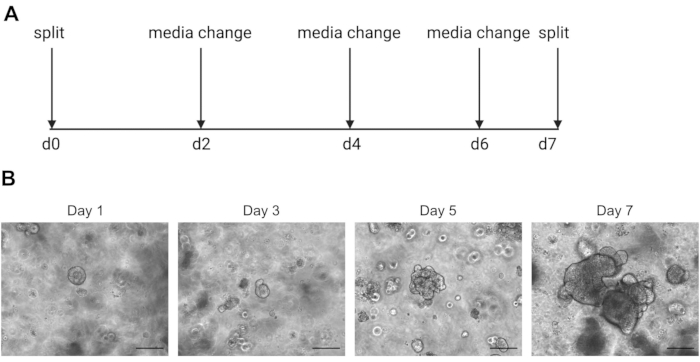
Abbildung 2: Schematische Organoid-Erhaltung und repräsentative Hellfeldbilder. (A) Schematische Darstellung zur Erhaltung und Weitergabe menschlicher Darmorganoide. (B). Repräsentative Hellfeldbilder der Tage 1, 3, 5 und 7 nach der Aufspaltung. Am Tag 7 werden die Organoide aufgrund der Ansammlung abgestorbener Zellen groß und dunkel und sind bereit, gespalten zu werden. Der Maßstabsbalken zeigt 25 μm an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
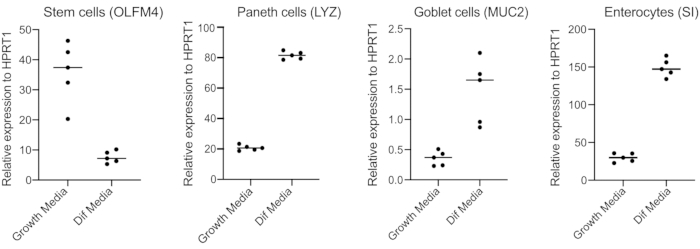
Abbildung 3: Repräsentative qPCR menschlicher Darmorganoide 4 Tage nach der Umstellung auf Differenzierungsmedien. Darmorganoide wurden in Wachstumsmedien gehalten oder für 4 Tage auf Differenzierungsmedien umgestellt. RNA wurde geerntet und qPCR wurde für Marker von Stammzellen (OLFM4), Paneth-Zellen (LYZ), Kelchzellen (MUC2) und Enterozyten (SI) durchgeführt. N = 5. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
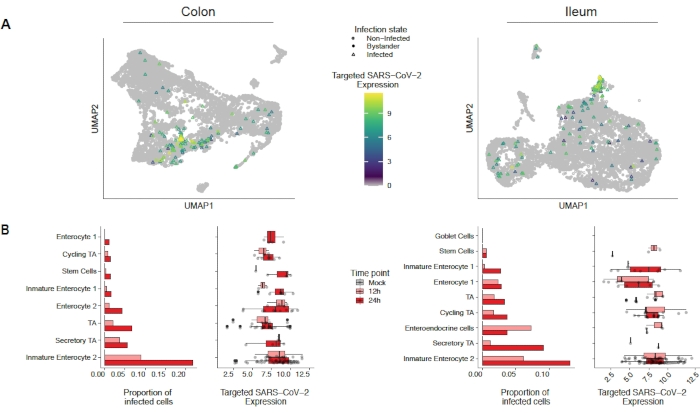
Abbildung 4: Identifizierung der mit SARS-CoV-2 infizierten Zellpopulation. Menschliche Organoide aus Dickdarm und Ileum wurden mit SARS-CoV-2 infiziert. Nach 12 und 24 h nach der Infektion wurden Zellen geerntet und einer Einzelzell-RNA-Sequenzierung unterzogen, um zu identifizieren, welche Zellpopulationen eine SARS-CoV-2-Infektion unterstützten. Es wurde festgestellt, dass die Virusinfektion im Laufe der Zeit zunimmt und hauptsächlich unreife Enterozyten 2 infiziert. Diese Zahl wurde von Triana et al.19modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
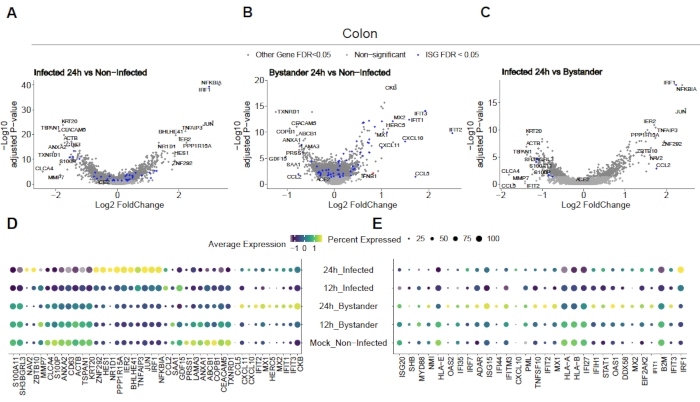
Abbildung 5: Bestimmung der intrinsischen angeborenen Immunantwort. Menschliche Darmorganoide wurden mit SARS-CoV-2 infiziert. Nach 12 und 24 h nach der Infektion wurden Zellen geerntet und einer Einzelzell-RNA-Sequenzierung unterzogen, um die intrinsische angeborene Immunantwort sowohl in viral infizierten als auch in nicht infizierten Nebenstehendenzellen zu bestimmen. SARS-CoV-2-infizierte Zellen zeigten eine starke entzündungsfördernde Reaktion, während nicht infizierte Umstehende eine Interferon-vermittelte Reaktion zeigten. Abbildung modifiziert von Triana et al.19. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Wachstumsmedien | |
| Verbindung | Endkonzentration |
| Anzeige DMEM/F12 | GlutaMAX (1X) |
| +GlutaMAX | HEPES 1 mM |
| +HEPES | Stift 10 U/ml |
| +P/S | Strep 10 μg/ml |
| L-WRN | 50 % nach Volumen |
| B27 | 01:50 |
| N-Acetyl-Cystein | 1 mM |
| EGF | 50 ng/ml |
| A83-01 | 500 nM |
| IGF-1 | 100 ng/ml |
| FGF-Basis | 50 ng/ml |
| Gastrin | 10 mM |
| Differenzierungsmedien | |
| Verbindung | Endkonzentration |
| Anzeige DMEM/F12 | GlutaMAX (1x) |
| +GlutaMAX | HEPES 1 mM |
| +HEPES | Stift 10 U/ml |
| +P/S | Strep 10 μg/ml |
| B27 | 01:50 |
| N-Acetyl-Cystein | 1 mM |
| R-Spondin | 5 % nach Volumen |
| Birne | 50 ng/ml |
| EGF | 50 ng/ml |
| Gastrin | 10 mM |
| A83-01 | 500 nM |
Tabelle 1: Medienzusammensetzung für Wachstums- und Differenzierungsmedien.
Diskussion
Enterische Krankheitserreger beginnen ihren Lebenszyklus am häufigsten, indem sie Darmepithelzellen von ihrer apikalen Seite mit Blick auf das Darmlumen infizieren. Während Organoide als gutes Modell zur Reproduktion der zellulären Komplexität und Organisation des Darmepithels anerkannt sind, machen ihre Organisation als dreidimensionale, geschlossene Strukturen ihre apikale Membran für den Erreger unzugänglich. Dieses Protokoll beschrieb Methoden zur Infektion von Darmorganoiden von ihrer apikalen Seite, ihrer basolateralen Seite oder beidem mit BLS-3-Erregern. Diese Protokolle können leicht angepasst werden, um jeden enterischen Erreger unter BSL-2- oder BLS-3-Eindämmung oder einem anderen Organoidmodell zu untersuchen, indem einige kritische Schritte befolgt werden, die unten hervorgehoben werden. Das oben beschriebene Verfahren dient zur Isolierung und Herstellung von Einzelzelltröpfchen gemäß den Vorschriften in Deutschland. Als Haftungsausschluss beschreibt dieses Protokoll nicht die Maßnahmen zur Handhabung der biologischen Sicherheit (Standardarbeitsanweisungen), die während der Arbeit unter BSL-3-Bedingungen ergriffen werden müssen. Es ist auch wichtig, darauf zu bestehen, dass die Vorschriften in anderen Ländern variieren können und dass die lokalen Behörden kontaktiert werden müssen, um sicherzustellen, dass alle lokalen Vorschriften eingehalten werden.
Einer der kritischen Schritte bei der Aussaat von Organoiden in zwei Dimensionen für apikale Infektionen ist die Kontrolle, dass sich Zellen ähnlich differenzieren, wenn sie als klassische dreidimensionale Organoide gezüchtet werden. Je nach enterischem Erreger könnte der Tropismus auf sehr seltene Zellen oder auf Zellen beschränkt sein, die hochdifferenziert werden müssen. In diesem Fall könnte die Verwendung eines zweidimensionalen Organoids, das sich nicht vollständig differenziert hat, zu dem Missverständnis führen, dass dieser enterische Erreger keine Darmorganoide infizieren kann. Es wird empfohlen, wenn möglich, Infektionen mit den drei Konfigurationen dieses Protokolls durchzuführen: 2D-Organoid nur für apikale Infektionen (Abschnitt 2), aufgebrochene 3D-Organoide für apikale und basolaterale Infektionen (Abschnitt 3) und vollständige 3D-Organoide nur für basolaterale Infektionen (Abschnitt 3). Dieser Ansatz wird helfen, den Eintrittsweg des Erregers (apikal vs. basolateral) zu erkennen und zu kontrollieren, dass ein ähnlicher Differenzierungsgrad erreicht wurde. Eine Alternative für die apikale 2D-Infektion ist die Mikroinjektion, bei der ein 3D-Organoid verwendet wird, der Erreger jedoch direkt in die apikale Seite abgegeben wird (siehe Bartfeld et al.27 für Details). Diese Methode erfordert einen erfahrenen Injektor, um sicherzustellen, dass der Erreger richtig platziert ist und die Organoide intakt bleiben. Die Mikroinjektion wird häufig in der BSL-2-Eindämmung verwendet und ist möglicherweise nicht für die BSL-3-Eindämmung geeignet.
Eine weitere wichtige Überlegung bei der Durchführung von Infektionsexperimenten in 2D-ausgesäten Organoiden ist die endgültige Zelldichte. Wie in Schritt 2.3 erwähnt, werden 100-150 Organoide in einer Vertiefung einer 48-Well-Platte oder einer Vertiefung einer 8-Well-Glasbodenkammerschlitten ausgesät. Abhängig von der Organoidlinie und von der Person, die mit den Organoiden umgeht, kann die Größe dieser Organoide signifikant unterschiedlich sein. Dies könnte zu sehr unterschiedlichen Zelldichten im 48-Well-Platten- oder 8-Well-Glasbodenkammerobjektträger führen. Abhängig vom enterischen Virus bevorzugen einige Viren spärlichere Zellen, während andere auch in der Lage sind, konfluente Zellen zu infizieren. Der molekulare Ursprung solcher Unterschiede in der Infektiosität für verschiedene Zellkonfluenzen ist nicht klar; Daher sollten Pilotexperimente durchgeführt werden, die darauf abzielen, die beste Zelldichte für den enterischen Erreger der Wahl zu finden, bevor eine weitere nachgelagerte Charakterisierung durchgeführt wird.
Häufig wird die FACS-Sortierung vor der Durchführung der Einzelzell-Tröpfchenemulsion durchgeführt. Dieser Schritt wird oft verwendet, um tote von lebenden Zellen und einzelne Zellen von Dubletten zu trennen. Bei der Arbeit mit BSL-3-Krankheitserregern ist es erforderlich, dass die Anlage mit einem geeigneten FACS-Sortierer ausgestattet ist, was nicht oft der Fall ist. Darüber hinaus haben nicht alle Zellen in einem Organoid die gleiche Größe, und es ist oft schwierig, zwischen einer Dublette oder einer größeren Zelle zu unterscheiden, wodurch das Risiko einer negativen Selektion gegen einen bestimmten Zelltyp besteht. Auch wird im Feld noch diskutiert, ob die Zeit, die für die Sortierung zwischen 5.000 und 10.000 pro Probe benötigt wird, zu einer signifikanten Veränderung des Transkriptprofils der einzelnen Zellen führen könnte. Während Zellfixierungsmethoden beschrieben wurden, die mit der Einzelzellsequenzierung kompatibel sind (z. B. Methanol und RNAassist), wurde beobachtet, dass dies zu einer Abnahme der Qualität der Sequenzierung führt18. Schließlich wird vermutet, dass auch das Sortieren von Zellen mittels Zelltodmarkern zu einem Bias führen kann. Angesichts der gerichteten Proliferation und Differenzierung der Zellen durch die Krypta-Zotten-Achse befinden sich die differenziertesten Zellen, die abgestoßen und freigesetzt werden, an der Spitze der Zotten. Diese Zellen sind oft positiv für verschiedene Marker von Zelltodwegen(z. B. Apoptose, Nekrose und Nekroptose); Wenn man sich jedoch die Rotavirus-Infektion des Mausdarms ansieht, ist die Spitze der Zotten das am meisten infizierte Gebiet28. Daher würde das Herausfiltern von Zellen, die positiv für Todesmarker aussehen könnten, zu einer negativen Selektion der infizierten Zellen führen, die die physiologische Infektion darstellen können. Derzeit gibt es keine gute Lösung für die Sortierung und Fixierung von Organoiden vor der Einzelzellsequenzierung. Die Verwendung lebender, unsortierter Zellen wird empfohlen, da weitere Studien erforderlich sind, um geeignete alternative Protokolle zu finden.
Die Einzelzellsequenzierung hat die Art und Weise, wie zelluläre Reaktionen ausgewertet werden können, revolutioniert. Diese Technik ermöglicht die Identifizierung von zelllinienspezifischen Reaktionen sowohl unter basalen Bedingungen als auch unter Pathogeninfektionen. Diese Methode hat Türen in vielen Bereichen geöffnet, die zuvor durch Massenauslesungen eingeschränkt waren. Während diese Methode sehr mächtig ist, hat sie ihre Grenzen. Eine wesentliche Einschränkung ist die umfangreiche bioinformatische Analyse, die nach der Sequenzierung erforderlich ist. Dies ist besonders wichtig bei der Analyse von Geweben und der Zuweisung von Zelltypen, bei denen derzeit keine Anmerkung vorhanden ist. Ein qualifizierter Bioinformatiker ist erforderlich, um alle Einzelzellstudien zu unterstützen.
Dieses Protokoll beschreibt, wie man menschliche Darmorganoide aussagt und behandelt, sie mit enterischen Krankheitserregern infiziert und scRNAseq durchführt. Die Anpassung dieses Ansatzes an andere Organe ist nun möglich, da für die meisten Organe Organoidmodellsysteme entwickelt wurden. Lungen- und Leberorganoide sind im Vergleich zu Darmorganoiden ähnlich organisiert und könnten daher mit einem analogen Ansatz auf diese Organoide übertragen werden. Die kritische Kontrolle besteht darin, zu validieren, dass diese Organoide, wenn sie in zwei Dimensionen gezüchtet oder aufgebrochen werden, eine ähnliche Differenzierung erreichen wie ihre 3D-Organoid-Gegenstücke. Die spezifischen Merkmale und Gene, die einen differenzierten Status definieren, sind für jedes Organmodell spezifisch. Andere Organoidmodelle wie Nieren- und Gefäßorganoide, große dichte Strukturen, benötigen zusätzliche Methoden, um diese Strukturen seriell in einzelne Zellen zu dissoziieren.
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen.
Danksagungen
Megan Stanifer und Steeve Boulant wurden durch Forschungsstipendien der Deutschen Forschungsgemeinschaft (DFG) gefördert: (Projektnummer 240245660, 278001972, 415089553 und 272983813 an Steeve Boulant und 416072091 an Megan Stanifer), baden-württemberg und das Bundesministerium für Bildung und Forschung BMBF 01KI20239B an MS und 01KI20198A und (NUM-COVID 19, Organo-Strat 01KX2021) an SB. Schematische wurden in BioRender erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Recombinant mouse noggin | Peprotech | Cat#250-38 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | Cat# G9145 | |
| 0.05% Trypsin-EDTA | Thermo Fischer Scientific | Cat#25300054 | |
| 24-well non-cell culture treated plate | Corning | Cat#3738 | |
| 8-well glass bottom chamber slide | iBIDI | Cart#80827 | |
| A83-01 | Tocris | Cat#2939 | |
| Advanced DMEM/F12 | Thermo Fischer Scientific | Cat# 12634010 | |
| B27 | Thermo Fischer Scientific | Cat#17504-044 | |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | Cat#1000202 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | Cat #1000127 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Single Cell 3′ Kit v3.1 | 10X Genomics | Cat#1000268 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Collagen from human placenta | Sigma Aldrich | Cat#C5533-5MG | |
| CYP34A forward | Eurofins | GATGGCTCTCATCCCAGACTT | Primers used to check for differentiation |
| CYP3A4 reverse | Eurofins | AGTCCATGTGAATGGGTTCC | Primers used to check for differentiation |
| DMEM/F12 | Thermo Fischer Scientific | Cat#11320074 | |
| EDTA | Sigma Aldrich | Car#E9884 | |
| Fast Read 102 counting slides | Biosigma | Cat# BVS100 | |
| Fetal Bovein Serum (FBS) | Capricorn | Cat#FBS-12A | |
| GlutaMAX | Thermo Fischer Scientific | Cat# 35050061 | |
| HEPES | Thermo Fischer Scientific | Cat3 15630080 | |
| L-WRN cells | ATCC | CRL-3276 | This cell line is used to make the conditioned media containg Wnt 3A, R-Spondin and Noggin. The protocol for the production of the conditioned media can be found on the manufacterures site. |
| MatriGel. GFR, LDEV free | Corning | Cat#354230 | |
| MUC-2 forward | Eurofins | TGTAGGCATCGCTCTTCTCA | Primers used to check for differentiation |
| MUC-2 reverse | Eurofins | GACACCATCTACCTCACCCG | Primers used to check for differentiation |
| N-acetyl-cysteine | Sigma Aldrich | Cat# A9165 | |
| OLMF4 forward | Eurofins | ACCTTTCCCGTGGACAGAGT | Primers used to check for differentiation |
| OLMF4 reverse | Eurofins | TGGACATATTCCCTCACTTTGGA | Primers used to check for differentiation |
| Penicillin/Streptomycin | Thermo Fischer Scientific | Cat#15140122 | |
| Recombinant human FGF basic | Peprotech | Cat#100-18B | |
| Recombinant human IGF-1 | BioLegend | Cat#590904 | |
| Recombinant mouse EGF | Thermo Fischer Scientific | Cat# PMG8043 | |
| SI forward | Eurofins | AATCCTTTTGGCATCCAGATT | Primers used to check for differentiation |
| SI reverse | Eurofins | GCAGCCAAGAATCCCAAT | Primers used to check for differentiation |
| TrypLE Express | Thermo Fischer Scientific | Cat#12605036 | |
| Y-27632 | Caymann Chemicals | Cat#10005583 |
Referenzen
- Kretzschmar, K., Clevers, H. Organoids: Modeling development and the stem cell niche in a dish. Developmental Cell. 38, 590-600 (2016).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141, 1762-1772 (2011).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Tekes, G., Ehmann, R., Boulant, S., Stanifer, M. L. Development of feline ileum- and colon-derived organoids and their potential use to support feline coronavirus infection. Cells. 9, (2020).
- Derricott, H., et al. Developing a 3D intestinal epithelium model for livestock species. Cell and Tissue Research. 375, 409-424 (2019).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nature Medicine. 26, 1077-1083 (2020).
- Sato, T., Clevers, H. Growing self-organizing mini-guts from a single intestinal stem cell: mechanism and applications. Science. 340, 1190-1194 (2013).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470, 105-109 (2011).
- de Graaf, M., van Beek, J., Koopmans, M. P. Human norovirus transmission and evolution in a changing world. Nature Reviews Microbiology. 14, 421-433 (2016).
- Leshem, E., et al. Real-world effectiveness of pentavalent rotavirus vaccine among bedouin and jewish children in southern Israel. Clinical Infectious Diseases : An Official Publication of the Infectious Diseases Society Of America. 62, 155-160 (2016).
- Karst, S. M. The influence of commensal bacteria on infection with enteric viruses. Nature Reviews Microbiology. 14, 197-204 (2016).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science. 353, 1387-1393 (2016).
- de Wit, E., van Doremalen, N., Falzarano, D., Munster, V. J. SARS and MERS: recent insights into emerging coronaviruses. Nature Reviews Microbiology. 14, 523-534 (2016).
- Scaldaferri, F., et al. The thrilling journey of SARS-CoV-2 into the intestine: From pathogenesis to future clinical implications. Inflammatory Bowel Diseases. 26, 1306-1314 (2020).
- Kipkorir, V., Cheruiyot, I., Ngure, B., Misiani, M., Munguti, J. Prolonged SARS-CoV-2 RNA detection in anal/rectal swabs and stool specimens in COVID-19 patients after negative conversion in nasopharyngeal RT-PCR test. Journal of Medical Virology. 92, 2328-2331 (2020).
- Lamers, M. M., et al. SARS-CoV-2 productively infects human gut enterocytes. Science. 369, 50-54 (2020).
- Stanifer, M. L., et al. Critical role of type III interferon in controlling SARS-CoV-2 Infection in human intestinal epithelial cells. Cell Reports. 32, 107863 (2020).
- Triana, S., et al. Single-cell transcriptomics reveals immune response of intestinal cell types to viral infection. bioRxiv. , (2020).
- Triana, S., et al. Single-cell analyses reveal SARS-CoV-2 interference with intrinsic immune response in the human gut. Molecular Systems Biology. 17, 10232 (2021).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50, 1-14 (2018).
- García-Rodríguez, I., Sridhar, A., Pajkrt, D., Wolthers, K. C. Put some guts into it: Intestinal organoid models to study viral infection. Viruses. 12, 1288 (2020).
- Stanifer, M. L., et al. Asymmetric distribution of TLR3 leads to a polarized immune response in human intestinal epithelial cells. Nature Microbiology. 5, 181-191 (2020).
- Lees, E. A., et al. Using human induced pluripotent stem cell-derived intestinal organoids to study and modify epithelial cell protection against salmonella and other pathogens. Journal of Visualized Experiments: JoVE. (147), e59478 (2019).
- Mahe, M. M., Sundaram, N., Watson, C. L., Shroyer, N. F., Helmrath, M. A. Establishment of human epithelial enteroids and colonoids from whole tissue and biopsy. Journal of Visualized Experiments: JoVE. (97), e52483 (2015).
- Fujii, M., et al. Human Intestinal organoids maintain self-renewal capacity and cellular diversity in niche-inspired culture condition. Cell Stem Cell. 23 (6), 787-793 (2018).
- Bartfeld, S., Clevers, H. Organoids as model for infectious diseases: Culture of human and murine stomach organoids and microinjection of Helicobacter Pylori. Journal of Visualized Experiments: JoVE. (105), e53359 (2015).
- Hernandez, P. P., et al. Interferon-lambda and interleukin 22 act synergistically for the induction of interferon-stimulated genes and control of rotavirus infection. Nature Immunology. 16, 698-707 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten