Method Article
Adaptação de organoides gastrointestinais para infecção por patógenos e sequenciamento de células únicas sob condições de nível de biossegurança 3 (BSL-3)
Neste Artigo
Resumo
Este protocolo descreve como infectar organoides intestinais humanos de seu lado apical ou basolateral para caracterizar interações hospedeiro/patógeno no nível de célula única usando a tecnologia de sequenciamento de RNA unicelular (scRNAseq).
Resumo
Organoides intestinais humanos constituem o melhor modelo celular para estudar infecções por patógenos do trato gastrointestinal. Esses organoides podem ser derivados de todas as seções do trato GI (gástrico, jejunum, duodeno, óleo, cólon, reto) e, após diferenciação, contêm a maioria dos tipos celulares que são naturalmente encontrados em cada seção individual. Por exemplo, organoides intestinais contêm enterócitos absorventes de nutrientes, células secretas (Cálice, Paneth e enteroendocrino), células-tronco, bem como todos os intermediários de diferenciação específicos da linhagem (por exemplo, tipos de células precoces ou imaturas). A maior vantagem no uso de organoides derivados do trato gastrointestinal para estudar doenças infecciosas é a possibilidade de identificar precisamente qual tipo de célula é alvo do patógeno entérico e abordar se as diferentes seções do trato gastrointestinal e seus tipos de células específicas respondem da mesma forma aos desafios do patógeno. Ao longo dos últimos anos, modelos gastrointestinais, bem como organoides de outros tecidos, têm sido empregados para estudar o tropismo viral e os mecanismos da patogênese. No entanto, utilizar todas as vantagens do uso de organoides ao empregar vírus altamente patogênicos representa um desafio técnico e requer rigorosas considerações de biossegurança. Além disso, como os organoides são frequentemente cultivados em três dimensões, o lado basolateral das células está voltado para o lado externo do organoide enquanto seu lado apical está voltado para o interior (lúmen) dos organoides. Esta organização representa um desafio para os patógenos entéricos, pois muitas infecções entéricas iniciam a partir do lado apical/luminal das células após a ingestão. O manuscrito a seguir fornecerá um protocolo abrangente para preparar organoides intestinais humanos para infecção com patógenos entéricos, considerando o lado da infecção (apical vs. basolateral) para realizar sequenciamento de RNA unicelular para caracterizar interações de hospedeiro/patógeno específicas do tipo celular. Este método detalha a preparação dos organoides, bem como as considerações necessárias para realizar este trabalho sob condições de contenção nível 3 (BSL-3).
Introdução
Estudar o tropismo específico do tipo celular e a resposta imune específica do tipo celular aos vírus entéricos humanos tem sido historicamente desafiador devido à falta de modelos celulares humanos primários. Essa limitação foi agora parcialmente erradicada com o desenvolvimento de organoides1. No caso do trato gastrointestinal, foram desenvolvidos modelos organoides gástricos e intestinais para humanos e várias outras espécies (porexemplo,murina, bovina, felina, morcego)2,3,4,5,6. Organoides intestinais reproduzem a arquitetura estrutural do epitélio intestinal humano e contêm estruturas cripta e villi, linhagens intestinais funcionais e até têm sido usadas para identificar linhagens celulares até então desconhecidas. Duas abordagens diferentes podem ser usadas para cultivar organoides intestinais. Primeiro, as células de hastes intestinais contendo criptas são isoladas de ressecções teciduais ou biópsias e cultivadas sob condições específicas de cultura (por exemplo, Wnt3A, R-spondin, Noggin e EGF) para expandir e, em seguida, diferenciar as células-tronco para a maioria das linhagens celulares intestinais (porexemplo,, enterócitos, células panételes, células de Cálice, células enteroendrinas)7. Este método permite o isolamento de organoides de todas as seções do trato gastrointestinal(porexemplo, estômago, duodeno, jejunum, íleo e cólon). O segundo método baseia-se em células-tronco pluripotentes ou embrionárias induzidas pelo homem, que são então diferenciadas em um processo stepwise em células epiteliais intestinais8. Esses organoides induzidos à base de células-tronco são frequentemente descritos como sendo mais embrionários na natureza em comparação com organoides derivados do paciente. Embora todos esses modelos organoides tenham sido fundamentais para desvendar as pistas de desenvolvimento necessárias para formar o trato intestinal, seu uso em pesquisas de doenças infecciosas ainda está em sua infância.
O vírus entérico é um termo amplo que abrange todos os vírus, que infectam através do trato gastrointestinal, como picornavírus(porexemplo, EV-71), reovírus (por exemplo, rotavírus) e calicivírus (por exemplo, norovírus)9. Os vírus entéricos iniciam seu ciclo de vida infeccioso através da ingestão de alimentos e água contaminados, o que deixa as pessoas em países em desenvolvimento em alto risco devido à descarga de resíduos não tratados no meio ambiente e à falta de cuidados médicos após o início da infecção10. Dependendo do tipo de patógeno, a infecção pode levar à gastroenterite, vômitos e/ou diarreia aquática devido ao vazamento do revestimento intestinal. Os norovírus humanos são um patógeno entérico altamente prevalente e altamente infeccioso, que leva a mais de 600 milhões de infecções e 15 milhões de internações em todo o mundo11. Os organoides têm sido fundamentais para a pesquisa do norovírus, pois apoiam a infecção e a replicação do norovírus humano, que antes era incapaz de ser cultivado nos modelos de cultura celular padrão12.
Nas últimas duas décadas, os coronavírus emergiram como patógenos humanos-chave13. Esta família inclui os MERS altamente patogênicos, SARS-CoV-1 e SARS-CoV-2, que requerem restrições rigorosas de nível de segurança ao realizar pesquisas sobre esses vírus. Curiosamente, embora todos esses três patógenos sejam reconhecidos principalmente por seus sintomas respiratórios induzidos e angústia, agora é evidente que esses vírus não infectam apenas o trato respiratório, mas também outros órgãos. Uma patologia importante induzida em pacientes infectados pelo SARS-CoV-2 além do problema respiratório é a presença de sintomas gastrointestinais14. Uma fração de pacientes infectados pelo SARS-CoV-2 apresenta tais sintomas, variando de diarreia muito leve a grave. Além disso, genomas SARS-CoV-2 podem ser detectados em fezes e biópsias do trato gastrointestinal de pacientes infectados15. É importante ressaltar que a presença de sintomas gastrointestinais não se limita ao SARS-CoV-2, pois também foram observados em pacientes infectados pelo MERS e SARS-CoV-1. Para entender como o SARS-CoV-2 induz a angústia gastrointestinal e identifica precisamente o tropismo do SARS-CoV-2 no trato gastrointestinal, os organoides intestinais humanos têm sido uma ferramenta-chave e agora são explorados para desvendar respostas específicas do tipo celular a este patógeno16,17.
O perfil transcricional de uma população celular (sequenciamento de RNA em massa) tem sido uma prática padrão ao avaliar infecções por patógenos tanto de linhas celulares imortalizadas quanto com organoides. Embora isso nos permita determinar mudanças globais em resposta a patógenos (por exemplo, a regulação das citocinas), o RNAseq em massa não nos permite determinar por que células específicas em uma população são mais propensas à infecção do que outras. O sequenciamento de RNA unicelular (scRNAseq) tornou-se uma ferramenta poderosa para desvendar programas transcricionais específicos da linhagem celular e pode ser usado para determinar como esses programas suportam ou reprimem a infecção por vírus18,19. A primeira descrição do scRNAseq foi em 2009 e foi usada para avaliar os perfis de transcrição das diferentes células encontradas em um blastomerede camundongos 20. Essas tecnologias foram expandidas e podem ser implementadas através de diversas plataformas diferentes. As versões iniciais desta tecnologia aplicaram o classificador celular ativado pela fluorescência (FACS) para separar células individuais para sequenciamento, que muitas vezes era limitado a placas de 96 ou 384 poços, dando assim 300 células individuais para analisar por amostra21. Esses métodos foram agora avançados pelas plataformas de sequenciamento unicelular, que usam um dispositivo microfluido para encapsular células únicas em gotículas individuais com código de barras contendo contas. Esta tecnologia permite que até 10.000 células sejam capturadas por condição amostral.
A combinação da tecnologia organoides com a scRNAseq nos permite estudar como os patógenos entéricos impactam o trato gastrointestinal de uma maneira específica do tipo celular. No entanto, várias considerações técnicas e de biossegurança precisam ser tomadas. Em primeiro lugar, os métodos de cultura organoides clássicos (3D) organoides, embutidos em uma matriz extracelular (ECM)) expõem o lado basolateral das células epiteliais para o exterior do organoide. À medida que os patógenos entéricos iniciam sua infecção através da ingestão de alimentos/água contaminados, a infecção mais frequentemente inicia a partir do lado apical das células, que não é acessível nesses organoides intestinais 3D. Portanto, os organoides precisam estar preparados para tornar o lado apical acessível para infecção por patógenos, seja através da semeadura 2D, expondo diretamente o lado apical das células, ou através da microinjeção22,23. Em segundo lugar, para realizar scRNAseq de amostras biológicas infectadas, é importante considerar sua natureza infecciosa. Embora métodos para corrigir células e inativar patógenos antes do isolamento unicelular para rnaseq subsequente tenham sido propostos, esses métodos muitas vezes levam a uma diminuição na qualidade de sequenciamento18. O protocolo abaixo descreverá várias abordagens para infectar organoides intestinais com vírus entéricos considerando o lado da infecção (infecção apical vs. basolateral)(Figura 1). Além disso, o protocolo incluirá um fluxo de trabalho para dissociar e isolar células únicas de organoides infectados com vírus altamente patogênicos para scRNAseq. O protocolo destacará as principais etapas que precisam ser implementadas ao trabalhar sob condições de contenção de nível 3 (BSL-3) de biossegurança para evitar a geração de aerossóis e a contaminação potencial.
Protocolo
O tecido humano foi recebido a partir de ressecção de cólon ou biópsias de ileum do Hospital Universitário Heidelberg para o seguinte protocolo. Este estudo foi realizado sob as recomendações do Hospital Universitário Heidelberg com consentimento por escrito informado de todos os sujeitos de acordo com a Declaração de Helsinque. Todas as amostras foram recebidas e mantidas de forma anônima. O protocolo foi aprovado pela Comissão de Ética do Hospital Universitário Heidelberg sob o protocolo S-443/2017.
1. Manutenção e passagem de organoides intestinais e colonos
ATENÇÃO: Os organoides intestinais humanos são derivados do tecido humano ou de células-tronco pluripotentes/embrionárias induzidas e, como tal, é necessária aprovação ética. Os regulamentos específicos do país precisam ser seguidos. O material humano geralmente não é testado e, portanto, é frequentemente considerado material BSL-2. As condições adequadas de contenção precisam ser confirmadas no país em que o experimento ocorre.
- Preparar organoides intestinais e colonos a partir de tecidos isolados e/ou biópsias usando métodos descritos anteriormente2. Além disso, podem ser encontrados detalhes mais técnicos sobre como preparar organoides a partir de material derivado do paciente ou de iPSCs em Lees et al. e Mahe et al.24,25.
- Uma vez estabelecidas as culturas organoides, siga a rotina de divisão abaixo descrita para preparar organoides para realizar infecções com patógenos entéricos.
- Sementes 20-100 organoides em 50 μL de solução 100% ECM em placas tratadas de cultura não-tecidual de 24 poços. Adicione 500 μL de mídia de crescimento(Tabela 1) por poço.
- Mude a mídia a cada 2 dias removendo 250 μL da mídia antiga e adicionando 250 μL da nova mídia de crescimento a cada poço. Aqueça a mídia à temperatura ambiente antes de mudar a mídia.
ATENÇÃO: A mídia fria liqueficará a solução ECM que contém organoides. - Passagem os organoides uma vez por semana quando o interior começa a escurecer devido ao acúmulo de células mortas. Um exemplo disso pode ser encontrado na Figura 2.
- No dia da divisão, remova a solução ECM de -20 °C e descongele no gelo.
- Coloque a nova placa de cultura celular vazia que será usada para semear os organoides depois de se dividir a 37 °C (isso requer um mínimo de 1h para ser quente e pode ser aquecido durante a noite). Aqueça a mídia cultural à temperatura ambiente.
- Remova a mídia de crescimento com uma pipeta P1000 e adicione 500 μL de soro fisiológico a frio de 1x tamponado de fosfato (PBS) em cada poço por 3 min para liquefazer parcialmente a solução ECM e dissociar da placa.
- Para garantir a interrupção total da solução ECM, use uma pipeta P1000 (configuração a 450 μL). Pipeta para cima e para baixo 10 vezes para resuspensar a solução PBS, ECM e organoides, transferir os organoides resuspended em um tubo cônico de 15 mL e colocá-los no gelo.
- Colete vários poços dos mesmos organoides no mesmo tubo cônico.
- Se vários organoides diferentes (doadores diferentes, seções diferentes, pré-tratamento diferente, etc.) forem divididos ao mesmo tempo, colete-os em diferentes tubos cônicos. Durante a coleta, mantenha os tubos cônicos no gelo.
- Gire as amostras a 500 x g por 5 min a 4 °C. Remova cuidadosamente o PBS com uma pipeta para manter a pelota organoide na parte inferior.
- Evite usar um sistema de resíduos de vácuo com uma pipeta equipada, pois a pelota de organoides é muito solta e pode ser facilmente aspirada ao remover o PBS muito rapidamente.
- Adicione 1 mL de 0,05% de trippsina ao tubo cônico e organoides resuspend por pipetar para cima e para baixo 10 vezes com uma pipeta P1000. Incubar o tubo cônico contendo organoides a 37 °C por 3 min.
- Adicione 2 mL de mídia DMEM/F12 contendo 10% de soro bovino fetal (FBS) e 1% penicilina/estreptomicina para parar a digestão e resuspend para interromper os organoides pipetando para cima e para baixo 10 vezes com uma pipeta P1000.
- Gire as amostras a 500 x g por 5 min a 4 °C. Remova os tubos cônicos da centrífuga e armazene-os no gelo.
- Remova cuidadosamente a mídia/trippsina com uma pipeta descartável de 5 mL para manter a pelota organoide na parte inferior. Deixe cerca de 500 μL de mídia e remova isso com uma pipeta P1000.
- Uma vez que a mídia/trippsina tenha sido removida de todos os tubos cônicos, remova a placa tratada de cultura não-celular de 24 poços da incubadora de 37 °C e coloque-a sob a capa de cultura celular.
- Adicione 100% de solução de ECM (mantida no gelo) ao tubo cônico com os organoides divididos na proporção de 1:3 a 1:5, dependendo dos hábitos de crescimento dos organoides doadores individuais. (Por exemplo, se um poço contendo 20-100 organoides em uma gota de 50 μL de solução ECM foi aprovado, resuspensar a pelota organoide em 150-250 μL de solução ECM gelada.)
- Semente 50 μL de solução ECM/mistura organoide por poço da placa tratada de cultura não-celular de 24 poços pré-aquecida a 37 °C.
NOTA: A solução ECM polimerizará muito rapidamente depois de aquecida. Mantenha a solução ECM fria o tempo todo (armazenada no gelo) durante o uso. Quando os organoides tiverem sido resuspendidos, sementes imediatamente em uma nova placa. Para iniciantes, recomenda-se armazenar uma caixa de pontas de pipeta a -20 °C para permitir tempo extra para a semeadura. - Incubar a placa de 24 poços a 37 °C por 10-15 min para permitir que a solução ECM polimerize.
- Após a polimerização, adicione 500 μL de mídia de crescimento(Tabela 1)26 a cada poço e incuba os organoides a 37 °C. Verifique os organoides diariamente por microscopia. Mude a mídia a cada 2 dias como na etapa 1.4.
2. Preparação de organoides em duas dimensões (2D) para infecção apical
NOTA: O seguinte protocolo descreverá como semear organoides intestinais como uma monocamada de células em uma placa de cultura celular para infectar células epitélios intestinais de seu lado apical. Use a placa de 48 poços para os experimentos de sequenciamento e o slide de câmara de 8 poços para controlar a infecção usando abordagens de imunofluorescência.
- Cresça e mantenha organoides como descrito na seção 1.
- Antes de semear organoides em 2D, cubra as placas de 48 poços e o deslizamento de câmara de 8 poços com 200 μL de 2,5% de colágeno humano em água por poço por 1h a 37 °C.
NOTA: Os organoides intestinais são melhor semeados em placas de 48 poços e em lâmina de câmara de 8 poços de fundo de vidro. A experiência mostrou que eles não infectam bem em placas de 96 poços. As pastilhas transwell também podem ser usadas para permitir infecção apical e basolateral em 2D. Se forem utilizadas transwells, monitore a resistência elétrica trans-epitelial (TEER) antes da infecção para confirmar uma monocamada confluente. Normalmente, os organoides intestinais têm uma barreira apertada com um TEER de 450-600 Ohm/cm2. - Semente 100-150 organoides em cada poço de uma placa de 48 poços ou para um poço de um slide de câmara de fundo de vidro de 8 poços.
- Para estimar o número de organoides, conte o número de organoides presentes na queda de 50 μL da solução ECM da placa de 24 poços preparada na seção 1. Em média, são necessários 1-2 poços, o que dará aproximadamente 15.000-30.000 células.
- Para interromper a solução ECM e tentarpsinizar os organoides, siga os passos 1.8-1.17.
- Remova o colágeno humano das placas de 48 poços e o deslizamento de câmara de 8 poços de vidro.
- Resuspenha a pelota organoide no tubo cônico em 250 μL de mídia de crescimento/bem e adicione a mistura aos poços revestidos de colágeno. Coloque a placa em uma incubadora de 37 °C.
NOTA: Ao realizar os experimentos, várias condições são comparadas (mock vs. infectado +/- tratamento de interesse). Para minimizar a variabilidade entre cada poço de organoides semeados em 2D, colete o número total de organoides necessários no mesmo tubo cônico. A coleta de organoides de até 12 poços de uma placa de 24 poços pode usar 1 mL de trippsina e 2 mL de mídia neutralizante. Ao usar 12-24 poços, aumente para 2 mL de trippsina e 4 mL de mídia neutralizante. - Depois de 48 horas, retire a placa da incubadora e coloque-a na capa de cultura celular. Remova a mídia de crescimento e substitua-a por 250 μL por poço de mídia de diferenciação(Tabela 1). Repita esta mudança de mídia 48 h depois.
- Após 48h, confirme a diferenciação (quatro dias pós-switch para mídia de diferenciação) extraindo RNA, fazer cDNA com 250 ng de RNA, e realizar qPCR baseado em verde SYBR usando primers para células-tronco (OLFM4 e/ ou SMOC2), células de cálice (MUC2), células enteroendrinas (CHGA) e enterócitos (SI e/ou CYP3A4)(Figura 3).
NOTA: Ao mudar da mídia de crescimento para a mídia de diferenciação, os organoides diminuirão sua expressão de marcadores de células-tronco (OLFM4 e/ou SMOC2) e aumentarão a expressão de células de cálice (MUC2), células enteroendócrinas (CHGA) e marcadores enterócitos (SI e/ou CYP3A4) por qPCR. Prepare um poço extra e mantenha com mídia de crescimento para comparar o nível de expressão de cada marcador específico do tipo celular em mídia de crescimento versus diferenciação. - Após a confirmação da diferenciação, os organoides estão prontos para infecção apical. Mova os organoides para a contenção do nível de biossegurança necessária para o patógeno de escolha.
ATENÇÃO: Consulte as normas locais e procedimentos operacionais padrão do instituto ao manusear patógenos sob contenção BSL-2 ou BSL-3. - Para infectar os organoides intestinais cultivados em 2D a partir de seu lado apical, remova a mídia com uma pipeta P1000 e adicione o patógeno diluído na multiplicidade de infecção necessária para o experimento em mídia de diferenciação (o volume mínimo de mistura de patógeno/mídia é de 50 μL por poço e o volume máximo é de 250 μL por poço).
- Permita que a infecção prossiga por 2h a 37 °C com uma mesa de roqueiro localizada na incubadora de cultura celular ou balançando manualmente a placa a cada 15-20 min. Otimize este tempo com base no patógeno de escolha.
- Depois de 2h, remova a mídia com uma pipeta P1000 e substitua-a por 250 μL por poço de mídia de diferenciação fresca. Incubar células a 37 °C até o ponto de tempo do sequenciamento unicelular.
- Para preparar células para sequenciamento de células únicas, prossiga para a seção 4.
3. Preparação de organoides em três dimensões (3D) para infecção apical e basolateral
- Cultivar organoides como descrito na seção 1 em uma placa tratada de cultura não celular de 24 poços.
- Dois dias após a passagem, retire a placa da incubadora e coloque em um capô de cultura celular.
- Remova a mídia de crescimento com uma pipeta P1000, substitua-a por 500 μL/bem de mídia de diferenciação pré-aquecida à temperatura ambiente e coloque a placa na incubadora de 37 °C.
- Após 48h, substitua por mídia de diferenciação fresca (500 μL/well).
NOTA: Os organoides são mantidos em meios de diferenciação por um total de 4 dias antes da infecção. - Confirme a diferenciação conforme descrito na etapa 2.9.
- Após a confirmação de que a diferenciação ocorreu, os organoides estão prontos para infecção.
- Descongele eCM no gelo. Aqueça a mídia de diferenciação à temperatura ambiente e aqueça uma placa de 24 poços a 37 °C.
- Para realizar infecções, remova os organoides da solução ECM.
NOTA: Remova o máximo possível de solução ECM, pois os vírus preferem aderir à solução ECM em vez de células. Se houver muita solução de ECM remanescente em torno dos organoides, então a infectividade será severamente impactada. - Interrompa o ECM adicionando 500 μL de PBS frio 1x e incubando por 3 min. Use um P1000 para pipeta para cima e para baixo 10x. Combine organoides em um tubo e centrífuga a 500 x g por 5 min. Remova o PBS com um P1000.
NOTA: Para minimizar a variabilidade entre cada condição de infecção, combine os organoides provenientes de vários poços de uma placa de 24 poços no mesmo tubo cônico. Por exemplo, se oito condições de infecção forem necessárias, oito poços de uma placa de 24 poços (contendo ~100 organoides cada) serão combinados e posteriormente divididos em oito poços de uma nova placa de 24 poços após a interrupção da agulha (ver passo 3.12). - Resuspend os organoides em 1 mL de mídia de diferenciação. Para infecção apical e basolateral seguem o passo 3.10.1 e para infecção basolateral seguem apenas o passo 3.10.2.
- Conecte uma agulha de 27 G a uma seringa de 1 mL e resuspenque a pelota seis vezes para permitir a interrupção dos organoides e para que a infecção ocorra tanto nos lados apical quanto basolateral. Prossiga para a etapa 3.11.
NOTA: Os organoides devem ser os organoides clássicos de cisto e polvo com um centro escuro quando observados antes da interrupção da agulha. Após a interrupção, esses organoides aparecerão menores, com menos ou nenhum interior escuro. Ao realizar a infecção, sempre haverá um mínimo de duas condições (simulação e infecção), um mínimo de dois poços de uma placa de 24 poços será inicialmente combinado em um tubo cônico de 15 mL para interrupção à base de agulha (explicando por que um mínimo de 1 mL é usado para resuspensão). Ao executar mais de duas condições, combine até 10 poços de uma placa de 24 poços em um único tubo cônico de 15 mL e resuspenque em 1 mL de mídia de diferenciação para interrupção da agulha. Após a interrupção, adicione 500 μL de mídia de diferenciação por n + 1 (por exemplo, se usar 10 poços, em seguida, cubra com 4,5 mL para ter 5,5 mL no total). Se forem necessárias mais de 10 condições, use vários tubos cônicos. Recomenda-se uma seringa de 1 mL, pois os organoides são melhor interrompidos com este volume da seringa. Uma alternativa para uma seringa é empregar uma ponta P1000 bem achatada. - Resuspend os organoides em 500 μL de mídia de diferenciação por poço. Tenha cuidado para não interromper os organoides e certifique-se de que o lado apical não esteja acessível. Prossiga para a etapa 3.11.
- Conecte uma agulha de 27 G a uma seringa de 1 mL e resuspenque a pelota seis vezes para permitir a interrupção dos organoides e para que a infecção ocorra tanto nos lados apical quanto basolateral. Prossiga para a etapa 3.11.
- Transfira 500 μL de suspensões organoides por poço para uma placa de cultura celular de 24 poços usando uma pipeta P1000 e mova a placa para a contenção do nível de biossegurança necessária para o patógeno de escolha.
ATENÇÃO: Consulte as normas locais e os procedimentos operacionais padrão do instituto ao manusear patógenos sob contenção BSL-2 ou BSL-3. Sempre realize a interrupção da agulha dos organoides fora do BSL-3 para evitar possíveis acidentes com a agulha. As regulamentações locais também podem impedir o uso de agulhas no BSL-3. - Adicione o patógeno diluído em meios de diferenciação para alcançar a multiplicidade de infecção necessária para o experimento. Não exceda um volume total de 500 μL (para ter 1 mL total na placa de 24 poços).
- Permita que a infecção prossiga por 2h (desta vez precisará ser adaptada ao patógeno de escolha) a 37 °C com uma mesa de roqueiro localizada na incubadora de cultura celular ou balançando manualmente a placa a cada 15-20 min.
- Após a incubação de 2 h, colete os organoides em um tubo de 1,5 mL por condição e gire a 500 x g por 5 min a 4 °C.
- Remova a mídia que contém patógeno com uma pipeta P1000 e armazene-a no gelo. Lave a pelota organoide uma vez com PBS.
- Resuspend os organoides em 50 μL de solução 100% ECM (que antes era descongelada no gelo), placa em uma placa tratada de cultura não-celular de 24 poços pré-aquecida a 37 °C e incubar a placa de 24 poços a 37 °C por 10-15 min para permitir que a solução ECM se polimerize.
- Após a polimerização, adicione 500 μL de mídia de diferenciação à temperatura ambiente a cada poço e incuba a 37 °C até a colheita para sequenciamento unicelular.
NOTA: Se a colheita precisar ocorrer mais de 48h após a semeadura, mude a mídia a cada 2 dias, removendo a mídia antiga e substituindo-a por 500 μL de mídia de diferenciação fresca. No entanto, a experiência tem mostrado que, uma vez que os organoides são cultivados sob meios de diferenciação, eles não sobreviverão por muito mais de 48 h.
4. Preparação de solução celular única e preparação de gems de gel em condições de biossegurança nível 3 (BSL-3)
NOTA: A conclusão das seguintes etapas requer que o equipamento de sequenciamento de célula única(Tabela de Materiais) e uma máquina PCR capaz de lidar com reações de 100 μL estejam presentes dentro de uma instalação BSL-3. Os organoides são cultivados conforme descrito na seção 1 e infectados como descrito nas seções 2-3, dependendo do patógeno e da rota de entrada. Em um momento pré-determinado pós-infecção, organoides são colhidos. Abaixo está descrito o método utilizado para a colheita de organoides intestinais.
- Antes de colher organoides infectados, remova as contas de gel de célula única(Tabela de Materiais) a partir de -80 °C e aqueça até a temperatura ambiente (pelo menos 30 minutos antes).
- Além disso, equilibre o reagente RT, reduzindo a enzima B e RT (todas armazenadas a -20 °C) à temperatura ambiente. Resuspenque o oligo do interruptor de modelo, conforme descrito nas instruções do fabricante.
NOTA: Todas as etapas a seguir devem ser realizadas em relação aos regulamentos locais de biossegurança seguindo o procedimento operacional padrão estabelecido para o patógeno considerado. Como regra geral para o trabalho BSL-3, deve-se garantir que o exterior de todos os vasos (placas, tubos, etc.) que saem de uma capa de cultura celular sejam devidamente desinfetados. O mesmo se aplica às mãos/luvas da pessoa que realiza os experimentos. - Para realizar infecções em organoides semeados em 2D (seção 2) prosseguem para a etapa 4.4. Para realizar infecções em organoides 3D (seção 3) proceda às seções 4.5.
- Para infecções de organoides 2D, traga a placa de cultura celular para dentro do capô e remova a mídia de diferenciação com uma pipeta P1000.
- Adicione 250 μL de 1x PBS à temperatura ambiente a cada poço.
- Remova o PBS com uma pipeta P1000 e adicione 250 μL de enzima de dissociação de temperatura ambiente (por exemplo, TrypLE Express) a cada poço de uma placa de 48 poços. Troque as luvas, limpe a placa e coloque a placa com a enzima dissociação na incubadora de 37 °C.
- Observe a dissociação celular por microscopia a cada 5 minutos.
NOTA: Aproximadamente, leva 15 minutos para dissociar organoides intestinais cultivados em 2D em células únicas. Este tempo precisará ser ajustado, pois dependerá de quantos organoides foram inicialmente semeados e infectados, bem como no patógeno, pois alguns patógenos são mais citopáticos e fazem com que os organoides dissociassem muito mais rápido do que outros. - Após a confirmação de uma aparente suspensão de célula única, traga a placa de volta para o capô de cultura celular e pare a digestão adicionando 250 μL de mídia DMEM/F12 contendo 10% de FBS por poço de uma placa de 48 poços.
- Colete as células em um tubo cônico de 15 mL usando uma pipeta P1000.
- Troque as luvas, limpe o tubo e gire as amostras a 500 x g por 5 min a 4 °C.
- Remova cuidadosamente a enzima de mídia/dissociação com um P1000 para manter a pelota celular na parte inferior. Resuspend células únicas em um volume mínimo de PBS contendo 0,1% de BSA. Para organoides intestinais, resuspenque em 250 μL para cada poço da placa de 48 poços.
- Passe as suspensões celulares em um tubo FACS com um filtro para remover quaisquer grandes aglomerados e coloque o tubo FACS contendo células no gelo.
- Prossiga para a etapa 4.6 para continuar com a contagem de células e a preparação da mistura mestre.
- Para infecções de organoides 3D, remova a placa da incubadora e coloque-a na capa da cultura celular.
- Remova a mídia de diferenciação de cada poço da placa de 24 poços com uma pipeta P1000. Adicione 500 μL de 1x PBS gelado a cada poço e incubar por 3 minutos no gelo.
- Para garantir a interrupção total da solução ECM, use uma pipeta P1000 (definida para 450 μL). Pipeta para cima e para baixo 10 vezes para resuspensar a solução PBS, ECM e organoides; transfira os organoides resuspended em um tubo cônico de 15 mL e coloque no gelo. Colete cada condição de infecção em seu próprio tubo cônico de 15 mL.
NOTA: O uso de um tubo cônico de 15 mL dá uma pelota celular melhor e mais distinta do que um tubo cônico de 50 mL ou um tubo de 1,5 mL. - Troque as luvas e remova o tubo do capô da cultura celular. Limpe a parte externa do tubo.
- Gire as amostras a 500 x g por 5 min a 4 °C.
- Mova o tubo de volta para a capa de cultura celular e remova pbs com uma pipeta P1000 para evitar reaprentar a pelota organoides da parte inferior do tubo.
- Resuspende a pelota em 1 mL de enzima dissociação (por exemplo, TrypLE Express). Troque as luvas, limpe o tubo e incuba as amostras a 37 °C.
- A cada 10 minutos por 30 minutos, mova o tubo de volta na capa da cultura celular e resuspense os organoides pipetando para cima e para baixo com uma pipeta P1000 10 vezes.
NOTA: Normalmente, os organoides intestinais requerem cerca de 30 minutos para formar uma única suspensão celular quando infectados em 3D. - Para determinar quando os organoides são dissociados em células únicas, tome 10 μL da suspensão organoide usando uma pipeta p10.
- Coloque a suspensão em um slide de contador de células plásticas descartáveis. Sele a porta de entrada da amostra com fita clara.
- Troque as luvas e limpe o lado de fora do balcão da célula. Use um microscópio brightfield para determinar se uma única suspensão celular é feita.
- Após a confirmação de uma única suspensão celular, pare a digestão adicionando 1 mL de mídia DMEM/F12 contendo 10% de FBS e pipeta para cima e para baixo 10 vezes com uma pipeta P1000.
- Troque as luvas, limpe o tubo e gire as amostras a 500 x g por 5 min a 4 °C.
- Remova cuidadosamente a enzima de mídia/dissociação com uma pipeta P1000 para evitar reaprentar a pelota da célula da parte inferior do tubo.
- Resuspenha as células únicas em um volume mínimo de PBS contendo 0,1% de BSA. Para organoides intestinais resuspend em 250 μL.
- Passe as suspensões celulares em um tubo FACS com um filtro para remover quaisquer grandes aglomerados e coloque o tubo FACS contendo células no gelo.
- Prossiga para a etapa 4.6 para continuar com a contagem de células e a preparação da mistura mestre.
- Determine o número de células por μL adicionando 10 μL de suspensão celular a uma câmara de contagem de células plásticas descartáveis.
- Sele a porta de entrada da amostra com fita clara antes de remover a amostra do capô de cultura celular, pois nesta fase, a suspensão celular ainda é infecciosa.
- Troque as luvas, limpe a câmara de contagem de células e conte o número da célula usando um microscópio de campo brilhante.
- Dentro da capa de cultura celular, prepare uma mistura mestre de Reagente RT, Template Switch Oligo, Reducing Agent B e RT enzima C em um tubo de 1,5 mL de acordo com as instruções do fabricante, dependendo do número de amostras no experimento. Para cada amostra, alíquota de 33,4 μL de mistura mestre em um tubo PCR e armazenar no gelo.
- Adicione as células e a água à mistura mestre de acordo com o número da célula alvo, conforme descrito nas instruções do fabricante.
NOTA: 50%-60% das células são normalmente recuperadas (ou seja, ao carregar 10.000 células no chip, 5.000-6.000 células são usadas para análise). Portanto, sempre carregue 10.000 células. Se a densidade celular não permitir isso, centrifugar as células e resuspend em um volume menor. Tome cuidado para não sobrecarregar o chip, pois isso resultará em entupimento do chip. Além disso, se as células não forem adequadamente dissociadas, haverá um risco aumentado de obter múltiplas células por conta, que precisarão ser removidas no processamento de bioinformática a jusante. - Mude as luvas, mova o controlador de célula única para o capô da cultura celular, e prepare o chip, pois o chip só está coberto com uma junta que não está selada.
NOTA: A máquina precisa estar dentro do capô para evitar exposição a suspensões celulares infectadas e aerossóis potenciais. - Adicione o chip de célula única ao suporte do chip e encha as pistas nãousadas com 50% de glicerol.
- Adicione a mistura mestre, as contas e o óleo de particionamento nas pistas utilizadas para amostras de acordo com as instruções do fabricante.
- Cubra o chip com a junta, carregue o chip no controlador e inicie o programa.
NOTA: Recomenda-se carregar apenas seis das oito pistas. Problemas com emulsões impróprias ocorrem frequentemente nas faixas um e oito. A empresa diz que todas as pistas são independentes, e isso não deve ocorrer; no entanto, se possível, evite essas duas pistas e execute dois chips se oito amostras forem necessárias. - Após a conclusão do programa, remova o chip e a junta.
- Use uma pipeta multicanal e transfira 100 μL das emulsões para um tubo PCR limpo. Certifique-se de que cada poço tenha uma cor branca uniforme indicando uma emulsão completa.
- Troque as luvas, limpe os tubos e transfira o tubo PCR para uma máquina PCR que possa suportar reações de 100 μL. Executar o programa: 53 °C para 45 min; 85 °C por 5 min.
- Na conclusão, armazene as amostras a 4 °C. Neste ponto, processe a reação de acordo com as instruções do fabricante ou armazene a 4 °C durante 3 dias ou -20 °C durante 1 semana.
- Após 5 min a 85 °C, a maioria dos vírus envoltos será inativada, remova o CDNA do BSL-3 de acordo com o procedimento normal de funcionamento e realize os preparativos da biblioteca em um laboratório BSL-1.
NOTA: Este experimento precisa ser realizado como três réplicas biológicas (por exemplo, em três dias diferentes) porque a extensão da diferenciação de cada tipo de célula pode variar ligeiramente entre cada experimento. As bibliotecas de sequenciamento resultantes poderiam ser indexadas de forma diferente e sequenciadas em uma sequência de sequência.
Resultados
Preparação de organoides para sequenciamento unicelular
Os resultados de sequenciamento de células únicas são altamente dependentes do uso de células de boa qualidade. Para garantir que os organoides sejam de boa qualidade, eles devem ser devidamente mantidos e observados diariamente para determinar quando estão prontos para serem divididos(Figura 2). O tempo de divisão dos organoides é dependente do doador; alguns doadores crescem mais rapidamente e precisam ser divididos a cada 5 dias, enquanto outros são mais lentos e precisam ser divididos a cada 10 dias. Em média, os organoides são divididos uma vez por semana quando os centros ficam escuros(Figura 2B). Se os organoides se tornarem muito grandes e acumularem muitas células mortas no centro, o organoide morrerá.
Organoides são mantidos em uma mídia que contém altas quantidades de Wnt3A. Isso suporta o nicho de células-tronco e promove que os organoides continuem a crescer e proliferar. Nessas condições de crescimento, os organoides contêm altas quantidades de células-tronco e células amplificadoras de trânsito e uma menor quantidade de populações celulares diferenciadas, como enterócitos maduros, células de cálice e células enteroendócrinas. No entanto, para imitar a complexidade celular encontrada dentro do intestino humano, é importante empurrar a diferenciação celular e produzir mais dessas células. Isso é feito alterando as condições de mídia e removendo o Wnt3A, e reduzindo R-Spondin e Noggin (Tabela 1). Normalmente, a diferenciação celular em relação a enterócitos, células de cálice e células enteroendócrinas requer 4 dias de mídia de diferenciação(Figura 3). É fundamental obter uma boa diferenciação; caso contrário, avaliar o tropismo patógeno e as respostas específicas do tipo celular se tornarão difíceis.
Confirmação da inativação do patógeno BSL-3
A inativação total deve ser confirmada com o patógeno de escolha e validada que é seguro remover o cDNA do BSL-3. Para o SARS-CoV-2, a inativação total do vírus foi validada tomando 100 μL de SARS-CoV-2 e incubando-o em uma máquina PCR por 5 min a 85 °C. O vírus foi então adicionado de volta às células vero ingênuas, e a infecção por vírus foi comparada com o vírus não-térmico tratado por imunofluorescência e ensaios de placa a 24 h, 48 h e 72 h pós-infecção para garantir que todas as partículas não fossem mais infecciosas. Esses resultados foram enviados à agência reguladora local e, após sua aprovação, foi realizado o experimento unicelular e o processamento de CDNA.
Resultados de sequenciamento de células únicas
Para avaliar como o SARS-CoV-2 infecta organoides humanos de cólon e íleo, foi realizado sequenciamento unicelular. Os organoides foram preparados como descritos acima e infectados em formato 2D para permitir a infecção apical pelo SARS-CoV-2. As células infectadas foram colhidas às 12h e 24 h pós-infecção e processadas para sequenciamento unicelular, conforme descrito acima. A análise dos dados de sequenciamento unicelular nos permitiu determinar que apenas uma subpopulação (enterocyto imaturo 2) de células epiteliais intestinais humanas suportava a infecção do SARS-CoV-2 (Figura 4). Além disso, como nem todas as células de uma população foram infectadas, tanto células infectadas quanto células de espectadores não infectadas foram analisadas(Figura 5). Esses resultados mostraram que o SARS-CoV-2 induziu uma cascata de sinal pró-inflamatório em células infectadas, enquanto as células de espectadores não infectadas mostraram uma resposta imune mediada por interferon. Além disso, o SCRNA-Seq mostrou que as células infectadas não conseguiam sentir interferons devido ao bloqueio mediado pelo vírus da via(Figura 5). Não foi possível obter essas informações ao usar sequenciamento de RNA em massa.

Figura 1: Esquema representando os três métodos diferentes para preparar organoides intestinais humanos para infecção com patógenos entéricos. A infecção apical pode ser alcançada através da semeadura de organoides intestinais em 2D. Uma infecção apical e basolateral pode ser realizada interrompendo o organoide 3D. Finalmente, uma única infecção basolateral pode ser realizada infectando organoides intestinais 3D intactos. Cada um desses métodos pode ser usado para gerar amostras para sequenciamento de células únicas. Clique aqui para ver uma versão maior desta figura.
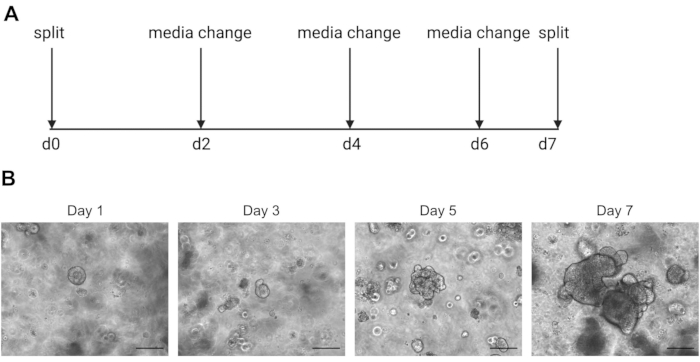
Figura 2: Esquema de manutenção organoide e imagens representativas de brightfield. (A) Esquema para manutenção e passagem de organoides intestinais humanos. (B) Imagens representativas de brightfield dos dias 1, 3, 5 e 7 pós-divisão. No dia 7, os organoides se tornam grandes e escuros devido ao acúmulo de células mortas e estão prontos para serem divididos. A barra de escala indica 25 μm. Clique aqui para ver uma versão maior desta figura.
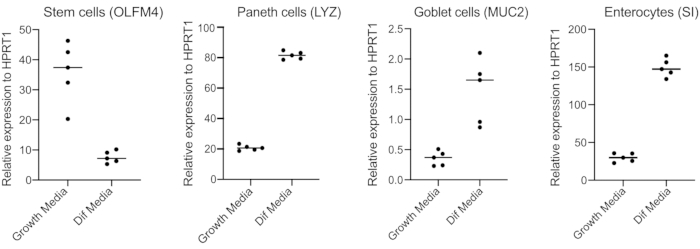
Figura 3: Representante qPCR de organoides intestinais humanos 4 dias após a mudança para mídia de diferenciação. Os organoides intestinais foram mantidos em meios de crescimento ou mudaram para mídia de diferenciação por 4 dias. O RNA foi colhido e o qPCR foi realizado para marcadores de células-tronco (OLFM4), células paneth (LYZ), células de cálice (MUC2) e enterócitos (SI). N = 5. Clique aqui para ver uma versão maior desta figura.
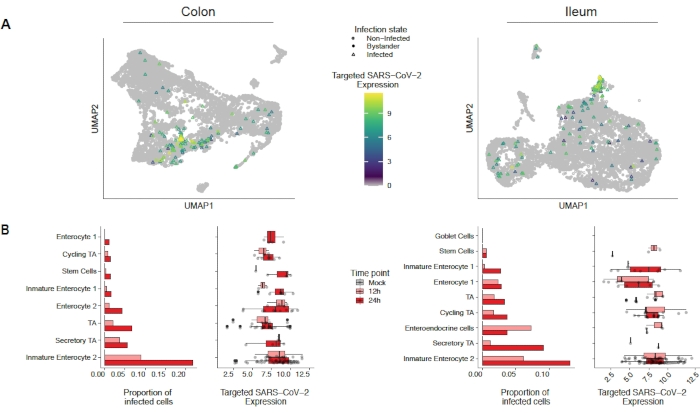
Figura 4: Identificação da população celular infectada pelo SARS-CoV-2. Organoides derivados de cólon humano e íleo foram infectados com SARS-CoV-2. Após 12 e 24 h, células pós-infecção foram colhidas e submetidas ao sequenciamento de RNA unicelular para identificar quais populações celulares apoiaram a infecção pelo SARS-CoV-2. A infecção por vírus aumentou com o tempo e infectou principalmente enterócito imaturo 2. Este número foi modificado a partir de Triana et al.19. Clique aqui para ver uma versão maior desta figura.
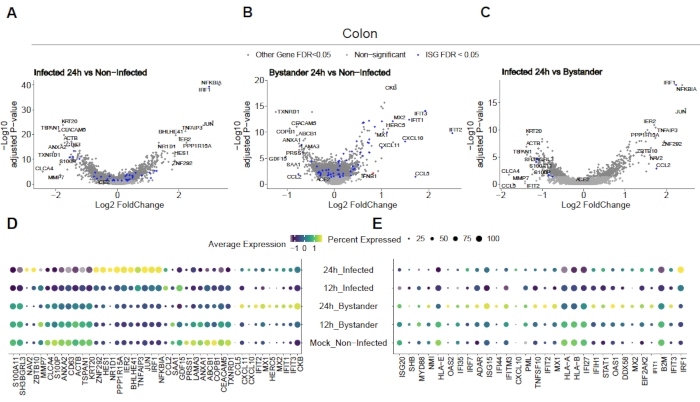
Figura 5: Determinação da resposta imune intrínseco inata. Organoides derivados do cólon humano foram infectados com SARS-CoV-2. Após 12 e 24 h, células pós-infecção foram colhidas e submetidas ao sequenciamento de RNA de células únicas para determinar a resposta imune intrínseco inata em células de espectadores viralmente infectadas e não infectadas. As células infectadas pelo SARS-CoV-2 apresentaram uma forte resposta pró-inflamatória, enquanto as células de espectadores não infectadas apresentaram uma resposta mediada por interferon. Figura modificada de Triana et al.19. Clique aqui para ver uma versão maior desta figura.
| Mídia de Crescimento | |
| Composto | Concentração final |
| Ad DMEM/F12 | GlutaMAX (1X) |
| +GlutaMAX | HEPES 1 mM |
| +HEPES | Caneta 10 U/mL |
| +P/S | Estreptocos 10 μg/mL |
| L-WRN | 50% em volume |
| B27 | 01:50 |
| N-acetil-cisteína | 1 mM |
| EGF | 50 ng/mL |
| A83-01 | 500 nM |
| IGF-1 | 100 ng/mL |
| FGF básico | 50 ng/mL |
| Gastrin | 10 mM |
| Mídia de diferenciação | |
| Composto | Concentração final |
| Ad DMEM/F12 | GlutaMAX (1x) |
| +GlutaMAX | HEPES 1 mM |
| +HEPES | Caneta 10 U/mL |
| +P/S | Estreptocos 10 μg/mL |
| B27 | 01:50 |
| N-acetil-cisteína | 1 mM |
| R-spondin | 5% em volume |
| Cabeça | 50 ng/mL |
| EGF | 50 ng/mL |
| Gastrin | 10 mM |
| A83-01 | 500 nM |
Tabela 1: Composição de mídia para mídia de crescimento e diferenciação.
Discussão
Patógenos entéricos geralmente iniciam seu ciclo de vida infectando células epiteliais intestinais de seu lado apical voltado para o lúmen do intestino. Embora os organoides sejam bem reconhecidos como um bom modelo para reproduzir a complexidade celular e a organização do epitélio intestinal, sua organização como estruturas tridimensionais e fechadas tornam sua membrana apical inacessível ao patógeno. Este protocolo descreveu métodos para infectar organoides intestinais de seu lado apical, seu lado basolateral, ou ambos com patógenos BLS-3. Esses protocolos podem ser facilmente adaptados para estudar qualquer patógeno entérico sob contenção BSL-2 ou BLS-3 ou qualquer outro modelo organoide seguindo algumas etapas críticas que são destacadas abaixo. O método descrito acima é para o isolamento e preparação de gotículas unicelulares de acordo com as regulamentações na Alemanha. Como isenção de responsabilidade, este protocolo não descreve as medidas de tratamento de biossegurança (procedimentos operacionais padrão) que precisam ser tomadas enquanto trabalham sob condições BSL-3. Também é importante insistir que as regulamentações podem variar em outros países e que as autoridades locais devem ser contatadas para garantir que todas as regulamentações locais sejam respeitadas.
Um dos passos críticos na semeadura de organoides em duas dimensões para infecção apical é controlar que as células se diferenciarão da mesma forma em comparação com quando cultivadas como organoides tridimensionais clássicos. Dependendo do patógeno entérico, o tropismo pode ser restrito a células muito raras ou a células que precisam ser altamente diferenciadas. Neste caso, o uso de um organoide bidimensional que não se diferenciava totalmente poderia resultar na má conclusão de que este patógeno entérico não pode infectar organoides intestinais. Sugere-se, se possível, a realização de infecções utilizando as três configurações deste protocolo: organoide 2D apenas para infecção apical (seção 2), organoides 3D abertos para infecção apical e basolateral (seção 3), e organoides 3D completos apenas para infecção basolateral (seção 3). Esta abordagem ajudará a discernir a rota de entrada do patógeno (apical vs. basolateral) e também controlará que um nível semelhante de diferenciação foi alcançado. Uma alternativa para infecção apical 2D é a microinjeção, que usará um organoide 3D, mas fornecerá o patógeno diretamente para o lado apical (ver Bartfeld et al.27 para obter detalhes). Este método requer um injetor qualificado para garantir que o patógeno esteja devidamente colocado, e os organoides permaneçam intactos. A microinjeção é comumente usada na contenção BSL-2 e pode não ser adequada para contenção BSL-3.
Uma consideração importante adicional na realização de experimentos de infecção em organoides semeados em 2D é a densidade celular final. Como mencionado na etapa 2.3, 100-150 organoides serão semeados em um poço de uma placa de 48 poços ou um poço de um deslizamento de câmara de vidro de 8 poços. Dependendo da linha organoide e da pessoa que manuseia os organoides, o tamanho desses organoides pode ser significativamente diferente. Isso pode resultar em densidades celulares muito diferentes na placa de 48 poços ou em deslizamento de câmara de vidro de 8 poços. Dependendo do vírus entérico, alguns vírus preferem células mais esparsas, enquanto outros também serão capazes de infectar células confluentes. A origem molecular de tais diferenças na infectividade para diferentes confluências celulares não é clara; portanto, experimentos piloto com o objetivo de encontrar a melhor densidade celular para o patógeno entérico de escolha devem ser realizados antes de realizar uma caracterização mais a jusante.
Muitas vezes a classificação FACS é realizada antes de realizar a emulsão de gotícula de célula única. Esta etapa é frequentemente usada para separar mortos de células vivas e células únicas de doublets. Ao trabalhar com patógenos BSL-3, requer que a instalação esteja equipada com um classificador FACS apropriado, o que não é frequentemente o caso. Além disso, nem todas as células em um organoide têm o mesmo tamanho, e muitas vezes é difícil discriminar entre um doublet ou uma célula maior, causando assim o risco de seleção negativa contra um tipo de célula específica. Além disso, ainda há discussão no campo se o tempo necessário para a triagem entre 5.000-10.000 para cada amostra poderia resultar em uma modificação significativa do perfil de transcrição das células individuais. Embora tenham sido descritos métodos de fixação celular compatíveis com sequenciamento unicelular (por exemplo, metanol e RNAassist), observou-se que isso leva a uma diminuição na qualidade do sequenciamento18. Finalmente, suspeita-se que a triagem de células usando marcadores de morte celular também pode levar a um viés. Dada a proliferação direcional e diferenciação das células através do eixo cripta-villi, as células mais diferenciadas, que serão derramadas e liberadas, estão localizadas na ponta do villi. Essas células são muitas vezes positivas para diferentes marcadores de vias de morte celular (porexemplo, apoptose, necrose e necroptose); no entanto, ao olhar para a infecção rotavírus do intestino do camundongo, a ponta do villi é a área mais infectada28. Assim, filtrar células que possam parecer positivas para marcadores de morte resultaria em uma seleção negativa das células infectadas que podem representar a infecção fisiológica. Atualmente, não há uma boa solução para classificar e fixar organoides antes do sequenciamento unicelular. O uso de células vivas e não sortidas é recomendado, pois são necessários estudos adicionais para encontrar protocolos alternativos adequados.
O sequenciamento unicelular revolucionou a forma como as respostas celulares podem ser avaliadas. Esta técnica permite a identificação de respostas específicas da linhagem celular, tanto em condições basais quanto sob infecções por patógenos. Este método abriu portas em muitos campos que antes eram limitados por leituras em massa. Embora este método seja muito poderoso, ele tem suas limitações. Uma limitação fundamental é a extensa análise bioinformática que é necessária a jusante do sequenciamento. Isso é especialmente fundamental ao analisar tecidos e atribuir tipos de células onde atualmente não há anotação. Ter um bioinformático qualificado é necessário para apoiar todos os estudos unicelulares.
Este protocolo descreve como semear e lidar com organoides intestinais humanos, infectá-los com patógenos entericos e realizar scRNAseq. Adaptar essa abordagem a outros órgãos agora é possível, já que sistemas de modelo organoide foram desenvolvidos para a maioria dos órgãos. Os organoides pulmonares e hepáticos são igualmente organizados em comparação com organoides intestinais, e como tal, o uso de uma abordagem análoga poderia ser transposto para esses organoides. O controle crítico será validar que, quando cultivados em duas dimensões ou abertos, esses organoides alcançam diferenciação semelhante à de suas contrapartes organoides 3D. As características e genes específicos que definem um status diferenciado são específicos para cada modelo de órgão. Outros modelos organoides, como organoides renais e vasculares, grandes estruturas densas, precisarão de métodos adicionais para dissociar essas estruturas em células únicas.
Divulgações
Os autores não declaram interesses financeiros concorrentes.
Agradecimentos
Megan Stanifer e Steeve Boulant foram apoiadas por bolsas de pesquisa da Deutsche Forschungsgemeinschaft (DFG): (Projeto número 240245660, 278001972, 415089553 e 272983813 para Steeve Boulant e 416072091 para Megan Stanifer), o estado de Baden-Wuerttemberg e o Bundesministerium für Bildung und Forschung BMBF 01KI20239B para MS e 01KI20198A e NUM (-COVID 19, Organo-Strat 01KX2021) para SB. Os esquemas foram criados em BioRender.
Materiais
| Name | Company | Catalog Number | Comments |
| Recombinant mouse noggin | Peprotech | Cat#250-38 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | Cat# G9145 | |
| 0.05% Trypsin-EDTA | Thermo Fischer Scientific | Cat#25300054 | |
| 24-well non-cell culture treated plate | Corning | Cat#3738 | |
| 8-well glass bottom chamber slide | iBIDI | Cart#80827 | |
| A83-01 | Tocris | Cat#2939 | |
| Advanced DMEM/F12 | Thermo Fischer Scientific | Cat# 12634010 | |
| B27 | Thermo Fischer Scientific | Cat#17504-044 | |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | Cat#1000202 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | Cat #1000127 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Chromium Next GEM Single Cell 3′ Kit v3.1 | 10X Genomics | Cat#1000268 | Used in the preparation of single cell solution and preparation of Gel beads-in-emulsion (GEM) |
| Collagen from human placenta | Sigma Aldrich | Cat#C5533-5MG | |
| CYP34A forward | Eurofins | GATGGCTCTCATCCCAGACTT | Primers used to check for differentiation |
| CYP3A4 reverse | Eurofins | AGTCCATGTGAATGGGTTCC | Primers used to check for differentiation |
| DMEM/F12 | Thermo Fischer Scientific | Cat#11320074 | |
| EDTA | Sigma Aldrich | Car#E9884 | |
| Fast Read 102 counting slides | Biosigma | Cat# BVS100 | |
| Fetal Bovein Serum (FBS) | Capricorn | Cat#FBS-12A | |
| GlutaMAX | Thermo Fischer Scientific | Cat# 35050061 | |
| HEPES | Thermo Fischer Scientific | Cat3 15630080 | |
| L-WRN cells | ATCC | CRL-3276 | This cell line is used to make the conditioned media containg Wnt 3A, R-Spondin and Noggin. The protocol for the production of the conditioned media can be found on the manufacterures site. |
| MatriGel. GFR, LDEV free | Corning | Cat#354230 | |
| MUC-2 forward | Eurofins | TGTAGGCATCGCTCTTCTCA | Primers used to check for differentiation |
| MUC-2 reverse | Eurofins | GACACCATCTACCTCACCCG | Primers used to check for differentiation |
| N-acetyl-cysteine | Sigma Aldrich | Cat# A9165 | |
| OLMF4 forward | Eurofins | ACCTTTCCCGTGGACAGAGT | Primers used to check for differentiation |
| OLMF4 reverse | Eurofins | TGGACATATTCCCTCACTTTGGA | Primers used to check for differentiation |
| Penicillin/Streptomycin | Thermo Fischer Scientific | Cat#15140122 | |
| Recombinant human FGF basic | Peprotech | Cat#100-18B | |
| Recombinant human IGF-1 | BioLegend | Cat#590904 | |
| Recombinant mouse EGF | Thermo Fischer Scientific | Cat# PMG8043 | |
| SI forward | Eurofins | AATCCTTTTGGCATCCAGATT | Primers used to check for differentiation |
| SI reverse | Eurofins | GCAGCCAAGAATCCCAAT | Primers used to check for differentiation |
| TrypLE Express | Thermo Fischer Scientific | Cat#12605036 | |
| Y-27632 | Caymann Chemicals | Cat#10005583 |
Referências
- Kretzschmar, K., Clevers, H. Organoids: Modeling development and the stem cell niche in a dish. Developmental Cell. 38, 590-600 (2016).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141, 1762-1772 (2011).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Tekes, G., Ehmann, R., Boulant, S., Stanifer, M. L. Development of feline ileum- and colon-derived organoids and their potential use to support feline coronavirus infection. Cells. 9, (2020).
- Derricott, H., et al. Developing a 3D intestinal epithelium model for livestock species. Cell and Tissue Research. 375, 409-424 (2019).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nature Medicine. 26, 1077-1083 (2020).
- Sato, T., Clevers, H. Growing self-organizing mini-guts from a single intestinal stem cell: mechanism and applications. Science. 340, 1190-1194 (2013).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470, 105-109 (2011).
- de Graaf, M., van Beek, J., Koopmans, M. P. Human norovirus transmission and evolution in a changing world. Nature Reviews Microbiology. 14, 421-433 (2016).
- Leshem, E., et al. Real-world effectiveness of pentavalent rotavirus vaccine among bedouin and jewish children in southern Israel. Clinical Infectious Diseases : An Official Publication of the Infectious Diseases Society Of America. 62, 155-160 (2016).
- Karst, S. M. The influence of commensal bacteria on infection with enteric viruses. Nature Reviews Microbiology. 14, 197-204 (2016).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science. 353, 1387-1393 (2016).
- de Wit, E., van Doremalen, N., Falzarano, D., Munster, V. J. SARS and MERS: recent insights into emerging coronaviruses. Nature Reviews Microbiology. 14, 523-534 (2016).
- Scaldaferri, F., et al. The thrilling journey of SARS-CoV-2 into the intestine: From pathogenesis to future clinical implications. Inflammatory Bowel Diseases. 26, 1306-1314 (2020).
- Kipkorir, V., Cheruiyot, I., Ngure, B., Misiani, M., Munguti, J. Prolonged SARS-CoV-2 RNA detection in anal/rectal swabs and stool specimens in COVID-19 patients after negative conversion in nasopharyngeal RT-PCR test. Journal of Medical Virology. 92, 2328-2331 (2020).
- Lamers, M. M., et al. SARS-CoV-2 productively infects human gut enterocytes. Science. 369, 50-54 (2020).
- Stanifer, M. L., et al. Critical role of type III interferon in controlling SARS-CoV-2 Infection in human intestinal epithelial cells. Cell Reports. 32, 107863 (2020).
- Triana, S., et al. Single-cell transcriptomics reveals immune response of intestinal cell types to viral infection. bioRxiv. , (2020).
- Triana, S., et al. Single-cell analyses reveal SARS-CoV-2 interference with intrinsic immune response in the human gut. Molecular Systems Biology. 17, 10232 (2021).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50, 1-14 (2018).
- García-Rodríguez, I., Sridhar, A., Pajkrt, D., Wolthers, K. C. Put some guts into it: Intestinal organoid models to study viral infection. Viruses. 12, 1288 (2020).
- Stanifer, M. L., et al. Asymmetric distribution of TLR3 leads to a polarized immune response in human intestinal epithelial cells. Nature Microbiology. 5, 181-191 (2020).
- Lees, E. A., et al. Using human induced pluripotent stem cell-derived intestinal organoids to study and modify epithelial cell protection against salmonella and other pathogens. Journal of Visualized Experiments: JoVE. (147), e59478 (2019).
- Mahe, M. M., Sundaram, N., Watson, C. L., Shroyer, N. F., Helmrath, M. A. Establishment of human epithelial enteroids and colonoids from whole tissue and biopsy. Journal of Visualized Experiments: JoVE. (97), e52483 (2015).
- Fujii, M., et al. Human Intestinal organoids maintain self-renewal capacity and cellular diversity in niche-inspired culture condition. Cell Stem Cell. 23 (6), 787-793 (2018).
- Bartfeld, S., Clevers, H. Organoids as model for infectious diseases: Culture of human and murine stomach organoids and microinjection of Helicobacter Pylori. Journal of Visualized Experiments: JoVE. (105), e53359 (2015).
- Hernandez, P. P., et al. Interferon-lambda and interleukin 22 act synergistically for the induction of interferon-stimulated genes and control of rotavirus infection. Nature Immunology. 16, 698-707 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados