JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
살아있는 포유류 세포의 세포에서 단일 막 단백질의 다 색 지역화 현미경
요약
여기, 우리 살아있는 세포의 세포에 단일 막 단백질의 다 색 지역화에 대 한 프로토콜을 제시. Fluorophores 연결할 자체 라벨 단백질 사용 됩니다. 단백질, 세포 기관이 동일한의 다른 막 구획에 있는 ~ 18의 정밀도로 지역화할 수 nm.
초록
Subcompartments 세포에 있는 단백질의 지역화에 대 한 지식을 그들의 특정 한 기능을 이해 하는 것이 결정적 이다. 여기, 우리는 지역화를 생성 하 고이 단백질의 지도 추적 하 여 단백질을 위해 액세스할 수 있는 microcompartments의 결정에 대 한 수 있도록 슈퍼 해상도 기법 제시. 또한, 멀티 컬러 지역화 현미경으로 지역화 및 추적 프로필 다른 subcompartments에 있는 단백질의 얻을 수 있습니다 동시에. 기술은 살아있는 세포에 대 한 특정 이며 단일 모바일 막 단백질의 반복적인 이미지에 기반. 관심사의 단백질 유전자 특정, 소위 자체 라벨 태그와 융합 된다. 이 태그는 기판 공유 방식으로 반응 하는 효소. 이러한 기판에 활용 된 형광 염료입니다. 형광과 효소 태그 단백질의 반응 레이블이 단백질에 기판 결과 표시. 여기, Tetramethylrhodamine (TMR)와 실리콘 Rhodamine (선생님)는 효소의 기질에 부착 된 형광 염료로 사용 됩니다. NM 범위에 오후에 기질 농도 사용 하 여 하위 화학 량 론 라벨 이루어집니다 뚜렷한 신호 귀착되는. 이러한 신호 ~ 15-27와 지역화는 nm 정밀도. 기술을은 사용할 수 막 투과성 염료와 효소 레이블이 자체적으로 지정 하는 것이 레 퍼 토리에 의해 색상 수는 제한 하는 의하여 단일 분자의 멀티 컬러 이미징 할 수 있습니다. 우리 품질 관리 효소 (Pten)의 지역화를 결정 하 여 기술의 타당성을 보여줍니다-다른 막 단백질에 관하여 처리 하는 동안 다른 미토 콘 드 리아 구획에 니 1 (PINK1)을 유발. 단일 분자 무서 워 또는 공동 추적에 의해 다르게 레이블이 단일 단백질 간의 진정한 실제 상호 작용에 대 한 테스트는 제한 하지만, 낮은 라벨도 두 개의 인접 한 단백질을 동시에 표시 하는 데에 대 한 확률 감소 때문입니다. 기술은 단백질 막 구획에 이미징에 대 한 강한 동안, 대부분의 경우 높은 모바일 수용 성 단백질의 지역화가 결정 적합 하지 않다.
서문
이 프로토콜의 목표는 지역화 하 고 단일 막 단백질 라이브 셀 내부를 추적 하는 이미징 방법 제공. 추적 및 지 방화 현미경 검사 법 (TALM)1,2이 메서드를 호출 하 고. 확률적 광학 재건 현미경 (폭풍)3 와 같이 형광 Photoactivation 지역화 현미경 검사 법 ((F) 팜)4,5, TALM 단일 분자 기반 형광 지역화 기술 이다. 그러나, 그것은 동일의 반복적인 이미지와 함께 조합에 있는 막 단백질의 이동성 표시 계시 한다 다른 위치에 분자 microcompartment 모바일 단백질에 대 한 액세스할 수 있는 방식으로 구분 됩니다. 즉, 단백질의 가능한 국부 세포 기관이의 건축 및 단백질1의 이동성에 의해 설정 됩니다. 메서드는 지역화 및 탄도 이미징 모바일 단백질에 의해 지도 계시 한다 때문에 다양 한 다른 슈퍼 해상도 기법6,,78 보완. 라벨은 se 당 비 형광은 유전자 조작된 융해 단백질을 사용 하 여 기반으로 합니다. 이 융해 단백질은 자체 효소 반응 covalently 염료에 활용 된 기판 레이블이 지정 됩니다. 이 절차는 라벨도 될 수 있는 이점이 있다 추가 된 기판의 크기에 의해 제어. 또한, 그것은 선택한 활용된 염료에 따라 형광의 색상을 다양 하 게 있습니다. 여러 자체 라벨 효소-태그는 사용할 수9. 또 다른 장점은, 사용 하는 자체 효소 태그 라벨 활용된 염료를 일반적으로 더 안정적이 고 밝은 형광 단백질1 와 개별 단백질 따라서 기록 될 수 있습니다 더 이상 보다는 더 정확 하 게 때까지 그들은 표백. 모바일 단백질의 궤적의 녹음 및 확산 계수10,11의 추출에 대 한 수 있습니다.
여기, 우리는 미토 콘 드 리아 멤브레인 단백질, TALM의 타당성 입증 하지만 그것은 또한 다른 세포 유형12,13를 포함 하 여 다른 내부 및 여분 cellular 막 단백질에 적용할 수 있습니다. 우리 다 색 TALM 추가 기존 슈퍼 해상도 형광 현미경 검사 법 기술14,15, 보완성에 다른 subcompartments에 있는 단백질의 동시 구별에 대 한 허용 표시 16. TALM 라이브 셀 이미징17와 호환 됩니다. 사진-물리학 선택한 rhodamines Tetramethylrhodamine (TMR)와 실리콘-Rhodamien (선생님)의 특히 그들의 밝기와 안정성, 수 있게 기록 단일 막 단백질 지역화 (및 궤적) 지도 제공 하는 여러 프레임에 걸쳐. 그러나 모션 블러는 너무 높은 때문에 프레임 마다 수집 된 광자는 적절 한 지역화를 위해 너무 낮은, TALM 높은 확산 계수와 수용 성 단백질의 지역화에 대 한 제한 됩니다. 게다가, TALM phototoxic 효과 감소 예 폭풍 또는 자극 방출 소모 (호텔 위치) 현미경6,7, 보다 전원을 적게 필요 합니다. 이것은 phototoxic 스트레스는 종종 organellar 형태학18 및 따라서 이동성 분석19영향을 때문에 중요 한. 합계에서는, 선물이 살아있는 세포에 다 색 TALM 지역화 현미경 방법 폭풍/호텔 위치 사이의 간격을 채우는 기술 (F) 팜 및 photobleaching 후 단백질 이동성 형광 복구 등을 분석 하는 기법 (졸라)20 / ,21, 형광 상관 분광학 (FCS)22, 그리고 형광 상관 분광학 (FCCS)11,23크로스.
Access restricted. Please log in or start a trial to view this content.
프로토콜
다음 프로토콜 로컬 기관 연구 윤리 위원회의 지침을 따릅니다.
1입니다. 방법
-
세포 배양
- 배양 세포, 예를 들면 HeLa 세포 (인간의 자 궁 경부 암), T25 셀 문화 플라스 크 포함 하는 37 ° C, 5% CO2성장 매체의 5 mL에.
참고: 이미지에 대 한 준비 coverslips (1.3과 1.4 단계 참조)에 셀을 분할 하 고 영상 매체에 유지.
- 배양 세포, 예를 들면 HeLa 세포 (인간의 자 궁 경부 암), T25 셀 문화 플라스 크 포함 하는 37 ° C, 5% CO2성장 매체의 5 mL에.
-
세포 transfection
참고: 사용 가능한 태그 단백질을 안정적으로 나타내는 셀 라인을 피하기 위해 강력한 overexpression24 . 일시적인 transfection에 대 한 플라스 미드 DNA transfection에 대 한 사용 금액을 적응. 예를 들어 Ca2 + 인산 transfection25 를 사용 하는 경우에 셀 (80-90 %confluency) 플라스 미드 DNA의 2.5-5 µ g으로 3.5 cm 세포 문화 접시에 transfect. 더블 transfection를 수행할 때 각 플라스 미드 구조 당 2.5 µ g를 사용 합니다.- 듀얼 컬러 실험에 대 한 하나의 자체 라벨 단백질의 안정적인 식이 셀 라인을 사용 하 고 뚜렷이 다른 자체 라벨 단백질17인코딩 플라스 미드와 transfect.
참고: 여기, 듀얼 컬러 실험, HeLa 세포 사용 되었다 안정적 PINK1-헤일로-태그와 Tom20-fSNAP 셀프 라벨링 단백질을 표현.
- 듀얼 컬러 실험에 대 한 하나의 자체 라벨 단백질의 안정적인 식이 셀 라인을 사용 하 고 뚜렷이 다른 자체 라벨 단백질17인코딩 플라스 미드와 transfect.
-
Coverslips의 청소
- 장소는 비 커에 coverslips입니다. coverslips를 포함 하는 비 커에 H2O의 30 mL를 추가 하 고 부드럽게 흔들어 그들의 표면에서 먼지를 제거 하.
- 핀셋으로는 coverslips를 수집 하 고 질소의 흐름과 함께 그들을 건조.
- 플라즈마 청소, 예를 들어, coverslips의 표면에 유기 오염을 제거 합니다.
참고: 추가 유리 재료의 오염을 피하기 위해, 처리는 coverslips의 중 장갑을 착용 합니다.
주의: coverslips 플라즈마 청소 여 청소를 하는 경우는 coverslips의 위 측에만 청소; 폴 리-L-리-폴 리 에틸렌 글리콜-아르기닌-글리신-aspartate (PLL-말뚝-RGD) (섹션 1.4)로 코팅 한 셀 시드 (섹션 1.5)이이 측면을 사용 합니다.
-
PLL-말뚝-RGD와 Coverslip 코팅
참고: PLL-말뚝-RGD는 폴 리 에틸렌 글리콜 (3000 다)와 cysteine-glycine-arginine-glycine-aspartate-serine (CGRGDS) 펩 티 드 연결 된 폴 리-L-리 신 (PLL) 파생. PLL 부정 청구 유리 표면에 고 못 브러시를 형성 한다. 충전된 형광 염료의 불특정 바인딩 대폭 줄어듭니다. 또한, RGD 모티브 integrin 수용 체의 신호 펩 티 드를 모방 하 고 그로 인하여 그렇지 않으면 쉽게 고착 하지 않는 셀의 integrin 중재 준수를 촉진.- 앞에서 설명한26으로 PLL-말뚝-RGD를 준비 합니다. 즉, 1 mL의 PBS에 PLL-말뚝-RGD의 0.8 mg 분해. 깨끗 한 coverslip의 상부에 PLL-말뚝-RGD 솔루션의 10 µ L를 추가 합니다.
- 두 번째 coverslip 고 (위에 있는 PLL-말뚝-RGD 드롭)는 첫 번째 coverslip;에 거꾸로 그것의 깨끗 한 표면 배치 이 두 개의 coverslips 사이 PLL-말뚝-RGD 솔루션 사이에 발생 합니다.
- 신중 하 게 비 커에 샌드위치 coverslips를 배치 하 고 먼지가 없는 건조 한 환경에서 실 온에서 1 h에 품 어.
- 1 시간 후 물으로 coverslips를 완전히 커버 하는 비이 커에 H2O의 30 mL를 추가 합니다.
- Coverslips는 서로에서 분리 될 때까지 부드럽게 비 커를 흔들.
- 핀셋을 사용 하 여 물에서 coverslips를 수집 하 고 질소 가스의 흐름에 따라 그들을 건조.
참고: 코팅된 coverslips 저장할 수 있습니다 건조, 살 균 유리 뚜껑이 있는 페 트리 접시에에서 며칠에 대 한.
-
이미징에 대 한 시료의 준비
- PLL-말뚝-RGD 코팅 표면 위 35 mm 셀 문화 접시에 단일 코팅된 coverslips 전송 및 영상 위에 매체의 2 개 mL를 추가.
- 영상 매체와 코팅된 coverslip 셀 문화 접시에 2 mL에 각 막 단백질에 자체 라벨 태그를 표현 하는 ~ 500000 trypsinized 셀 (200-500 µ L)를 추가 합니다. 얻기 위해 셀의 균일 분배를 보장 하기 위해 손으로 부드럽게 악수를 균일 하 게 셀 레이어.
- 80 %confluency이 때까지 셀에서 37 ° C, 5% CO2 를 품 어.
참고: 셀 샘플 영상 전과 transfection 전에 1 한 일 시드 되어야 합니다. 안정적으로 표현 하는 관심사의 단백질, 세포 이미징 하기 전에 2 일 시드할 수 있습니다. 나중에, 성장 하는 coverslip에 셀만 몇 군데.
-
태그 단백질의 라벨
참고: 형광 기판 물 무료 DMSO에 해체 해야 한다. 1 µ M 형광 기판의 재고 솔루션을 사용 하 여 최종 레이블 농도 0.2-30 nM17때 좋습니다. 세포 내부 막 단백질 이미징, 막 투과성 형광 기판 사용 합니다.- 따뜻한 물 욕조에 37 ° C에 영상 매체를.
- 뚜껑 2 mL 튜브에 미리 데워 진된 영상 매체의 1 mL를 플라스틱. 1 µ M 재고 솔루션 최종 라벨 솔루션을 준비 하에서 형광 기판의 0.2-30 µ L를 추가 (최종 농도: 0.2-30 nM).
- 소용돌이 10 라벨 솔루션 s.
- 대체는 coverslip에 셀으로 35mm 문화 접시에 있는 매체 (단계 1.5 참조) 준비의 1 mL에 의해 라벨링 솔루션.
- 20-30 분 동안 37 ° C, 5% CO2 라벨 솔루션에서 셀을 품 어.
- 셀 2 mL PBS의 한 번, 다음 두 번 매체 이미지의 2 mL 씻으십시오. 마지막으로, 셀 요리에 신선한 영상 매체의 1 mL를 플라스틱 하 고 적어도 1 시간에 대 한 37 ° C, 5% CO2 배양 기에 다시 샘플을 넣어. 이미징, 전에 영상 매체를 한 번 더 교환 합니다.
참고: 처음으로 실험을 실행 하는 경우 상용 세포 기관이 특정 염료27,28와 세포 얼룩 자체 레이블된 organellar 막 단백질의 올바른 대상으로 확인 합니다. 이 경우에, 자체 라벨 효소에 대 한 기판의 100-200 nM을 사용 하 여 강한 신호를 생성 하도.
-
형광 구슬 샘플의 준비
참고: 광 유를 결정 하 고 다른 채널의 이미지 정렬, 다 색 형광 구슬 (0.1 µ m) 사용 됩니다. 기록 된 이미지와 함께 두 개의 방출 채널에 대 한 관계 변환 매트릭스를 생성 됩니다.- 구슬 순수한 H2o. 1%의 솔루션을 희석
- 장소 5 방울 청소 coverslip에 5 개의 다른 위치에서 형광 구슬 준비 솔루션 (단계 1.3 참조).
- 건조 한 깨끗 한 벤치에 형광 구슬 샘플을 보자.
참고: 샘플 다시 사용할 수 있습니다; 따라서, 오염 방지 4 ° c.에 그것을 유지 하는 알루미늄 호 일로 샘플을 커버
2입니다. 현미경
-
실험적인 체제
참고: 듀얼 컬러 단일 분자 이미징에 대 한 기본적인 현미경 시스템은 거꾸로 현미경에 기반: 그것은 다중 mode 광학 편광 단일 총 내부 반사 (사격)으로 monomode 섬유를 유지를 통해 결합 하는 두 개의 레이저 장비 콘덴서, TIRF, polyband 방출 필터, 이미지 스플리터와 매우 민감한 카메라 (그림 1)를 위한 석유 집중 목표. 사격 콘덴서는 입사 각도의 지속적인 튜닝는 피-, 높은 경사를 전환할 필요 고 적 층된 광학 시트 (매우 경사 얇은 조명 (힐로)29)와 함께 TIRF 흥분 모드 최적화 침투 깊이입니다. 이미지는 매우 민감한 냉각된 검출기 시스템 획득, 예를 들어, 백업 조명 전자 멀티 충전 결합 된 장치 (EMCCD) 카메라 (양자 효율 QE > 90%) 또는 sCMOS 카메라 (QE > 80-90%).- 그 나중 실험, 예를 들어, 10000 프레임 실험에 기록 하는 경우, 비드 샘플 또한 10000 프레임을 기록으로 동일한 조건 하에서 형광 구슬 (단계 2.2 참조) 이미징 하 여 광학 드리프트를 결정 합니다. 광학 드리프트의 결정에 대 한 첫 번째 프레임과 마지막 인수 프레임 (그림 1B)에 구슬의 위치를 비교 합니다. 필요한 경우 나중에 광학 드리프트30 이미지 시리즈를 수정 및/또는 드리프트 안정적인 환경을 사용.
- 주황색과 빨간색 형광 플러스 오렌지 형광 및 빨간 형광에 대 한 적절 한 방출 필터에 대 한 적절 한 색 성 빔 스플리터, 예를 들어, 필터 큐브를 장비. 적당 한 필터 이미지 스플리터를 장비. 두 채널 (그림 1C)에 단일 색상 샘플을 기록 하 여 다른 채널에 신호를 한 채널에서의 가능한 누수를 확인 합니다.
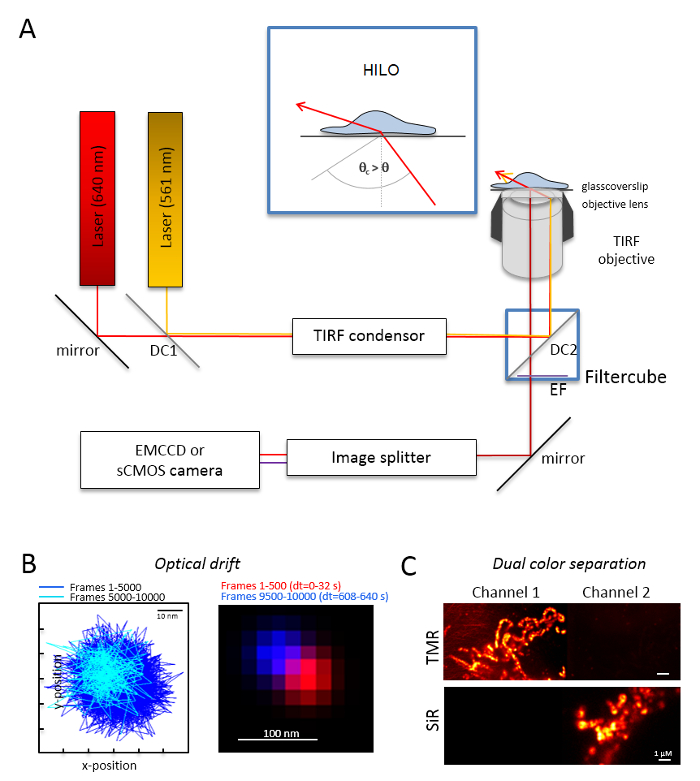
그림 1 : 광학 레이아웃입니다 다 색 추적 및 지역화 (TALM) 오렌지와 빨간색 미터. (A) 두 개 이상 여기 레이저, TIRF 콘덴서, TIRF 적당 한 목표, 이미지 스플리터와 민감한 카메라 거꾸로 현미경 설치. 삽입: 셀 안에 세포를 자극, 입사 빔 각도 설정 되어야 합니다 매우 달성 하기 TIRF에 대 한 중요 한 각도 경사와 광학 시트 조명 (힐로)을 적 층 하는 보다 작은. D c 1: Dichroic 거울 1; D c 2: Dichroic 거울 2입니다. EF: 방출 필터입니다. 다음 실험으로 동일한 프레임 속도 10000 프레임에 대 한 형광 구슬의 위치를 이미징 하 여 광학 드리프트에 (B) 테스트 (여기: 15 Hz). 처음 500 프레임 및 마지막 500 프레임의 연결 된 위치는 드리프트를 보여줍니다. 또한, 빨간색과 파란색에서 첫 번째 및 마지막 프레임의 위치와 병합 된 이미지를 최소한의 드리프트를 보여줍니다. 드리프트 신호의 중심 사이의 거리 시간으로 나눈 총 녹음, 여기 125 오후/미 신호, 여기 TMR 및 선생님의 명확한 구분에 (C) 검사입니다. 두 채널 (채널 1에 TMR)과 채널 2에서 경 3, 000 프레임에서 누적 합계 이미지 생성 되었다. 선생님HTL Tom20 HaloTag와 TMRHTL OxPhos 복잡 한 V-HaloTag에에 붙어 있었다. 색상은 거짓 색상입니다. 바 규모 = 100 nm (B) 및 1 개의 µ m (C). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
-
이미지 분할의 물리적 맞춤 이미지 생성
참고: 장착 하는 coverslip에 준비 된 표본에 대 한 성가 샘플 홀더 수 사용 (그림 2A). 먼지, 등 샘플에 빠지지 않으려면 장소 문화 접시의 뚜껑 느슨하게 챔버, 위에 설치 하는 경우. 동일한 샘플 홀더 형광 구슬 또는 셀; coverslip 마운트를 사용할 수 있습니다. 세포는 군데 추가 0.5-0.8 mL 영상 매체의. 이미지 분할 2로 이미지를 분할 또는 더 괴기 하 게 채널을 분리 하 고 동일한 카메라에 그들 측면-의해-측면을 프로젝트. 이 프로세스는 잠재적으로 통과 뚜렷한 광학 경로 때문 채널 사이의 체계적인 왜곡을 소개 하 고 직접 colocalization 분석을 방해. 따라서, 실제 정렬 및 두 번째, 수행 후 보정 변환 매트릭스를 정렬. 두 정렬 프로세스에 대 한 형광 구슬 homogenously 보기의 필드에 걸쳐 배포 될 한다.- 소계 (PTFE) 사이 샘플 홀더에 형광 구슬 가진 준비 된 샘플을 마운트-링과 빨간 고무 링 (그림 2A).
- 현미경, 모든 하드웨어 구성 요소, 그리고 현미경 검사 법에 필요한 모든 소프트웨어를 시작 합니다.
- 소 프로 파 놀과 접촉 보풀 조직 지우기와 목적과 coverslip 아래쪽을 청소. 다음 두 항목 모두 신선한 보풀 조직으로 건조. 대물 렌즈의 눈동자에 침수 기름 작은 물방울을 놓습니다.
- 하는 coverslip 하단의 연락처 기름 현미경 스테이지에 비드 샘플 샘플 홀더를 배치 합니다. 전송 빛 또는 레이저를 사용 하 여 구슬에 초점.
- 두 개의 형광 채널에서 비슷한 신호 강도 달성 하기 위해 두 여기 레이저의 파워를 조정 합니다. 많은 독특한 형광 신호 영역에 대 한 검색.
- 카메라 제어 소프트웨어를 사용 하 여 형광 채널의 병합 된 보기를 생성 합니다. 다음 수동으로 기울기 두 형광 채널 (그림 2B)에서 신호의 최고의 오버레이 달성 하기 위해 이미지 스플리터의 내부 거울 이미지 분배기에 나사를 사용 합니다.
참고: 주의! 카메라의 동적 범위를 초과 하지 마십시오.
-
맞춤 소프트웨어 공간 변환을 수행 하 여 괴기 하 게 분리 된 채널을
참고: 다음 부분 후 교정 맞춤 및 우리의 소프트웨어 플러그인 (요청 시 사용 가능)와 지역화 절차를 보여 줍니다.- TIRF 현미경 제어 소프트웨어를 시작 하 고 라이브 스트림 모드에서 개별 채널을 표시 하려면 선택 합니다. 모든 채널 (그림 2C)에 스냅숏 이미지 흥미로운 형광을 가져가 라.
- 이 스냅숏 이미지를 사용 하 여 변환 매트릭스를 생성 ( 그림 2참조).
참고: 변환 매트릭스 공간 변환, 일반적으로 유사 하나, 번역 (단일 포인트 소스 두 채널 사이에서 신호 발산)에 대 한 해결을 위해 사용 됩니다. - 소프트웨어 분석 플러그인을 시작 ( 그림 2C참조 하십시오 우리의 실험실에서 요청 시 얻어질 수 있다).
- 소프트웨어에 형광 구슬 (단계 2.2 참조) 이전에 기록 된 듀얼 컬러 이미지를 로드 합니다. 형광 채널의 사용된 방향을 선택 합니다. '예' '이미지 보정'에 대 한 질문 받을 때 클릭 하 고 이전 찍은 스냅샷 선택 합니다.
- 단위 변환 요인 (픽셀 크기, 프레임 속도, 광자 변환 인수)를 정의 하려면 "장치 관리자"를 엽니다.
- "지역화 관리자"를 엽니다. 지점 확산 기능 (PSF)를 먼저 확인 합니다. 단추: "PSF 반경". "PSF 견적" 창이 열리면 정의 숫자 조리개와 최대 방출. 시작 "견적 PSF 반경"를 클릭 하 여. 평가 상자를 얻은 실험 PSF. 정의 수락, 디플레이션 루프의 수 및 얼마나 많은 코어 컴퓨터의 계산을 위해 사용 된다. "지역화" 2D 대칭 가우스 함수 (그림 2C) 단일 입자의 강도 분포를 피팅 시작을 누릅니다.
- "수락" 얻은 실험 PSF. 정의 평가 상자, 디플레이션 루프의 수 및 얼마나 많은 코어 컴퓨터의 계산을 위해 사용 된다. "지역화" 2D 대칭 가우스 함수 (그림 2C) 단일 입자의 강도 분포를 피팅 시작을 누릅니다.
- "교정 관리자"를 엽니다. 두 채널의 렌더링된 병합 된 이미지에서 원래 신호 및 지역화 된 센터 표시 됩니다. "" 모드를 선택 합니다. 수동으로 연결 선을 그려 같은 형광 구슬에서 유래 하는 두 개의 채널에 지역화 된 센터의 해당 쌍을 연결 합니다.
- 보기의 필드를 통해 모든 배포 해당 신호를 연결 합니다. 이 후, "동의"를 누릅니다. 보정을 저장 합니다.
참고: 공간 변환 각 형광 구슬에서 샘플 이며 사이 보간됩니다. 추출 된 변환 함수 이후에 그들은 그들의 지역화 정밀도 내에서 중첩 되도록 실험 듀얼 컬러 단일 분자 지 방화를 수정 하는 데 사용 되는 변위 필드 Δr(x,y)를 나타냅니다. 공간 변환 매트릭스는 일반적으로 번역, 스케일링, 및 회전 나노미터의 정확도와 채널 간의 해결 관계 한 고이 수동 일대일 매핑 (그림 2C)에서 추정 될 수 있다.
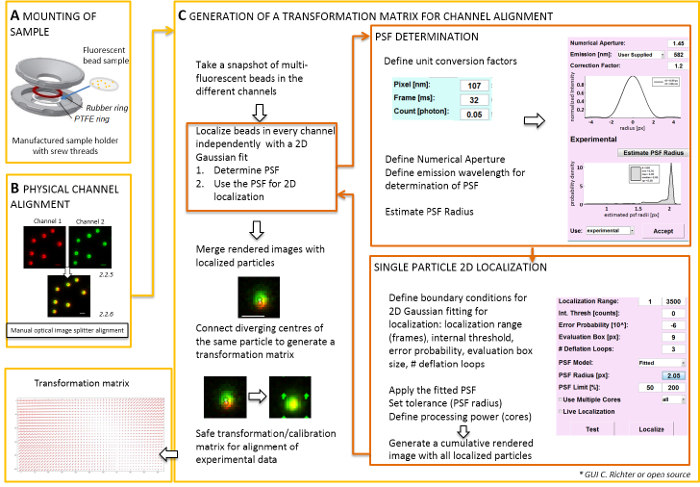
그림 2 : 듀얼 컬러 정렬에 대 한 워크플로. (A) 형광 구슬 coverslip 샘플 홀더는 PTFE와 고무 링 사이 마운트되어. 다음 챔버의 상단 및 하단 부분 함께 나올입니다. 이미지 분할에 의해 생성 되는 채널 조회의 (B) 실제 맞춤입니다. 두 개의 채널에 구슬 (0.1 µ m)에서 형광 신호를 기록 (녹색 및 빨강, 거짓 색) 병합 됩니다. 다른 신호의 최고의 오버레이 달성 될 때까지 광학 이미지 분배기에 해당 나사는 수동으로 설정 되어 (노란색, 하단 패널). (C) post-processive 채널 정렬에 대 한 변환 매트릭스의 세대. 입자의 정확한 지역화, 지점 확산 기능 (PSF) 방출 파장 및 목표의 숫자 조리개에 대 한 의존에 확인할 필요가 있다. 센터는 PSF의 맞는 대칭 2 차원 가우스에 의해 분석 된 강도 프로 파일에 의해 결정 수 있습니다. 신호 피크의 결과 지역화 다음 원래, 흐리게 신호에 예상 된다. 병합된 이미지에 두 개의 채널에서 신호의 지역화 된 센터 나중 실험 데이터의 post-processive 정렬에 사용 되는 변환 매트릭스를 생성에 연결 됩니다. 스케일 바 = 1 개의 µ m (B, C). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
-
미토 콘 드 리아 멤브레인 단백질의 단일 분자 이미징
참고: 모든 실험 상 온에서 수행 됩니다. T-세포 또는 비 부착 한 세포 해야 될 움직일 수 agarose에서31이미징 하기 전에.- 고무 및 PTFE 반지 (그림 3A) 사이 coverslip에 부착 세포 견본을 탑재 합니다. 0.5-0.8와 챔버를 입력 매체를 이미징 하는 mL.
- 2.2.2–2.2.5 단계를 반복 합니다.
- 조명 각도 TIRF 현미경 힐로 시트32 (힐로 모드, 그림 1A)을 통해 관심의 특정 영역을 흥분 시키기 위해 사격 모드에 대 한 중요 한 각도 보다 작은 입사 각도 만드는 소프트웨어 제어를 조정 합니다.
주의: 레이저 빔으로 직접적인 눈 접촉을 피하십시오! - EM 이득을 설정 하 고 프레임 마다 충분 한 광자를 수집 하는 실험에 대 한 적당 한 노출 시간을 선택 합니다.
- 레이저 파워 달성 이후 지역화 정밀 직접 S/N33 (그림 3C)에 해당 (S/N) 비율 (그림 3B), 소음에 높은 신호를 설정 합니다.
- 겹치지 않는, 길쭉한 미토 콘 드리 아와 단일 분자 신호 (3D 그림; 세포 주변에 있는 영역을 찾기 보조 비디오 1)입니다. 없는 단일 분자 신호 표시 되는 경우 단일 분자 신호 (그림 3E)의 외관에 결과 표백까지 기다립니다.
- 신호 수까지 기록 합리적인 계속 (보통 형광 염료, 그림 3 층의 표백 동작에 따라 1000-10000 프레임) 너무 낮습니다.
- 영상 처리 소프트웨어 및 미토 콘 드리 아 구조 확인 적어도 1000 기록 된 프레임 (그림 3)의 이미지를 렌더링된 누적 합계를 생성 하 여 시작 합니다.
참고: 가장 빠른 가능한 프레임 속도 판독 영역에 의해 결정 됩니다. 1 개 채널 보기의 필드 256 x 512 픽셀, 그리고 쿼드-색상 256 x 256 픽셀을 위해 듀얼 컬러 이미지 분배기 (512 x 512 픽셀)으로 감소 된다. 따라서, 두 가지 색상에 대 한 이미지 분배기를 사용 하 여, 이것이 30 Hz. 설정 프레임 전송 모드는 가장 낮은 가능한 판독 시간을 달성 하기. - 소프트웨어 분석 플러그인 및 부하 원시 데이터를 시작 합니다. 채널 방향 및 로드 이미지를 선택 합니다. 단계의 2.3.9 때 "보정 이미지"에 대 한 변환 매트릭스를 사용 합니다. 채널 별도로 표시 됩니다.
- 각 채널에 대 한 단위 변환 요소를 정의 하기 전에 "장치 관리자"를 엽니다. 각 채널에 대 한 "지역화 관리자"를 엽니다. 다음 계산에 사용 되는 컴퓨터의 얼마나 많은 코어 설정 및 사용 조건에 대 한 이론적인 PSF 추가 평가 상자, 디플레이션 루프의 수를 정의 합니다. 마지막으로, 지역화 된 단일 입자 (그림 3 H;를 "지역화"를 누릅니다 보조 비디오 2)입니다.
- 참고 프로그램 마지막으로 보여주는 누적 해상도 이미지를 생성 합니다 모든 입자 (그림 3I) 지역화.
- 예를 들어, 오픈 소스 소프트웨어 또는 요청 시 사용할 수 있는 소프트웨어에 의해 분석을 수행 합니다.
- 예를 들어, 다중 표적 추적10 모두 지역화 된 채널에 단일 분자를 추적
참고: 단계 2.4.13 경계 조건을 올바르게 설정 하는 관심사의 단백질의 diffusibility에 대 한 예비 (실험) 지식이 필요 합니다. 일반적으로, 올바른 경계 조건을 찾는 반복 프로세스입니다.
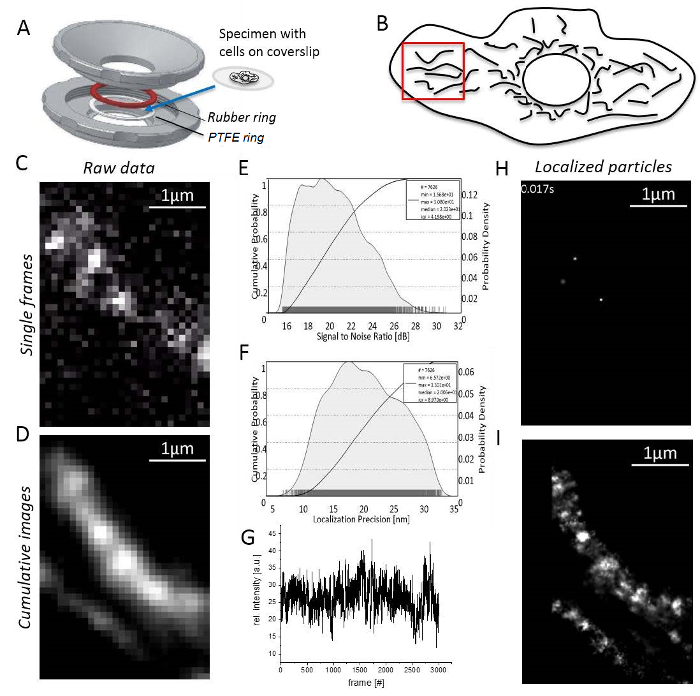
그림 3 : 단일 분자 지 방화 현미경 검사 법 중 단계. (A) A coverslip 표본으로 만든 샘플 홀더 (J. Bereiter에 의해 한 설계)의 상단 및 하단 부분 (회색) 사이 탑재. (적색) 고무 링 및 PTFE 반지 (화이트) 인감 시스템에서 위와 아래는 coverslip 때 샘플 홀더 부품 볼트 함께. TMR 신호 잡음 비율 (B) 신호. (C) 모든 지역화 된 입자에 대 한 지역화 정밀 히스토그램을 계산. (D) 이미징, 여기, 명확 하 게 분리 된 미토 콘 드리 아와 세포 주변에 대 한 합리적인 영역의 선택. (E) 녹화 및 이미지 처리: 고유한 단일 분자 신호와 단일 프레임 표시 됩니다 (여기서, CV-HaloTag/TMRHTL 의 단일 분자 기록 했다). (F) 강도의 TMR 녹화 시간 동안. (G) 3000 프레임, 처리 되지 않은 이미지를 누적 합계. (H) 입자의 이력서-HaloTag/TMRHTL 단일 프레임에서 2D 가우스 기능 지역화. (나) 누적, 합계 이미지 3000 프레임에서 모든 지역화 된 CV-HaloTag/TMRHTL 입자를 렌더링. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
Access restricted. Please log in or start a trial to view this content.
결과
멀티 컬러 이미징 및 colocalization 분석은 단백질의 하위 organellar 지역화가 결정을 도울 수 있다. 우리가 시연이 이전 cytosolic 인산 가수분해 효소와 tensin homologue, PINK1, 미토 콘 드 리아 프로 테아 제17에 의해 그것의 처리로 인해 다른 하위-미토 콘 드리 아 위치는. PINK1 미토 콘 드 리아 기능34,35보장 하는 중요 한 ?...
Access restricted. Please log in or start a trial to view this content.
토론
여기, 듀얼 컬러 단일 분자 지역화 모바일 막 단백질의 기술을 제시 했다. 다음 프로토콜, 막 단백질 자체 라벨 TMR 및 그들의 각각 기판에 활용 된 선생님 rhodamine 염료와 반응 하는 단백질에 융합 된다. Rhodamine 밝은 photostable 염색 하 고 반복적인 이미징1따라서 허용 합니다. 성공적인 성능에 대 한 여러 조건 및 중요 한 주제 마음에 보관 해야 합니다.
첫째, 그?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 없다.
감사의 말
저자 생물 물리학 그룹 및 Jacob Piehler Wladislaw 콜 기술 지원 및 자료의 준비에 대 한 지속적인 지원과 사용 하기 위해 현미경을 제공 하기 위한 CellNanOs 보드 나 브루 크의 대학에 감사 하 고 싶습니다. 프로젝트는 SFB 944에 의해 투자 되었다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| (2-(4-(2-hydroxyethyl)-1-piperazinyl)-ethanesulfonic acid, 1 M) (HEPES) | Biochrom | #1104E | |
| DC1: Dichroid beam splitter | Chroma | 640 dcxr | NC506031 |
| DC2: Polychroic Mirror, beamsplitter | Chroma | zt405/488/561/640rpc | discontinued |
| Dulbecco´s Phosphate-Buffered Saline (PBS) 1x (w/o Ca & Mg) | Sigma-Aldrich & Co. | #RNBF8311 | |
| Earle´s MEM without phenol red, without L-Glutamine and without NaHCO3 containing 1% FBS, 0.1% HEPES, 0.1% NEAA, 0.1% Alanyl-L-Glutamine and 34.78% sodium hydrogen carbonate (NaHCO3 0.75g/l) | Imaging medium | ||
| Earle´s minimum essential medium (MEM) with phenol red, containing 1% Fetal Bovine Serum Superior (FBS), 0.1% HEPES (2-(4-(2-hydroxyethyl)-1-piperazinyl)-ethanesulfonic acid, 1 M), and 0.1% non-essential amino acids (NEAA) | Growth medium | ||
| EF: Emission filter quadbandpass | AHF analysentechnik | F72-866 | Brightline HC 446 nm/523 nm/600 nm/677 nm |
| EMCCD camera | Andor | Andor iXON 897 | EMCCD camera |
| Emission filter QuadView filter cubes, orange | AHF analysentechnik | F39-637 | bandpass 582 - 619 nm |
| Emission filter QuadView filter cubes, red | Chroma | bandpass 655 - 725 nm (HQ 690/70) | |
| FBS (Fetal bovine serum) superior | Biochrom | S0615 | |
| Fluorescent beads: TetraSpeck™ Microspheres, 0.1 µm, fluorescent blue/green/orange/dark red | Thermo Fisher Scientific | T7279 | fluorescent microspheres |
| Glutamine | Biochrom | #0951C | |
| HeLa cells | DSMZ | ACC-57 | Cervical carcinoma cells from patient Henrietta Lacks |
| Hela cells CI::paGFP, stable | Muster et al., PLOSOne 2010 | ||
| Hela cells CV g::Halo7-Tag, stable | Appelhans et al., NanoLett 2012 | ||
| Hela cells Tom20::Halo7-Tag, stable | Appelhans et al., NanoLett 2012 | ||
| Image splitter | Photometrics | Dual-View QV2 | image splitter emission |
| Imaging processing software | ImageJ2 / Fiji | freeware | |
| Immersion Oil - ImmersolTM 518 F (ne = 1.518, ve = 45) | Carl Zeiss Jena GmbH | 444960-0000-000 | |
| Inverted epifluorescence microscope | Olympus IX-71/73/83 | ||
| Laser 561 nm, 200 mW | CrystaLaser | CL-561-200 | 561 nm emission |
| Laser 642 nm, 140 mW | Omicron | Luxx-642-140 | 642 nm emission |
| MATLAB | MathWorks | version R2013a | |
| MEM with Earle's Balanced Salt Solution 2.2 g/L NaHCO3, stable glutamine w/o PR | Biochrom | FG-0385 | |
| MEM with Earle's Balanced Salt Solution with 2.2 g/L NaHCO3, stable glutamine, Phenolred | Biochrom | FG-0325 | |
| MitoTracker® Deep Red FM | Thermo Fisher Scientific | M22426 | dye |
| MitoTracker® Green FM | Thermo Fisher Scientific | M7514 | dye |
| Multi-mode-optical polarization maintaining monomode fiber | Pointsource/Qioptiq | KineFLEX | |
| NHS-PEG-MAL, Rapp Polymer | Rapp Polymere GmbH Tübingen | coverslip coating | |
| non-essential amino acids (NEAA) | Biochrom | #0802E | |
| PEG 800 (Polyethylene glycol) 10 % | Carl Roth GmbH | Art No. 0263.1 | coverslip coating |
| Penicillin/Streptomycin | Biochrom | #0122E | |
| Plasmid for PINK1-Halo7-Tag expression | Beinlich et al., ACS Chemical Biology 2015 | ||
| Poly-L-lysine (1.2 mg/ml) | Sigma-Aldrich & Co. | Cat. No.P9155 | coverslip coating |
| RGD Peptide (Ac-CGRGDS-COOH) | Coring System Diagnostix GmbH, Gernsheim | coverslip coating / Intergrin receptor motif | |
| Silicon Rhodamine linked to HaloTag®-Ligand (SiRHTL) | personal gift from Kai Johnson | dye | |
| Software analysis plugin | self-written C. P. Richter, Biophysik Osnabrück | SLIMFAST 16g | |
| Tetramethylrhodamine / SNAP-Cell® TMR-Star linked to SNAP-Ligand (TMRstar) | New England Biolab® | S9105S | dye |
| Tetramethylrhodamine linked to HaloTag®-Ligand (TMRHTL) | Promega | G8251 | dye |
| TIRF condensor | Olympus | Cell^TIRF MITICO System | TIRF condensor |
| TIRF microscope controlling software | Olympus cellSens 1.12 | ||
| TIRF objective | Olympus | 150x oil objective (N.A. 1.45; Olympus UAPO) | |
| Trypsin/EDTA 10x | Biochrom | #0266 | |
| Water H2O 99,5 % Rotipuran® Low organic | Carl Roth GmbH | Art. No. HN57.1 |
참고문헌
- Appelhans, T., Richter, C., Wilkens, V., Hess, S., Piehler, J., Busch, K. Nanoscale organization of mitochondrial microcompartments revealed by combining tracking and localization microscopy. Nano Letters. 12 (2), 610-616 (2012).
- Appelhans, T., Busch, K. Single Molecule Tracking and Localization of Mitochondrial Protein Complexes in Live Cells. Methods Mol Biol. 1567, 273-291 (2017).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods. 3 (10), 793-795 (2006).
- Gould, T. J., Verkhusha, V. V., Hess, S. T. Imaging biological structures with fluorescence photoactivation localization microscopy. Nat Protoc. 4 (3), 291-308 (2009).
- Pennacchietti, F., Gould, T. J., Hess, S. T. The Role of Probe Photophysics in Localization-Based Superresolution Microscopy. Biophys J. 113 (9), 2037-2054 (2017).
- Wegel, E., Göhler, A., Lagerholm, B. C., Wainman, A. Imaging cellular structures in super-resolution with SIM, STED and Localisation Microscopy: A practical comparison. Scientific reports. , Available from: https://www.nature.com/articles/srep27290?WT.feed_name=subjects_physical-sciences (2016).
- Pellett, P., et al. Two-color STED microscopy in living cells. Biomedical Optics Express. 2 (8), (2011).
- Ishigaki, M., et al. STED super-resolution imaging of mitochondria labeled with TMRM in living cells. Mitochondrion. 28, 79(2016).
- Liss, V., Barlag, B., Nietschke, M., Hensel, M. Self-labelling enzymes as universal tags for fluorescence microscopy, super-resolution microscopy and electron microscopy. Scientific Reports. 5, 17740(2015).
- Sergé, A., Bertaux, N., Rigneault, H., Marguet, D. Dynamic multiple-target tracing to probe spatiotemporal cartography of cell membranes. Nat Methods. 5 (8), (2008).
- Appelhans, T., Busch, K. B. Dynamic imaging of mitochondrial membrane proteins in specific sub-organelle membrane locations. Biophysical reviews. 9 (4), 345-352 (2017).
- Wilmes, S., et al. Triple-color super-resolution imaging of live cells: resolving submicroscopic receptor organization in the plasma membrane. Angewandte Chemie. 51 (20), 4868-4871 (2012).
- Niewidok, B., et al. Single-molecule imaging reveals dynamic biphasic partition of RNA-binding proteins in stress granules. J Cell Biol. , (2018).
- Wurm, C. A., Jakobs, S. Differential protein distributions define two sub-compartments of the mitochondrial inner membrane in yeast. FEBS Lett. 580 (24), 5628-5634 (2006).
- Schmidt, R., Wurm, C. A., Punge, A., Egner, A., Jakobs, S., Hell, S. W. Mitochondrial cristae revealed with focused light. Nano Lett. 9 (6), 2508-2510 (2009).
- Kukat, C., Wurm, C. A., Spahr, H., Falkenberg, M., Larsson, N. G., Jakobs, S. Super-resolution microscopy reveals that mammalian mitochondrial nucleoids have a uniform size and frequently contain a single copy of mtDNA. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13534-13539 (2011).
- Beinlich, F., Drees, C., Piehler, J., Busch, K. Shuttling of PINK1 between Mitochondrial Microcompartments Resolved by Triple-Color Superresolution Microscopy. ACS chemical biology. 10 (9), 1970-1976 (2015).
- Shim, S., et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes. Proceedings of the National Academy of Sciences. 109 (35), 13978-13983 (2012).
- Sbalzarini, I., Mezzacasa, A., Helenius, A., Koumoutsakos, P. Effects of Organelle Shape on Fluorescence Recovery after Photobleaching. Biophysical Journal. 89 (3), 1482-1492 (2005).
- Reits, E., Neefjes, J. From fixed to FRAP: measuring protein mobility and activity in living cells. Nature Cell Biology. 3 (6), E145-E147 (2001).
- Goehring, N., Chowdhury, D., Hyman, A., Grill, S. FRAP Analysis of Membrane-Associated Proteins: Lateral Diffusion and Membrane-Cytoplasmic Exchange. Biophysical Journal. 99 (8), 2443-2452 (2010).
- Bacia, K., Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: principles and applications. Cold Spring Harbor protocols. 2014 (7), 709-725 (2014).
- Sukhorukov, V., Dikov, D., Busch, K., Strecker, V., Wittig, I., Bereiter-Hahn, J. Determination of protein mobility in mitochondrial membranes of living cells. Biochimica et biophysica acta. 1798 (11), 2022-2032 (2010).
- Kim, T. K., Eberwine, J. H. Mammalian cell transfection: the present and the future. Anal Bioanal Chem. 397 (8), 3173-3178 (2010).
- Graham, F. L., van der Eb, A. J. A new technique for the assay of infectivity of human adenovirus 5 DNA. Virology. 52 (2), 456-467 (1973).
- Wedeking, T., et al. Spatiotemporally Controlled Reorganization of Signaling Complexes in the Plasma Membrane of Living Cells. Small. 11 (44), 5912-5918 (2015).
- Mironov, S. L., Ivannikov, M. V., Johansson, M. [Ca2+]i signaling between mitochondria and endoplasmic reticulum in neurons is regulated by microtubules. From mitochondrial permeability transition pore to Ca2+-induced Ca2+ release. J Biol Chem. 280 (1), 715-721 (2005).
- Poot, M., et al. Analysis of mitochondrial morphology and function with novel fixable fluorescent stains. J Histochem Cytochem. 44 (12), 1363-1372 (1996).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells (vol 5, pg 159). Nature Methods. 5 (5), 455(2008).
- Elmokadem, A., Yu, J. Optimal Drift Correction for Superresolution Localization Microscopy with Bayesian Inference. Biophys J. 109 (9), 1772-1780 (2015).
- Barlag, B., et al. Single molecule super-resolution imaging of proteins in living Salmonella enterica using self-labelling enzymes. Sci Rep. 6, 31601(2016).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nature Methods. 5 (2), 159-161 (2008).
- Mortensen, K. I., Churchman, L. S., Spudich, J. A., Flyvbjerg, H. Optimized localization analysis for single-molecule tracking and super-resolution microscopy. Nat Methods. 7 (5), 377-381 (2010).
- Jin, S., Youle, R. J. PINK1- and Parkin-Mediated Mitophagy at a Glance. Journal of Cell Science. 125, 795-799 (2013).
- Yamano, K., Youle, R. J. PINK1 is degraded through the N-end rule pathway. Autophagy. 9 (11), 1758-1758 (2013).
- Wiedemann, N., et al. Machinery for protein sorting and assembly in the mitochondrial outer membrane. Nature. 424 (6948), 565-571 (2003).
- Thompson, R., Larson, D., Webb, W. Precise Nanometer Localization Analysis for Individual Fluorescent Probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유