JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
필드 이펙트 바이오센싱 기술을 이용한 분자 샤페론 Hsp90과 클라이언트 단백질 키나제 Cdc37 사이의 생체분자 상호작용 탐구
요약
전계 효과 바이오 센싱 (FEB)은 생체 분자 상호 작용을 검출하기위한 라벨이없는 기술입니다. 결합 표적이 고정되는 그래핀 바이오센서를 통해 전류를 측정한다. FEB 기술은 Hsp90과 Cdc37 사이의 생체 분자 상호 작용을 평가하는 데 사용되었으며 두 단백질 간의 강력한 상호 작용이 검출되었습니다.
초록
생체 분자 상호 작용은 기능적으로 관련된 생물학적 사건을 조절하고 조정함으로써 수많은 세포 과정에서 다양한 역할을합니다. 단백질, 탄수화물, 비타민, 지방산, 핵산 및 효소와 같은 생체 분자는 생명체의 기본 빌딩 블록입니다. 그들은 무수한 삶의 사건을 동기화하기 위해 생물 시스템의 복잡한 네트워크로 모입니다. 단백질은 전형적으로 그들의 기능을 수행하기 위해 복잡한 상호작용 네트워크를 이용한다; 따라서 세포 및 유기체 수준에서 세포에서의 중요성을 밝히기 위해 그러한 상호 작용을 평가하는 것이 필수적입니다. 이 목표를 향해 우리는 특정 생체 분자 상호 작용을 결정하기 위해 빠르게 부상하는 기술인 FEB(Field Effect BioSensing)를 도입합니다. FEB는 벤치탑, 라벨 프리, 신뢰할 수있는 생체 분자 검출 기술로서 특정 상호 작용을 결정하고 고품질의 전자 기반 바이오 센서를 사용합니다. FEB 기술은 바이오 센서 표면에 사용되는 생체 적합성 나노 물질로 인해 나노 몰 범위의 상호 작용을 모니터링 할 수 있습니다. 개념 증명으로서, 열 충격 단백질 90 (Hsp90)과 세포 분열 사이클 37 (Cdc37) 사이의 단백질 - 단백질 상호 작용 (PPI)이 해명되었다. Hsp90은 ATP 의존성 분자 샤페론으로 많은 단백질의 폴딩, 안정성, 성숙 및 품질 관리에 필수적인 역할을하므로 여러 가지 중요한 세포 기능을 조절합니다. Cdc37은 단백질 키나제 특이적 분자 샤페론으로 간주되며, 이는 단백질 키나아제를 Hsp90으로 특이적으로 인식하고 모집하여 하류 신호 전달 경로를 조절하기 때문입니다. 따라서 Cdc37은 Hsp90의 공동 샤페론으로 간주됩니다. 샤페론-키나제 경로(Hsp90/Cdc37 복합체)는 세포 성장을 촉진하는 다발성 악성종양에서 과다활성화된다; 따라서 암 치료의 잠재적 인 표적입니다. 본 연구는 Hsp90/Cdc37 모델 시스템을 이용한 FEB 기술의 효율성을 입증한다. FEB는 두 단백질 사이에 강한 PPI를 검출하였다(세 번의 독립적인 실험에서 0.014 μM, 0.053 μM 및 0.072 μM의 KD 값 ). 요약하면, FEB는 라벨이 없고 비용 효율적인 PPI 검출 플랫폼으로, 빠르고 정확한 측정을 제공합니다.
서문
생체 분자 상호 작용 :
단백질은 유기체의 필수적인 부분이며 세포 대사, 세포 구조, 세포 신호, 면역 반응, 세포 부착 등과 같은 수많은 분자 경로에 참여합니다. 일부 단백질은 독립적으로 기능을 수행하지만, 대부분의 단백질은 적절한 생물학적 활성을 조정하기 위해 결합 인터페이스를 사용하여 다른 단백질과 상호 작용합니다1.
생체분자 상호작용은 주로 관련된 단백질의 뚜렷한 구조적 및 기능적 특성(2)에 기초하여, 예를 들어, 단백질 표면, 복합체 안정성, 또는 상호작용의 지속성(3)에 기초하여 분류될 수 있다. 필수 단백질과 생체 분자 상호 작용에서의 역할을 확인하는 것은 분자 수준 4에서 생화학 적 메커니즘을 이해하는 데필수적입니다. 현재, 이러한 상호작용5를 검출하기 위한 다양한 접근법이 있다: 시험관내 6, 실리코7, 살아있는 세포8, 생체외9, 및 생체내10에서 각각은 그 자신의 강점과 약점을 갖는다.
생체내 분석은 실험 도구(11)로서 전체 동물을 사용하여 수행되고,the ex vivo 분석은 자연 조건의 최소한의 변화를 제공함으로써 조절된 외부 환경에서 조직 추출물 또는 전체 기관(예를 들어, 심장, 뇌, 간)에 대해 수행된다. 생체내 및 생체외 연구의 가장 일반적인 적용은 그들의 전반적인 안전성 및 효능을 보장함으로써 인간 시험 전에 잠재적인 약리학적 제제의 약동학, 약력학 및 독성 효과를 평가하는 것이다12.
생체분자 상호작용은 또한 살아있는 세포 내에서 검출될 수 있다. 살아있는 세포를 이미징하면 특정 생화학 적 경로의 반응을 실행할 때 동적 상호 작용을 관찰 할 수 있습니다13. 더욱이, 생체발광 또는 형광 공명 에너지 전달과 같은 검출 기술은 세포(14) 내에서 이러한 상호작용이 언제 어디서 발생하는지에 대한 정보를 제공할 수 있다. 살아있는 세포에서의 검출은 중요한 세부 사항을 제공하지만, 이러한 검출 방법론은 광학 및 라벨에 의존하며, 이는 네이티브 생물학을 반영하지 않을 수 있습니다. 그들은 또한 시험관 내 방법보다 덜 제어되며15를 수행하기 위해 전문 지식이 필요합니다.
실리코 전산 방법은 주로 시험관내 실험 전에 표적 분자의 대규모 스크리닝에 사용된다. 계산 예측 방법, 컴퓨터 기반 데이터베이스, 분자 도킹, 정량적 구조-활성 관계, 및 기타 분자 역학 시뮬레이션 접근법은 실리코 도구(16)에서 잘 확립된 것 중 하나이다. 힘든 실험 기술과 비교할 때, in silico 도구는 높은 감도로 쉽게 예측을 할 수 있지만 예측 성능의 정확도는 떨어집니다17.
시험관내 분석은 표준 생물학적 맥락 밖의 미생물 또는 생물학적 분자로 수행됩니다. 시험관 내 방법을 통해 생체 분자 상호 작용을 묘사하는 것은 단백질 기능과 세포 기능의 복잡한 네트워크 뒤에있는 생물학을 이해하는 데 중요합니다. 바람직한 분석 방법론은 단백질의 고유 특성, 운동 값, 및 상호작용의 모드 및 강도(18,19)에 따라 선택된다.
Hsp90/Cdc37 상호 작용:
Hsp90과 Cdc37을 연결하는 샤페론-키나제 경로는 종양 생물학(20)에서 유망한 치료 표적이다. Hsp90은 세포 주기 조절, 단백질 조립, 세포 생존 및 신호전달 경로에서 중심적인 역할을 한다. 그들의 기능을 위해 Hsp90에 의존하는 단백질은 Cdc37과 같은 공동 샤페론을 통해 복합체화를 위해 Hsp90으로 전달된다. Hsp90/Cdc37 복합체는 대부분의 단백질 키나제의 폴딩을 제어하고 다수의 세포내 신호전달 네트워크(21)를 위한 허브로서 기능한다. 이는 급성 골수모구성 백혈병, 다발성 골수종 및 간세포암종22,23을 포함하는 다양한 악성종양에서의 그의 상승된 발현으로 인해 유망한 항종양 표적이다.
일반적으로 사용되는 시험관내 생체분자 상호작용 검출 기술
공동면역침전(co-IP)은 생물학적 관련 상호작용(24)을 확인하기 위해 항원-항체 특이성에 의존하는 기술이다. 이 방법의 주요 단점은 낮은 친화도 상호작용 및 동역학적 값24를 검출할 수 없다는 것이다. 등온 적정 열량계 (ITC), 표면 플라즈몬 공명 (SPR), 생물층 간섭 측정 (BLI) 및 FEB 기술과 같은 생물 물리학 적 방법이 운동 값을 결정하기 위해 바람직하다.
ITC는 생체 분자 상호 작용을 특성화하기위한 완전한 열역학 분석과 함께 결합 에너지의 결정에 기초한 생물 물리학 적 검출 방법입니다25. ITC의 주요 이점은 표적 단백질의 어떠한 표지 또는 고정도 필요로 하지 않는다는 것이다. ITC가 직면하는 주요 어려움은 한 실험에 필요한 고농도의 표적 단백질과 작은 결합 엔탈피26으로 인한 비공유 복합체 분석의 어려움입니다. SPR 및 BLI 둘 모두는 센서 표면 상의 표적 분자의 고정화에 의존하는 라벨이 없는 생물물리학적 기술이며, 이어서 고정화된 표적27,28 위에 분석물의 후속 주사가 뒤따른다. SPR에서, 생체분자 상호작용 동안 굴절률의 변화는 측정된다27; BLI에서, 반사광에서의 간섭은 시간(28)의 함수로서 파장의 변화로서 실시간으로 기록된다. SPR과 BLI는 모두 높은 특이성, 감도 및 검출 기능(29)을 제공하는 공통적인 이점을 공유한다. 두 방법 모두에서, 표적 단백질은 바이오센서 표면 상에 고정화되고, 따라서, 표적의 천연 입체형태가 약간 손실될 수 있고, 이는 특이적 대 비특이적 상호작용(30)을 구별하는 것을 어렵게 만든다. BLI는 고가의 일회용 광섬유 바이오센서를 사용하여 타겟을 고정화하고, 따라서 비용이 많이 드는 기술(31)이다. 이러한 잘 정립 된 생체 분자 검출 도구와 비교할 때, FEB 기술은 운동 특성화를 통해 실시간으로 생체 분자 검출을 위해 낮은 나노 몰 농도를 사용하여 신뢰할 수 있고 라벨이없는 플랫폼을 제공합니다. FEB 기술은 또한 ITC에서 직면 한 버블 링 문제를 극복하고 SPR 또는 BLI에 비해 비용 효율적입니다.
전계 효과 트랜지스터(FET) 기반 바이오센서는 다양한 생물 의학 응용 분야를 제공하여 생체 분자 상호작용을 감지하는 새로운 분야입니다. FET 시스템에서, 표적은 바이오센서 칩에 고정화되고, 상호작용은 전도도(32)의 변화에 의해 검출된다. 효율적인 전자 바이오센서의 개발에서 고려되어야 할 독특한 특징은 센서 표면(33)을 제작하는데 사용되는 코팅 물질의 반전도성 성질 및 화학적 안정성과 같은 물리화학적 특성이다. FET에 사용되는 실리콘과 같은 종래의 재료들은 적절한 기능(34)을 위해 트랜지스터 채널과 특정 환경 사이에 끼워진 산화물 층들을 필요로 하기 때문에 센서의 감도를 제한하였다. 더욱이, 실리콘 트랜지스터는 높은 염수 환경에 민감하기 때문에 자연 환경에서 생물학적 상호작용을 측정하기가 어렵다. 그래핀 기반 바이오 센서는 우수한 화학적 안정성과 전기장을 제공하기 때문에 대안으로 제시됩니다. 그래 핀은 탄소의 단일 원자 층이기 때문에 반도체로서 매우 민감하고 생물학적 용액과 화학적으로 양립 할 수 있습니다. 이들 특성들 모두는 호환가능한 전자 바이오센서들(35)을 생성하는 것이 바람직하다. 그래핀 코팅 바이오센서가 제공하는 생체분자의 놀라운 초고로딩 잠재력은 그래핀 기반 바이오센서 FEB 기술의 개발로 이어진다.
FEB 기술의 원리 : FEB는 결합 표적이 고정되어있는 그래핀 바이오 센서를 통해 전류를 측정하는 라벨이없는 생체 분자 검출 기술입니다. 고정화된 단백질과 분석물 사이의 상호작용은 실시간으로 모니터링되는 전류의 변화를 초래하여, 정확한 동역학적 측정(36)을 가능하게 한다.
계측: FEB 시스템은 그래핀 전계 효과 트랜지스터(gFET) 센서 칩과 실험 전반에 걸쳐 일정한 전압을 적용하는 전자 리더기로 구성됩니다(그림 1). 분석물은 바이오센서 표면에 고정화된 표적 단백질에 용액으로 도포된다. 상호 작용이 발생하면 전류의 변화가 실시간으로 측정되고 기록됩니다. 분석물 농도가 증가함에 따라, 결합된 분석물의 분율 또한 증가하여, 전류에서 더 높은 변화를 야기할 것이다. 계측기(Table of Materials)와 함께 제공된 자동화된 분석 소프트웨어를 사용하여 I-Response가 측정되고 바이오센싱 장치(BU)37로 기록됩니다. I-Response는 고정화된 타겟과 분석물질의 상호작용 시 실시간으로 측정되는 바이오센서 칩을 통한 전류(I)의 변화로 정의된다. FEB 자동 분석 소프트웨어는 C-Response가 정전 용량(C)의 변경을 기록하는 동적 상호 작용 이벤트에 대한 I-응답 및 C-응답을 모두 분석할 수 있습니다. I-응답 및 C-응답 둘 다의 변동은 결합된 분석물의 분율에 직접적으로 대응하며, KD 값을 생성하기 위해 추가로 분석될 수 있다. 자동화된 분석 소프트웨어의 기본 기본 설정은 I-Response입니다.
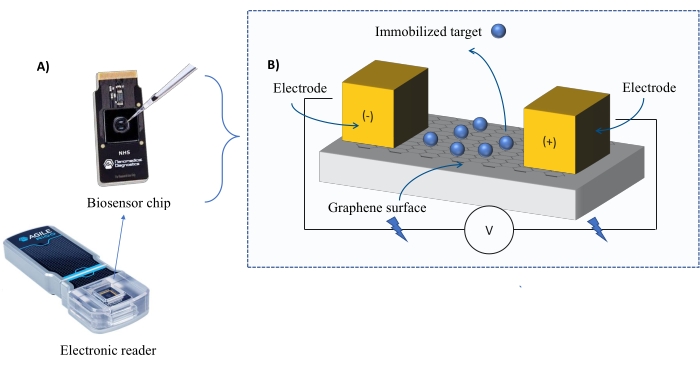
그림 1: 실험 설정 개요 . (A) 그래핀 기반 칩 및 전자 리더기. (B) 칩 성분의 개요. 칩은 시스템에 전류를 공급하는 두 개의 전극에 부착됩니다. 칩의 표면은 그래핀으로 덮여 있으며, 활성화되면 표적을 결합 할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
방법론:
초기에, 활성화된 바이오센서 칩이 FEB 장치에 삽입되고(도 1) 이어서 아래에 요약된 단계들의 실행이 뒤따른다: (1) 캘리브레이션: 실험은 기준선 평형 반응을 생성하기 위해 1x 포스페이트 완충 식염수(PBS; pH = 7.4)를 사용하는 시스템 교정으로 시작한다. (2) 연관성 : 분석물이 칩에 도입되고 결합 포화도에 도달 할 때까지 I- 응답이 모니터링됩니다. (3) 해리: 분석물은 1x PBS를 사용하여 해리된다. (4) 재생성: 분석물질의 잔재물은 1x PBS를 사용하여 제거한다. (5) 세척: 칩으로부터 결합 및 비결합 분석물의 철저한 제거를 위해 1x PBS를 사용하여 총 5개의 세척이 수행된다.
분석:
데이터 분석은 계측기와 함께 제공되는 완전 자동화된 소프트웨어를 사용하여 수행됩니다. 자동화된 분석소프트웨어는 KD 값을 갖는 힐 핏 플롯을 생성합니다. 힐 핏 플롯은 분석물 농도의 함수로서 표적 단백질에 대한 분석물의 연관성을 기술한다. 반-최대 반응이 달성되는 농도는 KD 값 에 비례한다. 낮은KD 값은 높은 결합 친화도를 나타내고 그 반대의 경우도 마찬가지입니다.
FEB 실험에서 얻은 데이터를 검증하기 위해 I-응답은 데이터 검토/내보내기 소프트웨어를 사용하여 각 분석물 농도에 대한 각 판독 지점에서 추출되며 아래에 설명된 대로 다른 통계 분석 소프트웨어( 자료 표 참조)로 내보낼 수 있습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
참고: 본 연구에 사용된 재조합 단백질인 Hsp90 및 Cdc37이 상업적으로 수득되었다( 표 참조).
1. 칩 활성화
참고: 실험에 사용되는 모든 재료는 재료 표에 나열되어 있습니다. 준비된 모든 용액을 멸균 0.2 μm 필터를 통해 여과하십시오.
- 15 mL 튜브에서 2.5 mL의 1 M 2-(N-모르폴리노)에탄술폰산(MES) 완충액(pH = 6.0)에 EDC 2 mg을 첨가하여 1-에틸-3-(3-디메틸아미노프로필)카르보디이미드(EDC) 용액을 제조하였다. 별도의 15 mL 튜브에서 1 M MES 버퍼 (pH = 6.0) 2.5 mL에 설포-NHS 6 mg을 첨가하여 N-하이드록시설포숙신이미드 (sulfo-NHS) 용액을 준비하십시오. 각 용액의 Aliquot 50 μL를 독립 튜브에 넣고 향후 사용을 위해 -20 °C에서 보관하십시오.
- 동일한 부피의 EDC 모래 설포-NHS 용액 (EDC 50 μL + 50 μL의 sulfo-NHS)을 위아래로 피펫팅하여 혼합하십시오 (볼텍스 방지).
참고: EDC/sulfo-NHS의 혼합 용액은 적절한 칩 기능화를 위해 효과적인 가교를 유지하기 위해 30분 이내에 사용해야 합니다. - 회사에서 공급한 바이오센서 칩(5.7 cm x 2.4 cm; 자료표 참조)을 뚜껑이 달린 유리 페트리 접시에 넣습니다. 칩 활성화와 관련된 모든 기능화 단계는 페트리 접시 내에서 수행하는 것이 좋습니다. 50 μL의 1 M MES 버퍼 (pH = 6.0)를 바이오센서 칩에 적용하고, 실온에서 1분 동안 인큐베이션한 다음, 버퍼를 흡인한다.
- 50μL의 EDC/sulfo-NHS 용액을 센서 칩에 즉시 바릅니다. 페트리 접시를 덮고 실온에서 15분 동안 배양하십시오. 칩에서 EDC/sulfo-NHS 용액을 흡인합니다.
- 칩을 50 μL의 1 M MES 완충액 (pH = 6.0)으로 1회 헹구고; MES 버퍼를 흡인한다.
2. 표적 단백질 고정화
- 칩 2x를 1x PBS의 50 μL로 헹구었다(pH=7.4). 칩으로부터 PBS를 흡인하고, 표적 분자인 Hsp90 (50 μL; 500 nM)을 첨가한다.
참고: 버퍼 불일치는 전체 실험을 망칠 수 있습니다. 따라서, 실험 전에, 표적 분자가 교정에 사용된 것과 동일한 완충액 (예를 들어, 1x PBS (pH = 7.4))에 있는지 확인하는 것이 중요하다. 필요한 경우, 실험 전에 밤새 투석하여 완충액 교환을 수행하십시오. 이 실험에서, 재조합 단백질, Hsp90 및 Cdc37 둘 다에 대해, 4°C에서 적절한 완충액 교환을 갖는 1x PBS (pH = 7.4; 물질의 표 참조)에 대해 밤새 투석을 수행하였다. 표적 물질(이 경우 Hsp90)의 농도는 상이한 실험 프로토콜과 표적 물질(단백질/펩티드/리간드)의 성질에 따라 달라질 수 있다. - 유리 페트리 접시를 덮고 실온에서 30 분 동안 배양하십시오. 표적 분자를 함유하는 용액을 흡인하고, 1x PBS (pH = 7.4)의 50 μL로 3x 헹구었다. 칩으로부터 1x PBS (pH = 7.4) 용액을 흡인한다.
- 칩에 50 μL의 퀀치 1(1x PBS(pH = 7.4) 중의 3.9 mM 아미노-PEG5-알코올) 용액을 첨가한다. 유리 페트리 접시를 덮고 실온에서 15분 동안 인큐베이션한다. 칩에서 Quench 1 솔루션을 흡인하십시오.
- 칩에 50 μL의 퀀치2 (1 M 에탄올아민 (pH = 8.5)) 용액을 첨가한다. 유리 페트리 접시를 덮고 실온에서 15 분 동안 배양하십시오. 흡인물 퀀치 2 용액을 칩으로부터 5x 1x PBS의 50 μL를 사용하여 칩 5x를 헹구고, 마지막 PBS 액적을 센서에 남긴다.
3. 분석물 시료 준비
- 원하는 농도 범위에서 Cdc37에 대한 분석물 희석 시리즈를 준비하십시오. 첫 번째 실험을 위해, 다음의 농도가 사용되었다: 25 nM, 50 nM, 100 nM, 200 nM, 400 nM, 800 nM, 1,000 nM, 2,000 nM, 3,000 nM, 및 5,000 nM. 두 번째 실험을 위해, 0.4 nM 내지 200 nM 범위의 상이한 농도 세트가 사용되었다.
- 신뢰할 수있는 KD 값을 얻기 위해 적어도 여덟 개의 상이한 분석물 농도를 포함하도록 실험을 설계한다. 분석물 단백질의 상이한 희석물을 교정에 사용된 것과 동일한 완충액 및 표적 단백질로 제조하는 단계; 여기서 1x PBS (pH = 7.4)입니다.
4. 활성화 된 바이오 센서 칩을 FEB 장치에로드
주: FEB 장치는 LED 조명 표시가 있는 리더기와 바이오센서 칩을 삽입하는 카트리지로 구성됩니다.
- 표적 단백질 고정화 후, 활성화된 칩을 USB를 통해 컴퓨터에 연결된 장치의 카트리지에 삽입하십시오. 칩 삽입 후 FEB 장치가 실험 준비가 되었음을 나타내는 녹색 LED 표시등이 리더에 표시됩니다. FEB 장치가 연결된 컴퓨터에 회사에서 제공하는 자동화 소프트웨어( 자료 표 참조)를 설치하여 아래에 설명된 대로 단계별로 실험을 모니터링합니다.
5. 실험 실행
- 자동화 된 소프트웨어에서 실험 실행 모듈을 누르고 재생성 또는 기타 원하는 프로토콜 로 10 포인트를 선택하십시오. 운영자 이름, 실험 이름, 날짜 (예 : Yana, Hsp90 + Cdc37, 14.03.2021)를 입력하십시오. 재생 완충제 (예를 들어, PBS 완충제); 고정화된 표적 (예를 들어, Hsp90); 용액 중의 분석물 (예를 들어, Cdc37). 자세한 내용은 보충 그림 S1을 참조하십시오.
- 소프트웨어에 표시된 실험 시작 단추를 누르고 아래에 설명된 대로 자동화된 소프트웨어에 표시된 지침을 따릅니다.
참고: 이 소프트웨어는 완전히 자동화되고 사용자 친화적이며 실험 전반에 걸쳐 사용자를 단계별로 안내합니다. 팝업 창이 화면에 나타나고 실험의 각 단계에서 더 진행하라는 지침이 표시됩니다. 이 소프트웨어는 실험 전반에 걸쳐 각 분석물 농도에 대한 교정, 분석물 연관, 해리, 재생 및 세척 (5x)에서 연속적으로 각 반복적 인 단계에 대한 지침을 제공합니다. - 계측기 교정을 수행합니다. 이렇게 하기 위해, 칩으로부터 남아있는 PBS 용액을 흡인하고 50 μL의 교정 완충액(1x PBS; pH = 7.4)을 적용한다. 계속 버튼을 누르고 교정 단계가 완료될 때까지 5분 동안 기다립니다. 이 소프트웨어는 교정 단계(5분)에 대해 결정된 끝점을 후속 조치에 대한 경고 경보와 함께 표시합니다.
- 다음으로, 분석물 연관을 수행한다. 이렇게 하려면, 칩으로부터 교정 완충액을 흡인하고 가장 낮은 분석물 농도 (25 nM의 Cdc37)의 50 μL를 적용한다. 계속 단추를 누르고 연결 단계가 완료될 때까지 5분 동안 기다립니다. 소프트웨어는 계속 진행하기 위해 경고 경보와 함께 연결 단계 (5 분)의 끝점을 표시합니다.
- 분석물 해리를 수행합니다. 이렇게 하기 위해, 분석물 용액을 칩으로부터 흡인하고 50 μL의 해리 완충액(1x PBS; pH = 7.4)을 적용한다. 계속 버튼을 누르고 해리 단계 지속 시간(5분)이 완료될 때까지 5분 동안 기다립니다. 이 소프트웨어는 해리 단계 (5 분)의 끝점을 후속 조치에 대한 경고 경보와 함께 표시합니다.
- 그런 다음 칩 재생성을 수행하십시오. 해리 용액을 칩으로부터 흡인하고 50 μL의 재생 완충액 (1x PBS; pH = 7.4)을 적용한다. 계속 단추를 누르고 재생성 단계 지속 시간(30초)이 완료될 때까지 30초 동안 기다립니다. 이 소프트웨어는 후속 조치에 대한 경고 경보와 함께 재생 단계 (30 초)의 끝점을 표시합니다.
- 마지막으로 칩을 씻으십시오. 칩으로부터 재생 용액을 흡인하고, 50 μL의 세척 완충액(1x PBS; pH = 7.4)을 칩에 적용한다. 칩에서 용액을 흡인하고이 5x를 반복하십시오. 세척 버퍼의 마지막 방울을 칩에 놓고 계속 버튼을 누르고 소프트웨어 디스플레이에서 세척 단계 지속 시간이 완료 될 때까지 30 초 동안 기다리십시오.
참고: 소프트웨어는 세척 단계(30초)의 끝점을 경고와 함께 표시하여 실험의 다음 주기를 진행합니다. - 사용된 각 분석물 농도에 대해 상기 단계를 반복하는 단계; 교정, 분석물 결합, 해리, 재생 및 세척 (5x)의 다섯 단계는 하나의 사이클을 구성합니다. 여기에 나타낸 실험을 위해, 우리는 10개의 분석물 농도 (25 nM 내지 5,000 nM 또는 0.4 nM 내지 200 nM 범위; 그림 2).
6. 분석
- 실험이 끝날 때 자동화된 분석 소프트웨어의 맨 위에 있는 분석 단추를 누릅니다. 모든 실험 포인트가 포함된 표시 창이 나타납니다. 창에서 규정 된 프로토콜에 사용 된 분석물 농도가 올바른지 확인하십시오.
- 분석 실행 단추를눌러 KD 값을 자동으로 생성합니다. 이 소프트웨어는 평형에서의 해리 상수 인 KD 값이 계산되는 해당 I-Response에 대해 분석물 농도를 플로팅하여 Hill fit 플롯을 생성합니다.
- 아래에 설명된 대로 데이터 검토/내보내기 소프트웨어를 사용하여 다른 통계 분석 소프트웨어에서 분석을 위해 원시 데이터를 내보냅니다.
- 실험이 끝날 때 자동으로 생성된 R1R 파일(예: Hsp90 + Cdc37 14.03.2021)을 데스크톱의 새 폴더에 복사합니다. 회사에서 제공하는 데이터 검토/내보내기 소프트웨어를 엽니다( 보충 그림 S2A 참조).
- 데이터 처리 > 처리 R1R 파일을 클릭하> 데이터 검토 / 내보내기 소프트웨어의 홈 화면에서 확인.
- 6.3.1단계에서 바탕 화면에 만든 R1R 파일이 들어 있는 폴더를 선택합니다. 를 누르고 OK 버튼을 누릅니다. 이렇게 하면 원본 R1R 데이터의 복사본이 만들어져 원본 파일을 재정의하지 않고 데이터를 검토하고 편집할 수 있습니다.
- 데이터 검토/내보내기 소프트웨어의 홈 화면에 표시된 데이터 처리 아이콘을 누릅니다. 처리 된 R1R 파일로드를 누르> OK입니다. 처리된 R1R 파일이 포함된 6.3.1단계에서 만든 동일한 폴더를 선택합니다. OK를 누릅니다. 이 단계에서는 실험 파일이 들어 있는 폴더를 검토할 준비가 되었습니다.
- 데이터 검토/내보내기 소프트웨어의 홈 화면에서 데이터 분석을 누릅니다. 교정을 선택> 편집한 R1R 파일을 정상> 보정 합니다( 보충 그림 S2B 참조). 이 단계에서는 첫 번째 보정 단계에 따라 모든 데이터 요소를 보정하여 기준선을 만듭니다.
- 데이터 검토/내보내기 소프트웨어의 홈 화면에서 데이터 분석을 누릅니다. R1R 파일 검토 및 편집을 선택> 확인. 데이터 요소를 검토하거나, 점을 삭제하거나, 단계 삭제/추가 단추를 사용하여 단계를 추가합니다 . 교정 단계가 기준선에 있고 연관 단계가 최고점에 있는지( 보충 그림 S3 참조) 모든 단계가 올바른 위치에 있는지 확인한 다음 저장 버튼을 눌러 모든 변경 사항을 저장합니다.
- 데이터 검토/내보내기 소프트웨어의 홈 화면에서 데이터 분석을 누릅니다. 데이터 분석/플롯을 선택하고 내보낼 단계를 선택합니다( 보충 그림 S4 참조). 데이터를 내보내기 전에 보정 단계(예: 단계 1, 단계 6 등)를 뺀 후 각 연관 단계(예: 단계 2, 단계 7 등)를 내보내기 목록에 추가합니다.
- 이 데이터 내보내기를 누릅니다. 이 소프트웨어는 각 트랜지스터의 각 분석 물 농도 데이터 포인트에 대한 I-Response가 포함 된 스프레드 시트 파일을 생성합니다 (모든 트랜지스터가 작동하는 경우 각 농도 포인트에 대해 세 가지 다른 I 응답 값을 갖습니다). 이 스프레드 시트 파일을 사용하여 통계 소프트웨어의 데이터를 추가로 분석하십시오.
- 통계 분석 소프트웨어를 엽니다. 세 개의 Y 값으로 XY 테이블을 작성하십시오. 만들기를 누릅니다. 이 테이블의 데이터(X, Y 값)는 6.3.8단계에서 만든 스프레드시트 파일에서 복사됩니다. x축은 분석물 농도(사용된 농도에 따라 다름)에 해당하며, 세 개의 Y 값은 회사에서 제공하는 데이터 검토/내보내기 소프트웨어에서 얻은 I-응답에 해당합니다.
- 이 데이터 분석을 누릅니다. 전체 부위 > 결합 포화> 비선형 회귀(곡선 적합)> XY 분석을 선택>니다. 소프트웨어는 자동으로 데이터를 분석하고, KD 값을 생성하고, 데이터 포인트의 그래프를 생성합니다.
Access restricted. Please log in or start a trial to view this content.
결과
실험 1의 결과:
표적 단백질 Hsp90(500 nM)을 전술한 바와 같이 표적 고정화 프로토콜에 따라 칩에 고정화시켰다. 첫 번째 실험을 위해, 25 nM 내지 5,000 nM 범위의 분석물 단백질, Cdc37의 10 농도를 문헌에서 이용가능한 데이터에 기초하여 제조하였다 ( 표 1 참조).
실험의 단계는 I-Response에서 발생하는 변경 사항을 따라 실시간으로 모니터링할 수 있습니다(<...
Access restricted. Please log in or start a trial to view this content.
토론
이 연구에서, Hsp90과 Cdc37 사이의 생체분자 상호작용을 결정하기 위해 FEB 기술(실시간 운동 특성화 접근법)을 사용하는 타당성을 평가하였다. 초기 탐구 실험 (첫 번째 실험)은 적절한 분석물 농도를 선택하는 것이 실험의 중요한 부분이며 실험은 KD 값 위와 아래의 농도 점을 포함하여 설계되어야한다고 제안했으며, 이는 문헌에서 사용 가능한 데이터를 기반으로 예측되었습니다.
Access restricted. Please log in or start a trial to view this content.
공개
저자는 이해관계, 재정적 또는 다른 이유로 어떠한 충돌도 선언하지 않습니다.
감사의 말
이 연구는 Binational Science Foundation (BSF)에서 S.K.S. 및 N.Q.에 대한 보조금으로 지원되었습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA | Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA | T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 | White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA | |
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 | White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA | Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 | Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA | Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA | Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA | Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA | |
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA | |
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA | |
| Statistical software | GraphPad, Prism | NA CAS number: NA | Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 | White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA | Tip 1-200 µl, in bulk, 1,000 pcs |
참고문헌
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083(2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438(2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213(2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), San Diego, Calif. 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , Springer International Publishing. 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882(2014).
- Vu, C. -A., Chen, W. -Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214(2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434(2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era...? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유