Method Article
인간 조혈 줄기 및 전구 세포에서 발암성 이형접합체 기능 획득 돌연변이 공학
요약
조혈 줄기 및 전구 세포 (HSPC)의 체세포 돌연변이를 충실하게 모델링하는 새로운 전략은 조혈 줄기 세포 생물학 및 혈액 악성 종양을 더 잘 연구하는 데 필요합니다. 여기에서는 CRISPR/Cas9의 사용과 이중 rAAV 공여체 형질도입을 결합하여 HSPC에서 이형접합체 기능 획득 돌연변이를 모델링하는 프로토콜이 설명됩니다.
초록
평생 동안 조혈 줄기 및 전구 세포 (HSPC)는 체세포 돌연변이를 획득합니다. 이러한 돌연변이 중 일부는 증식 및 분화와 같은 HSPC 기능적 특성을 변경하여 혈액 악성 종양의 발병을 촉진합니다. 재발하는 체세포 돌연변이의 기능적 결과를 모델링, 특성화 및 더 잘 이해하려면 HSPC의 효율적이고 정확한 유전자 조작이 필요합니다. 돌연변이는 유전자에 해로운 영향을 미치고 기능 상실 (LOF)을 초래할 수 있으며, 대조적으로 기능을 향상 시키거나 기능 획득 (GOF)이라고하는 특정 유전자의 새로운 특성으로 이어질 수도 있습니다. LOF 돌연변이와 달리 GOF 돌연변이는 거의 독점적으로 이형 접합 방식으로 발생합니다. 현재의 게놈 편집 프로토콜은 개별 대립 유전자의 선택적 표적화를 허용하지 않아 이형 접합 GOF 돌연변이를 모델링하는 능력을 방해합니다. 여기에서는 효율적인 DNA 공여체 주형 전달을 위해 CRISPR/Cas9 매개 상동성 지향 복구 및 재조합 AAV6 기술을 결합하여 인간 HSPC에서 이형접합체 GOF 핫스팟 돌연변이를 엔지니어링하는 방법에 대한 자세한 프로토콜을 제공합니다. 중요하게도, 이 전략은 이중 형광 리포터 시스템을 사용하여 성공적으로 이형접합체로 편집된 HSPC의 추적 및 정제를 허용합니다. 이 전략은 GOF 돌연변이가 HSPC 기능과 혈액 악성 종양으로의 진행에 어떻게 영향을 미치는지 정확하게 조사하는 데 사용할 수 있습니다.
서문
클러스터형 규칙적인 간격의 짧은 회문 반복(CRISPR)/Cas9 기술의 개발로 새롭고 매우 강력한 기기가 과학자 툴킷에 추가되었습니다. 이 기술은 게놈의 정밀한 엔지니어링을 가능하게 하며 연구 목적(Hsu et al.1에서 검토됨)뿐만 아니라 최근에는 임상 환경 2,3,4로 성공적으로 번역된 것으로 입증되었습니다. CRISPR/Cas9 편집 전략은 Cas9 단백질과 단일 가이드 RNA(sgRNA)5,6,7의 활성에 의존합니다. 숙주 세포에서 Cas9 단백질은 sgRNA 서열과 상보적인 DNA의 특정 부위로 안내되고 DNA 이중 가닥 절단(DSB)을 도입합니다. DSB가 생성되면 발생할 수 있는 두 가지 주요 및 경쟁 복구 메커니즘, 즉 비상동 말단 접합(NHEJ)과 상동성 지향 복구(HDR)가 있습니다. NHEJ는 오류가 발생하기 쉽고 주로 사용되는 복구 메커니즘으로 삽입 및 삭제 (indels)로 이어지는 반면, HDR은 자매 염색 분체를 복구 템플릿으로 사용하여 매우 정확하지만 세포주기8의 S 또는 G2 단계로 제한됩니다. 게놈 엔지니어링에서 HDR은 Cas9 유도 DSB의 양쪽 DNA 말단과 동일한 상동성 팔에 의해 측면에 있는 기증자 주형을 제공함으로써 DNA의 표적 변형에 활용될 수 있습니다(그림 1). HDR에 사용되는 기부자 템플릿 유형은 편집 효율성에 큰 영향을 줄 수 있습니다. 인간 HSPC의 유전 공학을 위해, 아데노-관련 바이러스 혈청형 6 (AAV6)은 최근 단일-가닥DNA 주형의 전달을 위한 우수한 매개체로 기술되었다 9,10.
CRISPR/Cas9 게놈 공학은 유해한 돌연변이(11)를 교정하기 위해 치료적으로 사용될 수 있지만, 암 발생을 모델링하기 위해 DNA에 병원성 돌연변이를 도입하는 데에도 활용될 수 있다(12). 백혈병과 같은 혈액 암은 건강한 HSPC에서 체세포 돌연변이의 순차적 획득을 통해 발생합니다13,14. 초기 유전적 사건은 클론 증식 이점으로 이어져 불확정 전위(CHIP)의 클론 조혈을 초래합니다.15,16. 돌연변이의 추가 획득은 결국 백혈병 변형과 질병의 발병으로 이어질 것입니다. 체세포 돌연변이는 자가 재생, 생존, 증식 및 분화를 제어하는 유전자에서 찾을 수 있습니다17.
게놈 엔지니어링을 통해 개별 돌연변이를 건강한 HSPC에 도입하면 이 단계적 백혈구 발생 과정을 정확하게 모델링할 수 있습니다. 급성 골수성 백혈병(AML)18,19과 같은 골수성 신생물에서 발견되는 제한된 수의 재발성 돌연변이로 인해 이 질병은 특히 게놈 엔지니어링 도구를 사용하여 요약하기 쉽습니다.
체세포 돌연변이는 하나의 대립 유전자 (단일 대립 유전자 / 이형 접합 돌연변이) 또는 두 대립 유전자 (이중 대립 유전자 / 동형 접합 돌연변이)에서만 나타날 수 있으며 유전자의 기능에 중대한 영향을 미칠 수 있으며, 이는 기능 상실 (LOF) 또는 기능 획득 (GOF)을 유발할 수 있습니다. LOF 돌연변이는 유전자의 LOF를 감소(하나의 대립유전자가 영향을 받는 경우) 또는 완전(두 대립유전자가 모두 영향을 받는 경우)으로 이어지는 반면, GOF 돌연변이는 유전자의 활성화 또는 새로운 기능을 증가시킵니다. GOF 돌연변이는 전형적으로 이형접합체20이다.
중요하게도, 접합성 (이종 접합체 대 동형 접합체)은 돌연변이를 충실하게 모델링하려는 시도에 중요한 영향을 미칩니다. 따라서, 이형접합체 핫스팟 GOF 돌연변이를 조작하기 위해서는 유전자의 단 하나의 대립유전자만을 표적화하는 조작이 필요하다. 오류가 발생하기 쉬운 NHEJ는 다양하고 예측할 수없는 생물학적 결과를 초래할 수있는 다양한 길이21의 indels로 이어집니다. 그러나 NHEJ는 DSB 도입 후 세포에 의해 사용되는 주된 복구 프로그램이기 때문에 현재 HSPC를 조작하는 데 사용되는 대부분의 CRISPR / Cas9 플랫폼은 유전 적 결과를 정확하게 예측할 수 없습니다22,23. 대조적으로, HDR을 통한 게놈 엔지니어링을 위한 재조합 아데노-관련 바이러스(rAAV) 벡터 기반 DNA 공여체 템플릿의 사용과 결합된 CRISPR/Cas9-매개 이중 가닥 절단(DSB)의 도입은 인간 HSPC에서 돌연변이의 대립유전자 특이적 삽입을 허용합니다(11,24). 개별 대립유전자에 대한 별개의 형광 리포터와 결합된 돌연변이 및 야생형(WT) 서열의 동시 통합을 수행하여 이형접합체 유전자형을 선택할 수 있습니다(그림 2). 이 전략은 재발성, 백혈병, 이형접합체 GOF 핫스팟 돌연변이가 HSPC 기능, 질병 시작 및 진행에 미치는 영향을 정확하게 특성화하는 강력한 도구로 활용할 수 있습니다.
이 기사에서는 1 차 인간 HSPC에서 반복적으로 돌연변이 된 이형 접합체 GOF 돌연변이의 효율적인 엔지니어링을위한 자세한 프로토콜이 제공됩니다. 이 전략은 CRISPR/Cas9와 이중 AAV6 형질도입의 사용을 결합하여 이형접합체 GOF 돌연변이의 전향적 생성을 위한 WT 및 돌연변이 DNA 기증자 템플릿을 제공합니다. 예로서, 칼레티큘린(CALR) 유전자에서 재발성 제1형 돌연변이(52 bp 결실)의 공학은도 25를 나타낼 것이다. CALR의 엑손 9에서 이형 접합체 GOF 돌연변이는 본 태성 혈소판 증가증 (ET) 및 원발성 골수 섬유증 (PMF)26과 같은 골수 증식 성 장애에서 반복적으로 발견됩니다. CALR은 새로 합성 된 단백질의 접힘 과정에서 주로 품질 관리 기능을 갖는 소포체 상주 단백질입니다. 그 구조는 단백질의 샤페론 기능에 관여하는 아미노 (N) 말단 도메인과 프롤린이 풍부한 P 도메인, 칼슘 저장 및 조절27,28에 관여하는 C- 도메인의 세 가지 주요 도메인으로 나눌 수 있습니다. CALR 돌연변이는 +1 프레임 시프트를 일으켜 새로운 확장 된 C- 말단 말단의 전사와 소포체 (ER)- 머무름 신호 (KDEL)의 손실로 이어집니다. 돌연변이 CALR은 트롬보포이에틴 (TPO) 수용체에 결합하여 증식 증가와 함께 TPO 비의존적 신호전달을 유도하는 것으로 나타났습니다29.
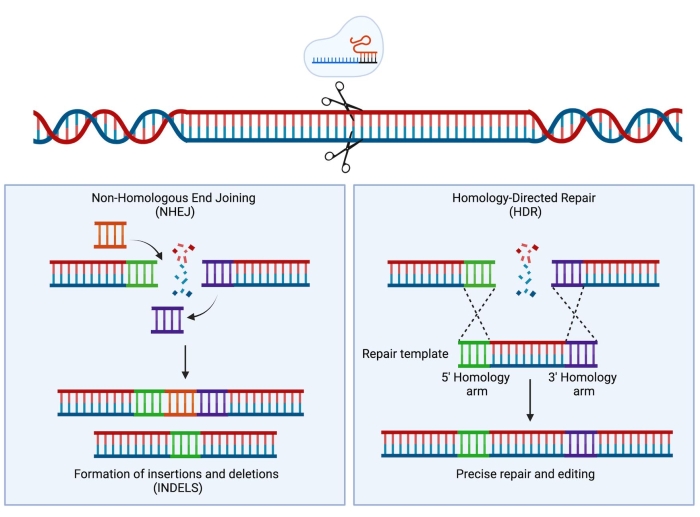
그림 1: NHEJ 및 HDR 복구. DNA에 이중 가닥 절단이 도입된 후 NHEJ 및 HDR 복구 메커니즘의 단순화된 개략적 표현. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
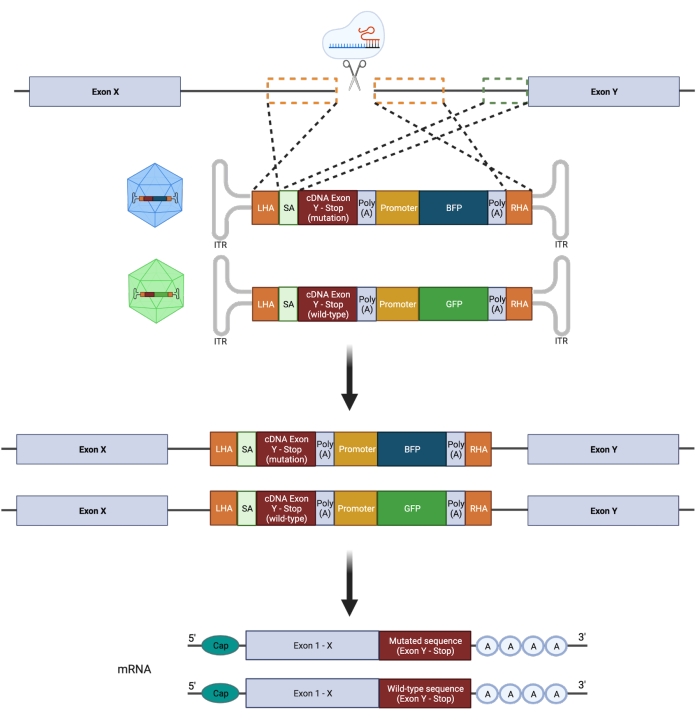
그림 2: 이대립유전자 HDR 편집 전략의 개략도. 기증자 주형을 표적 대립 유전자에 통합 한 후 기능하는 mRNA로 번역하는 것을 보여주는 개략적 표현. 주황색 점선 상자는 왼쪽 상동 팔 (LHA) 및 오른쪽 상 동성 팔 (RHA)에 해당하는 영역을 나타냅니다. HA의 이상적인 크기는 각각 400bp입니다. 녹색 점선 상자는 SA 시퀀스에 해당하는 영역을 나타냅니다. SA의 크기는 150bp입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
이 프로토콜은 건강한 기증자 유래 CD34+ HSPC의 사용이 필요하며 지역 기관 검토 위원회(IRB)의 윤리적 승인과 서명된 정보에 입각한 동의가 필요합니다. 이 프로토콜에 사용된 CD34+ HSPC는 만삭 분만(>임신 34주)의 제대혈(UCB)에서 분리되었습니다. 분만 전에 산모로부터 정보에 입각한 동의를 얻었고 그라츠 의과 대학에서 UCB 수집에 대한 윤리적 승인을 받았습니다(IRB 승인: 31-322 ex 18/19). 이 프로토콜에 사용 된 재료의 전체 목록은 재료 표에서 찾을 수 있습니다.
1. sgRNA 설계 및 절단 효율 평가
- 온라인 데이터베이스 도구(예: COSMIC, https://cancer.sanger.ac.uk/cosmic)에서 원하는 돌연변이의 위치와 올바른 전사체를 검색합니다.
- sgRNA 설계 도구를 사용하여 돌연변이(엑소닉 표적화) 또는 이전 인트론(인트론 표적화)에 인접한 sgRNA를 선택합니다. 이 프로토콜에서는 Benchling의 온라인 도구가 사용됩니다. sgRNA 설계에 사용할 수 있는 온라인 도구 목록은 재료 표를 참조하십시오.
- 데이터베이스에서 가져오기>> DNA 서열 >가져오기 + (만들기) 옵션을 선택합니다. 원하는 유전자 예를 들어 CALR을 입력하고 > 검색> 올바른 전사체(예: CALR-201-ENST00000316448)를 선택하여 가져오기> 인간을 선택합니다. DS 브레이크를 도입할 관심 영역(예: 인트론 7)을 선택합니다.
- 화면 오른쪽에서 CRISPR 옵션을 선택하고 가이드 설계 및 분석을 선택합니다. 단일 가이드를 선택하고 가이드 길이를 20bp로 유지하고 SpCas9(NGG)로 편집하기 위해 PAM 시퀀스를 유지합니다.
- 높은 목표 점수(원하는 궤적에서 편집할 가능성이 높음)와 높은 비목표 점수(원치 않는 궤적에서 편집할 가능성이 낮음)를 가진 가이드를 선택합니다. 가장 성능이 좋은 sgRNA를 찾기 위해 테스트할 가이드를 세 개 이상 선택하십시오. sgRNA를 상용 공급업체에서 화학적으로 변형된 합성 sgRNA로 주문하십시오.
참고: 이 단계에서는 초기 선별을 위해 소량의 합성 sgRNA를 주문하는 것이 좋습니다. 우수한 성능의 sgRNA가 확인되면 선택된 sgRNA의 더 큰 순서로 진행합니다. - 해동 2 x 10 5-5 x 105 CD34+ HSPC. 세포를 10% 항생제(예: 페니실린/스트렙토마이신)가 보충된 예열된 RPMI1640 mL로 옮깁니다.
참고: 순도가 90%인 CD34+ HSPC를 사용하여 재현 가능한 최상의 결과를 얻을 수 >. - 실온(RT)에서 350 x g 에서 10분 동안 원심분리합니다. 혈구계로 세포를 세고 0.2% 페니실린/스트렙토마이신(P/S), 100ng/mL 트롬보포이에틴(TPO), 100ng/mL 줄기세포 인자(SCF), 100ng/mL FMS 유사 티로신 키나제 3 리간드(FLT3L), 100ng/mL 인터루킨-6(IL-6), 35nM UM171 및 0.75μM 스템레게닌1(SR1)이 보충된 SFEM II 배지에 세포를 2.5 x 105 세포/mL의 농도로 현탁합니다. 37 °C/5% CO2에서 48 h -72 h 동안 배양합니다.
참고: 지금부터 전체 매체를 HSPC 보존 매체라고 합니다. - 15mL 튜브에 세포를 수집하고 세포를 세십시오. 트리판 블루 배제에 의한 세포 생존율을 확인한다.
- 시작하기 전에 형질 주입 시스템을 켜고 큐벳 옵션을 선택하십시오. HSPC에 적합한 프로그램 및 핵 절제 용액을 선택하십시오 ( 재료 표 참조). 2 x 10 5 및5 x 10사이에서 6 개의 세포를 하나의 100 μL 큐벳에서 핵 제거 할 수 있습니다. WT 대조군으로서 사용하기 위해 48 h 동안 배양물에 보관하기 위해 소수의 세포(예를 들어, 1 x 105-2 x 105)를 따로 보관한다.
- RNP 복합체를 준비하십시오. 1.5mL 튜브에 15μg의 Cas9와 8μg의 sgRNA(몰비 1:2.5)를 넣고 25°C에서 가열 블록에서 10분 동안 배양합니다.
- RNP 복합체가 인큐베이션되는 동안, 세포를 RT에서 350 x g 에서 5분 동안 원심분리하고, 피펫을 사용하여 상청액을 버린다. 세포를 100 μL의 뉴클레오펙션 용액에 현탁시킨다.
- 세포를 RNP 복합체와 혼합하고 큐벳으로 옮깁니다. 큐벳을 부드럽게 두드려 전사 중에 형성되었을 수 있는 최종 기포를 제거합니다.
- 큐벳을 형질주입 시스템의 홀더에 삽입하고 DZ-100 프로그램으로 세포를 전기천공합니다.
- 전기천공 직후 P/S 없이 예열된 HSPC 머무름 배지 400μL를 추가합니다.
- 미세 전달 피펫으로 세포를 P/S 없이 예열된 HSPC 보존 배지가 들어 있는 배양 플레이트로 옮깁니다. 세포 수에 따라 0.25 x 10 6과 1 x 10 6 cells/mL 사이의 밀도에 도달하기 위해 적절한 배양 플레이트(24웰, 12웰 또는6웰 플레이트)를 사용합니다.
- 플레이트를 37 °C / 5 % CO2의 인큐베이터로 옮깁니다. 핵 세포를 6-8 시간 동안 배양하십시오.
- 6-8시간 후, 기존 배지를 제거하고 P/S가 보충된 새로운 예열된 HSPC 보존 배지로 교체합니다. 세포를 2.5 x 10 5 및 5 x 105 사이의 농도로 현탁시키고 세포 배양 플레이트(24, 12 또는 6-웰 플레이트)로 옮깁니다. 세포를 37°C/5%CO2에서 48시간 동안 배양합니다.
- 2 x 10 5 셀을 수확하고 RT에서 350 x g에서5 분 동안 원심 분리합니다. 시작하기 전에 가열 블록을 65 ° C 및 98 ° C로 설정하십시오.
- 상청액을 버리고 세포를 1.5mL 튜브의 1x DPBS 1mL에 현탁시킵니다. RT에서 350 x g 로 5분 동안 원심분리합니다.
- 상청액을 버리고 세포를 50 μL의 DNA 추출 용액에 현탁시킨다. 15 초 동안 소용돌이.
- 65°C에서 6분 동안 배양합니다. 15초 동안 와동시키고 98°C에서 2분 동안 배양한다.
- 중앙에 DSB와 함께 약 400-600 bp의 앰플리콘을 생성하는 프라이머를 사용하여 PCR에 의해 DSB의 영역을 증폭합니다. PCR 반응을 위해 추출된 DNA 0.5-1 μL를 사용하십시오.
참고 : DNA 추출 용액의 구성으로 인해 DNA 농도는 분광 광도법으로 정확하게 정량화 할 수 없습니다. - DNA 사다리가 있는 PCR 산물을 100V에서 1.5% 아가로스 겔에서 45-60분 동안 실행합니다. 젤을 청색광 또는 UV 투과기에 놓습니다.
- 제조업체의 지침에 따라 시판되는 키트를 사용하여 겔에서 올바른 크기의 DNA 밴드를 추출합니다( 재료 표 참조).
- PCR로부터의 정방향 프라이머 또는 역방향 프라이머를 사용하여 Sanger 시퀀싱에 의해 샘플을 서열분석한다. 길이가 400-600bp인 PCR 산물의 경우 총 부피 15μL에서 5ng/mL 농도의 시퀀싱을 위해 75ng의 DNA가 필요합니다.
- sgRNA에 의해 생성된 삽입 및 결실을 식별하여 편집 효율을 계산하도록 설계된 프로그램에 시퀀싱 파일을 업로드하여 sgRNA의 편집 효율을 분석한다. 진행할 최고 성능의 sgRNA를 선택합니다.
참고: 전용 온라인 도구는 재료 표에 나열되어 있습니다. 이 분석에는 형질감염된 및 WT HSPC의 .ab1 파일, sgRNA 서열 및 PAM 서열이 필요합니다.
2. 상동성 지향 복구(HDR) 벡터 구성
- HDR 템플릿 디자인
참고 : WT 시퀀스 용 템플릿과 변형 된 시퀀스 용 템플릿의 두 가지 HDR 템플릿을 설계해야합니다.- 원하는 유전자의 게놈 서열과 코딩 서열(CDS)을 분자 클로닝에 적합한 소프트웨어로 가져옵니다(전용 도구는 재료 표에 나열되어 있음). 게놈 서열 파일로부터, DSB의 5' 말단에서 이상적으로 400 bp를 선택하여 좌측 상동성 팔(HA)을 설계한다. 이 시퀀스를 새 파일에 붙여 넣습니다.
- 인트론이 sgRNA로 표적화되는 경우 스플라이스 수용체(SA) 서열을 포함해야 합니다. 표적 인트론의 마지막 150bp를 선택하고 왼쪽 HA 뒤에 붙여 넣습니다. 엑손이 직접 표적화되는 경우 이 시퀀스가 필요하지 않습니다(그림 3).
- CDS 파일에서 관심 있는 cDNA를 선택합니다. 엑소닉 서열이 표적화되는 경우 cDNA가 DSB 부위의 바로 하류에서 시작하고 관심 유전자의 다음 엑손과 정지 코돈을 모두 포함하는지 확인하십시오. 인트론이 표적화되는 경우 cDNA가 다음 엑손의 첫 번째 코돈으로 시작하는지 확인하십시오. 코돈-최적화된 cDNA를 SA 서열 뒤에 삽입한다. 이를 위해 전용 온라인 도구를 사용하십시오 (재료 표).
- 관심있는 cDNA (예를 들어, SV40 또는 bGH) 이후에 3' 폴리아데닐화 (PolyA) 신호를 삽입한다(표 1). PolyA에 이어서, 형광 단백질에 대한 프로모터 서열 (예를 들어, 비장 포커스 형성 바이러스 [SFFV] 또는 폴리유비퀴틴 C [UBC], 표 1)을 삽입한다.
- 형광 단백질(즉, GFP, BFP, 또는 mCherry)에 대한 서열을 삽입한다. 두 번째이지만 다른 PolyA 신호를 삽입합니다.
참고: 다른 PolyA 서열을 삽입하면 플라스미드의 박테리아 재조합 또는 주형에 있는 두 개의 동일한 서열로 인한 서열 정렬 문제와 같은 문제를 피할 수 있습니다. - 게놈 서열 파일에서 DSB의 3' 말단에서 이상적으로 400bp를 선택하여 올바른 HA를 설계합니다. 두 번째 PolyA 뒤에 이 시퀀스를 삽입합니다.
- 전체 템플릿의 사본을 만들고 원하는 돌연변이의 서열을 포함하도록 관심 있는 cDNA를 수정합니다.
- 형광 단백질을 다른 형광 단백질로 교환한다. 예를 들어, GFP가 WT 템플릿에 사용된 경우 돌연변이 템플릿에 대해 BFP 또는 mCherry와 교환합니다. HDR 템플릿을 구성하고 pAAV-MCS 플라스미드(또는 다른 적절한 백본)에 클로닝합니다.
참고: 어셈블리에 필요한 단편은 PCR로 생산하거나 상업적으로 주문할 수 있으며 인접한 단편과 겹치는 서열을 포함해야 합니다. 관심있는 돌연변이가 점 돌연변이, 작은 삽입 또는 작은 결실인 경우, 돌연변이된 cDNA는 WT cDNA를 함유하는 HDR 주형 상에서 부위-지시된 돌연변이유발을 수행함으로써 PCR에 의해 생성될 수 있다. - 유능한 대장균 을 열 충격 방법을 사용하여 조립 된 제품으로 변형시킵니다. 시작하기 전에 박테리아를 얼음에서 10분 동안 해동시킵니다.
- 50μL의 박테리아에 2μL의 조립 된 제품을 첨가하십시오. 내용물을 부드럽게 튕겨서 섞습니다.
- 샘플을 얼음 위에 30분 동안 놓습니다. 샘플을 30초 동안 42°C로 설정된 열블록으로 옮깁니다.
- 샘플을 얼음에 5 분 동안 옮깁니다. 450μL의 실온 SOC 배지를 추가하고 샘플을 37°C에서 1시간 동안 배양합니다.
- 샘플을 암피실린이 함유된 LB 한천 플레이트에 펼칩니다. 각 샘플에 대해 100μL의 세 가지 다른 희석된 박테리아 용액(희석되지 않음, 1:5 및 1:10)을 뿌려 선택할 수 있는 단일 콜로니가 있는 LB 한천 플레이트를 얻습니다. 플레이트를 37°C에서 밤새 인큐베이션한다.
- 다음날 샘플 당 3 개의 식민지를 선택하십시오. 콜로니를 앰피실린이 보충된 LB 배지 4mL가 들어 있는 캡이 있는 15mL 튜브로 옮기고 37°C의 셰이커에서 밤새 배양합니다.
- 각 콜로니에서 500μL의 박테리아 용액을 분취하여 나중에 사용할 수 있도록 냉장고에 보관하십시오. 제조업체의 지침에 따라 플라스미드 DNA를 추출하기 위해 미니 준비를 수행합니다.
- Sanger 시퀀싱을 위해 샘플을 보내 플라스미드의 올바른 조립을 확인합니다. 플라스미드 전체에 분포된 충분한 프라이머를 사용하여 중단 없는 서열 확인을 보장하고, 이상적으로는 정방향 및 역방향 판독으로 각 영역을 두 번 덮습니다.
- 암피실린이 보충된 LB 배지 200mL에 올바르게 조립된 플라스미드를 포함하는 박테리아 용액 200μL를 추가하고 37°C에서 밤새 쉐이커에서 배양합니다.
- 제조업체의 지침에 따라 플라스미드 DNA를 추출하기 위해 미디 또는 맥시 준비를 수행합니다. 플라스미드 DNA를 -20 °C에서 보관하십시오.
- 재조합 AAV6 제제
참고: WT HDR 템플릿이 있는 rAAV6에 대한 것과 변형된 HDR 템플릿이 있는 rAAV6에 대해 하나씩 두 개의 개별 AAV6 준비를 수행해야 합니다. 이 섹션에서는 하나의 바이러스만 준비하는 데 필요한 단계에 대해 설명합니다.- HEK293T 세포를 해동하고 세포를 10% FBS, 1% P/S 및 25mM HEPES가 보충된 예열된 DMEM 10mL로 옮기고 RT에서 350 x g 에서 5분 동안 원심분리합니다.
- 세포를 1 x 105 cells/mL의 농도로 현탁시키고 적합한 플라스크(예를 들어, 175cm2 플라스크)로 옮긴다. 플라스크를 37°C/5%CO2의 인큐베이터로 옮깁니다.
참고 : HEK293T 세포는 세포가 완전히 회복되고 충분한 수의 세포를 얻을 수 있도록 미리 해동해야합니다 (하나의 바이러스를 생산하려면 11 x 107 세포가 필요함). 최소 3 회 계대 후에 세포를 사용하고 세포를 20 계대 미만으로 유지하는 것이 좋습니다. HEK293T 세포는 일주일에 세 번 분할해야 합니다. 세포를 유지하는 동안 이들은 70%-80% 컨플루언스를 초과해서는 안 됩니다. AAV 제제에 사용 된 DMEM에는 이미 L- 글루타민과 나트륨 피루 베이트가 보충되어야합니다. - 50mL 튜브에 세포를 수집하고 RT에서 350 x g 에서 5분 동안 원심분리합니다.
- 10% FBS, 1% P/S 및 25mM HEPES가 보충된 20mL의 DMEM에 세포를 현탁하고 혈구계로 계산합니다. 죽은 세포 배제를 위해 트리판 블루를 사용하십시오.
- 175cm2 플라스크에 10% FBS, 1% P/S 및 25mM HEPES가 보충된 DMEM 20mL에 3 x 106개의 세포를 시드합니다. 하나의 바이러스를 생산하려면 적어도 4 개의 175cm2 플라스크를 준비하십시오. 세포를 3일 동안 37°C/5%CO2에서 배양한다.
참고: 이 단계는 주말에 셀을 확장할 수 있으므로 금요일에 수행하는 것이 가장 좋습니다. - HEK293T 세포를 수확하고 계수한다.
- 바이러스 중 하나를 준비하려면 10% FBS, 1% P/S 및 25mM hepes가 보충된 DMEM 20mL에 각각 11 x 106 HEK293T가 포함된 10개의 150mm 접시를 준비합니다.
- 접시를 37 ° C / 5 % CO2 의 인큐베이터에 24 시간 동안 놓습니다. 기존 배지를 조심스럽게 버리고 10% FBS, 25mM hepes 및 1mM 부티르산 나트륨이 보충된 무항생제 DMEM 20mL로 교체합니다. 계속하기 전에 셀의 합류도가 80 %를 넘지 않는지 확인하십시오.
- 형질주입 혼합물을 포함할 두 개의 15mL 튜브를 준비합니다. 튜브 1에 환원된 혈청 배지 5mL, rAAV6 돌연변이 HDR 플라스미드 60μg 및 pDGM6(헬퍼 플라스미드) 220μg을 추가합니다. 튜브 2에 환원된 혈청 배지 5mL와 1mg/mL 폴리에틸렌이민 용액(PEI, 형질주입 시약) 1120μL를 추가합니다.
- 튜브 2의 내용물을 튜브 1에 추가하고 30초 동안 소용돌이합니다. PEI 미셀에 DNA가 적절하게 캡슐화되도록 RT에서 15분 동안 배양합니다.
알림: 용액을 20분 이상 보관하지 마십시오. - 각 접시에 용액 1.1mL를 조심스럽게 적가하고 부드럽게 소용돌이 치며 분배합니다. 접시를 37 ° C / 5 % CO2 의 인큐베이터에 48 시간 동안 놓습니다.
- 48시간 후, 각 접시에 0.5M EDTA 250μL를 넣고 접시를 인큐베이터에 10분 동안 둡니다. 접시에서 세포를 씻어 수확하고 500mL 원심분리 튜브 또는 여러 개의 50mL 튜브로 옮깁니다.
- 2,000 x g 에서 4 °C에서 10분 동안 원심분리합니다. 상청액을 폐기하십시오. 상청액의 완전한 제거를 보장하기 위해, 4°C에서 1분 동안 2,000 x g 에서 다시 원심분리한다.
- 남아 있는 상청액을 폐기하십시오. 잔류 상청액은 바이러스 정제를 손상시킬 수 있습니다.
- 와동하여 펠릿을 풀고 제조업체의 지침에 따라 AAV 정제 키트(재료 표 참조)를 사용하여 바이러스를 추출합니다. 대안적으로, 요오드 구배 초 원심 분리30,31에 의한 추출을 수행하십시오.
- 정제된 바이러스를 분취하고, -80°C에서 보관한다. 최적의 형질도입 조건을 결정하기 위해 AAV를 기능적으로 적정하거나 디지털 액적 PCR(ddPCR)32로 AAV 역가를 정량화합니다.
- rAAV6 WT HDR 플라스미드의 제조를 위해 이 과정을 반복한다.
- ddPCR에 의한 AAV 적정
- 시작하기 전에 가열 블록을 65 ° C 및 98 ° C로 설정하십시오.
- 바이러스 5μL에 DNA 추출 용액 15μL를 첨가하여 바이러스 DNA를 추출합니다. 15 초 동안 소용돌이.
- 65°C에서 6분 동안 배양합니다. 15 초 동안 소용돌이. 98°C에서 2분 동안 배양합니다.
- 추출된 DNA(바이러스 DNA의 1:4 희석)를 사용하여 ddPCR을 위해 뉴클레아제가 없는(nf)H2O로 연속 희석액(1:400, 1:40,000, 1:160,000, 1:640,000)을 준비합니다.
참고: ddPCR이 즉시 수행되지 않는 경우 희석액을 -20°C에서 보관할 수 있습니다. - ddPCR에 대한 희석액 중 세 가지를 선택합니다. 사용할 희석액의 선택은 바이러스의 농도에 따라 다릅니다. 바람직하게는, 기계의 검출 한계 내에 있는 최상의 희석을 찾기 위해 세 가지 상이한 희석물(예를 들어, 1:40,000, 1:160,000 및 1:640,000)을 사용한다.
- 작업하는 동안 시약을 얼음 위에 보관하십시오. 마스터 믹스를 준비하십시오 (모든 샘플은 중복으로 측정됩니다). 각 반응에 대해 프로브용 ddPCR 슈퍼믹스 12.5μL, dUTP 없음, 프라임타임 표준 qPCR 분석 1.25μL, AAV-ITR(재료 표 참조) 및 6.25μL nf-H2O를 준비합니다.
- 5μL의 DNA와 20μL의 마스터 믹스를 혼합합니다. 1:40,000, 1:160,000 및 1:640,000 희석에 대해 이 작업을 수행합니다.
- 카트리지 홀더에 카트리지를 넣습니다. 70μL의 액적 생성 오일을 오일로 표시된 행의 프로브에 추가합니다. 거품이 발생하지 않도록 주의하십시오.
- 샘플로 표시된 행에 샘플 부피의 20μL를 추가합니다. 거품이 발생하지 않도록 주의하십시오.
- 개스킷으로 카트리지를 밀봉하고 액적 발생기에 넣으십시오. 기계의 시작 버튼을 눌러 액적을 생성하고 개스킷을 제거한 다음 다중 채널 피펫으로 생성된 액적 40μL를 96웰 플레이트로 조심스럽게 옮깁니다. 물방울과 기포가 파괴되지 않도록 천천히 작업하십시오.
- PCR 플레이트 실러를 사용하여 피어스 호일(빨간색 줄무늬가 위를 향함)로 96웰 플레이트를 밀봉합니다. 플레이트를 열 순환기에 놓습니다.
- 볼륨을 40μL로, 뚜껑 온도를 105°C로, 램프 속도를 2°C/s로 설정합니다. 표 2에 설명된 대로 PCR 프로그램을 실행하십시오.
- 사용하기 30분 전에 액적 판독기를 켜십시오. 바탕 화면에서 소프트웨어를 엽니다.
- 플레이트 레이아웃을 입력하고 실험 탭에서 ABS 를 선택하고 슈퍼믹스 탭에서 ddPCR 슈퍼믹스 를 선택합니다. 그런 다음 먼저 PRIME을 누른 다음 소프트웨어에서 시스템 플러시 를 클릭하여 시스템을 시작합니다.
- 플레이트를 리더에 입력하십시오. 실행을 시작하고 완료되면 후속 분석을 위해 데이터를 CSV 파일로 스프레드시트로 내보냅니다.
참고: 소프트웨어에서 생성된 숫자는 μL당 게놈 사본(GC)입니다. 여기에 희석 계수를 곱해야 합니다: GC/μL x 5(마스터 믹스에서 DNA 희석) x 초기 희석(즉, 40,000 또는 160,000).
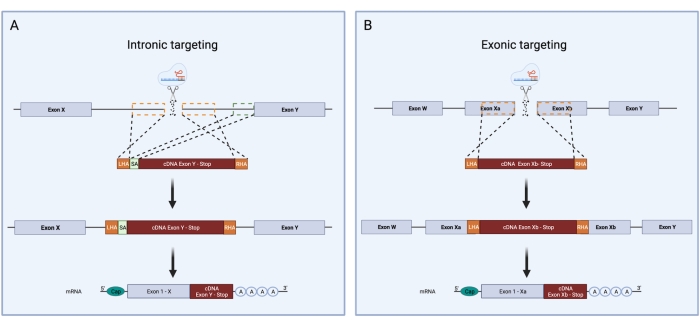
그림 3: CRISPR/Cas9 HDR 녹인 중 인트론 및 엑소닉 타겟팅의 개략적인 개요. CRISPR/Cas9 HDR 녹인을 위한 인트로닉 및 엑소닉 타겟팅 전략 간의 개략적 비교. (A) 인트론 표적화 동안, 이중 가닥 절단이 DNA의 인트론에 도입된다. HDR 템플릿은 LHA, cDNA 서열 및 RHA로 구성됩니다. 인트론 표적화는 추가적으로 3' 스플라이스 부위, 분기점 및 폴리피리미딘 관을 포함하는 슬라이스 수용체의 존재를 필요로 합니다. 이렇게 하면 올바른 접합이 가능합니다. 녹색 점선 상자는 SA 시퀀스에 해당하는 영역을 나타냅니다. SA의 크기는 150bp입니다. (B) 엑소닉 타겟팅은 엑손에서 직접 이중 가닥 절단의 생산에 의존합니다. HDR 템플릿은 LHA, cDNA 서열 및 RHA로 구성됩니다. 주황색 점선 상자는 LHA 및 RHA에 해당하는 영역을 나타냅니다. HA의 이상적인 크기는 각각 400bp입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 발기인 | ||||
| 이름 | 길이 | 묘사 | ||
| 증권 시세 표시기 | 492 bp | 비장 초점 형성 바이러스 프로모터. 강력한 포유류 프로모터. 구성적으로 표현 | ||
| 증권 시세 표시기 | 400 bp | 인간 유비퀴틴 C 유전자로부터 유래된 프로모터. 구성적으로 표현됩니다. | ||
| 증권 시세 표시기 | 508 bp | 거대 세포 바이러스에서 유래 한 프로모터. 인핸서 영역을 포함할 수 있다. 구성적으로 표현됩니다. 강력한 포유류 프로모터. 침묵시킬 수 있습니다. | ||
| EF-1a | 1182 bp | 인간 진핵 번역 신장 인자 1 알파 프로모터. 구성적으로 표현됩니다. 강력한 포유류 프로모터. | ||
| 증권 시세 표시기 | 200-300 bp | EF-1 알파 인트론리스 약식 | ||
| 전능신교 | 584 bp | 하이브리드 포유동물 프로모터는 CMV 조기 인핸서 (C), 닭 베타 액틴 프로모터 (A), 및 토끼 베타글로빈 유전자에 대한 스플라이스 수용체 (G)를 포함한다. 구성적으로 표현됩니다. | ||
| 폴리아데닐화(PolyA) 신호 | ||||
| 약어 | 길이 | 묘사 | ||
| SV40 폴리아 | 82-122 bp | 유인원 바이러스 40 폴리아데닐화 신호 | ||
| bGH 폴리아 | 224 bp | 소 성장 호르몬 폴리아데닐화 신호 | ||
| rbGlob PolyA | 56 bp | 토끼 베타 글로빈 폴리아데닐화 신호 | ||
표 1: 프로모터 및 폴리아데닐화 신호.
| 걸음 | 온도 | 시간 | 사이클 |
| 효소 활성화 | 95°C | 10분 | 1배 |
| 변성 | 94°C | 30초 | 42배 |
| 어 닐 링 | 60°C | 1분 | |
| 확장 | 72°C | 30초 | |
| 효소 비활성화 | 98°C | 10분 | 1배 |
| 들다 | 4°C | ∞ |
표 2: 디지털 액적 PCR 프로그램.
3. HSPC의 편집
- HSPC의 형질주입 및 형질도입
- CD34+ HSPC를 해동하고 RT에서 350 x g 의 10분 동안 1% P/S. 원심분리기가 보충된 사전 예열된 RPMI 10mL로 세포를 옮깁니다.
- 세포를 HSPC 머무름 배지에 2.5 x 10 5 cells/mL의 농도로 현탁시키고 37°C/5 % CO2에서 48시간 -72 시간 동안 배양합니다.
- 15mL 튜브에서 세포를 수확합니다. 세포를 계수하고 트리판 블루 배제에 의한 세포 생존율을 확인한다. 2 x 10 5 및5 x 10사이에서 6 개의 세포가 단일 큐벳에서 핵 형성 될 수 있습니다. 계산된 셀 번호에 따라 적절한 수의 큐벳을 준비하십시오.
- RNP 복합체를 준비하십시오. 1.5mL 튜브에 15μg의 Cas9와 8μg의 sgRNA(몰비 1:2.5)를 넣고 25°C에서 가열 블록에서 10분 동안 배양합니다.
- RNP 복합체가 배양되는 동안, 세포를 350 x g 에서 5분 동안 원심분리하고 상청액을 버린다.
- 세포를 100 μL의 뉴클레오펙션 용액에 현탁시킨다. 세포를 RNP 복합체와 혼합하고 큐벳으로 옮깁니다.
- 큐벳을 부드럽게 두드려 전사 중에 형성되었을 수 있는 잔류 기포를 제거합니다.
- 큐벳을 형질감염 시스템의 홀더에 삽입하고 sgRNA 설계 섹션에서 이전에 설명한 대로 세포를 전기천공합니다.
- 전기천공 직후, P/S가 없는 예열된 HSPC 머무전 배지 400μL를 추가하고 미세 전달 피펫으로 세포를 P/S가 없는 예열된 HSPC 머무름 배지 500μL가 포함된 조직 배양 플레이트로 옮깁니다. 세포 수에 따라 0.25 x 10 6과 1 x 106 cells/mL 사이의 밀도에 도달하기 위해 적절한 배양 플레이트(24웰, 12웰 또는6웰 플레이트)를 사용합니다. 플레이트를 37 °C / 5 % CO2의 인큐베이터로 옮깁니다.
- 얼린 rAAV가 들어 있는 바이알을 얼음 위에서 해동합니다. 각 rAAV6의 최적 양을 세포 현탁액에 피펫팅하여 세포를 형질도입합니다. 높은 형질 도입 효율을 얻기 위해 전기 천공 후 20-30 분 이내에 형질 도입을 수행하십시오.
참고 : 각 바이러스의 최적 농도 (WT HDR 템플릿의 경우 하나의 바이러스와 돌연변이 된 HDR 템플릿의 경우 다른 별도의 바이러스)는 실험적으로 결정되어야합니다. 보통, 5,000-10,000 GC/셀 (예를 들어, 1 x 106 셀에 대해 5 x 109 GC)은 높은 형질도입을 초래한다. - 세포 현탁액을 피펫과 부드럽게 혼합합니다. 형질도입된 세포를 37°C/5%CO2에서 6-8시간 동안 배양한다. 6-8 시간 후, 튜브에서 세포를 수집하고 RT에서 350 x g 에서 5 분 동안 원심 분리합니다.
- 기존 배지를 폐기하고 P/S가 보충된 새로운 예열된 HSPC 보유 배지로 교체하십시오.
- 세포를 2.5 x 10 5-5 x 105 cells/mL 사이의 농도로 현탁시키고 조직 처리된 세포 배양 플레이트(24, 12 또는 6웰 플레이트)로 옮깁니다. 분류를 진행하기 전에 세포를 37°C/5%CO2에서 48시간 동안 배양한다.
- 이형접합 GOF 돌연변이를 지닌 조작된 HSPC의 유동 분류
- 15mL 튜브에서 세포를 수확하고 RT에서 350 x g 에서 5분 동안 원심분리합니다. 상청액을 제거하고, 세포를 0.1% BSA를 함유하는 1mL의 DPBS에 현탁시키고, RT에서 350 x g 에서 5분 동안 다시 원심분리한다.
- 상청액을 버리고 세포 수에 따라 적절한 부피의 DPBS + 0.1% BSA에 세포를 현탁시킨다.
참고: 분류기 막힘 위험을 줄이기 위해 최소 부피 200μL로 세포를 현탁하고 1 x 107 cells/mL를 초과하지 않는 것이 좋습니다. - 세포를 캡이 장착 된 멸균 FACS 튜브로 옮깁니다. 7-AAD 또는 기타 생존 염료(형광 단백질과의 상용성에 따라 다름)를 생존/사멸 세포 배제를 위해 세포 현탁액에 추가합니다.
- 형광 리포터 단백질에 대해 이중 양성인 살아있는 세포를 200μL의 HSPC 머무름 배지가 들어 있는 수집 튜브에 분류합니다.
참고: 분류 절차 중에 적절한 게이팅을 보장하기 위해 AAV로만 형질도입된 셀로 구성된 적절한 컨트롤을 추가해야 합니다. - 분류된 세포를 RT에서 350 x g 로 5분 동안 원심분리하고 배양에서 추가 확장을 위해 2.5 x 105 cells/mL 농도의 HSPC 보존 배지에 세포를 현탁하거나 기능 분석에 세포를 직접 사용합니다.
4. 유전자 편집 성공 확인
- 게놈 DNA 추출
- 시작하기 전에 가열 블록을 65 ° C 및 98 ° C로 설정하십시오. 2 x 10 5 게놈 편집 및 정렬 정제 세포를 수확하고 350 x g에서5분 동안 원심분리합니다.
- 앞서 설명한 대로 gDNA를 추출합니다. DNA 용액을 PCR에 직접 사용하거나 -20 ° C에서 보관하십시오.
- 인-아웃 PCR
- 5' 삽입 부위를 증폭하기 위해 2개의 프라이머를 디자인한다(도 4A): 프라이머 전방 1은 좌측 상동성 아암 외부의 게놈 유전자좌를 표적화하고; 프라이머 역방향 2는 통합 시퀀스를 표적으로 합니다.
- 인-아웃 PCR의 경우 반응당 다음 PCR 믹스를 준비합니다.
10 μL의 PCR 마스터 믹스 (2x, 재료 표 참조)
1 μL 정방향 프라이머 1 (10 μM 스톡)
1 μL 프라이머 리버스 2 (10 μM 스톡)
추출된 DNA 0.5-1.0 μL
뉴클레아제가 없는H2O내지 최종 부피 20 μL
참고: 하나 이상의 반응의 경우, 피펫팅 오류를 설명하기 위해 하나의 추가 반응을 포함하는 마스터 믹스를 준비하는 것이 좋습니다. 모의 처리된 샘플에서 비주형 대조군 및 주형 DNA를 포함하는 것이 중요합니다. - 적절한 열 순환 프로그램으로 PCR 반응을 실행하십시오(제조업체 지침 참조).
알림: 먼저 최적의 어닐링 온도에 대해 프라이머 쌍을 테스트하는 것이 좋습니다. 이것은 Tm을 계산 한 다음 기울기 온도를 수행 할 수있는 열 순환기로 PCR을 실행하여 수행 할 수 있습니다. - 100V에서 45-60분 동안 1.5% 아가로스 겔에서 DNA 사다리가 있는 PCR 산물을 실행합니다(그림 4B). 젤을 청색광 또는 UV 투과기에 놓습니다. 젤에서 밴드를 빼십시오.
- DNA 겔 추출 키트를 사용하여 밴드에서 DNA를 추출합니다. 추출된 PCR 샘플을 Sanger 시퀀싱을 위한 적절한 프라이머와 함께 보내 내인성 유전자 좌위에서 원하는 cDNA의 정확하고 원활한 통합을 확인합니다.
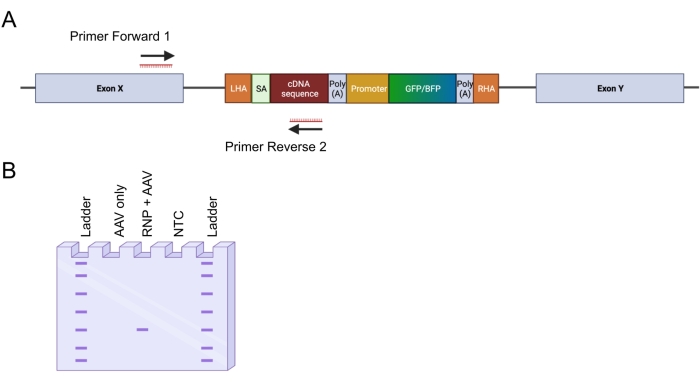
그림 4: 인-아웃 PCR에 의한 게놈 통합 검증. (A) 인-아웃 PCR 전략의 개략적 표현. 묘사 된 전략에서 두 개의 프라이머가 설계되었습니다. 프라이머 포워드 1은 LHA 외부의 게놈 유전자좌를 표적으로 하고, 프라이머 역방향 2는 코돈 최적화 서열을 표적으로 한다. (B) 아가로스 겔 전기영동의 개략적인 표현. 성공적으로 편집된 세포(RNP + AAV)만 인아웃 PCR 동안 PCR 산물을 생성하는 반면, 편집되지 않은 샘플(AAV만 해당)은 PCR 산물을 생성하지 않습니다. 약어 : NTC = 비 템플릿 제어. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
상술한 프로토콜을 적용함으로써, 이형접합체 제1형 CALR 돌연변이가 제대혈 유래 HSPC에 재현가능하게 도입되었다. 이 돌연변이는 엑손 9 (CALR의 마지막 엑손)에서 52bp 결실로 구성되며, 이는 +1 프레임 시프트를 초래하여 새로운 양전하를 띤 C- 말단 도메인26,33의 번역으로 이어집니다. 내인성 유전자 좌위에서 CALR 돌연변이를 도입하기 위해 Exon 9의 업스트림 인트로닉 표적화 전략이 채택되었는데, 이는 Cas9 유도 DSB가 HDR 메커니즘을 통해 복구되지 않은 경우 코딩 서열의 원치 않는 변경을 피할 수 있기 때문입니다. 이러한 특정한 경우에, 인트론 7에 대한 sgRNA는 유리한 상동성 아암과 조합된 높은 온-타겟 및 낮은 오프-타겟 서열의 이용 가능성으로 인해 설계되었다 (서열 반복의 결여; 그림 5A).
그런 다음 두 개의 기증자 템플릿을 AAV6 벡터로 설계 및 패키징했습니다. 내인성 엑손으로부터 통합된 cDNA로의 정확한 스플라이싱을 가능하게 하기 위해, 공여자 주형은 (i) 3' 스플라이스-부위, 분기점 및 폴리피리미딘 관을 포함하는 SA 서열, (ii) WT(CALRWT) 또는 돌연변이된 서열(CALRDEL ) 정지 코돈을 포함하는, (iii) 유인원 바이러스 40 (SV40) polyA 신호, (iv) 별개의 내부 프로모터의 조절하에 형광 단백질을 암호화하는 서열, 비장 포커스형성 바이러스 (SFFV) 프로모터, 이어서 (v) 소 성장 호르몬 (bGH) polyA 신호. CALR WT cDNA를 함유하는 공여체 템플릿은 GFP 카세트를 함유하도록 설계된 반면, CALRDEL cDNA 서열을 함유하는 공여체 템플릿은 BFP 카세트를 함유하도록 설계되었다. 전체 구축물은 좌우 HA에 의해 측면에 있었다(도 5A).
RNP 복합체를 사용한 형질감염 및 rAAV6 바이러스로의 형질도입 이틀 후, 세포를 유동 세포분석에 의해 분석하였다. 4개의 주요 집단이 검출될 수 있었다: (i) HDR 기반 게놈 편집이 없는 세포를 나타내는 GFP 또는 BFP를 발현하지 않는 세포, (ii) WT 구축물만을 통합한 세포를 나타내는 GFP에 대해서만 양성인 세포, (iii) 돌연변이된 구축물만을 통합한 세포를 나타내는 BFP에 대해서만 양성인 세포, 및 (iv) GFP 및 BFP 이중 양성 세포, 는 WT 및 돌연변이된 서열 모두를 통합한 세포를 나타낸다(도 5B). 이형접합체 유형 1 CALR 돌연변이를 갖는 HSPC의 순수한 집단을 얻기 위해, 이중 양성 세포를 유동 세포분석에 의해 분류하였다. 2개의 WT 서열이 녹인된 HSPC를 대조군 세포(GFP+ mCherry+; 그림 5B). 대조군 세포로서 사용하기 위한 유효한 대안은 세이프 하버 유전자좌에서 형광 단백질의 이중대립유전자 통합을 갖는 HSPC일 것이다(즉, AAVS1; 도시되지 않음). 분류된 HSPC에 대해 수행된 트리판 블루 배제에 의한 세포 계수는 세포의 90% 이상이 생존 가능하다는 것을 나타내었다.
구축물의 원활한 온-타겟 통합은 인-아웃 PCR 전략을 적용하여 확인하였다(도 6A). 이 특정 경우에, 우리는 녹 인 CALR WT 서열 (도 6A에서 겔 전기 영동의 레인 1)과 녹 인 CALRDEL 서열 (도 6A의 겔 전기 영동의 레인 2)에 대한 두 개의 분리 된 인-아웃 PCR을 수행했다. 겔 밴드에서 추출한 DNA에 대해 수행된 Sanger 시퀀싱은 CALRDEL/WT HSPC에서 WT 및 돌연변이 서열의 올바른 삽입을 확인했습니다(그림 6B).
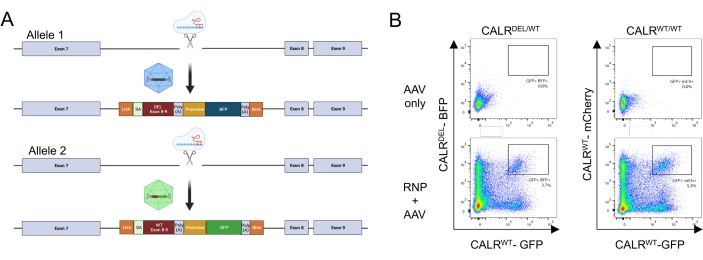
그림 5: 이형접합 CALR 돌연변이를 갖는 HSPC의 생성. (A) 이형접합 CALR 돌연변이의 삽입을 위한 편집 전략을 묘사하는 대표적인 도식. RNP 복합체는 CALR 유전자의 엑손 7과 엑손 8 사이의 인트론을 표적으로 합니다. 돌연변이된 엑손 8-9 및 BFP를 포함하는 AAV와 WT 엑손 8-9 및 GFP를 포함하는 두 개의 AAV는 공여체 복구 주형으로 작용하며 하나의 대립유전자에서 돌연변이된 서열의 통합과 나머지 대립유전자에 WT 서열의 통합을 촉진합니다. (B) HSPC의 형질감염 및 형질도입 후 GFP 및 BFP 또는 GFP 및 mCherry 48h의 발현을 나타내는 대표적인 유세포분석 플롯. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
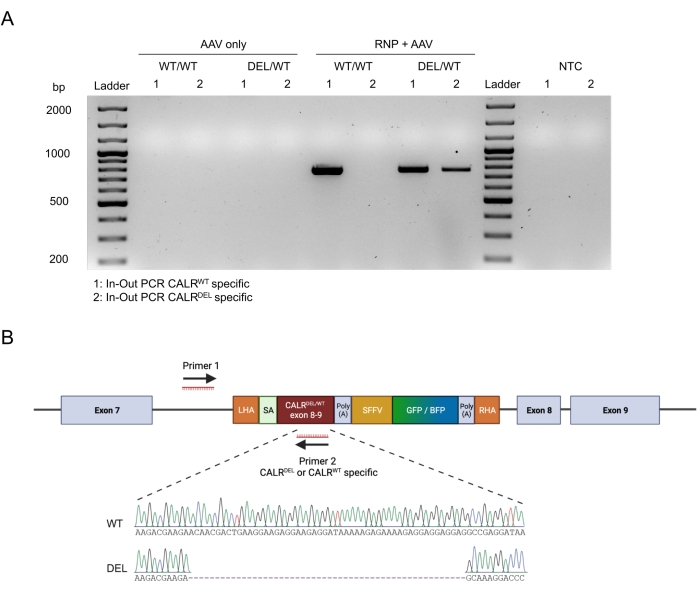
그림 6: HSPC에서 성공적인 이형접합체 CALR 돌연변이의 검증. (A) AAV 대조군, CALR WT/WT 및 CALRDEL/WT에서 추출한 게놈 DNA에 대해 수행된 인-아웃 PCR 산물의 겔 전기영동. 100 bp DNA 사다리를 사용하였다. 약어 : NTC = 비 템플릿 제어. (B) WT 및 돌연변이 서열의 성공적인 통합을 확인하는 CARRDEL/WT에서 수행된 인아웃 PCR로부터 얻은 Sanger 시퀀싱 결과. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
인간 1차 HSPC의 효율적이고 정확한 유전자 조작은 정상적인 조혈, 그리고 가장 중요한 것은 조혈 세포의 백혈병 변형에 영향을 미치는 과정을 탐구하고 이해할 수 있는 좋은 기회를 나타냅니다.
이 프로토콜에서, 재발성 이형접합체 GOF 돌연변이를 발현하도록 인간 HSPC를 조작하는 효율적인 전략이 설명되었다. 이 절차는 CRISPR/Cas9 기술과 rAAV6 벡터를 DNA 주형의 기증자로 활용하여 WT 및 돌연변이 DNA 서열을 내인성 유전자 좌위에 정확하게 삽입했습니다. 조작된 cDNA(WT 및 돌연변이)를 분리된 형광 리포터 단백질과 결합하면 최종 이형접합체 상태의 세포를 농축하고 추적할 수 있습니다.
이 전략은 자주 사용되는 렌티바이러스(LV) 기반 방법에 비해 몇 가지 이점을 제공합니다. 한 가지 주요 이점은 CRISPR/Cas9 기반 시스템이 내인성 유전자좌에서 정밀한 편집을 허용하여 내인성 프로모터 및 조절 요소를 보존할 수 있다는 것입니다. 이것은 세포에서 편집 된 유전자의 발현에서 균질성을 유도하며, LV 기반 방법을 사용할 때 거의 달성 할 수없는 목표입니다. LV 벡터를 사용한 유전자 전달은 전사 활성 부위34에 대한 선호도와 유전자의 반무작위 통합을 유도한다. 이것은 전달 된 유전자의 과발현과 편집 된 세포 간의 이질성으로 해석되어 결국 돌연변이 및 유전자 상호 작용의 역할을 조사하고 분석하는 데 어려움을 초래할 수 있습니다. 두 번째 이점은 설명된 시스템이 부위 특정 편집 시스템이기 때문에 삽입 돌연변이유발(35)의 위험을 제거한다는 것이다.
이중 형광 리포터 전략은 두 대립 유전자에서 성공적으로 편집 된 세포의 정확한 농축 및 추적을 허용하며, 하나의 대립 유전자는 WT cDNA를 통합하고 다른 대립 유전자는 돌연변이 된 cDNA 서열을 통합합니다. 단일 리포터만을 발현하는 세포는 단일 대립유전자 통합 또는 동일한 형광 리포터와의 HDR 템플릿의 이중대립유전자 통합을 나타냅니다. 두 시나리오 모두 단일 세포 유래 클론을 생산하고 개별적으로 분석하는 경우에만 정확하게 구별할 수 있습니다. 그러나 HSPC는 시험관 내에서 증식 능력이 제한적이며, 장기간 배양 상태를 유지하면 HSPC는보다 성숙한 자손으로 분화하기 시작하고자가 재생 및 생착 능력을 잃습니다. 이것은 원하는 이형 접합 돌연변이를 보유하고있는 단일 세포 클론을 선택하고 확장하는 것을 불가능하게 만든다. 이형접합체 돌연변이를 지닌 세포에 대한 이중 형광 단백질 전략 및 유세포분석에 의한 농축의 적용은 연장된 시험관 내 배양에 의해 유도된 문제를 우회할 수 있게 한다.
이 특정 예에서, 이형 접합 CALRDEL / WT 돌연변이를 운반하는 HSPC의 순수한 집단을 얻기 위해 HSPC가 효율적으로 조작되고 분류 될 수 있음을 성공적으로 입증했습니다.
그러나 이 시스템은 공학적 이형접합체 프레임시프트 돌연변이에 국한되지 않고 미센스 및 넌센스 돌연변이를 포함한 다른 돌연변이 유형을 생성하기 위해 쉽게 채택될 수도 있습니다. WT를 함유하는 AAV의 상이한 조합 또는 상이한 형광 리포터 단백질을 갖는 돌연변이된 서열을 적용함으로써, 이 시스템은 또한 동형접합 돌연변이(돌연변이 cDNA를 운반하지만 상이한 형광 리포터를 운반하는 2개의 rAAV를 사용한 동시 형질도입) 또는 심지어 돌연변이의 교정(WT cDNA를 운반하지만 상이한 형광 리포터를 갖는 2개의 AAV를 사용한 동시 형질도입)의 도입에도 활용될 수 있다. 또한이 전략이 발암 성 GOF 돌연변이의 도입에만 국한되지 않는다는 점을 언급하는 것이 중요합니다. 실제로, 기술된 프로토콜은 유전자 녹아웃, 유전자 대체(36,37), 전이유전자(즉, 키메라 항원 수용체)의 표적화된 녹인(knock-in)38), 심지어 질병을 유발하는 돌연변이(11,39)의 교정을 포함하는 다수의 대안적 전략에 활용될 수 있다.
CRISPR/Cas9 및 AAV6를 다중 형광 리포터와 결합하는 전략은 T 세포, 형질모세포양 수지상 세포, 유도만능줄기세포, 신경 줄기세포 및 기도 줄기 세포를 포함한 다른 많은 세포 유형에도 적용 가능한 것으로 나타났습니다. 24,38,40,41,42,43,44 . 이 전략은 우수한 키메라 항원 수용체 (CAR) T 세포의 생산을 위해 구현될 수 있다. 예를 들어, CAR T 세포에서 TGFBR2 유전자의 CRISPR/Cas9-매개 녹아웃이 억제성 TGF-β 풍부한 종양 미세환경에서 이들의 기능을 크게 증가시킨다는 것이 최근에 발표되었다45. 이러한 접근법은 CAR을 발현하도록 T 세포를 조작하고 TGFBR2 유전자의 두 대립 유전자 모두에 CAR을 특이적으로 삽입함으로써 TGFBR2 유전자를 녹아웃하는 원스텝 프로토콜을 제공할 수 있다. 더욱이, 이러한 접근법은 또한 T 세포 수용체 알파 상수 (TRAC) 유전자46,47에 CAR을 통합함으로써 보편적인 CAR T 세포를 생성하는데 유용할 수 있다.
재현성을 높이고 세포의 효율적인 편집을 보장하려면 몇 가지 중요한 고려 사항을 처리해야 합니다. 세포의 성공적인 편집을 보장하기 위한 주요 중요 포인트는 (i) sgRNA의 선택, (ii) HDR 템플릿의 설계 및 (iii) rAAV6 생산에 있습니다.
우수한 성능의 sgRNA를 선택하는 것은 HDR 템플릿이 통합될 수 있는 최대 대립 유전자 수를 결정하기 때문에 중요합니다. 현재 사용할 수 있는 수많은 소프트웨어로 인해 후보 sgRNA에 대한 검색이 단순화되었습니다. 관심 영역을 선택함으로써, 소프트웨어는 각각 원하는 유전자좌 및 원치 않는 유전자좌에서 편집할 기회를 나타내는 온-타겟 스코어 및 오프-타겟 스코어를 갖는 일련의 sgRNA를 제안할 수 있다. 이러한 점수는 이전에 공개된 점수 모델(48,49)에 기초하여 계산된다. 이것이 우수한 성능의 sgRNA를 선택하기 위한 좋은 출발점이지만, 실리코에서 예측된 성능이 시험관 내에서 효율적인 sgRNA와 항상 일치하는 것은 아니기 때문에 sgRNA의 성능을 확인해야 합니다. 따라서 최상의 sgRNA를 찾을 가능성을 높이기 위해 최소 3개의 sgRNA를 설계하고 테스트하는 것이 좋습니다. 실제로 우수한 성능의 sgRNA가 확인되면 HDR 템플릿의 설계를 진행하는 것이 좋습니다.
HDR 템플릿을 디자인할 때 예방 조치를 고려해야 합니다. 왼쪽 및 오른쪽 상동성 암(각각 LHA 및 RHA)은 각각 sgRNA 절단 부위의 업스트림 및 다운스트림에 걸쳐 있어야 하며, HA가 짧을수록 HDR 주파수가 감소할 수 있습니다. HDR을 통해 도입할 수 있는 cDNA의 크기는 AAV의 패키징 기능에 따라 달라지며, 이는 약 4.7kb입니다. HDR 템플릿 내에 필수적인 수많은 요소 (LHA, RHA, SA, PolyA, 프로모터 및 형광 리포터 서열)로 인해, 돌연변이 또는 WT cDNA를 위한 나머지 공간은 제한적이다. 이것은 원하는 돌연변이가 유전자의 3' 말단 근처 또는 전체적으로 짧은 CDS를 갖는 유전자에 위치하는 경우에는 문제가 되지 않는다. 그러나, 돌연변이가 긴 CDS (AAV의 나머지 패킹 공간 초과)를 갖는 유전자의 전사 개시 측 (TSS) 근처에있는 경우,이 설명 된 접근법은 실현 가능하지 않을 수있다. 이 문제를 피하기 위해 HDR 템플릿을 두 개의 AAV로 분할하는 전략이 최근 Bak과 동료들에 의해 개발되었습니다. 이 전략은 큰 유전자(50)의 최종 원활한 통합을 얻기 위해 2개의 개별 HDR-매개 통합에 의존한다.
바이러스의 품질과 역가는 세포의 성공적인 게놈 공학을 만들거나 깨뜨릴 수 있는 추가 요소입니다. 최적의 수율을 위해서는 HEK293T가 배양 상태를 유지하면서 완전한 컨플루언스에 도달하지 않도록 하는 것이 중요합니다. 이상적으로는 HEK293T 세포가 70%-80% 컨플루언시에 도달할 때 분할되어야 합니다. 또한 HEK293T는 바이러스 생성 능력을 감소시킬 수 있으므로 장기간 배양해서는 안 됩니다. 새로운 HEK293T 세포는 20 회 계대 후에 해동해야합니다. 높은 바이러스 역가를 얻는 것은 실험의 효율성과 재현성을 높이는 데 중요합니다. 낮은 바이러스 역가는 HSPC의 형질도입에 필요한 대량의 rAAV 용액으로 해석됩니다. 일반적으로, 핵 제거된 세포에 첨가된 rAAV 용액은 HSPC 머무름 배지의 총 부피의 20%를 초과해서는 안 된다. 더 많은 양의 AAV 용액은 세포 사멸 증가, 증식 감소 및 형질 도입 효율 장애로 이어질 수 있습니다. 따라서 바이러스 역가가 낮은 경우 바이러스를 더욱 집중시키는 것이 좋습니다.
요약하면, 이 프로토콜은 추가 이중 형광 리포터와 함께 CRIPSR/Cas9 및 rAAV6 공여체 템플릿을 동시에 사용하여 인간 HSPC를 정확하고 효율적으로 조작할 수 있는 재현 가능한 접근 방식을 제공합니다. 이 접근법은 정상적인 조혈 줄기 세포 생물학과 돌연변이가 백혈구 생성에 기여하는 것을 연구하는 데 훌륭한 도구임이 입증되었습니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 연구는 오스트리아 과학 기금 (FWF, 번호 P32783 및 I5021)의 AR에 대한 보조금으로 지원됩니다. A.R.에 대한 추가 자금은 오스트리아 내과 학회 (Joseph Skoda Fellowship), 오스트리아 혈액 종양 학회 (OeGHO; 임상 연구 보조금) 및 MEFOgraz. T.K.는 백혈병 및 림프종 학회의 특별 펠로우입니다.
자료
| Name | Company | Catalog Number | Comments |
| 175 cm2 Cell Culture Flask, Vent Cap, TC-treated | Corning | 431080 | |
| 150 mm x 25 mm dishes | Corning | 430599 | |
| 293T | DSMZ | ACC 635 | https://www.dsmz.de/collection/catalogue/details/culture/ACC-635 |
| 4D Nucleofector Core Unit | Lonza | - | For nucleofection of human HSPCs use the DZ-100 program. |
| 4D Nucleofector X Unit | Lonza | - | |
| 500 ml Centrifuge Tube | Corning | 431123 | |
| 7-AAD | BD Biosciences | 559925 | |
| AAVpro Purification Kit | Takara | 6666 | |
| Alt-R S.p. Cas9 Nuclease V3 | Integrated DNA Technologies (IDT) | 1081058 | |
| Avanti JXN-30 Ultracentrifuge | Beckman Coulter | - | |
| Benchling sgRNA design tool | Online tool for sgRNA design: http://www.benchling.com/crispr | ||
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906-100G | |
| C1000 Touch Thermal Cycler | Bio-Rad | - | |
| Chemically modified synthetic sgRNA | Synthego | Website: https://www.synthego.com/products/crispr-kits/synthetic-sgrna Sequence for the sgRNA targeting intron 7 of CALR: 5’-CGCCTGTAATCCTCGCCCAG-3’ An 80 nucleotide SpCas9 scaffold is added to the 20 nucleotide RNA sequence to complete the sgRNA. Chemical modifications of 2'-O-Methyl are added to the first and last 3 bases and 3' phosphorothioate bonds are added in the first 3 and last 2 bases. *Alternatively chemically modified synthetic sgRNAs can be acquired from IDT (https://eu.idtdna.com/site/order/oligoentry/index/crispr) and Trilink (https://www.trilinkbiotech.com/custom-oligos) | |
| CHOPCHOP sgRNA design tool | Online tool for sgRNA design: http://chopchop.cbu.uib.no | ||
| Costar 24-well Clear TC-treated Multiple Well Plates | Corning | 3526 | |
| CRISPick sgRNA design tool | Online tool for sgRNA design: https://portals.broadinstitute.org/gppx/crispick/public | ||
| CRISPOR sgRNA design tool | Online tool for sgRNA design: http://crispor.tefor.net | ||
| ddPCR 96-Well Plates | Bio-Rad | 12001925 | |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DG8 Cartridges for QX200/QX100 Droplet Generator | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 Droplet Generator | Bio-Rad | 1863009 | |
| DreamTaq Green PCR Master Mix (2X) | Thermo Scientific | K1081 | |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Dulbecco’s Modified Eagle Medium (DMEM) with high glucose | Sigma-Aldrich | D6429-6X500ML | |
| Dulbecco’s Phosphate Buffered Saline (DPBS) | Sigma-Aldrich | D8537-500ML | |
| FACSAria Fusion | BD Biosciences | - | |
| Falcon 5 mL Round Bottom | Corning | 352054 | |
| Fetal Bovine Serum (FBS) Good Forte (heat inactivated), 500 ml | Pan Biotech | P40-47500 | |
| FlowJo 10.8.0 | BD Biosciences | - | |
| GenAgarose L.E. | Inno-train | GX04090 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| Gibson Assembly Master Mix | New England Biolabs Inc. (NEB) | E2611L | |
| HEK293T | |||
| HEPES solution | Sigma-Aldrich | H0887-100ML | |
| ICE | Synthego | https://ice.synthego.com | |
| IDT codon optimization tool | IDT | https://www.idtdna.com/pages/tools/codon-optimization-tool | |
| IDT sgRNA design tool | Online tool for sgRNA design: https://www.idtdna.com/site/order/designtool/index/CRISPR_CUSTOM | ||
| LB Broth (Lennox) EZMix powder microbial growth medium | Sigma-Aldrich | L7658-1KG | |
| LB Broth with agar (Lennox) EZMix powder microbial growth medium | Sigma-Aldrich | L7533-1KG | |
| Midori Green Advance | Nippon Genetics | MG04 | |
| Monarch Plasmid Miniprep Kit | NEB | T1010L | |
| Monarch DNA Gel Extraction Kit | NEB | T1020L | |
| NEB 5-alpha Competent E. coli (High Efficiency) | NEB | C2987U | |
| Nuclease-Free Water, 5X100 ml | Ambion | AM9939 | |
| NucleoBond Xtra Midi | Macherey-Nagel | 740410 | |
| Opti-MEM, Reduced Serum Medium, 500 ml | Gibco | 31985070 | |
| P3 Primary Cell 4D-Nucleofetor X Kit L | Lonza | V4XP-3024 | The Lonza Primary P3 solution is supplied as a 2.25 mL P3 Primary Cell Nucleofector Solution and 0.5 mL Supplement 1. To reconstitute, add the Supplement 1 to the P3 Primary Cell Nucleofector Solution and mix. |
| pAAV-MCS2 | Addgene | 46954 | |
| PCR Plate Heat Seal Foil, pierceable | Bio-Rad | 1814040 | |
| pDGM6 | Addgene | 110660 | |
| Penicillin-Streptomycin (P/S) | Gibco | 15140122 | |
| Polyethylenimine (PEI) | Polysciences | 23966 | Add 50 mL of PBS 4.5 pH (made with HCl) to 50 mg of PEI in a tube. Dissolve by placing the tube in a 70°C water bath and vortexing every 10 minutes until the solution is dissolved. After the solution has reached RT, filter sterilze through a 0.22 μm filter, make 1120 μL aliquots, and store at -80°C. |
| Polystyrene Test Tube, with Snap Cap | |||
| Primers | Eurofins | - | Primers were ordered from Eurofins (eurofinsgenomics.eu) as unmodified salt free custom oligos. The primers were designed by using PRIMER-Blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) Primer 1 Fwd: AAGTGATCCGTTCGCCATGAC; Primer 2 Rev CALR WT specific: ACGTCCTCTTCCTCGTCCTC; Primer 2 Rev CALR DEL specific: CCAACCCTGGAGACACGCTTC |
| PrimeTime qPCR Primer Assay | IDT | - | PrimeTime qPCR Probe Assays (1 probe/2 primers) that can be ordered from IDT (https://eu.idtdna.com/site/order/qpcr/assayentry). Scale: Std - qPCR Assay 500 reactions; Primer 1 Forward (5'-3') : GGAACCCCTAGTGATGGAGTT; Primer 2 Reverse (5'-3'): CGGCCTCAGTGAGCGA; Probe (5'-3'): CACTCCCTCTCTGCGCGCTCG; 5' Dye/3' Quencher: FAM/ZEN/IBFQ; Primer to probe ratio: 3.6 |
| PX1 PCR Plate Sealer | Bio-Rad | 1814000 | |
| QuantaSoft Software | Bio-Rad | ||
| Quick Extract DNA Extraction Solution | Lucigen | QE0905T | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | ||
| Recombinant human Flt3-ligand | Peprotech | 300-19 | |
| Recombinant human IL-6 | Peprotech | 200-06 | |
| Recombinant Human SCF | Peprotech | 300-07 | |
| Recombinant Human TPO | Peprotech | 300-18 | |
| RPMI 1640 | Sigma-Aldrich | R8758-6X500ML | |
| SnapGene | Dotmatics | Molecular cloning software https://www.snapgene.com *Alternatively also Benchling (https://www.benchling.com) and Geneious (https://www.geneious.com) can be used. | |
| Soc outgrowth medium | NEB | B9020S | |
| Sodium-butyrate | Sigma-Aldrich | B5887-1G | |
| Stem Regenin 1 (SR1) | Biogems | 1224999 | |
| StemSpan SFEM II | STEMCELL Technologies | 9655 | |
| TAE Buffer (Tris-acetate-EDTA) 50X | Thermo Scientific | B49 | |
| TIDE | http://shinyapps.datacurators.nl/tide/ | ||
| Trypan blue 0.4% | Sigma-Aldrich | T8154-100ML | |
| TrypLE (with phenol red), 500 ml | Thermo Scientific | 16605-028 | |
| UltraPure 0.5: EDTA, pH 8.0, 100 ml | Thermo Scientific | 15575-038 | |
| UM171 | STEMCELL Technologies | 72914 | |
| Vector Builder codon optimization tool | Vector Builder | https://en.vectorbuilder.com/tool/codon-optimization.html |
참고문헌
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing transthyretin amyloidosis. New England Journal of Medicine. 385 (6), 493-502 (2021).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. New England Journal of Medicine. 384 (3), 252-260 (2021).
- Stadtmauer, E. A., et al. CRISPR-engineered T cells in patients with refractory cancer. Science. 367 (6481), (2020).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Veldwijk, M. R., et al. Pseudotyped recombinant adeno-associated viral vectors mediate efficient gene transfer into primary human CD34+ peripheral blood progenitor cells. Cytotherapy. 12 (1), 107-112 (2010).
- Song, L., et al. High-efficiency transduction of primary human hematopoietic stem cells and erythroid lineage-restricted expression by optimized AAV6 serotype vectors in vitro and in a murine xenograft model in vivo. PLoS One. 8 (3), 58757 (2013).
- Dever, D. P., et al. CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells. Nature. 539 (7629), 384-389 (2016).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Jan, M., et al. Clonal evolution of preleukemic hematopoietic stem cells precedes human acute myeloid leukemia. Science Translational Medicine. 4 (149), (2012).
- Corces-Zimmerman, M. R., Hong, W. J., Weissman, I. L., Medeiros, B. C., Majeti, R. Preleukemic mutations in human acute myeloid leukemia affect epigenetic regulators and persist in remission. Proceedings of the National Academy of Sciences of the United States of America. 111 (7), 2548-2553 (2014).
- Jaiswal, S., et al. Clonal hematopoiesis and risk of atherosclerotic cardiovascular disease. New England Journal of Medicine. 377 (2), 111-121 (2017).
- Genovese, G., et al. Clonal hematopoiesis and blood-cancer risk inferred from blood DNA sequence. New England Journal of Medicine. 371 (26), 2477-2487 (2014).
- Papaemmanuil, E., et al. Genomic classification and prognosis in acute myeloid leukemia. New England Journal of Medicine. 374 (23), 2209-2221 (2016).
- Cancer Genone Atlas Research Network. Genomic and epigenomic landscapes of adult de novo acute myeloid leukemia. New England Journal of Medicine. 368 (22), 2059-2074 (2013).
- Ball, M., List, A. F., Padron, E. When clinical heterogeneity exceeds genetic heterogeneity: thinking outside the genomic box in chronic myelomonocytic leukemia. Blood. 128 (20), 2381-2387 (2016).
- Varmus, H. E. The molecular genetics of cellular oncogenes. Annual Review of Genetics. 18, 553-612 (2003).
- Cox, D. B. T., Platt, R. J., Zhang, F. Therapeutic genome editing: Prospects and challenges. Nature Medicine. 21 (2), 121-131 (2015).
- Tothova, Z., et al. Multiplex CRISPR/Cas9-based genome editing in human hematopoietic stem cells models clonal hematopoiesis and myeloid neoplasia. Cell Stem Cell. 21 (4), 547-555 (2017).
- Mandal, P. K., et al. Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell. 15 (5), 643-652 (2014).
- Bak, R. O., Dever, D. P., Porteus, M. H. CRISPR/Cas9 genome editing in human hematopoietic stem cells. Nature Protocols. 13 (2), 358-376 (2018).
- Foßelteder, J., et al. Human gene-engineered calreticulin mutant stem cells recapitulate MPN hallmarks and identify targetable vulnerabilities. Leukemia. , (2023).
- Nangalia, J., et al. Somatic CALR mutations in myeloproliferative neoplasms with nonmutated JAK2. New England Journal of Medicine. 369 (25), 2391-2405 (2013).
- Merlinsky, T. R., Levine, R. L., Pronier, E. Unfolding the role of calreticulin in myeloproliferative neoplasm pathogenesis. Clinical Cancer Research. 25 (10), 2956-2962 (2019).
- Belčič Mikič, T., Pajič, T., Zver, S., Sever, M. The contemporary approach to CALR-positive myeloproliferative neoplasms. International Journal of Molecular Sciences. 22 (7), 3371 (2021).
- How, J., Hobbs, G. S., Mullally, A. Mutant calreticulin in myeloproliferative neoplasms. Blood. 134 (25), 2242-2248 (2019).
- Grieger, J. C., Choi, V. W., Samulski, R. J. Production and characterization of adeno-associated viral vectors. Nature Protocols. 1 (3), 1412-1428 (2006).
- Zolotukhin, S., et al. Recombinant adeno-associated virus purification using novel methods improves infectious titer and yield. Gene Therapy. 6 (6), 973-985 (1999).
- Aurnhammer, C., et al. Universal real-time PCR for the detection and quantification of adeno-associated virus serotype 2-derived inverted terminal repeat sequences. Human Gene Therapy Methods. 23 (1), 18-28 (2011).
- Klampfl, T., et al. Somatic mutations of calreticulin in myeloproliferative neoplasms. New England Journal of Medicine. 369 (25), 2379-2390 (2013).
- Bulcha, J. T., Wang, Y., Ma, H., Tai, P. W. L., Gao, G. Viral vector platforms within the gene therapy landscape. Signal Transduction and Targeted Therapy. 6 (1), 1-24 (2021).
- Montini, E., et al. The genotoxic potential of retroviral vectors is strongly modulated by vector design and integration site selection in a mouse model of HSC gene therapy. The Journal of Clinical Investigation. 119 (4), 964-975 (2009).
- Vaidyanathan, S., et al. Targeted replacement of full-length CFTR in human airway stem cells by CRISPR-Cas9 for pan-mutation correction in the endogenous locus. Molecular Therapy. 30 (1), 223-237 (2022).
- Cromer, M. K., et al. Gene replacement of α-globin with β-globin restores hemoglobin balance in β-thalassemia-derived hematopoietic stem and progenitor cells. Nature Medicine. 27 (4), 677-687 (2021).
- Wiebking, V., et al. Genome editing of donor-derived T-cells to generate allogenic chimeric antigen receptor-modified T cells: Optimizing αβ T cell-depleted haploidentical hematopoietic stem cell transplantation. Haematologica. 106 (3), 847-858 (2021).
- Wilkinson, A. C., et al. Cas9-AAV6 gene correction of beta-globin in autologous HSCs improves sickle cell disease erythropoiesis in mice. Nature Communications. 12 (1), 1-9 (2021).
- Dever, D. P., et al. CRISPR/Cas9 genome engineering in engraftable human brain-derived neural stem cells. iScience. 15, 524-535 (2019).
- Laustsen, A., et al. Interferon priming is essential for human CD34+ cell-derived plasmacytoid dendritic cell maturation and function. Nature Communications. 9 (1), 1-14 (2018).
- Bak, R. O., et al. Multiplexed genetic engineering of human hematopoietic stem and progenitor cells using CRISPR/Cas9 and AAV6. eLife. 6, 27873 (2017).
- Nakauchi, Y., et al. The cell type-specific 5hmC landscape and dynamics of healthy human hematopoiesis and TET2-mutant preleukemia. Blood Cancer Discovery. 3 (4), 346-367 (2022).
- Vaidyanathan, S., et al. selection-free gene repair in airway stem cells from cystic fibrosis patients rescues CFTR function in differentiated epithelia. Cell Stem Cell. 26 (2), 161-171 (2020).
- Tang, N., et al. TGF-β inhibition via CRISPR promotes the long-term efficacy of CAR T cells against solid tumors. JCI Insight. 5 (4), 133977 (2020).
- Georgiadis, C., et al. Long terminal repeat CRISPR-CAR-coupled "universal" T cells mediate potent anti-leukemic effects. Molecular Therapy. 26 (5), 1215-1227 (2018).
- Ren, J., et al. Multiplex genome editing to generate universal CAR T cells resistant to PD1 inhibition. Clinical Cancer Research. 23 (9), 2255-2266 (2017).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Doench, J. G., et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nature Biotechnology. 34 (2), 184-191 (2016).
- Bak, R. O., Porteus, M. H. CRISPR-mediated integration of large gene cassettes using AAV donor vectors. Cell Reports. 20 (3), 750-756 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유