É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Purificação do M. magneticum Strain AMB-1 proteína associada Magnetosome MamAΔ41
Neste Artigo
Resumo
Mama é uma proteína Magnetosome único associado que foi mostrado para ser envolvido na ativação magnetosome. Aqui apresentamos o protocolo de purificação da Mama exclusão mutante (MamAΔ41) de M. magneticum AMB-1.
Resumo
Bactérias magnetotáticas compreendem um grupo diverso de microorganismos aquáticos que são capazes de se orientar ao longo campos geomagnético. Este comportamento é acreditado para ajudar sua busca por ambientes adequados
Protocolo
1. Clonagem e expressão de Mama no Gene E. coli
O mutante do gene foi amplificado mamAΔ41 usando a reação em cadeia da polimerase (PCR) de DNA genômico de Magnetospirillum magneticum AMB-1, com primers: 5'-GCATTACGCATATGGACGACATCCGCCAGGTG-3 'e 5'-GCGCGGCAGCCATA-TGGCATACG-3'. Nos fragmentos de DNA amplificado, um site NcoI foi introduzido no códon de iniciação ATG eo códon de terminação foi substituído por um site SCOI. Os fragmentos foram digeridos com NcoI e SacI e clonados no respectivos sites de pET52b (+), dando origem a pET52bMamAΔ41-AMB1. Nesta construção, o gene mamAΔ41 foi fundida em-frame com o 10-Sua tag no C-terminal. O plasmídeo foi electroporated em E. coli estirpe BL21. E. coli estirpe BL21 abrigar pET52bMamAΔ41 foi cultivado em auto-indução (15) meio contendo ampicilina (50 mg / ml) 310 ° K por 3 horas. A temperatura de cultivo foi então deslocada de 310 ° a 300 ° K e mantido por um adicional de 48 h, a 300 ° K. As células foram colhidas por centrifugação a 5465g por 10 minutos a 277 ° K. 8 cultura de litros produzidos 60 gramas de pellet celular molhado.
2. Os cálculos de bioinformática
Cálculos de peso molecular (MW), de acordo com a seqüência de aminoácidos ea absorção previsto de 1mg/1ml de proteína em 280nm, utilizando o servidor ProtParam (http://www.expasy.ch/tools/protparam.html). Para 10His-Tag-MamAΔ41 o MW é 22529 Da (240 aminoácidos) e para MamAΔ41 (com 9 aminoácidos esquerda após sua remoção por tag-trombina) o Mw é 20596.5Da (187 aminoácidos). A absorção previsto de 1mg/1ml de proteína em 280nm para 10His-Tag-MamAΔ41 é 0,595 e para MamAΔ41 é 0,579. Além disso, a seqüência de aminoácidos não contém resíduos de cisteína. Portanto, agentes redutores não são necessários durante o processo de purificação.
3. Purificação da MamAΔ41
- Para a preparação de solução estoque, pesar e preparar (200 ml cada) nas seguintes concentrações: Imidazol (MW 228,2 g / mol) solução 4M, NaCl (Mw 58,44 g / mol) solução 5M, Tris-HCl (Mw 121,3 g / mol ) solução 1M, ajustar o pH = 8. Adicionar água destilada duas vezes (DDW) e misture usando um vortex até que a solução é clara. Soluções de filtro com filtro 0.22μm e manter em temperatura ambiente.
- Prepare e filtrar 6 buffers frescos a partir de soluções estoque (Tabela 1).
- Diluir e suspender as células em tampão Um volume em 1:2 ratio, gramas de células para buffer (ml).
- Adicionar 10μl para cada 10gr de bactérias de DNase I (1 mg / ml) a fim de quebrar fragmentos de DNA na amostra. Adicionar 1 ml para cada 20gr de bactérias de EDTA coquetel inibidor de protease livre. Incubar por 20 min no gelo.
- Interrompido células por dois ciclos de imprensa francesa a 25.000 psi. Ao contrário de sonicação, a imprensa francesa tem como objetivo em alto volume (10ml>) extrusão de células, forçando as células sob alta pressão através de um orifício fino. O pistão deve ser resfriado em gelo por 10 minutos antes do seu uso e de montagem devido ao acúmulo de temperatura.
- Transferência de células lisadas em tubos de ultracentrífuga 25ml e equilibrá-los com precisão (50mg) com tampão A até que a amostra atinge a linha de tubo s pescoço.
- Restos celulares separado por centrifugação a 270.000 g Ultra por 1 hora a 277 ° K.
- Prepare um caseiro gravidade Ni coluna NTA (2,5 cm de diâmetro), adicionando 4 ml de Ni NTA, que são preserva em etanol 20%, na coluna. Deixe o líquido cair completamente, em seguida, lavar com 40 ml de DDW e deixe o líquido cair completamente. Pré-equilibrada a resina na coluna com 40 ml de tampão A.
- Aplicar a fração solúvel de ultra-centrifugação de tubos sobre a gravidade coluna NTA Ni. Deixe o líquido cair, recolher flow-through (proteínas não ligada) e preservar a 277k.
- Coletar amostra de pellet dos tubos de ultracentrifugação usando uma pequena dica para acabar com o pellet. Misture a ponta em 50μl de 5% β-mercaptoetanol em SDS-PAGE 2X tampão de amostra.
- Lavar a coluna com 100 ml de tampão B. Deixe a queda de líquidos, coleta de fluxo e preservar a 277k.
- Prepare 15 tubos eppendorf marcados (1,5 ml).
- Eluição - Válvula coluna próxima e então carregar 1ml de tampão C na coluna; incubar por 3 min seguido de abertura da válvula ea vazão através da recolha. Repita este passo três vezes.
- Eluir resto de suspensão de 1 ml incrementos tampão C e fechar a válvula.
- OD medida em 280nm das frações e avaliar localização do pico de proteína, se o OD ainda é> 0,3, as amostras adicionais são necessários.
- SDS-PAGE análise das frações coletadas. Tal análise deve ser realizada também com os exemplos a seguir, as proteínas unbound flow-through, lavagem de fluxo e ultra-centrifugação da amostra de sedimento. Mix 10μl de cada amostra com 10μl de 5% β-mercaptoetanol em SDS-PAGE 2X tampão de amostra. Coloque as amostras em 15% SDS-PAGE e correr por 45 min em 180 Volts.
- Manchar o gel em 15 ml de solução InstantBlue, agitar durante 1 hora. Certifique-se que a caixa de plástico é coberto e protegido da luz.
- Avaliar gel, se a expressão da proteína é elevado e específico de acordo com as faixas indicativas, mesclagem selecionado frações eluídas.
- A fim de remover 10-Seu-tag, adicionar trombina bovina (10 unidades / mL), 10μl de trombina por 1 mg de proteína, de acordo com a recomendação do fabricante para a amostra combinada.
- A diálise é utilizada para a remoção de excesso de Imidazol e para diminuir as concentrações de NaCl para os níveis necessários durante a ligação de íons de câmbio. Para diálise, corte comprimento desejado, de 7 de peso molecular de corte kD (MWCO) tubos de diálise. Lavar com DDW e faça um nó em uma extremidade (ou selo com clipes). Transferência de solução de proteína fundiram no saco, deixando algum espaço na parte superior e grampo para cima.
- Tubo de submergir a diálise em 4 tampão de diálise Litros refrigerados (buffer D). Agitar a solução lentamente durante a noite com 277 ° K.
- Tire o tubo de diálise do copo, faça um pequeno corte e transferência da proteína para 50ml tubo.
- Cromatografia de troca iônica: montar pré-embalados MonoQ 4.6/100 coluna PE em Cromatografia Rápida Desempenho líquido (FPLC). Lavar a coluna com 5 volumes coluna (CV) de água filtrada DDW. A coluna equilibrada com 5 CV de tampão D.
- Lavar o loop de injeção com DDW (dois volumes loop) e repita com tampão D. Load a proteína no circuito de injeção.
- Injectar a amostra de proteínas (vazão de 1 ml / min) e iniciar a coleta de frações de SDS-PAGE detecção de proteínas sem limites.
- Se o volume inicial de proteína (passo 3.23) é maior do ciclo de injeção, repita os passos de 3,25-3,26, sem lavar o loop, até que toda a amostra é carregada na coluna.
- Lavar a coluna com 3 CV de tampão C, a fim de eliminar as proteínas sem limites.
- Eluir a proteína com um gradiente linear de 60 min 40 2000mm NaCl entre buffers D & E (E quando buffer de 100% é o fim do gradiente), o fluxo de taxa de 1 ml / min (embora maiores taxas de fluxo pode ser usado no custo de volumes maiores, mas menos concentrado picos, proteína). Coletar picos de proteínas como eles aparecem no cromatograma 280nm.
- Coluna de pré-embalados de acordo limpa para a fabricação de recomendação.
- SDS-PAGE análise das frações coletadas. Tal análise deve ser realizada com as seguintes amostras, as proteínas ilimitada de fluxo e proteína picos de fluxo contínuo. Executar o SDS-PAGE e mancha como falar em passos de 3,17-3,18.
- Avaliar gel para a existência de bandas de proteínas na Mw previsto de acordo com a proteína marcador pré-manchado. Mesclar frações de eluição selecionadas que contêm a proteína em questão.
- Diálise das amostras fundidas, a fim de reduzir as concentrações de NaCl necessária para cromatografia por exclusão de tamanho. Realizar a diálise, como mencionado nos passos 3,21-3,23 contra tampão F.
- Concentrar a amostra de proteína usando um Vivaspin-15 10000 MWCO a 8 mg / ml em uma centrífuga de mesa, 4000 rpm. Validar concentração desejo medindo OD em 280nm em cubetas de quartzo. Se a proteína é muito concentrada diluir com tampão F e medir novamente.
- Montar tamanho exclusão coluna pré-embalados, HiLoad 26/60 Superdex 200, em FPLC; lavar a coluna com 3 CV de água filtrada DDW. A coluna equilibrada com 3 CV de tampão F.
- Lavar ciclo de injeção com 5ml DDW (dois volumes loop) seguiu pelo mesmo volume com tampão F. Não coloque mais de 4 ml da amostra de proteína no ciclo de injeção.
- Injectar a amostra de proteínas (vazão de 3,5 ml / min) e iniciar a coleta de frações de proteínas de pico como eles aparecem se o cromatograma 280nm. Deixe o fluxo de buffer até atingir o volume da coluna.
- Se o volume inicial de proteína (passo 3.23) é maior do ciclo de injeção, repita os passos de 3,25-3,26, sem lavar o loop, até que toda a amostra é carregada na coluna.
- Coluna de pré-embalados limpo de acordo com a recomendação de fabricação.
- SDS-PAGE análise das frações coletadas. Tal análise deve ser realizada com picos nas amostras de proteínas. Executar o SDS-PAGE e mancha como falar em passos de 3,17-3,18.
- Avaliar gel: se picos de proteínas são específicas, na Mw correta previsto de acordo com a proteína marcador pré-coradas e apenas uma banda é visível mesclagem selecionado frações eluídas da proteína em questão.
- Concentre-se amostra de proteína usando um Vivaspin-15 10000 MWCO a> 20 mg / ml em uma centrífuga de mesa, 4000 rpm. Validar concentração desejo medindo OD em 280nm. O MamAΔ41 purificada foi então concentrada a 26,5 mg / ml para a cristalização.
- Confirme a pureza da amostra e identificação de proteínas usando Matrix-assisted laser de dessorção / ionização (MALDI-TOF).
- Se a proteína é purificada de acordo com o espectrômetro de massa e SDS-PAGE, divida o MamAΔ41 concentrado para tubos eppendorf pré-marcada, 25-50μl de proteína em cada um.
- Flash congelados em nitrogênio líquido e armazenar a 193 ° K.
4. Resultados representante
Quando este protocolo é feito corretamente deve-se altamente purificado, tamanho amostras de proteína homogênea e concentrada. Estas amostras de proteínas são, então, pronto para ensaios de cristalização, bem como estudos bioquímicos, tais como cinética enzimática, afinidade de ligação e muito mais. Aqui descritos são resultados representativos dos principais passos no protocolo de purificação. SDS-PAGE análise do perfil de eluição de Ni-NTA coluna de afinidade deve revelar muito mais proteína expressa na fração solúvel em MW apropriado de ~ 22kDa (Figura 1). Esta análise SDS-PAGE deve também revelar mínimo, se qualquer proteína nas proteínas ilimitada de fluxo, lavagem de fluxo e ultra-centrifugação da amostra de sedimento. Se grandes bandas da proteína apropriada aparecem nestas amostras deve-se considerar a preparação de buffer errado, problemas no rompimento celular ou problemas na cultura de auto-indução condições e crescimento.
Cromatografia de troca iônica é pré-formados a fim de separar entre o nosso proteína desejável outras proteínas E.coli que estavam vinculados à resina de níquel (devido a interações eletrostáticas ou histidina / negativo aminoácidos loops ricos em proteínas) e proteína His-Tagged uncut desejável. Esta coluna separa proteínas de acordo com a afinidade de ligação à resina com carga positiva no aumento da concentração de NaCl. Proteína altamente carregadas negativamente vai eluir em maiores concentrações de NaCl appose para moderar os com carga negativa. As vantagens desta coluna são de alta vazão e capacidade de ligação. O cromatograma de troca iônica (Figura 2) revela uma boa separação entre 3 populações proteínas no aumento da concentração de NaCl. SDS-PAGE análise é necessária a fim de determinar MW de cada população, isolando a uma desejável e avaliar se as etapas de purificação que são necessários mais. A primeira população é MamAΔ41 (~ 20 kDa), enquanto a segunda população é MamAΔ41 + Tag His (~ 22kDa) que não foi clivada pela trombina bovina e à terceira população é indetectável no SDS-PAGE, devido à baixa concentração. Se os picos de proteínas não são claramente separados deve-se considerar a preparação de buffer errado ou alterar a inclinação do gradiente de NaCl.
Cromatografia por exclusão de tamanho é pré-formados a fim de separar entre o nosso proteína desejável a outras proteínas de células E. coli que foram ligados à resina Ni-NTA e não foram separados durante a cromatografia de troca iônica. Esta coluna separa proteínas de acordo com seu tamanho. Grandes proteínas vai eluir em menores volumes de eluição oposição a pequenos que serão eluídos em volumes maiores. O cromatograma coluna (Figura 3) revela uma boa separação da proteína desejável como uma população principal. A população parece eluir no tamanho apropriado com um monômero MW de ~ 20 kDa. A presença de ~ 80 kDa banda (E.coli proteína típica que se liga Ni-NTA) é detectado em SDS-PAGE antes de carregá-coluna e desaparece durante essa rodada. Desde que a concentração desta proteína kDa ~ 80 é baixo, para começar, a sua população não aparece no cromatograma, devido à sua diluição e SDS-PAGE análise é necessária para determinar a eficiência de purificação. Recomendamos o carregamento de uma amostra diluída e concentrada do pico principal do SDS-PAGE para um certamente poderia determinar a presença de uma proteína pura no MW apropriado. Por esta fase, a proteína é purificada e sua homogeneidade deve ser avaliada por MALDI-TOF. Esta análise revelou uma proteína de 20.347 MW e outra em Da Mw de 10176 Da (Figura 4). O ~ proteína de 10 kDa é o ~ proteína kDa 20 dobrou cobrado. O MW previsto de MamAΔ41 com os 9 aminoácidos esquerda após sua Tag-remoção foi 20596,5 Da. Comparando-o com o obtido MALDI-TOF MW descobrimos que eles são 248 Da distante. Essa diferença pode ser resultado de erros MALDI-TOF medição e / ou devido à degradação comum do metionina primeiro eo segundo glicina. Para concluir, a proteína é altamente purificada e pode ser utilizado nos experimentos posteriores.
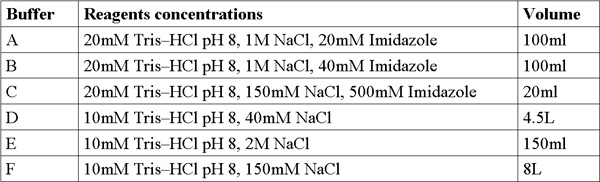
Tabela 1. Formulações Buffer.
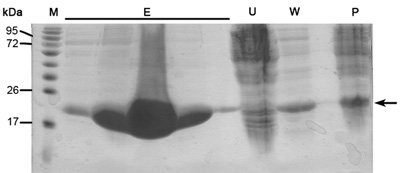
Figura 1. Representante análise SDS-PAGE de Ni-NTA purificação coluna Da direita para a esquerda;. P-ultra-centrifugação pellet (3.11 passo protocol), W-lavagem (3,12 etapa protocol), U-unbounded proteínas (3.10 passo protocol), E- cinco pistas de eluição samples (3,14 etapa protocol), M - marcador de proteína (números indicam MW). Seta indica MamAΔ41.
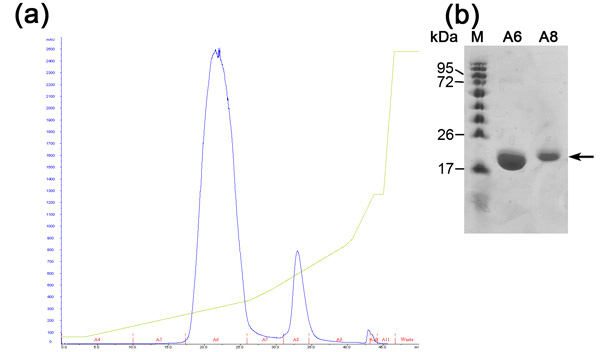
Figura 2. Análise de cromatografia de troca iônica etapa (a) de troca iónica cromatograma (coluna MonoQ); Blue - 280nm absorção, representa a concentração de proteína, Green-concentração de NaCl, Vermelho - indicação das frações coletadas. (B) Representante análise SDS-PAGE da etapa de purificação de troca iônica. Da esquerda para a direita; M - marcador de proteína (números indicam Mw), A6 - fração primeiro pico, segundo A8-fração de pico.

Figura 3. Tamanho análise de cromatografia de exclusão (a) a exclusão Tamanho (Superdex coluna 200) cromatograma; Blue - 280nm absorção, representa a concentração de proteína, a indicação Red das frações coletadas. (B) Representante análise SDS-PAGE do tamanho do passo de purificação de exclusão. Da esquerda para a direita; proteína M-marcador (números indicam MW), Prei pré-injeção de amostra, A4-6 fração combinada coletados a partir do primeiro pico, A4-6D-diluído fracção recolhida, a partir do pico.
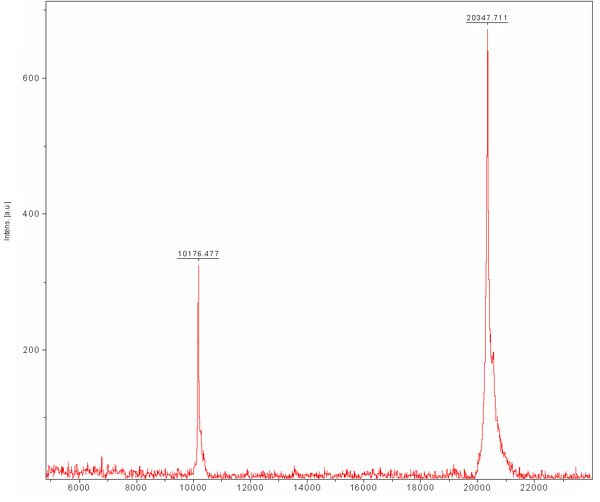
Figura 4. Matrix-assisted laser de dessorção / ionização (MALDI-TOF) espectro de massa de purificada MamAΔ41. A matriz é o ácido Sinapic (SA). MamAΔ41 mostrado em 20347 Da, também mostrado é a espécie dobrou acusado de MamAΔ41 em 10176 Da.
Discussão
Purificação de proteínas é o passo principal em todas as proteínas estruturais ou estudos bioquímicos. Uma vez que cada proteína é o único com seu próprio comportamento, é preciso definir suas propriedades e modificar sua purificação de acordo. Proteína-alvo deve ser analisado como um primeiro passo para a purificação utilizando ferramentas de bioinformática. Eles são usados para calcular o objectivo ponto iso-elétrico, avaliar a sua necessidade de redução / oxidação ambiente e sua necessida...
Agradecimentos
Reconhecemos Dr. Amir Aharoni por seu apoio e Davidov Geula, Noam Grimberg e Chen Guttman para os seus conselhos e comentários.
Materiais
| Name | Company | Catalog Number | Comments | |
| French Press | Equipment | Thermo Fisher Scientific, Inc. | FA-078A | |
| Pressure cell | Equipment | Thermo Fisher Scientific, Inc. | FA-032 | |
| Ultra-centrifuge | Equipment | Sorvall, Thermo Scientific | Discovery 90SE | |
| Rottor | Equipment | Beckman Coulter Inc. | Ti60 | |
| Ultra-centrifuge tubes; PC-Bottle+Cap Assay 26.3ml | Equipment | Beckman Coulter Inc. | BC-355618 | |
| 2.5cm diameter, Glass Econo-Column Chromatography Columns | Equipment | Bio-Rad | 737-2521 | |
| Ni-NTA His Bind resin | Equipment | Novagen, EMD Millipore | M0063428 | |
| Spectrophotometer | Equipment | Amersham | Ultraspec 2100 pro | |
| Quartz cuvette | Equipment | Hellma | 104-QS | |
| Fast Performance Liquid Chromatography- AKTA purifier 10 | Equipment | GE Healthcare | 28-4062-64 | |
| Ion exchange column – MonoQ 4.6/100 PE | Equipment | GE Healthcare | 10025543 | |
| Size exclusion pre-packed column-HiLoad 26/60 Superdex 200 | Equipment | GE Healthcare | 17-1071-01 | |
| Centricon - Vivaspin15 – 10,000 MWCO | Equipment | Sartorius AG | VS1501 | |
| Table centrifuge | Equipment | Thermo Fisher Scientific, Inc. | IEC CL30R | |
| MALDI-TOF | Equipment | Bruker Corporation | Reflex IV | |
| Tris-HCl (hydrotymethyl) aminomethane | Reagent | BioLab | 20092391 | |
| Sodium Chloride | Reagent | FRUTROM | 235553470 | |
| Imidazole | Reagent | Alfa Aesar | 288-32-4 | |
| EDTA free protease inhibitors cocktail | Reagent | Sigma-Aldrich | P-8849 | |
| Dnase I (Deoxyribonuclease I) | Reagent | Sigma-Aldrich | DN-25 | |
| Bovine Thrombin | Reagent | Fisher Scientific | BP25432 | |
| Glycine | Reagent | BioLab | 07132391 | |
| Soudim Dodecyl Sulfate (SDS) | Reagent | BioLab | 19822391 | |
| Beta-mercapt–thanol | Reagent | Sigma-Aldrich | M-3148 | |
| InstantBlue | Reagent | Expedeon | 1SB01L | |
| PageRuler Prestained Protein Ladder | Reagent | Fermentas | SM0671 |
Referências
- Faivre, D., Schuler, D. Magnetotactic Bacteria and Magnetosomes. Chem Rev. 108, 4875-4898 (2008).
- D'Andrea, L. D., Regan, L. TPR proteins: the versatile helix. Trends Biochem Sci. 28, 655-662 (2003).
- Young, J. C., Barral, J. M., Hartl, U. l. r. i. c. h., F, . More than folding: localized functions of cytosolic chaperones. Trends Biochem Sci. 28, 541-547 (2003).
- Brocard, C., Hartig, A. Peroxisome targeting signal 1: is it really a simple tripeptide?. Biochim Biophys Acta. 1763, 1565-1573 (2006).
- Fransen, M., Amery, L., Hartig, A., Brees, C., Rabijns, A., Mannaerts, G. P., Van Veldhoven, P. P. Comparison of the PTS1- and Rab8b-binding properties of Pex5p and Pex5Rp/TRIP8b. Biochim Biophys Acta. 1783, 864-873 (2008).
- Baker, M. J., Frazier, A. E., Gulbis, J. M., Ryan, M. T. Mitochondrial protein-import machinery: correlating structure with function. Trends Cell Biol. 17, 456-464 (2007).
- Mirus, O., Bionda, T., von Haeseler, A., Schleiff, E. Evolutionarily evolved discriminators in the 3-TPR domain of the Toc64 family involved in protein translocation at the outer membrane of chloroplasts and mitochondria. J Mol Model. 15, 971-982 (2009).
- Gatsos, X., Perry, A. J., Anwari, K., Dolezal, P., Wolynec, P. P., Likic, V. A., Purcell, A. W., Buchanan, S. K., Lithgow, T. Protein secretion and outer membrane assembly in Alphaproteobacteria. FEMS Microbiol Rev. 32, 995-1009 (2008).
- Tiwari, D., Singh, R. K., Goswami, K., Verma, S. K., Prakash, B., Nandicoori, V. K. Key residues in Mycobacterium tuberculosis protein kinase G play a role in regulating kinase activity and survival in the host. J Biol Chem. 284, 27467-27479 (2009).
- Edqvist, P. J., Broms, J. E., Betts, H. J., Forsberg, A., Pallen, M. J., Francis, M. S. Tetratricopeptide repeats in the type III secretion chaperone, LcrH: their role in substrate binding and secretion. Mol Microbiol. 59, 31-44 (2006).
- Grunberg, K., Muller, E. C., Otto, A., Reszka, R., Linder, D., Kube, M., Reinhardt, R., Schuler, D. Biochemical and proteomic analysis of the magnetosome membrane in Magnetospirillum gryphiswaldense. Appl Environ Microbiol. 70, 1040-1050 (2004).
- Komeili, A., Vali, H., Beveridge, T. J., Newman, D. K. Magnetosome vesicles are present before magnetite formation, and MamA is required for their activation. Proc Natl Acad Sci USA. 101, 3839-3843 (2004).
- Okuda, Y., Fukumori, Y. Expression and characterization of a magnetosome-associated protein, TPR-containing MAM22, in Escherichia coli. FEBS Lett. 491, 169-173 (2001).
- Taoka, A., Asada, R., Sasaki, H., Anzawa, K., Wu, L. F., Fukumori, Y. Spatial localizations of Mam22 and Mam12 in the magnetosomes of Magnetospirillum magnetotacticum. J Bacteriol. 188, 3805-3812 (2006).
- Studier, F. W. Protein production by auto-induction in high density shaking cultures. Protein Expression and Purification. 41, 207-234 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados