Method Article
Usando um contador de células automatizadas para simplificar Estudos da Expressão Gênica: Knockdown siRNA de Expressão IL-4 Gene Dependente em células NAMALWA
Resumo
Este procedimento descreve um fluxo de trabalho rápido e fácil de introduzir em siRNA difícil de transfecção de linhas celulares e siga expressão gênica por PCR em tempo real. Uso de um contador de células automatizado, multi-prato bem eletroporação, e da estação de eletroforese automatizada fornecer resultados rápidos e confiáveis, sem a necessidade de manipulação robótica caro.
Resumo
O uso de siRNA knockdown mediado gene continua a ser uma importante ferramenta em estudos de expressão gênica. estudos estão sendo conduzidos siRNA não só para estudar os efeitos da downregulating genes individuais, mas também para interrogar vias de sinalização e de outras redes de interação complexas. Estas análises via requerem o uso de modelos celulares relevantes e métodos que causam menos perturbação da fisiologia celular. Eletroporação é cada vez mais sendo usado como uma forma eficaz de introduzir siRNA e outros ácidos nucléicos em difícil transfecção de linhas celulares e pilhas, sem alterar a via de sinalização sob investigação. Existem várias etapas fundamentais para uma experiência bem sucedida siRNA, e existem maneiras de simplificar o trabalho, melhorando a qualidade de dados em vários estágios experimentais. Para ajudar você a começar com o seu projeto de siRNA knockdown mediado gene, vamos demonstrar como realizar um estudo completo do percurso recolha e contagem de células antes da eletroporação através de pós-real-time PCR transfecção análise de expressão gênica. O estudo a seguir investiga o papel do ativador transcricional STAT6 em IL-4 expressão gênica dependente de CCL17 em uma linha de células de linfoma de Burkitt (NAMALWA). As técnicas demonstradas são úteis para uma ampla gama de siRNA baseado em experiências com ambos os aderentes e células em suspensão. Também vamos mostrar como otimizar contagem de células com o contador de células automatizado TC10, como electroporate várias amostras simultaneamente, usando o sistema de eletroporação MXcell, e como a avaliação simultânea da qualidade e quantidade de RNA com o sistema de eletroforese Experion automatizado.
Protocolo
1. Preparação e contagem das células NAMALWA
- Remover as células da garrafa de cultura e centrifugar as células a pelota. Remover as células sobrenadante e ressuspender em um volume conhecido de PBS. As células utilizadas neste protocolo são células em suspensão. Se você estiver usando células aderentes terá de trypsinize antes de coletar.
- Para determinar o número de células vivas, mancha uma alíquota da suspensão de células, misturando-o 01:01 com uma solução 0,4% de azul de tripano e Pipete 10 da mistura sobre uma lâmina de contagem, e depois inserir o slide na célula automatizada TC10 contador (Bio-Rad Laboratories, Inc).
- O contador de células TC10 vai autofocus para assegurar a contagem mais precisa, e depois prosseguir com a contagem. Ela determina automaticamente a densidade de células vivas e células totais da amostra.
- Selecione ver a imagem para mostrar as células na tela principal. Em seguida, selecione a calculadora de diluição para ter o contador de células TC10 calcular automaticamente a quantidade de mistura de células que serão necessários.
- Este experimento está usando um total de três siRNAs: uma siRNA controle e dois siRNAs diferentes visando o gene STAT6, além de controles untransfected. Isto requer 3,6 ml na concentração final de 5 X 10 6 células por ml.
- Digite a concentração desejada e os valores de volume final na calculadora de diluição, e calcula o volume de suspensão de células necessárias com base na contagem de células vivas para essa amostra. Alternativamente, realizar o cálculo manualmente. Em seguida, transferir o volume necessário de mistura de células para esses experimentos em um tubo novo.
- Girar a misturas de células para agregar as células e remover todos os PBS. Ressuspender em 3,6 ml de tampão de eletroporação Gene Pulser (Bio-Rad Laboratories). Transferência de 800 mL alíquotas em tubos contendo os siRNAs apropriado e misture delicadamente. As células estão agora prontos para eletroporação.
2. Electroporating as células em placas de 96 poços
- A placa de 96 poços eletroporação permite que o replica para todos os quatro tratamentos de ser electroporated juntos.
- Transferência de 150 mL da suspensão de células nos poços apropriados de uma placa de eletroporação de 96 poços.
- Inserir a placa na câmara de eletroporação MXcell placa e feche a tampa.
- Electroporate usando configurações otimizadas para a linha de celular e de buffer a ser utilizado. Estas células são electroporated usando uma onda de decaimento exponencial com as definições do aparelho de 250 V, 350 mF, e 1000 Ω de resistência.
- Depois de eletroporação é completa, pipeta para cima e para baixo suavemente em cada bem para misturar e depois transferir as células a uma placa de cultura de tecidos contendo pré-aquecido mídia. NAMALWA células são cultivadas em RPMI (Life Technologies), suplementado com FCS 7,5% e 1% piruvato.
- Amostras de controle que não foram electroporated são tomadas a partir da suspensão de células remanescentes e adicionado à cultura de pratos pré-aquecido meio de cultura de forma idêntica para as células electroporated.
- Células incubar durante a 37 ° C com 5% de CO 2.
- Após 24 horas, um conjunto de células foi induzida com concentração de 10 ng / ml final IL-4 recombinante (R & D Systems). O segundo conjunto de células controle só recebem mídia adicional. Todas as células são incubadas uma hr mais 24 a 37 ° C e 5% CO 2.
3. Extrato e verificar RNA
- Após um total de 48 horas de crescimento, contar as células em cada poço, e transferir uma alíquota de cada bem a um tubo de microcentrífuga para a extração de RNA, e congelar uma alíquota de segundo para análise de proteínas western blotting ou outros.
- Centrifugar amostras para agregar as células, e, em seguida, remover e descartar o sobrenadante de cada tubo.
- Prosseguir com a extração de RNA. Normalmente usamos o total Aurum RNA kit (Bio-Rad) de acordo com o protocolo incluídos.
- Ao monitorar siRNA knockdown mediado gene é especialmente importante para verificar a qualidade RNA RNA degradado, porque vai dar resultados enganosos. A relação 260/280 não indica a integridade do RNA, portanto, é importante para executar as amostras em um gel ou dispositivo microfluídicos para confirmar RNA não são degradadas.
- Para avaliar a qualidade e quantidade em uma única etapa, essas amostras serão analisadas com o sistema de eletroforese Experion automatizado (Bio-Rad).
- Primeiro, as amostras de calor. Então, primeiro, carga e vórtice do Experion chip de RNA. Inserir o chip na estação eletroforese Experion e começar a correr.
- Após a corrida é completa, o software irá exibir Experion resultados automaticamente na forma de um electroferograma tabela de resultados, (que exibe RNA concentração, a relação ribossomal, RNA eo número indicador de qualidade), e gel virtual (que é semelhante a um RNA tradicional gel). De alta qualidade total de amostras de RNA terá tipicamente um RQI de 8-10.
4. PCR em tempo real
- Após verificar a qualidade RNA, prossiga com as reações cDNA. Este experimento usou o iScript um passo-RT-PCR mix (Bio-Rad) para combinar a síntese de cDNA e PCR em tempo real em uma reação.
- Faça um mix master contendo todos os componentes de reação que não seja os modelos RNA para cada conjunto de primers, e distribuir esses na placa de PCR.
- Após diluição da RNA a uma concentração uniforme, adicione a amostras de RNA nas cavidades apropriadas. Cada reação é configurado em triplicado.
- Selar a placa, e colocá-lo no CFX384 sistema de PCR em tempo real (Bio-Rad). Selecione o protocolo apropriado e começar a correr.
- Uma vez que a corrida é completa, a expressão do gene é determinada pela comparação de expressão gênica do gene de interesse normalizado para um gene nas células de referência electroporated com siRNA experimental para a normalização das células electroporated mesmo com um controle siRNA.
5. Análise de knockdown siRNA
- A expressão de CCL17 foi monitorado usando quantitativa PCR em tempo real para investigar o papel do ativador transcricional STAT6 em IL-4 indução dependente de CCL17. Para este experimento, GAPDH foi utilizado como gene de referência. Genes de referência alternativo ou genes múltiplos de referência pode ser usado da mesma maneira.
- O software Gerenciador CFX automaticamente normalizado CCL17 expressão a expressão do gene de referência a partir da mesma amostra e automaticamente comparados os valores resultantes normalizada a tratamentos de controle simplesmente selecionando GAPDH como "referência" e ao tratamento controle (siRNA controle sem indução de IL-4) como "controle" na janela de configurações experiência do gerente de software CFX.
- Sem indução de IL-4, a transcrição de CCL17 permaneceu em baixa, níveis constitutivos comparável ao tratamento controle.
- Células induzidas com IL-4 e manter um caminho STAT6 normal (ie, siRNA controle ou tratamentos controle untransfected) mostraram 10/08 indução dobra de CCL17 expressão.
- Células induzidas com IL-4, mas com STAT6 expressão inibida pela introdução de qualquer siRNA STAT6 mostrou expressão CCL17 apenas cerca de 2 vezes maior do que as células uninduced confirmando o papel do STAT6 no IL-4 expressão induzida de CCL17.
6. Resultados representante
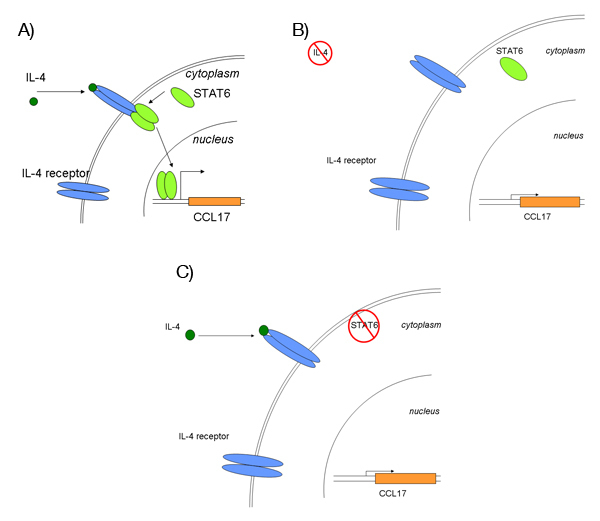
Figura 1. Visão geral da via IL-4 sinalização.
Um Binding) de IL-4 ao seu receptor leva a dimerização e ativação de STAT6. Ativado STAT6 transloca para o núcleo e ativa a transcrição de seus genes-alvo, como CC17.
B) Sem IL-4 indução, transcrição de CCL17 permanecerá em baixa, níveis constitutivos.
C) Depois de siRNA mediada pelo silenciamento do gene STAT6, o tratamento com IL4 deve levar a pouco ou nenhum aumento no CCL17 expressão.
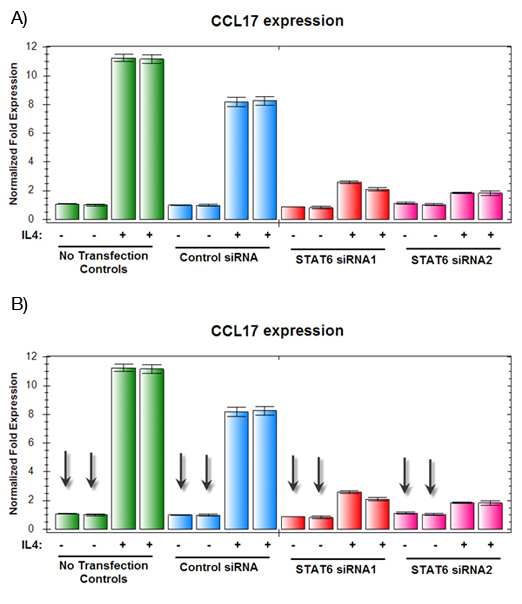
Figura 2. Indução de CCL17 por IL4 medida por diferentes condições quantitativas por PCR em tempo real.
A) As amostras cultivadas sem IL-4 teve um baixo nível de expressão constitutiva, independentemente da expressão do gene.
B) Como mostrado em verde e azul, as amostras de controle normalmente respondeu ao tratamento com IL-4, com uma indução 10/08 dobra de CCL17. No entanto, as células transfectadas com siRNAs segmentação STAT6, mostrado em vermelho e rosa, tinha diminuído CCL17 expressão em resposta a IL-4, apoiando a idéia de que STAT6 desempenha um papel na expressão de IL-4 induzida de CCL17.
Discussão
Este artigo demonstrou como electroporate siRNAs em uma difícil transfectar linhagem de células de suspensão e como seguir a expressão gênica nessas células. Ao fazer este procedimento é importante lembrar de manter as coisas o mais consistente possível em todas as amostras.
Divulgações
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados