Method Article
Dimorfismo Sexo músculo esquelético de Proteomics
Neste Artigo
Resumo
Um conjunto simples e direta de métodos para isolar e determinar a identidade das proteínas mais abundantes expressa no músculo esquelético. Cerca de 800 spots são discernidos em um gel de duas dimensões de 10 mg do músculo, o que permite a determinação de gênero específico expressão da proteína. Esses métodos produzem resultados equivalentes na maioria dos tecidos.
Resumo
Contração bruta no músculo esquelético é determinado principalmente por um número relativamente pequeno de proteínas contráteis, no entanto, este tecido é também extremamente adaptáveis a fatores ambientais, tais como uma hipertrofia por meio de exercícios de resistência e atrofia por desuso. É, assim, apresenta remodelação e adaptação ao stress (calor, isquemia, metais pesados, etc.) 2,3. Podem ocorrer danos ao músculo por uma força muscular exercendo ao mesmo tempo o alongamento, o chamado 4 contração excêntrica. As proteínas contráteis pode ser prejudicada em tais esforços e precisa ser reparado, degradadas e / ou ressintetizamos; estas funções não fazem parte das proteínas contráteis, mas de outras proteínas muito menos abundante na célula. Para determinar qual subconjunto de proteínas está envolvida na melhoria deste tipo de dano, um proteoma global deve ser estabelecida antes do exercício 5 e seguido, após o exercício para determinar o diferencialexpressão da proteína e, assim, destacar proteínas candidato nas adaptações ao dano e sua reparação. Além disso, a maioria dos estudos do músculo esquelético têm sido realizados sobre o macho da espécie e, portanto, pode não ser representativa do músculo feminino.
Neste artigo apresentamos um método para extrair proteínas reproduzível dos músculos masculinos e femininos, e separando-os por eletroforese bidimensional em gel seguido de alta resolução de imagem digital 6. Isto fornece um protocolo para spots (e proteínas posteriormente identificado) que mostram uma diferença estatisticamente significativa (p <0,05), duas vezes aumentar ou diminuir, aparecer ou desaparecer a partir do estado do controle. Estes são, então, excisadas, digeridos com tripsina e separados por alta pressão cromatografia líquida acoplada a um espectrômetro de massa (LC / MS) para identificação de proteínas (LC / MS / MS) 5. Esta metodologia (Figura 1) pode ser usado em muitos tecidos com pouca ou nenhuma modificação (fígado, cérebro, ouvirt etc.).
Protocolo
1. Preparação de amostras
- Use tecido fresco, congelado ou flash-RNAlater tratados de camundongos como matéria-prima. Blot fora qualquer excesso com um conservante Kimwipe antes de prosseguir.
- Remova todos os tendões e fáscia muscular e em todo o mince o tecido com um único lâminas de barbear afiada por 2 min em placas de vidro refrigerada em uma câmara fria (4 ° C) até que o tecido é bem picada ou em pó. Recolher a amostra para um tubo de microcentrífuga pré-pesados e determinar o peso do tecido. Não use mais de 10 mg do tecido como o seu material de partida.
- Adicionar 45 mL tampão de extração (8 M uréia, 50 mM DTT, CHAPS 4%, 0,2% ampholytes transportadora 08/05, 0,0002% de azul de bromofenol) por um tecido mg picada, à temperatura ambiente. Se o volume for superior a 350 mL, adicione 350 mL inicialmente e homogeneizar (passo 1.4), em seguida, adicione o restante do buffer para minimizar respingos.
- Homogeneizar o tecido sete vezes com um pilão motorizado (Kontes Corp) por 15 segundos a4 ° C, com dois minutos de resfriamento sobre o gelo entre cada homogeneização.
- Centrifugar as amostras a 15.600 xg por 30 minutos a 4 ° C. Salve o sobrenadante e medir o seu volume. A célula de detritos pellet é descartado.
- Precipitar as proteínas no sobrenadante com acetona gelada grau reagente (3 vezes v: relação v). Vortex por 15 segundos. Centrífuga para 15.600 xg por 10 segundos para formar pelotas, e ressuspender em tampão de extração com o motor Kontes e hastes de polipropileno mexendo (BioSpec Prod)
- Repita o passo 1,6.
- Quer continuar com a estimativa de concentração de proteínas das amostras ou armazená-los a -20 ° C.
2. Estimativa de concentração de proteína
Use o ensaio Lowry 7 ou 8 do ensaio BCA (Pierce); apontar para cerca de 4,5 mg ml -1.
3. Focalização isoelétrica
- Retire uma tira IPG ReadyStrip (11 cm, pH 5-8, Bio-Rad)de -20 ° C e deixe-se equilibrem em temperatura ambiente por 10-15 min.
- Vortex da amostra e pipeta 600-800 g (volume total 200 mL) de amostra de proteína em uma linha reta na extremidade traseira de um canal na bandeja de reidratação de amostra, deixando cerca de 1 cm em cada extremidade.
- Usando pinça, retire a tampa de plástico da tira IPG - certifique-se de lidar com apenas as extremidades da tira e evitar qualquer contato com o gel. Nota final básicos da tira e posicione-o no lado esquerdo da bandeja. Coloque a tira de IPG gel lado-down no topo da amostra, evitar bolhas prendendo embaixo. Permita que ele se hidratar durante uma hora.
- Sobreposição com 2,5 mL de óleo mineral (BioRad). Cubra o tabuleiro com a tampa, e deixe hidratar durante a noite a temperatura ambiente.
- Coloque um pavio de papel em ambas as extremidades do canal bandeja de foco e molhados cada um com 10 mL de água Millipore.
- Retire IPG strip e mantenha verticalmente por cerca de 10 segundos a drenar o óleo. Blot a b plásticoacking com um Kimwipe.
- Coloque o lado gel IPG strip para baixo e com final básicos para a esquerda na bandeja de foco. Cobrir a faixa com 2,5 mL de óleo mineral.
- Coloque a bandeja para dentro da célula IEF PROTEAN (Bio-Rad) e um programa de um protocolo de 3 etapas (Tabela 1) à temperatura de célula padrão de 20 ° C, uma corrente máxima de 50 mA / strip, e não há tempo de reidratação.
Tensão Tempo Volt-Hrs Rampa Passo 1 250 20 min Linear Passo 2 8000 2,5 hr Linear Passo 3 8000 40000 Rápido Total 6,5 hr 40000 Tabela 1. Três etapas protocolo de PROTEAN célula IEF de 11 centímetros IPG tiras.
- Pegue as tiras IPG para fora da célula IEF usando uma pinça. Blot o apoio de plástico com uma Kimwipe e coloque o lado gel IPG strip-se em uma bandeja de reidratação limpo. Você pode cobrir a bandeja de reidratação e envolvê-la com filme plástico e guarde a - 80 ° C ou prosseguir.
4. SDS eletroforese em gel de poliacrilamida
- Transferência da faixa (até lado gel) em uma bandeja de equilíbrio 11 cm.
- Adicionar 4 mL de tampão de redução (6 M uréia, 2% SDS, 0,05 M Tris / HCl pH 8,8, glicerol 20%, DTT 2%) para o canal e equilibrar a tira durante 10 min com agitação suave.
- Remova o tampão de redução, adicionar 4 mL de tampão de alquilação (6 M uréia, 2% SDS, 0,05 M Tris / HCl pH 8,8, glicerol 20%, 2,5% iodoacetamida) e equilibrar a tira durante 10 minutos com agitação suave.
- Lave a tira IPG por imersão a strip brevemente em um tampão SDS X rodando (25 mM Tris, 192 mM de glicina, 0,1% SDS, pH 8.2).
- Lay o lado gel strip para cima da placa traseira do Critério de 11 cm pré-moldados de 10,5% -14% Tris-HCl em gel de poliacrilamida SDS (Bio-Rad) e empurre-o suavemente de modo que ele faz pleno contato com o gel SDS; certifique-se não haja bolhas entre as duas superfícies gel.
- Sobreposição da tira IPG com 1 mL de solução de agarose derretida (0,5% agarose, 25 mM Tris, 192 mM de glicina, 0,1% SDS, de azul de bromofenol) e deixe o agarose solidificar por 5 minutos.
- Carga de 5 mL de proteína marcadora 10-225 kDa (USB, P / N: 76740) no único poço como padrões de peso molecular.
- Execute o gel em tampão SDS (25 mM Tris, 192 mM de glicina, 0,1% SDS, pH 8,2) em 200 V à temperatura ambiente ou em uma câmara fria (4 ° C) usando a migração frente corante azul de bromofenol para monitorar a eletroforese.
- Remova o gel do elenco de plástico e mancha durante a noite, agitando suavemente em Coomassie Brilliant BlueR-250 mancha (45,5% de metanol, 45,5% dH2O, 9,0% de ácido acético, 0,25% Coomassie Brilliant Blue R-250).
- Agitar o gel em uma solução Destain (45,5% de metanol, 45,5% dH 2 O, 9,0% de ácido acético) duas vezes por 3 horas cada um e em Destain 2 (metanol 5%, 90% dH 2 O, 5% de ácido acético) durante a noite. Armazenar o gel em 7% de ácido acético para análise.
5. Gel de imagem e análise
- Capturar imagens dos géis usando o programa de software One Quantidade que opera o sistema de imagem VersaDoc (BioRad). Cortar as áreas da imagem de modo uniforme para todos os géis em sua experiência.
- Carregar as imagens recortadas no programa PDQuest software 8.0 para criar um conjunto de experimento. Siga o assistente de configuração da experiência para definir os parâmetros para detecção de ponto e ponto de correspondência através do gel. Inspecionar e refinar o local correspondência resultados manualmente, se necessário.
- Realizar uma análise quantitativa e estatística dos géis para detectar manchas de proteínas combinado com two-fold ou superior diferenças estatisticamente significativas na expressão entre os dois grupos gel. Crie uma análise conjunto com estes pontos de interesse e cortá-los, utilizando um cortador local EXQuest (BioRad) para tripsina digestão e LC-MS identificação baseada em proteína.
6. Em gel-digestão tríptica
Para esta etapa um Pierce modificado In-Gel Digest tríptico Kit (# 89871X, Pierce) protocolo é usado.
- Adicionar 200 mL de solução de descoloração (25 mM de bicarbonato de amônio, acetonitrila 50%) para as velas em gel. Vortex e incubar as amostras a 37 ° C por 30 minutos com agitação. Remova e elimine cuidadosamente a solução.
- Repita o passo 6.1.
- Adicionar 30 mL de recém-preparados buffer (50 mM Tris [2-carboxietil] fosfina, 22,5 mM de bicarbonato de amônio) para os tubos contendo as velas em gel e incubar a 60 ° C por 10 minutos. Reduzindo
- Permitem que as amostras para esfriar e, depois, remover e descartar o buffer de redução.
- Adicionar 30 mL de tampão de alquilação (100 iodoacetamida mM, 20 mM de bicarbonato de amônio, preparado imediatamente antes da utilização em papel alumínio envolto em tubos. Incubar as amostras no escuro à temperatura ambiente por 1 hora.
- Remova e descarte o buffer de alquilação. Lave as velas gel pela adição de 200 mL de solução de descoloração a cada tubo. Incubar as amostras a 37 ° C por 15 minutos.
- Remova e descarte a solução de descoloração e repita a lavagem.
- Adicionar 50 mL de acetonitrila para as velas em gel e incube-os por 10 minutos à temperatura ambiente, que lhes permitam reduzir seca, em seguida, remova cuidadosamente a acetonitrila.
- Repita 6,8 passo mais uma vez. Pedaços de gel devem ser brancos e pequenos.
- Permitem que pedaços de gel para secar em um concentrador de CentriVap (Labconco, modelo EX1245) por 10 minutos.
- Engrossar as peças gel, adicionando 10 mL de solução de tripsina ativado (10 tripsina ng / mL, bicarbonato de amônio 25 mM). Incubar à temperatura ambiente por 15 minutos.
- Adicionar 25 mL tampão de digestão (25 mM de bicarbonato de amônio) para os tubos. Incubar as amostras à temperatura ambiente durante a noite com agitação.
- Sonicate as amostras por 10 minutos (Branson, modelo banho sonicador 2510) e spin-las antes de remover o sobrenadante para um novo tubo. Salve o sobrenadante.
- Para continuar a extrair os peptídeos, adicionar 10 mL de 0,1% de ácido fórmico e incubar por 5 minutos em temperatura ambiente com agitação. Sonicate as amostras por 10 minutos, recolher o sobrenadante e combinam com sobrenadante salvou na etapa 6.13.
7. Dessalinização microescala de extratos de peptídeo
- Remover bicarbonato de amônio e outros sais nos extratos peptídeo com a ajuda de PepClean C-18 Colunas Spin (ThermoFisher) de acordo com as instruções do fabricante.
- Peptídeos eluir duas vezes com 20 mL de acetonitrila 70%, evaporar as amostras secas por centrifugação no Concentrador CentriVap por 1 hora e ressuspenderos peptídeos em 10 mL de ácido fórmico 0,1% para análise de MS.
8. Identificação de proteínas por espectrometria de massa HPLC-coupled
- 5 mL separado da mistura de peptídeos em um cromatógrafo líquido de alta performance (HPLC) mais de 40 minutos, utilizando um gradiente linear de acetonitrila 20-50% com 0,2% de ácido fórmico em um Pepswift coluna PS-DVB monolítica (Dionex) acoplado a um nanospray I de origem para um espectrômetro de massa ion trap (LCQ Deca XP Max, Thermo Fisher Scientific) a uma vazão de 400 nL / min na ponta.
- Detectar peptídeo MS1 sinais entre 400-1400 m / z e permitir a três picos de íon mais intenso a ser isolado para MS2 seqüenciamento por CID com a exclusão dinâmica.
- Para identificação de proteínas, analisar os resultados através de um peptídeo Sequest pesquisa de banco de dados do mouse de referência (BioWorks software, Thermo-Fisher Scientific) com uma modificação cisteína estática alquilados e modificações dinâmicas para fosforilação (serina, treonina, tirosina), methylamento (histidina), oxidação (metionina), ADP ribosylation (arginina), e N-terminal acetilação. Aplicar critérios conservadores de correspondência (por exemplo, a tolerância peptídeo definido para 2 AMU e 1,00 tolerância fragmento, proteínas contêm pelo menos 2 peptídeos passar um Xcorr Carga vs Estado de filtro [z = 1 / x = 1,5, z = 2 / x = 2,00, z = 3 / x = 2,50, z = 4 / x = 3,00] e uma probabilidade de p <0,05) 5 manualmente e inspecionar os espectros de MS para a identificação de proteínas confiante.
9. Resultados representativos:
Masculino e feminino não exercidas, músculo esquelético murino (bíceps braquial) foi extraído e separado em um mapa bidimensional 5, o primeiro nível do músculo comparativa proteoma (Figura 2). Uma vez visualizada usando alta resolução de imagem digital; cerca de 800 spots de proteínas foram detectadas em cada um. Usando o mapa do sexo masculino proteoma como linha de base, é claro que existem inúmeros pontos que a mudança em abundância no proteoma do sexo feminino. As intensidades de ponto são medidas da mudança nos valores de proteína (Figura 3). As identidades dos spots de proteínas que mudam mais de duas vezes (para cima ou para baixo) e são estatisticamente significativos (p <0,05) com um n = 5 camundongos, são determinados por análise de seqüência de aminoácidos por espectrometria de massa de cromatografia líquida acoplada. Estes resultados mostram que as mulheres demonstram um decréscimo abundância em enzimas de açúcar metabolismo energético e na enzima creatina quinase (CK) isoformas, um sistema de fornecimento de energia em diferentes músculos. Seres humanos e animais mostram maior sexo masculino níveis séricos de CK em repouso e exercício seguinte 9,10, mas os níveis séricos de CK não necessariamente correlacionado com a quantidade de perturbação miofibrilar 11,12. Este dimorfismo de gênero na abundância celular da CK no músculo bíceps braquial murino é uma novela finding5 e pode responder à fisiologicamente diferentes níveis séricos de CK.
Números:
g1.jpg "/>
Figura 1. Um esquema geral do protocolo para a proteômica comparativa. O protocolo está subdividido em três grupos: preparação de amostras, análise de amostra e, identificação de proteínas. As extremidades de cada um destes três grupos de protocolos também são razoáveis lugares pausa, embora os melhores resultados são garnered se o protocolo geral é realizada sem parar.
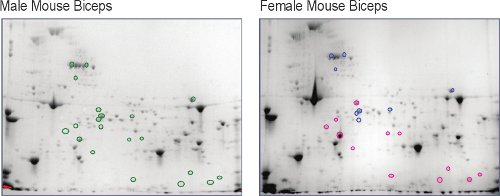
Figura 2. Comparação de proteína murina bíceps braquial feminino pontos em relação aos pontos idênticos em bíceps braquial do sexo masculino (verde círculos o). Spots de que o aumento maior ou igual a duas vezes nas fêmeas estão circulados vermelho (o) e aqueles que diminuíram menos do que ou igual a duas vezes estão circulados azul (o).

Figura 3 Análise Gel Intensity Spot:. Eixo X, do sexo femininopré-exercício (controle, n = 5); eixo Y, única fêmea luta 0 grupo de pontos de tempo h (n = 5). Linha de regressão: coeficiente de correlação = 0,919; inclinação = 0,976; interceptar = -0,0293. Pontos acima da linha vermelha e abaixo da linha azul mudar mais de + / - duas vezes.
Discussão
A LC / MS proteômica método aqui apresentado é um protocolo mais confiável e reprodutível para uma rápida análise do primeiro nível do proteoma do músculo esquelético. Que permite uma comparação razoavelmente expediente da especificidade de gênero. Proteínas baixa abundância exigiria um fracionamento da amostra do músculo para eliminar o maior número de proteínas contráteis quanto possível, aumentando assim as proteínas pouco abundantes. Adaptando o perfil proteoma pode ser realizado com diferentes pH tiras gama IPG e géis de gradiente alternativo percentual, se desejar. Mais sensíveis manchas fluorescentes existe, mas é recomendável que cada laboratório determine a linearidade destas manchas em seu sistema. Para uma análise mais rigorosa de expressão da proteína do sistema DIGE (bidimensional In-Gel sistema de eletroforese, a GE Healthcare Life Sciences) pode ser usado, no entanto, este não adiciona um nível de complexidade da metodologia. Além disso, o protocolo aqui descrito pode ser usado com a maioria dos animal tecidos para os quais um genoma foi seqüenciado, bem como de seqüenciamento de novo de uma proteína a partir de qualquer fonte, desde que ele pode ser resolvido em um gel de duas dimensões, uma vez que se sobrepõem fragmentos enzimática pode ser gerado usando enzimas de elevada pureza, além à tripsina. Amostras a partir de fontes de outros tecidos pode exigir algumas alterações na metodologia de extração, especialmente se procurar proteínas da membrana e hidrofóbicas, no entanto, temos tido bons resultados usando este protocolo com cardíacas, fígado, rim e tecidos cerebrais. Outras modificações pós-traducionais podem ser detectados através da modificação do espectro de massa critérios de pesquisa do banco de dados. Em suma, temos apresentado um protocolo razoavelmente detalhadas inicial para análises de proteoma.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos a Yutian Gan para o uso da Figura 3. Este trabalho foi financiado pela National Science Foundation 0420971; do Smith College Blakeslee, Wilens e fundos de Holmes, e da Howard Hughes Medical Institute.
Materiais
| Name | Company | Catalog Number | Comments | |
| Nome do reagente | Tipo | Companhia | Número de catálogo | Comentários |
|---|---|---|---|---|
| PepClean C-18 Giro Colunas | Consumível | ThermoFisher Científico | PI-89870 | |
| Pepswift monolítica coluna (100um x 5 cm) | Consumível | Dionex | 162348 | |
| Critério de pré-moldados 10,5% -14% Tris-HCl SDS géis de poliacrilamida | Consumível | BioRad | 345-0106 | |
| Readystrip tiras IPG | Consumível | BioRad | PH variando faixas | |
| Ácido acético | Reagente | Fisher Scientific | A465-1 | |
| Acetona | Reagente | Pharmco | 329000 | |
| Acetonitrila | Reagente | Fisher Scientific | A955 | |
| Agarose | Reagente | BioRad | 163-2111 | Overlay solução |
| Bicarbonato de amônio | Reagente | Fluka | 40867 | |
| Azul de bromofenol | Reagente | Fisher Scientific | BP114 | |
| Bio-Lyte Ampholytes | Reagente | BioRad | PH variando faixas | |
| CHAPS | Reagente | USB | 13361 | |
| Commassie blue R-250 | Reagente | ThermoFisher Científico | 20278 | |
| Ditiotreitol (TDT) | Reagente | USB | 15395 | |
| Ácido fórmico | Reagente | ThermoFisher Científico | 28905 | |
| Glicerina | Reagente | Sigma-Aldrich | G6279 | |
| Glicina | Reagente | USB | 16407 | |
| Iodoacetamida | Reagente | Sigma-Aldrich | I6125 | |
| Metanol | Reagente | Fisher Scientific | A452 | |
| Óleo mineral | Reagente | BioRad | 163-2129 | |
| Dodecil sulfato de sódio | Reagente | Sigma-Aldrich | L6026 | |
| Tris base de | Reagente | Sigma-Aldrich | 93349 | |
| Tris [2-carboexyethyl] fosfina | Reagente | ThermoFisher Científico | 77720 | |
| Tripsina Endoproteinase, TPCK tratado, grau MS | Reagente | Perfurar | 90055 | modificado |
| Uréia | Reagente | USB | 75826 | |
| Água | Reagente | Burdick & Jackson | 365 | |
| Concentrador CentriVap | Tool | Labconco | ||
| Exquest cortador local | Ferramenta | BioRad | ||
| LCQ Deca XP Max ion espectrômetro de massa armadilha | Ferramenta | ThermoFisher Científico | ||
| Motorizada pilão | Ferramenta | Kontes Corp | ||
| Polipropileno mexendo | Ferramenta | BioSpec Prod | ||
| varas | ||||
| PROTEAN célula IEF | Ferramenta | BioRad | ||
| Sonicador | Ferramenta | Branson | ||
| Além disso Surveyor HPLC sistema | Ferramenta | ThermoFisher Científico | ||
| VersaDoc imagem sistema | Ferramenta | BioRad |
Referências
- Fehrenbach, E., Mooren, F. C., Völker, K. . Molecular and Cellular Exercise Physiology. , 199-217 (2005).
- Mooren, F. C., Mooren, F. C., Völker, K. The Cell. Molecular and Cellular Exercise Physiology. , 3-18 (2005).
- Thompson, H. S., Clarkson, P. M., Scordilis, S. P. The repeated bout effect and heat shock proteins: intra-muscular HSP27 and HSP70 expression following two bouts of eccentric exercise in humans. Acta. Physiol. Scand. 174, 47-56 (2002).
- McHugh, M. P. Recent advances in the understanding of the repeated bout effect: the protective effect against muscle damage from a single bout of eccentric exercise. Scand. J. Med. Sci. Sports. 13, 88-97 (2003).
- Metskas, L. A., Kulp, M., Scordilis, S. P. Gender Dimorphism in the Exercise-Naïve Murine Skeletal Muscle Proteome. Cell Molec. Biol. Lett. 15, 507-516 (2010).
- Lopez, J. L. Two-dimensional electrophoresis in proteome expression analysis. J. Chromatogr. B. 849, 190-202 (2007).
- Lowry, O. H., Rosenburg, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J. Biol. Chem. 193, 265-275 (1951).
- Smith, P. K., Krohn, R. I., Hermanson, G. T., Mallia, A. K., Gartner, F. H., Provenzano, M. D., Fujimoto, E. K., Goeke, N. M., Olson, B. J., Klenk, D. C. Measurement of Protein Using Bicinchoninic Acid. Anal. Biochem. 150, 76-85 (1985).
- Norton, J. P., Clarkson, P. M., Graves, J. E., Litchfield, P. L., Kirwan, J. Serum creatine kinase activity and body composition in males and females. Hum. Biol. 57, 591-598 (1985).
- Amelink, G. J., Kamp, H. H., Bär, P. R. Creatine kinase isoenzyme profiles after exercise in the rat: sex-linked differences in leakage of CK-MM. Pflügers. Arch. 412, 417-421 (1988).
- Kendall, B., Eston, R. Exercise-induced muscle damage and the potential protective role of estrogen. Sports Med. 32 (2), 103-123 (2002).
- Tiidus, P. M. Can oestrogen influence skeletal muscle damage, inflammation, and repair?. Br. J. Sports Med. 39, 251-253 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados