É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fabricação e uso de microarranjos microambiente (MEArrays)
Neste Artigo
Resumo
Um método de triagem combinatória funcional para obtenção de conhecimentos sobre os impactos da composição molecular de microambientes sobre as funções celulares é descrito. O método tira partido de microarray existentes baseados em tecnologias para gerar matrizes de microambientes definidos combinatórias que suportam a adesão de células e análise funcional.
Resumo
As interações entre as células e seu microambiente circundante tem consequências funcionais para o comportamento celular. No nível de célula única, microambientes distintos podem impor diferenciação, migração, proliferação e fenótipos, e sobre o nível do tecido processa o microambiente complexo como a morfogénese e tumorigénese 1. Não só o celular e molecular conteúdo de microambientes impacto que as células dentro, mas assim como a elasticidade 2 e 3 geometria do tecido. Definido como a soma total de célula-célula,-ECM, e solúveis em interacções dos factores, para além das características físicas, o microambiente é complexa. Os fenótipos de células dentro de um tecido são parcialmente devido ao seu conteúdo genómico e em parte devido às interacções com o microbiota combinatórias. Um grande desafio é unir combinações específicas de componentes microambientais com comportamentos distintos. ent "> Aqui, nós apresentamos o microambiente microarray plataforma (MEArray) para a célula baseada em triagem funcional de interacções com microambientes combinatórias 4. O método permite um controlo simultâneo da composição molecular e o módulo de elasticidade, e combina a utilização de microarray amplamente disponíveis tecnologias e micropatterning. telas MEArray requerem tão pouco quanto 10.000 células por matriz, o que facilita estudos funcionais de tipos de células raras, tais como as células progenitoras adultas. Uma limitação da tecnologia é que microambientes tecidos inteiros não pode ser completamente recapitulado em MEArrays. No entanto, a comparação das respostas no mesmo tipo de célula a numerosas microambientes relacionados, por exemplo, combinações de pares de proteínas de ECM que caracterizam um dado tecido, irá fornecer informações sobre a forma como os componentes do microambiente eliciar tecido-específicos fenótipos funcionais.
MEArrays podem ser impressos usando uma grande variedade de recombinante grofactores wth, citoquinas, e proteínas purificadas de ECM, e suas combinações. A plataforma é limitado apenas pela disponibilidade de reagentes específicos. MEArrays são passíveis de tempo caduco análise, mas na maioria das vezes são usados para análises de ponto final de funções celulares que são mensuráveis com sondas fluorescentes. Por exemplo, a síntese do DNA, a apoptose, a aquisição de estados diferenciados, ou a produção de produtos de genes específicos são normalmente medidos. Resumidamente, o fluxo de base de um experimento MEArray é preparar as lâminas revestidas com substratos de impressão e para preparar a placa mestre de proteínas que estão a ser impressa. Em seguida, as matrizes são impressas com um robô microarray, as células são deixadas a ligar, crescer em cultura, e, em seguida, são quimicamente fixados ao atingir o ponto de extremidade experimental. Ensaios fluorescentes ou colorimétricas, fotografados com microscópios tradicionais ou scanners microarray, são utilizados para revelar relevantes fenótipos celulares e moleculares (Figura 1).
Protocolo
1. Preparação impressão Substratos
A decisão para usar polidimetilsiloxano (PDMS) com revestimento ou de poliacrilamida (PA)-lâminas revestidas depende dos parâmetros importantes do design experimental. O módulo de elasticidade dos dois polímeros pode ser ajustado para imitar as rigidezes de tecidos diferentes, alterando a relação de base / cura de PDMS, e o rácio de acrilamida / bis-acrilamida de PA. PDMS pode imitar os tecidos mais rígidos na gama de 1-10MPa (por exemplo, cartilagem, a córnea, e as paredes das artérias), e de PA pode imitar os tecidos macios no intervalo entre 100 Pa-100kPa (por exemplo, da mama, do cérebro, do fígado, da próstata e) 5. PDMS é barato, fácil de preparar, e a geometria dos elementos impressos serão idênticos para a cabeça dos pernos de impressão. Assim, o tamanho e forma dos elementos pode ser controlado com precisão utilizando pinos com geometrias de ponta diferentes. PDMS é mais hidrofóbica do que PA, o que faz com que alguns desafios durante a manipulação de células e immunoscontendo os passos, e podem ser incompatíveis com algumas linhas de células. Porque PA é um hidrogel e uma superfície não-incrustante nativa, as células só juntar aos pontos em que existem proteínas que a adesão de células de suporte. A geometria dos elementos impressos sobre géis de PA não seguir precisamente a geometria da cabeça de alfinete, geralmente eles tornam-se círculos devido à difusão, independentemente da geometria da cabeça de alfinete, que é usado. Impressão de contacto parâmetros de tempo e de diâmetro do pino pode ser determinada empiricamente por tamanho em géis de funcionalidade óptima PA.
Polidimetilsiloxano (PDMS)
- Em um copo de plástico descartável combinar Sylgard 184 base de elastómero de silicone com o agente de cura a uma razão de 10:1, misturar vigorosamente com uma espátula de madeira ou de plástico, em seguida, depressor desgaseificar em um sino de vácuo à temperatura ambiente durante 30 min.
- Centro de uma lâmina de microscópio padrão sobre o mandril de vácuo accionada de um dispositivo de revestimento de centrifugação, em seguida, 0,5 ml garoa do polímero elastómero misturado para o centro da superfície de deslizamento. Girar em6000 rpm durante 60 segundos.
- Curar as lâminas PDMS revestidos em um forno 70 ° C ou sobre uma placa quente digitais (protegida do pó) durante 4 horas a durante a noite.
- Lâminas curados podem ser utilizados imediatamente ou armazenados durante vários meses, de uma caixa de lâmina que está selado no interior de um saco de plástico Ziploc e mantida dentro de uma gaveta. O PDMS atrai pó então ele deve ser bem protegidos contra a circulação de ar do quarto.
- Nota: Nitrilo ou outros sem látex luvas devem ser usadas quando se trabalha com o kit de elastômero PDMS. Contato acidental com luvas de látex vai inibir a polimerização PDMS.
Poliacrilamida (PA)
- NaOH ataque: Colocar as lâminas em bloco de aquecimento a 80 ° C. Adicionar 1 mL de NaOH 0,1 N em cada lâmina, certificando-se a cobrir a superfície da lâmina inteira. Deixe a evaporar NaOH (uma película branca devem formar na superfície de deslizamento). Uma vez que o gel PA só pode anexar firmemente em superfícies NaOH gravadas, o gel PA vai destacar durante a secagem passo se o surf slide inteiroace não está coberto por NaOH. Se a superfície da lâmina não foi completamente coberto, repetir-se adicionando 1 ml de 0,1 N NaOH. As lâminas podem então ser armazenado à temperatura ambiente (RT) durante vários dias. Nota: Uma alternativa ao ataque químico NaOH é de ozono ou plasma limpar as lâminas.
- 3-aminopropiltrietoxissilano revestimento (APES): Em um exaustor, Colocar as lâminas em um prato de 15 ml e adicionar 300 uL APES em cada slide gravado NaOH. Deixe os macacos reagem com o NaOH desliza por 5 min. Superior desta vez vai causar dificuldade em lavagem reagente APES que não reagiu. Lavar a APES completamente com água desionizada duas a três vezes em ambos os lados das lâminas. Se a lavagem não é completa, APES será oxidado por glutaraldeído, para formar um depósito castanho em lâminas no passo 1.8.
- Oxidação glutaraldeído: Aspirar todas as soluções a partir das superfícies de deslizamento. Em cada prato de 15 cm, adicionar 25 ml de glutaraldeído a 0,5% em PBS. Se reagir durante 30 minutos numa área escura. Após 30 minutos, aspirar tudo o glutaraldeído e uso não-lint trabalhotoalhetes Atory (Kimwipes por exemplo) para cuidadosamente secar os slides. As lâminas podem então ser armazenado à temperatura ambiente durante até um dia.
- Preparação do gel: Após a preparação de misturas de PA, incluindo acrilamida, bis-acrilamida e 2 O em DDH acordo com a tabela abaixo, desgaseificar durante 30 min, e em seguida colocar misturas de PA em gelo para retardar a polimerização. Adicionar APS e TEMED e misture bem antes de fazer direito os géis. Pipeta misturas PA sobre as superfícies de deslizamento e local 24 mm x 50 mm, um número de lamelas no topo da PA. Evite pressionar lamínula e lâmina de vidro em conjunto e evitar a formação de bolhas. Para géis> 40.000 Pa usar 100 ul, para outros géis usar 350 ul.
| Módulo desejado (Pa) | % De acrilamida | Bis-acrilamida% | Acrilamida do estoque de 40% (ml) | Bis-acrilamida a partir de estoque 2% (ml) | Água desionizada (ml) | APS (ul) | TEMED (ul) |
| 480 ± 160 | 3 | 0,06 | 0,75 | 0,3 | 8,95 | 100 | 10 |
| 4470 ± 1190 | 5 | 0,15 | 1,25 | 0,75 | 8 | 100 | 10 |
| 40.400 ± 2390 | 8 | 0,48 | 2 | 2,4 | 5,6 | 100 | 10 |
Adaptado de 6,7.
- Deixe o gel PA polimerizar por 2 horas, e depois remover lamínulas em água deionizada.
- Lavar as lâminas de gel de PA em grandes frascos Coplan em água durante a noite (~ 8 horas), para remover acrilamidas não reagidos.
- Lâminas secos em um forno de 37 ° C por 2-4 horas, ou até completamente PA gel endurece.
- PA gel de lâminas podem ser armazenadas a 4 ° C, durante um mês, uma caixa deslizante vedado.
2. Preparação de proteína Placa Mestre
- Todos shoul proteínasd ser aliquotadas em stocks de soluções 10X nos tampões recomendados pelo fornecedor e armazenado a -80 ° C. A maioria das proteínas de ECM são solúveis em água desionizada, mas o pH poderá ter de ser ajustada com gotas de ácido acético. A maioria dos factores de crescimento, citocinas, e os domínios extracelulares dos receptores recombinantes são preparadas em PBS com BSA, mas as condições do fabricante irá variar. Filtrar as alíquotas de proteína através de um de 0,45 4 mm filtro de seringa de nylon (Nalgene), antes do armazenamento.
- Projetar uma placa mestre de acordo com combinações de proteínas desejadas e diluições. As células aderentes geralmente dependem da presença de pelo menos um ECM compatível para mediar a adesão celular, mas os anticorpos para epítopos de superfície celular também pode mediar ligação às vezes. É uma boa ideia para adicionar corante FITC livre ou uma proteína fluorphore-conjugado a pelo menos um poço de modo a que as matrizes podem ser facilmente orientada mais tarde.
- Preparar a placa mestre diluindo as combinações de proteína com tampão de impressão composta por 100 mMTris-acetate/20% glycerol/0.05% de Triton-100X pH 5,2. Tipicamente, cada poço de uma placa de 384 poços não contém mais de 10 ul.
- Grave o conteúdo de cada poço em cada placa mestre em uma guia do arquivo de base de dados e fornecer delimitado cada prato principal com um número de identificação único. A data de seis dígitos, seguido pelas iniciais do designer muitas vezes serve o propósito (MMDDYYinitial). Uma vez que os volumes são pequenos assim, alíquotas de proteína, pode ser usado eficientemente para gerar um grande número de placas de replicados. Recomenda-se que as placas mestras são armazenadas à temperatura de -80 ° C e cada placa mestra deve submeter-se a não mais de dois ciclos de congelação-descongelação.
3. Impressão MEArray
- MEArrays pode ser impresso com robôs de impressão mais convencionais microarray. Impressoras pinos Quill que usam silicone ou pinos de aço inoxidável funcionam bem, mas a viscosidade proteína pode ser problemático. Impressoras capilares são robôs microarray ideais de impressão para esta aplicação, como eles funcionamBem, com soluções de proteína viscosos.
- Para atingir poder estatístico bom dentro de uma matriz, 10 a 12 pontos replicatas de cada microambiente é recomendado. Uma tal concepção permite a comparação de actividade num microambiente em relação ao outro na mesma matriz simples usando o teste T de estatísticas. Dunnette teste pode ser usado para comparar a actividade de um ambiente de controle com microambientes outros. Esta concepção funciona melhor quando um fenótipo funcional tem sido associada com o micro-ambiente de controle antes de realizar as experiências MEArray.
- Umidade deve ser mantida em torno de 50%. Controlo da humidade é importante, porque uma baixa humidade pode secar a solução para dentro os pinos ou nas cavidades da placa mestre causando deposição ineficiente dos substratos de impressão. A umidade pode ser controlada eficazmente por draping o robô com os não-porosa folhas de plástico e usando tanto umidificador e um umidificador de-definido para manter a umidade de 50%. Plattens de impressão pode ser arrefecido useful para preservar algumas proteínas, mas o cuidado deve ser tomado para evitar a formação de condensação nos slides.
- Cada matriz impressa deve ser rotulado com rótulos congelador à prova de slides codificados com um número de série que consiste de identificador da placa mestre seguido por um número de três dígitos (MMDDYYinitial-nnn). Como cada matriz é usado ou distribuído, detalhes de seus tratamentos experimentais devem ser mantidos em um banco de dados. Rastreamento das datas de impressão e os números de ciclos de congelamento e descongelamento das placas mestre vai ajudar a identificar as condições ideais para manter a reprodutibilidade.
- MEArrays impressos devem ser armazenados em caixas de slide selados a -20 ° C durante não mais de um mês. Reprodutibilidade visivelmente declina.
4. Cultura de células de mamíferos em MEArrays de Análise Funcional
- Anexar câmaras de cultura: Para limitar o volume dos meios de comunicação e os números de cultura de células necessário no MEArrays, uma câmara de plástico é fitted rodear a matriz impressa. Para muitas matrizes, uma câmara única a partir de uma lâmina de duas câmaras (Nunc), que contém uma área de 4,2 cm 2, podem ser usados. Retire as câmaras do slide câmara de manufaturados e cortar as câmaras no meio com uma lâmina de barbear. Usar uma seringa de 3 ml para aplicar uma camada fina de silicone do aquário (DAP) à extremidade de uma câmara e pressionar na superfície de um MEArray. Evite colocar a câmara de silicone aplicada aquário sobre as características da matriz.
- De bloqueio e de lavagem: MEArrays precisam de ser bem enxaguados para remover monómeros que não reagiram, os quais podem ser tóxicos para as células. Se PDMS-lâminas revestidas foram utilizados, em seguida, as regiões entre as características impressas primeiro necessita de ser bloqueado com uma camada de incrustação não-para impedir a adesão celular; incubar as matrizes em Pluronic F108 a 1% (BASF) em água ou em BSA a 2% em água durante 15 minutos sob vácuo. Lâminas de gel de PA não requerem um passo de bloqueio. Em todos os casos, lavar matrizes com meios de cultura de células três vezes por cinco minutos (depende escolha de mídiasobre as células utilizadas, mas o uso de antibióticos é recomendado, independentemente dos meios de comunicação ou de células). Géis PA exigem incubação adicional de 30 min na mídia para hidratar o gel.
- Fixação das células: Quatro a cinco matrizes pode caber dentro de um único 15 centímetros placa de Petri estéril. Cobrir a placa de Petri com uma tampa para manter as matrizes estéril. Adicionar metade do volume de meio final ao MEArray adicionando as células em meio a uma concentração final de 10.000 a 1.000.000 de células / ml. Células atribui às características impressas a velocidades diferentes, dependendo da composição do microambiente impresso. Verifique para fixação uniforme exibindo as matrizes através de um microscópio invertido etapa em 15 a 20 minutos de intervalo. Agitando os MEArrays frente e para trás, as células ligadas de uma forma padronizada pode ser distinguida das células flutuantes, não acopladas.
- Remoção das células não ligadas: Na PA revestidos MEArrays, as células não ligadas podem ser aspirados e os meios de comunicação podem ser substituídos com um volume adequado. Em PDMS revestido MEArrays, os meios nunca pode ser completamente removida do poço, porque as células secar e morrer quase imediatamente. Assim, em PDMS revestido MEArrays, as células não ligadas deve ser removido por um processo de trocas sucessivas de metade do volume de meio até que todas as células não ligadas são removidas, como determinado por exame microscópico. O efeito de humedecimento de PDMS é menos proeminente quando soro contendo meios de comunicação é utilizado em relação ao meio definido, e quando a BSA é utilizado para bloquear as áreas não impressas em comparação com Pluronics F108.
- As células podem ser cultivadas em MEArrays colocados dentro pratos de Petri de 15 cm durante muitos dias, com alterações normais dos meios. Mudanças de mídia em slides PDMS deve ser feito com sucessivas mudanças de metade do volume de mídia.
- Fixadores comuns, tais como o paraformaldeído e metanol / acetona, são compatíveis com os sistemas de MEArray. Quando a coloração de células de PA revestido MEArrays, fixadores podem ser adicionados e lavada da mesma forma que seria um procedimento de coloração convencional.No entanto, quando da coloração das células em PDMS revestido MEArrays, a superfície deve permanecer molhado, mesmo durante a fixação. Aspirar metade dos media e substituir com um fixador. Repita o processo algumas vezes, até o poço é preenchido com uma maioria de fixador. Após a fixação, o fixador é gradualmente substituído da mesma maneira com tampão de bloqueio, que é apropriado para a próxima etapa de análise.
- Imunomarcação é vulgarmente utilizado para analisar as funções celulares. Rotinas de coloração pode variar, mas quando se trabalha com os MEArrays PDMS, é necessário executar todos os passos de lavagem e aspiração como acima, mudando gradualmente as soluções e nunca permitindo que a superfície de-molhada. De molhar vai causar artefatos na coloração.
- As câmaras podem ser removidas com o auxílio de uma lâmina de barbear. As lamelas podem ser montados no topo da MEArrays coradas utilizando Fluoromount-G (Southern Biotech). A detecção pode ser realizada com a maioria dos scanners de fluorescência multicolor microarray ou em microscopia confocal com motorizadomodos de aquisição de azulejos de imagem.
5. Resultados representativos
Um exemplo de modelado deposição de proteína num impresso PDMS-coasted MEArray pinos usando um quadrado com ponta de silicone sobre um pino de eixo oco robô microarray-impressão é mostrado na Figura 2. Deposição de várias proteínas que estão impressas pode ser verificado por imunofluorescência utilizando anticorpos (Figura 2A). As diluições das soluções de proteína na placa mestre reflectem a quantidade (intensidade de fluorescência) que é depositado sobre a superfície do substrato de impressão (Figura 2B). As células devem anexar aos recursos impressos de forma óbvia padronizada (Figura 2C).
Um exemplo de uma experiência que mostra que as diluições MEArray inversas de duas proteínas microambiente eliciada perfis de expressão específicas de queratina em uma proteína de forma dependente da concentração, num humano multipotentes p epitelial mamariarogenitor linha de células (células D920), é mostrada na Figura 3. Lotes de bolhas são úteis para determinar se os fenótipos específicos são impostas sobre as células replicam as características de uma série de diluições. Por exemplo, se uma molécula particular de um microambiente provoca um fenótipo distinto, uma vez que o componente instrutivo foi diluído suficiente para um fundo de um ECM neutro o fenótipo deve mudar ou desaparecer. Detecção de imunofluorescência de queratina 8 e 14 de queratina proteínas dos filamentos intermediários foi realizada com um Axon 4200A (Molecular Devices) scanner microarray. Doze diluições em série repetição foram impressas em cada MEArray eo log 2 Proporção de queratina 8 a queratina 14 intensidade média de fluorescência foi representado graficamente como uma trama de bolha para dar uma ideia realista de variação e reprodutibilidade do sinal. É mostrado dados de um MEArray que foi fixada após as células tinham ligado e as células não ligadas foram lavadas (Figura 3A) e, após 24 horas de culture (Figura 3B). Para esta análise relativamente pequena, uma ANOVA one-way foi usada para determinar a variação do sinal de média em cada ponto do tempo, e agrupados bicaudais t-testes foram utilizados para determinar se as diversas diluições de colagénio tipo I humano recombinante e P- caderina causado mudanças na expressão de queratina. Não houve variação da média entre as células sobre as características apenas após fixação, no entanto, não houve diferenças significativas na expressão de queratina entre as células após 24 h de exposição aos diferentes microambientes. T-testes verificaram que tipo de alta concentração de colágeno I produziu maior expressão 8 queratina, enquanto que altas concentrações de P-caderina provocou uma forte queratina 14 sinal após 24 horas. Este resultado foi consistente com relatos prévios que P-caderina contendo microambientes imporão de K14 que expressam o fenótipo mioepitelial em bi-potentes células progenitoras mamárias 4.
Um exemplo de um Scanne inteirod MEArray impressa numa 40,000 Pa PA gel é apresentado na Figura 4.

Figura 1. Um diagrama de fluxo do processo de MEArray. Em primeiro lugar, o substrato de impressão são preparados quer com PDMS ou PA. Em segundo lugar, as placas de mestre está preparado e anotado em um banco de dados. Em terceiro lugar, os MEArrays são impressas e codificadas com os números de série. Quarta, câmaras de cultura estão ligados, são blocos de superfícies e / ou lavado, em seguida, as células podem se conectar e células não ligados são lavados. Em quinto lugar, as células podem ser tratadas com a coloração ou bio-ensaio após um período de incubação com base no desenho experimental. Finalmente, as Imagens de MEArray podem ser obtidas e analisadas com scanner apropriado e software.
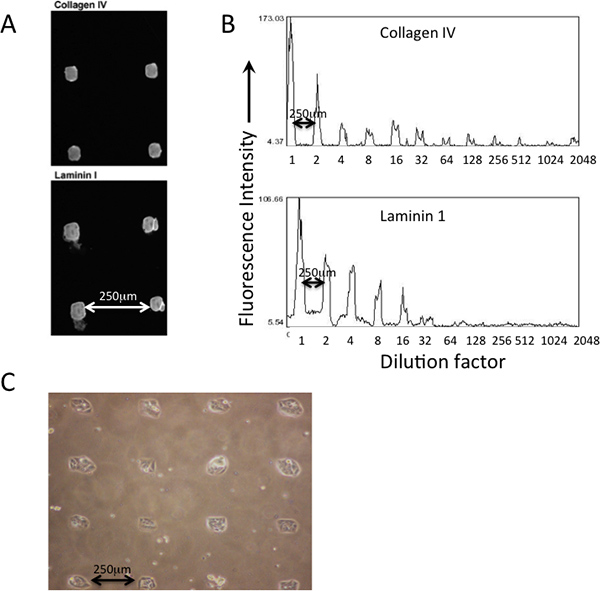
Figura 2. DEPOSition e relativa abundância de proteínas de impressos pode ser verificada com imunocoloração antes da fixação das células. A) Os anticorpos que reconhecem o colagénio tipo IV e laminina-111 foram utilizados para verificar a sua presença em características impressas de um MEArray. B) Utilizando uma média de pixel recurso de análise de intensidade de NIH ImageJ software, a abundância relativa das duas proteínas ao longo de uma série de diluições, a partir de uma solução de proteína de 200 ug / ml, pode ser avaliada qualitativamente. C) micrografia Fase D920 células aderidas em forma de quadrado características de um impresso MEArray PDMS-revestido.
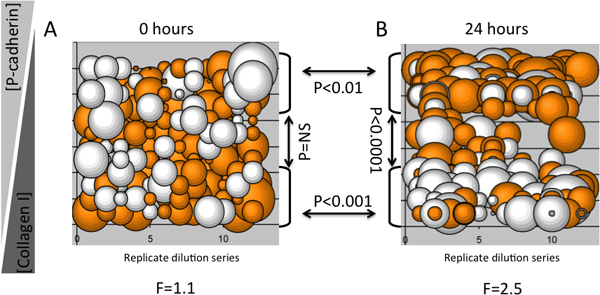
Figura 3. Um exemplo de uma análise MEArray utilizando as alterações na expressão da queratina em uma linha de células progenitoras multipotentes como funções do tempo e do microambiente. Cada bolha representa relações de queratina 8 e 14 níveis de proteína de queratina 10-15 células ligados a uma FEAtura de uma MEArray. Expressão foi determinada com sondas de imunofluorescência. A) Mostra os rácios de queratina nas células apenas após a fixação, e B) mostra as razões de queratina após 24 horas em uma matriz que foi plaqueada em paralelo. A concentração máxima de ambas as proteínas foi de 200 ug / mL e diluído duas vezes. O diâmetro de uma bolha representa a magnitude da razão de log 2 da queratina 8 e intensidade média de queratina 14, e o código de cores laranja e branco indica valores> 0 e <0, respectivamente. F-valores para uma análise de variância e P-valores de T-testes, e suportes com setas identificando as populações comparadas, são mostrados.

Figura 4. Um exemplo de uma varredura MEArray adquiridos utilizando um modo de aquisição de telhado em um microscópio confocal de laser. HCC1569 células que permitem a incorporação do análogo de DNA EdU durante 4 h antes da fixação.DAPI (azul) e Edu (vermelho) são mostrados.
Discussão
O método aqui apresentado permite MEArray análises funcionais da célula e interacções microambiente combinatórias 4. Análise MEArray combina o uso de tecnologias micropatterning básicos, biologia celular, e robôs de microarranjos de impressão e dispositivos de análise que estão disponíveis em instalações multiusuários muitos. MEArray telas são compatíveis com tipos de células mais aderentes, embora formulações meios isentos de soro podem necessitar de ser ajustado em alguns casos, de modo...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
ML é apoiado pela NIA (R00AG033176 e R01AG040081) e pelo Laboratório de Pesquisa e Desenvolvimento Dirigido, EUA Departamento de contrato de Energia # DE-AC02-05CH11231.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários (opcional) |
| Lâminas de vidro 25 mm x 75 mm | VWR | 48311-600 | |
| Lamínulas de vidro (no.1) 24 mm x 50 mm | VWR | 48393-241 | |
| Prato de coloração (ou frasco Coplan) | VWR | 25461-003 | |
| Placas de petri (15 cm) | BD Falcon | 351058 | |
| NaOH (1,0 N) | Sigma-Aldrich | S2567 | |
| APES (> 98% de (3-aminopropil) trietoxissilano) | Sigma-Aldrich | A3648 | |
| Glutaraldeído | Sigma-Aldrich | G7651 | 50% em água |
| APS (> Persulfato de amónio 98%) | Sigma-Aldrich | A3678 | Preparar uma solução de trabalho de 10% com DDQ 2 O |
| TEMED (N, N, N ', N'-tetrametiletilenodiamina) | Sigma-Aldrich | T9281 | |
| Acrilamida (40%) | Sigma-Aldrich | A4058 | |
| Bis-acrilamida (2% w / v) | Fisher biorreagentes | BP1404-250 | |
| 0,45 um filtro de seringa de nylon de 4 mm | Nalgene | 176-0045 | |
| FITC | Sigma-Aldrich | F4274 | |
| PDMS (polidimetilsiloxano) | Dow Corning | 3097358-1004 | Sylgard kit Elastômero 184 via Adesivos Ellsworth |
| 2-lâminas de câmara | NUNC | 177380 | |
| Pluronic F108 | BASF | 30089186 | |
| Aquselante arium | Dow Corning | DAP 00688 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Copos plásticos descartáveis | |||
| Abaixadores de língua | |||
| Luvas de borracha nitrílica | |||
| Caixas de plástico de slides de microscópio | |||
| Spin-coater | WS-400B-6NPP/LITE | Laurell Technologies Corporation | |
| Forno | |||
| Hotplate Digital | |||
| 384 poços | Uma marca adequada para o robô microarray | ||
| Microarray robô impressão | |||
| Fase invertido e microscópio de fluorescência | |||
| Axon microarray scanners | Molecular Devices | Existem várias configurações |
Referências
- Bissell, M. J., Labarge, M. A. Context, tissue plasticity, and cancer: are tumor stem cells also regulated by the microenvironment. Cancer Cell. 7, 17-23 (2005).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev. Cell. 6, 483-495 (2004).
- LaBarge, M. A. Human mammary progenitor cell fate decsions are products of interactions with combinatorial microenvironments. Integrative Biology. 1, 70-79 (2009).
- Kim, H. N. Patterning Methods for Polymers in Cell and Tissue Engineering. Annals of biomedical engineering. , (2012).
- Boudou, T., Ohayon, J., Picart, C., Pettigrew, R. I., Tracqui, P. Nonlinear elastic properties of polyacrylamide gels: implications for quantification of cellular forces. Biorheology. 46, 191-205 (2009).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current protocols in cell biology. Chapter 10, Unit 10 (2010).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat. Methods. 2, 119-125 (2005).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol. Syst. Biol. 2, 37 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados