Method Article
Matrix-assisted laser dessorção / ionização Time of Flight (MALDI-TOF) espectrometria de massa Análise de proteínas intactas maior que 100 kDa
Neste Artigo
Resumo
Medição de massa exata representa um passo importante durante a investigação de proteínas. De matriz assistida por laser de dessorção / ionização (MALDI) Espectrometria de massa (MS) pode ser utilizado para tal determinação e a sua principal vantagem é a tolerância para os sais, detergentes e contaminantes. Aqui, ilustramos uma abordagem acessível para a análise de proteínas maiores do que 100 kDa, por MALDI-MS.
Resumo
Efetivamente determinar massas de proteínas é fundamental para muitos estudos biológicos (por exemplo, para investigações de biologia estrutural). A determinação da massa exacta permite avaliar a correcção das sequências primárias de proteínas, a presença de mutações e / ou modificações pós-traducionais, a possível degradação de proteínas, a homogeneidade da amostra, e o grau de incorporação de isótopos no caso da etiquetagem (por exemplo, 13 C a rotulagem ).
Electropulverização ionização (ESI) Espectrometria de massa (MS) é amplamente utilizado para a determinação da massa de proteínas desnaturadas, mas a sua eficácia é afectado pela composição do tampão de amostra. Em particular, a presença de sais, detergentes, e os contaminantes compromete severamente a eficácia da análise de proteína por ESI-MS. De matriz assistida por dessorção / ionização por laser (MALDI) A EM é uma alternativa atractiva, devido à sua tolerância ao sal e a simplicidade de aquisição de dados e interpretaçãoção. Além disso, a determinação da massa de proteínas heterogéneas grandes (maiores do que 100 kDa) é mais fácil por MALDI-MS, devido à ausência de sobreposição de distribuição de estados de carga elevados, que estão presentes no espectro de ESI.
Aqui nós apresentamos uma abordagem acessível para análise de proteínas maiores que 100 kDa por MALDI-tempo de vôo (TOF). Nós ilustrar as vantagens da utilização de uma mistura de duas matrizes (isto é, ácido 2,5-di-hidroxibenzóico e ácido α-ciano-4-hidroxicinâmico) e a utilidade do método de camada fina, como abordagem para a deposição da amostra. Discutimos, também, o papel fundamental da matriz e pureza solvente, das normas utilizadas para a calibração, a energia do laser, e do tempo de aquisição. No geral, nós fornecemos informações necessárias para um novato para a análise de proteínas intactas maiores que 100 kDa por MALDI-MS.
Introdução
Biologia estrutural conta com a produção de proteínas de alta qualidade 1 e, portanto, precisa ser combinada com técnicas eficientes e confiáveis para análise de proteínas 2,3. No nosso espectrometria de massa (MS) no interior do laboratório um instituto de biologia estrutural precisamos confirmar sequência primária de proteínas, avaliar a presença de mutações e modificações pós-tradução, a degradação de proteínas, a homogeneidade da amostra, e a qualidade de marcação isotópica (por exemplo deuterado proteínas para estudos de ressonância magnética nuclear, 4). Desde biólogos estruturais usar proteólise limitada de distinguir domínios estruturalmente rígidas a partir de peças flexíveis, precisamos caracterizar de forma confiável tais proteínas truncadas usando o MS.
Quando biomoléculas são analisadas por MS, duas abordagens possíveis são utilizados para ionizar suavemente tais moléculas pesadas e instáveis. Ionização por Electrospray (ESI) ioniza as moléculas directamente a partir dofase líquida 5; de matriz assistida por laser de desabsorção de ionização (MALDI) exige que as biomoléculas são co-cristalizado com moléculas orgânicas de absorção de ultravioletas (isto é, moléculas de matriz) 6.
ESI de tempo-de-voo (TOF) MS acoplada a cromatografia líquida tornou-se uma técnica de rotina para a análise de proteínas intactas, pois permite a determinação da massa com elevada precisão (≤ 50 ppm). No entanto, é muito sensível à composição do tampão de amostra (em particular, aos sais e detergentes) e os contaminantes (por exemplo, polímeros), que são por vezes difíceis de eliminar, fazendo com que a supressão do sinal do analito.
MALDI-TOF MS representa uma alternativa eficaz para ESI-MS, porque seu desempenho é menos afetado por componentes do tampão, detergentes e contaminantes, e permite a determinação da massa de proteína intacta, com precisão suficiente (≤ 500 ppm) para a validação de seqüência. Após Protein digestão, MALDI-TOF MS pode ser também utilizado para analisar os péptidos obtidos para posterior confirmação da sequência primária por o assim chamado "impressões digitais da massa dos péptidos".
Em nossas mãos, a determinação da massa de proteínas intactas, que são maiores que 100 kDa e heterogêneo (devido a modificações ou truncamentos), é mais fácil por MALDI-MS do que por ESI-MS. Isto é devido à falta de distribuição de estados de carga sobrepostas presentes no espectro de ESI. Além disso, uma vez que o processo MALDI não é dependente do tamanho do analito 7, método que os rendimentos de alta sensibilidade, quando uma massa de biomolécula é acima de 100 kDa. Análises notáveis de proteínas intactas foram realizados usando instrumentos caseiros entre o final da década de 1980 e princípio da década de 1990, 8-11.
MALDI-TOF pode também ser usado como ferramenta de rastreio para avaliar a qualidade de amostras de proteína, porque requer menos tempo de preparação da amostra e é menos susceptível a INTERFERÊNces, devido às impurezas comuns (por exemplo, sais). Depois de uma primeira avaliação rápida por MALDI-MS, uma amostra pode ser ainda analisada por ESI-TOF para determinar a massa com uma maior precisão. Além disso, MALDI gera íons contendo menos encargos que ESI e, portanto, a aquisição e interpretação de dados MALDI é mais simples. Isso permite que os alunos trabalham em biologia estrutural para analisar suas proteínas recombinantes apenas depois de um breve treinamento.
Dois factores principais que influenciam a qualidade dos espectros de MALDI: a matriz e da técnica utilizada para a deposição da matriz (por exemplo, gotículas secas 8 e fina camada 12-16,17,18). A matriz orgânica único [por exemplo, ácido sinapinico (SA) 19-21 ou-4-hidroxicinâmico α-ciano ácido (α-CHCA) 14,22,23] é muitas vezes usado para o exame MS de proteínas intactas e complexos de proteínas com ligação cruzada . Usando α-CHCA, grupo Chait anteriormente apresentado um protocolo detalhado para preparing uma camada ultra-fina antes da análise MALDI de proteínas solúveis e de membrana 14,15. Recentemente, Gorka et al. Ilustrada do revestimento alvo à base de grafite para melhorar a análise de MALDI de péptidos e proteínas, utilizando α-CHCA como matriz 24.
Aqui, nós apresentamos um protocolo simples para a análise de proteínas intactas por MALDI-TOF MS, utilizando uma mistura de duas matrizes de: ácido 2,5-di-hidroxibenzóico (DHB) e α-CHCA 25. Foram avaliados sistematicamente o desempenho da mistura de DHB-CHCA em comparação com as matrizes de SA e α-CHCA, para o controlo de proteínas intactas. A mistura de matriz permite uma melhor resolução (ou seja, os picos de proteína são muito mais nítidas). Além disso, a presença de vários iões encargos intensas (isto é, M +2 H + 2, H 3 H 3 +, etc) possibilita uma determinação de massa mais preciso porque a resolução dos instrumentos de MALDI-TOF axial é mais elevada com menor massa sobre a carga (m / z) 26. Isto é particularmente útil para a determinação de pesos moleculares de proteínas maiores do que 100 kDa.
Maior sensibilidade também é atingido utilizando a mistura DHB-CHCA (0,5 pmol de proteína manchado no alvo MALDI).
Como mencionado acima, um fator importante que deve ser considerado quando se utiliza instrumento MALDI é a deposição de matriz. Laugesen et al. Proposto o uso de uma mistura de DHB-CHCA pela primeira vez 25, utilizando a deposição das gotículas secas. No entanto, observou-se melhores resultados (por exemplo, sensibilidade muito mais elevado) quando se utilizou o método de camada fina que a primeira camada é formada pela α-CHCA dissolvido em acetona. A camada fina método 12,27 implica a formação de um substrato homogéneo de cristais de matriz sobre o alvo de MALDI, o qual foi descrito num vídeo Júpiter anteriormente 15. Em seguida, a amostra é depositada sobre esse substrato, e finalmentematriz adicional é depositado (ver a seguir). Neste artigo, nós também ilustrar como para depositar a amostra sobre o alvo MALDI, mas também como limpar a meta de 15, para recristalizam, e preparar as matrizes.
Para concluir nosso objetivo é oferecer todas as informações necessárias para a análise de proteínas intactas para os cientistas (em particular, os biólogos estruturais), que precisam avaliar a qualidade das proteínas produzidas de forma rápida e simples e que não estão tão familiarizados com MALDI-TOF MS. Como previsto, em meados de 1990 28, MS teve um impacto sobre o aumento da pesquisa biológica, como a sua acessibilidade para os biólogos tem aumentado. Esperamos que as informações que nós fornecemos será útil para fazer MALDI-TOF acessível para os biólogos e cientistas que gostariam de começar a utilizar a espectrometria de massa.
Protocolo
1. Preparação de Amostras de proteínas: Buffer Exchange (Opcional)
5-25 uL de concentrações micromolares de proteína (1 a 20 | iM) são necessários. Permuta de tampão pode ser levada a cabo utilizando os dispositivos de ultrafiltração centrífuga (por exemplo, Vivaspin, Sartorius) ou colunas de filtração em gel de microcentrífuga (por exemplo, Micro Bio-Spin 6 colunas de cromatografia, Bio-Rad) 29,30. Passos troca de tampão pode ser repetida 2 a 3 vezes. Descrição detalhada de troca de tampão foi apresentado anteriormente 29,30. Por exemplo, nós utilizamos 20 mM de tris (hidroximetil) aminometano (ou seja, Tris) pH 8 como tampão final.
Nota: Este passo pode ser omitido em muitos casos. Deve ser realizado quando uma amostra de proteína contém moléculas ou tampões que podem interferir fortemente com a detecção MS [por exemplo, glicerol, 4 - (2-hidroxietil)-1-piperazinoetanossulfónico ácido, HEPES]. É também útil para melhorar a qudade dos espectros.
2. A recristalização de matrizes de modo a melhorar a sua pureza (Opcional)
- Verter 10 ml de etanol a 40% (EtOH) num frasco de Pyrex.
- Adicionar 600 mg de uma matriz.
- Utilizando um banho de água e de um aquecedor de resistência, aquecer a solução de matriz e mexa até que a matriz é completamente dissolvido usando uma vareta de vidro ou de um agitador magnético.
- Repita o passo 2 até que você tenha uma solução saturada.
Nota: O uso de uma muito grande volume de solvente ou dissolução da matriz muito abaixo do ponto de ebulição da solução irá causar um fraco rendimento de cristais ou sem cristais de todo.
- Deixar a solução arrefecer lentamente. Pode deixar à temperatura ambiente durante várias horas e em seguida a 4 ° C durante a noite. Nota: se os cristais não são formados, a cristalização pode ser induzida por raspagem do interior do recipiente (logo abaixo da superfície da solução), utilizando uma vareta de agitação de vidro.
- Recolha dos cristais por filtração da matriz.
- Lavar os cristais com uma quantidade mínima de solvente gelada e filtra-los.
- Permitir que os cristais para secar. Pode utilizar vácuo para este passo.
3. Limpeza de MALDI aço inoxidável Alvo
- Lavar a placa MALDI com metanol (MeOH) e limpe com um pano de limpeza feito especialmente para aplicações de laboratório (por exemplo, Kimwipe).
- Lavar o alvo com H 2 O e limpe-a com um tecido de limpeza.
- Insira a placa de MALDI em um copo de 600 ml e cobri-lo com 50% de EtOH.
- Sonicar o alvo na solução de EtOH durante 10 minutos num banho de ultra-sons.
- Se houver resíduos remanescentes na placa, repetir os passos 1-4, mais uma vez.
- Finalmente, lavar o alvo com MeOH (ou com água, consulte a nota abaixo), incliná-lo de uma forma que todo o líquido é coletado em um tecido de limpeza. Deixe a seco alvo à temperatura ambiente ou usando um azotofluxo de gás.
Nota: Um procedimento semelhante foi anteriormente descrito 15.
Nota: O solvente (MeOH ou água) utilizado para o último enxaguamento do alvo determina algumas características alvo. Se o alvo é lavado com MeOH, a amostra se espalha sobre o alvo muito mais do que pelo uso da água.
4. α-CHCA fina camada da solução: Preparação e deposição no alvo MALDI
- Dissolve-α CHCA em acetona, a fim de se obter uma solução saturada.
- Ao lado (sem qualquer pipeta) mergulhar uma ponta 10 ul (por exemplo GELoader Tip, Eppendorf) em solução saturada de α-CHCA: uma pequena quantidade da solução fluirá na ponta (devido a capilaridade).
- Toque muito rapidamente (ou seja, 1 segundo) o alvo MALDI com ponteira e depositar a solução de acetona α-CHCA no alvo MALDI. Isto irá constituir a fina camada de matriz, onde a proteínaamostra vai ser depositado. Tenta gerar um ponto de camada fina mais pequeno possível.
Nota: Ao utilizar SA como matriz, preparamos uma camada fina utilizando uma solução saturada de SA em acetona.
5. Preparação de Soluções: Natrix SA, α-CHCA, DHB e CHCA_DHB Mistura 25
- Prepara-se uma solução de 20 mg / ml SA em ACN, 0,1% de TFA (70:30, v / v).
- Prepare a 20 mg / ml solução α-CHCA em ACN e 5% de ácido fórmico (70:30, v / v) [denominado como "solução α-CHCA"].
- Prepara-se uma solução / DHB ml 20 mg em ACN e 0,1% de ácido trifluoroacético (TFA) (70:30, v / v) [denominado como "solução DHB"].
- Misture "solução α-CHCA" e "solução de DHB" na proporção de 1:1 (vol / vol), para obter a "solução CHCA_DHB".
Nota: Pode-se misturar "solução α-CHCA" e "solução DHB" em diferentes proporções, ao analisar as proteínas com massa inferior a 100 kDa. Para example uma proporção de 40:60 entre α-CHCA e DHB (vol / vol) produz uma melhor resolução, mas menor sensibilidade (ver discussão).
6. Deposição de amostra no alvo MALDI
- Depósito de 0,5 ul de amostra de proteína em camada fina α-CHCA previamente preparada. Imediatamente depois disso, adicionar 0,5 ml da solução de matriz (isto é, a solução ou o SA solução α-CHCA ou a "mistura CHCA_DHB"). Isto implica misturar a amostra e a matriz sobre o alvo.
Nota: Normalmente uma mistura da amostra de proteína com a matriz em uma proporção de 1:1. Esta relação é fundamental para a boa qualidade dos espectros de MALDI. Se você gostaria de testar diferentes concentrações das amostras, você pode usar a solução de ácido ACN_formic (descrito acima) para diluir a amostra.
Nota: Se você preferir, você pode misturar a amostra ea matriz em um tubo e, em seguida, depositar o misto "sample_matrix soluçãoção "na camada fina.
Nota: Sugerimos que você testar diferentes concentrações das amostras porque diluindo a amostra permite reduzir a interferência de contaminantes. Para diluir a amostra, você pode usar a solução de ácido ACN_formic (descrito acima) para diluir a amostra.
7. Calibrant Deposição
- Depósito 0,5 ul do padrão de calibração (por exemplo, "padrão de proteína II", Bruker Daltonics, Bremen), em seguida, adicionar 0,5 mL da solução de matriz.
Nota: Coloque o calibrador sobre a placa de MALDI ao lado dos pontos de amostragem de proteína (ver discussão).
8. MALDI Spectra Aquisição de Calibrant e amostras de proteínas
Uma vez que as amostras ea calibração são secas, você pode observar os "spots" sob um microscópio. Esta observação é opcional e pode ser interessante principalmente para os novatos. Para adquirir os espectros da amostra, insira ªe alvo no instrumento MALDI-TOF e escolher os parâmetros instrumentais adequadas que são diferentes para cada tipo de equipamento. Para obter o mais adequado set-up deve-se seguir as recomendações do fabricante. Como considerações gerais ao analisar proteínas intactas, deve-se utilizar o instrumento no modo linear (não no modo refletor, o que é apropriado para análises peptídeos). Normalmente utilizamos o nosso instrumento no modo positivo de lítio. Além disso, um parâmetro chave é a "extração de íons pulsada", que melhora a resolução instrumental 31. Em termos simples, a "extracção de iões pulsado" pode ser definida como uma pausa entre a geração de iões de amostra e o tempo em que os iões são acelerados para o detector. Ao analisar proteínas intactas, deve-se utilizar uma "extração de íons pulsada" (por exemplo, 500 nanossegundos) maior do que quando se observa peptídeos (por exemplo, 80 ns).
Escolha o intervalo adequado m / z, adquirir os espectros of a calibração, e calibrar o instrumento. Quando você adquirir o espectro de suas amostras, use a intensidade do laser apropriado (ver discussão).
Resultados
Analisou-se uma proteína intacta (região do cromossomo 1 manutenção proteína, CRM1; peso molecular: 123386 Da) usando duas matrizes diferentes, dois métodos de deposição, e a mesma intensidade do laser (Figura 1). SA e uma mistura CHCA_DHB foram utilizados como matrizes. A mistura produziu os espectros de massa de maior qualidade, em termos de relação sinal-ruído e de sensibilidade (Figuras 1A e B). Em particular, foi possível detectar 0,5 pmol de proteína depositada no alvo MALDI (Figura 1C). A mesma quantidade de proteína era apenas detectável utilizando SA (Figura 1D). Além disso, utilizando a mistura de matrizes, o método de escolha para a deposição de matriz é a abordagem "fina camada" (Figura 1A). Usando o método "seco gota", não se detectou qualquer proteína com uma massa superior a 100 kDa (dados não mostrados). Utilizando a mistura CHCA_DHB (Figuras 1A e 1C), se multiplicam carregada de iões do Protein foram observados, permitindo a determinação da massa com maior precisão porque a resolução de pico em instrumentos de MALDI-TOF axiais é inversamente proporcional à m / z 26.
Analisamos também monomérica beta-galactosidase (116.300 Da) (Figura 2). Os espectros CHCA_DHB eram melhores do que os espectros SA em termos de sensibilidade e resolução. Além disso, a presença de iões de cargas múltiplas (M + 2, M 3 e M 4 + +) permitiu confirmar a massa da proteína com maior precisão (Figuras 2A e 2C). Utilizando a abordagem de camada fina, foi comparado o desempenho da mistura CHCA_DHB, SA e α-CHCA sozinho para a análise de uma imunoglobulina, IgG (148500 Da) (Figura 3). Quando correlacionar os diferentes dados, sinais de proteína foram maiores e com melhor resolução no espectro CHCA_DHB. Para concluir, o uso da mistura CHCA_DHB e o método de deposição de camada fina demonstraram melhorias significativas na qualidade de espectros de massa de proteína intacta grande adquiridos utilizando um instrumento MALDI TOF.
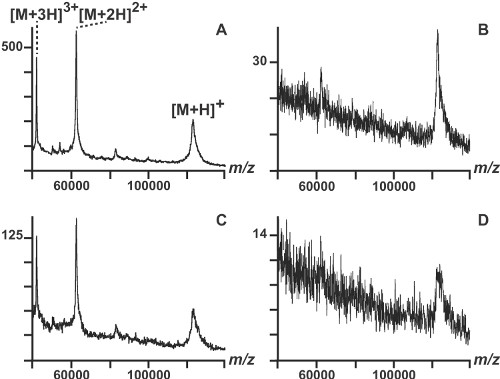
Figura 1. Análise MALDI-TOF da região cromossomo intacto manutenção uma proteína, (CRM1), peso molecular: 123.386 Da. A) 1 pmol de CRM1 foi analisada usando a mistura DHB_CHCA como matriz B) quantidade: 1. Pmole; matriz:. SA C) valor: 0,5 pmole; matriz: D) quantidade DHB_CHCA: 0,5 pmole; matriz:. SA. O sinal dos picos, expressa numa escala de intensidade de unidade arbitrária, é normalizado em relação ao valor máximo actual de cada espectro.
pg "src =" / files/ftp_upload/50635/50635fig2.jpg "/>
Figura 2. Análise MALDI-TOF de intacta beta-galactosidase (116.300 Da). A) quantidade: 20 pmol; DHB_CHCA matriz B) quantidade:. 20 pmol; matriz:. SA C) quantidade: 5 pmol; matriz: D) quantidade DHB_CHCA:. 5 pmol; matriz: SA.

Figura 3. Análise MALDI-TOF de uma imunoglobulina intacta, IgG (148.500 Da); montante:. 1,7 pmol Os espectros obtidos com a mistura CHCA_DHB eram muito mais intenso (máximo: 8200 unidade arbitrária, au) do que os espectros SA (máximo: 1.200 au) e espectros α-CHCA (máximo: 4500 au)
Discussão
Foi apresentado um protocolo de detalhe para adquirir espectros de massa de alta qualidade de grandes proteínas intactas com pesos moleculares maiores do que 100 kDa, utilizando um instrumento de MALDI-TOF. Um certo número de aspectos importantes relativos à preparação da amostra deve ser considerada com cuidado, a fim de melhorar a qualidade dos espectros de massa. Matrizes e solventes de alta pureza são de importância fundamental, porque todos os contaminantes presentes nas matrizes e solventes estão concentrados no alvo MALDI após evaporação do solvente. A fim de melhorar a pureza das matrizes MALDI é possível purificá-los usando métodos clássicos de recristalização (ver acima). Recomenda-se também a preparar as soluções recém-matrizes (pelo menos uma vez por semana) para melhorar a cristalização da matriz e para evitar a degradação da matriz.
A abordagem de deposição de matriz é muito crítico para a qualidade final dos espectros. Como descrito acima, o método de camada fina é o nosso métodof escolha 12. Além disso, nós otimizamos a abordagem para depositar a primeira camada. Quando a matriz é dissolvido em acetona para obter uma solução saturada, a propanona permite rápida evaporação do solvente, mas também faz com que o tamanho da primeira camada muito difícil de controlar. Sugerimos utilizar uma dica GELoader tão pouco pincel que você mergulha na solução matrix_acetone para obter um volume mínimo que você depositar rapidamente no alvo. O tamanho da primeira camada de algum modo controla o tamanho final da mancha de MALDI: um pequeno ponto (ou seja, 0,5-0,75 mm de diâmetro) é preferível, porque, neste caso, a concentração da amostra é maior do que no caso de uma grande mancha. A obtenção de um pequeno ponto difere da abordagem camada ultra-fina anteriormente proposto em um vídeo JOVE 15. Em Fenyo et al. A solução camada fina está espalhada por todo o alvo MALDI usando o lado de uma ponteira. Essa abordagem requer testar a fina camada que deverá satisfazer critérios específicos(Por exemplo, velocidade de evaporação do solvente). Se o critério não for atendido, o alvo MALDI deve ser lavado e uma nova camada fina deve estar preparado. Isso faz com que a preparação bastante trabalhoso para um novato. Além disso, a deposição da mistura de amostra / matriz na placa requer a aspiração do excesso de solvente, utilizando uma linha de vácuo e um passo de lavagem utilizando TFA 15 é necessário, tornando-o Fenyo et al. Preparação um pouco mais difícil do que a nossa abordagem.
A relação de volume entre a matriz e a amostra é muito importante para uma análise bem sucedida de proteínas intactas por MALDI-TOF. Depois de testar proporções diferentes, sugerimos usar uma proporção de 1:1. A matriz diferente: proporção da amostra poderia comprometer a qualidade do espectro, provavelmente devido à cristalização não homogêneo. A ordem na qual matriz e a amostra são depositadas também é crítico. Depois de depositar a camada fina da matriz, a amostra é depositada e imediatamente após (antes da SAMPle local torna-se seca) a mistura CHCA_DHB é adicionado. Este procedimento foi definido como "método sanduíche" 27, em que a amostra só é depositado entre duas camadas de matriz. Como alternativa, a solução CHCA_DHB e a amostra pode ser misturada no tubo de 0,5 ml e, em seguida, detectadas na fina camada de CHCA 12. Isto produz uma mancha com uma camada homogénea de cristais resultantes em espectros de alta qualidade. Quando a análise de proteínas com uma massa inferior a 100 kDa, a concentração de DHB pode ser aumentada (por exemplo, 60:40 DHB: CHCA), o que pode produzir mais nítida pico, mas poderão comprometer a sensibilidade de medição (isto é, picos de menor intensidade).
Para uma correcta calibração do instrumento, que é essencial para depositar os padrões de calibração muito perto dos pontos de amostragem, permitindo que se obtenha uma melhor precisão de massa. A "pseudo-processo de calibração interna" foi sugerido anteriormente 15: após a aquisição de um espectro de amostra, o i ponto de calibraçãos tiro do laser e do sinal de calibração são adicionados ao espectro da amostra. Isto permite-lhe calibrar internamente o espectro da amostra usando os picos de calibração.
A energia do laser devem ser também cuidadosamente controlado durante a aquisição espectros. Na maioria dos casos, mais elevada a energia aumenta a sensibilidade, mas diminui a resolução. Sugerimos utilizando o mínimo de energia possível, a laser, sem comprometer a sensibilidade da aquisição. Além disso, para melhorar a qualidade espectral, pode-se adquirir mais tiros em cada ponto de amostra. Normalmente, os mais tiros você adquirir (1000-2000 tiros), quanto maior a intensidade do sinal. Quando bater o ponto com o laser, é preciso encontrar a posição correta (ou seja, "sweet spot"), uma vez que a amostra não é homogeneamente distribuída. Você pode atirar na mesma área até que o sinal está aumentando, e, em seguida, tentar encontrar uma outra área que contém a amostra.
Em conclusão, o elevado grau de pureza de matrizes e solventos, os métodos utilizados para a amostra e deposição matrizes no alvo, a posição dos padrões utilizados para a calibração, a intensidade de energia do laser, o tempo de aquisição (número de disparos / spot) são fatores críticos que se deve ter em conta quando análise de proteínas intactas por MALDI-TOF MS.
Autor Contribuições
LS e EBE projetou os experimentos, realizados os experimentos de espectrometria de massa, analisou os dados, e escreveu o manuscrito.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos ao Dr. Christophe Masselon (iRTV, CEA, Grenoble) e membros da infecção viral e Grupo Câncer no IBS, para a sua avaliação crítica do manuscrito e para discussões úteis. Somos gratos a Cyril Dian para o presente tipo de proteína CRM1. Este trabalho científico teve lugar na facilidade de espectrometria de massa de Grenoble Centre Instruir (ISBG; UMS 3518 CNRS-CEA-UJF-EMBL). Foi apoiado financeiramente pela Infra-estrutura francesa para Iniciativa de Biologia Estrutural Integrado (Frisbi, ANR-10-InSb-05-02), GRAL (ANR-10-LabX-49-01) [no âmbito da Parceria Grenoble para Biologia Estrutural] e por o Centro Nacional Francês de Pesquisa Científica (CNRS).
Materiais
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Trizma hydrochloride | Sigma | T3253 | |
| Trizima | Sigma | T6066 | |
| Micro Bio-Spin 6 chromatography columns | Bio-Rad | 732-6221 | |
| Vivaspin 500 | Sartorius | VS0122 | various molecular weight cut off |
| Methanol | Fluka | 14262 | |
| Ethanol | Fluka | 02860 | |
| KIMTECH SCIENCE Precision Wipes Tissue Wipers | Kimberly Clark | 05511 | |
| α-cyano-4-hydroxycinnamic acid | Sigma Aldrich | C8982 | |
| Acetone | Sigma Aldrich | 650501 | |
| 2,5-dihydroxybenzoic acid | Fluka | 39319 | |
| GELoader Tips | Eppendorf | 0030 001.222 | |
| Acetonitrile | Sigma Aldrich | 34998 | |
| Formic acid | Fluka | 09676 | |
| Trifluoroacetic acid | Sigma Aldrich | 302031 | |
| Protein Standard II | Bruker Daltonics | 207234 | It contains Trypsinogen, Protein A, Serum Albumin-Bovine |
| Immunoglobulin G | ABSciex | GEN602151 | |
| [header] | |||
| EQUIPMENT | |||
| Water purifier PURELAB Ultra Analytic | ELGA LabWater | 89204-052 | |
| Autoflex MALDI-TOF instrument | Bruker Daltonics | ||
| MALDI-TOF target | Bruker Daltonics | ||
Referências
- Rupp, B. . Biomolecular Crystallography : Principles, Practice, and Application to Structural Biology. , (2010).
- Cohen, S. L., Chait, B. T. Mass spectrometry as a tool for protein crystallography. Annual review of biophysics and biomolecular structure. 30, 67-85 (2001).
- Chait, B. T. Mass spectrometry--a useful tool for the protein X-ray crystallographer and NMR spectroscopist. Structure. 2, 465-467 (1994).
- Gans, P., et al. Stereospecific isotopic labeling of methyl groups for NMR spectroscopic studies of high-molecular-weight proteins. Angew Chem Int Ed Engl. 49, 1958-1962 (2010).
- Wilm, M. Principles of electrospray ionization. Molecular & cellular proteomics : MCP. 10, M111 009407 (2011).
- Urban, P. L., Amantonico, A., Zenobi, R. Lab-on-a-plate: extending the functionality of MALDI-MS and LDI-MS targets. Mass spectrometry reviews. 30, 435-478 (2011).
- De Hoffmann, E., Stroobant, V. . Mass Spectrometry: Principles and Applications. , (2007).
- Karas, M., Hillenkamp, F. Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons. Analytical chemistry. 60, 2299-2301 (1988).
- Spengler, B., Cotter, R. J. Ultraviolet laser desorption/ionization mass spectrometry of proteins above 100,000 daltons by pulsed ion extraction time-of-flight analysis. Analytical chemistry. 62, 793-796 (1990).
- Beavis, R. C., Chait, B. T. High-accuracy molecular mass determination of proteins using matrix-assisted laser desorption mass spectrometry. Analytical chemistry. 62, 1836-1840 (1990).
- Hillenkamp, F., Karas, M., Beavis, R. C., Chait, B. T. Matrix-assisted laser desorption/ionization mass spectrometry of biopolymers. Analytical chemistry. 63, 1193A-1203A (1991).
- Beavis, R. C., Chait, B. T. Matrix-assisted laser desorption ionization mass-spectrometry of proteins. Methods in enzymology. 270, 519-551 (1996).
- Xiang, F., Beavis, R. C. A method to increase contaminant tolerance in protein matrix-assisted laser desorption/ionization by the fabrication of thin protein-doped polycrystalline films. Rapid Communications in Mass Spectrometry. 8, 199-204 (1994).
- Cadene, M., Chait, B. T. A robust, detergent-friendly method for mass spectrometric analysis of integral membrane proteins. Analytical chemistry. 72, 5655-5658 (2000).
- Fenyo, D., et al. MALDI sample preparation: the ultra thin layer method. J. Vis. Exp.. (3), e192 (2007).
- Gabant, G., Cadene, M. Mass spectrometry of full-length integral membrane proteins to define functionally relevant structural features. Methods. 46, 54-61 (2008).
- Hook, P., et al. Long range allosteric control of cytoplasmic dynein ATPase activity by the stalk and C-terminal domains. The Journal of biological chemistry. 280, 33045-33054 (2005).
- Vorm, O., Roepstorff, P. Peptide sequence information derived by partial acid hydrolysis and matrix-assisted laser desorption/ionization mass spectrometry. Biological mass spectrometry. 23, 734-740 (1994).
- Beavis, R. C., Chait, B. T. Cinnamic acid derivatives as matrices for ultraviolet laser desorption mass spectrometry of proteins. Rapid Communications in Mass Spectrometry. 3, 432-435 (1989).
- Madler, S., Boeri Erba, ., E, R., Zenobi, MALDI-ToF Mass Spectrometry for Studying Noncovalent Complexes of Biomolecules. Topics in current chemistry. , (2012).
- Wortmann, A., Pimenova, T., Alves, S., Zenobi, R. Investigation of the first shot phenomenon in MALDI mass spectrometry of protein complexes. The Analyst. 132, 199-207 (2007).
- Beavis, R. C., Chaudhary, T., Chait, B. T. Alpha-Cyano-4-hydroxycinnamic Acid as a Matrix for Matrix-assisted Laser Desorption Mass Spectrometry. Organic Mass Spectrometry. 27, 156-158 (1992).
- Liu, Z., Schey, K. L. Optimization of a MALDI TOF-TOF mass spectrometer for intact protein analysis. Journal of the American Society for Mass Spectrometry. 16, 482-490 (2005).
- Gorka, J., Bahr, U., Karas, M. Graphite supported preparation (GSP) of alpha-cyano-4-hydroxycinnamic acid (CHCA) for matrix-assisted laser desorption/ionization mass spectrometry (MALDI-MS) for peptides and proteins. Journal of the American Society for Mass Spectrometry. 23, 1949-1954 (2012).
- Laugesen, S., Roepstorff, P. Combination of two matrices results in improved performance of MALDI MS for peptide mass mapping and protein analysis. Journal of the American Society for Mass Spectrometry. 14, 992-1002 (2003).
- Sparkman, D. O. . Mass Spectrometry Desk Reference. , (2000).
- Kussmann, M., Roepstorff, P. Sample preparation techniques for peptides and proteins analyzed by MALDI-MS. Methods Mol Biol. 146, 405-424 (2000).
- Wang, R., Chait, B. T. High-accuracy mass measurement as a tool for studying proteins. Current opinion in biotechnology. 5, 77-84 (1994).
- Kirshenbaum, N., Michaelevski, I., Sharon, M. Analyzing large protein complexes by structural mass spectrometry. J. Vis. Exp. (40), e1954 (2010).
- Hernandez, H., Robinson, C. V. Determining the stoichiometry and interactions of macromolecular assemblies from mass spectrometry. Nature protocols. 2, 715-726 (2007).
- Vestal, M. L., Juhasz, P., Martin, S. A. Delayed extraction matrix-assisted laser desorption time-of-flight massspectrometry. Rapid Communications in Mass Spectrometry. 9, 1044-1050 (1995).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados