Method Article
Snapshot Genome-wide de Reguladores da cromatina e Estados em
Neste Artigo
Resumo
A questão de como os reguladores e os estados de cromatina cromatina afetar o genoma in vivo é essencial para a nossa compreensão de como o diagnóstico precoce decisões destino celular são feitas no embrião em desenvolvimento. Abordagem mais popular para investigar características de cromatina em um nível-se global definida aqui para embriões de Xenopus o chip-Seq-.
Resumo
The recruitment of chromatin regulators and the assignment of chromatin states to specific genomic loci are pivotal to cell fate decisions and tissue and organ formation during development. Determining the locations and levels of such chromatin features in vivo will provide valuable information about the spatio-temporal regulation of genomic elements, and will support aspirations to mimic embryonic tissue development in vitro. The most commonly used method for genome-wide and high-resolution profiling is chromatin immunoprecipitation followed by next-generation sequencing (ChIP-Seq). This protocol outlines how yolk-rich embryos such as those of the frog Xenopus can be processed for ChIP-Seq experiments, and it offers simple command lines for post-sequencing analysis. Because of the high efficiency with which the protocol extracts nuclei from formaldehyde-fixed tissue, the method allows easy upscaling to obtain enough ChIP material for genome-wide profiling. Our protocol has been used successfully to map various DNA-binding proteins such as transcription factors, signaling mediators, components of the transcription machinery, chromatin modifiers and post-translational histone modifications, and for this to be done at various stages of embryogenesis. Lastly, this protocol should be widely applicable to other model and non-model organisms as more and more genome assemblies become available.
Introdução
The first attempts to characterize protein-DNA interactions in vivo were reported about 30 years ago in an effort to understand RNA polymerase-mediated gene transcription in bacteria and in the fruit fly1,2. Since then, the use of immunoprecipitation to enrich distinct chromatin features (ChIP) has been widely adopted to capture binding events and chromatin states with high efficiency3. Subsequently, with the emergence of powerful microarray technologies, this method led to the characterization of genome-wide chromatin landscapes4. More recently, chromatin profiling has become even more comprehensive and high-resolution, because millions of co-immunoprecipitated DNA templates can now be sequenced in parallel and mapped to the genome (ChIP-Seq)5. As increasing numbers of genome assemblies are available, ChIP-Seq is an attractive approach to learn more about the genome regulation that underlies biological processes.
Here we provide a protocol to perform ChIP-Seq on yolk-rich embryos such as those of the frog Xenopus. Drafts of the genomes of both widely used Xenopus species—X. tropicalis and X. laevis—have now been released by the International Xenopus Genome Consortium6. The embryos of Xenopus species share many desirable features that facilitate and allow the interpretation of genome-wide chromatin studies, including the production of large numbers of high-quality embryos, the large size of the embryos themselves, and their external development. In addition, the embryos are amenable to classic and novel manipulations like cell lineage tracing, whole-mount in situ hybridisation, RNA overexpression, and TALEN/CRISPR-mediated knockout technology.
The following protocol builds on the work of Lee et al., Blythe et al. and Gentsch et al.7-9. Briefly, Xenopus embryos are formaldehyde-fixed at the developmental stage of interest to covalently bind (cross-link) proteins to their associated genomic DNA. After nuclear extraction, cross-linked chromatin is fragmented to focus subsequent sequencing on specific genomic binding or modification sites, and to minimize the contributions of flanking DNA sequences. Subsequently, the chromatin fragments are immunoprecipitated with a ChIP-grade antibody to enrich those containing the protein of interest. The co-immunoprecipitated DNA is stripped from the protein and purified before creating an indexed (paired-end) library for next-generation sequencing (NGS). At the end, simple command lines are offered for the post-sequencing analysis of ChIP-Seq data.
Protocolo
NOTA: Todos os trabalhos Xenopus cumpre integralmente com os animais do Reino Unido (Scientific Procedures) Act 1986, tal como aplicado pelo Instituto Nacional de Pesquisa Médica MRC.
1. Preparações
- Estimar o número de embriões necessários para o experimento chip (veja a discussão).
- Preparar as soluções seguintes que são armazenados à temperatura ambiente: 500 ml de 10x de Marc modificação de Ringer (MMR), sem EDTA, pH ajustado para 7,5 e esterilizado por autoclave (1 M de NaCl, KCl 20 mM, 20 mM de CaCl2, 10 mM de MgSO4, 50 mM de HEPES pH 7,5) a 10, 1 ml de tampão de eluição de SDS (50 mM Tris-HCl pH 8,0, EDTA 1 mM, SDS a 1%) e 1 ml de tampão de carregamento de DNA 5x (0,2% Orange G, 30% de glicerol, 60 mM de EDTA pH 8,0).
- Preparar as soluções seguintes que são armazenados a 4 ° C: 50 ml de tampão de HEG (50 mM HEPES-KOH, pH 7,5, EDTA 1 mM, pH 8,0, glicerol a 20%), 500 ml de tampão de extracção de E1 (50 mM de HEPES-KOH pH 7,5, 150 mM de NaCl, 1 mM de EDTA. 10% de glicerol, 0,5% de Igepal CA-630, 0,25% de Triton X-100), E2 (10 mM Tris-HCl pH 8,0, NaCl 150 mM, EDTA 1 mM, EGTA 0,5 mM), e E3 (10 mM Tris-HCl pH 8,0, NaCl 150 mM, EDTA 1 mM, 1% de Igepal CA-630, 0,25% de Na-desoxicolato a 0,1% SDS), 500 ml de tampão RIPA (50 mM de HEPES-KOH, pH 7,5, LiCl 500 mM, EDTA 1 mM , 1% de Igepal CA-630, 0,7% desoxicolato de Na) e 50 ml de tampão TEN (10 mM Tris-HCl pH 8,0, EDTA 1 mM, NaCl 150 mM).
- Pontuação e clipe de 15 ml tubo de poliestireno cônico na marca de 7 ml. Use este tubo para conter extractos nucleares submetidos a ultra-sons.
- Para a análise pós-sequenciamento, usar um computador operacional multicore Unix-style com pelo menos 8 GB de RAM e espaço livre em disco de 500 GB. Instale o seguinte software localmente a maioria dos quais são usados na linha de comando: FastQC, Illumina casava-1.8 filtro de qualidade, Bowtie 11, SAMtools 12, Homer 13, MACS2 14, IGV 15,16, Cluster3 17, Java TreeView, BLAST + 18, e b2g4pipe 19. Verifique as instruções de instalação e requisitos para compiladores e software de terceiros.
- Construir um índice Bowtie para alinhar curto NGS lê ao genoma Xenopus. Um exemplo é mostrado aqui para o X. tropicalis v7.1 genoma (Novembro de 2011), que pode ser baixado como um arquivo FASTA (genome.fa) do servidor ftp Xenbase (/ pub / Genomics / JGI). Mover o arquivo FASTA para o subdiretório índice de Bowtie.
- Use a seguinte linha de comando (aqui depois que o personagem prompt>) para gerar arquivos de índice xenTro7:
> Bowtie-build /path/to/bowtie/index/genome.fa xenTro7
> Export BOWTIE_INDEXES = / path / to / bowtie / index /
- Use a seguinte linha de comando (aqui depois que o personagem prompt>) para gerar arquivos de índice xenTro7:
- Baixe o arquivo de anotação gene (GTF) a partir do browser UCSC Genome ou o site espelho no servidor NIMR para versões mais recentes do genoma (genomes.nimr.mrc.ac.uk) através de ferramentas / Table Browser. Use o arquivo genoma FASTA eo arquivo GTF personalizar HOMER para Xenopus (por exemplo, X. tropicalis genoma v7.1, - nome xenTro7).
- Como alternativa, use pacotes HOMER pré-construídos em algumas versões antigas do genoma Xenopus.
> LoadGenome.pl -nome xenTro7 -org nulo -fasta /path/to/genome.fa -gtf path / to / genes.gtf
- Como alternativa, use pacotes HOMER pré-construídos em algumas versões antigas do genoma Xenopus.
- Crie uma trilha genoma (.genome arquivo) para o navegador do genoma IGV fazendo o upload de um arquivo FASTA indexada (genome.fa com arquivo genome.fa.fai na mesma pasta) eo arquivo de anotação (genes.gtf). Criar um índice genoma andaime (genome.fa.fai) para o genoma de Xenopus, como se segue:
> Samtools faidx /path/to/genome.fa - Use BLAST + para passar Gene Ontology (GO) termos de várias espécies de modelo (humano, rato, peixe-zebra, mosca da fruta e leveduras) sobre genes de Xenopus da seguinte forma:
- Baixe todas as sequências codificadoras (CDS) como um único arquivo FASTA (cds.fa) a partir do browser UCSC Genome via Tools/ Browser Mesa e atualização BLAST + com o banco de dados BLAST pré-formatado de proteínas não redundantes (NR):
> Update_blastdb.pl nr - Pesquisar humana (txid9606), rato (txid10090), peixe-zebra (txid7955), mosca da fruta (txid7227) e leveduras (txid4932) proteínas do site do NCBI via sua função de pesquisa avançada (http: http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?CMD=search&DB=journals. gov / proteína / avançado) e enviar a lista GI resultante (identificador de sequência) (sequence.gi.txt) para o computador.
- Atribuir genes de Xenopus para as proteínas mais semelhantes a partir da lista GI executando BLASTx com uma certa esperar (E) cutoff valor (aqui 10 -20). Certifique-se o formato de saída é xml (-outfmt 5 out blastx_results.xml). Faça uso de economia de tempo threads (-num_threads) que se correlacionam com o número de núcleos de computadores disponíveis.
> Blastx -db / path / to / nr -gilist /path/to/sequence.gi.txt -consulta / path / to / cds.fa -evalue 1e-20
-outfmt 5 out /path/to/blastx_results.xml -num_threads [# tópicos] - Abra o arquivo b2gPipe.properties da pasta b2g4pipe com um editor de texto e atualizar as propriedades de banco de dados para Dbacces.dbname = b2go_sep13 e Dbacces.dbhost = publicdb.blast2go.com. B2g4pipe executado a partir da pasta de instalação.
> Java Xmx1000m cp *: ext / *: es.blast2go.prog.B2GAnnotPipe -em /path/to/blastx_results.xml
resultados Check-out / xenTro7 prop b2gPipe.properties -v -annot
NOTA: extratos Este programa GO termos para cada BLAST bater e atribui-los a correspondentes genes de Xenopus (xenTro7.annot). As configurações de banco de dados mais atualizados podem ser encontrados em Ferramentas / Configurações Gerais / Configurações DataAccess da aplicação Blast2GO Java Web Start (ver 9.11.1).
- Baixe todas as sequências codificadoras (CDS) como um único arquivo FASTA (cds.fa) a partir do browser UCSC Genome via Tools/ Browser Mesa e atualização BLAST + com o banco de dados BLAST pré-formatado de proteínas não redundantes (NR):
2. Chromatin Cross-linking
- Fertilizar ovos de Xenopus, de-geleia e cultura embryos de acordo com protocolos padrão 20.
- Transferir os embriões dejellied (máximo de 2.500 X. laevis ou 10.000 X. tropicalis) no estágio de desenvolvimento de interesse para um 8 ml frasco da amostra de vidro com tampa e lave-as brevemente uma vez com 0.01x MMR.
- Fixar os embriões com 1% de formaldeído em 0.01x MMR (por exemplo, adicionar 225 ul de 36,5-38% de formaldeído a 8 ml 0.01x MMR) para 15 a 40 min à temperatura ambiente (ver discussão para o tempo de fixação e o número de embriões necessária por experiência CHIP).
NOTA: O formaldeído é corrosivo e altamente tóxico. É perigoso em caso de olho e contato com a pele, indigestão, e inalação. Use a coifa ao adicionar formaldeído para o frasco. - Pare a fixação por lavagem brevemente os embriões três vezes com MMR 0.01x frio. Não deixe OS Embry fazer contato com a superfície do líquido porque a tensão superficial faz com que eles se rompem.
- Alíquota os embriões em 2 ml microtubos no gelocom um máximo de 250 embriões por tubo, que ocupam um volume de aproximadamente 250 ul (X. tropicalis) ou 600 ul (X. laevis) antes de incubação.
- Pipeta afastado tanto MMR 0.01x possível. Passe para o próximo passo, se você continuar imediatamente com a secção 3.
- Equilibrar embriões em 250 ul de tampão de HEG frio. Uma vez que os embriões terem assentado no fundo do tubo de remover tanto líquido quanto possível e encaixe por congelação em azoto líquido. Armazenar a -80 ° C.
3. Chromatin Extração
NOTA: A seguir à extracção da cromatina reticulado a partir de embriões de Xenopus funciona mais eficientemente com os tempos de fixação indicados no passo 2.3 e de 50 a 80 X. tropicalis ou 25 a 40 X. laevis embriões por ml de tampão de extracção de E1, E2 e E3. Cada passo de extracção é repetida, de modo que é necessário o dobro do volume calculado de tampão. Para aumento de escala, usar vários microcentri 2 mltubos Fuge ou 50 ml tubos de centrífuga. Manter as amostras em gelo e tampões durante a extracção da cromatina.
- Suplemento de quantidades adequadas de tampões E1, E2 e E3, com DTT 1 mM e comprimidos inibidores de protease. Se realizar chip com um anticorpo específico para phospho, mais buffers suplemento com NaF mM 5 e 2 mM Na 3 VO 4.
- Homogeneizar embriões fixos com E1 por pipetagem cima e para baixo. Homogenatos de centrifugação numa centrífuga refrigerada (4 ° C) a 1000 xg durante 2 min (5 min ou no caso da utilização de tubos de 50 ml). Aspirar o sobrenadante e quaisquer lípidos ligados à parede.
- Ressuspender pelotas em E1. Manter as amostras em gelo durante 10 min. Sobrenadantes de centrífuga e descartar como no passo 3.2.
- Ressuspender em pelotas E2. Sobrenadantes de centrífuga e descartar como no passo 3.2.
- Repita o passo 3.4, mas manter as amostras em gelo durante 10 min antes da centrifugação.
- Ressuspender pelotas na E3. Manter as amostras em gelo durante pelo menos 10 min. Centrífuga e descartar supernatants como no passo 3.2.
NOTA: Nesta fase, os resuspensions deve tornar-se bastante transparente. Os detergentes aniónicos em E3 extrair núcleos reticulados, tornando mais das plaquetas da gema restantes solúveis. - Ressuspender piscina e pelotas de núcleos reticulados (normalmente de cor castanha a partir dos grânulos de pigmento insolubilizado) em um volume total de 1 ml de E3. Diluir a amostra com E3 para 2 ou 3 ml, se afigura-se muito viscosa e difícil de pipeta. Manter em gelo ou a 4 ° C para prosseguir com o passo 4 ou no mesmo dia seguinte. Encaixe por congelação em azoto líquido e armazenar a -80 ° C para uso posterior.
4. Chromatin Fragmentação
NOTA: A sonicação é usado tanto para solubilizar e ao cisalhamento da cromatina reticulado. Aqui estão os parâmetros para executar o Misonix Sonicator 3000 equipado com um microtip cônico 1/16 polegadas e sistema de som. Se estiver usando outros sonicators, siga as recomendações dos fabricantes para tosquiarreticulado cromatina ou usar 6 a 12 W durante 4 a 8 minutos no total.
- Transferir a amostra nuclear a partir do passo 3.7 em um tubo de custom-built para ultra-sons (passo 1.4). Manter a amostra refrigerada durante a sonicação, tendo o tubo ligado a um copo de plástico de 800 ml cheio com água gelada através de uma curta braçadeira termómetro.
- Colocar o recipiente sobre uma tomada de laboratório. Ajustar o macaco de modo que a micro-pipeta sonicador é submerso na amostra de cerca de dois terços do volume e profundidade centrado sem tocar na parede do tubo.
- Sonicar a amostra durante 7 minutos, no total, interrompido a cada 30 segundos com um mínimo de pausas. Definir poder de 1.0. Iniciar a sonicação e aumentar imediatamente o ajuste de potência (normalmente 2 a 4) para atingir uma leitura de 9 a 12 W. Pausa imediatamente se a amostra começa a espumar. Reposicionar tubo e reinicia quando a espuma desapareceu completamente.
- Transfira a cromatina cortado em pré-refrigerados 1,5 ml microtubos e rotação à velocidade máxima (> 15,000 xg) durante 5 min a 4 ° C.
- Transferir o sobrenadante para pré-arrefecida de 1,5 ml tubos de microcentrífuga. Recolha 50 ul de sobrenadante (contendo idealmente a cromatina de cerca de 400.000 ou mais núcleos) para visualizar o grau de fragmentação de cromatina (ponto 5). Use o restante do sobrenadante para Chip (secção 6).
- Armazenar as amostras a 4 ° C durante até um dia. Amostras Snap-congelantes como alíquotas (um por cada experimento ChIP) em azoto líquido durante a armazenagem a longo prazo a -80 ° C.
5. Imagem Chromatin Fragmentação
- Adicionar 50 ul de tampão de eluição de SDS, 4 ul de NaCl 5 M e 1 ml de proteinase K (20 ug / ul) e 50 ul do sobrenadante a partir do passo 4.6.
- Incubar durante 6 a 15 horas (O / N) num forno de hibridação ajustado para 65 ° C.
- Purifica-se o ADN utilizando um kit de purificação de PCR comercial. Se necessário, usar 3 M de acetato de sódio (pH 5,2) para ajustar o pH, tal como recomendado pelo fabricante. Eluir aADN duas vezes com 11 ul de tampão de eluição (10 mM Tris-HCl pH 8,5).
- Adicionar 0,4 uL de RNase A (20 ug / uL) e 5 uL de tampão de carregamento de DNA 5x antes de executar a totalidade da amostra ao lado de uma de 100 bp e uma escada de DNA de 1 kb de um gel de agarose a 1,4% por electroforese. Para melhores resultados, gel mancha com uma solução segura coloração ácido nucleico após eletroforese.
6. Chromatin Immunoprecipitation
NOTA: Nesta secção, usam baixa retenção de tubos de 1,5 ml de microcentrífuga e, pelo menos, 1 ml de solução tampão indicada por tubo para lavar as esferas magnéticas durante 5 min a 4 ° C. Antes de remover o tampão a partir das esferas, deixam os tubos no suporte magnético por 20 a 30 segundos de cada vez ou até que a solução se torne clara.
- Transferência de 10 a 30 ul de sobrenadante (cromatina cortado) a partir do passo 4.6 para um novo tubo para ser usado mais tarde como a amostra de entrada, o que corresponde a aproximadamente 1% da cromatina total utilizado para a apara. Armazenar a 4° C até amostras de chips estão prontos para reverter ligações cruzadas.
- Transferir a cromatina restante para um novo tubo. Para experiências ChIP-qPCR que requerem um controle anticorpo, distribuir volumes iguais de cromatina para dois tubos.
- Adicione o anticorpo ChIP-grade (ou o controle anticorpo correspondente) a cromatina. Como um guia, use cerca de 1 mg de anticorpo por um milhão de células que expressam o epitopo de interesse.
- Para estimar com mais precisão a quantidade de anticorpos necessária por experimento Chip, executar o mesmo chip com diversas quantidades de anticorpos (por exemplo, 0,25 mg, 1 mg e 2,5 mg) e comparar o rendimento em loci controle negativo e positivo por Chip-qPCR (veja seção 10). Como um controlo de anticorpo, utilizar soro normal de isotipo e as mesmas espécies como em animal hospedeiro ao anticorpo.
- Incubar num rotor (10 rpm) O / N a 4 ° C.
- Lavar uma quantidade adequada de pérolas magnéticas de anticorpo-compatível uma vez com E3 durante 5 min umat 4 ° C. Verificar as especificações do fabricante para a capacidade de ligação do anticorpo dos grânulos (normalmente 5 a 20 ul de pérolas de se ligar 1 ug de anticorpo de IgG).
- Adicionar pérolas lavadas para o anticorpo cromatina pré-incubadas. Além disso incubar num agitador rotativo (10 rpm) durante 4 horas.
- Wash miçangas quatro vezes dez vezes (CHIP-Seq) com tampão RIPA pré-refrigerados (CHIP-qPCR) ou, em seguida, uma vez com tampão pré-refrigerados TEN.
- Apenas realizar esta etapa se realizando um experimento ChIP-Seq.
- Ressuspender lavado beads em 50 ul de tampão TEN por tubo. Piscina todos os cordões de uma única experiência do chip ao transferi-los para um novo tubo. Utilizar a cremalheira magnética e refrigerado (4 ° C) centrifugação a 1.000 xg, para recolher os grânulos no fundo do tubo. Descartar tanto líquido quanto possível, sem perturbar o pelete de contas.
- Material de tira sai aos seus grânulos pela ressuspensão as contas em 50 a 100 mL de tampão de eluição SDS e vortexing-los continuamente com um termomisturador (1.000 rpm) durante 15 min a 65 ° C. Depois disso centrífuga à velocidade máxima (> 15.000 x g) durante 30 seg. Transferir o sobrenadante (ChIP eluato) para um novo tubo.
- Repita o passo anterior e combinar os eluatos de chip.
7. Chromatin reverso Cross-linking e purificação de DNA
- Adicionar tampão SDS a eluição suficiente da amostra de entrada (passo 6.1) para atingir o volume da amostra de chip, que é de 100 a 200 ul (6,10 passo). Suplemento ambas as amostras de chip e de entrada com 1/20 volume de 5 M NaCl. Incubar as amostras durante 6 a 15 horas (O / N) a 65 ° C num forno de hibridação.
- Adicionar 1 volume de tampão TE e ARNase A a 200 ng / ml. Incubar durante 1 hora a 37 ° C.
- Adicionar proteinase K a 200 jig / ml. Incubar durante 2 a 4 horas a 55 ° C.
- Purifica-se o ADN por extracção com fenol: clorofórmio: álcool isoamílico a extracção seguida por precipitação com etanol como descrito anteriormente 9. Para Chip-Seq, adicione 32ul de tampão de eluição (10 mM Tris-HCl, pH 8,5) para dissolver o sedimento de DNA. Deixar as amostras em gelo durante 30 minutos para assegurar que o DNA é completamente dissolvido.
NOTA: kits de purificação de PCR comercial tem menor recuperação de ADN mas são mais convenientes, e pode ser utilizada para amostras ChIP-qPCR. - Para Chip-Seq, determinar a concentração de 1 ml de chip e entrada de DNA usando métodos baseados em fluorometria. Seguir as instruções do fabricante e certificar-se de que a concentração de ADN cai dentro da gama de detecção fiável do fluorómetro.
8. ChIP-Seq Biblioteca Construção e Validação
NOTA: Os métodos atuais para preparação biblioteca DNA permitir a construção de bibliotecas de alta complexidade para NGS 1-2 ng. À custa de alguma complexidade, as bibliotecas podem ser feitas a partir de tão pouco como 50 pg de ADN (ver Tabela específico de Materiais / Equipamento). Use a mesma quantidade de DNA tanto para chip e biblioteca de entrada. Resumidamente, a MAKe indexado (emparelhado-end) bibliotecas chip-Seq, chip e DNA de entrada precisa ser reparado-end, ligado a adaptadores especiais (ver Tabela de materiais específicos / equipamentos) e PCR amplificado selecionado-size.
- Siga as orientações do fabricante para fazer bibliotecas chip-Seq. Veja a discussão para outras recomendações.
- Eluir cada biblioteca em 12 mL de tampão de eluição e determinar a concentração de 1 ml de cada biblioteca chip e entrada usando um fluorímetro. Esperar concentrações de 5 a 25 ng / ul. Considere reduzir o número de ciclos de PCR (menos do que 18 ciclos) se as concentrações são mais elevadas do que 25 ng / ul.
NOTA: quantificação exata é fundamental para alcançar os melhores resultados NGS. Bibliotecas com concentrações tão baixas como 1 ng / mL após 18 ciclos de PCR pode ser sequenciado, mas freqüentemente são de baixa complexidade. - Use 1 ml de biblioteca para determinar a distribuição de tamanho de fragmento e para verificar se há contaminação adaptador dímero (faixa em torno de 120 pb) por cà base de quadril electroforese capilar. Repetir a fase sólida purificação imobilização reversível com uma taxa de grânulos-a-amostra de 1: 1 (em vez de 1,6: 1), se a biblioteca contém dimeros de adaptador.
- Execute qPCR em validado loci controlo positivo e negativo (ver secção 10) para verificar se as tendências de enriquecimento DNA semelhantes são observados antes e após a preparação da biblioteca. Enviar de controle de qualidade bibliotecas aprovados para sequenciamento.
9. Análise Pós-sequenciação e Visualização de Dados
NOTA: Hoje em dia, NGS é muitas vezes realizado por in-house ou instalações de sequenciamento comerciais (ver discussão para algumas orientações NGS). A saída padrão são únicos ou múltiplos arquivos gzip comprimidos FASTQ (*) .fastq.gz armazenar milhões de sequenciamento lê. Normalmente, multiplexado lê já estão separados de acordo com seu índice e cada leitura contém um identificador de seqüência e uma pontuação de controle de qualidade (Phred + 33 para Illumina 1.8+) para cada base chamada. Esta abordagem aqui é apenas um dos muitos aspectos como analisar dados NGS. O leitor é convidado a verificar se qualquer uma das seguintes linhas de comando exigir mudanças como este campo está avançando rapidamente e as atualizações estão ocorrendo regularmente.
- Concatenar arquivos FASTQ gzip comprimidos e verificar a qualidade dos dados de sequenciamento utilizando o script FastQC. Executar isso e mais um dos seguintes comandos, tanto para chip e seqüenciamento de entrada de dados (Exemplos mostrados para ChIP) a partir do terminal:
> Cat /path/to/*.fastq.gz> ChIP.fastq.gz
> Fastqc ChIP.fastq.gz
NOTA: Os dados brutos do sequenciamento de sucesso de uma biblioteca ChIP-Seq de alta complexidade deve passar a maioria dos testes. Falhas são originários principalmente de pobres corridas de sequenciação e artefatos experimentais, tais como a amplificação PCR tendenciosa ou contaminação adaptador. Um certo grau de duplicação (redundância) é esperado como redundante lê pode representar bona fide enriquecimento DNA 21. No entanto, pode-se restringir as etiquetas de leitura mais tarde - a extremidade 5 'ou lê - a um por par de base para eliminar qualquer redundante lê sem afectar a sensibilidade de detecção de picos (passo 9.4) 21. - Dados pré-processo de sequenciamento para eliminar a contaminação do adaptador (homerTools guarnição -3 ) permitindo que um descasamento (-mis 1). Use as primeiras 20 bases do (indexados) adaptador (5 'para 3') proximal ao fragmento de ADN de interesse quando da ligação (mostrada por adaptador listados na Tabela de Materiais específicos / Equipamentos).
> Gzip -CD ChIP.fastq.gz | fastq_illumina_filter -vn> ChIP.fastq
> homerTools guarnição -3 GATCGGAAGAGCACACGTCT -mis um -min 36 ChIP.fastq
NOTA: A remoção de filtrado lê (N) só é necessária por padrão em arquivos FASTQ gerados pela Illumina 1.8. Omita o comando fastq_illumina_filter (ie,. '| Fastq_illumina_filter -vn')Se uma versão mais antiga do que 1,8 gerou o identificador de seqüência. - Alinhe pré-processado lê para o genoma de referência (xenTro7) usando gravata borboleta. Apenas manter mapeados exclusivamente lê (-m 1) usando as configurações padrão, ou seja, duas máximas descasamentos nos primeiros 28 bases e um Phred + 33 pontuação total qualidade de todos os desencontros por leitura de máxima 70. Relatório de alinhamento em formato SAM (-S) . Aumentar o número de megabytes por thread (--chunkmbs) se a memória pedaço está esgotado:
> Bowtie -m 1 -S -p [# tópicos] --chunkmbs [eg 200] xenTro7 ChIP.fastq.trimmed> ChIP.sam
NOTA: Bowtie espera Phred + 33 índices de qualidade, por padrão. Inclua a opção - phred64-quals se o arquivo FASTQ foi gerado com Phred + 64 índices de qualidade por Illumina com mais de 1.8. - Use dois comandos Homer para transformar o alinhamento (SAM) arquivo em um arquivo figurão (.bw):
> MakeTagDirectory Chip / -single -tbp 1 ChIP.sam
> MakeUCSCfile Chip /-bigWig / path / to / genome.fa.fai -fsize 1e20 -norm 1E7 -o ChIP.bw
NOTA: A transformação requer o índice de andaime (genome.fa.fai) do genoma de referência (passo 1.8). Aqui, o perfil está limitada a uma marcação por par de bases (-tbp 1) e normalizados para 10 milhões lê (1E7 -norm). figurão é um do formato preferido para visualizar dinamicamente os perfis de cromatina com um navegador genoma, tais como IGV (passo 9.12). - Determinar a distribuição de tags (CHIP -d /) em marcos genômicos (por exemplo, +/- 10 kb com 25 caixas de pb, -Si ze 20000 -hist 25), como o início da transcrição (tss, exemplo mostrado aqui) e terminação ( tts) locais. Execute o script perl annotatePeaks.pl HOMER com anotações de Xenopus xenTro7 (passo 1.7):
> AnnotatePeaks.pl tss xenTro7 -Tamanho 20000 -hist 25 -d Chip /> ChIP_tagDensity.tss - Encontrar picos significativos de enriquecimento de ADN entre ChIP(-t ChIP.sam) e de entrada (Input.sam -c) na X. tropicalis genoma usando MACS2 com um 1% FDR cutoff (q 0,01) e fragmentos de DNA (após sonicação) de 200 pb (--bw = 200) para a construção de modelos. Adicione o --broad bandeira para esta linha de comando se espera de uma ampla distribuição do recurso de cromatina de interesse, tais como marcas de histonas ou RNA polimerase.
> Macs2 callpeak -t ChIP.sam -c Input.sam -f SAM -n ChIP -g 1.4376e9 q 0,01 --bw = 200
NOTA: O tamanho efetivo do X. tropicalis montagem do genoma v7.1 é de cerca de 1.437.600 mil bp (1.4376e9 -g). MACS2 gera um arquivo de BED (ChIP_peaks.bed) alistar picos com suas localizações genômicas. - Compare vários perfis de cromatina em forma de um mapa de calor em cluster:
- Criar uma matriz de distribuição de tag de diretórios densidade tag de interesse (-d Chip / other_ChIP /) em picos MACS2 (por exemplo, +/- 1 kb com 25 caixas pb, -Tamanho 2000 -hist 25 -ghist):
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -Tamanho 2000 -hist 25 -ghist -d Chip / other_ChIP /> ChIP.matrix - Use a interface gráfica do usuário de Cluster3 para fazer o upload de arquivos ChIP.matrix e hierarquicamente agrupar as densidades de tag com base na distância euclidiana mínima para o centróide mais próximo. Abra o arquivo CTD gerada em Java TreeView para visualizar o clustering.
- Criar uma matriz de distribuição de tag de diretórios densidade tag de interesse (-d Chip / other_ChIP /) em picos MACS2 (por exemplo, +/- 1 kb com 25 caixas pb, -Tamanho 2000 -hist 25 -ghist):
- Encontre romance e motivos de ligação previamente conhecidas, que são enriquecidas em cimeiras de pico +/- 100 bp (-size 200). Use annotatePeaks.pl para mapear ocorrências motivo e para traçar densidades motivo:
> FindMotifsGenome.pl ChIP_peaks.bed xenTro7 ChIP_motifs / -Tamanho 200 -p [# tópicos]
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -m motif1.motif> ChIP_peaks.motif1
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -m motif1.motif -Tamanho 800 -hist 25> motif1.density
NOTA: O infe roteiro findMotifsGenome.plrs o enriquecimento a partir da comparação de selecionados aleatoriamente sequências fundo gene-centric. A novela motivo mais enriquecido é guardado sob motif1.motif no formato de uma matriz de pesos posição. O leitor é convidado a justificar estes resultados com outros métodos de novo descoberta motivo, como cisFinder 22 e 23 MEME. - Anotar picos calculando sua distância para o gene mais próximo e por determinação da sua normalizado contagem de leitura dentro de 400 janelas pb (-size 400):
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -size 400 -d Chip / input /> ChIP_peaks.genes - Resumir a saída usando o seguinte comando awk para listar o número (N), a localização ea contagem de leitura normalizada individual (R) e todos (LR) picos por (alvo) gene mais próximo.
> Awk 'BEGIN {FS =' t '} $ 7> = -5000 && $ 7 <= 1000
{N [$ 8] + = 1; R [$ 8] + = $ 9; LR [$ 8] = LR [$ 8] ', &# 39; 7 $ '(' $ 9 ')'} END {for (i em N)
{Print i ' t' N [i] ' t' R [i] ' t' substr (LR [i], 2)}} 'ChIP_peaks.genes> ChIP_peaks.summary
NOTA: O número após $ refere-se ao número da coluna, que podem precisar de modificar o ficheiro para caber ChIP_peaks.genes criado na etapa anterior. Este exemplar roteiro filtra os picos para além de 5 kb a montante e jusante 1 kb do TSS. 7 dólares, US $ 8 e US $ 9 referem-se a distância para o TSS, o identificador do gene ea contagem normalizado leitura per pico, respectivamente. - Realizar a análise de termos GO enriquecidas entre genes-alvo da seguinte forma:
- Inicie a interface gráfica do usuário de Blast2GO a partir da linha de comando via Java Web Start (javaws) 19.
> Javaws http://blast2go.com/webstart/blast2go1000.jnlp - Siga as instruções dos desenvolvedores para carregar a anotaçãoarquivar para genes de Xenopus (xenTro7.annot) como gerados no 1.9.4 e um arquivo simples de genes-alvo identificados. Certifique-se de que os mesmos identificadores de genes são usados em ambos os arquivos.
- Inicie a interface gráfica do usuário de Blast2GO a partir da linha de comando via Java Web Start (javaws) 19.
- Visualize os perfis de cromatina, acrescentando figurão (ChIP.bw, Input.bw) e arquivos de cama (ChIP_peaks.bed) em IGV como faixas. Dados do complemento com RNA-Seq rastreia se disponível para a mesma fase de desenvolvimento. Exceto resultados como uma sessão.
- Use a programação plataformas R (www.r-project.org) ou MATLAB para manipular mais e visualizar dados como gerado acima. Alternativamente, traçar pequenos conjuntos de dados com Excel.
10. ChIP-qPCR para Testing chip e Confirmando ChIP-Seq
- Utilizar a plataforma on-line Primer3 para conceber iniciadores vizinhas aproximadamente 100 pb de DNA a 60 ° C (T m) para ambos os loci de controlo positivo (pico-específicos) e negativos. Confirmar a especificidade do iniciador utilizando o in silico busca PCR implementado para o Browser UCSC Genome.
- Criar uma curva padrão de 8 pontos de diluições de três vezes a partir de entrada de cerca de 1% ou utilizar o 2 - Método ΔΔC (t), 8,24 para a quantificação do ADN de enriquecimento.
- Execute-PCR em tempo real em triplicatas técnicas para todas as amostras, ou seja, chip de controle e, se necessário, as amostras da curva padrão.
- Lote enriquecimento de ADN como a percentagem de ADN de entrada ou como uma proporção de ChIP contra amostra de controlo em ambos os loci de controlo positivas e negativas.
Resultados
Resultados equivalentes aos resultados aqui apresentados são esperados se o protocolo é bem executado e o anticorpo em uso é de qualidade ChIP-grade (ver discussão). Este protocolo permite a extracção de núcleos a partir de embriões de Xenopus fixado em formol e o corte eficiente de cromatina por ultra-sons (Figura 1A-C). Cromatina cortados mostra uma distribuição assimétrica dos fragmentos de DNA que variam normalmente de 100 a 1000 pb e com um pico entre 300 e 500 pb (Figura 1C). A 50 pg mínima de DNA imunoprecipitado é necessário para fazer com sucesso uma biblioteca ChIP-Seq emparelhado-end indexado com inserções de DNA de tamanho similar (Figura 2A). A biblioteca deve ser desprovido de dímeros do adaptador, que pode ser visto no electroferograma em aproximadamente 120 pb.
Após a sequenciação por síntese, pré-processado lê são mapeados para o genoma (Figura 2B, C). Em uma experiência bem-sucedida com X. embriões tropicalis, normalmente 50 a 70% de um único final lê de 40 pb podem ser mapeados exclusivamente para a montagem do genoma de v7.1 maximamente com dois desemparelhamentos. Enquanto entrada lê alinhar bastante uniforme em todo o genoma, o alinhamento dos ChIP lê resultados em enriquecimentos específico de cadeia que ladeiam o recurso de cromatina de interesse. Isto porque todos os fragmentos são sequenciados a partir da extremidade 5 '(Figura 2C) 25. Estendendo o alinhamento na direcção de leitura para um tamanho médio de fragmento produz perfis precisos para os dispositivos individuais de cromatina, tais como os eventos de ligação de factor de transcrição. Estas ocupações de ADN aparecem como picos quando visualizados em IGV ou qualquer outro navegador genoma compatível. Chamadores de pico como MACS são usados para determinar a localização destes picos (Figura 3A). Desta forma, dezenas de milhares de locais de ligação foram determinadas no X. tropicalis genoma para T-box fatores de transcrição como Vegt 26. ChIP-qPCR experimentos deve confirmar o enriquecimento local encontrou por Chip-Seq (Figura 3B).
Experimentos chip-Seq permitem explorar as características do genoma de recursos de cromatina. Por exemplo, o cálculo da distribuição de leitura sobre os elementos genômicos, como início da transcrição e locais de terminação pode destacar as preferências de ligação espaciais ao redor de genes (Figura 3C). Da mesma forma, um mapa de calor de distribuições de leitura em locais de pico é utilizado para comparar diferentes características de cromatina numa escala de todo o genoma (Figura 3D). Certos fatores de transcrição ligam DNA sequence-especificamente. De novo motivo análise do DNA genômico picos subjacentes pode recuperar esse tipo de informação, incluindo motivos co-enriquecido de potenciais co-fatores (Figura 3E). A grande maioria dos genes-alvo mostram ocupação DNA em um menor, em vez de nível superior (Figura 3F). Este recurso livre de escala parece ser bastante comum entre os trfatores anscription e sugere que apenas uma pequena fração dos genes-alvo são diretamente regulados com relevância biológica 27,28. A análise dos termos GO enriquecidos ou outros atributos, tais como a expressão diferencial de genes alvo pode revelar mais insights sobre a função biológica da característica da cromatina no embrião de Xenopus (Figura 3G).
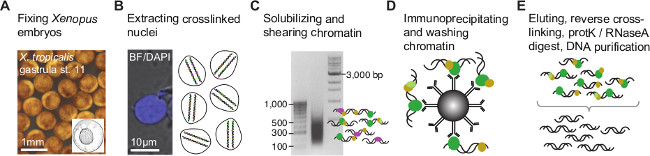
Figura 1. Chromatin procedimento immunoprecipitation para embriões de Xenopus. (A) Os embriões estão no estágio de desenvolvimento de interesse para ligar covalentemente (cross-link) quaisquer proteínas associadas com DNA genômico fixo-formaldeído. Após a extração nuclear (B), a cromatina reticulada é fragmentada para diminuir DNA genômico de ligação ou locais de modificação da cromatina, minimizando o DN flanqueandoUma sequência (C). Subsequentemente, os fragmentos de cromatina são imunoprecipitadas com um anticorpo ChIP grau de enriquecimento daqueles contendo o epitopo de interesse (D). O DNA co-imunoprecipitou é retirada da proteína e purificado (E) antes de criar a biblioteca de fragmentos de chip para NGS (Figura 2). Por favor, clique aqui para ver uma versão maior desta figura.

Figura 2. ChIP-Seq preparação biblioteca, sequenciamento-by-síntese, mapeamento e pico de chamada. (A) O electroferograma exibe uma biblioteca boa ChIP Seq com moldes de DNA de 250 a 450 pb. Estes modelos implica a inserção de ADN de interesse ladeado pelo universal (58 pb) e o (63 pb) do adaptador indexado. (B) Milhões de grupos, com cada grupo contendo modelos idênticos, são base seqüenciado por base, na presença de todos os quatro nucleotídeos que possuem reversível, fluorophore distinta e propriedades de terminação idênticos. As imagens fluorescentes são processados em tempo real, para ligar as bases correspondentes, os quais, em última análise são reunidos em lê. (C) Apenas lê que o mapa de forma única para o genoma de Xenopus são mantidos. Como todos os fragmentos são sequenciados a partir da extremidade 5 ', o mapeamento de ChIP lê resulta em picos específica de cadeia simples que flanqueiam o recurso de cromatina de interesse. Desse modo, os chamadores de pico detectar o enriquecimento que se origina de imunoprecipitação e estender a lê para um comprimento médio fragmento de localizar com precisão características de cromatina. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 3. Um exemplo de análise pós-sequenciamento e visualização de dados por meio do fator T-box zygotic transcrição Vegt (zVegT). Todas as lidas contagens mostradas aqui são normalizados para 10 milhões exclusivamente mapeados e não redundante lê. (A) Trecho do perfil de todo o genoma de zVegT obrigatório em X. tropicalis gastrula embriões (estágio 11-12,5 após Nieuwkoop e Faber 29). Cada pico, um engavetamento de prorrogado lê, representa um local de ligação. Estes picos são denominadas por MACS2 com uma taxa de detecção de falsas (FDR) de menos do que 1%. Cada gene MESP mostra zVegT muito proximal ea montante obrigatório, mas apenas Mespa e mespb são expressos por esse estágio (dados de RNA-Seq 30). (B) os níveis de ocupação DNA de zVegT conforme determinado pelo chip-qPCR em vários loci (incluindo um não -bound região 0,5 kb a montante do β -actina) confirm o enriquecimento específico encontrado por Chip-Seq. Comparar resultados para Mespa com pico chamado (barra vermelha) em (A). O nível de ocupação de ADN é visualizado como uma percentagem de entrada para ambos, o chip com o anticorpo Vegt (policlonal de coelho do isotipo IgG) e o chip com o anticorpo de controlo (IgG de coelho normal). As barras de erro reflectem o desvio padrão de duas réplicas biológicas. (C) METAGENE análise mostra ligação preferencial zVegT (tags binned mais de 25 pb) do promotor em relação a qualquer outra região genómica em torno e dentro dos corpos de genes. (D) mostra Heatmap k-significativo agrupadas níveis (k = 5) de ocupação DNA (tags finalmente resolvido mais de 25 pb) de zVegT e Smad2 / Smad3 (dados do chip-Seq 31) em relação a todas as regiões zVegT-bound em fase de gástrula. O heatmap é log 2 base e centrado em 5 tags por bp. (E) De novo a análise descobre o motivo canônica fator de transcrição T-box motivo de ligação em 38% dos zVegT-regiões com destino se a pontuação motivo subjacente é normalizado para uma taxa de detecção de 5% em sequências de fundo. O mapa de densidade mostra maior enriquecimento para o motivo T-box no centro de locais de ligação zVegT, ao passo que o motivo de ligação de Smad2 / Smad3 canónica é dificilmente enriquecido. (F) histograma mostra os níveis de ocupação de ADN zVegT, que são calculados para cada gene alvo de todos os picos (+/- 200 pb) entre 5 kb a montante [-]. e 1 kb a jusante [+] de sítios de início da transcrição correspondente (G) Top 300 genes com os maiores níveis de ocupação DNA dentro -5 kb e um kb são enriquecido para os processos biológicos de desenvolvimento embrionário precoce. Estes vão termos estão em linha com a função putativa de zVegT. A FDR é baseado em um teste exato de duas caudas de Fisher e corrigida para testes múltiplos. Por favor, clique aqui para ver uma versão maior desta figura.
Discussão
Nosso protocolo descreve como fazer e analisar os perfis de cromatina do genoma a partir de embriões de Xenopus. Ele abrange todas as etapas a partir de proteínas de ligação cruzada para loci endógena in vivo para o processamento de milhões de lê representando locais genômicas enriquecidas em silico. Desde um número crescente de projectos de genoma estão disponíveis, este protocolo deve ser aplicável a outros modelos e não-modelo organismos. A seção experimental mais importante, o que diferencia este protocolo para além de trabalhos anteriores 8,31,33,34, é o procedimento pós-fixação para extrair núcleos cruzada. Ele facilita a solubilização cromatina eficiente e de corte e fácil upscaling. Juntamente com as eficiências melhoradas de preparação da biblioteca este protocolo permite a construção de bibliotecas de ChIP-SEQ de alta complexidade da metade a dois milhões de células que expressam o epítopo associado cromatina de interesse. Para as experiências de ChIP-qPCR, alguns dez mil destas células são normalmente suficientepara verificar se há enriquecimento DNA em talvez seis loci genômicos distintas. Estes números são estimativas conservadoras, mas podem variar dependendo do nível de expressão da proteína, a qualidade do anticorpo, eficiência, e acessibilidade epitopo de ligação cruzada. Como um guia, um único embrião de Xenopus contém cerca de 4000 células na fase mid-blástula (8.5, depois de Nieuwkoop e Faber 29), 40.000 células na fase de gástrula tardia (12) e 100.000 células na fase tailbud precoce (20).
O tempo de fixação exata para immunoprecipitation eficiente precisa ser determinada empiricamente por Chip-qPCR (seção 10). Em geral, os tempos de fixação mais longos são necessários se o experimento envolve X. laevis embriões, primeiros estágios de desenvolvimento, e fracos (ou indiretas) propriedades de ligação de DNA. No entanto, não é recomendável que fixa embriões Xenopus mais de 40 min, ou o processamento de mais embriões do que o indicado (seção 3), com cromatina de corte torna-se menos eficiente. É importante nãopara usar qualquer glicina após a fixação como essa etapa comum para matar formaldeído pode fazer extração nuclear a partir de embriões de gema-rico muito difíceis. Actualmente, a razão para isto não é conhecido. É concebível que o aducto de formaldeído-glicina reage ainda com N-terminal de amino ou grupos de resíduos de arginina 35.
O anticorpo é a chave para qualquer experimento chip e controles suficientes precisam ser realizados para mostrar sua especificidade para o epitopo de interesse (ver orientações por Landt et al. 36). Se nenhum anticorpo ChIP grau está disponível, a introdução das proteínas de fusão marcadas com epítopo correspondente pode ser uma alternativa legítima como estas proteínas endógenas podem ocupar locais de ligação 37. Neste caso, os embriões não injectados são melhores para utilizar como controlo negativo, em vez de um chip com soro não-específico. Esta estratégia pode também ser aplicado se a proteína de interesse é expressa em níveis baixos, resultando em fraca recuperação de enriDNA Ched.
Como para fazer bibliotecas ChIP-Seq, devido à baixa quantidade de ADN a ser utilizado, é recomendável optar por procedimentos que reduzam o número de etapas de limpeza e de combinar reacções de manter qualquer perda de ADN a um mínimo. Os adaptadores e os iniciadores têm de ser compatíveis com a sequenciação múltipla e a plataforma NGS (ver Tabela específico de Materiais / Equipamento). Se utilizando Y-adaptadores (contendo braços longos de cadeia simples), é crítico para pré-amplificar a biblioteca de três a cinco rondas de PCR antes das inserções de ADN-seleccionando tamanho (ex., De 100 a 300 pb) por electroforese em gel. Extremidades de cadeia simples causar fragmentos de DNA para migrar de forma heterogênea. Ensaio executado com diversas quantidades de ADN de entrada (por exemplo, 0,1, 0,5, 1, 2, 5, 10 e 20 ng) são recomendados para determinar o número total de ciclos de PCR (inferior ou igual a 18 ciclos) necessária para fazer um tamanho -Selecionadas biblioteca de 100 a 200 ng. A redução do número de ciclos de PCR torna a sequenciação de redundant lê menos provável. Fase sólida contas de imobilização reversíveis são bons limpeza reagentes para se recuperar de forma eficiente o DNA de interesse e de forma confiável remova todas as placas livres e dímeros de ligação e reações de PCR.
Em termos de número, tipo e comprimento de leituras, em torno 20-30000000 single-end lê de 36 bp é suficiente para a maioria dos experimentos chip-Seq para cobrir todo o genoma Xenopus com profundidade suficiente. As máquinas NGS mais prevalentes são rotineiramente capaz de atender a esses critérios. No entanto, ela pode ser benéfica para aumentar o número de leituras, se uma ampla distribuição de leituras é esperado, conforme observado com modificações de histonas, em vez de picos agudos. Para muitas experiências ChIP-Seq, 4-5 bibliotecas diferente indexados podem ser combinadas e sequenciadas, um fluxo de pista de células utilizando uma máquina de NGS de alto desempenho. Algumas vezes também é conveniente para aumentar o comprimento e a sequência de leitura ambas as extremidades do molde de ADN (emparelhado-final) para aumentar mappability when análise cromatina dentro de regiões genômicas repetitivas.
Este protocolo foi aplicado com sucesso a uma grande variedade de características de cromatina, tais como factores de transcrição, mediadores de sinalização e histonas modificações pós-traducionais. No entanto, os embriões adquirir um crescente grau de heterogeneidade celular como elas se desenvolvem e perfis de cromatina se tornam mais difíceis de interpretar. Passos promissores têm sido feitos em Arabidopsis e Drosophila a paisagens cromatina tecidos especificamente perfil por meio da extração de tipo específico de células de núcleos 38,39. Nosso protocolo inclui uma etapa de extração nuclear, o que poderia abrir caminho para tecido-específicas ChIP-Seq em outros embriões.
Divulgações
The authors declare that they have no competing financial interests.
Agradecimentos
We thank Chris Benner for implementing the X. tropicalis genome (xenTro2, xenTro2r) into HOMER and the Gilchrist lab for discussions on post-sequencing analysis. I.P. assisted the GO term analysis. G.E.G and J.C.S. were supported by the Wellcome Trust and are now supported by the Medical Research Council (program number U117597140).
Materiais
| Name | Company | Catalog Number | Comments |
| 1/16 inch tapered microtip | Qsonica | 4417 | This microtip is compatible with Sonicator 3000 from Misonix and Q500/700 from Qsonica. |
| 8 ml glass sample vial with cap | Wheaton | 224884 | 8 ml clear glass sample vials for aqueous samples with 15-425 size phenolic rubber-lined screw caps. |
| Adaptor | e.g., IDT or Sigma | NA | TruSeq universal adaptor,
AATGATACGGCGACCACCGAG ATCTACACTCTTTCCCTACAC GACGCTCTTCCGATC*T. TruSeq indexed adaptor, P-GATCGGAAGAGCACACGTC TGAACTCCAGTCAC ‐NNNNNN‐ ATCTCGTATGCCGTCT TCTGCTT*G. *, phosphorothioate bondphosphate group at 5' end. NNNNNN, index (see TruSeq ChIP Sample Preparation Guide for DNA sequence). Order adaptors HPLC purified. Adaptors can be prepared by combining equimolar amounts (each 100 µM) of the universal and the indexed adaptor and cooling them down slowly from 95 °C to room temperature. Use 1.5 pmol per ng of input DNA. Store at -20 °C. |
| b2g4pipe (software) | Blast2GO | non-commercial | http://www.blast2go.com/data/blast2go/b2g4pipe_v2.5.zip |
| BLAST+ (software) | Camacho et al. | non-commercial | http://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastDocs& DOC_TYPE=Download |
| Bowtie (software) | Langmead et al. | non-commercial | http://bowtie-bio.sourceforge.net/index.shtml |
| cisFinder (software) | Sharov et al. | non-commercial | http://lgsun.grc.nia.nih.gov/CisFinder/ |
| Chip for capillary electrophoresis | Agilent Technologies | 5067-1504 | Load this chip with 1 µl DNA for library quality control. Store at 4 °C. |
| Chip-based capillary electrophoresis system | Agilent Technologies | G2940CA | The Agilent 2100 BioAnalyzer is used to check the quality of ChIP-Seq libraries. Keep reagents at 4 °C. |
| ChIP-Seq library preparation kit (KAPA Hyper Prep Kit) | Kapa Biosystems | KK8504 | Kit contains KAPA end repair and A-tailing enzyme mix, end Repair and A-tailing buffer, DNA ligase, ligation buffer, KAPA HiFi HotStart ReadyMix (2X), and KAPA library amplification primer mix (10X) (see also PCR primers). Adaptors are not included. Store at -20 °C. |
| ChIP-Seq library preparation kit (alternative, ThruPLEX-FD Prep Kit) | Rubicon Genomics | R40048 | Kit uses their own stem-loop adaptors and primers. This kit eliminates intermediate purification steps and is as sensitive as the KAPA Hyper Prep Kit. Store at -20 °C. |
| Cluster3 (software) | de Hoon et al. | non-commercial | http://bonsai.hgc.jp/~mdehoon/software/cluster |
| FastQC (software) | Simon Andrews | non-commercial | http://www.bioinformatics.babraham.ac.uk/projects/fastqc |
| Fluorometer | life technologies | Q32866 | Qubit 2.0 Fluorometer |
| Fluorometer reagents | life technologies | Q32851 | The kit provides concentrated assay reagent, dilution buffer, and pre-diluted DNA standards for the Qubit fluorometer. Store DNA standards at 4 °C, buffer and dye at room temperature. |
| Formaldehyde | Sigma | F8775-4X25ML | Formaldehyde solution, for molecular biology, 36.5-38% in H2O, stabilised with 10-15% methanol. Store at room temperature. CAUTION: Formaldehyde is corrosive and highly toxic. |
| Gel (E-Gel EX agarose , 2%) | life technologies | G4010 | Pre-cast gel with 11 wells, openable format. Leave one lane between ladder and library empty to avoid cross-contamination. Store gels at room temperature. |
| Gel electrophoresis system | life technologies | G6465 | E-Gel iBase and E-Gel Safe Imager combo kit for size-selecting ChIP-Seq libraries. |
| Gel extraction kit | Qiagen | 28706 | Store all reagents at room temperature. Use 500 µl of QG buffer per 100 mg of 2% agarose gel slice to extract DNA. Use MinElute columns (from MinElute PCR purification kit) to elute DNA twice. |
| HOMER (software) | Chris Benner | non-commercial | http://homer.salk.edu/homer/index.html |
| Hybridization oven | Techne | FHB1D | Hybridizer HB-1D |
| IGV (software) | Robinson et al. | non-commercial | http://www.broadinstitute.org/igv/home |
| Illumina CASAVA-1.8 quality filter (software) | Assaf Gordon | non-commercial | http://cancan.cshl.edu/labmembers/gordon/fastq_illumina_filter |
| Java TreeView (software) | Alok Saldanha | non-commercial | http://jtreeview.sourceforge.net |
| Laboratory jack | Edu-Lab | CH0642 | This jack is used to elevate sample in sound enclosure for sonication. |
| Ladder, 100 bp | New England BioLabs | N3231 | Keep 1x solution at room temperature. Store stock at -20 °C. |
| Ladder, 1 kb | New England BioLabs | N3232 | Keep 1x solution at room temperature. Store stock at -20 °C. |
| Low-retention 1.5-ml microcentrifuge tubes | life technologies | AM12450 | nonstick, RNase-free microfuge tubes, 1.5 ml |
| MACS2 (software) | Tao Liu | non-commercial | https://github.com/taoliu/MACS |
| Magnetic beads | life technologies | 11201D | These Dynabeads are superparamagnetic beads with affinity purified polyclonal sheep anti-mouse IgG covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 11203D | These Dynabeads are superparamagnetic beads with affinity purified polyclonal sheep anti-rabbit IgG covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 10001D | These Dynabeads are superparamagnetic beads with recombinant protein A covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 10003D | These Dynabeads are superparamagnetic beads with recombinant protein G covalently bound to the bead surface. Store at 4 °C. |
| Magnetic rack | life technologies | 12321D | DynaMag-2 magnet |
| MEME | Bailey et al. | non-commercial | http://meme.nbcr.net/meme/ |
| Na3VO4 | New England BioLabs | P0758 | Sodium orthovanadate (100 mM) is a commonly used general inhibitor for protein phosphotyrosyl phosphatases. Store at -20 °C. |
| NaF | New England BioLabs | P0759 | Sodium fluoride (500 mM) is commonly used as general inhibitor of phosphoseryl and phosphothreonyl phosphatases. Store at -20 °C. |
| NGS machine | Illumina | SY-301-1301 | Genome Analyzer IIx |
| NGS machine (high performance) | Illumina | SY-401-2501 | HiSeq |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2028 | Use as control for goat polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2025 | Use as control for mouse polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2027 | Use as control for rabbit polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Nucleic acid staining solution | iNtRON | 21141 | Use RedSafe nucleic acid staining solution at 1:50,000. Store at room temperature. |
| Orange G | Sigma | O3756-25G | 1-Phenylazo-2-naphthol-6,8-disulfonic acid disodium salt. Store at 4 °C. |
| PCR primers | e.g., IDT or Sigma | Primers to enrich adaptor-ligated DNA fragments by PCR: AATGATACGGCGACCACCGA*G and CAAGCAGAAGACGGCATACGA*G, phosphorothioate bond. Primers designed by Ethan Ford. Combine primers at 5 µM each. Use 5 µl in a 50 µl PCR reaction. Store at -20 °C. | |
| MinElute PCR purification kit | Qiagen | 28006 | for purification of ChIP-qPCR and shearing test samples. Store MinElute spin columns at 4 °C, all other buffers and collection tubes at room temperature. |
| Phenol:chloroform:isoamyl alcohol (25:24:1, pH 7.9) | life technologies | AM9730 | Phenol:Chloroform:IAA (25:24:1) is premixed and supplied at pH 6.6. Use provided Tris alkaline buffer to raise pH to 7.9. Store at 4 °C. CAUTION: phenol:chloroform:isoamyl alcohol is corrosive, highly toxic and combustible. |
| Primer3 (software) | Steve Rozen & Helen Skaletsky | non-commercial | http://biotools.umassmed.edu/bioapps/primer3_www.cgi |
| Protease inhibitor tablets | Roche | 11836170001 | cOmplete, Mini, EDTA-free. Use 1 tablet per 10 ml. Store at 4 °C. |
| Protease inhibitor tablets | Roche | 11873580001 | cOmplete, EDTA-free. Use 1 tablet per 50 ml. Store at 4 °C. |
| Proteinase K | life technologies | AM2548 | proteinase K solution (20 µg/µl). Store at -20 °C. |
| RNase A | life technologies | 12091-039 | RNase A (20 µg/µl). Store at room temperature. |
| Rotator | Stuart | SB3 | Rotator SB3 |
| SAMtools (software) | Li et al. | non-commercial | http://samtools.sourceforge.neta |
| Solid phase reversible immobilisation beads | Beckman Coulter | A63882 | The Agencourt AMPure XP beads are used to minimise adaptor dimer contamination in ChIP-Seq libraries. Store at 4 °C. |
| Sonicator 3000 | Misonix/Qsonica | Newer models are now available. Q125, Q500 or Q700 are all suitable for shearing crosslinked chromatin. | |
| Sound enclosure | Misonix/Qsonica | optional: follow the manufacturer's recommendation to obtain the correct sound enclosure. | |
| Thermomixer | eppendorf | 22670000 | Thermomixer for 24 x 1.5 mL tubes. Precise temperature control from 4 °C above room temperature to 99 °C. |
Referências
- Gilmour, D. S., Lis, J. T. Detecting protein-DNA interactions in vivo: distribution of RNA polymerase on specific bacterial genes. Proc Natl Acad Sci U S A. 81 (14), 4275-4279 (1984).
- Gilmour, D. S., Lis, J. T. In vivo interactions of RNA polymerase II with genes of Drosophila melanogaster. Mol Cell Biol. 5 (8), 2009-2018 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Ren, B., et al. Genome-wide location and function of DNA binding proteins. Science. 290 (5500), 2306-2309 (2000).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Hellsten, U., et al. The genome of the Western clawed frog Xenopus tropicalis. Science. 328 (5978), 633-636 (2010).
- Lee, T. I., Johnstone, S. E., Young, R. A. Chromatin immunoprecipitation and microarray-based analysis of protein location. Nature Protocols. 1 (2), 729-748 (2006).
- Blythe, S. A., Reid, C. D., Kessler, D. S., Klein, P. S. Chromatin immunoprecipitation in early Xenopus laevis embryos. Dev Dyn. 238 (6), 1422-1432 (2009).
- Gentsch, G. E., Smith, J. C. Investigating physical chromatin associations across the Xenopus genome by chromatin immunoprecipitation. Cold Spring Harb Protoc. 2014 (5), (2014).
- Ubbels, G. A., Hara, K., Koster, C. H., Kirschner, M. W. Evidence for a functional role of the cytoskeleton in determination of the dorsoventral axis in Xenopus laevis eggs. J Embryol Exp Morphol. 77, 15-37 (1983).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Mol Cell. 38 (4), 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9 (9), R137 (2008).
- Robinson, J. T., et al. Integrative genomics viewer. Nat Biotechnol. 29 (1), 24-26 (2011).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Brief Bioinform. 14 (2), 178-192 (2013).
- Imoto, S., Nolan, J., Bioinformatics Miyano, S. . 20 (9), 1453-1454 (2004).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Sive, H., Grainger, R., Harland, R. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Chen, Y., et al. Systematic evaluation of factors influencing ChIP-seq fidelity. Nat Methods. 9 (6), 609-614 (2012).
- Sharov, A. A., Ko, M. S. H. Exhaustive search for over-represented DNA sequence motifs with CisFinder. DNA Res. 16 (5), 261-273 (2009).
- Bailey, T. L., et al. MEME SUITE: tools for motif discovery and searching. Nucl Acids Res. 37 (2), W202-W208 (2009).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10 (10), 669-680 (2009).
- Gentsch, G. E., et al. In vivo T-box transcription factor profiling reveals joint regulation of embryonic neuromesodermal bipotency. Cell Rep. 4 (6), 1185-1196 (2013).
- Barabasi, A. L., Oltvai, Z. N. Network biology: understanding the cell's functional organization. Nat Rev Genet. 5 (2), 101-113 (2004).
- Biggin, M. D. Animal transcription networks as highly connected, quantitative continua. Dev Cell. 21 (4), 611-626 (2011).
- Nieuwkoop, P. D., Faber, J. . Normal table of Xenopus laevis (Daudin): a systematical and chronological survey of the development from the fertilized egg till the end of metamorphosis. , (1994).
- Akkers, R. C., et al. A hierarchy of H3K4me3 and H3K27me3 acquisition in spatial gene regulation in Xenopus embryos. Dev Cell. 17 (3), 425-434 (2009).
- Yoon, S. J., Wills, A. E., Chuong, E., Gupta, R., Baker, J. C. . HEB and E2A function as SMAD/FOXH1 cofactors. Genes Dev. 25 (15), 1654-1661 (2011).
- Jallow, Z., Jacobi, U. G., Weeks, D. L., Dawid, I. B., Veenstra, G. J. Specialized and redundant roles of TBP and a vertebrate-specific TBP paralog in embryonic gene regulation in Xenopus. Proc Natl Acad Sci U S A. 101 (37), 13525 (2004).
- Buchholz, D. R., Paul, B. D., Shi, Y. -. B. Gene-specific changes in promoter occupancy by thyroid hormone receptor during frog metamorphosis. Implications for developmental gene regulation. J Biol Chem. 280 (50), 41222-41228 (2005).
- Wills, A. E., Guptaa, R., Chuonga, E., Baker, J. C. Chromatin immunoprecipitation and deep sequencing in Xenopus tropicalis and Xenopus laevis. Methods. 66 (3), 410-421 (2014).
- Metz, B., et al. Identification of formaldehyde-induced modifications in proteins: reactions with model peptides. J Biol Chem. 279 (8), 6235-6243 (2004).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Res. 22 (9), 1813-1831 (2012).
- Mazzoni, E. O., et al. Embryonic stem cell-based mapping of developmental transcriptional programs. Nat Methods. 8 (12), 1056-1058 (2011).
- Deal, R. B., Henikoff, S. A simple method for gene expression and chromatin profiling of individual cell types within a tissue. Dev Cell. 18 (6), 1030-1040 (2010).
- Bonn, S., et al. Tissue-specific analysis of chromatin state identifies temporal signatures of enhancer activity during embryonic development. Nat Genet. 44 (2), 148-156 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados