Method Article
Detecção de alto rendimento de patógenos respiratórios em espécimes animais por Nanoscale PCR
Neste Artigo
Resumo
testes de alto rendimento de DNA e RNA patógenos base por nanoescala PCR é descrito usando um canino sindrômica e painel de PCR respiratória equina.
Resumo
escala nanoliter PCR em tempo real usa multiplexação espacial para permitir que vários ensaios para ser executado em paralelo em uma única chapa, sem os inconvenientes típicos de combinação de reações juntos. Nós projetamos e avaliou um painel com base nesse princípio para identificar rapidamente a presença de agentes de doenças comuns em cães e cavalos com doença respiratória aguda. Este manuscrito descreve um fluxo de trabalho PCR de diagnóstico nanoescala para preparação de amostras, amplificação e análise de sequências alvo de patógenos, com foco em procedimentos que são diferentes das reações escala microlitro. No painel respiratória apresentados, 18 ensaios foram, cada um configurado em triplicado, com capacidade para até 48 amostras por placa. Um fluxo de trabalho de extração e pré-amplificação universal foi otimizado para preparação de amostras de alto rendimento para acomodar vários matrizes e DNA e RNA patógenos base. Os dados representativos são apresentados para um alvo de ARN (influenza A matriz) e um alvo de ADN (herpesvírus equino1). A capacidade de testar com rapidez e precisão para um grupo abrangente, baseada em síndrome de patógenos é uma ferramenta valiosa para melhorar a eficiência e ergonomia de testes de diagnóstico e para o diagnóstico de doença respiratória aguda e gestão.
Introdução
Com a demanda para a detecção rápida e abrangente de múltiplos agentes em diagnósticos clínicos para seres humanos e animais, um único organismo métodos diagnósticos moleculares para detecção de patógenos são pesados a menos que usado para o grande número de amostras a ser testado para uma única doença. No contexto veterinário, metodologias de diagnóstico de elevado rendimento, são particularmente importantes devido à necessidade adicional para cobrir agentes patogénicos a partir de uma grande variedade de espécies. OneHealth abordagens para a gestão de doenças de origem alimentar e vigilância patógeno emergente são exemplos de necessidades de testes que incluem um número de bactérias, vírus (DNA ou RNA base), parasitas e fungos. O desafio de combinação de testes para vários analitos juntos (multiplexação), a fim de melhorar a eficiência do teste é uma possível perda de sensibilidade e um grande fardo para a otimização e validação de ensaios.
escala nanoliter em tempo real, reação em cadeia da polimerase (PCR) é um alternativo para a prática de multiplexagem que permite muitas reacções separadas para executar simultaneamente na mesma amostra 1. A plataforma OpenArray é uma aplicação deste princípio 2; que combina a tecnologia de microarray com PCR em tempo real. Com base no conceito de multiplexagem espacial, cada amostra é testada para um grande número de alvos em separado por meio de furos de passagem. Esta plataforma foi inicialmente utilizada com a ligação de ADN de cadeia dupla baseada cyanine química 2 e está agora disponível para a química à base de sonda utilizando sondas de extinção escuros 3. Esta plataforma tem sido principalmente utilizado para a caracterização genética em seres humanos e animais 4 5. Recentemente, foi adaptado para a detecção de ADN e ARN agentes patogénicos contidos no sangue por Grigorenko et ai. 6, utilizando um procedimento em dois passos de transcrição reversa / amplificação pré-(pré- amplificador).
Descrevemos aqui um procedimento baseado em pré-amplificador de um passo para a detecção de ambos os tipos de patógenos no sec respiratóriaretions e uma variedade de outros tipos de amostras que podem ser realizados inteiramente em um dia de trabalho normal. Após a conclusão do pré-amplificador de um-passo, as amostras são transferidas para uma placa de 384 poços, que é o formato aceite pelo sistema de manuseamento de líquidos automatizado que vem com este nanoescala plataforma de PCR. O sistema desenha a mistura principal e a amostra através da superfície de até quatro placas (192) amostras e controlos de cada vez. Na sequência deste processo de carregamento, nós descrevemos como amostras são amplificados e analisados utilizando uma planilha macro que resume a média de 3 repetições técnicos para cada combinação de amostra / target em uma tabela que se encaixa em uma página impressa.
Um método comum para a análise de ADN extraído e / ou de ARN a partir de amostras usando esta plataforma foi estabelecida. Uma grande variedade de amostras foram extraídas, sujeitos a transcrição reversa, e pré-amplificado num formato de 96 poços, minimizando o potencial de erros. amostras respiratórias testadas incluíram swab nasal, compressas faringe profundas,lavagens trans-traqueal, lavado broncoalveolar e tecido pulmonar. Uma vez que alguns dos agentes testados podem também estar presentes no sangue periférico ou fezes, que incorporou os tipos de amostras para o processo. Este fluxo de trabalho simplificado ajuda a economizar tempo e recursos em relação à execução experimentos em várias placas, permitindo testes eficiente através da detecção do gene patógeno e toxina à base de painel no lugar do teste individual. O painel respiratória demonstrado aqui teve 18 ensaios de cada um impresso em triplicado através de furos, com capacidade para até 48 amostras por placa. Os patógenos equinos detectados incluídos adenovírus eqüino 1 e 2, vírus da arterite equina, vírus da rinite equina A e B, vírus de herpes equina tipos (MAT) 1 e 4, e Streptococcus equi. Os agentes patogénicos caninos incluídos coronavírus canino respiratória (betacoronavirus), vírus da cinomose canina, adenovírus canino, vírus parainfluenza canina, pneumovírus canino, Bordetella bronchiseptica e Mycoplasma cynos. UMAUm ensaio universal da gripe e um controlo interno (MS2 ARN fago) também foram incluídos.
Protocolo
Não há seres humanos ou animais experimentais foram utilizados para o desenvolvimento deste protocolo. Os controlos foram gerados por purificação de fragmentos amplificados confirmou-sequência e transcrição in vitro para alvos de ARN. As amostras clínicas foram submetidos a testes de diagnóstico de rotina para o Centro de Saúde de diagnóstico Cornell Animal.
Projeto 1. Placa
- Use um software de desenho de primers para verificar se cada ensaio em conformidade com em tempo real as condições de ciclismo PCR à base de sonda padrão com 60 ° C de recozimento usando a ferramenta de teste de sonda de primer (selecionar a sonda quantificação ajuste com parâmetros padrão).
NOTA: O alvo RNA representante utilizado foi adaptado da gripe universal Um ensaio de matriz alvo publicada por Shu et al 7.. Os iniciadores e sonda são as seguintes: Iniciador directo: GACCRATCCTGTCACCTCTGAC, Reverse Primer: AGGGCATTYTGGACAAAKCGTCTA, Probe: Fam-TGCAGTCCTCGCTCACTGGGCACG-QSY. O ensaio DNA representante usado aqui foi adaptada frosou o método de detecção eqüino tipo de herpesvírus 1 (EHV-1), publicado por Elia et al. 8. Os iniciadores e sonda são as seguintes: Iniciador directo: GCTCTCAGGTTTTACGACATC, Reverse Primer: CTTTACCCAGGCCCTTGAAA, Probe: FAM-TCAACGTGGACAATACCGCAGTGATTAT-QSY. - Encomendar placas em nanoescala de amplificação por PCR na configuração desejada.
NOTA: Para este estudo, utilizou-se o formato de 18x3 a expressão do gene (Tabela 1). Fornecer sequências para todos os iniciadores e sondas (ou IDs de ensaio inventariados) para cada alvo para o fabricante.
2. Ácido Nucleico Extracção
- Extracto de ácido nucleico total (DNA e RNA) por qualquer método desejado.
NOTA: Um kit de extracção à base de esférulas magnéticas automatizado foi utilizado aqui de acordo com as instruções do fabricante (ver a tabela de materiais para mais detalhes). - Preparar as amostras como se segue:
- Limpe a parte externa de cada recipiente da amostra com 10% de água sanitária e seque bem. Limpe f enluvadadicas inger com lixívia a 10% entre as amostras para evitar a contaminação cruzada.
- Ponto nasal ou zaragatoas faríngeas profundas em qualquer recipiente estéril, selado (tal como um tubo de recolha de sangue topo vermelho) com algumas gotas de solução salina adicionada para prevenir a dessecação, antes da transformação.
NOTA: algodão, plástico, madeira de cabo, e Dacron e outros cotonetes sintéticos são aceitáveis, mas evitar swabs de alginato de cálcio. - Para cotonetes e lavagens traqueais, adicionar de Dulbecco Modified Eagle Médium (DMEM), de modo que existe, pelo menos, 1-2 ml de líquido. Vortex a zaragatoa e DMEM vigorosamente no tubo. Em seguida, utilizar uma pipeta para transferir cerca de 1 ml de meio para um novo tubo.
- tecidos mecanicamente lisam (100-200 mg em 1 ml de DMEM) usando um disruptor de tecido de acordo com as instruções do fabricante, seguido por centrifugação durante 3 minutos a 825 x g. Transferir 500 ul do sobrenadante para um novo tubo.
- Para as fezes, combinar a 400 mg de fezes com 800 ul de 1x SA tamponada com fosfatoA linha a pH 7,4 (PBS). Vortex a suspensão durante 1 minuto ou mais, até que a amostra é homogeneizada ou completamente suspenso. Uma vez homogeneizada, Centrifugar a suspensão durante 10 min a 18000 xg, em seguida, transferir 400 ul a um tubo novo.
- Para o sangue não coagulado todo, vortex a amostra e fazer uma alíquota de 250 ul. Adicione seis gotas de reagente lítico e vortex para misturar. Incubar 15 min a 37 ° C.
- Prepare a lise e lavagem tampões de acordo com as instruções do fabricante. Adicionar MS2 fago ou um controlo interno de escolha para o tampão de lise como um controlo positivo interno 9 extracção.
- Adicionar 235 ul de tampão de lise e 175 ul de amostra (ou PBS para o controlo negativo) a cada tubo de esferas. Prosseguir com o protocolo do fabricante.
NOTA: ácido nucleico purificada foi eluída total de aqui em 90 ul.
3. transcrição reversa / Pré-amplificação (pré-amplificador)
NOTA: Mantenha todos os reagentes e as amostras utilizadas noo pré-amplificador de reacção em gelo em todos os momentos. Na sequência pré-amplificador, manter todos os reagentes à temperatura ambiente.
- Montar o DNA eluído e / ou ARN, mistura de reacção de PCR, a água livre de nuclease, como um controlo negativo, e padrões combinadas de controlo de amplificação positivo.
NOTA: Até 48 amostras pode ser executado em uma placa de amplificação incluindo controles (incluir pelo menos um controle de extração negativo para cada placa de extracção a partir do qual as amostras deverão ser puxado). - Imprimir um mapa da amostra. Tem uma segunda verificação pessoa que o jogo disposição do mapa e da amostra.
- Em um tubo de 1,5 ml, adicione o seguinte para preparar a mistura principal pré-amplificador.
NOTA: Um volume final de 14 uL foi utilizada aqui.- Adicionar uma mistura de iniciadores pré-misturados de cada alvo de tal modo que a concentração final de cada iniciador é de 900 nM.
NOTA: A piscina utilizado aqui foi preparado a uma concentração 10x, e 1,4 jil adicionado por reacção. - Adicionar iniciadores aleatórios para uma concentração final de 600 nm ou 0,1x.
- Adicionar 2x mistura de RT-qPCR para metade do volume final (7 ul aqui).
- Misturar os componentes mestre Misture em vortex e girando suavemente.
- Adicionar uma mistura de iniciadores pré-misturados de cada alvo de tal modo que a concentração final de cada iniciador é de 900 nM.
- Realizar o pré-amplificador, em uma placa de 96 poços padrão usando o lado esquerdo da placa apenas (colunas 1-6).
- Adicionar 8,5 uL de mistura principal pré-amplificador e 5,5 ul de amostra de ADN / ARN a cada poço.
- Depois de combinar todos os reagentes (amostras, controles positivos e controlos negativos com pré-amplificador mix), selar completamente a placa de 96 poços padrão com um selo adesivo transparente. Remova qualquer excesso de vedação usando uma lâmina de barbear para evitar a formação de lacunas de vedação durante o ciclismo. Centrifugar a placa selada por 20 s usando uma placa de controle giratório PCR. Verificar para assegurar que todos os reagentes são combinados no fundo dos poços da placa.
- Executar o ensaio de pré-amplificador em um termociclador convencional sob as seguintes condições: 15 min a 50 ° C; 1 min a 95 ° C; 20 ciclos de 15 seg a 95 ° C, depois 2 minutos a 60 ° C; 99,9 & #176; C durante 10 min; manter a 4 ° C. Programa estas condições antes de iniciar.
- Ligue o termociclador convencional, selecione o programa de ciclos de pré-amplificador e iniciar o programa. Colocar a placa de 96 poços no termociclador apenas quando a porção inferior do termociclador (não a tampa) atinja a temperatura necessária para a produção de cDNA (50 ° C) através da monitorização da leitura de temperatura no ecrã. Antes disso, manter o frio placa para minimizar o risco de produzir resultados falsos positivos.
- Uma vez que o pré-amplificador de execução é concluída, verifique se a placa permaneceu selado.
- Proceder imediatamente para o passo 4.1. Para armazenar esta placa durante a noite, realizar a diluição, tal como descrito nas etapas 4.2 a 4.5, excepto em vez de cobrir a placa frouxamente, selar a placa com uma vedação adesiva clara e colocar dentro de um saco de zíper de bloqueio, armazenado a 4 ° C.
4. Diluição de Preamp Placa
- Remover o número apropriado de placas de amplificação from freezer (até 4 podem ser executados ao mesmo tempo). Não abra a embalagem. Permitir que a placa de aquecer durante pelo menos 15 min à temperatura ambiente. Registe o número e lote número de série da placa.
NOTA: placas não abertos podem permanecer em temperatura ambiente por até 24 horas, mas para as melhores práticas, removê-los do congelador não mais do que 30 minutos antes do necessário. - Centrifugar a placa pré-amplificador concluída durante 20 segundos usando a placa de controle giratório PCR. Ligue a máquina de amplificação e do computador e iniciar o programa associado.
- Remova todos os itens desnecessários de uma área de configuração PCR. Coloque luvas duplas e tampas da luva.
- Retire o selo da placa pré-amplificador e remova cuidadosamente e elimine as luvas externas. Dilui-se a produtos pré-amplificador a 1: 5 pela adição de 56 ul de tampão TE a cada poço das colunas 1-6 da placa pré-amplificador 96 poços e mistura-se pipetando para cima e para baixo. Ir apenas para o primeiro batente da pipeta, a aspiração a partir do fundo e de distribuição superior para cima, mas não criandobolhas. Adicionar tampão TE para todos os 6 colunas independentemente de poços não utilizados.
- Selar a placa ou com um adesivo transparente ou selo de alumínio e centrifugar-lo por 20 segundos usando a placa de controle giratório PCR. Defina este prato para o lado (vagamente coberto) até o passo 6.1 está completa. Descarte restantes luvas e capas de manga.
5. Preparação para 384 poços Transferência para a placa de amplificação
- Configure os materiais necessários para cobrir e selar a placa de amplificação: tampa, macho seringa de fluido de imersão, prima placa, garrafa de etanol esguicho, e lenços de laboratório que não solte fiapos. Retire a tampa da seringa e bloquear uma pequena ponta de plástico na seringa (requer força). Ejetar uma pequena quantidade de fluido de imersão em uma toalha de papel para remover o acúmulo de ar (que é ok se algumas pequenas bolhas de ar permanecer na ponta). Utilizar o fluido de imersão dentro de 60 minutos de retirar a seringa do saco de alumínio selado a vácuo. Uma vez que a seringa é aberta, não volte a colocar a tampa para uso posterior.
- Verificaque o caixote do lixo do manipulador de carga líquida placa está vazio e abrir a caixa de dicas. Escrever a data de vencimento das dicas sobre a tampa da caixa dica quando uma nova caixa é aberta, e garantir que as dicas não são expirado. Ligue o manipulador de líquidos e computador e abra o software manipulador de líquido, que irá realizar um auto-teste.
- Clique em "Setup and Load". Clique em "Browse" para selecionar o arquivo CSV criado a partir do software Rastreador de Amostra. Se o arquivo não é mostrado, vá para o "Instrumento / Editar Preferências" e clique em "Change" por "placa de amostra de pasta de arquivos". Selecione a pasta onde o arquivo CSV é salvo. Em seguida, clique em "Configuração e Load", depois em "Procurar" e selecione o arquivo.
- Em seguida, clique em "Procurar" ao lado da "placa de retenção Posição 1" e selecione o arquivo TPF (fornecido pelo fabricante) que tem o mesmo número de placa.
Transferência 6. Líquido Handler
Nota: Não comece a fienchendo a placa de 384 poços até que todos os passos acima estão completas. A evaporação é uma preocupação com pequenos volumes - não deixe que a placa de 384 poços sentar descoberto com líquido dentro.
- Uma vez que todas as etapas acima são completados colocados em novas luvas duplos, bem equipados e manga de cobre. Em seguida, adicionar 2,5 mL de amplificação de mistura principal para uma placa de 384 poços.
- Transferir 2,5 mL de produto pré-amplificador diluído para a placa de 384 poços de acordo com o mapa na Tabela 2 e cobrir imediatamente a placa firmemente com um selo de alumínio. Descarte luvas externas e coberturas luva.
- Centrifugar a placa de 384 poços durante 20 segundos na placa fiandeira PCR.
- Não retire o selo de alumínio, mas colocar a placa de 384 poços no manipulador de líquido.
- Abra o pacote contendo uma placa de amplificação encerrado e colocá-lo cuidadosamente no manipulador de líquido na primeira ranhura, com o número de série à direita - mantenha o caso pelas bordas sem tocar a superfície of a placa. Utilizar placas desembrulhados dentro de uma hora de abertura.
Nota: O objectivo do processo é o de proteger a placa de ser tocado, uma vez que isso poderia perturbar as pequenas quantidades de iniciadores e sondas no interior dos furos de passagem. - Remova o selo de alumínio da placa de 384 poços dentro do manipulador de líquidos uma vez que tudo está no lugar. Clique em Avançar e em seguida, verificar todas as caixas que cada etapa é concluída. Clique em OK para iniciar a execução.
- Feche a porta para o manipulador de líquidos e iniciar imediatamente o processo de enchimento. Fique ao lado do manipulador de líquidos e prosseguir imediatamente para a próxima etapa uma vez que a placa está cheia.
- Enquanto o manipulador de líquidos está em execução, remova o filme protetor da parte inferior de uma tampa caso.
NOTA: O adesivo na parte inferior da tampa da caixa é coberta por uma fita de vermelho e uma película de protecção. O filme precisa ser removido primeiro a acessar a fita vermelha.- Deixar o filme superior no lugar até que o passo 6.15. Primeiro a fita vermelha, puxando ligeiramente para releasum e que a partir do adesivo.
- Remova o (preenchido) placa de amplificação carregado a partir do manipulador de líquidos e gentilmente colocá-lo na imprensa placa com o número de série no lado direito.
- Coloque a tampa na parte superior da placa com a extremidade recortada para a direita e o adesivo na parte inferior (que aponta para a placa).
- Puxe para baixo a alavanca de imprensa placa de fechá-lo com cuidado; a luz pisca durante 20 segundos. Não toque na placa de imprensa durante este tempo. Assim que a luz fica verde, levante a alavanca com cuidado e retire cuidadosamente a placa selada, segurando-o nas bordas.
- Enquanto que prende a placa de amplificação selado verticalmente pelas extremidades do caso, inserir imediatamente a ponta para seringas para o porto de carga, no final do processo, em seguida, distribuir o fluido de imersão lentamente em um movimento contínuo suave para preencher o espaço entre a placa e o tampa. Deixe uma pequena bolha de ar no canto uma vez que a placa está imerso.
- Embora continuando a hvelha a placa verticalmente pelas extremidades, selar o porto de carga, inserindo a ficha na porta e torcer no sentido horário ficha, aplicando pressão suficiente até que a alça quebra. Aderência das bordas do caso firmemente de modo a que a força de ruptura do cabo não causar o caso de queda. Se uma placa cair, descartá-lo.
- Limpe a caixa com um pano que não solte fiapos foi completamente pulverizada com etanol. Para secar o caso, limpe a caixa para baixo com um pano limpo limpe. Gentilmente lidar com o caso; certifique-se de não aplicar pressão sobre o vidro acima dos poços.
- Traga a placa selada à máquina de amplificação. Sob a guia "Instrumento", selecione "Console Instrumento". Seleccione a máquina de amplificação, em seguida, clique no botão "Open Door".
- Coloque a placa de amplificação no primeiro slot do adaptador de placa, verificando se o adaptador está correctamente alinhada com o A1 no canto superior esquerdo. Oriente a placa com o código de barras voltado para cima e em direçãoa parte frontal do instrumento. Clique no botão "Feche a porta". Note-se a utilização da bandeja de adaptador - há um limite de 10 utilizações.
- Na guia Início, na parte inferior da tela, sob o menu Executar, selecione OpenArray. Clique em "Obter IDs de placa." As caixas vazias serão automaticamente preenchidos com informações sobre a placa. Para começar a correr, clique em "Start Run".
- Feche o software de manipulador de líquidos e desligue o manipulador de líquidos. Coloque a tampa da ponta sobre o rack de ponta. Descartar as dicas do caixote do lixo e limpá-lo. Limpo e descontaminar todos os itens, incluindo a superfície de trabalho, pipeta, balde de lixo e caixas de sugestões.
- Selar a placa pré-amplificador com um selo adesivo transparente e armazenar a -20 ° C.
Análise 7. Resultado
- Uma vez que o prazo tenha terminado, salve o arquivo e, em seguida, clique no "X" no separador com o nome da execução, na parte inferior da tela para fechar o arquivo.
- Clique em "Porta Aberta"No topo da tela para abrir a porta. Remova a placa de amplificação, verificar que nenhum fluido de imersão vazou. Clique em" Feche a porta "no topo da tela para fechar a porta.
- Clique em "Abrir" e selecione o arquivo de execução para abri-lo. Pressione o botão verde Analisar no canto superior direito da tela. Clique em "Exportar" no lado esquerdo da tela e, em seguida, "Start Export" para exportar os resultados (como um arquivo .txt). Minimizar o arquivo depois que se abre. Em seguida, clique em "Export QC Imagens".
- Rever imagens QC no programa de análise de imagem de acordo com os seguintes critérios:
- Avaliar a qualidade do carregamento com PRE-READ_CHANNEL_4.tiff e pós-READ_CHANNEL_4.tiff, verificando se não há manchas escuras ou manchas.
NOTA: Um corante detectadas por este canal é adicionado pelo fabricante para os iniciadores e sondas, que é libertado para a solução quando a reacção é carregado. A leitura neste canal indica que as reações foram adequadamentecarregados e que os iniciadores e sonda misturados com corante foram presente. - Verifique se há vazamentos ou agitação para a placa após o carregamento usando S02_C001_t03_p001_m1_x2_e1_cp # _spotfind.tiff. Se a imagem estiver escura, aumentar o brilho (pressione Ctrl + Shift + C para abrir controles) até que toda a placa é visível. Verifique se a imagem parece uniforme, sem sombras, bolhas, ou amostras deslocadas.
- Avaliar a colocação da tampa e detritos sob a tampa (pontos brilhantes) usando STAGE2_CYCLE1_CHANNEL_1.tiff e STAGE2_CYCLE40_CHANNEL_1.tiff.
- Avaliar a qualidade do carregamento com PRE-READ_CHANNEL_4.tiff e pós-READ_CHANNEL_4.tiff, verificando se não há manchas escuras ou manchas.
- Opcional: Abra uma planilha contendo o Macro Resultados (arquivo de código Suplementar). No arquivo de dados exportados, clique direito sobre a aba na parte inferior da tela e selecione "Mover ou Copiar". No campo "reservar", selecione "Resultados Macro.xlsm". Clique em "Mover para acabar" e marque a caixa "Criar cópia". Em seguida, clique em OK.
- Se a opção Resultados Macro não aparecer no campo "reservar", simply copiar o conteúdo da planilha arquivo de dados exportados (clique na célula superior esquerda para destacar todas as células e Ctrl-C) e colá-lo em uma nova aba.
- Excluir o guia de idade "Export" e, em seguida, mudar o nome do guia recém-adicionado como "Export".
- Clique em "Alt-F8" (ou clique em Exibir e Macros), em seguida, selecione "Macro" e clique em "Executar". Em seguida, repita isso para Macro2, clicando em "Alt-F8", em seguida, selecionando "Macro2" e clicar em "Run".
- Renomeie o arquivo Resultados Macro usando as informações relevantes (data e número de série), colocar esta informação acima da tabela, e salvar como o mesmo nome do arquivo na pasta Resultados exportado. Finalmente, imprima resultados tabela gerado pelo macro.
- No software do aparelho de amplificação, verifique as curvas para cada amostra e controle contra a mesa macro, selecionando Análise / Amplification Plot no lado esquerdo da tela. Em seguida, selecione a guia Sample, e clique em cada amostra aconfirmar que existem 2 ou 3 curvas de amplificação positivos para cada um dos resultados positivos na tabela.
- Desligue a máquina de amplificação.
Resultados
Os resultados são mostrados nas Figuras 1 e 2 por um ensaio representativo de ARN (matriz da gripe) e ensaio de ADN (EHV-1) com uma combinação de controlos positivos e amostras clínicas enviadas para testes de diagnóstico de rotina. Os sinais de fluorescência emitidas ao longo deste reaccionais típicas estão mostradas na Figura 1, que representa graficamente os valores de fluorescência em bruto por ciclo. Todos os limiares ciclo relativa (TC) foram gerados automaticamente pelo software do aparelho de amplificação. a fluorescência de fundo foi usado como uma medida separada para avaliação da qualidade do carregamento em vez de incorporá-lo nas leituras de fluorescência antes de calcular os valores de Ct.
O limite de detecção (LOD) para cada alvo foi calculada com base na média geral dos valores Ct do nível de detecção de 95% e de 2 desvios-padrão em uma associação de controlos paratodos os alvos. A diluição mais elevada em que, pelo menos, 95% das réplicas foram positivos para os ensaios representativos foi de 50 cópias; detecção de 5 cópias foi bem sucedida em 50% de repetições. Nem o ensaio foi detectada abaixo de uma cópia. Os limites de determinação para o ensaio de RNA e DNA foram calculados os valores Ct de 21,92 e 20,05. Estes valores foram considerados os pontos de corte para os valores de relatórios como positivas vs. suspeito (potencialmente não repetível).
Outros aspectos do desempenho analítico dos alvos foi avaliada utilizando diluições em série de controlos positivos reunidas são executados em três dias diferentes (Figura 2). As eficiências médias dos ensaios de ARN e de ADN representativos eram 101,1% e 106,6%; a eficiência média geral para todos os alvos foi 101,3%. Os ensaios também teve boa linearidade (R 2> 0,98). Variação dentro replicar orifícios tipicamente estava dentro de desvios-padrão de 0,2 para ambas as amostras clínicas ded controlos. Não foi observada nenhuma reactividade cruzada entre qualquer um dos objectivos.
A planilha Resultados Finais no arquivo suplementar mostra resultados quantitativos representativos para 10 amostras de diagnóstico clínico e 4 controles. As amostras clínicas são um subconjunto dos tipos de amostras testados rotineiramente incluindo nasal / amostras de orofaringe, tecido pulmonar, e as amostras fecais. Um (equina) amostra fecal neste conjunto mostra um controlo interno MS2 falha, que indica a presença de inibidores na amostra. Isto é tipicamente controlado por diluição da amostra eluiu-se e / ou a re-extracção. Como é o caso aqui, o controlo de amplificação negativa deve ser negativo para todos os alvos, eo controle de extração negativo só deve conter o controlo interno. No conjunto clínica, amostras produziu valores Ct para betacoronavirus, Bordetella bronchiseptica, cinomose vírus A, vírus parainfluenza canina, pneumovírus canino, ea gripeUMA.
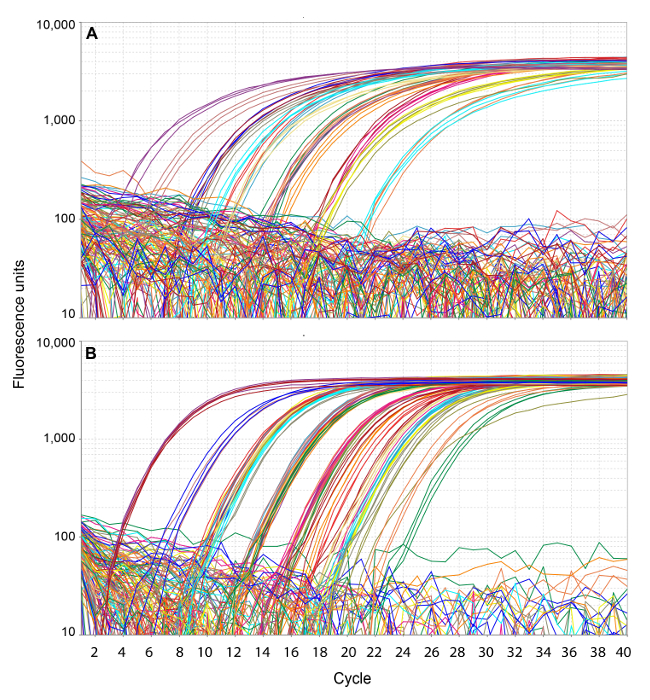
Figura 1. Os gráficos de amplificação. As leituras de fluorescência são representados graficamente contra a ciclos de amplificação de RNA para o ensaio (A) e ensaio de DNA (B). Triângulos são mostrados denotando valores Ct. Por favor clique aqui para ver uma versão maior desta figura.
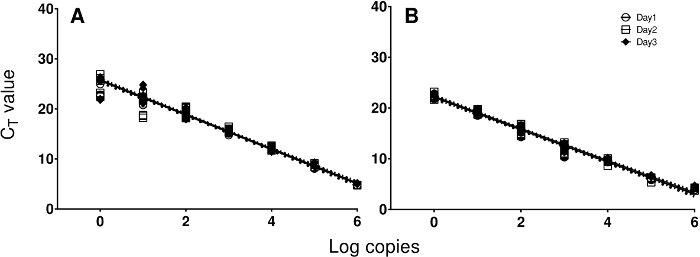
Figura 2. As curvas padrão. As curvas padrão do ensaio de RNA e DNA testado em três dias diferentes são representados, a fim de demonstrar a linearidade e gama de amplificação, utilizando os controlos positivos. Ciclo valores limiar (Ct) em função de log (10) do número de cópias de ARN (A) ou de DNA ( B) padrão. Por favor clique aqui para ver uma versão maior desta figura.

Tabela 1. Esquema de os locais-alvo e placa de amplificação. As placas utilizadas para ensaios de PCR em tempo real nanoescala nesta plataforma de lâmina de microscópio são de tamanho e estão dispostos em 48 submatrizes de 64 furos de passagem, com um total de 3072 orifícios para as reacções individuais. Um sub-disposição é mostrado aqui, com 18 alvos, em triplicado. Cada amostra é adicionada a uma sub-disposição pelo manipulador de líquidos. As placas são revestidas com compostos hidrófilos e hidrófobos para reter os reagentes em furos de passagem através de tensão superficial. O chip de aço inoxidável é "photolithographically estampados e wet-gravado para formar uma matriz rectilínea de 3072 usinado-micro, 320 mícrons diamete. r buracos de 33 nl cada "2 abreviações para as metas são as seguintes: BCOR, coronavírus respiratório canino (betacoronavirus); BORD, Bordetella CAV bronchiseptica, adenovírus canino; CPIV, vírus parainfluenza canina; CPNv, pneumovírus canino; DISTA / B, canino vírus da cinomose A e B; EADV1 / 2, adenovírus eqüino 1/2; EAV, vírus eqüino arterite; EHV-1/4, eqüino tipo de herpesvírus 1/4; ERVA / B, equina vírus rinite A / B; IVM, influenza A matriz; MCYNOS, Mycoplasma cynos; MS2, controle interno (MS2 RNA fago); sequ, Streptococcus equi.
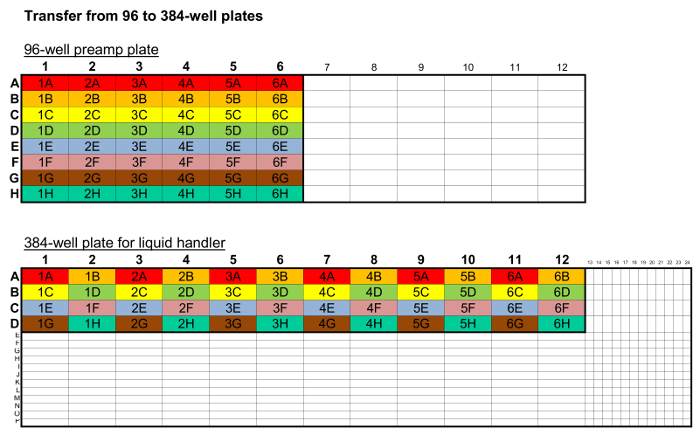
Tabela 2. Transferir a placa de mapa. Um mapa de transferência de placa com código de cores para a utilização de uma pipeta de 8 canais fixa para transferir a partir do pré-amplificador de placa de 96 cavidades para a placa de 384 poços é mostrado. Alternativamente, pode ser utilizada uma pipeta ajustável. O mesmo procedimento é realizado cada vez independentemente de h OW muitas amostras são na placa.
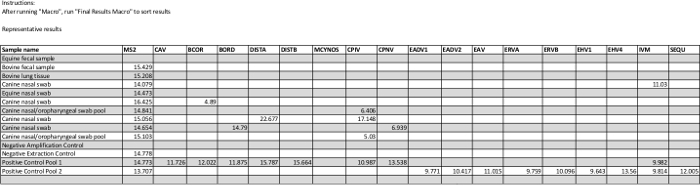
Arquivo suplementar. Um arquivo de planilha habilitado para macro que contém duas macros para resultados de formatação em uma tabela de resumo (conforme descrito no protocolo) é fornecido. A tabela de resultados típico gerado pelas macros está incluído no arquivo (em Resultados Finais). As duas macros irá preencher os nomes da amostra na primeira coluna (até 48 amostras) e a média dos três valores de Ct para cada alvo para aqueles com resultados positivos; note que os alvos que não amplificam aparecem em branco nesta tabela. Isto pode ser facilmente impressa sobre um pedaço de papel e usada como uma referência para a verificação dos dados em bruto de forma eficiente. Na guia resultados finais, resultados representativos para 10 amostras clínicas e 4 controles são mostrados. Abreviações de nomes de ensaio são listados na legenda da Tabela 1.www.jove.com/files/ftp_upload/54781/supplemental_code_file_R1.xlsm "target =" _ blank "> Clique aqui para baixar esse arquivo.
Discussão
Este procedimento foi utilizado para testes de rotina de agentes patogénicos respiratórios no nosso laboratório, ao longo de seis meses (2-3 vezes por semana). Nós também tivemos sucesso usando o mesmo procedimento para criação de perfis de patógenos entéricos em amostras fecais e bactérias isoladas em um prato personalizado separadamente. Uma vez que as placas são produzidas, a equipe experiente pode completar as etapas 2-7 no prazo de um dia de trabalho normal. Os passos mais críticos são a vedação adequada da placa pré-amplificador, a transferência das amostras entre as placas pré-amplificado (que deve ser feito rapidamente para evitar a evaporação), e o revestimento final da placa de amplificação. Utilização da folha de cálculo macro como um guia para a análise dos resultados (verificação curvas para as amostras e controlos) foi também crítica que reduziu significativamente a quantidade de tempo e papelada necessária para este processo. A planilha macro fornecida é um exemplo; ele precisaria ser modificado ou re-criada para placas com diferentes configurações. Isso pode facilmente ser performed por alguém com conhecimento básico de planilha.
Devido à quantidade muito pequena de amostra carregada na placa de amplificação (33 nl), a pré-amplificação é necessária. Na optimização deste protocolo (não mostrado), comparou-se um número de parâmetros de Pré-amplificação, incluindo a mistura principal, a adição de iniciadores aleatórios, número de ciclos, o tempo de recozimento, e a diluição antes da amplificação. Cada alvo tinha suas próprias condições ideais, e os descritos aqui representam aquelas que produziram o melhor limite de detecção geral para o nosso painel. Este painel abrange uma ampla gama de alvos patogénicos e tipos de amostras que são encontrados nos testes de diagnóstico veterinário de rotina, mas na modificação dos procedimentos Pré-amplificação podem ser necessários para diferentes painéis. As condições de amplificação optimizado pelo fabricante baseiam-se num tempo real protocolo de PCR com base em sonda com um padrão de 10 min a 95 C de activação de enzimas ° seguido de desnaturação a 95 ° C e AnnEaling / extensão a 60 ° C. Os iniciadores e as sondas são pré-impresso sobre as placas também utilizando condições optimizadas pelo fabricante e, portanto, não necessitam de titulação. A combinação dos passos de transcrição reversa e de pré-amplificação de todas as amostras (independentemente do tipo de alvo) foi necessário a fim de manter a eficiência do fluxo de trabalho. Tendo em todas as amostras de transcrição reversa também é benéfico para maximizar a sensibilidade. Além disso, o uso de uma mistura principal para o pré-amplificador que é otimizado para inibidores minimizando permite versatilidade na combinação de diferentes tipos de amostras na mesma placa.
O Center for Disease Control (CDC) ensaio de matriz de influenza descrito por Shu et al. 7 e adaptado aqui para reações escala nanoliter é uma gripe universal Um ensaio que é apropriado para testar amostras de seres humanos e animais de companhia. Ele foi projetado para a detecção universal do gene matriz de todos os vírus da gripe A utilizando rea escala microlitroficções. Ela tem sido usada em todo o mundo como parte de um painel de vírus CDC Influenza Humana e um Painel de gripe suína CDC. O ensaio 8 EHV-1 adaptada aqui detecta um importante patógeno respiratório de cavalos que podem causar abortos e doença neurológica (revisado por Pusterla e Hussey 10). O significado de se adaptar estes ensaios a uma plataforma de alto rendimento com um controlo interno independente da espécie é que eles podem ser incorporados em uma abordagem de vigilância OneHealth. Tendo ambos os ensaios em uma plataforma de testes de alto rendimento irá facilitar a preparação para emergências em instalações clínicas, abrigos e eventos de desempenho.
Os resultados descritos acima foram representativas de todos os alvos na placa com a excepção de Mycoplasma cynos, que mostraram consideravelmente mais variação no desempenho analítico. Isto era provavelmente devido à temperatura sub-óptima de fusão (T m) dos iniciadores, o que deve ser idealmente de 58-60 &# 176; C (a sonda Tm ideal é 68-70 ° C). Uma limitação desta plataforma é que ela leva mais tempo para projetar e fabricar as placas do que para encomendar sondas individuais, o que limita a capacidade de modificar rapidamente sequências. Outra limitação de PCR em tempo real, em geral, é a incapacidade de detectar novos agentes patogénicos ou inesperados. Isto pode ser ultrapassado, em certa medida através da concepção de ensaios que correspondem a sequências em várias espécies, mas metodologias baseadas inteiro de sequenciamento do genoma imparciais são mais adequados para a descoberta de novos agentes 11,12.
Nanoescala PCR em tempo real permite que um novo paradigma para, em vez de testes de espécies baseada, que é útil para reduzir o custo dos reagentes e trabalhistas em diagnóstico molecular de alto rendimento à base de síndrome. Testes de painel em grande escala por esta abordagem pode facilitar os esforços de vigilância OneHealth tais como os descritos por Dunne e Gurfield 13 e Moutailler et al. 14. Recolha de amostras de esfregaço cedo,geralmente dentro de 3 dias do início clínico, proporciona a melhor oportunidade para identificar a presença de patógenos respiratórios. Infecciosa surgimento da doença é muitas vezes imprevisível, e testes que podem ser realizados em espécies diferentes sem a necessidade de modificação de reagentes são ideais para a preparação. As aplicações futuras desta tecnologia são susceptíveis de estar em tipagem patogénica, de perfis de resistência antimicrobiana, e outros painéis à base de diagnósticos clínicos da síndrome. Nanoescala PCR em tempo real é mais útil para uma rápida triagem, de alto rendimento de vários tipos de amostras e de agentes patogénicos, e serão complementadas com abordagens baseadas em seqüenciamento imparciais ou parcialmente tendenciosos para identificar novas e emergentes patógenos.
Divulgações
Marcia Slater e Elen Ortenberg são funcionários da Thermo Fisher Scientific Inc., que produz reagentes e instrumentos utilizados neste artigo.
Agradecimentos
O trabalho sobre os ensaios de patógenos respiratórios descritos aqui foi apoiado por fundos de desenvolvimento interno Cornell Centro de Diagnóstico de Saúde Animal. Desenvolvimento da nanoescala fluxo de trabalho PCR e sistemas de garantia de qualidade associados foi parcialmente financiado (FOA PA-13-244) e realizado em colaboração com o Laboratório de Investigação Veterinária da Food and Drug Administration e Response Network (FDA Vet-LIRN), Grant No. 1U18FD005144- 01. As taxas de publicação foram patrocinados pela VWR e Quanta Biosciences. Agradecemos Gabrielle Nickerson, Roopa Venugopalan, Veldina Camo, Xiulin Zhang, Jinzhi Yu, Weihua Wang, e Katrina Walker para a sua assistência com a escrita e revisão do protocolo. Agradecemos a Mike Carroll para filmar as entrevistas do autor. Nós finalmente agradecer ao editor e três revisores anônimos por seus comentários.
Materiais
| Name | Company | Catalog Number | Comments |
| Zap-OGlobin II lytic reagent | Beckman Coulter | 7546138 | Optional reagent for preparing blood samples. |
| Extraction kit (MagMAX Total Nucleic Acid Isolation Kit) | Thermo-Fisher/Applied Biosystems | AM1840 | Other extraction kits appropriate for the desired sample types may be substituted. This kit comes with PBS, lysis buffer, and wash buffer. |
| 2x One-Step RT-qPCR mix (qScript XLT One-Step RT-qPCR ToughMix) | Quanta Biosciences | 95134-500 | |
| Gene-specific primer pool | IDT | custom order | Can be generated by the user or purchased with the amplification plates. |
| Random primer mix | New England Biolabs | S1330S | |
| Standard 96-well plate | Thermo-Fisher/Applied Biosystems | N8010560 | This can be any plate that fits into the conventional PCR machine used. |
| Amplification master mix (OpenArray master mix) | Thermo-Fisher/Applied Biosystems | 4462164 | |
| Clear adhesive seal | Thermo-Fisher | 430611 | |
| Foil seal | Excel Scientific | AF-100 | |
| 384-well plate | Thermo-Fisher/Applied Biosystems | 4482221 | |
| Amplification plate (QuantStudio 12K Flex OpenArray plate) | Thermo-Fisher/Applied Biosystems | 4470813 | Can be customized with any assays conforming to standard TaqMan conditions. |
| Accessories kit | Thermo-Fisher/Applied Biosystems | 4469576 | Contains the case lids, plugs, and immersion fluid. |
| AccuFill tips | Thermo-Fisher/Applied Biosystems | 4457246 | |
| Amplification machine (QuantStudio 12K Flex Real-Time PCR System) | Thermo-Fisher/Applied Biosystems | 4471090 | |
| Liquid handler (OpenArray AccuFill System) | Thermo-Fisher/Applied Biosystems | 4457243 | This is purchased with the amplification machine. |
| Primer design software (Primer Express) | Thermo-Fisher/Applied Biosystems | 4363991 | See manufacturer's instructions for detailed primer and probe specifications. |
| Image analysis program (ImageJ) | NIH | Available at http://imagej.nih.gov/ij/ | |
| Spreadsheet program (Excel) | Microsoft | Available at https://products.office.com/en-us/excel | |
| DMEM | Thermo-Fisher/Gibco | 11965-092 | |
| Tissue disruptor (TissueLyser II) | Qiagen | 85300 | |
| Conventional thermal cycler (Veriti) | Thermo-Fisher/Applied Biosystems | 4375786 | Any conventional thermal cycler with a heated lid can be used. |
| PCR plate spinner | VWR | 89184-608 | This device has only one speed (2,500 rpm); the rotor starts when the lid is closed and stops when the button is pushed. |
| TE buffer | Millipore | 8890-100ML | 10 mM Tris(hydroxymethyl)aminomethane hydrochloride, 1 mM Ethylenediaminetetraacetic acid, pH 8.0 |
Referências
- Kalinina, O., Lebedeva, I., Brown, J., Silver, J. Nanoliter scale PCR with TaqMan detection. Nucleic Acids Res. 25 (10), 1999-2004 (1997).
- Morrison, T., et al. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34 (18), e123 (2006).
- Bustin, S. A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. J Mol Endocrinol. 29 (1), 23-39 (2002).
- McCall, M. N., et al. A benchmark for microRNA quantification algorithms using the OpenArray platform. BMC bioinformatics. 17 (1), 138 (2016).
- Pozzi, A., Previtali, C., Cenadelli, S., Gandini, L., Galli, A., Bongioni, G. Genetic traceability of cattle using an OpenArray genotyping platform. Anim Genet. 47 (1), 133-134 (2016).
- Grigorenko, E., et al. Multiplex screening for blood-borne viral, bacterial, and protozoan parasites using an OpenArray platform. J Mol Diagn. 16 (1), 136-144 (2014).
- Shu, B., et al. Design and performance of the CDC real-time reverse transcriptase PCR swine flu panel for detection of 2009 A (H1N1) pandemic influenza virus. J Clin Microbiol. 49 (7), 2614-2619 (2011).
- Elia, G., et al. Detection of equine herpesvirus type 1 by real time PCR. J Virol Methods. 133 (1), 70-75 (2006).
- Dreier, J., Störmer, M., Kleesiek, K. Use of Bacteriophage MS2 as an Internal Control in Viral Reverse Transcription-PCR Assays. J Clin Microbiol. 43 (9), 4551-4557 (2005).
- Pusterla, N., Hussey, G. S. Equine Herpesvirus 1 Myeloencephalopathy. Vet Clin North Am Equine Pract. 30 (3), 489-506 (2014).
- Firth, C., Lipkin, W. I. The genomics of emerging pathogens. Annu Rev Genomics Hum Genet. 14, 281-300 (2013).
- Lecuit, M., Eloit, M. The potential of whole genome NGS for infectious disease diagnosis. Expert Rev Mol Diagn. 15 (12), 1517-1519 (2015).
- Dunne, G., Gurfield, N. Local Veterinary Diagnostic Laboratory, a Model for the One Health Initiative. Vet Clin North Am Small Anim Pract. 39 (2), 373-384 (2009).
- Moutailler, S., et al. Co-infection of Ticks: The Rule Rather Than the Exception. PLOS Negl Trop Dis. 10 (3), e0004539 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados