É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação da Transcitose Intestinal de Isolados de Bacteremia de Escherichia coli Neonatal
Neste Artigo
Resumo
Escherichia coli causa sepse em neonatos que ingerem a bactéria na época do nascimento. O processo envolvido na capacidade da E. coli de viajar do trato entérico para a corrente sanguínea é pouco compreendido. Este modelo in vitro avalia a capacidade das cepas de E. coli de viajar através das células epiteliais intestinais.
Resumo
Os recém-nascidos ingerem cepas maternas de E. coli que colonizam seu trato intestinal na época do parto. Cepas de E. coli com a capacidade de se deslocar através do intestino invadem a corrente sanguínea do recém-nascido, causando bacteremia com risco de vida. A metodologia aqui apresentada utiliza células epiteliais intestinais polarizadas cultivadas em pastilhas semipermeáveis para avaliar a transcitose de isolados de bacteremia neonatal de E. coli in vitro. Este método usa a linhagem celular intestinal T84 estabelecida que tem a capacidade de crescer até a confluência e formar junções apertadas e desmossomos. Após atingir a confluência, as monocamadas maduras de T84 desenvolvem resistência transepitelial (TEER), que pode ser quantificada usando um voltímetro. Os valores de TEER estão inversamente correlacionados com a permeabilidade paracelular de componentes extracelulares, incluindo bactérias, através da monocamada intestinal. A passagem transcelular de bactérias (transcitose), por outro lado, não altera necessariamente as medidas do TEER. Neste modelo, a passagem bacteriana através da monocamada intestinal é quantificada por até 6 h pós-infecção, e medições repetidas de TEER são feitas para monitorar a permeabilidade paracelular. Além disso, esse método facilita o uso de técnicas como a imunocoloração para estudar as mudanças estruturais em junções apertadas e outras proteínas de adesão célula a célula durante a transcitose bacteriana através do epitélio polarizado. O uso deste modelo contribui para a caracterização dos mecanismos pelos quais a E. coli neonatal transcita através do epitélio intestinal para produzir bacteremia.
Introdução
Escherichia coli é a causa mais comum de sepse de início precoce em recém-nascidos 1,2,3. A taxa de mortalidade da bacteremia neonatal por E. coli pode chegar a 40%, sendo a meningite uma possível complicação associada a graves incapacidades de neurodesenvolvimento2. A ingestão de cepas maternas de E. coli pelo recém-nascido pode produzir bacteremia neonatal; esse processo tem sido replicado em modelos animais 2,4. Uma vez ingeridas, as bactérias patogênicas viajam do lúmen intestinal neonatal através da barreira intestinal e entram na corrente sanguínea, causando septicemia. Cepas neonatais invasivas de E. coli que produzem bacteremia variam em sua capacidade de invadir células epiteliais intestinais 1,5. No entanto, sua capacidade de transcitar o epitélio intestinal após a invasão não foi completamente caracterizada.
Este modelo de transcitose intestinal é um método in vitro útil para emular a passagem bacteriana através do epitélio intestinal. O objetivo geral dos métodos apresentados neste manuscrito é comparar a capacidade de isolados neonatais de E. coli de transcitose o epitélio intestinal. O modelo aqui descrito utiliza células T84, que são células imortalizadas de adenocarcinoma intestinal humano 6,7. As células T84 são cultivadas até a confluência em uma membrana semipermeável com dois compartimentos separados. A justificativa para o uso dessa técnica é que, como acontece in vivo, essas células intestinais polarizam e desenvolvem junções apertadas maduras 6,8. O lado em contato com a membrana torna-se o lado basal. O lado oposto das células torna-se o lado apical, assemelhando-se ao lúmen intestinal, onde os patógenos ingeridos aderem e invadem. A membrana transwell é permeável a bactérias, mas as células intestinais polarizadas formam junções apertadas, o que prejudica o movimento paracelular bacteriano9. Assim, este método proporciona a vantagem de um ambiente controlado in vitro utilizando uma linhagem celular humana para estudar o processo de transcitose bacteriana, incluindo a via transcelular. Enquanto outros métodos existem para investigar a transcitose de bactérias através do epitélio intestinal, o método transwell apresentado aqui fornece maior facilidade e acessibilidade. Técnicas alternativas, como as que utilizam amostras ex vivo configuradas em sistemas de câmara Ussing, estão disponíveis. No entanto, utilizam espécimes de tecido que podem não ser facilmente acessíveis, principalmente se a pesquisa pretende estudar a fisiologia humana10. Os organoides intestinais representam outro exemplo de alternativa in vitro para o estudo das interações hospedeiro-bactéria11. Embora as monocamadas organoides também possam ser usadas no sistema transwell para estudar a transcitose bacteriana, elas requerem o isolamento e o crescimento de células-tronco e o uso de fatores de crescimento específicos para induzir a diferenciação12. Assim, seu uso é mais demorado e associado a maiores custos em comparação com o método transwell descrito neste manuscrito.
A avaliação da passagem bacteriana através do epitélio intestinal usando este sistema de transwell in vitro tem sido realizada com sucesso para vários patógenos. Esses estudos têm demonstrado a utilidade do sistema transwell utilizando células T84 para caracterizar a transcitose de bactérias através do epitélio intestinal polarizado13,14,15. No entanto, a aplicação deste método transwell para comparar a capacidade de transcitose de cepas neonatais de E. coli produtoras de bacteremia não foi descrita em detalhes. Este manuscrito fornece a outros pesquisadores um protocolo de transwell padrão que é confiável e fácil de usar e não requer recursos que são muito caros.
Para comparar a capacidade de cepas neonatais invasivas de E. coli de transcitar o epitélio intestinal, o lado apical da monocamada epitelial intestinal pode ser infectado com um número conhecido de células bacterianas. Após a incubação, o meio no lado basal do epitélio pode ser coletado e as bactérias quantificadas para determinar a quantidade de transcitose bacteriana ao longo do tempo. Neste manuscrito, os métodos apresentados são utilizados para estudar a capacidade de transcitose de cepas clínicas neonatais de E. coli recuperadas de recém-nascidos hospitalizados com bacteremia. Os critérios de inclusão para a seleção desses isolados clínicos neonatais para estudos de transcitose já foram publicados anteriormente 1,2,16. Quando este método é realizado usando diferentes cepas de E. coli, suas habilidades de transcitose podem ser comparadas. Através deste processo, o modelo de transcitose intestinal fornece dados valiosos para caracterizar os fatores de virulência de E. coli que contribuem para o processo multipasso que culmina no desenvolvimento da bacteremia neonatal.
Protocolo
NOTA: Execute todas as manipulações das células, bactérias, placas e reagentes T84 em um gabinete de segurança de Nível de Biossegurança 2 (BSL-2) para evitar a contaminação. Use áreas separadas e incubadoras para todo o trabalho envolvendo células T84 estéreis, células T84 infectadas e E. coli. Os isolados clínicos de E. coli testados com os métodos aqui descritos foram obtidos seguindo as diretrizes do Comitê de Ética em Pesquisa de nossa instituição 1,16.
1. Preparação de inserções de transcitose com células T84 (aproximadamente 1-2 semanas antes do experimento)
- Grow American Type Culture Collection (ATCC) células T84 em meio de cultura de tecidos (TCM + antibióticos) consistindo de Dulbecco's Modified Eagle Medium: Mistura de nutrientes F-12 de Ham: mistura de nutrientes F-12 de Ham: 50% cada), 5% de soro fetal bovino e 1% (100 U/mL) de penicilina/estreptomicina dupla mistura de antibióticos. Incubar as células a 37 °C com 5% de CO2.
NOTA: Uma variação desta formulação de meio sem penicilina/estreptomicina (MTC sem antibióticos) é utilizada para as etapas posteriores (secção 2 e seguintes) do procedimento. Certifique-se de que a formulação correta seja usada para cada etapa. - Trabalhando dentro de um gabinete de biossegurança (BSC), semeie as células T84 em inserções transwell de cultura de células de membrana de polietileno tereftalato com poros de 3 μm feitos para placas de 24 poços. Inclua replicações de inserção transwell para cada condição experimental desejada, além de controles não infectados para monitorar possíveis contaminações.
- Em uma placa de 24 poços projetada para conter inserções transwell, preencha o número desejado de poços coletores com 1 mL de TCM + antibióticos.
- Em cada poço, coloque uma inserção transwell.
- Semeia estas inserções com 1 x 105 células T84 suspensas em 500 μL de MTC + antibióticos. Quantifique o número de células utilizando um hemocitômetro com coloração azul de tripano ou um contador celular automatizado17.
- Incubar as placas transwell contendo inserções semeadas nas mesmas condições em que as células foram cultivadas.
- Verificar com microscopia de luz se as monocamadas começaram a se tornar confluentes após a semeadura da pastilha, aproximadamente 48 h após a semeadura.
- A cada 2 dias após a semeadura, medir e registrar a resistência elétrica transepitelial (TEER) usando um medidor epitelial de volt/ohm (EVOM) para avaliar a maturidade da monocamada. Uma vez que as inserções atinjam um TEER de pelo menos 1.000 Ω·cm2, elas são consideradas prontas para o ensaio18.
NOTA: As inserções normalmente levam de 7 a 10 dias para atingir este TEER após a semeadura. As células T84 apresentarão 100% de confluência sob microscopia de luz, uma vez atingida essa resistência.- Armazene a sonda EVOM com os eletrodos submersos em 0,15 M KCl quando não estiver em uso.
- Antes de medir o TEER, descontamine a sonda do eletrodo submergindo-a em 5 mL de etanol a 70% em um tubo cônico de 50 mL por 10-15 min. Remova a sonda, agite o excesso de etanol e deixe-o secar ao ar dentro do BSC por 10 min. Reter o tubo de etanol.
- Teste o EVOM e a sonda colocando a sonda seca descontaminada em um poço estéril contendo 1 mL de TCM + antibióticos com uma inserção estéril no interior contendo 500 μL de TCM + antibióticos. Certifique-se de que a leitura do EVOM esteja <200 Ω. Registre esse valor em branco para usá-lo nos cálculos de resistência posteriores descritos na etapa 1.3.6.
- Remova a sonda do tubo de MTC + antibióticos. Reter este tubo para o armazenamento da sonda durante todo o experimento.
- Abaixe suavemente a sonda na primeira inserção com o eletrodo longo no poço coletor e o eletrodo curto dentro da inserção. Permita que o eletrodo longo toque o fundo do poço coletor, mas não empurre para baixo, pois isso pode perturbar a monocamada epitelial.
- Repita este processo para medir e registrar a resistência em Ohms (Ω) para cada inserção. Quando terminar, descontamine a sonda no etanol submergindo-a por mais 10-15 min. Em seguida, mova a sonda descontaminada de volta para a solução KCl para armazenamento. Subtraia a resistência em branco obtida na etapa 1.3.3 de cada valor obtido de cada pastilha que contenha células T84. Multiplique a resistência resultante (Ω) para cada pastilha pela área do fundo de cada pastilha (cm 2) para obter a medida final do TEER (Ω·cm2).
- Uma vez que o TEER atinja pelo menos 1.000 Ω·cm2, a monocamada epitelial está madura e pronta para ensaios de infecção.
- À medida que o TEER amadurece, forneça às células um meio fresco a cada 1-2 dias.
- Em uma nova placa de 24 poços, adicione 1 mL de MTC + antibióticos a um poço para cada inserção semeada que está sendo preparada.
- Usando pinças estéreis, transfira cuidadosamente as inserções para os poços recém-reabastecidos.
- Substitua a mídia nas inserções.
- Remova a mídia antiga das inserções inclinando a placa e usando um aspirador de vácuo interno para remover suavemente a mídia com uma ponta de pipeta ao longo do lado da inserção. O aspirador permite a regulação da sucção de baixo nível para evitar a ruptura das células. Não permita que a ponta da pipeta toque a parte inferior da pastilha, pois isso interromperá a monocamada epitelial em desenvolvimento.
- Adicione 500 μL de MTC + antibióticos às pastilhas. Visualize a monocamada com microscopia de luz para verificar se ela permanece intacta.
- A cada 1-2 dias, meça o TEER em cada pastilha, conforme descrito acima nas etapas 1.3.2-1.3.6.
2. Preparação das células T84 1 dia antes da experiência com MTC sem antibióticos
- Meça e registre o TEER no dia anterior ao experimento.
- Substitua o TCM da mesma maneira que durante a preparação e manutenção anteriores da célula. No entanto, a MTC sem antibióticos é usada em preparação para a infecção (1 mL no poço da placa e 500 μL na inserção).
3. Culturas de E. coli (iniciadas 1 dia antes do experimento)
CUIDADO: Use precauções de nível de biossegurança 2 (BSL-2) ao trabalhar com cepas clínicas patogênicas de E. coli.
- Pegue um tubo cônico marcado de 15 mL com 5 mL de caldo de lisogenia estéril (LB) e use uma alça estéril para inocular o caldo com uma colônia de uma cepa bacteriana (E. coli). Repita este processo, criando um tubo de cultura durante a noite para cada cepa a ser testada.
- Incubar a cultura noturna, com as tampas dos tubos soltas, em agitador de incubadora (250 rpm, 37 °C).
4. Preparação do inóculo de E. coli , células epiteliais e materiais (na manhã do experimento)
NOTA: Utilize MTC sem antibióticos aquecidos até 37 °C a partir deste ponto.
- Adicione 250 μL de cada cultura LB durante a noite a 25 mL de MTC sem antibióticos em um tubo cônico de 50 mL (um por cepa individual). Mantenha as tampas dos tubos soltas. Coloque estes novos tubos de cultura no agitador nas mesmas configurações (250 rpm, 37 °C) por exatamente 2 h. Execute as subetapas restantes enquanto aguarda.
- Meça o TEER em cada inserção, conforme descrito nas etapas 1.3.2-1.3.6. Registre-os como os TEERs no tempo (t) = 0 h.
- Mova as inserções para poços em uma nova placa e altere a mídia usando a técnica descrita na etapa 1.4.3. No entanto, desta vez, preencha os novos poços coletores com 500 μL de MTC sem antibióticos e preencha as inserções com 400 μL de MTC sem antibióticos. Mantenha as inserções dentro da incubadora de cultura de tecidos até o momento da infecção.
- Estabelecer um número suficiente de placas quadradas de ágar LB para aquecer à temperatura ambiente (RT) para posterior chapeamento e quantificação bacteriana.
5. Inoculação das células (início do experimento)
- Após exatamente 2 h, remova as culturas bacterianas matinais do agitador e centrifuga-as por 10 min (1.900 x g, 4 °C).
NOTA: Para todas as etapas a seguir, mantenha todas as suspensões bacterianas no gelo para minimizar o crescimento. - Ressuspeite a pastilha bacteriana na MTC sem antibióticos. Use um espectrofotômetro para ajustar a densidade óptica (OD) para 0,7-0,9 e diluir ainda mais com MTC sem antibióticos para uma concentração de 1 x 106 unidades formadoras de colônias (UFC)/mL (aproximadamente diluição 1:100). Use esta suspensão bacteriana para infectar cada inserção com 1 x 105 UFC por volume de 100 μL.
- Rotule a placa transwell e infecte cada inserção com 100 μL do inóculo ajustado por OD (total de 1 x 105 UFC por inserção). O ensaio já começou. Anote a hora e registre-a como t = 0 h.
- Chapear a suspensão bacteriana de inóculo para quantificar a UFC/mL utilizando o método de diluição da via, chapeando alíquotas de 10 μL em placas quadradas de ágar LB19.
6. Quantificação da transcitose
- A cada 30 minutos após a inoculação, encha novos poços com 500 μL de MTC sem antibióticos. Transfira as inserções para esses novos poços usando um conjunto diferente de pinças estéreis para cada cepa bacteriana diferente.
- Colete a mídia do poço coletor usado para cada inserção em tubos rotulados separados. Coloque esses tubos no gelo. Devolva as placas transwell para a incubadora entre os pontos de tempo.
- Para cada inserção, combine os meios coletados de t = 0,5 h, t = 1 h, t = 1,5 h e t = 2 h e vórtice brevemente. Chapear o meio coletado em placas de ágar LB utilizando o método de diluição da via para quantificar a quantidade de bactérias transcitadas nas primeiras 2 h do experimento.
NOTA: Recuperar as bactérias a cada 30 minutos e mantê-las no gelo garante que o crescimento bacteriano nos poços coletores seja minimizado e as medições sejam feitas em bactérias predominantemente transcitadas. - Coloque as placas de ágar LB de diluição de via marcadas na incubadora bacteriana a 37 °C sem CO2 suplementar e devolva a placa de transwell T84 à incubadora de cultura de tecidos.
- Em t = 4 h, repita o passo 6,3 combinando os meios coletados de t = 2,5 h, t = 3 h, t = 3,5 h e t = 4 h.
- Em t = 6 h, repita o passo 6,3 combinando os meios coletados de t = 4,5 h, t = 5 h, t = 5,5 h e t = 6 h. Além disso, em t = 6 h, chapear o meio dos poços de controle.
7. Fim do experimento
- Meça e registre o TEER ao final do experimento, t = 6 h. Use o procedimento descrito nas etapas 1.3.2-1.3.6.
- Descontamine a sonda submergindo-a em etanol a 70% por 10-15 min. Descarte as inserções ou salve/processe-as para aplicações adicionais, se desejar. Permitir que as placas de ágar LB incubem durante a noite e desinfetar e/ou eliminar com segurança todos os outros materiais utilizados.
- Após a incubação durante a noite, conte as colônias bacterianas manualmente nas placas LB de diluição da trilha para determinar a quantidade de inóculo e a quantidade de transcitose de E. coli. Certifique-se de que as placas de controle não mostram nenhum crescimento bacteriano.
Resultados
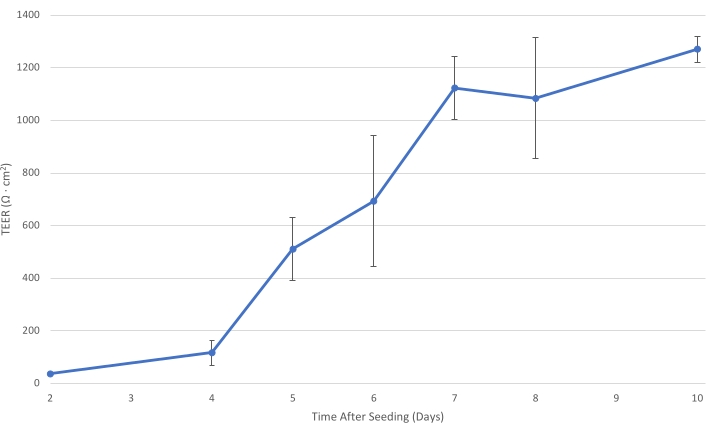
Figura 1: T84 TEER ao longo do tempo. À medida que a camada de células T84 amadurece na pastilha, a resistência elétrica da monocamada aumenta. Em um TEER de pelo menos 1.000 Ω·cm2, a camada celular está suficientemente desenvolvida para diminuir o transporte bacteriano paracelular e permitir a medição do trânsito bacteriano pri...
Discussão
Esse método é derivado de técnicas utilizadas em gastroenterologia e doenças infecciosas20. Modelos in vitro da barreira epitelial intestinal têm sido utilizados para elucidar os mecanismos pelos quais o conteúdo luminal interage com esse componente relevante da imunidade inata 6,8. As interações hospedeiro-patógeno de E. coli neonatal invasiva também têm sido caracterizadas separadamente por meio de análises ...
Divulgações
Nenhum.
Agradecimentos
Este trabalho foi apoiado por uma bolsa de estudante Sarah Morrison emitida pela Escola de Medicina da Universidade de Missouri-Kansas City para IA.
Materiais
| Name | Company | Catalog Number | Comments |
| 10,000 U/ mL Penicillin/Streptomycin Mixture | Fisher Scientific | 15-140-122 | |
| 15 mL sterile conical tubes | MidSci | C15B | |
| 2 mL microcentrifuge tubes | Avant | AVSS2000 | |
| 50 mL sterile polypropylene conical tubes | Falcon | 352070 | |
| Aspirator | Corning | 4930 | |
| Biosafety Cabinets | Labconco | 30441010028343 | Three of these are used in the method: one for sterile tissue work, one for infected tissue work, and one for bacterial work. |
| Centrifuge | Sorvall | Legend RT | |
| Disposable inoculation loops | Fisherbrand | 22363605 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-084 | |
| Epithelial Volt/Ohm Meter | World Precision Instruments | EVOM2 | |
| Fetal Bovine Serum | Fisher Scientific | 10437028 | |
| Ham's F-12 Nutrient Mixture | Gibco | 11765-047 | |
| Hemacytometer | Sigma Aldrich, Bright Line | Z359629 | |
| Incubator shaker | New Brunswick | Innova 4080 | |
| Incubators | Thermo Scientific | 51030284 | Three of these are used in the method: one for sterile tissue culturing, one for infected tissue culturing, and one for bacterial incubation. |
| Lysogeny broth | Difco | 244610 | |
| Lysogeny broth agar | IBI Scientific | IB49101 | |
| Nikon Eclipse TS2R Microscope | Nikon | ||
| Spectrophotometer | Unico | 1100RS | |
| T84 Intestinal Cells | American Tissue Culture Collection | CCL248 | |
| Tissue culture inserts, with polyethylene trephthalate membrane, 3 µm pores, 24 well format | Falcon | 353096 | |
| Tissue culture plate, 24 wells | Falcon | 353504 | |
| Trypan blue stain | Fisher Scientific | T10282 |
Referências
- Shakir, S. M., Goldbeck, J. M., Robison, D., Eckerd, A. M., Chavez-Bueno, S. Genotypic and phenotypic characterization of invasive neonatal Escherichia coli clinical isolates. American Journal of Perinatology. 31 (11), 975-982 (2014).
- Cole, B. K., et al. Route of infection alters virulence of neonatal septicemia Escherichia coli clinical isolates. PloS One. 12 (12), 0189032 (2017).
- Stoll, B. J., et al. Early-onset neonatal sepsis 2015 to 2017, the rise of Escherichia coli, and the need for novel prevention strategies. Journal of the American Medical Association Pediatrics. 174 (7), 200593 (2020).
- Dalgakiran, F., Witcomb, L. A., McCarthy, A. J., Birchenough, G. M., Taylor, P. W. Non-invasive model of neuropathogenic Escherichia coli infection in the neonatal rat. Journal of Visualized Experiments. (92), e52018 (2014).
- Williams, M., et al. Whole-genome sequencing-based phylogeny, antibiotic resistance, and invasive phenotype of Escherichia coli strains colonizing the cervix of women in preterm labor. BMC Microbiology. 21 (1), 330 (2021).
- Schoultz, I., Keita, &. #. 1. 9. 7. ;. The intestinal barrier and current techniques for the assessment of gut permeability. Cells. 9 (8), 1909 (2020).
- Devriese, S., et al. T84 monolayers are superior to Caco-2 as a model system of colonocytes. Histochemistry and Cell Biology. 148 (1), 85-93 (2017).
- Buckley, A., Turner, J. R. Cell biology of tight junction barrier regulation and mucosal disease. Cold Spring Harbor Perspectives in Biology. 10 (1), 029314 (2018).
- Awad, W. A., Hess, C., Hess, M. Enteric pathogens and their toxin-induced disruption of the intestinal barrier through alteration of tight junctions in chickens. Toxins. 9 (2), 60 (2017).
- Vancamelbeke, M., Vermeire, S. The intestinal barrier: A fundamental role in health and disease. Expert Review of Gastroenterology & Hepatology. 11 (9), 821-834 (2017).
- Aguilar, C., et al. Organoids as host models for infection biology - A review of methods. Experimental and Molecular Medicine. 53 (10), 1471-1482 (2021).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiology Spectrum. 9 (1), 0000321 (2021).
- Gavin, H. E., Beubier, N. T., Satchell, K. J. The effector domain region of the Vibrio vulnificus MARTX toxin confers biphasic epithelial barrier disruption and is essential for systemic spread from the intestine. PLoS Pathogens. 13 (1), 1006119 (2017).
- Kobayashi, H., et al. Aeromonas sobria serine protease decreases epithelial barrier function in T84 cells and accelerates bacterial translocation across the T84 monolayer in vitro. PloS One. 14 (8), 0221344 (2019).
- Kalischuk, L. D., Inglis, G. D., Buret, A. G. Campylobacter jejuni induces transcellular translocation of commensal bacteria via lipid rafts. Gut Pathogens. 1 (1), 2 (2009).
- Cole, B. K., Ilikj, M., McCloskey, C. B., Chavez-Bueno, S. Antibiotic resistance and molecular characterization of bacteremia Escherichia coli isolates from newborns in the United States. PloS One. 14 (7), 0219352 (2019).
- Cadena-Herrera, D., et al. Validation of three viable-cell counting methods: Manual, semi-automated, and automated. Biotechnology Reports. 7, 9-16 (2015).
- den Hartog, G., et al. Apurinic/apyrimidinic endonuclease 1 restricts the internalization of bacteria into human intestinal epithelial cells through the inhibition of Rac1. Frontiers in Immunology. 11, 553994 (2020).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. Biotechniques. 23 (4), 648-650 (1997).
- Lievin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: Lessons from cultured, fully differentiated human colon cancer cell lines. Microbiology and Molecular Biology Reviews. 77 (3), 380-439 (2013).
- Kaczmarek, A., Budzynska, A., Gospodarek, E. Detection of K1 antigen of Escherichia coli rods isolated from pregnant women and neonates. Folia Microbiologica. 59 (5), 419-422 (2014).
- Kalita, A., Hu, J., Torres, A. G. Recent advances in adherence and invasion of pathogenic Escherichia coli. Current Opinion in Infectious Diseases. 27 (5), 459-464 (2014).
- McCool, D. J., Marcon, M. A., Forstner, J. F., Forstner, G. G. The T84 human colonic adenocarcinoma cell line produces mucin in culture and releases it in response to various secretagogues. Biochemical Journal. 267 (2), 491-500 (1990).
- Resta-Lenert, S., Barrett, K. E. Enteroinvasive bacteria alter barrier and transport properties of human intestinal epithelium: Role of iNOS and COX-2. Gastroenterology. 122 (4), 1070-1087 (2002).
- Elatrech, I., et al. Escherichia coli LF82 differentially regulates ROS production and mucin expression in intestinal epithelial T84 cells: Implication of NOX1. Inflammatory Bowel Diseases. 21 (5), 1018-1026 (2015).
- El-Aouar Filho, R. A., et al. Heterogeneous family of cyclomodulins: Smart weapons that allow bacteria to hijack the eukaryotic cell cycle and promote infections. Frontiers in Cellular and Infection Microbiology. 7, 208 (2017).
- Hopkins, A. M., Walsh, S. V., Verkade, P., Boquet, P., Nusrat, A. Constitutive activation of Rho proteins by CNF-1 influences tight junction structure and epithelial barrier function. Journal of Cell Science. 116, 725-742 (2003).
- Shiou, S. R., et al. Erythropoietin protects intestinal epithelial barrier function and lowers the incidence of experimental neonatal necrotizing enterocolitis. Journal of Biological Chemistry. 286 (14), 12123-12132 (2011).
- Newburg, D. S., Ko, J. S., Leone, S., Nanthakumar, N. N. Human milk oligosaccharides and synthetic galactosyloligosaccharides contain 3’-, 4-, and 6'-galactosyllactose and attenuate inflammation in human T84, NCM-460, and H4 cells and intestinal tissue ex vivo. Journal of Nutrition. 146 (2), 358-367 (2016).
- Burns, J. L., Griffith, A., Barry, J. J., Jonas, M., Chi, E. Y. Transcytosis of gastrointestinal epithelial cells by Escherichia coli K1. Pediatric Research. 49 (1), 30-37 (2001).
- Raut, B., Chen, L. J., Hori, T., Kaji, H. An open-source add-on EVOM((R)) device for real-time transepithelial/endothelial electrical resistance measurements in multiple transwell samples. Micromachines. 12 (3), 282 (2021).
- McCarthy, A. J., Stabler, R. A., Taylor, P. W. Genome-wide identification by transposon insertion sequencing of Escherichia coli K1 genes essential for in vitro growth, gastrointestinal colonizing capacity, and survival in serum. Journal of Bacteriology. 200 (7), 00698 (2018).
- Sayoc-Becerra, A., et al. The JAK-inhibitor tofacitinib rescues human intestinal epithelial cells and colonoids from cytokine-induced barrier dysfunction. Inflammatory Bowel Diseases. 26 (3), 407-422 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados