É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem de células vivas de cérebros de larvas de terceiro ínstar de Drosophila melanogaster
Neste Artigo
Resumo
Aqui, discutimos um fluxo de trabalho para preparar, dissecar, montar e obter imagens de cérebros de explantes vivos de larvas de terceiro ínstar de Drosophila melanogaster para observar a dinâmica celular e subcelular sob condições fisiológicas.
Resumo
As células-tronco neurais de Drosophila (neuroblastos, RNs doravante) sofrem divisões assimétricas, regenerando o neuroblasto auto-renovador, ao mesmo tempo em que formam uma célula-mãe do gânglio diferenciador (GMC), que sofrerá uma divisão adicional para dar origem a dois neurônios ou glia. Estudos em RNs descobriram os mecanismos moleculares subjacentes à polaridade celular, orientação fusiforme, auto-renovação e diferenciação de células-tronco neurais. Essas divisões celulares assimétricas são facilmente observáveis através de imagens de células vivas, tornando os RNs larvais ideais para investigar a dinâmica espaço-temporal da divisão celular assimétrica em tecidos vivos. Quando devidamente dissecados e imageados em meio suplementado com nutrientes, os RNs em cérebros explantes dividem-se robustamente por 12-20 h. Os métodos descritos anteriormente são tecnicamente difíceis e podem ser desafiadores para aqueles que estão começando na área. Aqui, um protocolo é descrito para a preparação, dissecção, montagem e imagem de explantes cerebrais de larvas de terceiro ínstar vivas usando suplementos de corpo gorduroso. Problemas potenciais também são discutidos, e exemplos são fornecidos de como essa técnica pode ser usada.
Introdução
A divisão celular assimétrica (DCA) é o processo pelo qual componentes subcelulares como RNA, proteínas e organelas são particionados desigualmente entre as células filhas 1,2. Esse processo é comumente visto em células-tronco, que sofrem DCA para dar origem a células-filhas com diferentes destinos de desenvolvimento. Drosófila Os RNs dividem-se assimetricamente para produzir um RN, que retém sua caule, e uma célula-mãe ganglionar (GMC). O GMC sofre novas divisões para produzir neurônios diferenciadores ou glia3. RNs em divisão assimétrica são abundantes no cérebro em desenvolvimento de larvas de terceiro ínstar, que são prontamente observados por microscopia. No estágio larval de terceiro ínstar, há cerca de 100 RNs presentes em cada lobo cerebral central3,4,5,6.
A divisão celular assimétrica é um processo altamente dinâmico. Protocolos de imagem de células vivas têm sido utilizados para medir e quantificar a dinâmica da polaridade celular 7,8,9,10, a orientação do fuso 11,12,13, a dinâmica do córtex da actomiosina14,15,16,17,18, a biologia dos microtúbulos e centrossomos 19,20,21,22,23,24,25,26,27, e membrana 10,28 e dinâmica da cromatina29. Descrições qualitativas e quantitativas de DCA baseiam-se em métodos e protocolos robustos para a divisão de imagens de RNs em cérebros vivos intactos. O protocolo a seguir descreve métodos para preparar, dissecar e obter imagens de cérebros de larvas de terceiro ínstar para imagens de células vivas in vivo usando duas abordagens de montagem diferentes. Esses métodos são mais adequados para pesquisadores interessados na dinâmica espaço-temporal de divisões de células-tronco, bem como divisões em outras células cerebrais, pois permitem observações de curto e longo prazo de eventos celulares. Além disso, essas técnicas são facilmente acessíveis aos recém-chegados ao campo. Nós demonstramos a eficácia e adaptabilidade desta abordagem com cérebros de larvas expressando microtúbulos fluorescentemente marcados e proteínas de fusão cortical. Além disso, discutimos métodos de análise e considerações para aplicação em outros estudos.
Protocolo
OBS: A Figura 1 mostra os materiais necessários para a realização deste estudo.
1. Considerações e preparativos para a experiência
- Evitar que as larvas se superlotem.
NOTA: A qualidade do cérebro das larvas de explantes está diretamente relacionada com a saúde e qualidade das larvas antes da dissecção. Larvas desnutridas por superlotação geralmente produzem cérebros de baixa qualidade30.- Certifique-se de que não mais do que 20-30 larvas estão presentes por prato de tampa de refeição para evitar a desnutrição. Exemplos disso podem ser vistos na Figura 2.
- Filtre e alíquota o meio de Schneider antes de usar.
- Para cada dissecção, preparar imagens frescas e meio de dissecção suplementando o meio de inseto de Schneider com 1% de soro de crescimento bovino (BGS). Um volume de 5 mL de dissecção e meio de imagem geralmente é suficiente para um experimento de imagem.
- Aquecer o meio suplementado à temperatura ambiente (TR) antes de usar.
- Considere a duração do filme a ser coletado e use-a para fatorar a suplementação do meio de imagem, a abordagem de montagem e as configurações de aquisição do microscópio.
NOTA: Em condições ideais, os RNs em cérebros suplementados apenas com BGS se dividirão robustamente por mais de 3 h.- Complemente o meio de imagem adicionando tecidos corporais de gordura larval ao meio de imagem para suportar divisões após 4 h ao conduzir experimentos que exigem filmes mais longos.
NOTA: Os corpos gordos secretam mitógenos que suportam a proliferação NB31, e os corpos gordurosos inteiros de 10 larvas são suficientes para suportar quatro a cinco cérebros. Além disso, amostras fotografadas com uma lâmina ligada à membrana foram mostradas para se dividir por mais de 10 h13,32, enquanto amostras fotografadas com uma lâmina de vários poços geralmente se dividem com menos frequência (observações não publicadas). - Alternativamente, implemente um meio de imagem mais complexo para filmes mais longos, como descrito anteriormente33. Minimize os fotodanos ajustando o tempo de exposição, a potência do laser e a frequência de amostragem para obter melhores resultados.
- Complemente o meio de imagem adicionando tecidos corporais de gordura larval ao meio de imagem para suportar divisões após 4 h ao conduzir experimentos que exigem filmes mais longos.
2. Estadiamento e coleta das larvas (Figura 2)
- Cruzar moscas virgens fêmeas de 1-5 dias com moscas adultas adultas de 1-7 dias para produzir progênie com o genótipo desejado. Para um rendimento ideal, cruze 10-15 virgens fêmeas com 5-10 machos. Deposite essas moscas em uma gaiola de moscas com uma tampa de refeição (Figura 2A-C) e incube a 25 °C.
- Troque a tampa da refeição diariamente. Isso evita que as tampas das refeições fiquem superlotadas de larvas, o que reduz a qualidade dos cérebros dissecados.
- Se a tampa da refeição estiver significativamente coberta de larvas (ou seja, >30), divida essa tampa de refeição ao meio e substitua uma metade por uma tampa de refeição fresca que também tenha sido cortada ao meio. Alternativamente, troque as tampas das refeições com mais frequência (ou seja, a cada 12 h em vez de a cada 24 h). Exemplos de tampas de refeição superpovoadas podem ser vistos na Figura 2E, F.
- Incubar a tampa da farinha com as larvas a 25 °C até que as larvas atinjam a idade desejada.
3. Dissecção do corpo adiposo larval (Figura 3)
NOTA: Este protocolo descreve dissecações usando uma placa de dissecção de 3 poços.
- Pipetar ~400 μL de dissecção e meio de imagem em cada poço de uma placa de dissecção de 3 poços.
- Lave dez larvas selvagens de 72-96 h bem alimentadas, segurando-as suavemente com pinças de dissecção e mergulhando-as dentro e fora da solução de dissecção no poço mais inferior até que todas as partículas alimentares tenham sido lavadas. Após o enxágue, mova as larvas limpas para o meio bem.
- Usando uma pinça, segure a larva pelos ganchos da boca. Com o outro conjunto de pinças, rompa um lado da cutícula da larva.
- Essa ruptura fará com que os corpos gordurosos se espalhem para fora da larva. Os corpos gordurosos são esbranquiçados e semitranslúcidos e terão estrutura em forma de rede (Figura 3I). Os corpos gordos também tenderão a grudar em si mesmos e nas pinças de dissecção. Uma vez identificado, colete o máximo de gordura possível de cada larva e transfira-o com a pinça para o poço superior com 400 μL de meio de dissecção RT.
4. Dissecção cerebral larval (Figura 3)
- Lavar as larvas experimentais em dissecção e meio de imagem como acima para libertá-las de resíduos alimentares. Para melhores resultados, evite armazenar larvas não dissecadas na solução de dissecção. Isso fará com que as larvas "se afoguem" e afetará negativamente a qualidade dos cérebros dissecados.
- Usando uma pinça, segure a larva pelos ganchos da boca. Com outro conjunto de pinças, corte/rasgue suavemente aproximadamente 1/3 da larva do lado posterior (Figura 3A). Isso fará com que elementos do trato digestivo, corpos adiposos, tecido conjuntivo e sistema nervoso "estourem" do lado rompido da larva (Figura 3B).
- Usando uma pinça, segure a larva pelos ganchos da boca. Com o outro conjunto de pinças, escove suavemente a cutícula em direção aos ganchos da boca enquanto "empurra" para dentro com a pinça segurando os ganchos da boca até que toda a larva seja virada do avesso. Esse movimento é semelhante a virar uma meia "do avesso" (Figura 3C, D).
- Inverta a larva para que o sistema nervoso central e outros tecidos fiquem voltados para fora enquanto ainda estão conectados à cutícula. Nesta etapa, localize o sistema nervoso central (SNC) para evitar a remoção acidental. Com o uso de uma pinça, remova suavemente todo o tecido não pertencente ao SNC, deixando apenas o SNC e o cérebro presos à cutícula (Figura 3E).
- O cérebro será ligado à cutícula através de conexões axonais. Usando tesouras de microdissecção, corte essas conexões axonais para liberar o cérebro da cutícula. Para fazer isso, primeiro corte suavemente abaixo dos lobos cerebrais (Figura 3F). Repita com as conexões sob o cordão nervoso ventral.
NOTA: Esta etapa pode ser feita com pinças se a tesoura de microdissecção não estiver disponível. Tome cuidado especial ao usar pinças para garantir que o tecido cerebral não seja esticado demais durante a remoção da cutícula, porque o estresse mecânico afetará negativamente a saúde do cérebro. - Transfira o cérebro dissecado para um poço com dissecção e meio de imagem. Para experimentos de imagem com mais de 3 h, use dissecção e meio de imagem suplementado com corpos gordurosos como descrito acima. Dissecar as larvas em lotes para manter o tempo de dissecção inferior a 20 min.
5. Montagem e geração de imagens (Figura 4)
- Para aquisição de imagens com uma lâmina34 ligada à membrana:
- Coletar cérebros dissecados e corpos gordurosos isolados no último poço da placa de dissecação.
- Monte metade da lâmina colocando uma membrana permeável a gás sobre a parte de trás da lâmina e pressione o anel dividido no centro, mantendo-o no lugar (Figura 4A-C).
- Usando uma micropipeta de 200 μL, transfira até 10 cérebros dissecados e o máximo de corpo gordo possível (veja acima) em ~130-140 μL de dissecção e meio de imagem para a membrana. Certifique-se de depositar o meio com as amostras no centro da membrana permeável a gás (Figura 4D, E).
- Orientar os cérebros para a população de RNs a serem imageados e para o tipo de microscópio a ser utilizado (Figura 4E). Posicione a amostra o mais próximo possível da objetiva do microscópio. Por exemplo, para obter imagens de RNs nos lobos cerebrais centrais, oriente os cérebros de tal forma que os lobos cerebrais estejam mais próximos da objetiva (Figura 4H).
- Uma vez que os cérebros estão orientados, coloque suavemente uma tampa de vidro em cima da solução na membrana. Isso fará com que a solução contendo os cérebros e corpos gordurosos se espalhe por toda a membrana (Figura 4F).
- Limpe a solução excessiva segurando um tecido de laboratório perto da borda da lamínula. A quantidade ideal de solução é alcançada quando os cérebros tocam a lamínula sem serem esmagados. Se a reorientação for necessária nesta etapa, mova cuidadosamente a tampa para mover os cérebros.
- Imobilize a lamínula aplicando vaselina derretida ao longo das bordas da lamínula com um pincel. Deixe a geleia solidificar (Figura 4G).
- Para aquisição de imagens com uma lâmina de imagem de vários poços (Figura 4):
- Adicionar 400 μL de meio de imagem a um poço de uma lâmina de vários poços (no experimento aqui realizado, uma micro[μ]-lâmina de câmara de 8 poços foi usada; Figura 4I). Transferir os corpos gordurosos previamente dissecados para este poço (ver passo 3.4).
- Deposite até 10 cérebros em um aglomerado próximo ao centro do poço (Figura 4J).
- Orientar os cérebros para a população de RNs a serem imageados e para o tipo de microscópio utilizado, conforme descrito no passo 5.1.4 (Figura 4K). Organize as amostras de modo que fiquem próximas umas das outras. Isso minimizará a distância que o estágio deve se mover entre as amostras, o que reduz a deriva da amostra durante a aquisição.
- Uma vez que os cérebros foram orientados no poço, permita que os cérebros se acomodem por 2-5 min. Isso aumenta sua estabilidade durante o transporte/imagem. Preparar o microscópio para aquisição durante este tempo.
- Cubra a μ-slide com a tampa da lâmina e transfira-a para o microscópio. Comece a aquisição com a menor potência de laser e tempo de exposição possível para minimizar o fotoclareamento.
6. Melhores práticas de processamento e gerenciamento de dados
- Processar os dados conforme necessário de acordo com o software de análise disponível.
- Para o exemplo mostrado aqui, salve os dados adquiridos com o software SlideBook como um arquivo de imagem do SlideBook (.sld).
- Para converter em tipo de arquivo proprietário do Ismiris (.ims) usando o Imaris File Converter, abra o Imaris File Converter em uma janela separada. Clique e arraste os arquivos .sld para a seção "entrada" do Imaris File Converter.
- Determine o local de saída desejado para os arquivos convertidos e clique em "Iniciar tudo".
- Após a conversão, visualize e anote os dados no software Imaris.
NOTA: Alternativas para análise de imagens podem ser usadas no lugar de Imaris, como Fiji (https://hpc.nih.gov/apps/Fiji.html), Aivia (https://www.aivia-software.com/), Volocity (https://www.volocity4d.com/), ou outros.
- Retenha o máximo possível dos dados originais para manter registros adequados. Por exemplo, se o software de aquisição for salvo em um formato de arquivo, mas for convertido em um formato diferente para análise, mantenha a versão adquirida dos dados.
- Para análise de dados, mantenha um registro do maior número possível de detalhes sobre a amostra e as configurações de aquisição. As principais informações a serem retidas, incluem o genótipo das larvas dissecadas, a idade das larvas antes da dissecção, o estado da tampa da refeição em que foram criadas, a potência do laser usada durante a imagem, o tempo de exposição, o tempo de aquisição e a resolução temporal.
7. Exemplo de quantificação do comprimento do ciclo celular (Figura 5)
NOTA: neste exemplo, larvas expressando o marcador de polaridade Pins (Pins::EGFP16) e a proteína de ligação a microtúbulos Júpiter25 (cereja::Júpiter13) foram fotografadas. A análise subsequente foi realizada utilizando-se o software Imaris.
- Abra o filme usando o software de análise de imagem de sua escolha. Percorra a duração do filme para identificar NBs em divisão e rotule-os para referência futura. Identificar os RNs em divisão por seus distintos fusos mitóticos (Figura 5C-E).
- Identifique um estágio do ciclo celular de referência para determinar o comprimento do ciclo celular. Neste exemplo, a metáfase é usada como referência.
- Determine manualmente o número de quadros entre metáfases sucessivas e converta-o em minutos ou horas para determinar o tempo necessário para completar um ciclo celular.
- Faça isso pegando a resolução temporal do filme e multiplicando-a pelo número de quadros entre as metáfases. Por exemplo, se a resolução temporal do filme é de um quadro a cada 5 min, e as metáfases são observadas no quadro 13 e no quadro 35, o tempo entre essas metáfases seria de 110 min ([35 − 13] × 5).
- Plote os dados com qualquer software apropriado. Os dados aqui apresentados foram plotados utilizando o software PRISM.
8. Exemplo de quantificação do alinhamento do fuso celular (Figura 5)
Observação : neste exemplo, a análise é executada usando o software Imaris.
- Abra o arquivo de filme no Imaris ou em outro software de sua preferência. Percorra a duração do filme para identificar NBs em divisão e rotule-os para referência futura.
- Determinar o vetor formado pelos polos do fuso utilizando os centrossomos apicais e basais (representados por m), da seguinte forma:
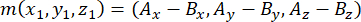
onde Ax, Ay e Az são as coordenadas do centrossomo apical, e Bx, By e Bz são as coordenadas do centrossomo basal. Da mesma forma, o eixo do vetor de divisão (representado por n) é formado pelo ponto médio dos pinos apicais::EGFP crescente e o córtex basal: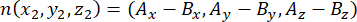
onde Ax, Ay e Az são as coordenadas do ponto médio dos Pinos::EGFP crescente, e Bx, By e Bz são as coordenadas do ponto médio do córtex basal. - Determine a magnitude dos vetores m e n:
Magnitude de m: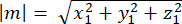
Magnitude de n: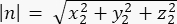
- Determine o produto ponto (representado por k) de m e n:
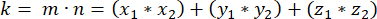
- Usando o produto ponto k e as magnitudes vetoriais m e n, determine o ângulo entre os vetores:
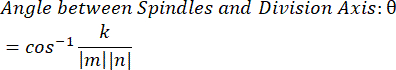
- Plote os dados no software de escolha. Os dados aqui apresentados foram preparados no Microsoft Excel e visualizados no PRISM.
Resultados
Dissecção e imagem de NBs do lobo cerebral central expressando Pins::EGFP e Cherry::Jupiter
Para mostrar este protocolo, larvas expressando Cherry::Jupiter13 e endogenamente marcadas Pins::EGFP16 (w; worGal4, UAS-cherry::jupiter/CyO; Pinos::EGFP/TM6B, Tb) foram fotografados por 4 h usando o protocolo descrito usando lâminas de imagem de múltiplos poços (Figura 5C,D). Dados adicionais foram obtidos de la...
Discussão
Este protocolo descreve uma abordagem para a obtenção de imagens de cérebros vivos de explantes de larvas de Drosophila melanogaster . O protocolo aqui descrito permite que cérebros de explantes sejam observados por 12-20 h sob as condições experimentais adequadas. Atenção especial deve ser dada à preparação das amostras e ao planejamento dos experimentos desejados. Como mencionado acima, um dos fatores mais críticos que determinam a qualidade do tecido dissecado é a saúde das larvas. Para alcança...
Divulgações
Os autores não têm divulgações financeiras a declarar.
Agradecimentos
Esta pesquisa é apoiada por R35GM148160 (C. C.) e um National Institutes of Health (NIH) Training Grant T32 GM007270 (R. C. S)
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
Referências
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados