Method Article
Отображение структурно-функциональных отношений неупорядоченных онкогенных факторов транскрипции с использованием транскриптомного анализа
В этой статье
Резюме
Внутренне неупорядоченные домены важны для функции фактора транскрипции онкогенного слияния. Чтобы терапевтически нацелиться на эти белки, требуется более подробное понимание регуляторных механизмов, используемых этими доменами. Здесь мы используем транскриптомику для отображения важных структурных особенностей внутренне неупорядоченного домена EWS в саркоме Юинга.
Аннотация
Многие виды рака характеризуются хромосомными транслокациями, которые приводят к экспрессии онкогенных факторов транскрипции слияния. Как правило, эти белки содержат внутренне неупорядоченный домен (IDD), слитый с ДНК-связывающим доменом (DBD) другого белка, и организуют широко распространенные транскрипционные изменения для содействия злокачественности. Эти слияния часто являются единственной повторяющейся геномной аберрацией в раковых заболеваниях, которые они вызывают, что делает их привлекательными терапевтическими мишенями. Тем не менее, нацеливание на онкогенные факторы транскрипции требует лучшего понимания механистической роли, которую низкосложные IDD играют в их функции. N-концевой домен EWSR1 представляет собой IDD, участвующий в различных факторах транскрипции онкогенного синтеза, включая EWS / FLI, EWS / ATF и EWS / WT1. Здесь мы используем РНК-секвенирование для исследования структурных особенностей домена EWS, важного для транскрипционной функции EWS/FLI в саркоме Юинга. Впервые выполнено шРНК-опосредованное истощение эндогенного слияния из клеток саркомы Юинга в паре с эктопической экспрессией различных EWS-мутантных конструкций. Затем РНК-секвенирование используется для анализа транскриптомов клеток, экспрессирующих эти конструкции, для характеристики функционального дефицита, связанного с мутациями в домене EWS. Интегрируя транскриптомный анализ с ранее опубликованной информацией о мотивах связывания ДНК EWS / FLI и геномной локализации, а также функциональными анализами на трансформирующую способность, мы смогли идентифицировать структурные особенности EWS / FLI, важные для онкогенеза, и определить новый набор генов-мишеней EWS / FLI, критически важных для саркомы Юинга. В данной работе демонстрируется использование РНК-секвенирования в качестве метода картирования структурно-функциональных отношений внутренне неупорядоченного домена онкогенных факторов транскрипции.
Введение
Подмножество раковых заболеваний, включая многие злокачественные новообразования в детском и подростковом возрасте, характеризуется хромосомными транслокациями, которые генерируют новые онкогены слияния1,2,3,4,5,6. Полученные в результате слияния белки часто функционируют как онкогенные факторы транскрипции, организуя широко распространенные изменения в транскрипционной регуляции для содействия опухолевомугенезу 7,8. Раковые заболевания с этими транслокациями обычно обладают спокойным мутационным ландшафтом, с небольшим количеством повторяющихся геномных аберраций, кроме патогномонального слияния4,9. Таким образом, непосредственное нацеливание на слитый белок является привлекательной терапевтической стратегией при этих заболеваниях. Однако эти онкогенные факторы транскрипции обычно состоят из низкосложного, внутренне неупорядоченного, транскрипционно активирующего домена, слитого с ДНК-связывающимдоменом (DBD)10,11,12,13,14. Как внутренне неупорядоченные домены (IDD), так и DBD этих белков оказались трудными для нацеливания с помощью обычных фармакологических подходов. Поэтому разработка новых терапевтических подходов требует более детального молекулярного понимания механизмов, используемых этими слияниями для аберрантного регулирования экспрессии генов.
N-концевая часть IDD EWSR1 обычно сливается с DBD при раке, включая EWS / FLI в саркоме Юинга, EWS / WT1 в диффузной мелкоклеточной опухоли и EWS / ATF1 в прозрачной клеточной саркоме мягких частей10. Механистическая роль EWS IDD в каждом из этих слияний не до конца понятна. Семейство слияний EWS/ETS, в частности EWS/FLI, является наиболее функционально охарактеризованным на сегодняшний день. EWS/FLI координирует общегенетные эпигенетические и транскрипционные изменения, приводящие к активации и подавлению тысяч генов7,11,15,16. Исследования показали, что IDD важен для набора как транскрипционных коактиваторов (таких как p300, WDR5 и комплекс BAF), так и ко-репрессоров (таких как комплекс NuRD)11,15,17. Слияние EWS IDD с C-концевой частью FLI1 придает новую ДНК-специфичность ETS DBD FLI1, так что слияние онкопротеина (EWS / FLI) связывается с повторяющимися GGAA-микросателлитными областями генома в дополнение к консенсусному мотиву ETS18,19,20. В сочетании с функцией рекрутирования коактиваторов эта возникающая ДНК-связывающая активность EWS/FLI способствует образованию de novo энхансеров в GGAA-микроспутниках дистальнее к стартовым участкам транскрипции (TSS) («энхансероподобные» микроспутники) и рекрутирует РНК-полимеразу II для содействия транскрипции на GGAA-микроспутниках, расположенных проксимально к TSS («промотороподобные» микроспутники)11,15,16,21.
Взятые вместе, эти данные привели нас к гипотезе о том, что дискретные элементы в области EWS способствуют набору различных сорегуляторов к различным типам сайтов связывания EWS / FLI. Тем не менее, распознавание этих элементов в части EWS EWS / FLI и то, как они функционируют, было затруднено очень повторяющимся и неупорядоченным характером области. Здесь мы используем ранее опубликованную систему спасения нокдауна в клетках саркомы Юинга для функционального отображения этих элементов в EWS IDD. В этой системе EWS/FLI истощается с помощью шРНК, нацеленной на 3'UTR гена FLI1, и экспрессия спасается с помощью различных конструкций МУТАНТНОЙ кДНК EWS/FLI, в которых отсутствуют 3'UTR7,17,22. Эти эксперименты были сосредоточены на конструкциях с различными делециями для отображения структурно-функциональной взаимосвязи между EWS IDD и важными онкогенными фенотипами, включая активацию репортерной конструкции GGAA-микросателлита, анализы формирования колоний и целевую валидацию EWS/FLI-активированных и -репрессированных генов7,17,22 . Однако эти исследования не смогли найти дискретные поддомены в EWS IDD в EWS / FLI, которые уникально важны либо для активации, либо для подавления. Все протестированные конструкции были либо способны активировать и подавлять специфические гены-мишени, что приводит к эффективному формированию колоний, либо не могли регулировать ни один из генов-мишеней EWS / FLI, что приводило к потере колониеобразования7,17,22.
Транскриптомный анализ, обеспечиваемый широким внедрением секвенирования следующего поколения, обычно используется для сравнения сигнатур экспрессии генов в двух условиях, часто в контексте скрининга или описательных исследований. Вместо этого мы хотели использовать возможность захвата данных экспрессии всего генома с использованием РНК-секвенирования (RNA-seq) для характеристики вклада IDD в функцию транскрипционного фактора. В этом случае RNA-seq сопряжен с системой нокдауна-спасения для изучения структурно-функциональной взаимосвязи домена EWS. Этот подход применим к другим факторам транскрипции слияния, включая другие слияния EWS или факторы транскрипции дикого типа с плохо изученной функцией, и имеет множество преимуществ по сравнению с другими анализами, используемыми для исследований функционального картирования, такими как репортерные анализы или целевая qRT-ПЦР. К ним относятся тестирование структурных детерминант функции в соответствующем контексте хроматина, способность тестировать несколько типов элементов ответа в одном анализе (т.е. активированные и репрессированные, GGAA-микросателлит и немироспутник и т.д.) и результирующая способность лучше обнаруживать частичную функцию.
Успешная реализация этого подхода зависит от клеточной системы, которая захватывает интересующие фенотипы (в данном случае клетки A673 с shRNA-опосредованным истощением EWS/FLI), и панели мутантных конструкций в векторе экспрессии, подходящем для клеточной системы (в данном случае pMSCV-гигро с различными мутантами EWS/FLI с тегами 3x-FLAG, которые будут доставлены ретровирусной трансдукцией). Вирусная трансдукция либо конструкций истощения на основе CRISPR, либо конструкций истощения на основе шРНК и конструкций экспрессии кДНК с соответствующим отбором для генерации стабильных клеточных линий, рекомендуется по сравнению с транзиторной трансфекцией. Последующая интерпретация результатов усиливается, когда транскриптомные данные могут быть сопряжены с другими данными, связанными с локализацией транскрипционного фактора и другими фенотипическими показаниями, если таковые имеются.
В данной работе мы применяем данный подход для характеристики активности мутанта DAF EWS/FLI14. Мутант DAF имеет 17 мутаций тирозина в аланин в повторяющихся областях EWS IDD EWS / FLI14. Этот конкретный мутант EWS был ранее зарегистрирован и не может активировать экспрессию репортерного гена при слиянии с ATF1 DBD14. Однако предварительные данные qRT-PCR свидетельствуют о том, что этот мутант смог активировать транскрипцию мишени EWS/FLI NR0B123. Описанный здесь транскриптомический подход позволил успешно обнаружить частичную функцию мутанта DAF. Связывая эти транскриптомные данные с информацией о мотивах связывания и распознавания EWS/FLI, мы также показываем, что мутант DAF сохраняет функцию при GGAA-микроспутниковых повторах. Эти результаты идентифицируют DAF как первый частично функциональный мутант EWS/FLI и выделяют функцию в немироспутниковых генах как важную для онкогенеза (как сообщалось23). Это демонстрирует силу этого транскриптомного подхода к картированию структуры и функции для обеспечения понимания функции онкогенных факторов транскрипции.
протокол
1. Установка панели конструкций in vitro
ПРИМЕЧАНИЕ: Этот шаг будет варьироваться в зависимости от конкретного белка, подлежащего анализу.
- Подготовьте аликвоты вируса к истощению и экспрессии конструкций по мере необходимости.
- Засейте 10 см чашку для посева тканей 3-5 x 106 клетками HEK293-EBNA или HEK293T для каждой конструкции, необходимой для вирусной трансдукции. Пусть клетки прилипают в течение ночи в модифицированной среде Dulbecco Eagle Media (DMEM), дополненной 10% фетальной бычьей сывороткой (FBS), пенициллином / стрептомицином / глютамином (P / S / Q) и 0,3 мг / мл G418.
ПРИМЕЧАНИЕ: Клетки HEK293-EBNA и HEK293T рекомендуются для вирусного производства, потому что они легко растут, имеют высокую эффективность трансфекции и эффективно экспрессируют рекомбинантные белки из эписомальных плазмид. Клетки должны быть между 50-70% сливающимися в день трансфекции. - Подготовьте трансфекционную смесь для каждой конструкции вирусной трансдукции. Соедините 2 мл восстановленной сывороточной среды с 90 мкл трансфекционного реагента.
ПРИМЕЧАНИЕ: Рекомендуется предварительное нагревание восстановленных сывороточных сред. - Добавьте по 10 мкг к трансфекционной смеси плазмиды вирусной упаковки (например, gag-pol), плазмиды вирусной оболочки (например, VSV-G) и одной из них либо истощения на основе CRISPR, либо истощения на основе шРНК, либо конструкции экспрессии кДНК (например, pMKO или pMSCV). Хорошо перемешать мягким пипетированием.
- Дайте трансфекционной смеси постоять в течение 20 минут при комнатной температуре. Удалите питательную среду HEK293-EBNA из блюд для посева тканей и добавьте 3 мл DMEM, дополненных 10% FBS, P / S / Q и 10 мМ пирувата натрия. К каждому блюду добавить по 2 мл трансфекционной смеси по каплям. Пусть клетки сидят в трансфекционных средах на ночь в инкубаторе при 37 °C и 5% CO2.
- На следующее утро добавьте 20 мл среды DMEM с 10% FBS, добавками P / S / Q и 10 мМ пирувата натрия. Инкубируют клетки в нем при 37 °C и 5% CO2 на ночь.
- На следующее утро замените среду 5 мл вирусной коллекционной среды (VCM) (DMEM, дополненный 10% инактивированным теплом FBS, P / S / Q и 20 мМ HEPES).
- Через 4 ч соберите VCM с пластин и храните в конической трубке объемом 50 мл на льду при 4 °C. Заменить 5 мл свежего VCM.
- Через 4 ч собрать VCM с пластин в ту же коническую трубку объемом 50 мл и хранить на льду при 4 °C. Замените 8 мл свежего VCM для ночного сбора.
- Утром собирают ВКМ с пластин и хранят в конической трубке объемом 50 мл на льду при 4 °C. Заменить 5 мл свежего VCM.
- Через 4 ч соберите VCM с пластин и храните в конической трубке объемом 50 мл на льду при 4°C. Заменить 5 мл свежего VCM. Через 4 ч соберите ВСМ с пластин и добавьте в коническую трубку объемом 50 мл.
- Aliquot собирает из 50 мл трубки в криотрубки (2 мл на аликвоту) после фильтрации через фильтр 0,45 мкм. Хранить вирусные аликвоты при -80 °C до использования.
ПРИМЕЧАНИЕ: Здесь протокол можно приостановить, а вирусные аликвоты можно хранить до готовности к использованию.
- Засейте 10 см чашку для посева тканей 3-5 x 106 клетками HEK293-EBNA или HEK293T для каждой конструкции, необходимой для вирусной трансдукции. Пусть клетки прилипают в течение ночи в модифицированной среде Dulbecco Eagle Media (DMEM), дополненной 10% фетальной бычьей сывороткой (FBS), пенициллином / стрептомицином / глютамином (P / S / Q) и 0,3 мг / мл G418.
- Семена клеток соответствующей плотности в чашке для культивирования тканей 10 см. Целевой показатель 50%-го слияния. Дайте клеткам прилипнуть в течение ночи, поместив в инкубатор при 37 °C, содержащий 5% CO2.
ПРИМЕЧАНИЕ: Для клеток A673 это 5 x 106 клеток в 10 мл среды DMEM с 10% FBS, добавками P / S / Q и 10 мМ пирувата натрия. Эти условия могут варьироваться в зависимости от скорости роста используемых клеток. - Истощают эндогенный фактор интереса. Если клеткам не нужно истощать эндогенный белок, представляющий интерес, перейдите к шагу 1.4.
- Разморозить вирусную аликвоту для трансдукции шРНК или конструкции CRISPR, нацеленной на интересующий белок. Быстро разморозьте замороженные аликвоты на водяной бане при температуре 37 °C.
- Добавьте 2,5 мкл 8 мг/мл полибрена к каждой вирусной аликвоте и перемешайте путем мягкого пипетирования. Удалите носители с пластин клеток и аккуратно добавьте вирусную аликвоту к пластине 10 см путем пипетки вдоль боковой стороны пластины. Покачайте пластину, чтобы распространить 2 мл вирусной аликвоты.
- Инкубировать при 37 °C в инкубаторе культуры тканей в течение 2 ч. Раскачивайте пластину каждые 30 минут, чтобы предотвратить высыхание любых областей пластины.
- Добавьте 5 мл среды DMEM с 10% FBS, добавкой P / S / Q и 10 мМ пирувата натрия с 5 мкл 8 мг / мл полибрена. Дайте клеткам высиживаться в течение ночи.
- Утром удаляют среду из клеток и пропускают клетки в среды, дополненные селекционным реагентом. При прохождении клеток засейте их таким образом, чтобы они росли в течение 48-72 ч и достигали 50% слияния.
ПРИМЕЧАНИЕ: Для клеток A673 с pSRP-iEF-2 клетки засевают в расщеплении 1:5 и отбирают в течение 72 ч с 2 мкг/мл пуромицина.
- Трансдукция конструкций выражений кДНК.
- Проверьте клетки, чтобы подтвердить 50-70% слияние.
- Разморозить вирусную аликвоту (ай) для трансдукции интересующей конструкции (конструкций) кДНК. Быстро разморозьте замороженные аликвоты на водяной бане при температуре 37 °C. Добавьте 2,5 мкл 8 мг/мл полибрена к каждой вирусной аликвоте и перемешайте путем осторожного пипетирования.
- Удалите среду из покрытых клеток и осторожно добавьте вирусную аликвоту к пластине 10 см, пипеткой вдоль боковой стороны пластины. Покачайте пластину, чтобы распространить 2 мл вирусной аликвоты.
- Инкубировать при 37 °C в инкубаторе культуры тканей в течение 2 ч. Раскачивайте пластину каждые 30 минут, чтобы предотвратить высыхание любых областей пластины.
- Добавьте 5 мл среды DMEM с 10% FBS, добавкой P / S / Q и 10 мМ пирувата натрия с 5 мкл 8 мг / мл полибрена. Дайте клеткам высиживаться в течение ночи.
- Утром удаляют медиа из клеток и проходят ячейки в среды двойного отбора. Растите и проходите клетки по мере необходимости в течение 7-10 дней, чтобы обеспечить двойной отбор и экспрессию конструкции кДНК.
ПРИМЕЧАНИЕ: Это разделение этого отрывка может потребовать оптимизации для разных клеточных линий. Для клеток A673 с pSRP-iEF-2 и конструкцией pMSCV-hygro клетки пропускают без расщепления на 2 мкг/мл пуромицина и 100 мкг/мл гигромицина.
2. Соберите клетки, проверьте экспрессию конструкций и настройте коррелятивные фенотипические анализы
- Через 7-10 дней двойного отбора собирают клетки в коническую трубку объемом 15 мл. Подсчитайте собранные клетки с помощью гемоцитометра. Aliquot собирала клетки для секвенирования РНК и проверки экспрессии конструкций кДНК.
ПРИМЕЧАНИЕ: Установите любые коррелятивные фенотипические анализы, требуемые исследуемым исследовательским вопросом. Колониеобразующие анализы являются примером коррелятивного фенотипического анализа, который используется здесь.- Соберите от 5 x 105 до 1 x 106 клеток для секвенирования РНК и 2 x 106 клеток для экстракции белка. Пеллетные ячейки центрифугированием при 1000 х г при 4 °C в течение 5 мин и удаляют надосадочное вещество.
- Вымойте гранулу 1 мл холодного PBS. Гранулы методом центрифугирования при 1000 х г при 4 °С в течение 5 мин и удаляют супернатант. Гранулы мгновенной заморозки в жидком азоте и хранение при -80 °C.
- Настройте любые корреляционные анализы с оставшимися клетками.
ПРИМЕЧАНИЕ: Протокол может быть приостановлен здесь с собранными образцами, хранящимися в морозильной камере -80 °C.
- Проверка нокдауна интересующего белка (если используется) и выражения панели конструкций.
- Гранулы оттаивания клеток для экстракции белка на льду. Повторное суспендирование клеток в ледяном холоде 500 мкл ядерного экстракционного буфера (20 мМ HEPES pH 7,9, 140 мМ NaCl, 10% глицерин, 1,5 мМ MgCl2,1 мМ ЭДТА, 1 мМ DTT, 1% IGEPAL) ингибитором протеазы. Дайте ему постоять 5 минут на льду.
- Ядра гранул методом центрифугирования при 1000 х г при 4 °C в течение 5 мин и удаления надосадочного вещества. Промывайте ядра в 500 мкл ледяного буфера для экстракции ядра (20 мМ HEPES pH 7,9, 140 мМ NaCl, 10% глицерина, 1,5 мМ MgCl2,1 мМ ЭДТА, 1 мМ DTT, 1% IGEPAL) ингибитором протеазы.
- Ядра гранул путем центрифугирования при 1000 х г при 4 °C в течение 5 мин и удаления супернатанта. Повторное суспендирование ядер в 200 мкл холодного буфера RIPA с ингибитором протеазы (регулировка объема буфера RIPA в соответствии с размером гранул). Дайте ему постоять на льду в течение 45-60 минут с энергичным вихрем каждые 15 минут.
- Гранулированный клеточный мусор центрифугированием при 16 000 х г при 4 °C в течение 45-60 мин. Сохранить супернатант и перенести в свежую холодную трубку
- Подготовьте образцы для электрофореза SDS-PAGE путем кипячения 5-10 мкг белка с 1x нагрузочным буфером в течение 5 мин. Запустите гель SDS-PAGE по мере необходимости для интересующего белка.
- Перенос на нитроцеллюлозную или PVDF-мембрану по мере необходимости для интересующего белка. Блокируют и промокают соответствующими первичными и вторичными антителами для подтверждения нокдауна эндогенного белка (если используется) и эктопической экспрессии конструкции кДНК.
ПРИМЕЧАНИЕ: Здесь протокол можно приостановить.
- Экстракт РНК. Оценка качества и количества РНК.
- Оттаивайте гранулы клеток на льду. Экстрагируйте общую РНК с помощью набора для экстракции на основе спиновой колонки кремнезема в соответствии с инструкциями производителя.
- Вкратце, лизируйте клетки, используя буфер лизиса из набора. Либо нанесите лизат на спиновую колонну кремнезема с коротким спином при >13000 об/мин в течение 30-60 секунд, либо удалите гДНК, применив лизат к колонке удаления гДНК с коротким спином при >13000 об/мин в течение 30-60 секунд.
- Выполните переваривание ДНК на колонке, если лизат был непосредственно нанесен на спин-колонку кремнезема. При использовании колонки для удаления гДНК нанесите элюат на кремнеземно-спиновую колонну с коротким отжимом при >13000 об/мин в течение 30-60 с.
- Промывайте РНК на колонке в соответствии с инструкциями производителя. Элютивная РНК в 30 мкл буфера элюирования.
- Оцените качество и количество РНК с помощью флуорометра или любого другого сопоставимого инструмента. Убедитесь, что соотношение 260/280 близко к 2 и что есть по крайней мере 2,5 мкг РНК для секвенирования.
ПРИМЕЧАНИЕ: По мере сбора реплик каждая реплика должна обрабатываться с использованием одного и того же протокола извлечения РНК. - Используйте небольшую аликвоту РНК для подтверждения стабильного нокдауна интересующего белка, если это необходимо, с помощью qRT-PCR. Храните оставшийся образец РНК при -80 °C.
- Соберите биологические реплики, повторяя шаги 1-2 до тех пор, пока не будет собрано 3-4 полных набора РНК. Убедитесь, что каждая реплика демонстрирует адекватную экспрессию конструкций кДНК и стабильное нокдаун эндогенного белка (если используется).
3. Секвенирование нового поколения
- Отправьте извлеченную РНК для секвенирования с использованием платформы секвенирования следующего поколения с целевым показателем 50 миллионов парных считываний пары оснований (bp) 150. Следуйте инструкциям предприятия по обработке образцов. Выбор для полиаденилированных РНК и специфического секвенирования нитей.
4. Конвейер выравнивания и подсчета расшифровок
ПРИМЕЧАНИЕ: Этот протокол предполагает, что после отправки и обработки образцов для каждого образца возвращается набор парных файлов FASTQ. Эти файлы часто сжимаются суффиксом "fastq.gz". Дальнейший анализ этих файлов FASTQ потребует доступа к высокопроизводительному вычислительному оборудованию (HPC) под управлением операционной системы Linux.
- Передача файлов
- Откройте терминал в среде высокопроизводительных вычислений с помощью PuTTY. Создайте каталог для анализа под названием "project".
- Перейдите в каталог "path_to/project" и создайте новый каталог для сжатых необработанных файлов fastq.gz под названием "fastq". Также сделайте каталог под названием «обрезанный». Это показано на рисунке S1A-C.
- Перенесите сжатые необработанные файлы fastq.gz из локального хранилища в каталог «path_to/project/fastq/» с помощью WinSCP или аналогичной программы. Убедитесь, что для каждого примера есть файлы "R1" и "R2", как показано на рисунке S1B.
- Необязательно: при необходимости установите TrimGalore. Задайте каталог, содержащий исполняемый файл trim_galore, в переменной среды PATH в Linux.
ПРИМЕЧАНИЕ: Низкокачественные считыватели и адаптеры обрезаны TrimGalore. В распоряжении гостей отеля TrimGalore https://github.com/FelixKrueger/TrimGalore. - Необязательно: перейдите в каталог загруженных пакетов программного обеспечения (например, «path_to/программное обеспечение»). Загрузите последнюю версию пакета TrimGalore с помощью команды «curl -fsSL https://github.com/FelixKrueger/TrimGalore/archive/[version].tar.gz -o trim_galore-[version].tar.gz».
- Необязательно: распакуйте файл tar.gz. Используйте команду "tar -xvzf trim_galore-[version_number].tar.gz".
- Необязательно: сделайте TrimGalore исполняемым. Используйте команду "chmod a+x path_to/software/TrimGalore-[version]/trim_galore". Убедитесь, что этот новый каталог находится в PATH. Используйте команду "export PATH=path_to/software/TrimGalore-[version]:$PATH".
- Перейдите к path_to/project/fastq/. Используйте TrimGalore для обрезки низкокачественных считываний из файлов fastq.gz с помощью команды, показанной на рисунке S1C.
ПРИМЕЧАНИЕ: Дополнительные флаги для этой команды могут быть актуальны и могут быть найдены здесь: https://github.com/FelixKrueger/TrimGalore/blob/master/Docs/
Trim_Galore_User_Guide.md - Проверьте наличие обрезанных файлов fastq.gz в каталоге path_to/project/trimmed. Убедитесь, что они называются sample1_R1_val_1.fq.gz и sample1_R2_val_2.fq.gz
- Выровняйте обрезанные файлы FASTQ с помощью STAR и сгенерируйте количество расшифровок.
ПРИМЕЧАНИЕ: STAR доступен по адресу https://github.com/alexdobin/STAR)- Необязательно: установите STAR версии 2.6 или более поздней. Установите исполняемый файл STAR в контуре.
- Необязательно: перейдите в каталог загруженных пакетов программного обеспечения (например, «path_to/программное обеспечение»).
- Необязательно: Загрузите пакет STAR с помощью команды "curl -SLO https://github.com/alexdobin/STAR/archive/[version].tar.gz". Распакуйте файл tar.gz.
- Необязательно: используйте команду "tar -xzf [version].tar.gz". Сделайте STAR исполняемым. Используйте команду "chmod a+x path_to/software/STAR-[version]/bin".
- Необязательно: убедитесь, что этот новый каталог находится в пути. Используйте команду "export PATH=path_to/software/STAR-[version_number]/bin/linux_x86_64_static:$PATH".
ПРИМЕЧАНИЕ: Руководство STAR доступно по адресу: (https://github.com/alexdobin/STAR/blob/master/doc/STARmanual.pdf). - Убедитесь, что есть индекс генома для использования с STAR. Поместите его в каталог, отдельный от каталога path_to/project/. Если индекс был ранее сгенерирован для предыдущих экспериментов, используйте его. В качестве альтернативы используйте соответствующий предварительно сгенерированный индекс, если он доступен здесь: http://refgenomes.databio.org/. В противном случае создайте новый индекс с помощью команды "STAR--runMode genomeGenerate", используя инструкции в руководстве STAR.
ПРИМЕЧАНИЕ: Для остальной части этого протокола путь к индексу STAR будет называться "path_to/STAR_index". - Перейдите в каталог path_to/project/. Создайте новый каталог с именем "STAR_output", как показано на рисунке S1D.
- Перейдите в каталог path_to/project/trimmed/. Используйте команду, показанную на рисунке S1D, чтобы запустить STAR, чтобы выровнять обрезанные файлы fastq.gz.
ПРИМЕЧАНИЕ: Этот шаг является наиболее вычислительно интенсивным, и рекомендуется выполнять его на кластере HPC с несколькими потоками (т.е. >16), предназначенными для задачи выравнивания. В зависимости от количества образцов и доступных вычислительных ресурсов этот шаг может занять от нескольких часов до нескольких дней. - Найдите необходимые выходные данные для следующих шагов, которые содержат счетчики на расшифровку, в следующем месте: path_to/project/STAR_output/sampleN_ReadsPerGene.out.tab.
ПРИМЕЧАНИЕ: В файле ReadsPerGene.out.tab столбец 1 содержит информацию о подсчитываемой функции. В колонке 2 содержатся незастегнутые счетчики чтения, в колонке 3 — счетчики чтения с прямым застреванием, а в колонке 4 — счетчики чтения с обратной очередью. Первые четыре строки этого файла будут содержать информацию о выровненных чтениях, которые не выровнялись по одному гену. Для этого протокола требуется количество непристегнутых счетчиков чтения. - Используйте RStudio (предпочтительно) или R в среде HPC для компиляции данных из строки 5 и ниже для столбцов 1 и 2 для каждого образца. Задайте для рабочего каталога значение "project" в R.
- Чтение в каждом файле ReadsPerGene.out.tab с помощью команды на рисунке S2A. Для первого столбца возьмите только символы перед "." в столбце "Ensembl gene ID" для удобства последующей обработки.
- Скомпилируйте счетчики из всех образцов в фрейм данных под названием "totcts" с помощью команд на рисунке S2B. Сохраните эту новую таблицу необработанных данных счетчика в виде файла с разделителями табуляции .txt, т.е. при желании sample_counts.txt с помощью команды "write.table".
ПРИМЕЧАНИЕ: Порядок идентификатора гена Ensembl одинаков для каждого файла ReadsPerGene.out.tab в разных образцах.
5. Дифференциальное выражение и последующий анализ
- Нормализуйте пакетные эффекты между сэмплами с помощью ComBat.
ПРИМЕЧАНИЕ: Существуют две возможные переменные, которые объясняют изменения в экспрессии генов, первая из которых является используемой конструкцией (т.е. образец), а вторая - внешними факторами, связанными с прохождением клеток во времени (т.е. партия). Рекомендуется выполнить этап нормализации выборок для пособного варианта с помощью ComBat С помощью R-пакета.- При необходимости установите и загрузите библиотеки для sva, DESeq2, AnnotationDBI, org. Hs.eg.db, pheatmap, RColorBrewer, genefilter, Cairo, ggplot2, ggbiplot, rgl и reshape2, как показано на рисунке S2C. Для установки используйте команду "install.packages" или Bioconductor в документации для каждого пакета.
- Сначала отфильтруйте данные только по тем генам, которые имеют хотя бы одно количество на чтение. Сохраните эту новую таблицу, чтобы обозначить фильтрацию, как показано на рисунке S2D.
ПРИМЕЧАНИЕ: Часто многие гены имеют очень низкое количество считываний или вообще не имеют его. - Подготовьте вторую таблицу для пакетной нормализации под названием "vars", как показано на рисунке S2E. Задайте для имен строк уникальные имена каждого образца. Задайте имена столбцов "sample", "batch" и "construct".
- Присвойте всем образцам уникальный номер в столбце «образец» от 1 до n, где n — это количество образцов. Присвойте номера партий всем образцам в столбце «пакет» таким образом, чтобы a_1 условий и b_1 условий присваивалось 1, а a_2 условий и условий b_2 присваивалось 2. Присвойте все обозначения условий всем образцам в колонке "конструкция" таким образом, чтобы все образцы "условие-а" были "А", а образцы условий-в - "В".
- Определите также пакетную переменную и конкретную матрицу нулевой модели для ComBat, как показано на рисунке S2F. Запустите ComBat с помощью команды, определенной на рисунке S2F.
- Далее курируйте данные, округляя до ближайшего целого числа. Также удаляют гены с отрицательным значением. Используйте команды, показанные на рисунке S3A.
ПРИМЕЧАНИЕ: Выходные данные пакетной нормализации будут иметь нецелое число считываний и некоторые гены с отрицательными значениями. Этот шаг является обязательным, поскольку анализ дифференциальных выражений нижестоящего потока не поддерживает отрицательное количество считываний. - Определите профиль дифференциального выражения для каждой конструкции с помощью DESeq2.
- Введите дизайн эксперимента для DESeq2, как показано на рисунке S3B. Создайте DESeqDataSet (dds) с помощью функции DESeqDataSetFromMatrix, оцените коэффициенты размера и запустите DESEq2, как показано на рисунке S3B.
ПРИМЕЧАНИЕ: Крайне важно, чтобы данные столбца, введенные для "условия", находились в том же порядке, что и столбец в матрице счетчика. - Чтобы оценить качество анализа, извлеките rlog-нормализованные счетчики, используемые DESeq2, как показано на рисунке S3B.
ПРИМЕЧАНИЕ: Во время анализа DESeq2 преобразует подсчеты с помощью «регуляризированного журнала», rlog, преобразования, чтобы уменьшить различия между образцами для генов с низким количеством (низкая информация), чтобы сохранить различия в генах с более высоким количеством в образцах (высокая информация). - При извлечении результатов для каждого транскрипционного профиля из результатов DESeq2 выполните парные сравнения со ссылкой либо на условие нокдауна, либо на базовый пустой вектор, как показано на рисунке S3C. Дополнительно измените эти результаты с помощью символов гена HGNC, как показано на рисунке S3D.
- Как показано на рисунке S3E,извлеките данные из результатов DESeq2. Экспорт в виде одного файла с идентификатором гена Ensembl, символом HGNC, выражением базового среднего и данными дифференциальной экспрессии для всех конструкций с log2FoldChange и необработанными и скорректированными p-значениями.
ПРИМЕЧАНИЕ: Использование скорректированного p-значения < 0,05 является рекомендуемой отсечкой для дифференциального выражения. - Оцените успешную нормализацию партии и сходство внутри образца. Проверьте кластеризацию образцов с помощью PCA и графиков расстояний от образца до образца с помощью нормализованных подсчетов rlog с использованием кода, показанного на рисунках S4A-B.
- Введите дизайн эксперимента для DESeq2, как показано на рисунке S3B. Создайте DESeqDataSet (dds) с помощью функции DESeqDataSetFromMatrix, оцените коэффициенты размера и запустите DESEq2, как показано на рисунке S3B.
- Используйте профили дифференциальных выражений для создания графиков вулканов с помощью кода на рисунке S4C. Оцените изменения в экспрессии генов между конструкциями.
- Используйте нормализованные подсчеты rlog и иерархическую кластеризацию для идентификации сигнатур генов, уникальных для различных конструкций. Используйте код, показанный на рисунке S4D.
- Извлеките 1000 наиболее изменчивых генов во всех конструкциях матрицы. Используйте pheatmap для выполнения неконтролируемой иерархической кластеризации ваших образцов на основе этих генов.
- Извлеките интересующие кластеры из дендрограммы, решив, на каком уровне появляются интересующие кластеры дендрограммы. Установите "k" равным количеству кластеров на этом уровне. Переназначьте тепловую карту, упорядоченную по кластерам, чтобы определить, какие кластеры представляют интерес, как показано на рисунке S5.
- Экспортируйте список генов, связанных с каждым кластером, как показано в таблице S1. Используйте эту информацию для определения генов в интересующих кластерах.
- Определите биологические роли для различных кластеров идентифицированных генов и сравните между классами. Это может быть выполнено с использованием различных инструментов биоинформатики. ToppGene24 используется здесь и находится в свободном доступе онлайн.
ПРИМЕЧАНИЕ: Есть много бесплатных инструментов, которые требуют только списка генов для копирования и вставки в поле на веб-сайте. Выберите аналитические инструменты, наиболее подходящие для исследуемых исследовательских вопросов. - Необязательно, если имеются данные о геномном связывании, которое управляет транскрипционным выходом для интересующего транскрипционного фактора, сравните транскрипционный ответ у генов, связанных с различными связывающими элементами, для дальнейшей оценки мутантной функции.
6. Сравнение с соответствующими фенотипами
- Сравните коррелятивные фенотипы с генерируемыми данными транскриптомного профиля и интерпретируйте соответствующим образом.
Результаты
Предварительные данные qRT-PCR свидетельствуют о том, что мутант EWS/FLI под названием DAF со специфическими мутациями тирозина в аланин в повторяющейся и неупорядоченной области EWS сохранял способность активировать гены-мишени EWS/FLI, но не смог подавить критические гены-мишени23. Чтобы лучше понять взаимосвязь между этими остатками в домене EWS и функцией EWS/FLI, был использован протокол, описанный выше и описанный на рисунке 1. A673 Клетки саркомы Юинга были вирусно трансдуцированы шРНК, нацеленной на 3'UTR FLI1,что привело к истощению эндогенного EWS / FLI. После четырех дней отбора функция EWS/FLI была спасена с помощью вирусной трансдукции различных мутантных конструкций EWS/FLI с тегами 3XFLAG, с пустым вектором в качестве элемента управления без спасения. Нефункциональный мутант, не имеющий домена EWS, называемый Δ22, использовался в качестве отрицательного контроля, а EWS/FLI дикого типа, называемый wtEF, использовался в качестве положительного элемента управления(рисунок 2A). В качестве тестовой конструкции использовался DAF, хотя при желании можно использовать более одной тестовой конструкции. Клетки отбирали в течение дополнительных 10 дней, чтобы позволить конструктной экспрессии стабилизироваться, а затем собирали для РНК (со стадией удаления гДНК), белковых и колониеобразующих анализов. Были собраны четыре реплики, и на рисунке 2B-Dпоказаны репрезентативные qRT-PCR и западные пятна, показывающие эффективный нокдаун и спасение. Следует отметить, что DAF-спасенные клетки не смогли сформировать колонии, как показано на рисунке 2E,что свидетельствует о нарушении онкогенной трансформации.
После завершения реплицированной валидации и фенотипических анализов РНК была представлена в Институт геномной медицины в Национальной детской больнице для подготовки библиотеки и секвенирования следующего поколения с ~ 50 миллионами парных считываний 150 bp. Данные были возвращены в виде файлов fastq.gz. Низкокачественные чтения были вырезаны из этих файлов с помощью TrimGalore, а STAR использовался для выравнивания считываний с геномом человека hg19 и подсчета показаний на ген. hg19 использовался для целей совместимости с другими кураторскими наборами данных для EWS/FLI, используемыми в последующем анализе. Эти счетчики считывания были объединены в единую матрицу подсчета для всех выборок, первые 6 строк которой показаны на рисунке 3.
Первоначально подсчеты проводились через DESeq2 без пакетной нормализации, однако визуальный осмотр расстояния от образца до образца показал потенциальные смешанные эффекты пакетов, как показано красными стрелками на рисунке 4A. Это, вероятно, возникло из-за биологической изменчивости, вызванной прохождением клеток в культуре, и различий в обработке каждой партии. Нормализация пакетных эффектов была выполнена с помощью ComBat и обычно рекомендуется. Расстояния между выборкой и выборкой пакетных нормализованных данных показаны на рисунке 4B. После пакетной нормализации DESeq2 использовался для создания транскрипционных профилей для трех конструкций (wtEF, Δ22 и DAF) относительно базового уровня. Обратите внимание, что в то время как «родительские» клетки A673 (имитация нокдауна и имитация спасения, называемая здесь «iLuc») были включены в дифференциальный анализ, ссылкой для этого эксперимента являются клетки с EWS / FLI-истощенными, называемые ячейками iEF. Транскрипционный профиль может быть сгенерирован для эндогенного белка здесь путем сравнения образца iLuc с iEF, и это может представлять интерес для понимания того, как работает система спасения, но это не является целью этого конкретного анализа. Транскрипционные профили, генерируемые для мутантов, включают положительные (wtEF) и отрицательные (Δ22) элементы управления по отношению к iEF, так что они должны функционировать в качестве ориентиров для других мутантов. Это важно, так как положительный контроль в этом примере не полностью повторял функцию эндогенной EWS/FLI, как обсуждалось в других местах7,23.
Анализ главных компонентов (PCA) на рисунке 5 предполагает, что транскрипционный профиль DAF является промежуточным между wtEF и Δ22, подтверждая частичную функцию. Более того, иерархическая кластеризация 1000 наиболее изменчивых генов по образцам показала, что DAF не смог подавить гены-мишени EWS/FLI и лишь частично сохранил активность активации генов, как показано на рисунках 6A и S5. Анализ ToppGene показал, что классы генов, которые активирует DAF, функционально отличаются от тех МИШЕНЕЙ, активируемых EWS/FLI, где DAF не функционирует(рисунок 6B). Интересно, что функция активированных генов, спасенных wtEF, но не DAF, по-видимому, связана с транскрипционным контролем и регуляцией хроматина. Основываясь на результатах анализов формирования колоний, гены из этой подписи основного гена должны быть дополнительно проанализированы на предмет их роли в онкогенезе, опосредованном EWS / FLI. Важность EWS/FLI-опосредованной генной репрессии была ранее описана17.
Известно, что EWS/FLI обладает уникальным сродством связывания к GGAA-микроспутниковым повторяющимся элементам19,22,и что связывание на этих элементах приводит к последующей регуляции генов11,15,18,20,22. Эти микроспутники были охарактеризованы как связанные с активацией или репрессией, и либо проксимальные к (< 5 кб) СТШ, либо дистальные к (> 5 кб) КСС25. Кроме того, существуют EWS/FLI-регулируемые гены с высоким сродством (HA) ETS мотивами, близкими к TSS23. С целью дальнейшего анализа характеристик функции DAF и того, какие типы генов, активированных EWS/FLI, DAF смог спасти, была проанализирована дифференциальная экспрессия генов, связанных с этими различными классами. Интересно, что DAF был наиболее способен спасти гены, активированные GGAA-микроспутником, но не смог спасти активированные гены вблизи участка ГК, как показано на рисунке 7. Как видно из иерархической кластеризации, DAF не может спасти репрессии, опосредованные EWS / FLI, во всех классах мотивов. Эти данные свидетельствуют о том, что DAF сохраняет достаточные структурные особенности EWS для связывания и активации с GGAA-микроспутниками, как проксимальными, так и дистальными к TSS. Это, вероятно, возникает из-за неповрежденного домена SYGQ, который считается важным для активности EWS / FLI при повторах GGAA11. Эти данные также свидетельствуют о том, что специфические тирозины, мутировавшие в DAF, играют важную, но плохо изученную роль в регуляции генов, опосредованной EWS / FLI с сайтов HA, а также в репрессии генов, что подчеркивает важную область дальнейшего исследования.

Рисунок 1: Рабочий процесс. Описание пошаговой процедуры выполнения сопоставления структуры-функции с помощью транскриптомики. Ячейки были впервые подготовлены для выражения набора конструкций, необходимых для отображения структуры-функции. После экспрессии клетки собирали для РНК и белка и анализировали на коррелятивные фенотипы. Экспрессия конструкций была проверена, и этот процесс повторялся 3-4 раза для сбора независимых биологических реплик. Затем РНК была представлена для секвенирования следующего поколения (NGS). Когда данные были получены, данные были обрезаны по качеству, выровнены, и было рассчитано количество на расшифровку. Контролировались пакетные эффекты, а транскриптомные сигнатуры и дифференциальная экспрессия определялись с помощью DESeq2. Может быть включена иерархическая кластеризация и последующий анализ, интегрирующий другие наборы данных -omics и различный путь или функциональный анализ. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
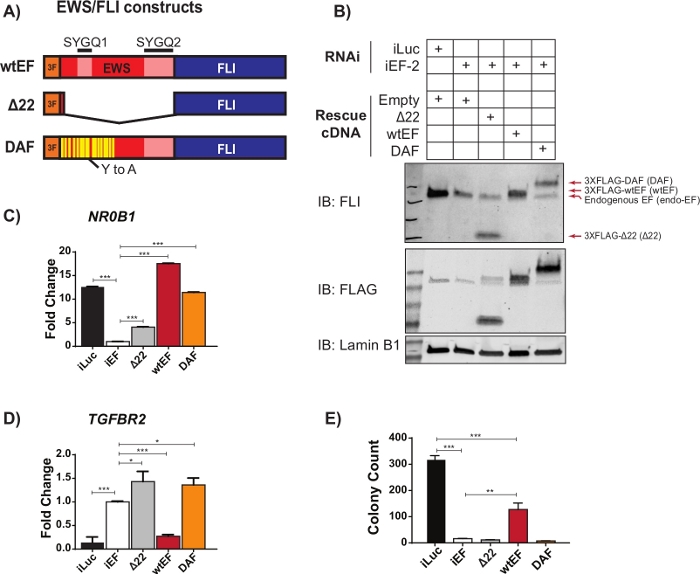
Рисунок 2: Валидация выражения конструкций и корреляционных анализов. (A) Схема, изображающая конструкции, протестированные в этом примере. (B) Валидация нокдауна эндогенного EWS/FLI и экспрессии 3X-FLAG-помеченных конструкций иммуноблотом. (С,D) Валидация конструктивной активности в активированном гене-мишени EWS/FLI(C) NR0B1и(D)репрессированном гене-мишени TGFBR2с помощью qRT-PCR. Данные представлены в виде среднего +/- стандартного отклонения. P-значения были рассчитаны с помощью теста честной значимости Туки. * p < 0,05, ** p < 0,01, *** p < 0,005 (E) Подсчет колоний из анализов мягкого агара, выполненных для оценки преобразующей активности конструкций. P-значения были рассчитаны с помощью теста честной значимости Туки. * p < 0,05, ** p < 0,01, *** p < 0,005. Этот рисунок адаптирован из Theisen, et al.23Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 3: Окончательные сопоставленные данные подсчета для анализа. Снимок экрана: первые 6 строк файла подсчета с подсчетом генов для всех образцов, подлежащих периодической нормализации и анализу. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
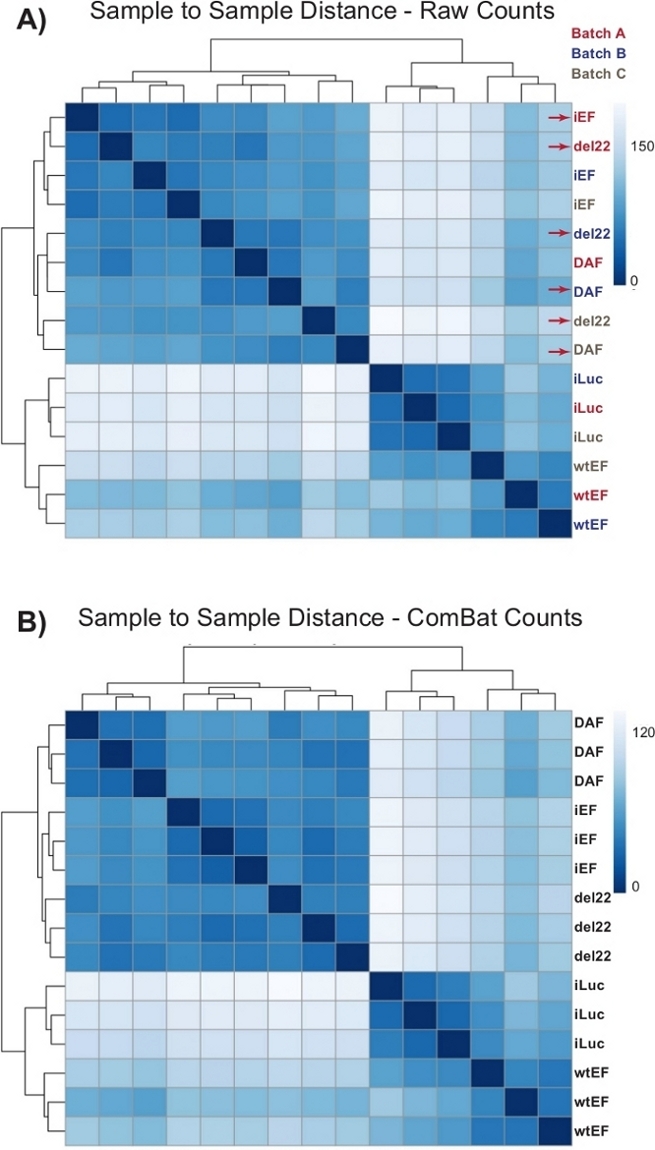
Рисунок 4: Тепловые карты расстояний от образца до образца. (A) График расстояния от образца до образца, показывающий кластеризацию выборки необработанных данных подсчета. Образцы, которые группируются как по партиям, так и по образцам, обозначаются красными стрелками. (B)График расстояния от образца до образца после пакетной нормализации с помощью ComBat. Здесь образцы из всех реплицируются вместе, независимо от пакета. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
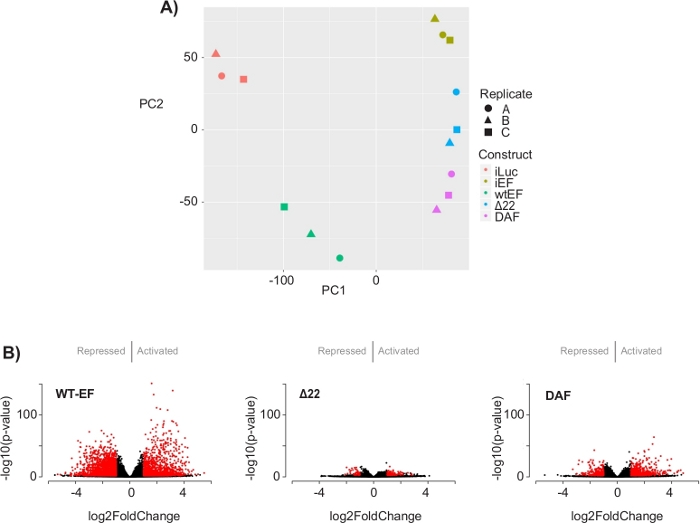
Рисунок 5: Результаты анализа дифференциальных выражений. (A)График принципиального компонентного анализа (PCA) транскриптомных сигнатур, сгенерированных для всех образцов, показывает сильную кластеризацию внутри выборки и демонстрирует, что DAF является промежуточным между положительным (wtEF) и отрицательным (Δ22) контролем. (B) Вулканические графики, показывающие -log(p-значение), построенные на основе log2FoldChange для генов в каждой конструкции. Гены с скорректированным p-значением < 0,05 и |log2(FoldChange)| > 1 считаются значительными и показаны красным цветом. Панель 5B адаптирована из Theisen, et al.23Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
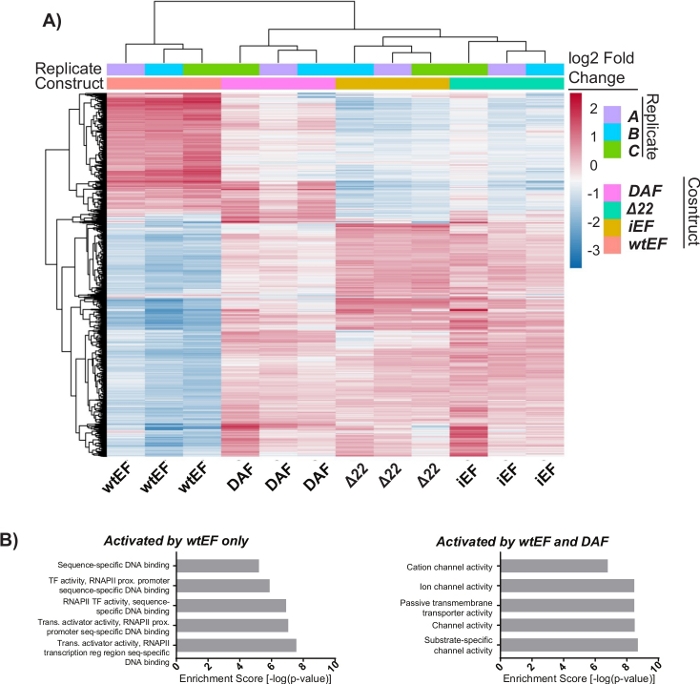
Рисунок 6: Иерархическая кластеризация для идентификации классов генов. Иерархическаякластеризация 1000 наиболее изменчивых генов во всех конструкциях и базовая линия, iEF, показывает, что DAF частично спасает активацию генов, опосредованную EWS / FLI. (B)Генная онтология (молекулярная функция) результаты ToppGene показывают функциональное обогащение генов, активированных EWS / FLI, которые либо спасены, либо не спасены DAF. Панель 6B адаптирована из Theisen, et al.23Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
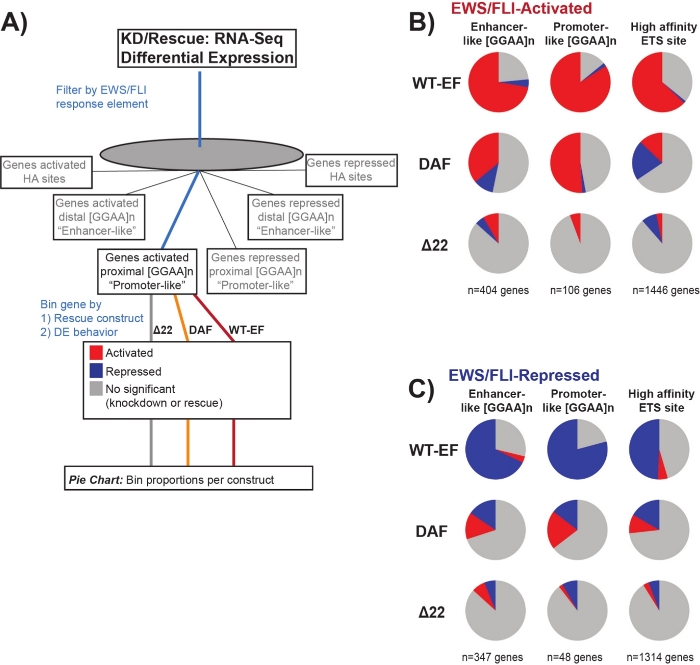
Рисунок 7: Подробный анализ различных элементов реакции фактора транскрипции на различные конструкции: (A) Схема, изображающая обработку данных, используемую для создания панелей (B) и (C) путем включения других доступных наборов данных с транскриптомными профилями здесь. (В,В) Компиляция, показывающая спасение различных классов прямых EWS/FLI-(B) активированных и(C)подавленных целей. Включенные гены были только генами с обнаруживаемой дифференциальной экспрессией эндогенной EWS/FLI. На каждой круговой диаграмме серым цветом изображена часть генов, которые не спасены конструкцией. Красным цветом изображена часть генов, которые дифференциально активированы, а синим — часть генов, которые дифференциально подавлены. Этот рисунок адаптирован из Theisen, et al.23Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Рисунок S1: Загрузка файлов fastq.gz в среду HPC, обрезка и выравнивание. Пожалуйста, нажмите здесь, чтобы загрузить этот рисунок.
Рисунок S2: Сопоставление количества считываний между выборками и выполнение пакетной нормализации с помощью ComBat. Пожалуйста, нажмите здесь, чтобы загрузить этот рисунок.
Рисунок S3: Запуск DESeq2 и извлечение результатов дифференциального анализа выражений. Пожалуйста, нажмите здесь, чтобы загрузить этот рисунок.
Рисунок S4: Анализ выходных данных. Пожалуйста, нажмите здесь, чтобы загрузить этот рисунок.
Рисунок S5: Иерархическая кластеризация для идентификации классов генов: Иерархическая кластеризация 1000 наиболее изменчивых генов во всех конструкциях и базовая линия, iEF, отсортированная по k кластерам. В данном случае k=7, но этот параметр задается пользователем, как показано на рисунке S4D. Пожалуйста, нажмите здесь, чтобы загрузить этот рисунок.
Таблица S1: Список генов (идентификатор гена Ensembl) с кластерной аннотацией. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Обсуждение
Изучение биохимических механизмов онкогенных факторов транскрипции критически важно для понимания заболеваний, которые они вызывают, и разработки новых терапевтических стратегий. Это особенно верно при злокачественных новообразованиях, характеризующихся хромосомными транслокациями, приводящими к слиянию факторов транскрипции. Домены, включенные в эти химерные белки, могут не иметь значимых взаимодействий с регуляторными доменами, присутствующими в белках дикого типа, что усложняет способность интерпретировать информацию о структуре-функции в контексте слияния26,27,28. Более того, многие из этих онкогенных синтезов характеризуются низкой сложностью внутренне неупорядоченных доменов10,13,29,30.
Домен EWS является примером такого внутренне неупорядоченного домена, который участвует в различных онкогенных слияниях10. Внутренне неупорядоченная и повторяющаяся природа препятствует усилиям по пониманию молекулярных механизмов, используемых областью EWS. Предыдущие усилия по исследованию структуры-функции в значительной степени прибегали к использованию различных мутантов в контексте репортерных генных анализов или в клеточных фонах, которые не могут повторить соответствующий клеточный контекст или не имеют каких-либо структурных вариаций, которые производят значимую частичную функцию11,17,25. Метод, представленный здесь, решает эти проблемы. Картирование структуры-функции выполняется в контексте клеток, связанных с заболеванием, а секвенирование следующего поколения позволяет транскриптомному профилированию оценивать функцию транскрипционного фактора в условиях нативного хроматина. В конкретном случае мутанта DAF EWS/FLI DAF, как сообщалось, проявлял небольшую активность в репортерных анализах с использованием изолированных элементов ответа, но проявлял активность в контексте полного промотора гена, либо в репортерном анализе, либо в нативном хроматине, предполагая интересный фенотип23. Использование метода, описанного здесь, более непосредственно решает вопрос о том, какой тип регуляторных элементов в геноме наиболее отзывчив в условиях заболевания. Тестируя все гены-мишени-кандидаты в их нативном контексте хроматина одновременно, транскриптомный подход с большей вероятностью идентифицирует конструкции с частичной функцией.
Неотъемлемая сила использования клеточного фона, связанного с заболеванием, является, пожалуй, самым большим ограничением этого метода. Одним из наиболее важных факторов является выбор подходящей клеточной системы для этих экспериментов. Многие клеточные линии, полученные от злокачественных новообразований с патогномоничными факторами транскрипции, не переносят сбивание этого фактора транскрипции, и во многих случаях, особенно для детского рака, истинная клетка происхождения остается спорной, а экспрессия онкогена в других клеточных фонах непомерно токсична31,32 . В этих случаях может быть полезно проводить эксперименты на другом клеточном фоне, если исследователь проявляет осторожность в интерпретации результатов и надлежащим образом подтверждает любые соответствующие результаты в более релевантном типе клеток.
Критически важно тщательно проверять стабильность и фенотипические последствия экспрессии онкогена и представлять для секвенирования только образцы, отвечающие строгим критериям. Здесь это включало вестерн-блот для подтверждения нокдауна и спасения, а также qRT-PCR небольшого числа известных генов-мишеней для проверки положительного контроля(рисунок 2). Также крайне важно уменьшить как можно большую вариабельность партии, тщательно выполняя клеточные и РНК-препараты как можно более похожим образом через каждую партию.
Метод, описанный здесь, становится особенно мощным в сочетании с другими типами геномных данных, которые говорят об общегеномной функции изучаемого фактора транскрипции. Будущие направления для этого типа структурно-функционального анализа будут расширены, чтобы включить ChIP-seq и ATAC-seq для определения связывания фактора транскрипции и любых индуцированных изменений доступности хроматина. Как набор, этот тип данных может указывать на то, где различные структурные компоненты онкогенного транскрипционного фактора способствуют различным аспектам функции (например, связывание ДНК против модификации хроматина против набора коконгуляторов). В целом, использование подходов на основе NGS для картирования структурно-функциональных отношений факторов транскрипции слияния может выявить новые идеи в биохимических детерминантах онкогенной функции этих белков. Это важно для углубления нашего понимания заболеваний, которые они вызывают, и для разработки новых терапевтических стратегий.
Раскрытие информации
SLL заявляет о конфликте интересов в качестве члена консультативного совета и акционера Salarius Pharmaceuticals. SLL также включена в список изобретателей по патентам США No. US 7,393,253 B2, «Методы и композиции для диагностики и лечения саркомы Юинга», и US 8,557,532, «Диагностика и лечение лекарственно-устойчивой саркомы Юинга». Это не меняет нашей приверженности политике JoVE в отношении обмена данными и материалами.
Благодарности
Это исследование было поддержано Высокопроизводительным вычислительным центром в Научно-исследовательском институте Эбигейл Векснер в Национальной детской больнице. Эта работа была поддержана Национальным институтом здравоохранения Национального института рака [U54 CA231641 to SLL, R01 CA183776 to SLL]; Alex's Lemonade Stand Foundation [Премия молодого исследователя ERT]; Пелотония [Стипендия ERT]; и Национальный совет по здравоохранению и медицинским исследованиям CJ Martin Overseas Biomedical Fellowship [APP1111032 to KIP].
Материалы
| Name | Company | Catalog Number | Comments |
| Wet Lab Reagents | |||
| anti-FLI rabbit pAb | Abcam | ab15289 | 1:500 |
| anti-lamin B1 rabbit pAb | Abcam | ab16048 | 1:2000 |
| Cell-based system for introduction of mutant constructs | Determined by cell system used | ||
| Cryotubes | For viral aliquots | ||
| DMEM | Corning Cellgro | 10-013-CV | For viral production |
| Fetal bovine serum | Gibco | 16000-044 | For viral production |
| G418 | ThermoFisher | 10131027 | For viral production |
| HEK293-EBNAs | ATCC | CRL-10852 | For viral production |
| HEPES | Gibco | 15630106 | |
| Hygromycin B | ThermoFisher | 10687010 | |
| M2 anti-FLAG mouse mAb | Sigma | F3165 | 1:2000 |
| Near IR-secondary antibodies | Li-Cor | ||
| Optimem | Gibco | 31985062 | For viral production |
| Penicillin/Streptomycin/Glutamine | Gibco | 10378-016 | For viral production |
| Polybrene | Sigma | TR-1003-G | For viral transduction |
| Puromycin | Sigma | P8833 | Stored at 2 mg/mL stock |
| RNeasy Plus kit | Qiagen | 74136 | Has gDNA removal columns |
| Selection reagents | As dictated by cell system used | ||
| Sodium Pyruvate | Gibco | 11360-070 | For viral production |
| Tissue culture media | Determined by cell system used | ||
| TransIT-LT1 | Mirus | MIR 2304 | For viral production |
| Software | |||
| Access to HPC environment | |||
| AnnotationDbi | 1.38.2 | ||
| Cairo | 1.5-10 | ||
| DESeq2 | 1.16.1 | ||
| genefilter | 1.58.1 | ||
| ggbiplot | 0.55 | ||
| ggplot2 | 3.1.1 | ||
| org.Hs.eg.db | 3.4.1 | ||
| pheatmap | 1.0.12 | ||
| PuTTY | |||
| R | 3.4.0 | ||
| RColorBrewer | 1.1-2 | ||
| reshape2 | 1.4.3 | ||
| rgl | 0.100.19 | ||
| R-studio | |||
| STAR | Version 2.6 or later | ||
| sva | 3.24.4 | ||
| TrimGalore! | |||
| WinSCP |
Ссылки
- Miettinen, M., et al. New fusion sarcomas: histopathology and clinical significance of selected entities. Human Pathology. 86, 57-65 (2019).
- Knott, M. M. L., et al. Targeting the undruggable: exploiting neomorphic features of fusion oncoproteins in childhood sarcomas for innovative therapies. Cancer and Metastasis Reviews. 38, 625-642 (2019).
- Yoshihara, K., et al. The landscape and therapeutic relevance of cancer-associated transcript fusions. Oncogene. 34, 4845-4854 (2015).
- Duesberg, P. H. Cancer genes generated by rare chromosomal rearrangements rather than activation of oncogenes. Medical Oncology and Tumor Pharmacotherapy. 4, 163-175 (1987).
- Dupain, C., Harttrampf, A. C., Urbinati, G., Geoerger, B., Massaad-Massade, L. Relevance of Fusion Genes in Pediatric Cancers: Toward Precision Medicine. Molecular Therapy - Nucleic Acids. 6, 315-326 (2017).
- Mitelman, F., Johansson, B., Mertens, F. The impact of translocations and gene fusions on cancer causation. Nature Reviews Cancer. 7, 233-245 (2007).
- Smith, R., et al. Expression profiling of EWS/FLI identifies NKX2.2 as a critical target gene in Ewing's sarcoma. Cancer Cell. 9, 405-416 (2006).
- Davicioni, E., et al. Identification of a PAX-FKHR gene expression signature that defines molecular classes and determines the prognosis of alveolar rhabdomyosarcomas. Cancer Research. 66, 6936-6946 (2006).
- Gröbner, S. N., et al. The landscape of genomic alterations across childhood cancers. Nature. 555, 321-327 (2018).
- Kim, J., Pelletier, J. Molecular genetics of chromosome translocations involving EWS and related family members. Physiological Genomics. 1, 127-138 (1999).
- Boulay, G., et al. Cancer-Specific Retargeting of BAF Complexes by a Prion-like Domain. Cell. 171, 163-178 (2017).
- Lessnick, S. L., Braun, B. S., Denny, C. T., May, W. A. Multiple domains mediate transformation by the Ewing’s sarcoma EWS/FLI-1 fusion gene. Oncogene. 10, 423-431 (1995).
- Leach, B. I., et al. Leukemia fusion target AF9 is an intrinsically disordered transcriptional regulator that recruits multiple partners via coupled folding and binding. Structure. 21, 176-183 (2013).
- Ng, K. P., et al. Multiple aromatic side chains within a disordered structure are critical for transcription and transforming activity of EWS family oncoproteins. Proceedings of the National Academy of Sciences U.S.A. 104, 479-484 (2007).
- Riggi, N., et al. EWS-FLI1 Divergent Chromatin Remodeling Mechanisms to Directly Activate or Repress Enhancer Elements in Ewing Sarcoma. Cancer Cell. 26, 668-681 (2014).
- Tomazou, E. M., et al. Epigenome Mapping Reveals Distinct Modes of Gene Regulation and Widespread Enhancer Reprogramming by the Oncogenic Fusion Protein EWS-FLI1. Cell Reports. 10, 1082-1095 (2015).
- Sankar, S., et al. Mechanism and relevance of EWS/FLI-mediated transcriptional repression in Ewing sarcoma. Oncogene. 32, 5089-5100 (2013).
- Gangwal, K., et al. Microsatellites as EWS/FLI response elements in Ewing's sarcoma. Proceedings of the National Academy of Sciences U.S.A. 105, 10149-10154 (2008).
- Gangwal, K., Close, D., Enriquez, C. A., Hill, C. P., Lessnick, S. L. Emergent Properties of EWS/FLI Regulation via GGAA Microsatellites in Ewing's Sarcoma. Genes & Cancer. 1, 177-187 (2010).
- Guillon, N., et al. The Oncogenic EWS-FLI1 Protein Binds In Vivo GGAA Microsatellite Sequences with Potential Transcriptional Activation Function. PLoS One. 4, 4932 (2009).
- Chong, S., et al. Imaging dynamic and selective low-complexity domain interactions that control gene transcription. Science. 361, (2018).
- Johnson, K. M., et al. Role for the EWS domain of EWS/FLI in binding GGAA-microsatellites required for Ewing sarcoma anchorage independent growth. Proceedings of the National Academy of Sciences U.S.A. 114, 9870-9875 (2017).
- Theisen, E. R., et al. Transcriptomic analysis functionally maps the intrinsically disordered domain of EWS/FLI and reveals novel transcriptional dependencies for oncogenesis. Genes & Cancer. 10, 21-38 (2019).
- Chen, J., Bardes, E. E., Aronow, B. J., Jegga, A. G. ToppGene Suite for gene list enrichment analysis and candidate gene prioritization. Nucleic Acids Research. 37, 305-311 (2009).
- Johnson, K. M., Taslim, C., Saund, R. S., Lessnick, S. L. Identification of two types of GGAA-microsatellites and their roles in EWS/FLI binding and gene regulation in Ewing sarcoma. PLOS One. 12, 0186275 (2017).
- Kim, P., Ballester, L. Y., Zhao, Z. Domain retention in transcription factor fusion genes and its biological and clinical implications: a pan-cancer study. Oncotarget. 8, 110103-110117 (2017).
- de Mendíbil, I. O., Vizmanos, J. L., Novo, F. J. Signatures of Selection in Fusion Transcripts Resulting from Chromosomal Translocations in Human Cancer. PLOS One. 4, 4805 (2009).
- Frenkel-Morgenstern, M., Valencia, A. Novel domain combinations in proteins encoded by chimeric transcripts. Bioinformatics. 28, 67-74 (2012).
- Hegyi, H., Buday, L., Tompa, P. Intrinsic Structural Disorder Confers Cellular Viability on Oncogenic Fusion Proteins. PLoS Computational Biology. 5, 1000552 (2009).
- Latysheva, N. S., Babu, M. M. Discovering and understanding oncogenic gene fusions through data intensive computational approaches. Nucleic Acids Research. 44, 4487-4503 (2016).
- Deneen, B., Denny, C. T. Loss of p16 pathways stabilizes EWS/FLI1 expression and complements EWS/FLI1 mediated transformation. Oncogene. 20, 6731-6741 (2001).
- Kendall, G. C., et al. PAX3-FOXO1 transgenic zebrafish models identify HES3 as a mediator of rhabdomyosarcoma tumorigenesis. eLife. 7, 33800 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены