Method Article
10ナノグラムのトータルRNAからシングルリードとペアエンドのmRNA - seqのイルミナライブラリ
要約
ここでは、T7リニアRNA増幅に基づく遺伝子発現解析用のライブラリーのシークエンシングの両方シングルリードとペアエンドイルミナのmRNA - seqの製造方法を説明します。このプロトコルは、トータルRNAを始めるのは10ナノグラムを必要とし、全体の転写産物を表す非常に一貫性のあるライブラリを生成します。
要約
mRNAのseqで全トランスクリプトームの配列決定は、現在、グローバルな遺伝子発現、突然変異、対立遺伝子特異的発現および他のゲノムワイドな解析を実行するために広く使用されている。 mRNAのSeqはさらに、非塩基配列ゲノムの遺伝子発現解析のためのゲートを開きます。 mRNAのSeqは高感度、広いダイナミックレンジを提供し、サンプル中の転写産物のコピー数を測定することができます。イルミナ社のGenome Analyzerは、単一の長いリードを特徴とする"ペアエンド"のアプローチは、両方の両端に配列決定されている。大比較的短いシーケンスの数(> 10 7)(<150 bp)の読み取りの配列決定を行い、選択的スプライシングのジャンクションを追跡することができます、挿 入や削除、およびde novoのトランスクリプトームアセンブリするのに便利です。
研究者が直面する大きな課題の一つは、出発原料の限られた量です。トータルRNA mを開始例えば、細胞がレーザーマイクロダイセクションで採取される実験では、利用可能なナノグラムの措置をAYがつきます。このようなサンプルからのmRNA - seqのライブラリーの調製は、1、2を説明するが、バイアスを導入することが重要なPCR増幅を伴うしている。最小限のPCR増幅を持つ他のRNA - seqのライブラリー構築の手順は3、4を発表したが、全体のRNAを開始するのマイクログラムの量を必要とされている。
ここでは重要なPCR増幅を回避し、トータルRNAのわずか10ナノグラムを必要とするライブラリの準備のためのmRNA - seqのシーケンスのためのイルミナGenome AnalyzerのIIのプラットフォーム用のプロトコルについて説明します。このプロトコルは、それが重要な増幅バイアスを導入せず、方向ライブラリーを作製することが示されたシングルエンドシーケンシング5、の説明で述べたと検証してきたが、ここでは、ペアエンドのプロトコルとして使用するためにさらにそれを検証する。我々は選択的に"T7造語、T7ベースのEberwineリニア増幅法を用いてトータルRNAを起動ポリアデニルメッセンジャーRNAを増幅LA"(T7リニア増幅)。mRNAの断片化されて増幅されたポリ、最終的なシーケンスのライブラリを生成するライゲーション転写し、アダプタを逆にする。双方単一の読み取りとペアエンドの実行では、配列がヒトのトランスクリプトーム6にマップされているとなるように正規化複数の実行からのデータを比較することができます。我々は試料7を比較するときRPKMに優れた尺度である百万ごとの転写産物の単位で遺伝子発現の測定(TPM)を、報告する。
プロトコル
1。トータルRNAを分離
- 細胞/組織に1ミリリットルトリゾールを追加し、必要に応じて18〜22ガーゼの針でホモジナイズする。
- 200μlのクロロホルムを追加し、4℃で30分間14,000 rpmでスピン℃、
- 上部の水層を取り出し、0.5μlのリニアアクリルアミドを追加し、その後500μlのイソプロパノールを加える。室温で20分間放置します。
- 4℃で14,000 rpmでスピン° C、70%エタノールで洗浄ペレット、および真空乾燥を。
- 1μlのヌクレアーゼフリー水と200μlのPCRチューブへの転送に再懸濁します。
2。二本鎖cDNAを準備する
- 上記1μlのRNAへのオリゴdT - T7逆転写プライマー(5' - GGCCAGTGAATTGTAATACGACTCACTATAGGGAGGCGGTTTTTTTTTTTTTTTTTTTTTTTT -3')100μMを1μl、70℃の熱を加える° C(ヒートブロック)で5分間、氷上でクールなスナップ用。
- 1μlの5 × FSのバッファー、0.5μlのDTT、0.5μlのdNTPミックスおよび0.5μlのRnaseOUTを(スーパースクリプトIIキットから)を追加します。
- 42〜ヒート° Cは、PCRのマシンで1分間、その後、0.5μlのスーパースクリプトII逆転写酵素を加える。 42℃1時間インキュベート℃に
- 70熱℃で10分間、クールに4℃
- 氷の上で、上記の反応に以下を追加します。
- ヌクレアーゼフリー水 - 22.75μL
- 5Xセカンドストランドバッファー - 7.5μlの
- dNTPミックス - 0.75μL
- 大腸菌DNAリガーゼ - 0.25μL
- 大腸菌のDNAポリメラーゼ - 1μL
- RnaseH - 0.25μL
° C、1μlのT4 DNAポリメラーゼを追加し、16でさらに10分間インキュベート16で2時間インキュベート℃に
- 1.5mlのエッペンドルフチューブに移し、80μlの水を加える。
- リニアアクリルアミド0.5μlを添加する。
- 72μlの3 M NH4OAcと冷100%エタノールを480μlを添加する。 -20℃で1時間沈殿物
- 4℃、14,000 rpmでスピン℃で30分間、1 mLの70%エタノールで洗浄、2の14,000 rpmで遠心分、そして約10分間乾燥した真空。
3。ポリ- in vitro転写によるmRNAを増幅する
- 3.5μLのヌクレアーゼフリー水でcDNAを上記の再懸濁します。
- 上記に、1μlのdNTPの各(合計4μL)、10 ×反応緩衝液1μlとT7ポリメラーゼを1μl、およびMegascriptキットからRnaseOUT 0.5μLを加える。
- 37℃のPCRマシンのC一晩。
- 次のステップの準備ができて。 -80℃で保存することができます℃に
4。増幅されたポリ- mRNAの断片化
- 上記の反応と4μlの10倍の断片化試薬に水26μlを添加する。
- 70℃で正確に7分間のPCRマシンの熱。
- 断片化の停止緩衝液5μl加え、氷上にサンプルを置く。
5。 RNAのクリーンアップ
- 上記の反応に、60μlの水とRNeasyアプライキットからのBuffer RLT350μlの、ピペッティングにより混合を追加。
- スピンカラムに250μlのエタノール、ピペッティングにより混合し、ピペットを追加。
- 20秒8000 rcfでスピン。
- RPE、20秒間スピンで1回洗浄、80%エタノール、2分間スピン、および5分のスピンドライカラムで2回目を洗う。
- 10μlのヌクレアーゼフリー水でRNAを溶出する。
6。 cDNA合成
単一の読み込みライブラリの最初の鎖の合成:
- PCRチューブに、以下を追加します。
- 断片化されたポリ- mRNAの - 10μlの
- をNotIランダムnonamerのプライマー - 1μL
5サーモサイクラーでの分、そして氷上で迅速な寒さのために70℃でインキュベートする。をNotI Nonamerのプライマー(5' - TGAATTCGCGGCCGCTCAAGCAGAAGACGGCATACGAGCTCTTCCGATCT NNNNNNNNN -3')。ランダムな領域がチップのSeqキットからイルミナのアダプタBのシーケンスの逆補数になるまで5'近位のシーケンスは、次のシーケンス間、NotI制限部位です。
- 5Xファーストストランドバッファー-4液
- DTT - 2μlの
- dNTP混合物* - 1.5μL
42℃2分間サーマルサイクラーに入れ° Cは、スーパースクリプトIII逆転写酵素を1μlを追加し、1時間、42℃でインキュベートする。
*代わりにdCTPの、5 - メチルdCTPをdNTP混合物で使用されていました。
ペアエンドライブラリの最初の鎖の合成:
- PCRチューブに、以下を追加します。
- 断片化されたポリ- mRNAの - 10μlの
- ランダムヘキサマープライマー - 1μL
5サーモサイクラーでの分、そして氷上で迅速な寒さのために65℃でインキュベートする。
- 上記に、氷(上付きfirststrand IIIキットから)に以下を追加します。
- 5Xファーストストランドバッファー - 4μL
- DTT - 2μlの
- のdNTP(からキット) - 1.5μL
45℃1分間サーマルサイクラーに入れ° Cは、スーパースクリプトIII逆転写酵素を1μlを追加し、1時間45℃でインキュベートする。
第二鎖合成(両方のライブラリ用):
- 上記に、(スーパースクリプトIIキットから)氷の上に以下を追加します。
- 水(RNaseフリー) - 91μL
- 5Xセカンドストランドバッファー - 30μlの
- のdNTP(キットから) - 3μlの
- 大腸菌DNAリガーゼ - 1μ
- 大腸菌のDNAポリメラーゼ - 4μL
- 大腸菌のRNase H - 1μL
反転してミックスチューブは、2時間16での短いスピンとインキュベート° Cを与える。
7。 cDNAの精製
- Zymo列、単一の読み取りのための水を40μlで溶出し、ペアエンドライブラリのための水を30μlのcDNAサンプルを精製する。
8。最後の修理
シングルリードのライブラリのための:
(イルミナチップのSeqサンプルプレップキットを使用してください)
- cDNAの上記の40μlのために、追加します。
- 10mMのATPとT4 DNAリガーゼバッファー - 5μlを
- dNTPミックス - 2μlを
- T4 DNAポリメラーゼ - 1μL
- クレノウ酵素(1U /μlに水で1:5に希釈) - 1μlの
- T4 PNK - 1μL
20℃で30分間サーマルサイクラーでインキュベート℃に
ペアエンドライブラリのための:
(イルミナペアエンドサンプルプレップキットを使用してください)
- cDNAを上記の30μlのために、追加します。
- RNaseをDNaseフリー水 - 45μlの
- 10mMのATPとT4 DNAリガーゼバッファー - 10μlの
- 10mMのdNTPミックス - 4μL
- T4 DNAポリメラーゼ - 5μlを
- Klenow酵素 - 1μL
- T4 PNK - 5μlの
20℃で30分間サーマルサイクラーでインキュベート℃に
9。 cDNAのクリーンアップ
- C cDNAを使用してZymo列を傾く。ペアエンドライブラリの、単一の読み取りとEB 32μlのためのEB 34μlの溶出。
10。 DNA断片の3'末端に'拠点を追加
シングルリードのライブラリのための:
- 以下の反応ミックスを調製する:
- DNAサンプル - 34μL
- クレノウバッファ - 5μlの
- dATPを - 10μlの
- クレノウエキソ - 1μL
37℃で30分間インキュベート℃、
ペアエンドライブラリのための:
- 以下の反応ミックスを調製する:
- DNAサンプル - 32μL
- クレノウバッファ - 5μlの
- dATPを - 10μlの
- クレノウエキソ - 3μlの
37℃で30分間インキュベート℃、
11。 cDNAのクリーンアップ
- cDNAを使用してzymo列をクリーンアップします。 EB10μlに溶出します。
12。アダプターライゲーション
jove_step">単一の読み取りライブラリの場合:(イルミナチップのSeqサンプルプレップキットを使用してください)
- 以下の反応ミックスを調製する:
- DNAサンプル - 10μlの
- アダプターオリゴミックス - 1μL
- 2X DNAリガーゼ緩衝液- -15μL
- DNAリガーゼ - 4μL
室温で15分間インキュベートする。アダプタは、氷と希薄化後1時20分に解凍してください。
ペアエンドライブラリのための:
(イルミナペアエンドサンプルプレップキットを使用してください)
- 以下の反応ミックスを調製する:
- DNAサンプル - 10μlの
- PEアダプターオリゴミックス - 10μlの
- 2X DNAリガーゼ緩衝液 - 25μlの
- DNAリガーゼ - 5μlの
20℃で15分間インキュベート℃、アダプタは、氷と希薄化後1時20分に解凍してください。
13。ライゲーション反応は、クリーンアップ
- ZYを使用してライゲーション反応をクリーンアップMOの列。ペアエンドライブラリのEBを5μlの別の溶出が続く単一の読み取りライブラリとEBの6μlのための水の44μlの溶出。
NotI消化を行う、唯一の単一の読み取りライブラリ用
- cDNAを - 44μL
- NEBバッファ3から5μlを
- BSA - 0.5μL
- をNotI - 1μL
37 ° C、およびzymoカラムを用いた精製反応で一晩に2時間インキュベートする。バッファEB 6μlの溶出は、EBの5μlの2番目の溶出が続きます。
14。サイズの選択/ゲル精製
Invitrogenから2%のSYBR安全なE -ゲルを使用して、次の手順を実行。
- プログラム0 -プレラン2分を実行します。
- インビトロジェン希釈1:4から1キロバイトプラスはしごをロードし、10μlをロードする。
- DNAサンプルのすべてをロードし、そして水10μlで空のレーンを埋める。
- プログラムを実行する1 -エーゲル2%は28分を実行します。
- サイズは200から300 bpのゲルSLICを選択します。新鮮なカミソリの刃を持つe。
15。ゲルスライスからDNAを溶出
- ゲルスライスを入れ重量を測定し、(キアゲンからゲル抽出キットを使用)ゲルの1倍量のBuffer QGの3つのボリュームを追加します。
- 50℃で10分間またはゲルスライスが溶けるまで。
- ispropanolの1ボリュームを追加し、反転またはピペッティングにより混和する。
- 、列に追加し、最高速度で1分間スピン、およびフロースルー捨てます。
- 、カラムにBuffer QGを500μLを追加し、最大速度で1分間スピン、およびフロースルーを捨てる。
- カラムにBuffer PEを750μlのを追加し、最高速度で1分間スピン、およびフロースルーを捨てる。
- 最高速度で1分間遠心。
- ペアエンドライブラリの、単一の読み取りと23μlのEB用EB 36μlの溶出。
16。 PCR
シングルリードのライブラリのための:
(イルミナチップのSeqサンプルプレップキットを使用してください)
- followinを準備グラムのPCR反応ミックス:
- DNA - 36μL
- 5X Phusionバッファ - 10μlの
- dNTPミックス - 1.5μL
- PCRプライマー1.1から1μL
- PCRプライマー2.1から1μL
- Phusionポリメラーゼ - 0.5μL
以下のPCRのプロトコルを使用します。
- 98℃で30秒° C
- の10サイクル:
- 98℃で10秒° C
- 65℃30秒° C
- 72℃で30秒
- 72℃5分° C
- 4℃ホールド° C
キットが使用されていること*初めて、EBバッファを使用してPCRプライマー1:2希釈する。
ペアエンドライブラリのための:
(イルミナペアエンドサンプルプレップキットを使用してください)
- 以下のPCR反応を準備します。
- DNA - 23μL
- Phusion DNAポリメラーゼ - 25μlの
- PCRプライマーPE 1.1から1μL
- PCRプライマーPE 2.1 -1μL
以下のPCRのプロトコルを使用して増幅する。
- 98℃で30秒° C
- の10サイクル:
- 98℃で10秒° C
- 65℃30秒° C
- 72℃で30秒
- 72℃5分° C
- 4℃ホールド° C
キットが使用されていること*初めて、EBバッファを使用してPCRプライマー1:2希釈する。
17。ライブラリのクリーンアップ
- zymo列を使用してライブラリをクリーンアップします。 EB 12μlの溶出
18。定量するライブラリ
- 量子ビットを使用してライブラリを定量する。それは、シーケンシングのための準備ができています。単一の読み込みライブラリのV4以上のイルミナ単一の読み取りクラスタ生成キットから配列決定用プライマーとペアエンドライブラリのV4以上のイルミナペアエンドクラスタ生成キットを使用してください。
19。データ分析
ボウタイ6 mに使用されたapはRefSeqの遺伝子セット(NCBIのビルド36.1)に読み込みます。シングルエンドの読み取り(30ヌクレオチド)とペアの端には、遺伝子のセットに10試合を許容すること、および読み取り当たり2個のミスマッチを許容するマップされた(42塩基)を読み取ります。百万分の一転写産物(TPM)の値がRSEM 7(期待値最大化によるRNA - Seqを)使用して遺伝子発現を測定するために得られた。
20。代表的な結果:我々は両方のシングルリードとペアエンドのトータルRNAを(図1)から始まる1μgを、100μgの、10μgの、1μgのとは100 pgから実行されるためT7LAライブラリを作った。我々のプロトコルの評価については、我々は、10μgの全RNAからこれらのコントロールライブラリを起動するT7 RNA増幅することなく単一の読み取りとペアエンドライブラリを作った"MinAmp"と呼ばれ、最小限の増幅を持っている。彼らが受ける唯一の増幅は、イルミナシーケンシングアダプターを連結するためのプロトコルの終わり近くにすべてのライブラリに共通するステップPCRの10サイクルです。使用されるすべてのRNAは、H14から分離されたヒト胚性幹細胞8。
まず、各種ライブラリ(表1と補足表1)によって同定された遺伝子の数を評価した。両方シングルリードとペアエンドライブラリの場合は、10 ngのT7LAライブラリは10以上のTPMに、10μgのMinAmpライブラリとしての遺伝子のほぼ同じ数を同定した。シングルリードのライブラリの場合には、10 ngのT7LAライブラリには、10μgの未増幅ライブラリによって識別された8500の遺伝子の100%を同定した。ペアエンドライブラリの場合、10 ngのT7LAライブラリには、10μgの未増幅ライブラリ(9267遺伝子の7961)によって同定された遺伝子の86%を同定した。未満は10 ngから作られたライブラリは、多くの遺伝子として同定することができませんでした。例えば、単一の読み取りのプロトコルで、1ngのライブラリにのみ識別さ〜10μgのMinAmpライブラリによって同定された遺伝子の50%を、私達は10 ngまでT7LAプロトコルで使用するためのトータルRNAの最小量を制限するように求める。また、ハウスキーピング遺伝子のマッピング、 GAPDH(図2)出発RNAの少なくとも10ngで行われたすべてのT7LAライブラリが極端な5'エクソンを含む、すべてのエクソンを同定することを示しています。 MinAmpライブラリーとは10 ng T7LAシングルリードとペアエンドライブラリの比較では、類似度の高い(それぞれスピアマン相関、R = 0.90と0.95、図3aとb)を示しています。我々はまた、10 ngのトータルRNAから成る2つの読み取りとペアエンドライブラリを比較し、それらがライブラリの両方のタイプが(非常によく似た遺伝子発現の署名を生成T7LAプロトコルを使用して作られたことを示す、非常に高い相関係数を(R = 0.92)であった図3c)。したがって、T7LAメソッドはMinAmpライブラリなどが、1000倍未満出発原料からのような信頼性の高い包括的なシーケンシングライブラリーを作製することができます。

図1ペアエンドのスキーマと単一の読み取りライブラリ調製プロトコルを。

図2すべての単一の読み取りとペアエンドライブラリのためのハウスキーピング遺伝子、GAPDH、のゲノムのブラウザの画像。単一の読み込みライブラリの左側のスケールバーは350総読み取りを示しています。ペアエンドライブラリのためのセンターのスケールバーは5000合計が読み取りを示しています。横軸はGAPDHのゲノム配列を表します。

図3各種ライブラリ(スピアマンの)の間で遺伝子発現の相関:ペアエンド間の単一の間A. 10 ngのT7LAを読み、10μgのMinAmpライブラリは両方のこれらのライブラリは非常によく似た遺伝子発現のパターンを(R = 0.90)を持っていることを示しているB. 10 ngのT7LAおよび10μgのMinAmpライブラリは、遺伝子発現プロファイルの類似度(R = 0.95)を示しています。遺伝子の元のCの相関は、10 ngのペアエンド〜10 ngの単一の読み取りライブラリpressionsはT7LA法(R = 0.92)が作成したこれらのライブラリ間の類似度の高い表示。
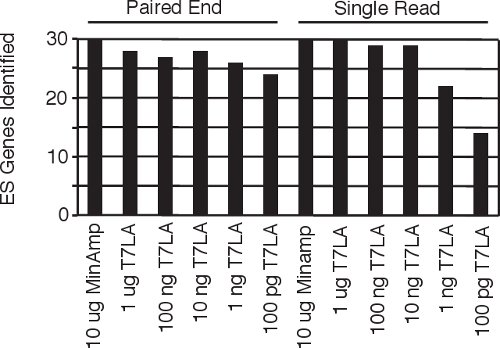
すべてのシングルリードとペアエンドライブラリーからのヒト胚性幹細胞特異的genes5の図4の同定。
| ライブラリ | 種類° | 生のクラスタ | フィルタを通過%クラスタ | ゲノムに整列% | %のエラーレート | 同定された遺伝子 |
| MinAmp 10ug | シングルリード | 225602 + / - 4952 | 65.48 + / - 2.58 | 47.61 + / - 0.53 | 0.62 + / - 0.06 | 8500 |
| 1ug T7LA | シングルリード | 144818 + / - 6513 | 82.21 + / - 6.45 | 48.09 + / - 0.27 | 0.42 + / - 0.03 | 8757 |
| 100ng T7LA | シングルリード | 27385 + / - 1818 | 81.33 + / - 11.75 | 44.46 + / - 4.53 | 0.49 + / - 0.10 | 8709 |
| 10ng T7LA | シングルリード | 11184 + / - 985 | 60.70 + / - 3.70 | 14.96 + / - 1.15 | 0.99 + / - 0.30 | 8589 |
| 1ng T7LA | シングルリード | 12695 + / - 1365 | 53.27 + / - 16.76 | 4.08 + / - 0.79 | 2.25 + / - 1.56 | 4720 |
| 100pg T7LA | シングルリード | 10390 + / - 1398 | 72.99 + / - 2.90 | 1.48 + / - 0.20 | 1.51 + / - 0.39 | 1121 |
| MinAmp 10ug | ペアエンドR1 | 95786 + / - 12937 | 90.77 + / - 2.79 | 58.50 + / - 0.95 | 0.94 + / - 0.38 | 9267 |
| ペアエンドR2 | 95786 + / - 12937 | 90.77 + / - 2.79 | 58.13 + / - 1.13 | 0.99 + / - 0.37 | ||
| 1ug T7LA | ペアエンドR1 | 297669 + / - 10196 | 91.35 + / - 0.36 | 46.89 + / - 0.14 | 0.47 + / - 0.01 | 7334 |
| ペアエンドR2 | 297669 + / - 10196 | 91.35 + / - 0.36 | 45.52 + / - 0.12 | 0.51 + / - 0.01 | ||
| 100ng T7LA | ペアエンドR1 | 205602 + / - 9932 | 90.53 + / - 0.76 | 63.44 + / - 1.00 | 0.48 + / - 0.02 | 8011 |
| ペアエンドR2 | 205602 + / - 9932 | 90.53 + / - 0.76 | 61.80 + / - 8.09 | 0.60+ / - 0.36 | ||
| 10ng T7LA | ペアエンドR1 | 214622 + / - 11155 | 89.98 + / - 1.13 | 56.32 + / - 1.94 | 0.80 + / - 0.26 | 7961 |
| ペアエンドR2 | 214622 + / - 11155 | 89.98 + / - 1.13 | 46.41 + / - 18.39 | 2.48 + / - 2.68 | ; | |
| 1ng T7LA | ペアエンドR1 | 144951 + / - 19841 | 90.54 + / - 1.19 | 3.91 + / - 0.16 | 8.71 + / - 0.86 | 8124 |
| ペアエンドR2 | 144951 + / - 19841 | 90.54 + / - 1.19 | 3.27 + / - 1.21 | 9.11 + / - 3.52 | ||
| 100pg T7LA | ペアエンドR1 | 187600 + / - 11759 | 89.52 + / - 1.11 | 1.78 + / - 0.05 | 13.42 + / - 0.50 | 6623 |
| ペアエンドR2 | 187600 + / - 11759 | 89.52 + / - 1.11 | 1.99 + / - 0.23 | 15.29 + / - 0.96 | ||
· R1とR2は前進であり、タグの順序を逆
*≥10 TPM
クラスタ番号は上の表1。情報、同定された遺伝子、エラーレート、単一の読み取りおよびペアエンドライブラリのパーセントの位置合わせ。
補足表1。すべてのサンプルのすべての遺伝子とそのTPM値のリスト、単一の読み取りとペア終了。
ディスカッション
ペアエンドライブラリを作るための現在のプロトコルは、1μgの9〜2.5μgのトータルRNAの10開始量の間に必要とする。ここでは、このメソッドは許可されている両方の単一の読み取りとペアエンドイルミナシーケンシングライブラリとショーを準備する私たちのリニアT7増幅系(T7LA)メソッドを提示するのに匹敵するデータを生成するトータルRNAのような低10 ngのからのライブラリの生成1000倍以上の出発原料(10μgのトータルRNA)から作られた最小限の増幅(MinAmp)ライブラリ。 10 ngのライブラリーは、遺伝子の類似の総数を特定するものではなく、また(図3aとb)類似している遺伝子発現の署名を生成しません。また、T7LA法により製造された単一の読み取りとペアエンドライブラリの両方は、研究者がどちらのプロトコルで作られたライブラリによって生成されたデータを比較できるようになる、(図3c)は互いに非常によく似ています。これらのライブラリは、ヒト胚性幹細胞のRNAから調製されて以来、我々が検索ライブラリのうち、30幹細胞特異的遺伝子のため、ほぼこれらの遺伝子のすべて(93から100パーセント)はこのように我々のプロトコルを検証する、トータルRNAを(図4)出発の少なくとも10 ngのから作られたライブラリによって識別されていることがわかります。我々は、我々のプロトコルは、特にそのようなフローソートされた細胞または出発物質が制限され、レーザーマイクロ解剖組織としての状況では、研究者にとって非常に有用ではないかと思います。このような状況では、我々のプロトコルは、我々のプロトコルが開始RNAの大きさの少なくとも3桁でほぼ同等の発現プロファイルを生成するので、はるかに大きいの開始量から作られたライブラリに匹敵する遺伝子発現データの生成をできるようになります。
開示事項
著者らは、JATが携帯電話ダイナミクス国際(CDI)の創設者、stockowner、コンサルタントおよびボードメンバーであることを開示している。彼はまたの科学顧問としての役割を果たし、タクティクスII幹細胞のベンチャーズの経済的利益を持っています。
謝辞
この作業は研究とウィスコンシン財団の大学Morgridge研究所からの資金によって支えられている。我々は編集支援のためクリスタイーストマンに感謝。
資料
| Name | Company | Catalog Number | Comments |
| 会社 | キット/試薬 | カタログ# | 特別なコメント |
| アンビオン | 断片化試薬 | AM8740 | |
| アンビオン | ライナーのアクリルアミド | AM9520 | |
| アンビオン | MEGAscript T7キット | AM1334 | |
| アンビオン | 非DEPCは、ヌクレアーゼフリーの水を扱わ | AM9932 | |
| Fermentas | dNTPのセット | R0181 | |
| Fermentas | メチル化のdNTP | R0431 | 唯一のシングルリードのサンプル調製用 |
| イルミナ | TruSeq SRクラスタ生成キットV5 | GD - 203 - 5001 | 唯一のシングルリードのサンプル調製用 |
| イルミナ | TruSeqのSeqキットV5 36サイクル | FC - 104 - 5001 | |
| イルミナ | チップのSeqサンプル調製キット | IP - 102 - 1001 | シングルリードサンプル調製用のみ。 1 CAT#E6040S設定NEB DNAサンプルプレップマスターミックスに置き換えることができます。 |
| イルミナ | TruSeq PEクラスタ生成キットV5 | PE - 203 - 5001 | ペアエンドサンプル調製用のみ |
| イルミナ | ペアエンドサンプル前処理キット | PE - 102 - 1001 | ペアエンドサンプル調製用のみ |
| インビトロジェン | 1キロバイトプラスはしご | 10787-018 | |
| インビトロジェン | 大腸菌のRNase H | 18021-014 | |
| インビトロジェン | RNaseのアウト | 10777-019 | |
| インビトロジェン | 第二鎖バッファー5倍 | 10812-014 | |
| インビトロジェン | 大腸菌のDNAポリメラーゼ | 18010-017 | |
| インビトロジェン | E -ゲルSYBR安全な2パーセント | G521802 | |
| インビトロジェン | 上付きIII(5X FSバッファおよび0.1M DTT付き) | 18080-085 | |
| インビトロジェン | トリゾール | 12183555 | |
| インビトロジェン | RNA quibitアッセイキット | Q32852 | |
| インビトロジェン | dsDNAをHS量子ビットアッセイキット | Q32851 | |
| インビトロジェン | 大腸菌DNAリガーゼ | 18052-019 | |
| インビトロジェン | T4 DNAポリメラーゼ | 18005-025 | |
| インビトロジェン | 超高純度のDNase RNaseの水 | 10977-015 | |
| インビトロジェン | スーパースクリプトII二本鎖cDNA合成キット | 11917-020 | |
| インビトロジェン | ランダムプライマー(Invitrogen社) | 48190-011 | ペアエンドサンプル調製用のみ |
| IDT | Not1Nonamer Bのプライマー | N / | シングルリードSAM用PLE準備のみ |
| IDT | オリゴdT T7 | N / | |
| NEB | NOT1消化キット | R0189S | 唯一のシングルリードのサンプル調製用 |
| キアゲン | をRNeasyアプライキット | 74204 | |
| キアゲン | RNeasyミニキット(50) | 74104 | |
| キアゲン | ゲル精製キット | 28604 | |
| キアゲン | DNase処理セット | 79254 | |
| キアゲン | したRNeasyアプライキット | 74204 | |
| Zymo研究 | DNAクリーンとコンセントレータ(250X) | D4014 |
参考文献
- Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., Wang, X., Bodeau, J., Tuch, B. B., Siddiqui, A., Lao, K., Surani, M. A. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Armour, C. D., Castle, J. C., Chen, R., Babak, T., Loerch, P., Jackson, S., Shah, J. K., Dey, J., Rohl, C. A., Johnson, J. M., Raymond, C. K. Digital transcriptome profiling using selective hexamer priming for cDNA synthesis. Nature Methods. 6, 647-649 (2009).
- Mortazavi, A., Williams, B. A., McCue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Mamanova, L., Andrews, R. M., James, K. D., Sheridan, E. M., Ellis, P. D., Langford, C. F., Ost, T. W., Collins, J. E., Turner, D. J. FRT-seq amplification-free, strand-specific transcriptome sequencing. Nature Methods. 5, 130-132 (2010).
- Sengupta, S., Ruotti, V., Bolin, J., Elwell, A., Hernandez, A., Thomson, J., Stewart, R. Highly consistent, fully representative mRNA-Seq libraries from ten nanograms of total RNA. Biotechniques. 49, 898-904 (2010).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome. Biology. 10, R25-R25 (2009).
- Li, B., Ruotti, V., Stewart, R. M., Thomson, J. A., Dewey, C. N. RNA-Seq gene expression estimation with read mapping uncertainty. Biotechniques. 26, 493-500 (2010).
- Thomson, J. A., Itskovitz-Eldor, J., Shapiro, S. S., Waknitz, M. A., Swiergiel, J. J., Marshall, V. S., Jones, J. M. Embryonic stem cell lines derived from human blastocysts. Science. 282, 1145-1147 (1998).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved