Method Article
Enriquecimiento de partículas de lipoproteínas nativas con microRNA y determinación posterior de su contenido absoluto/relativo de microRNA y su tasa de transferencia celular
En este artículo
Resumen
Aquí, se presenta un protocolo cuantitativo en tiempo real basado en la reacción en cadena de la polimerasa para la determinación del contenido nativo de microARN (absoluto/relativo) de las partículas de lipoproteína. Además, se demuestra un método para aumentar el nivel de microARN, así como un método para determinar la tasa de captación celular de las partículas de lipoproteínas.
Resumen
Las partículas de lipoproteínas son principalmente transportadores de lípidos y colesterol en el torrente sanguíneo. Además, contienen pequeñas cantidades de hebras de microRNA no codificante (miRNA). En general, miRNA altera el perfil de expresión proteica debido a las interacciones con messenger-RNA (mRNA). Por lo tanto, el conocimiento del contenido miRNA relativo y absoluto de las partículas de lipoproteína es esencial para estimar el efecto biológico de la captación de partículas celulares. Aquí, se presenta un protocolo cuantitativo en tiempo real de la reacción en cadena de la polimerasa (qPCR) para determinar el contenido absoluto de miRNA de las partículas de lipoproteínas, ejemplificado en las partículas de lipoproteínas nativas y enriquecidas con miRNA. El contenido de Mirna relativo se cuantifica utilizando tarjetas de matriz de microfluidos multiplacas. Además, este protocolo permite a los científicos estimar el miRNA celular y, por lo tanto, la tasa de captación de partículas de lipoproteínas. Un aumento significativo del nivel de miRNA celular es observable cuando se utilizan partículas de lipoproteína de alta densidad (HDL) cargadas artificialmente con miRNA, mientras que la incubación con partículas de HDL nativas no produce ningún efecto significativo debido a su contenido de miRNA bastante bajo. En cambio, la captación celular de partículas de lipoproteína de baja densidad (LDL), ni con miRNA nativa ni cargada artificialmente con ella, no alteró el nivel de miRNA celular.
Introducción
Las partículas de lipoproteína se componen de una monocapa de lípidos anfifílicos y colesterol que encierra un núcleo de ésteres de colesteryl y grasas de triglicéridos. Toda la partícula es estabilizada por las Apolipoproteínas incrustadas por membrana, que definen la funcionalidad biológica de la partícula. Las partículas de lipoproteína pueden distinguirse según su respectiva densidad creciente y, por lo tanto, disminuyendo el tamaño, a saber, como lipoproteínas de muy baja densidad (VLDL), lipoproteínas de densidad intermedia (IDL), LDL y partículas de HDL. A pesar del transporte de componentes insolubles en agua en el torrente sanguíneo, se ha demostrado que las partículas de HDL llevan hebras no codificantes de Mirna1,2. Las micro-RNAs son una clase de hebras de ARN cortas (generalmente dos docenas de nucleótidos), que degradan las hebras de mRNA complementarias intracelular y, de este modo, alteran el perfil de expresión de ciertas proteínas3,4,5, 6. Además, las alteraciones del perfil de miRNA se han encontrado en una variedad de enfermedades y, por lo tanto, el perfil es aplicable como un biomarcador para el diagnóstico y el pronóstico. El transporte extracelular de miRNAs entre células a través de partículas de lipoproteínas puede servir como un mecanismo adicional para la modulación del nivel de mRNA intercelular. Para estimar cuantitativamente el efecto biológico, se necesita conocimiento sobre el contenido miRNA absoluto y relativo de las partículas de lipoproteínas.
La PCR cuantitativa en tiempo real es un método adecuado y relativamente rápido para obtener esta información. Por lo tanto, el valor de cuantificación relativa (RQ) puede calcularse, y las diferencias relativas entre las diferentes muestras y las fracciones de lipoproteínas son estimable. Las tarjetas de matriz microfluidos multiwell son un método rápido y fácil de usar para determinar la presencia relativa (equivale al valor RQ) de miRNAs en una muestra. Las tarjetas de matriz de microfluidos multiwell constan de 96 o 384 cámaras de reacción individuales para las reacciones de qPCR individuales incrustadas en un dispositivo microfluidico. Cada cámara contiene la sonda de hidrólisis requerida y los imprimadores específicos de qPCR para un miRNA individual. Las ventajas son un tiempo de manipulación corto debido a la estandarización, un flujo de trabajo simple y un número reducido de pasos de pipeteo. Además, se minimiza el volumen de muestra necesario. A diferencia de la cuantificación relativa, el contenido absoluto de miRNA requiere una comparación de los resultados de la muestra de qPCR con curvas estándar de números absolutos conocidos de hebras de miRNA. Cabe señalar que, debido a su relativamente bajo contenido de miRNA, estándar y, por otra parte, incluso las técnicas de imágenes sensibles de una sola molécula no son factibles — el enriquecimiento artificial de las partículas de lipoproteína con miRNA es inevitable para estudiar celulares interacción de partículas de lipoproteínas y transferencia de miRNA. En cuanto a esto, la delipiación de la partícula HDL seguida con la posterior refundación7 permite la incorporación y, por lo tanto, el enriquecimiento con hebras de Mirna. El enriquecimiento similar de las partículas de LDL con miRNA no es factible debido a la hidrofobicidad de la proteína apoB-100, que es el componente principal de la partícula de LDL. Sin embargo, por adición del disolvente polar dimetilsulfóxido (DMSO), que es capaz de penetrar las membranas lipídicas, partículas de LDL pueden ser artificialmente cargados con miRNA hebras, así.
La microscopía de fuerza atómica de alta velocidad (HS-AFM) es una poderosa herramienta para la caracterización de especímenes biológicos que ofrecen una resolución temporal de subnanómetros espaciales y subsegundos8. Por lo tanto, es una técnica adecuada para el control de calidad de las partículas de lipoproteínas modificadas como partículas de lipoproteínas nativas/reconstituidas/etiquetadas pueden ser imágenes bajo un ambiente casi fisiológico.
Aquí, un protocolo basado en qPCR se presenta paso a paso para determinar el contenido de miRNA absoluto/relativo de partículas de lipoproteína y muestras celulares, lo que permite una estimación de la tasa de captación de partículas de lipoproteínas celulares. Por otra parte, se demuestra un método para el enriquecimiento de las partículas de lipoproteínas con miRNA. Este método puede ser adaptado para la manipulación general del contenido de lipoproteínas y, por lo tanto, demuestra la aplicabilidad de las partículas de lipoproteínas como objetivos para la administración de fármacos.
Protocolo
Las donaciones de sangre han sido aprobadas por el Comité de ética, Universidad médica de Viena (EK-Nr. 511/2007, EK-Nr. 1414/2016). La nomenclatura es de acuerdo con la información mínima para la publicación de los experimentos cuantitativos en tiempo real de PCR (MIQE)9 directrices.
1. aislamiento de partículas de lipoproteínas de sangre humana
- Preenfriar una ultracentrífuga a 4 ° c. Extraer sangre de voluntarios sanos después de ayunar durante la noche.
Nota: típicamente requerido son tres donantes, donando 80 mL cada uno, y los tubos de recolección de sangre que contienen ácido etilenediaminetetraacético (EDTA) como anticoagulante. - Centrifugar a 2.000 x g durante 20 min a 4 ° c y plasma de cosecha (fase superior); evitar las fuerzas de cizallamiento. Determinar el volumen total V y ajustar, si es necesario, a un múltiplo del volumen del tubo de centrifugación utilizando solución salina amortiguada con fosfato (PBS). Mida la masa de 1 mL 3x y calcule la densidad media ρ.
- Calcule la cantidad necesaria de bromuro de potasio (KBr) para ajustar la densidad con la siguiente ecuación10 (para la densidad deseada en gramos/mililitro, utilice A = 1,019, y para el volumen específico de kbr, use
 = 0,364 ml/g). Agregue el KBr al plasma y revuelva suavemente para evitar las fuerzas de cizallamiento hasta que el KBr se disuelva totalmente.
= 0,364 ml/g). Agregue el KBr al plasma y revuelva suavemente para evitar las fuerzas de cizallamiento hasta que el KBr se disuelva totalmente.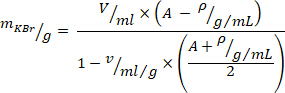
- Mida la densidad como se describe en el paso 1,2 y ajuste de nuevo, si es necesario, añadiendo más KBr. Llene y selle los tubos de centrifugación adecuados para la ultracentrifuga con plasma. Evite las burbujas de aire; de lo contrario, el tubo puede colapsar. Colocar los tubos en el rotor de acuerdo con las instrucciones del fabricante y centrifugar a 214.000 x g para 20 h a 4 ° c.
- Abra los tubos de acuerdo con las instrucciones del fabricante y deseche la fase superior que contiene VLDL e IDL. Determinar el volumen total V y ajustar, si es necesario, a un múltiplo del volumen del tubo de centrifugación utilizando PBS.
- Determine la densidad ρ de la fracción inferior. Calcule la cantidad necesaria de KBr para el ajuste de la densidad (utilice A = 1,063). Remover suavemente para evitar fuerzas de cizallamiento hasta que se disuelva KBr. Repita el paso 1,4.
- Retire y recoja la fase superior que contiene las partículas de LDL. Almacene la solución de partículas de LDL bajo una atmósfera de gas inerte a 4 ° c. Determinar el volumen total V y ajustar, si es necesario, a un múltiplo del volumen del tubo de centrifugación utilizando PBS. Determine la densidad ρ de la fracción inferior como se describe en el paso 1,2.
- Calcule la cantidad necesaria de KBr para el ajuste de la densidad (utilice A = 1,220) y agréguelo. Remover suavemente para evitar fuerzas de cizallamiento hasta que se disuelva KBr. Repita el paso 1,4.
- Retire y recoja la fase superior que contiene partículas de HDL. Determinar su volumen total V y ajustar, si es necesario, a un múltiplo del volumen del tubo de centrifugación utilizando PBS. Determine la densidad ρ. Se recomienda un segundo paso de centrifugación de la fase superior a 214.000 x g para 20 h a 4 ° c para eliminar la albúmina. Si es necesario, repita el paso 1,8. Retire y recoja la fase superior que contiene partículas de HDL.
- Preparar y preenfriar al menos 20 L de tampón de diálisis (0,9% NaCl, 0,1% EDTA [pH 7,4]) a 4 ° c. Tubos de diálisis prehúmedos (corte de peso molecular: 12 – 14 kDa) y agregue la solución de partículas LDL y HDL de acuerdo con las instrucciones del fabricante. Dialyze contra 5 L de tampón de diálisis a 4 ° c y cambie el buffer después de 1, 2 y 4 h.
- Después de 24 h, recupere las soluciones de partículas de lipoproteínas de los tubos de diálisis y determine la concentración de proteínas utilizando el ensayo de Bradford11 u otro apropiado. Almacene las soluciones de partículas de HDL y LDL en atmósfera de gas inerte a 4 ° c.
2. alícuts sintéticos de miRNA
Nota: cuando manipule oligonucleótidos de ARN, trabaje sin RNase. Trabaje sólo con consumibles plásticos frescos y desechables y siempre use guantes, que deben cambiarse con frecuencia. Utilice únicamente soluciones libres de nucleasas. Siempre trabaja en hielo.
- Gire hacia abajo el vial adquirido del fabricante a la fuerza máxima para formar un pellet del miRNA sintético liofilizado. Añadir un volumen adecuado de tampón Tris de 10 mM (hidroximetil) aminometano (TRIS), pH 7,5, para una concentración final de 10 μM (concentración de la culata) miRNA.
- Pipetear suavemente hacia arriba y abajo un par de veces para la resuspensión. Preparar alícullitas de 100 μL cada una en tubos estériles. Almacénelos a-20 ° c si no se utiliza inmediatamente. Evite el descongelamiento y congelación repetidos.
3. reconstitución de partículas HDL
-
Delipidation
- Prepare el buffer A (150 mM NaCl, 0,01% EDTA, 10 mM Tris/HCl [pH 8,0]). Preenfriar la centrífuga a-10 ° c. Preenfriar 100 mL de una mezcla de etanol: éter dietilo (3:2) a-20 ° c.
PRECAUCIÓN: Use el equipo de protección personal adecuado y trabaje en una campana de humos mientras maneja éter de dietilo, ya que es extremadamente inflamable y perjudicial para la piel. - Mezclar un volumen correspondiente a 5 mg de partículas HDL con 50 mL de etanol preenfriado: mezcla de éter dietilo (3:2) e incubar durante 2 h a-20 ° c. Centrifugar a 2.500 x g durante 10 min a-10 ° c.
- Desechar el sobrenadante, Resuspender el pellet en 50 mL de etanol preenfriado: mezcla de éter dietilo por vortexing e incubar por segunda vez durante 2 h a-20 ° c. Centrifugar de nuevo a 2.500 x g durante 10 min a-10 ° c.
Nota: si se desea, liofilizar el sobrenadante para un análisis del contenido de miRNA en la fracción lipídica de las partículas de HDL. - Secar el pellet bajo el flujo de gas N2 y resuspender en 250 μl de tampón A (ver paso 3.1.1). Determinar la concentración de proteína utilizando el ensayo de proteína de Bradford u otro apropiado y diluir hasta una concentración final de 1 mg de proteína/250 μL de tampón a.
Nota: el protocolo se puede pausar aquí. Almacene la solución durante la noche a 4 ° c bajo atmósfera de gas inerte.
- Prepare el buffer A (150 mM NaCl, 0,01% EDTA, 10 mM Tris/HCl [pH 8,0]). Preenfriar la centrífuga a-10 ° c. Preenfriar 100 mL de una mezcla de etanol: éter dietilo (3:2) a-20 ° c.
-
Reconstitución
- Preparar una solución de acción de fosfatidilcolina (PC) utilizando una mezcla de cloroformo: metanol (2:1) en una concentración de 5,6 mg de PC/mL. Del mismo modo, preparar soluciones de stock de oleato de colesterílico (5 mg de CO/mL) y colesterol (5 mg de C/mL). Almacene todas las soluciones a-20 ° c.
- En un tubo de vidrio limpio, mezclar 500 μL de PC, 100 μL de CO y 13,5 μL de C. Estos volúmenes corresponden a una relación molar aproximada de 100 PC: 22 CO: 4.8 C. seque la mezcla bajo el flujo de gas N2 mientras rota el tubo para producir una capa superficial homogénea.
Nota: el protocolo se puede pausar aquí. Almacenar el vial de vidrio (si se desea, es posible el almacenamiento) bajo atmósfera de gas inerte a-20 ° c. - Prepare una solución de espermina de 30 mM fresca en el buffer A. mezclar una alícuota (100 μL, 10 μM) de miRNA sintética (ver paso 2,2) con 100 μL de solución de espermina e incubar durante 30 min a 30 ° c.
Nota: para experimentos de control negativo, reemplace la solución Mirna y/o espermina con el mismo volumen de búfer A. - Disolver una alícuota de mezcla de PC/CO/C en la mezcla del paso 3.2.3.
- Preparar una solución de 30 mg/mL de desoxicolato sódico en el tampón A y añadir 50 μL a la solución desde el paso 3.2.4. Revuelva a 4 ° c durante 2 h.
- Añadir 250 μL de la solución de HDL delipidatada del paso 3.1.4. Este volumen corresponde a una relación molar aproximada de 100 PC: 22 CO: 4.8 C:1 de proteínas HDL delipidatadas. Revuelva a 4 ° c durante la noche.
-
Diálisis
- Preenfriar al menos 15 L de PBS a 4 ° c. Añadir 50 g de perlas adsorbentes a 800 mL de agua destilada doble (ddH2O) y remover durante 1 min. esperar 15 min, decantarse por el sobrenadante, y repetir el procedimiento con PBS.
- Casetes de diálisis prehúmedos (corte de peso molecular: 20 kDa) o tubos de diálisis apropiados y agregue la solución del paso 3.2.6, utilizando una jeringa de acuerdo con las instrucciones del fabricante.
- Añadir las perlas adsorbentes del paso 3.3.1 a 3 L de PBS y dializarse a 4 ° c. Cambie el buffer y las perlas después de 1 h y 2 h.
- Después de 24 h, recupere la solución de partículas HDL reconstituida (rHDL) y determine la concentración de proteínas utilizando el ensayo Bradford. Almacene la solución de partículas rHDL en atmósfera de gas inerte a 4 ° c.
4. etiquetado de partículas de LDL
- Preparar tampón de LDL 10x (1,5 M NaCl, 3 mM EDTA, 1 mM etilenglicol-bis (β-aminoetilo éter)-N, N, N ', N'-ácido tetraacético [EGTA, pH 7,4]) y almacenarlo a temperatura ambiente.
- Prepare una solución de espermina de 30 mM fresca en agua sin RNase. Mezclar una alícuota (100 μL, 10 μM) de miRNA sintética (ver paso 2,2) con 100 μL de solución de espermina e incubar durante 30 min a 30 ° c.
Nota: para experimentos de control negativo, reemplace el Mirna y/o la solución de espermina con el mismo volumen de 1 búfer de LDL. - Añada 100 μL de DMSO a la solución miRNA/spermine desde el paso 4,2 y diluya con 1,2 mL de buffer 1x LDL.
- Diluir la solución de partículas de LDL a una concentración final de aproximadamente 4 mg/mL con PBS y mezclar 450 μL con 50 μL de tampón 10x de LDL. Incubar durante 10 min en hielo.
- Combine la solución de partículas de LDL del paso anterior y la solución miRNA/spermine/DMSO (del paso 4,3) e incubarla durante 2 h a 40 ° c.
- Realice una diálisis similar a la descrita en la sección 3,3 y almacene la solución de partículas de LDL etiquetada en consecuencia.
5. control de calidad de partículas de lipoproteínas reconstituidas/etiquetadas
Nota: para el control de calidad, el diámetro y la forma general de las partículas de lipoproteínas se pueden determinar utilizando, por ejemplo, AFM o microscopía electrónica (EM). Aquí, HS-AFM se utiliza para medir la distribución de tamaño de partículas de lipoproteínas nativas/reconstituidas/etiquetadas.
- Diluir la solución de partículas de HDL/LDL en PBS (1:100 – 1:1000) e incubarla en mica recién rajada durante 5 min. Para quitar la mica12, presione la cinta adhesiva contra el sustrato y quite las capas de mica superiores tirando de la cinta.
Nota: dependiendo del instrumento AFM en particular, del área de observación y de la concentración inicial de partículas, el factor de dilución debe ajustarse para observar las partículas individuales. - Después de la incubación, enjuague la muestra con PBS y realice la toma de imágenes HS-AFM en PBS y en el modo de tapping con voladizos con constantes de resorte de kcant ≪ 0,2 N/m. Se recomienda utilizar tamaños de escaneo < 1 μm2 y mantener las fuerzas de imagen lo más bajas posible.
- Determine la altura de las partículas imágenes con respecto a la superficie de mica con el software adecuado.
- Cargue los datos en Gwyddion (Freeware), detecte las partículas a través del análisis de grano (Marque granos por umbral) y fije el umbral por encima del fondo del sustrato. Aplanar la imagen (eliminar fondo polinómico) eligiendo la opción excluir región enmascarada .
- Exporte los valores de altura de las partículas detectadas (distribución de diversas características de grano) y repita estos pasos para todas las imágenes grabadas. Realice análisis estadísticos creando histogramas o calculando las funciones de densidad de probabilidad de las alturas de partícula obtenidas.
- Repita los pasos 5.1 – 5.3 con partículas de lipoproteína nativas así como reconstituidas/etiquetadas y compare los resultados obtenidos para verificar la calidad de las partículas. Si observa residuos y/o conglomerados, deseche la muestra.
6. cultivo celular
- Cultivar células adherentes según un protocolo establecido (p. ej., ldlA7-SRBI13) hasta llegar a confluencias.
Nota: varias cámaras independientes dependiendo del número de experimentos (recomendado son dos cámaras por ajuste experimental) y experimentos de control negativo (recomendado son dos cámaras sin la adición de partículas de lipoproteína) son necesarios. Adicionalmente, se requieren dos cámaras para la determinación del número de célula. - Lave suavemente las células 3x con la solución de sal equilibrada (HBSS) de Hank precalentada para eliminar los desechos celulares y cubrir la capa celular con un volumen adecuado de medio de crecimiento libre de suero. Añadir un volumen adecuado de solución de partículas de lipoproteínas para alcanzar una concentración final de 50 μg/mL de partículas de lipoproteína. Incubar a 37 ° c y 5% CO2 para 16 h.
Nota: dependiendo del diseño experimental y la línea celular, el tiempo de incubación debe ser adaptado para lograr un aumento suficiente (es decir, medible) en el nivel de miRNA celular. - Lave suavemente las células 3x con HBSS precalentado (37 ° c) para eliminar los desechos celulares/partículas de lipoproteínas y cubra la capa celular con un volumen adecuado de medio libre de suero.
- Determine la densidad de la célula utilizando un método adecuado (p. ej., hemocitómetro, contador de celdas automatizado) en al menos dos cámaras independientes para calcular el número de celdas en el volumen de muestra del paso 6,3. Este número se utiliza para la normalización.
7. extracción de miRNA de muestras de partículas de células y lipoproteínas
Nota: la extracción de miRNA de las células se realiza utilizando el kit de extracción de miRNA con las siguientes modificaciones.
-
Las muestras celulares
- Preenfriar la centrífuga a 4 ° c. Añadir 350 μL de reactivo de lisis a dos cavidades que contengan células. Como experimento de control negativo, utilice una cámara sin células.
PRECAUCIÓN: utilice un equipo de protección personal adecuado y trabaje en una campana extractora mientras manipula el reactivo de lisis, ya que contiene fenol y tiocianato. - Espere de 3 a 5 minutos (dependiendo de la línea celular) para el desprendimiento de la célula. Si es necesario, verifique el desprendimiento de células con microscopía de campo claro. Piscina el contenido de las dos cámaras en un tubo de 1,5 mL.
- Utilizando una aguja de 20 G y una jeringa de 5 mL, homogeneizar/interrumpir la muestra 5x – 10x por aspiración e incubar durante 5 min. Añadir 140 μL de cloroformo (CHCl3), agitar vigorosamente durante 15 s e incubar durante 3 min.
- Centrifugar a 12.000 x g durante 15 min a 4 ° c. Después, deje de enfriar la centrífuga.
- Transfiera la fase acuosa superior a un nuevo tubo de 1,5 mL; evitar la mezcla de fase/contaminación ya que la interfase contiene ADN y la fase inferior contiene proteínas. Añadir 1.5 x el volumen de etanol al 100% y mezclar bien mediante pipeteo.
- Coloque una columna de centrifugado en un tubo de recogida de 2 mL y añada 700 μL de la mezcla del paso 7.1.5. Centrifugar a 8.000 x g durante 15 s a temperatura ambiente. Deseche el flujo y repita este paso con el volumen de muestra residual.
- Deseche el flujo y añada 700 μL del primer tampón de lavado a la columna de centrifugado. Centrifugar a 8.000 x g durante 15 s a temperatura ambiente.
- Deseche el flujo y añada 500 μL del segundo tampón de lavado a la columna de centrifugado. Centrifugar a 8.000 x g durante 15 s a temperatura ambiente. Repita este paso entero una segunda vez con 2 minutos de tiempo de centrifugación.
- Coloque la columna de centrifugado en un nuevo tubo de recogida de 2 mL y centrifugue a toda velocidad durante 1 minuto para secar la membrana.
- Coloque la columna de centrifugado en un tubo de recogida de 1,5 mL. Añadir 30 μL de agua sin RNase en el centro de la membrana para elución y centrifugarla a 8.000 x g durante 1 min. Repita todo este paso por segunda vez con el primer flujo a través de la solución de elución. El paso de transcripción inversa se realiza inmediatamente después de la extracción; de lo contrario, almacene las muestras de miRNA extraídas a-20 ° c.
- Preenfriar la centrífuga a 4 ° c. Añadir 350 μL de reactivo de lisis a dos cavidades que contengan células. Como experimento de control negativo, utilice una cámara sin células.
-
Las partículas de lipoproteína
- Ajuste el volumen de la muestra con la concentración de proteína más baja a 100 μL (= volumen máximo de muestra). Calcule los volúmenes de muestra de las otras muestras de acuerdo con esta normalización, inversamente a su concentración. Para experimentos de control negativo, utilice 100 μL de agua sin RNase.
Nota: la normalización no es necesaria, pero simplifica la comparación directa de los resultados durante el paso y el análisis de qPCR. - Preenfriar la centrífuga a 4 ° c. Añadir 700 μL de reactivo de lisis al volumen de muestra del paso 7.2.1.
- Realice la extracción de miRNA según los pasos 7.1.3 – 7.1.10.
- Ajuste el volumen de la muestra con la concentración de proteína más baja a 100 μL (= volumen máximo de muestra). Calcule los volúmenes de muestra de las otras muestras de acuerdo con esta normalización, inversamente a su concentración. Para experimentos de control negativo, utilice 100 μL de agua sin RNase.
8. la transcripción inversa
Nota: la transcripción inversa de miRNA se realiza utilizando un kit de transcripción inversa con las siguientes modificaciones.
- Descongelar los reactivos del kit y los imprimadores de transcripción inversa sobre hielo. Preparar la siguiente mezcla maestra en un tubo de reacción sobre hielo: 45,7 μL de H2O libres de rnasa, 16,5 μl de tampón de transcripción inversa 10x, 11 μl de enzima de transcripción inversa, 2,1 μl de inhibidor de la Rnasa y 1,7 μl de mezcla de dNTP. Mezclar suavemente, no hacer vórtice.
Nota: la escala depende de la cantidad de muestra; aquí, se calcula para 10 reacciones. - Etiquetar los tubos de 0,2 mL en consecuencia y mezclar 7 μL de la mezcla maestra del paso 8,1 con 5 μL de la muestra extraída del paso 7.1.10 y 3 μL de imprimación de transcripción inversa. Mezcle suavemente, Centrifugue en breve y almacene la mezcla sobre hielo.
- Para utilizar el mismo número de celda para cada línea de celda, reduzca el volumen de la muestra de la línea celular con un número de celda general más alto; utilizar esto como volumen residual para alcanzar el volumen total de la muestra de 5 μL de agua sin RNase.
Nota: las muestras de partículas de lipoproteínas ya están normalizadas en el paso 7.2.1. Para la preparación estándar de la curva, diluir una alícuota de miRNA secuencialmente en agua sin RNase y calcular el número de hebras por volumen de muestra. Se requieren al menos cinco puntos de datos dentro del rango de los valores resultantes del ciclo de cuantificación de muestreo (cq). Por lo general, los factores de dilución totales que oscilan entre 102 y 106 son adecuados. - Coloque los tubos en la máquina termocicladora e inicie el siguiente programa: 30 min a 16 ° c (paso de recocido), 30 min a 42 ° c (transcripción inversa), 5 min a 85 ° c (paso de fusión), y pausa a 4 ° c (almacenamiento). Realice el paso de qPCR inmediatamente después de la transcripción inversa; de lo contrario, almacene el ADN complementario (cDNA) sintetizado a partir de las muestras de miRNA a-20 ° c.
9. qPCR
- Realizar un qPCR del cDNA (transcrito inverso de miRNA) utilizando un ensayo comercialmente disponible (ver tabla de materiales) con las siguientes modificaciones.
- Descongelar todos los reactivos (Supermix, libre de RNase H2O), muestras de cDNA (del paso 8,4), y las imprimación en hielo. Preparar la siguiente mezcla maestra en un tubo de reacción sobre hielo: 75 μL de Supermix, 47,5 μL de H2O libre de rnasa, 7,56 μl de imprimación. Mezclar suavemente, no hacer vórtice.
Nota: la escala depende de la cantidad de muestra; aquí, se calcula para 10 reacciones. - Etiquetar los tubos de 0,2 mL en consecuencia y añadir 2 μL de la muestra de cDNA a 13 μL de la mezcla maestra y mezclar suavemente. En general, mida cada muestra 2x.
Nota: se requieren al menos dos muestras de control negativas, además, utilice agua sin RNase como muestra. Si la curva de calibración no se determina en la misma ejecución que la muestra, también se requiere una muestra de la medida de la curva de calibración. Se utiliza para calibrar cada corrida individual a la misma eficiencia de reacción. - Coloque los tubos en la máquina PCR e inicie el siguiente programa: 2 min a 50 ° c, 10 min a 95 ° c, 15 s a 95 ° c y 60 s a 60 ° c. Repite los dos últimos pasos del programa hasta 50X.
- Para un análisis de los valores de cq con el paquete de software de la máquina PCR, activar la normalización dynamictube (para la compensación de diferentes niveles de fondo utilizando la segunda derivada de cada rastro de la muestra) y ruido pendiente Corrección (normalización al nivel de ruido).
Nota: el valor de cq del control negativo debe ser varios ciclos superiores al valor más alto de la muestra cq .- Para un análisis de curva de calibración, determine el umbral para el cálculo de la cq para cada Mirna de las curvas estándar de cada Mirna individualmente, utilizando la función de umbral de búsqueda automática del paquete de software y manténla constante para cada miRNA específica.
- Para el análisis de muestras, si es necesario, compensar las diferentes eficiencias de reacción de la ejecución de la muestra con el punto de datos de la muestra de curva de calibración. El software calcula los valores de cq de las muestras a partir del nivel de umbral de la medida de la curva de calibración respectiva.
10. cálculo del contenido de miRNA
-
Curva de calibración
- Calcular, a partir del número inicial de hebras de miRNA por alícuota (100 μL de miRNA de 10 μM, peso molecular de la hoja de datos) y los siguientes pasos de dilución en serie, el número de hebras de miRNA en el volumen de la muestra (el volumen de muestra de 5 μL del paso 8,3).
Nota: suponiendo una eficiencia de transcripción inversa de 1, este número es igual al número de hebras de cDNA. - Calcule el número de hebras de cDNA en el volumen de muestra de 2 μL del paso 9,3. Considere de este modo el factor de dilución adicional de 7,5 (el volumen de muestra de 2 μL del paso 9,4 del volumen de muestra de 15 μL del paso 8,4).
- Trace el valor de cq del paso 9.5.1 en el número de hebras nhebras calculadas en el paso 10.1.2 en un trazado semilogarítmico base 10, y ajuste los puntos de datos con la siguiente curva de regresión (M = la pendiente de la curva de regresión, B = offset).
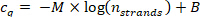
Confirme que el coeficiente de correlación (R2) de la línea es > 0,99.
- Calcular, a partir del número inicial de hebras de miRNA por alícuota (100 μL de miRNA de 10 μM, peso molecular de la hoja de datos) y los siguientes pasos de dilución en serie, el número de hebras de miRNA en el volumen de la muestra (el volumen de muestra de 5 μL del paso 8,3).
-
Las partículas de lipoproteína
- Calcule el número de hebras de miRNA por volumen de muestra utilizando el valor de c de muestra medido (paso 9.5.2) y la siguiente ecuación (M y B son los parámetros de la curva de calibración de la Mirna específica).
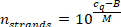
- Calcular el número correspondiente de partículas de lipoproteína en el volumen de la muestra, a partir del volumen en el paso 7.2.1 (100 μL), su concentración conocida, y los siguientes pasos de dilución (100 μL-> 30 μL de volumen de muestra en el paso 7.1.10-> 5 μL [dilución 1:6] muestra en el volumen total de 15 μL en el paso 8,4-> 2 μL [dilución 1:7.5] en el paso 9,4). Asuma un peso molecular de 250 kDa para partículas de HDL y 3 MDa para partículas de LDL y una recuperación del 100% de miRNA durante el paso de extracción de miRNA (ignorando cualquier contribución lipídica al peso molecular produce una ligera sobreestimación del número de hebras de miRNA por partícula de lipoproteína).
- Divida el número de hebras de miRNA del paso 10.2.1 por el número de partículas calculadas en el paso anterior para producir el número de hebras de miRNA por partícula de lipoproteína.
- Calcule el número de hebras de miRNA por volumen de muestra utilizando el valor de c de muestra medido (paso 9.5.2) y la siguiente ecuación (M y B son los parámetros de la curva de calibración de la Mirna específica).
-
Células
- Calcule el número de hebras de miRNA por volumen de muestra según el paso 10.2.1.
- Calcule el número correspondiente de células en el volumen de la muestra según el paso 10.2.2, comenzando con la concentración inicial del número de células medida en el paso 6,4.
- Divida el número de hebras de miRNA de 10.3.1 por el número de celdas calculadas en el paso anterior para producir el número de hebras de miRNA por célula.
- Calcule la tasa de captación de partículas de lipoproteínas dividiendo la cantidad total de hebras de miRNA del paso anterior después de la corrección para el nivel de miRNA de fondo de las células obtenidas de un experimento de control negativo por el ratio miRNA/partícula (paso 10.2.3 ) y el tiempo de incubación (16 h, ver paso 6,2).
11. matrices de microfluidos multiwell
-
la extracción de miRNA
- Realice la extracción de miRNA como se describe en el paso 7.
-
La transcripción inversa
- Descongelar los imprimadores de transcripción inversa, los componentes del kit de transcripción inversa y MgCl2 (25 mm) sobre hielo. Para ocho muestras, mezclar 8 μL de imprimadores de transcripción inversa (10x), 2,25 μL de dNTPs con dTTP (100 mM), 16,88 μL de transcriptasa inversa (50 U/μL), 9,00 μL de tampón de transcripción inversa 10x, 10,12 μL de MgCl2, 1,12 μl de inhibidor de RNase (20 U/μL) , y 1 μL de agua sin nucleasa.
- Mezclar suavemente y centrifugar brevemente. Añadir 4,3 μL de mezcla de reacción de transcripción inversa a 3,5 μL de miRNA extraída en un tubo y mezclar, girar hacia abajo e incubar sobre hielo durante 5 min. Coloque los tubos en una máquina termocicladora e inicie el siguiente programa: 16 ° c durante 2 min, 42 ° c durante 1 min , y 50 ° c para 1 s repetido 40x en total, y luego, como reacción de parada, 85 ° c durante 5 min y mantener a 4 ° c hasta que se detuvo.
-
Preamplificación
- Descongelar los cebadores sobre hielo e invertir y centrifugar brevemente. Mezclar la mezcla maestra de preamplificación (2x) girando la botella. Preparar la mezcla de reacción de preamplificación de acuerdo con la siguiente instrucción para ocho muestras: 112,5 μL de mezcla maestra de preamplificación (2x), 22,5 μL de imprimadores de preamplificación y 67,5 μL de agua sin nucleasa. Invierta y centrifugue brevemente.
- Mezclar 2,5 μL del producto de reacción de transcripción inversa desde el paso 11.2.2 con 22,5 μL de mezcla de reacción de preamplificación del paso anterior e invertir y centrifugar brevemente. Incubar las muestras sobre hielo durante 5 min.
- Colocar los tubos en una máquina termocicladora e incubar en las siguientes configuraciones: activación enzimática a 95 ° c durante 10 min, recocido a 55 ° c durante 2 min, extendiéndose a 72 ° c durante 2 min, repetida 12x: desnaturalización a 95 ° c durante 15 s y recocido/extendiéndose a 60 ° c durante 4 min , Inactivación enzimática a 99,9 ° c durante 10 min, y 4 ° c en espera.
- Girar hacia abajo, añadir 7,5 μL de 1x TE (pH 8,0) y 67,5 μL de agua sin nucleasa, invertir y centrifugar brevemente. Las muestras se pueden almacenar a-20 ° c durante un máximo de 1 semana.
-
Qpcr
- Mezclar la mezcla principal girando la botella. Preparar la mezcla de reacción PCR para una tarjeta: 450 μL de mezcla maestra, 441 μL de agua sin nucleasa y 9 μL de la muestra de preamplificación del paso 11.3.4. Invierta y centrifugue brevemente.
- Cargue cada depósito de llenado de la tarjeta de matriz de microfluidos multipozo con 100 μL de mezcla de reacción de PCR preparada según las instrucciones del fabricante y centrifugue 2x durante 1 min a 3.000 x g. La aceleración durante los dos pasos de centrifugación consecutivos es importante para llenar correctamente la tarjeta. Selle la tarjeta de acuerdo con las instrucciones del fabricante.
- Utilice un sistema de PCR con el siguiente programa: activación enzimática a 95 ° c durante 10 min y luego, repetida 40x: desnaturalización a 95 ° c durante 15 s y recocido/extendiéndose a 60 ° c durante 1 min.
- Importe el archivo de resultados del sistema PCR y calcule los valores de RQ utilizando el paquete de software del sistema con los siguientes ajustes de análisis: un valor de cq máximo admisible de 40,0, valores máximos de cq en los cálculos incluidos y valores atípicos entre réplicas excluidas. Active la tasa de descubrimiento falso de Benjamini – Hochberg como opción para el ajuste del valor p(corrección de la ocurrencia de falsos positivos14) y la normalización global como método de normalización (utilizando un valor de umbral mediano para todas las muestras 15).
Resultados
Un esquema general de aislamiento de partículas de lipoproteínas
La figura 1 muestra el esquema general del aislamiento de partículas de lipoproteínas a partir de sangre entera, utilizando la ultracentrifugación de flotación secuencial16. Si se desea, se pueden cosechar otras fracciones de partículas de lipoproteínas como las partículas VLDL y IDL durante este protocolo. El rotor de titanio de ángulo fijo en combinación con los tubos de sellado rápido de polipropileno es adecuado para resistir las fuerzas de centrifugación. Para evitar el colapso del tubo, es importante evitar las burbujas de aire en el tubo. La centrifugación se lleva a cabo a 4 ° c para minimizar la degradación de las proteínas. Normalmente a partir de plasma (60-80 mL por donante) de donaciones de sangre agrupadas de tres voluntarios, se puede esperar un rendimiento de volúmenes de solución de partículas de LDL y HDL de 3 mL cada uno con concentraciones en el rango de 1-3 mg/mL. Todo el procedimiento, a partir de la donación de sangre, tomó alrededor de 7 días.
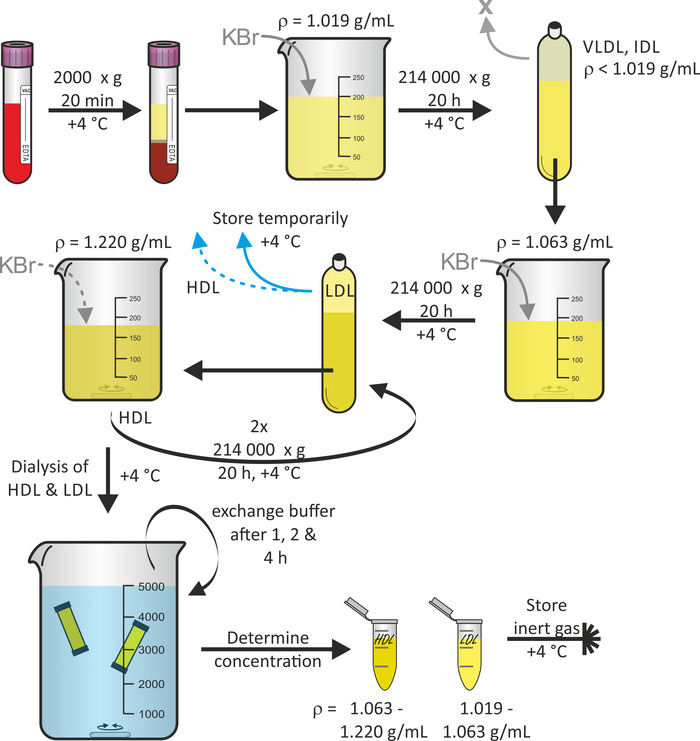
Figura 1: Diagrama de flujo de aislamiento de lipoproteínas. Centrifugar sangre de voluntarios sanos en tubos de contenedores de vacío y recolectar plasma (fase superior). Después del ajuste de su densidad a ρ = 1,019 g/ml usando KBR, Centrifugue la solución a 214.000 x g para 20 h a 4 ° c. Después del ajuste de la densidad de la fracción inferior a ρ = 1,063 g/ml usando KBR, Centrifugue la solución de nuevo a 214.000 x g para 20 h a 4 ° c. Almacene la fracción superior que contiene partículas de LDL temporales a 4 ° c. Después del ajuste de la densidad de la fracción inferior a ρ = 1,220 g/ml usando KBR, Centrifugue la solución dos veces a 214.000 x g para 20 h a 4 ° c. Recoja la fracción superior que contiene partículas de HDL, Dializa las soluciones de partículas de HDL y LDL y intercambia el tampón después de 1, 2 y 4 h. Después de 24 h, determinar la concentración de proteína y almacenar las muestras bajo gas inerte a 4 ° c. Por favor, haga clic aquí para ver una versión más grande de esta figura.
La reconstitución de partículas HDL
La reconstitución de partículas HDL se realizó de acuerdo con un protocolo previamente publicado por Jonas7. El primer paso fue la delipiación de partículas HDL como se muestra en la figura 2A, seguida por el segundo paso de la refundación (es decir, la reconstitución) como se muestra en la figura 2B, utilizando lípidos PC, Co, y C además de una mezcla de Mirna y spermina. Elegimos humano maduro miR-223 y miR-155 porque miR-223 muestra una gran abundancia y miR-155 es raro en partículas de lipoproteínas17. Normalmente, ambos pasos se realizan en dos días secuenciales. Durante la reconstitución, se podrían añadir otros componentes lipofílicos y/o anfifílicos como se desee. La evaporación completa del éter de etanol/dietilo y el disolvente de metanol/cloroformo de PC, CO y C fue fundamental. El último paso, como se muestra en la figura 2C, fue el procedimiento de diálisis para separar las partículas de HDL reconstituidas (RHDL) de los lípidos/Mirna/detergente libres. Esto tomó un adicional de 1-2 días. La adición de perlas absorbentes a la solución de diálisis mantuvo constante el gradiente de densidad a lo largo de la membrana de diálisis. Se puede esperar un rendimiento del 50% de las partículas de rHDL.
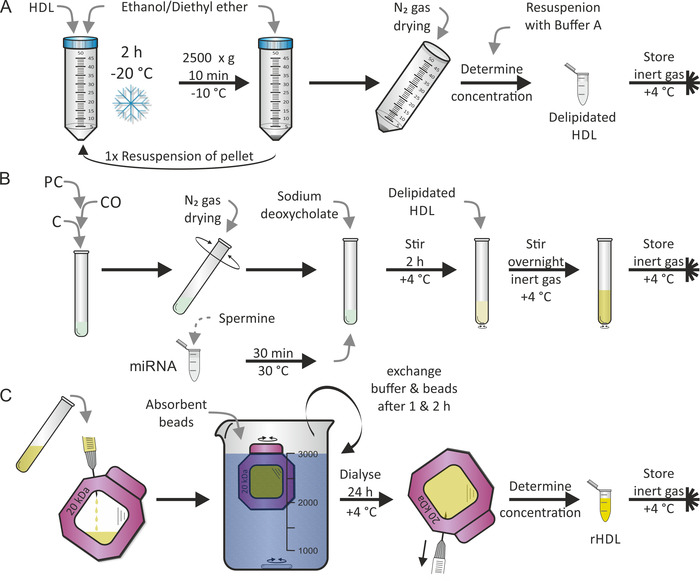
Figura 2: Diagrama de flujo de la reconstitución de partículas HDL. (A) delipidation: mezclar la solución de partículas HDL con etanol preenfriado/éter dietílico e incubar a-20 ° c durante 2 h. Después de desechar el sobrenadante, Resuspender el pellet y repita el procedimiento. Secar el pellet con N2 gas y resuspender en el buffer A. Después de la determinación de la concentración, almacene el HDL delipifado bajo atmósfera de gas inerte a 4 ° c. (B) reconstitución: después de mezclar la fosfatidil-colina (PC), el oleato de colesteryl (Co) y el colesterol (C), evaporan el disolvente usando N2 gas mientras giran el tubo. Incubar la alícuota de miRNA con solución de spermina durante 30 min a 30 ° c, añadir desoxicolato sódico y resuspender la película lipídica seca. Remover la muestra durante 2 h a 4 ° c, añadir la solución de HDL delipiada, y remover la muestra de nuevo, esta vez durante la noche a 4 ° c bajo atmósfera de gas inerte. (C) diálisis: transfiera la solución del panel B que contiene partículas de HDL reconstituida (RHDL) a una cámara de membrana de diálisis (corte de peso molecular: 20 kDa) y DIALIZARLO contra PBS y perlas absorbentes a 4 ° c. Intercambiar el buffer y las perlas después de 1 h y 2 h. recupere la solución de partículas rHDL después de 24 h, determine la concentración y almacene la muestra en atmósfera de gas inerte a 4 ° c. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Etiquetado de partículas de LDL
El etiquetado de las partículas de LDL con miRNA (figura 3) como se demostró para las partículas de HDL no fue factible debido a la hidrofobicidad de la proteína apob-100, que es el principal constituyente de la partícula de LDL. DMSO se utilizó para la penetración de la monocapa lipídica de la partícula de LDL y, por lo tanto, medió la Asociación miRNA. Todo el procedimiento tardó 1-2 días con un rendimiento cercano al 100%.
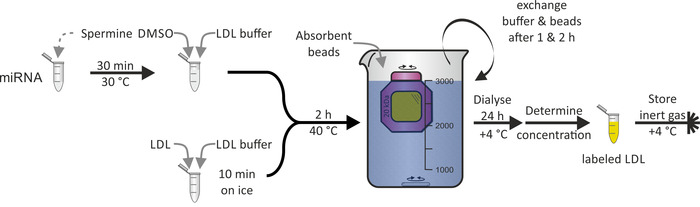
Figura 3: Diagrama de flujo de partículas de LDL etiquetado. Incubar la alícuota de miRNA con solución de spermina durante 30 min a 30 ° c y añadir DMSO y búfer de LDL. Incubar LDL muestra ingenio buffer LDL durante 10 min en hielo y añadir miRNA/spermine/DMSO mezcla. Después de la incubación a 40 ° c durante 2 h, transferir la solución a una cámara de membrana de diálisis (corte de peso molecular: 20 kDa) y dializarlo contra PBS y granos absorbentes a + 4 ° c. Búfer de intercambio y granos después de 1 & 2 h. recuperar la solución de partículas de LDL etiquetada después de 24 h, determinar la concentración y almacenar bajo atmósfera de gas inerte a + 4 ° c. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Control de calidad de partículas de lipoproteínas
HS-AFM se puede utilizar para examinar el tamaño y la forma de las partículas de lipoproteína nativas y reconstituidas/etiquetadas en la mica. Justo antes de su uso, mica tiene que ser recién hendecido (utilizar cinta adhesiva para eliminar la capa superior [s]) con el fin de proporcionar una superficie limpia y plana. Cuando se incuban partículas de HDL/LDL en mica, el factor de dilución (y/o el tiempo de incubación) debe ajustarse para observar las partículas individuales. Los clústeres no permiten la determinación de las cotas de partícula. Las partículas HDL son móviles en mica. Cuando se utiliza AFM convencional en lugar de HS-AFM, el protocolo de inmovilización debe adaptarse en consecuencia (tampón, recubrimiento superficial) para reducir la movilidad de partículas laterales. Durante el escaneo de la muestra, la fuerza de imagen debe mantenerse baja (modo de tapping) para evitar cualquier deformación de las partículas, que por lo tanto afectará a los valores medidos. Para el análisis de datos, se detectaron partículas mediante un algoritmo de umbral (p. ej., en Gwyddion: granos > marca por umbral) y su altura se determinó con respecto a la superficie de mica. La medición de la altura de las partículas es la forma más precisa de determinar el tamaño de las partículas, ya que las dimensiones laterales aparentes se amplían con la forma de la punta (ver imágenes ejemplares en la figura 4). Las funciones de densidad de probabilidad (PDF) de las alturas de partícula se calcularon para la evaluación estadística y la comparación de distribuciones de tamaño de las diversas partículas de lipoproteínas. Una comparación de partículas nativas y con la etiqueta de miRNA de LDL, como se muestra en la figura 4 , hace posible verificar la similitud principal entre las partículas de lipoproteínas etiquetadas y no etiquetadas (es decir, nativas) (etiquetadas como partículas de LDL sin la adición de Mirna/ las mezclas de espermatozoides se muestran como un control para el procedimiento de etiquetado en sí mismo). Todo el procedimiento tardó aproximadamente 1 día.
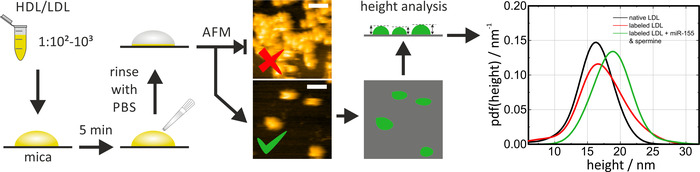
Figura 4: Diagrama de flujo y resultados representativos de las mediciones HS-AFM. Diluir la muestra de partículas de HDL/LDL en PBS (1:102-1:103) e incubarla en mica recién hendecida durante 5 min, seguida de un enjuague cuidadoso con PBS para eliminar las partículas libres (no electrostáticamente adsorbida). Realice imágenes HS-AFM y Compruebe la densidad de partículas en la superficie. Realice las mediciones en PBS a temperatura ambiente. La imagen superior de esta figura muestra una densidad de partículas demasiado alta; la imagen inferior es adecuada para el análisis. La altura de las partículas individuales se analizó después de la trilla y las partículas nativas (curva negra) y reconstituida/etiquetada (curva roja y verde) se compararon en una evaluación estadística. La barra de escala = 100 nm. Esta cifra ha sido modificada de Axmann et al.19. Por favor, haga clic aquí para ver una versión más grande de esta figura.
la extracción de miRNA, la transcripción inversa y la qPCR
La extracción de miRNA de lipoproteínas nativas/enriquecidas artificialmente o muestras celulares se realizó utilizando un kit de extracción de miRNA como se muestra en la figura 5A. Por la presente, un entorno sin RNase era crítico. Este paso tardó aproximadamente 1 h. la transcripción inversa de la muestra de miRNA extraída (figura 5b) se realizó utilizando procedimientos bioquímicos estándar descritos por el fabricante. Este paso tardó aproximadamente 1,5 h. Finalmente, la cantidad de cDNA obtenida durante el último paso se determinó utilizando qPCR (figura 5C). Una curva estándar, que relaciona los valores de cq obtenidos con el número de hebra de Mirna absoluto, arrojó el contenido absoluto de Mirna de la muestra inicial. Esto tardó aproximadamente 2,5 h.
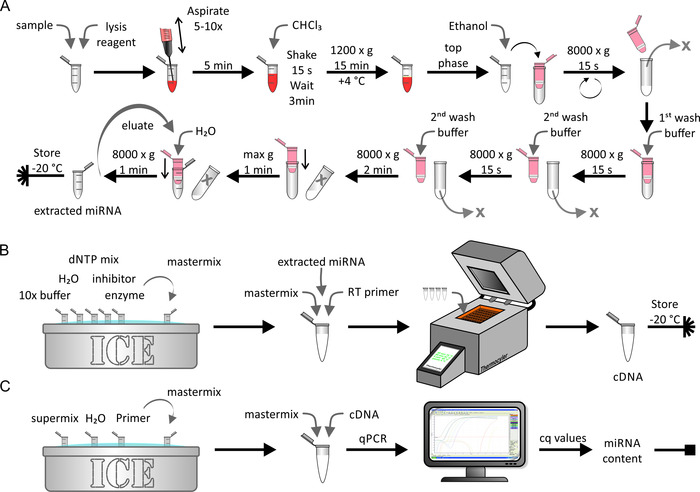
Figura 5: Diagrama de flujo de extracción de miRNA, transcripción inversa y qPCR. (A) extracción de Mirna: mezclar la muestra con reactivo de lisis y liarla mediante aspiración utilizando una jeringa. Incubar durante 5 min y añadir CHCl3. Agitar vigorosamente durante 15 s e incubar durante 3 min. Después de centrifugación a 1.200 x g durante 15 min a 4 ° c, recoja la fase superior y mezcle con etanol. Transfiera la solución a una columna de centrifugado (volumen máximo < 700 μL) y centrifugue a 8.000 x g durante 15 s. Deseche el eluyente y repita el último paso con el resto de la solución. Añada el primer tampón de lavado y centrifugue a 8.000 x g durante 15 s. Deseche el eluyente, añada el segundo tampón de lavado y centrifugue a 8.000 x g durante 15 s. Repita el último paso con un tiempo de centrifugación de 2 min. Seque aún más la membrana mediante centrifugación a velocidad máxima durante 1 min. Elute el miRNA con H2O y centrifugue a 80.000 x g durante 1 min. Almacene la muestra de Mirna extraída a-20 ° c. (B) transcripción inversa: descongelar el tampón 10x, H2O, dNTP Mix, inhibidor, y la enzima en el hielo y preparar la mezcla maestra. Añadir el miRNA extraído del panel a a la mezcla maestra y la imprimación de transcripción inversa y realizar la transcripción inversa utilizando una máquina termocicladora. Almacene el ejemplo de cDNA a-20 ° c. (C) qPCR: descongelar la Supermix, H2O, y imprimación sobre hielo y preparar la mezcla maestra. Agregue el cDNA del panel B a la mezcla maestra y realice qPCR. Analice los datos para obtener valores de cq y calcule el contenido absoluto de Mirna de la muestra (consulte la figura 6 y los resultados representativos para obtener más información). Por favor, haga clic aquí para ver una versión más grande de esta figura.
El contenido absoluto de miRNA y la velocidad de transferencia
El contenido absoluto de miRNA de partículas de HDL y LDL nativas y enriquecidas artificialmente se calculó a partir de los valores de cq de las muestras y una curva estándar de la Mirna respectiva, tal como se muestra en la figura 6. La figura 6A muestra los datos calculados por el software de análisis (con la normalización dynamictube activada [para la compensación de diferentes niveles de fondo utilizando la segunda derivada de cada traza de muestra] y la corrección de pendiente de ruido [normalización al nivel de ruido]). c los valoresq de las curvas estándar se determinaron utilizando la función de umbral auto-Find del paquete de software en la señal de fluorescencia normalizada medida por la máquina qPCR. Por la presente, el software maximizó el valor R del ajuste de la curva estándar. El nivel de umbral se mantuvo constante para cada análisis de muestra de miRNA específico. Posteriormente, los valores de cq se trazaron como una función del número de hebras de Mirna y se calculó una línea de regresión. Los valores de c de la muestra se determinaron con el mismo nivel de umbral que se muestra en la Figura 6B; las diferencias de eficiencia de reacción entre diferentes ejecuciones de qPCR fueron compensadas automáticamente por el software utilizando una muestra de curva de calibración adicional incluida en cada corrida. Utilizando la ecuación de la línea de regresión, se podría calcular la cantidad desconocida de miRNA en la muestra. El número de partículas de lipoproteínas se estimó a partir de la concentración inicial de proteínas y su peso molecular medio (MWHDL ~ 250 kDa). Por lo tanto, no se asumió ninguna contribución de lípidos al peso molecular, por lo tanto, el número de hebras de miRNA por partícula de lipoproteína se sobrestimó ligeramente. Además, se asumió una tasa de recuperación del 100% de miRNA durante el paso de extracción de miRNA. Además, se determinó el contenido de miRNA de las células antes y después de la incubación con partículas de HDL y se calculó la tasa de transferencia de miRNA como se muestra en la Figura 6C.
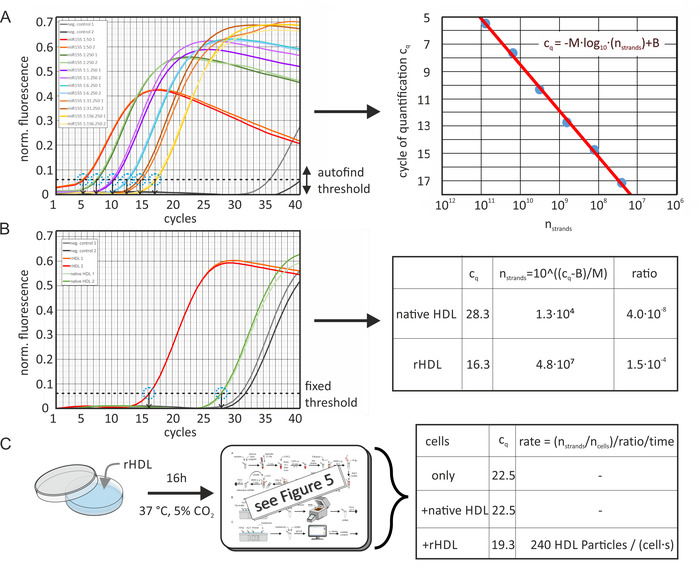
Figura 6: Diagrama de flujo de cálculo del contenido absoluto de miRNA y tasa de transferencia. (A) curva estándar para mir-155: una alícuota mir-155 (100 μl, 10 μm) se diluyó en serie con agua libre de ARN como se indicó. qPCR produjo valores cq para cada muestra de dilución serial (medido dos veces) utilizando la función de umbral de auto-Find del paquete de software. Los experimentos de control negativo (sin la adición de miRNA) arrojaron valores de cq de > 35. Los puntos de datos de los valores de cq como función del número de hebras de Mirna por volumen de muestra (calculado a partir de la concentración inicial y las diluciones en serie) fueron equipados con la ecuación presentada (línea roja, imagen derecha), produciendo M =-3,36 y B = 42,12. La eficacia de PCR determinada fue de 0,98. Las barras de error se calcularon a partir de los resultados de las repeticiones experimentales y eran más pequeñas que el diámetro del círculo de puntos de datos. (B) los valores de cq de las partículas de HDL nativas/enriquecidas artificialmente se determinaron con el mismo nivel de umbral que se determinó en el panel a y se convirtieron al número de hebras de Mirna en el volumen de muestra de qPCR. La proporción absoluta de miRNA de la muestra original se calculó a partir del número (concentración) de partículas de HDL en el volumen de la muestra (3,2 x 1011 partículas). (C) las muestras celulares (línea celular LDLA7-SRBI) fueron incubadas durante 16 h con partículas de HDL enriquecidas artificialmente (50 μg/ml) y analizadas de manera similar. Los valores cq determinados fueron 22,5, 22,5 y 19,3 sólo para las células, para las células INCUBADAS con HDL nativo, o para las células incubadas con solución de partículas RHDL (ambas 50 μg/ml), respectivamente. Estos valores se convirtieron en el número de hebras de miRNA como se hace en el panel B. El número de hebras de miRNA después de la incubación (7,3 x 106) se corrigió por sustracción del número de hebras de Mirna antes de la incubación (8,6 x 105). El resultado se dividió por el número de celdas en el volumen de la muestra (3.100), la proporción de partículas de miRNA (1,5 x 10-4) y el período de tiempo de incubación (16 h). Esto produjo la tasa de transferencia de partículas de lipoproteína a través de captación de miRNA (240 eventos de absorción de partículas HDL por célula y segundo). Esta cifra ha sido modificada de Axmann et al.19. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Matriz de microfluidos multiwell
Debido a los pequeños rendimientos de la extracción de miRNA, la transcripción inversa de la miRNA extraída fue seguida por un paso de preamplificación. Finalmente, se realizó qPCR, como se muestra en la figura 6. Para todos los pasos, se utilizaron procedimientos bioquímicos estándar según lo descrito por el fabricante. Aquí, se muestra una parte del perfil global de miRNA sobre las partículas de HDL de pacientes urómicos reclutados para un estudio sobre la influencia del MRC en el efluente del colesterol de los macrófagos18 . En este estudio, la capacidad de aceptador de colesterol de HDL o suero en, además de otros, 17 pacientes urómicos jóvenes adultos (etapas CKD 3-5) y 14 pacientes de hemodiálisis de adultos jóvenes sin enfermedades asociadas y controles coincidentes se midió. Para analizar los datos, se utilizaron los ajustes predeterminados (valor máximo admisible de CT: 40,0, incluyendo los valores máximos de CT en los cálculos y excluyendo los valores atípicos entre réplicas). Los valores Pse ajustaron utilizando la tasa de descubrimiento falso de Benjamini-Hochberg (corrección de la ocurrencia de falsos positivos), y como método de normalización, se seleccionó la normalización global , que encuentra los ensayos comunes entre todos los muestras para utilizar su mediana de CT para la normalización. En los resultados representativos, se representan algunos RQs de miRNAs aislados de HDLs de pacientes urómicos (los RQs de los controles son 1). Obviamente, miR-122 y miR-224 están muy expresados en las HDLs de pacientes urómicos. Todo este paso tardó aproximadamente 1 día.
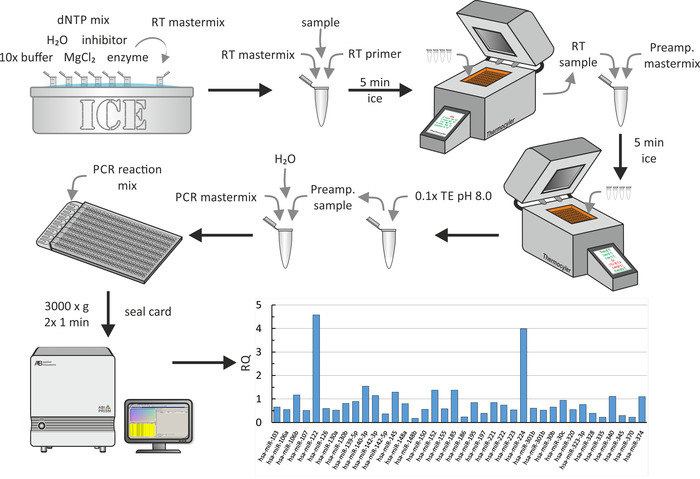
Figura 7: Diagrama de flujo y resultados representativos de la matriz de microfluidos multipozo. Después de la extracción de miRNA como se muestra en la figura 5A, mezclar la muestra de Mirna con imprimación de transcripción inversa y una mezcla principal que contiene 10x buffer, H2O, dNTP Mix, inhibidor, MgCl2, y la enzima. Después de la incubación en hielo durante 5 min, realice la transcripción inversa usando una máquina termocicladora. Añadir la mezcla maestra de preamplificación, incubar durante 5 min en hielo, y realizar preamplificación usando una máquina termocicladora. Añadir 0.1 x TE (pH 8,0) y mezclar una alícuota con mezcla maestra de PCR y H2O. Pipetear la mezcla de reacción PCR en el puerto de llenado de la matriz de microfluidos multipozo y girar dos veces a 3.000 x g durante 1 min cada una. Realice qPCR utilizando un sistema de PCR y analice los datos para producir valores de RQ (aquí, la figura muestra los valores de RQ de las partículas de HDL de pacientes con uremia en comparación con un grupo de control saludable18). Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
Aquí, el aislamiento de las fracciones de partículas de lipoproteínas de la sangre humana y la determinación de su contenido individual de miRNA se describe paso a paso. Es fundamental trabajar en un entorno libre de RNase mientras se maneja miRNA aislada y sintetizada: miRNA incrustada de partículas está obviamente protegida de la degradación enzimática. Como la relación miRNA/partícula de las partículas de lipoproteínas nativas es bastante baja, se requiere enriquecimiento artificial con miRNA para estudiar la captación de partículas holo de las células. Por lo tanto, la reconstitución de partículas HDL como se describió anteriormente7 se modifica para incorporar hebras de Mirna. Además, la separación de la fracción de lípidos y proteínas durante este procedimiento permite a los científicos examinar los componentes asociados a lípidos y proteínas de la partícula de lipoproteína19. De manera similar, se adapta el procedimiento de etiquetado de las partículas de LDL. Curiosamente, la adición de espermina — un estabilizador natural de los nucleótidos — no influyó en la relación miRNA/partícula. Cabe señalar que, en principio, el método permite la inplegamiento de otras sustancias que miRNA dentro de una partícula de lipoproteína. Por supuesto, hay un límite con respecto al tamaño físico de la sustancia basado en el tamaño total de HDL (diámetro: 5-12 Nm) y las partículas de LDL (diámetro: 18-25 nm).
En cuanto al control de calidad de las partículas de lipoproteínas reconstituidas/etiquetadas, el HS-AFM es un método aplicable para caracterizar las partículas de HDL/LDL en el nivel de partícula única. En comparación con el EM, permite tiempos de preparación más cortos y condiciones casi fisiológicas (húmedo, temperatura ambiente).
Debido a su inherente sensibilidad y amplificación, la qPCR es el método de elección para detectar bajas concentraciones de miRNA. Alternativamente, la microscopía de fluorescencia sensible a una molécula, que es capaz de detectar incluso moléculas individuales, no sería adecuada debido a las bajas concentraciones de, por ejemplo, hebras de miRNA etiquetadas con fluorescentemente por partícula. Por lo tanto, se ha encontrado que la proporción de hebras de miRNA por partícula de lipoproteína nativa es de 10 a8. El enriquecimiento artificial aumenta la relación por un factor de 10.000, lo que facilita la estimación de la tasa de captación de lipoproteínas celulares (no se detecta ninguna diferencia significativa usando partículas nativas de lipoproteína 19). La alta sensibilidad de la qPCR hace posible determinar esta tasa de captación midiendo el número de hebras de miRNA después del tiempo de incubación y la relación miRNA/partícula. Cabe señalar que el valor calculado ignora la degradación celular y la liberación de miRNA y, por lo tanto, representa al menos un límite inferior para la proporción de captación de partículas de lipoproteínas.
En el futuro, el método se puede adaptar para transferir sustancias farmacéuticas (especialmente también las lipofílicas) en las células y correlacionar su efecto biológico con la concentración intracelular (determinada a través de la tasa de captación de partículas de lipoproteínas).
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el proyecto del fondo científico austriaco P29110-B21, la "Hochschuljubiläumsstiftung der Stadt Wien Zur Förderung der Wissenschaft" proyecto H-3065/2011, el Fondo Europeo para el desarrollo regional (EFRE, IWB2020), el estado federal de Upper Austria, y el "Land OÖ Basisfinanzierung".
Materiales
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge | Beckman | Optima L-100 XP | Lipoprotein isolation |
| Ultracentrifuge rotor | Beckman | TI 55.2 | Lipoprotein isolation |
| Vacutainer (EDTA) | Becton Dickinson | 366643 | Lipoprotein isolation |
| Kaliumbromid | Sigma Aldrich | 2110 | Lipoprotein isolation |
| Ultracentrifuge tubes | Beckman | 342414 | Lipoprotein isolation |
| Ultracentrifuge tube sealer | Beckman | 342428 | Lipoprotein isolation |
| Sodiumchlorid | Carl Roth GmbH | P029.2 | Lipoprotein isolation |
| EDTA | Fisher Scientific | D/0650/50 | Lipoprotein isolation |
| Dialysis tubes, MWCO 12 - 14k | Spectra | 132700 | Lipoprotein isolation |
| synthetic miRNA | microSynth | - | synthetic miRNA |
| TRIS buffer pH 7 | Ambion | AM9850G | synthetic miRNA |
| TRIS buffer pH 8 | Ambion | AM9855G | synthetic miRNA |
| sterile tubes | Carl Roth GmbH | ENE8.1 | synthetic miRNA |
| Photometer | Eppendorf | Eppendorf Biophotometer | Bradford assay |
| Cuvette | Carl Roth GmbH | XK20 | Bradford assay |
| Coomassie G-250 Stain | BioRad GmbH | 161-0786 | Bradford assay |
| Sodiumchlorid | Carl Roth GmbH | 3957.1 | Delipitation |
| EDTA | Fluka Analytical | 03690-100ML | Delipitation |
| TRIS buffer pH 8.0 | Ambion | AM9855G | Delipitation |
| Ethanol 100% | AustrAlco | Ethanol Absolut 99.9% | Delipitation |
| Diethyl ether | Carl Roth GmbH | 3942.1 | Delipitation |
| Centrifuge | Thermo Scientific | Multifuge X3R | Delipitation |
| Chloroform | Carl Roth GmbH | 3313.1 | Reconstitution |
| Methanol | Carl Roth GmbH | 4627.1 | Reconstitution |
| Phosphatidylcholine | Sigma Aldrich GmbH | P3556-25mg | Reconstitution |
| Cholesterol oleate | Sigma Aldrich GmbH | C-9253-250mg | Reconstitution |
| cholesterol | Sigma Aldrich GmbH | C8667-500mg | Reconstitution |
| glass tubes | Carl Roth GmbH | K226.1 | Reconstitution |
| spermine | Sigma Aldrich GmbH | S3256-1G | Reconstitution |
| Thermomixer | Eppendorf | Thermomixer comfort | Reconstitution |
| Sodium deoxycholate | Sigma Aldrich GmbH | 3097-25G | Reconstitution |
| Magnetic stir bar | Carl Roth GmbH | 0955.2 | Reconstitution |
| 100µL micropipettes | Carl Roth GmbH | A762.1 | Reconstitution |
| PBS | Carl Roth GmbH | 9143.2 | Dialysis |
| Amberlite XAD-2 beads | Sigma Aldrich GmbH | 10357 | Dialysis |
| Slide-A-Lyzer casette (cut-off 20 kDa) | Thermo Scientific | 66003 | Dialysis |
| Syringe | Braun | 9161406V | Dialysis |
| Syringe needle | Braun | 465 76 83 | Dialysis |
| EGTA | Carl Roth GmbH | 3054.2 | Labeling of LDL particles |
| DMSO | life technologies | D12345 | Labeling of LDL particles |
| Atomic Force Microscope | RIBM | SS-NEX | Quality control of reconstituted/labeled lipoprotein particles |
| Muscovite Mica (V-1 Grade) | Christine Gröpl | G250-1/V1 | Quality control of reconstituted/labeled lipoprotein particles |
| AFM Cantilever | Nanoworld | USC-F1.2-k0.15 | Quality control of reconstituted/labeled lipoprotein particles |
| Gwyddion 2.49 | Czech Metrology Institute | http://gwyddion.net | Quality control of reconstituted/labeled lipoprotein particles |
| Chamber slides, Nunc Lab-Tek | VWR | 734-2122 | Cell culture |
| HBSS | Carl Roth GmbH | 9117.1 | Cell culture |
| Cell counter | Omni Life Science GmbH | Casy | Cell culture |
| miRNeasy Mini Kit | QIAGEN | 217004 | miRNA extraction |
| Centrifuge | Eppendorf | 5415R | miRNA extraction |
| 20G needle | Braun | Sterican 465 75 19 | miRNA extraction |
| 5ml syringe | Becton Dickinson | 309050 | miRNA extraction |
| PCR cabinet | Esco | PCR-3A1 | Reverse Transcription |
| TaqMan Reverse Transcription Kit | Thermo Scientific | 4366596 | Reverse Transcription |
| Rnase-free water | Carl Roth GmbH | T143 | Reverse Transcription |
| 0.2 ml tubes | Brand | 781305 | Reverse Transcription |
| Centrifuge | Carl Roth GmbH | Microcentrifuge AL | Reverse Transcription |
| Thermocycler | Sensoquest | Labcycler | Reverse Transcription |
| TaqMan Primer | Thermo Scientific | - | qPCR |
| iTaq Universal probe supermix | BioRad GmbH | 1725131 | qPCR |
| PCR machine | Corbett | Rotor-Gene RG-6000 | qPCR |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366596 | TaqMan Array |
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | TaqMan Array |
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 | TaqMan Array |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399933 | TaqMan Array |
| TaqMan Universal Master Mix II, no UNG | Applied Biosystems | 4440040 | TaqMan Array |
| TaqMan Advanced miRNA Human A Cards | Applied Biosystems | A31805 | TaqMan Array |
| Nuclease-free water | Thermo Scientific | R0581 | TaqMan Array |
| MgCl2 (25mM) | Thermo Scientific | R0971 | TaqMan Array |
| TE, pH 8.0 | Invitrogen | AM9849 | TaqMan Array |
| 7900HT Fast Real-Time PCR System | Applied Biosystems | 4351405 | TaqMan Array |
| TaqMan Array Card Sealer | Applied Biosystems | - | TaqMan Array |
Referencias
- Vickers, K. C., Palmisano, B. T., Shoucri, B. M., Shamburek, R. D., Remaley, A. T. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nature Cell Biology. 13 (4), 423-435 (2011).
- Tabet, F., et al. HDL-transferred microRNA-223 regulates ICAM-1 expression in endothelial cells. Nature Communications. 5, (2014).
- Wahid, F., Shehzad, A., Khan, T., Kim, Y. Y. MicroRNAs: Synthesis, mechanism, function, and recent clinical trials. Biochimica et Biophysica Acta - Molecular Cell Research. 1803 (11), 1231-1243 (2010).
- Carthew, R. W., Sontheimer, E. J. Origins and Mechanisms of miRNAs and siRNAs. Cell. 136 (4), 642-655 (2009).
- Fabian, M. R., Sonenberg, N., Filipowicz, W. Regulation of mRNA Translation and Stability by microRNAs. Annual Review of Biochemistry. 79 (1), 351-379 (2010).
- Filipowicz, W., Bhattacharyya, S. N., Sonenberg, N. Mechanisms of post-transcriptional regulation by microRNAs: Are the answers in sight?. Nature Reviews Genetics. 9 (2), 102-114 (2008).
- Jonas, A. Reconstitution of High-Density Lipoproteins. Methods in Enzymology. , 553-582 (1986).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Bustin, S. A., et al. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Patsch, J. R., Patsch, W. Zonal ultracentrifugation. Methods in Enzymology. 129 (1966), 3-26 (1986).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Yamamoto, D., et al. High-Speed Atomic Force Microscopy Techniques for Observing Dynamic Biomolecular Processes. Methods in Enzymology. 475, 541-564 (2010).
- Stangl, H., Cao, G., Wyne, K. L., Hobbs, H. H. Scavenger receptor, class B, type I-dependent stimulation of cholesterol esterification by high density lipoproteins, low density lipoproteins, and nonlipoprotein cholesterol. Journal of Biological Chemistry. 273 (47), 31002-31008 (1998).
- Hochberg, B. Controlling the False Discovery Rate: a Practical and Powerful Approach to Multiple Testing. Journal of the Royal Statistical Society. 57 (1), 289-300 (1995).
- Mestdagh, P., et al. A novel and universal method for microRNA RT-qPCR data normalization. Genome Biology. 10 (6), (2009).
- Schumaker, V. N., Puppione, D. L. 6] Sequential Flotation Ultracentrifugation. Methods in Enzymology. 128 (C), 155-170 (1986).
- Wagner, J., et al. Characterization of levels and cellular transfer of circulating lipoprotein-bound microRNAs. Arteriosclerosis, Thrombosis, and Vascular Biology. 33 (6), 1392-1400 (2013).
- Meier, S. M., et al. Effect of chronic kidney disease on macrophage cholesterol efflux. Life Sciences. 136, 1-6 (2015).
- Axmann, M., et al. Serum and Lipoprotein Particle miRNA Profile in Uremia Patients. Genes. 9 (11), 533 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados