Method Article
Arricchimento delle particelle di lipoproteina nativa con microRNA e successiva determinazione del loro contenuto di microRNA assoluto/relativo e della loro velocità di trasferimento cellulare
In questo articolo
Riepilogo
Qui viene presentato un protocollo quantitativo basato sulla reazione a catena della polimerasi in tempo reale per la determinazione del contenuto di micro-RNA nativo (assoluto/relativo) delle particelle di lipoproteina. Inoltre, è dimostrato un metodo per aumentare il livello di micro-RNA, nonché un metodo per determinare il tasso di captazione cellulare delle particelle di lipoproteina.
Abstract
Le particelle di lipoproteina sono prevalentemente trasportatori di lipidi e colesterolo nel sangue. Inoltre, contengono piccole quantità di filamenti di microRNA non codificante (miRNA). In generale, miRNA altera il profilo delle espressioni proteiche a causa delle interazioni con il Messaggero-RNA (mRNA). Pertanto, la conoscenza del contenuto di miRNA relativo e assoluto delle particelle di lipoproteina è essenziale per stimare l'effetto biologico dell'assorbimento delle particelle cellulari. Qui viene presentato un protocollo quantitativo basato sulla reazione a catena della polimerasi (qPCR) in tempo reale per determinare il contenuto di miRNA assoluto delle particelle di lipoproteina, esemplificato come mostrato per le particelle di lipoproteina arricchite con miRNA. Il contenuto di miRNA relativo è quantificato utilizzando schede di array microfluidici multiwell Program. Inoltre, questo protocollo consente agli scienziati di stimare il miRNA cellulare e, quindi, il tasso di captazione delle particelle lipoproteiche. Un aumento significativo del livello di miRNA cellulare è osservabile quando si utilizzano particelle di lipoproteina ad alta densità (HDL) caricate artificialmente con miRNA, mentre l'incubazione con particelle di HDL native non produce alcun effetto significativo a causa del loro contenuto di miRNA piuttosto basso. Al contrario, l'assorbimento cellulare delle particelle di lipoproteina a bassa densità (LDL), né con miRNA nativo né caricato artificialmente, non ha alterato il livello di miRNA cellulare.
Introduzione
Le particelle di lipoproteina sono composte da un monostrato di lipidi e colesterolo amphiphilici che racchiude un nucleo di esteri di colesteryl e grassi del trigliceride. L'intera particella è stabilizzata da apolipoproteine incorporate nella membrana, che definiscono la funzionalità biologica della particella. Le particelle di lipoproteina possono essere distinte in base alla loro rispettiva densità crescente e, quindi, diminuire le dimensioni, vale a dire come lipoproteina a bassissima densità (VLDL), lipoproteina a densità intermedia (IDL), LDL e particelle di HDL. Nonostante il trasporto di componenti insolubili in acqua nel sangue, è stato dimostrato che le particelle di HDL trasportano filamenti non codificanti di Mirna1,2. I micro-RNA sono una classe di brevi filamenti (di solito due dozzine di nucleotidi), che degradano i filamenti di mRNA intracellularmente complementari e, quindi, modificano il profilo di espressione di certe proteine3,4,5, 6. il Inoltre, le alterazioni del profilo di miRNA sono state trovate in una varietà di malattie e, pertanto, il profilo è applicabile come un biomarcatore per la diagnosi e la prognosi. Il trasporto extracellulare di Mirna tra le cellule attraverso le particelle lipoproteiche può fungere da meccanismo aggiuntivo per la modulazione del livello di mRNA intercellulare. Per stimare quantitativamente l'effetto biologico, è necessaria la conoscenza del contenuto di miRNA assoluto e relativo delle particelle di lipoproteina.
La PCR in tempo reale quantitativa è un metodo adatto e relativamente rapido per ottenere queste informazioni. Pertanto, il valore di quantificazione relativa (RQ) può essere calcolato e le differenze relative tra i diversi campioni e le frazioni di lipoproteina sono stimabili. Le schede di matrice microfluidica multiwell sono un metodo rapido e facile da usare per determinare la presenza relativa (equivale al valore RQ) dei miRNA in un campione. Le schede di matrice microfluidica multiwell consistono di 96 o 384 camere di reazione individuali per le singole reazioni qPCR incorporate in un dispositivo microfluidico. Ogni camera contiene la sonda di idrolisi richiesta e i primer qPCR specifici per un singolo miRNA. I vantaggi sono un breve tempo di gestione dovuto alla standardizzazione, un semplice flusso di lavoro e un numero ridotto di passaggi di pipettaggio. Inoltre, il volume campione richiesto è minimizzato. Contrariamente alla quantificazione relativa, il contenuto assoluto di miRNA richiede un confronto dei risultati dei campioni qPCR con curve standard di numeri assoluti noti di filamenti miRNA. Va notato che, a causa del loro contenuto di miRNA relativamente basso, le tecniche di imaging sensibili a singola molecola non sono realizzabili: l'arricchimento artificiale delle particelle di lipoproteina con miRNA è inevitabile per studiare interazione delle particelle di lipoproteina e trasferimento di miRNA. Per quanto riguarda questo, delipidazione della particella HDL seguita con successiva relipidazione7 permette l'incorporazione di e, quindi, arricchimento con filamenti Mirna. L'arricchimento simile delle particelle LDL con miRNA non è fattibile a causa dell'idrofobicità della proteina apoB-100, che è il costituente principale della particella LDL. Tuttavia, con l'aggiunta del solvente polare dimetilsolfossido (DMSO), che è in grado di penetrare le membrane lipidiche, le particelle di LDL possono essere caricate artificialmente con filamenti di miRNA pure.
La microscopia a forza atomica ad alta velocità (HS-AFM) è un potente strumento per la caratterizzazione di esemplari biologici che offrono una risoluzione temporale di subnanometro spaziale e subsecondo8. Quindi, è una tecnica ben adatta per il controllo di qualità delle particelle di lipoproteina modificate come particelle lipoproteiche native/ricostituite/etichettate possono essere immagine in un ambiente quasi fisiologico.
Qui, un protocollo basato su qPCR viene presentato passo-passo per determinare il contenuto di miRNA assoluto/relativo di particelle di lipoproteina e campioni di cellule, che consente una stima del tasso di assorbimento delle particelle di lipoproteina cellulare. Inoltre, è dimostrato un metodo per l'arricchimento delle particelle di lipoproteina con miRNA. Questo metodo può essere adattato per la manipolazione generale del contenuto di lipoproteine e, quindi, dimostra l'applicabilità delle particelle di lipoproteina come bersagli per la somministrazione di farmaci.
Protocollo
Le donazioni di sangue sono state approvate dal comitato etico dell'Università di medicina di Vienna (EK-Nr. 511/2007, EK-Nr. 1414/2016). La nomenclatura è conforme alle informazioni minime per la pubblicazione delle linee guida quantitative esperimenti PCR in tempo reale (MIQE)9 .
1. isolamento delle particelle di lipoproteina dal sangue umano
- Preraffreddare un ultracentrifuga a 4 ° c. Prelevare il sangue dai volontari sani dopo il digiuno notturno.
Nota: tipicamente richiesto sono tre donatori, donando 80 mL ciascuno, e tubi di raccolta del sangue contenenti acido etilendiamminetetraacetico (EDTA) come anticoagulante. - Centrifugare a 2.000 x g per 20 min a 4 ° c e al plasma di raccolta (fase superiore); evitare le forze di taglio. Determinare il volume totale V e regolare, se necessario, un multiplo del volume del tubo di centrifugazione utilizzando soluzione salina tampone fosfato (PBS). Misurare la massa di 1 mL 3x e calcolare la densità media ρ.
- Calcolare la quantità necessaria di bromuro di potassio (KBr) per la regolazione della densità con la seguente equazione10 (per la densità desiderata in grammi/millilitro, utilizzare a = 1,019 e per il volume specifico di kbr
 , usare = 0,364 ml/g). Aggiungere KBr al plasma e mescolare delicatamente per evitare forze di taglio fino a quando KBr è completamente sciolto.
, usare = 0,364 ml/g). Aggiungere KBr al plasma e mescolare delicatamente per evitare forze di taglio fino a quando KBr è completamente sciolto.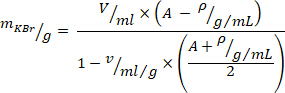
- Misurare la densità come descritto nel passaggio 1,2 e regolare nuovamente, se necessario, aggiungendo più KBr. tubi centrifughe di riempimento e tenuta adatti per ultracentrifugazione con plasma. Evitare eventuali bolle d'aria; in caso contrario, il tubo potrebbe crollare. Collocare i tubi nel rotore secondo le istruzioni del fabbricante e centrifugare a 214.000 x g per 20 ore a 4 ° c.
- Aprire i tubi secondo le istruzioni del produttore e scartare la fase superiore contenente VLDL e IDL. Determinare il volume totale V e regolare, se necessario, a un multiplo del volume del tubo di centrifugazione utilizzando PBS.
- Determinare la densità ρ della frazione inferiore. Calcolare la quantità richiesta di KBr per la regolazione della densità (utilizzare A = 1,063). Mescolare delicatamente per evitare forze di taglio fino a quando KBr non viene sciolto. Ripetere il passaggio 1,4.
- Rimuovere e raccogliere la fase superiore contenente particelle di LDL. Conservare la soluzione di particelle LDL in atmosfera gassosa inerte a 4 ° c. Determinare il volume totale V e regolare, se necessario, a un multiplo del volume del tubo di centrifugazione utilizzando PBS. Determinare la densità ρ della frazione inferiore come descritto nel passaggio 1,2.
- Calcolare la quantità richiesta di KBr per la regolazione della densità (utilizzare A = 1,220) e aggiungerla. Mescolare delicatamente per evitare forze di taglio fino a quando KBr non viene sciolto. Ripetere il passaggio 1,4.
- Rimuovere e raccogliere la fase superiore contenente particelle di HDL. Determinare il volume totale V e regolare, se necessario, a un multiplo del volume del tubo di centrifugazione utilizzando PBS. Determinare la densità ρ. Una seconda fase di centrifugazione della fase superiore a 214.000 x g per 20 ore a 4 ° c è raccomandata per rimuovere l'albina. Se necessario, ripetere il passaggio 1,8. Rimuovere e raccogliere la fase superiore contenente particelle di HDL.
- Preparare e preraffreddare almeno 20 L di tampone di dialisi (0,9% NaCl, 0,1% EDTA [pH 7,4]) a 4 ° c. Tubi di dialisi pre-bagnato (peso molecolare cut-off: 12 – 14 kDa) e aggiungere la soluzione di particelle LDL e HDL secondo le istruzioni del produttore. Dialyze contro 5 L di tampone di dialisi a 4 ° c e cambiare il buffer dopo 1, 2, e 4 h.
- Dopo 24 h, recuperare le soluzioni di particelle di lipoproteina dai tubi di dialisi e determinare la concentrazione proteica utilizzando il saggio di Bradford11 o un altro appropriato. Conservare le soluzioni di particelle HDL e LDL in atmosfera gassosa inerte a 4 ° c.
2. aliquote di miRNA sintetici
Nota: quando si gestiscono oligonucleotidi di RNA, lavorare senza RNasi. Lavorare solo con consumabili in plastica freschi e monouso e indossare sempre guanti, che dovrebbero essere cambiati frequentemente. Utilizzare solo soluzioni prive di nucleasi. Lavorare sempre sul ghiaccio.
- Far girare il flaconcino acquisito dal produttore alla massima forza per formare una pellet di miRNA sintetico liofilizzato. Aggiungere un volume appropriato di tampone Tris (TRIS) di 10 mM, pH 7,5, per una concentrazione finale di 10 μM (concentrazione di stock) di miRNA.
- Pipettare delicatamente su e giù un paio di volte per la risospensione. Preparare le aliquote di 100 μL ciascuno in tubi sterili. Conservarli a-20 ° c se non utilizzati immediatamente. Evitare lo scongelamento ripetuto e il congelamento.
3. ricostituzione delle particelle di HDL
-
Il delipidation
- Preparare il buffer A (150 mM NaCl, 0,01% EDTA, 10 mM Tris/HCl [pH 8,0]). Preraffreddare la centrifuga a-10 ° c. PreCool 100 mL di una miscela di etanolo: etere dietilico (3:2) a-20 ° c.
Attenzione: indossare adeguati dispositivi di protezione individuale e lavorare in una cappa aspirante durante la manipolazione di etere dietilico in quanto è estremamente infiammabile e dannoso per la pelle. - Miscelare un volume corrispondente a 5 mg di particelle HDL con 50 mL di etanolo preraffreddato: miscela di etere dietilico (3:2) e incubare per 2 h a-20 ° c. Centrifugare a 2.500 x g per 10 min a-10 ° c.
- Eliminare il surnatante, risospendere il pellet in 50 mL di etanolo preraffreddato: miscela di etere dietilico mediante vortexing e incubare una seconda volta per 2 h a-20 ° c. Centrifugare nuovamente a 2.500 x g per 10 min a-10 ° c.
Nota: se lo si desidera, lyophilize il supernatante per un'analisi del contenuto di miRNA nella frazione lipidica delle particelle di HDL. - Asciugare il pellet sotto il flusso di gas N2 e risospenderlo in 250 μl di tampone a (vedere il punto 3.1.1). Determinare la concentrazione proteica utilizzando il saggio proteico di Bradford o un'altra appropriata e diluirla in una concentrazione finale di 1 mg di proteina/250 μL di tampone A.
Nota: il protocollo può essere messo in pausa qui. Conservare la soluzione durante la notte a 4 ° c in atmosfera di gas inerte.
- Preparare il buffer A (150 mM NaCl, 0,01% EDTA, 10 mM Tris/HCl [pH 8,0]). Preraffreddare la centrifuga a-10 ° c. PreCool 100 mL di una miscela di etanolo: etere dietilico (3:2) a-20 ° c.
-
Ricostituzione
- Preparare una soluzione di stock di fosfatidilcolina (PC) utilizzando una miscela di cloroformio: metanolo (2:1) ad una concentrazione di 5,6 mg di PC/mL. Analogamente, preparare le soluzioni stock di colesteryl oleato (5 mg di CO/mL) e colesterolo (5 mg di C/mL). Conservare tutte le soluzioni a-20 ° c.
- In un tubo di vetro pulito, mescolare 500 μL di PC, 100 μL di CO e 13,5 μL di C. Questi volumi corrispondono a un rapporto molare approssimativo di 100 PC: 22 CO: 4.8 C. asciugare la miscela sotto il flusso di gas N2 mentre si ruota il tubo per produrre uno strato superficiale omogeneo.
Nota: il protocollo può essere messo in pausa qui. Conservare il flaconcino di vetro (se lo si desidera, lo stoccaggio è possibile) in atmosfera gassosa inerte a-20 ° c. - Preparare una soluzione di spermina 30 mm fresca nel tampone a. Miscelare un'aliquota (100 μl, 10 μm) di Mirna sintetico (vedere punto 2,2) con 100 μl di soluzione di spermina e incubare per 30 minuti a 30 ° c.
Nota: per esperimenti di controllo negativo, sostituire la soluzione di Mirna e/o spermina con lo stesso volume di buffer A. - Sciogliere un'aliquota del mix master PC/CO/C nella miscela dal punto 3.2.3.
- Preparare una soluzione di 30 mg/mL di desossicolato di sodio nel tampone A e aggiungere 50 μL alla soluzione dal punto 3.2.4. Mescolare a 4 ° c per 2 h.
- Aggiungere 250 μL di soluzione HDL delipidatata dal punto 3.1.4. Questo volume corrisponde a un rapporto molare approssimativo di 100 PC: 22 CO: 4.8 C:1 di proteine HDL delipidatate. Mescolare a 4 ° c durante la notte.
-
dialisi f
- Preraffreddare almeno 15 L di PBS a 4 ° c. Aggiungere 50 g di perle adsorbenti a 800 mL di acqua distillata doppia (ddH2O) e mescolare per 1 min. attendere 15 min, decantare il surnatante, e ripetere la procedura con PBS.
- Cassette di dialisi prebagnate (peso molecolare cut-off: 20 kDa) o tubi di dialisi appropriati e aggiungere la soluzione dal punto 3.2.6, utilizzando una siringa secondo le istruzioni del produttore.
- Aggiungere le perle adsorbenti dal passo 3.3.1 al 3 L di PBS e dialyze a 4 ° c. Cambiare il buffer e le perline dopo 1 h e 2 h.
- Dopo 24 h, recuperare la soluzione di particelle HDL ricostituita (rHDL) e determinare la concentrazione proteica utilizzando il saggio di Bradford. Conservare la soluzione di particelle rHDL in atmosfera gassosa inerte a 4 ° c.
4. etichettatura delle particelle LDL
- Preparare 10X tampone LDL (1,5 M NaCl, 3 mM EDTA, 1 mM glicole etilenico-bis (β-amminoetiletere)-N, N, n', acido N'-tetraacetico [EGTA, pH 7,4]) e conservarlo a temperatura ambiente.
- Preparare una soluzione di spermina 30 mm fresca in acqua priva di RNAsi. Miscelare un'aliquota (100 μl, 10 μm) di Mirna sintetico (vedere punto 2,2) con 100 μl di soluzione di spermina e incubare per 30 minuti a 30 ° c.
Nota: per esperimenti di controllo negativo, sostituire il Mirna e/o la soluzione di spermina con lo stesso volume di 1x tampone LDL. - Aggiungere 100 μL di DMSO alla soluzione di miRNA/Spermine dal punto 4,2 e diluirlo ulteriormente con 1,2 mL di 1x tampone LDL.
- Diluire la soluzione di particelle LDL ad una concentrazione finale di circa 4 mg/mL con PBS e miscelare 450 μL con 50 μL di tampone LDL 10x. Incubare per 10 min su ghiaccio.
- Combinare la soluzione di particelle LDL dal passaggio precedente e la soluzione miRNA/Spermine/DMSO (dal passaggio 4,3) e incubare per 2 h a 40 ° c.
- Eseguire la dialisi simile a quanto descritto nella sezione 3,3 e conservare la soluzione di particelle LDL etichettata di conseguenza.
5. controllo di qualità delle particelle lipoproteiche ricostituite/etichettate
Nota: per il controllo di qualità, il diametro e la forma generale delle particelle di lipoproteina possono essere determinati utilizzando, ad esempio, AFM o microscopia elettronica (EM). Qui, HS-AFM viene utilizzato per misurare la distribuzione delle dimensioni delle particelle lipoproteiche native/ricostituite/etichettate.
- Diluire la soluzione di particelle HDL/LDL in PBS (1:100 – 1:1000) e incubarla su mica appena sfalsata per 5 min. Per la tacitura della mica12, premere il nastro adesivo contro il substrato e rimuovere gli strati di mica superiori tirando il nastro.
Nota: a seconda del particolare strumento AFM, dell'area di osservazione e della concentrazione iniziale di particelle, il fattore di diluizione deve essere regolato per osservare le singole particelle. - Dopo l'incubazione, sciacquare il campione con PBS ed eseguire l'imaging HS-AFM in PBS e in modalità di maschiatura con cantilever con costanti di molla di kcant ≪ 0,2 N/m. Si consiglia di utilizzare le dimensioni di scansione < 1 μm2 e di mantenere le forze di imaging più basse possibile.
- Determinare l'altezza delle particelle immagine rispetto alla superficie di mica con un software idoneo.
- Caricare i dati in Gwyddion (freeware), rilevare le particelle attraverso l'analisi del grano (segnare grani per soglia) e impostare la soglia sopra lo sfondo del substrato. Appiattire l'immagine (rimuovere lo sfondo polinomiale) scegliendo l'opzione Escludi regione mascherata .
- Esporta i valori di altezza delle particelle rilevate (distribuzione delle varie caratteristiche del grano) e Ripeti questi passaggi per tutte le immagini registrate. Eseguire analisi statistiche creando istogrammi o calcolando le funzioni di densità di probabilità delle altezze delle particelle ottenute.
- Ripetere i passaggi 5.1 – 5.3 con le particelle lipoproteiche native e ricostituite/etichettate e confrontare i risultati ottenuti per verificare la qualità delle particelle. Se osservate detriti e/o conglomerati, scartate il campione.
6. coltura cellulare
- Coltivare le cellule aderenti secondo un protocollo stabilito (ad esempio, ldlA7-SRBI13) fino a raggiungere la confluenza.
Nota: diverse camere indipendenti a seconda del numero di esperimenti (raccomandati sono due camere per impostazione sperimentale) e esperimenti di controllo negativo (raccomandati sono due camere senza l'aggiunta di particelle di lipoproteina) sono necessari. Inoltre, due camere sono necessarie per la determinazione del numero di cella. - Lavare delicatamente le cellule 3x con la soluzione salina bilanciata di Hank (HBSS) per rimuovere i detriti cellulari e coprire lo strato cellulare con un volume appropriato di mezzo di crescita privo di siero. Aggiungere un volume appropriato di soluzione di particelle di lipoproteina per raggiungere una concentrazione finale di 50 μg/mL di particelle di lipoproteina. Incubare a 37 ° c e 5% CO2 per 16 h.
Nota: a seconda del progetto sperimentale e della linea cellulare, il tempo di incubazione deve essere adattato per ottenere un aumento sufficiente (cioè misurabile) del livello di miRNA cellulare. - Lavare delicatamente le cellule 3x con HBSS preriscaldato (37 ° c) per rimuovere le particelle di cellule/lipoproteine e coprire lo strato cellulare con un volume appropriato di mezzo privo di siero.
- Determinare la densità della cella utilizzando un metodo appropriato (ad esempio, hemocytometer, contatore cellulare automatizzato) in almeno due camere indipendenti per calcolare il numero di celle nel volume del campione dal passaggio 6,3. Questo numero viene utilizzato per la normalizzazione.
7. estrazione di miRNA da campioni di particelle di cellule e lipoproteine
Nota: l'estrazione di miRNA dalle cellule viene eseguita utilizzando il kit di estrazione miRNA con le seguenti modifiche.
-
Campioni di cellule
- Preraffreddare la centrifuga a 4 ° c. Aggiungere 350 μL di reagente di Lisi a due camere contenenti cellule. Come esperimento di controllo negativo, utilizzare una camera senza celle.
Attenzione: indossare adeguati dispositivi di protezione individuale e lavorare in una cappa aspirante durante la manipolazione del reagente di lisi in quanto contiene fenolo e tiocianato. - Attendere 3 – 5 min (a seconda della linea cellulare) per il distacco delle cellule. Se necessario, controllare il distacco delle cellule con la microscopia a campo chiaro. Pool il contenuto delle due camere in un 1,5 mL tubo.
- Usando un ago da 20 G e una siringa da 5 mL, omogeneizzare/disturbare il campione 5x – 10x per aspirazione e incubare per 5 min. aggiungere 140 μL di cloroformio (CHCl3), agitare vigorosamente per 15 s e incubare per 3 min.
- Centrifugare a 12.000 x g per 15 min a 4 ° c. In seguito, smettere di raffreddare la centrifuga.
- Trasferire la fase acquosa superiore ad un nuovo tubo 1,5 mL; evitare la miscelazione di fase/contaminazione in quanto l'interfase contiene DNA e la fase inferiore contiene proteine. Aggiungere 1.5 x il volume di 100% etanolo e mescolare accuratamente con il pipettaggio.
- Posizionare una colonna di spin in un tubo di raccolta da 2 mL e aggiungere 700 μL di miscela dalla fase 7.1.5. Centrifugare a 8.000 x g per 15 s a temperatura ambiente. Eliminare il flusso e ripetere questo passaggio con il volume residuo del campione.
- Eliminare il flusso e aggiungere 700 μL del primo tampone di lavaggio alla colonna di spin. Centrifugare a 8.000 x g per 15 s a temperatura ambiente.
- Eliminare il flusso e aggiungere 500 μL del secondo tampone di lavaggio alla colonna di spin. Centrifugare a 8.000 x g per 15 s a temperatura ambiente. Ripetere l'intero passo una seconda volta con 2 minuti di tempo di centrifugazione.
- Posizionare la colonna di spin in un nuovo tubo di raccolta da 2 mL e centrifugarlo a piena velocità per 1 minuto per asciugare la membrana.
- Posizionare la colonna di rotazione in un tubo di raccolta da 1,5 mL. Aggiungere 30 μL di acqua priva di RNAsi al centro della membrana per eluizione e centrifugare a 8.000 x g per 1 min. Ripetere l'intero passo una seconda volta con il primo flusso attraverso la soluzione di eluizione. La fase di trascrizione inversa viene eseguita immediatamente dopo l'estrazione; in caso contrario, conservare i campioni di miRNA estratti a-20 ° c.
- Preraffreddare la centrifuga a 4 ° c. Aggiungere 350 μL di reagente di Lisi a due camere contenenti cellule. Come esperimento di controllo negativo, utilizzare una camera senza celle.
-
Particelle di lipoproteina
- Impostare il volume del campione con la concentrazione proteica più bassa a 100 μL (= volume massimo del campione). Calcolare i volumi campione degli altri campioni in base a questa normalizzazione, inversamente alla loro concentrazione. Per esperimenti di controllo negativo, utilizzare 100 μL di acqua priva di RNAsi.
Nota: la normalizzazione non è necessaria, ma semplifica il confronto diretto dei risultati durante la fase di qPCR e l'analisi. - Preraffreddare la centrifuga a 4 ° c. Aggiungere 700 μL di reagente di lisi al volume campione del passo 7.2.1.
- Eseguire l'estrazione del miRNA secondo i passaggi 7.1.3 – 7.1.10.
- Impostare il volume del campione con la concentrazione proteica più bassa a 100 μL (= volume massimo del campione). Calcolare i volumi campione degli altri campioni in base a questa normalizzazione, inversamente alla loro concentrazione. Per esperimenti di controllo negativo, utilizzare 100 μL di acqua priva di RNAsi.
8. trascrizione inversa
Nota: la trascrizione inversa di miRNA viene eseguita utilizzando un kit di trascrizione inversa con le seguenti modifiche.
- Scongelare i reagenti del kit e i primer di trascrizione inversa sul ghiaccio. Preparare il seguente Mix Master in una provetta di reazione sul ghiaccio: 45,7 μL di RNase-Free H2O, 16,5 μl di 10X tampone di trascrizione inversa, 11 μl di enzima di trascrizione inversa, 2,1 μl di inibitore della rnasi e 1,7 μl di miscela dNTP. Mescolare delicatamente, non agitare.
Nota: la bilancia dipende dalla quantità del campione; qui, si calcola per 10 reazioni. - Etichettare di conseguenza i provette da 0,2 mL e mescolare 7 μL della miscela master dal passo 8,1 con 5 μL del campione Estratto da STEP 7.1.10 e 3 μL di primer di trascrizione inversa. Mescolare delicatamente, centrifugare a breve, e conservare la miscela sul ghiaccio.
- Per utilizzare lo stesso numero di cella per ogni linea cellulare, ridurre il volume del campione della linea cellulare con un numero di cella complessivo superiore; utilizzare questo come volume residuo per raggiungere il volume totale del campione di 5 μL di acqua priva di RNAsi.
Nota: i campioni di particelle di lipoproteina sono già normalizzati nel passaggio 7.2.1. Per la preparazione della curva standard, diluire un'aliquota di miRNA in sequenza in acqua priva di RNAsi e calcolare il numero di filamenti per volume campione. Obbligatori sono almeno cinque punti di dati all'interno dell'intervallo dei valori del ciclo di quantificazione (cq) risultante. Di solito, i fattori di diluizione totali che vanno da 102 a 106 sono adatti. - Collocare i tubi nella macchina termociclatrice e avviare il seguente programma: 30 min a 16 ° c (passo di ricottura), 30 min a 42 ° c (trascrizione inversa), 5 min a 85 ° c (fase di fusione) e pausa a 4 ° c (stoccaggio). Eseguire il passo qPCR immediatamente dopo la trascrizione inversa; in caso contrario, conservare il DNA complementare (cDNA) sintetizzato dai campioni di miRNA a-20 ° c.
9. qPCR
- Eseguire una qPCR del cDNA (trascritta in modo inverso da miRNA) utilizzando un saggio disponibile in commercio (vedere la tabella dei materiali) con le seguenti modifiche.
- Scongelare tutti i reagenti (SuperMix, RNase-Free H2O), cDNA campioni (dal passo 8,4), e i primer su ghiaccio. Preparare il seguente Mix Master in un tubo di reazione sul ghiaccio: 75 μL di SuperMix, 47,5 μL di H2O senza rnasi, 7,56 μl di primer. Mescolare delicatamente, non agitare.
Nota: la bilancia dipende dalla quantità del campione; qui, si calcola per 10 reazioni. - Etichettare di conseguenza i provette da 0,2 mL e aggiungere 2 μL del campione cDNA a 13 μL del mix master e mescolare delicatamente. In generale, misurare ogni campione 2x.
Nota: in aggiunta sono necessari almeno due campioni di controllo negativi: usare acqua priva di RNAsi come campione. Se la curva di calibrazione non è determinata nella stessa corsa del campione, è richiesto anche un campione della misurazione della curva di calibrazione. Viene utilizzato per calibrare ogni singola corsa alla stessa efficienza di reazione. - Collocare i tubi nella macchina PCR e avviare il seguente programma: 2 min a 50 ° c, 10 min a 95 ° c, 15 s a 95 ° c e 60 s a 60 ° c. Ripetere gli ultimi due passaggi del programma fino a 50x.
- Per un'analisi dei valori cq con il pacchetto software della macchina PCR, attivare la normalizzazione dynamictube (per la compensazione di diversi livelli di sfondo utilizzando il secondo derivato di ogni traccia di esempio) e la pendenza disturbo Correzione (normalizzazione al livello di rumorosità).
Nota: il valore cq del controllo negativo deve essere di diversi cicli superiori al valore cq di campionamento più alto.- Per un'analisi della curva di calibrazione, determinare la soglia per il calcolo del cq per ogni Mirna dalle curve standard di ogni Mirna singolarmente, utilizzando la funzione di soglia di ricerca automatica del pacchetto software e mantenerla costante per ogni miRNA specifico.
- Per l'analisi del campione, se necessario, compensare le diverse efficienze di reazione dell'esecuzione del campione con il punto dati dal campione della curva di calibrazione. Il software calcola i valori cq dei campioni dal livello di soglia della rispettiva misurazione della curva di calibrazione.
10. calcolo del contenuto di miRNA
-
Curva di taratura
- Calcolare, dal numero iniziale di filamenti miRNA per aliquota (100 μL di miRNA 10 μM, peso molecolare dalla scheda tecnica) e le successive fasi di diluizione seriale, il numero di filamenti di miRNA nel volume del campione (il volume campione di 5 μL dal punto 8,3).
Nota: supponendo un'efficienza di trascrizione inversa di 1, questo numero è uguale al numero di fili cDNA. - Calcolare il numero di filamenti cDNA nel volume campione di 2 μL dal passaggio 9,3. Si consideri quindi il fattore di diluizione aggiuntivo di 7,5 (il volume campione di 2 μL dal passo 9,4 dal volume campione di 15 μL dal punto 8,4).
- Tracciare il valore cq dal passo 9.5.1 contro il numero di fili nfili calcolati nel passaggio 10.1.2 in un grafico semilogarithmico base-10 e adattare i punti dati con la seguente curva di regressione (M = pendenza della linea lineare curva di regressione, B = offset).
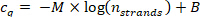
Verificare che il coefficiente di correlazione (R2) per la linea sia > 0.99.
- Calcolare, dal numero iniziale di filamenti miRNA per aliquota (100 μL di miRNA 10 μM, peso molecolare dalla scheda tecnica) e le successive fasi di diluizione seriale, il numero di filamenti di miRNA nel volume del campione (il volume campione di 5 μL dal punto 8,3).
-
Particelle di lipoproteina
- Calcolare il numero di filamenti miRNA per volume campione utilizzando il valore di campionamento cq misurato (Step 9.5.2) e la seguente equazione (M e B sono i parametri della curva di calibrazione del miRNA specifico).
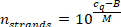
- Calcolare il numero corrispondente di particelle di lipoproteina nel volume del campione, partendo dal volume al punto 7.2.1 (100 μL), dalla concentrazione nota e dalle successive fasi di diluizione (100 μL-> 30 μL di volume campione nel passo 7.1.10-> 5 μL [diluizione 1:6] campione nel volume totale di 15 μL al passo 8,4-> 2 μL [diluizione 1:7,5] al punto 9,4). Assumere un peso molecolare di 250 kDa per le particelle HDL e 3 MDa per le particelle LDL e un 100% di recupero di miRNA durante la fase di estrazione di miRNA (ignorando qualsiasi contributo lipidico al peso molecolare produce una lieve sovrastima del numero di filamenti di miRNA per particelle di lipoproteina).
- Dividere il numero di filamenti di miRNA dal passo 10.2.1 per il numero di particelle calcolate nel passaggio precedente per produrre il numero di filamenti miRNA per particella di lipoproteina.
- Calcolare il numero di filamenti miRNA per volume campione utilizzando il valore di campionamento cq misurato (Step 9.5.2) e la seguente equazione (M e B sono i parametri della curva di calibrazione del miRNA specifico).
-
Cellule
- Calcolare il numero di filamenti miRNA per volume campione secondo il passo 10.2.1.
- Calcolare il numero corrispondente di celle nel volume del campione in base al passo 10.2.2, iniziando con la concentrazione del numero di cella iniziale misurata al passaggio 6,4.
- Dividere il numero di filamenti di miRNA da 10.3.1 per il numero di celle calcolate nel passaggio precedente per produrre il numero di filamenti miRNA per cella.
- Calcolare il tasso di assorbimento delle particelle di lipoproteina dividendo la quantità totale di filamenti di miRNA dal passaggio precedente dopo la correzione per lo sfondo livello di miRNA delle cellule ottenute da un esperimento di controllo negativo dal rapporto miRNA/particella (passo 10.2.3 ) e il tempo di incubazione (16 h, vedere il passo 6,2).
11. Array microfluidici multiwell
-
estrazione di miRNA
- Eseguire l'estrazione di miRNA come descritto al punto 7.
-
Trascrizione inversa
- Scongelare i primer di trascrizione inversa, i componenti del kit di trascrizione inversa e MgCl2 (25 mm) sul ghiaccio. Per otto campioni, mescolare 8 μL di primer di trascrizione inversa (10x), 2,25 μL di dNTPs con dTTP (100 mM), 16,88 μL di trascrittasi inversa (50 U/μL), 9,00 μL di 10X tampone di trascrizione inversa, 10,12 μL di MgCl2, 1,12 μl di inibitore della RNAsi (20 U/μl) e 1 μL di acqua priva di nucleasi.
- Mescolare delicatamente e centrifugare brevemente. Aggiungere 4,3 μL di miscela di reazione di trascrizione inversa a 3,5 μL di miRNA Estratto in un tubo e mescolare, girare verso il basso e incubare su ghiaccio per 5 minuti. Collocare i tubi in una macchina termociclatrice e avviare il seguente programma: 16 ° c per 2 min, 42 ° c per 1 min , e 50 ° c per 1 s ripetuto 40x in totale, e poi, come reazione di arresto, 85 ° c per 5 min e tenere a 4 ° c fino a quando si è fermato.
-
Preamplificazione
- Scongelare i primer sul ghiaccio e invertire e centrifugare brevemente. Mescolare il Master mix di preamplificazione (2x) agitando la bottiglia. Preparare la miscela di reazione di preamplificazione secondo le seguenti istruzioni per otto campioni: 112,5 μL di miscela Master preamplificazione (2x), 22,5 μL di primer di preamplificazione e 67,5 μL di acqua priva di nucleasi. Invertire e centrifugare brevemente.
- Mescolare 2,5 μL di prodotto di reazione di trascrizione inversa dal passo 11.2.2 con 22,5 μL di miscela di reazione di preamplificazione dal passaggio precedente e invertire e centrifugare brevemente. Incubare i campioni su ghiaccio per 5 min.
- Collocare i tubi in una macchina termociclatrice e incubare alle seguenti impostazioni: attivazione enzimatica a 95 ° c per 10 min, ricottura a 55 ° c per 2 min, estensione a 72 ° c per 2 min, ripetuta 12x: denaturazione a 95 ° c per 15 s e ricottura/estensione a 60 ° c per 4 min. , inattivazione enzimatica a 99,9 ° c per 10 min e 4 ° c in attesa.
- Girare verso il basso, aggiungere 7,5 μL di 1x TE (pH 8,0) e 67,5 μL di acqua priva di nucleasi, invertire e centrifugare brevemente. I campioni possono essere conservati a-20 ° c per un massimo di 1 settimana.
-
Qpcr
- Mescolare il mix master agitando la bottiglia. Preparare la miscela di reazione PCR per una scheda: 450 μL di miscela Master, 441 μL di acqua priva di nucleasi e 9 μL del campione di preamplificazione dal passo 11.3.4. Invertire e centrifugare brevemente.
- Caricare ogni serbatoio di riempimento della scheda di Array microfluidico multipozzetto con 100 μL di miscela di reazione PCR preparata secondo le istruzioni del fabbricante e centrifugare 2x per 1 min a 3.000 x g. L'accelerazione durante le due fasi di centrifugazione consecutive è importante per riempire correttamente la scheda. Sigillare la scheda secondo le istruzioni del fabbricante.
- Utilizzare un sistema PCR con il seguente programma: attivazione enzimatica a 95 ° c per 10 min e poi, ripetuta 40x: denaturazione a 95 ° c per 15 s e ricottura/estensione a 60 ° c per 1 min.
- Importare il file dei risultati dal sistema PCR e calcolare i valori RQ utilizzando il pacchetto software del sistema con le seguenti impostazioni di analisi: un valore massimo di cq consentito di 40,0, valori massimi di cq nei calcoli inclusi e outlier tra i replicati esclusi. Attivare il tasso di falsa scoperta Benjamini-Hochberg come opzione per la regolazione del valore p(correzione del verificarsi di falsi positivi14) e normalizzazione globale come metodo di normalizzazione (utilizzando un valore di soglia mediana per tutti i campioni 15).
Risultati
Uno schema generale di isolamento delle particelle di lipoproteina
La Figura 1 Mostra lo schema generale dell'isolamento delle particelle di lipoproteina a partire dal sangue intero, utilizzando la flottazione sequenziale ultracentrifugazione16. Se lo si desidera, altre frazioni di particelle di lipoproteina come le particelle VLDL e IDL possono essere raccolte durante questo protocollo. Il rotore in titanio ad angolo fisso in combinazione con tubi di tenuta rapida in polipropilene è idoneo a sopportare le forze di centrifugazione. Per evitare il collasso del tubo, è importante evitare le bolle d'aria nel tubo. La centrifugazione viene effettuata a 4 ° c per minimizzare la degradazione delle proteine. Di solito a partire dal plasma (60-80 mL per donatore) di donazioni di sangue in pool di tre volontari, si può aspettare una resa di volumi di soluzione di particelle LDL e HDL di 3 mL ciascuno con concentrazioni nell'intervallo di 1-3 mg/mL. L'intera procedura, a partire dalla donazione di sangue, ha impiegato circa 7 giorni.
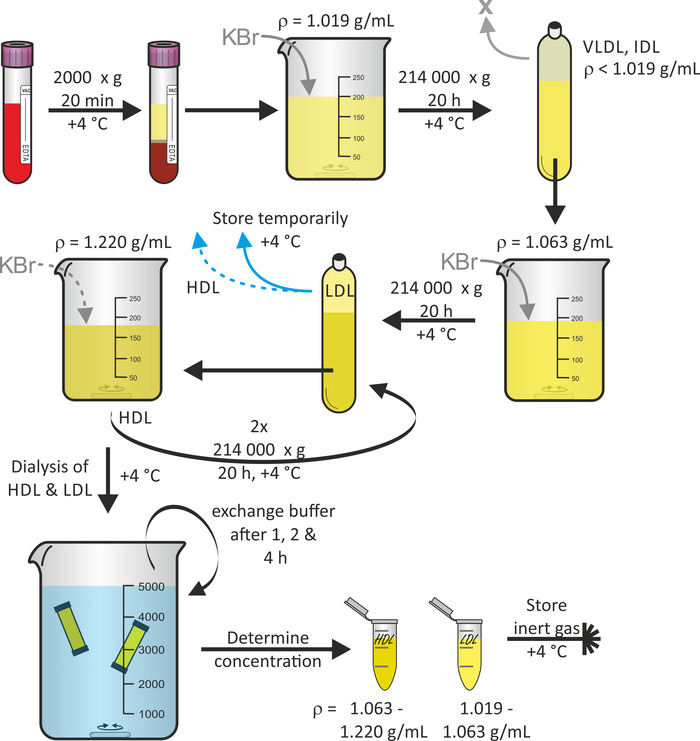
Figura 1: Diagramma di flusso dell'isolamento delle lipoproteine. Centrifugare il sangue da volontari sani in tubi per contenitori sottovuoto e raccogliere il plasma (fase superiore). Dopo la regolazione della sua densità a ρ = 1,019 g/ml con KBR, centrifugare la soluzione a 214.000 x g per 20 ore a 4 ° c. Dopo la regolazione della densità della frazione inferiore a ρ = 1,063 g/ml con KBR, centrifugare nuovamente la soluzione a 214.000 x g per 20 h a 4 ° c. Conservare la frazione superiore contenente particelle LDL temporanee a 4 ° c. Dopo la regolazione della densità della frazione inferiore a ρ = 1,220 g/ml con KBR, centrifugare la soluzione due volte a 214.000 x g per 20 h a 4 ° c. Raccogliere la frazione superiore contenente particelle di HDL, dialyze sia le soluzioni di HDL e LDL particelle e scambiare il buffer dopo 1, 2, e 4 h. Dopo 24 h, determinare la concentrazione proteica e conservare i campioni sotto gas inerte a 4 ° c. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Ricostituzione di particelle HDL
La ricostituzione delle particelle HDL è stata eseguita secondo un protocollo precedentemente pubblicato da Jonas7. Il primo passo è stato la delipidazione delle particelle HDL come mostrato nella Figura 2a, seguita dalla seconda fase di relipidazione (cioè, ricostituzione) come mostrato nella Figura 2B, utilizzando PC lipidico, Co, e C in aggiunta a una miscela di Mirna e Spermine. Abbiamo scelto l'uomo maturo miR-223 e miR-155 perché miR-223 Mostra una grande abbondanza e miR-155 è raro in particelle di lipoproteina17. In genere, entrambi i passaggi vengono eseguiti in due giorni sequenziali. Durante la ricostituzione, possono essere aggiunti altri componenti lipofilici e/o anfifilici come desiderato. La completa evaporazione di etanolo/etere etilico e metanolo/cloroformio solvente di PC, CO e C è stata critica. L'ultimo passo — come mostrato nella Figura 2C— è stata la procedura di dialisi per separare le particelle HDL ricostituite (rhdl) da lipidi/Mirna/detersivi liberi. Questo ha richiesto ulteriori 1-2 giorni. L'aggiunta di perle assorbenti alla soluzione di dialisi mantenuto il gradiente di densità lungo la membrana di dialisi costante. Si può aspettare una resa di 50% di particelle di rHDL.
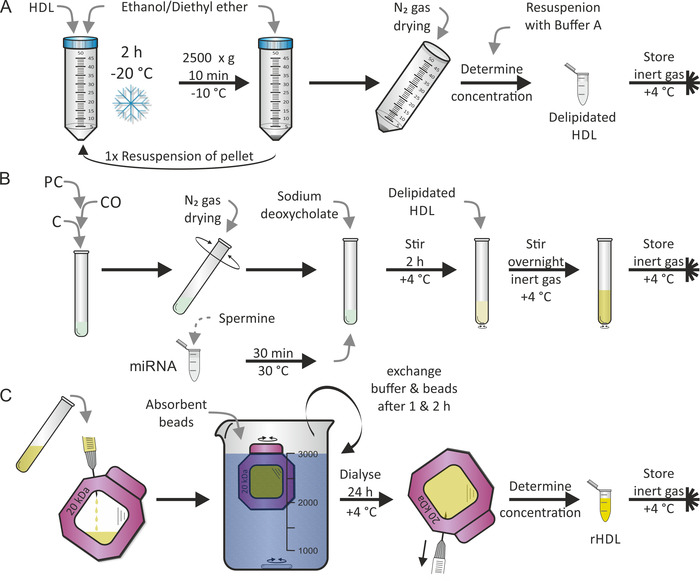
Figura 2: Diagramma di flusso della ricostituzione delle particelle HDL. (A) delipidazione: mescolare la soluzione di particelle HDL con etanolo preraffreddato/etere dietilico e incubare a-20 ° c per 2 h. Dopo aver scartati il surnatante, risospendere il pellet e ripetere la procedura. Asciugare il pellet con N2 gas e risospenderlo nel buffer a. Dopo la determinazione della concentrazione, conservare l'HDL delipidatato in atmosfera gassosa inerte a 4 ° c. B) ricostituzione: dopo la miscelazione di fosfatidil-colina (PC), colesteryl oleato (CO), e colesterolo (C), evaporare il solvente utilizzando N2 gas durante la rotazione del tubo. Incubare l'aliquota di Mirna con la soluzione di spermina per 30 minuti a 30 ° c, aggiungere il desossicolato di sodio e risospendere il film lipidico essiccato. Mescolare il campione per 2 ore a 4 ° c, aggiungere la soluzione di HDL delipidatata e mescolare nuovamente il campione, questa volta durante la notte a 4 ° c in atmosfera di gas inerte. (C) dialisi: trasferire la soluzione dal pannello B contenente particelle HDL ricostituite (rhdl) a una camera a membrana di dialisi (peso molecolare cut-off: 20 kDa) e DIALYZE contro PBS e perle assorbenti a 4 ° c. Scambiare il tampone e le perle dopo 1 h e 2 h. recuperare la soluzione di particelle rHDL dopo 24 h, determinare la concentrazione, e conservare il campione in atmosfera di gas inerte a 4 ° c. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Etichettatura delle particelle LDL
L'etichettatura delle particelle LDL con miRNA (Figura 3) come dimostrato per le particelle di HDL non era fattibile a causa dell'idrofobicità della proteina apoB-100, che è il principale costituente della particella LDL. DMSO è stato utilizzato per la penetrazione del monostrato lipidico della particella LDL e, quindi, mediato l'associazione di miRNA. L'intera procedura ha richiesto 1-2 giorni con una resa vicina al 100%.
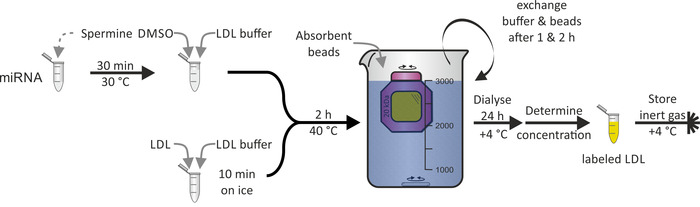
Figura 3: Diagramma di flusso dell'etichettatura delle particelle LDL. Incubare l'aliquota di Mirna con la soluzione di spermina per 30 minuti a 30 ° c e aggiungere il tampone DMSO e LDL. Incubare LDL campione Wit tampone LDL per 10 min su ghiaccio e aggiungere miRNA/Spermine/DMSO miscela. Dopo l'incubazione a 40 ° c per 2 h, trasferire la soluzione in una camera di membrana di dialisi (peso molecolare cut-off: 20 kDa) e dialyze contro PBS e perline assorbenti a + 4 ° c. Tampone di scambio e perline dopo 1 & 2 h. recuperare la soluzione di particelle LDL etichettati dopo 24 h, determinare la concentrazione e conservare in atmosfera di gas inerte a + 4 ° c. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Controllo di qualità delle particelle di lipoproteina
HS-AFM può essere utilizzato per esaminare le dimensioni e la forma delle particelle lipoproteiche native e ricostituite/etichettate su mica. Poco prima dell'uso, la mica deve essere appena sfalsata (utilizzare nastro adesivo per rimuovere lo strato superiore [s]) al fine di fornire una superficie pulita e piatta. Quando si incubano particelle HDL/LDL su mica, il fattore di diluizione (e/o il tempo di incubazione) deve essere regolato per osservare le singole particelle. I cluster non consentono una determinazione delle dimensioni delle particelle. Le particelle HDL sono mobili su mica. Quando si utilizza AFM convenzionale invece di HS-AFM, il protocollo di immobilizzazione deve essere adattato di conseguenza (tampone, rivestimento superficiale) per ridurre la mobilità delle particelle laterali. Durante la scansione del campione, la forza di imaging deve essere mantenuta bassa (modalità di maschiatura) per evitare qualsiasi deformazione delle particelle, che influenzerà di conseguenza i valori misurati. Per l'analisi dei dati, le particelle sono state rilevate tramite un algoritmo di soglia (ad esempio, in Gwyddion: grani > Mark by Threshold) e la loro altezza è stata determinata rispetto alla superficie di mica. Misurare l'altezza delle particelle è il modo più preciso per determinare le dimensioni delle particelle, poiché le dimensioni laterali apparenti sono ampliate dalla forma della punta (vedere immagini esemplari in Figura 4). Le funzioni di densità di probabilità (PDF) delle altezze delle particelle sono state calcolate per la valutazione statistica e il confronto delle distribuzioni delle dimensioni delle varie particelle di lipoproteina. Un confronto tra le particelle di LDL con etichetta nativa e miRNA, come mostrato nella Figura 4 , consente di verificare la somiglianza principale tra le particelle di lipoproteina etichettate e non etichettate (cioè native) (contrassegnate con particelle LDL senza l'aggiunta di Mirna/ le miscele di spermina sono mostrate come un controllo per la procedura di etichettatura stessa). L'intera procedura ha richiesto circa 1 giorno.
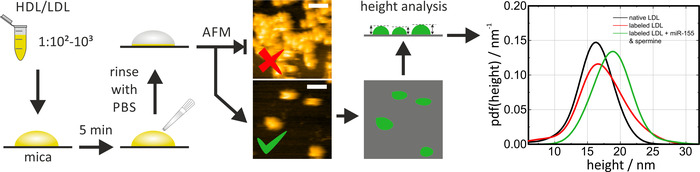
Figura 4: diagramma di flusso e risultati rappresentativi delle misurazioni HS-AFM. Diluire il campione di particelle HDL/LDL in PBS (1:102-1:103) e incubarlo sulla mica appena sfalsata per 5 minuti, seguita da un accurato risciacquo con PBS per rimuovere le particelle libere (non elettrostaticamente adsorbite). Eseguire l'imaging HS-AFM e controllare la densità delle particelle sulla superficie. Effettuare le misurazioni in PBS a temperatura ambiente. L'immagine superiore di questa figura mostra una densità di particelle troppo elevata; l'immagine inferiore è adatta per l'analisi. L'altezza delle singole particelle è stata analizzata dopo la soglia e le particelle native (curva nera) e ricostituite/etichettate (curva rossa e verde) sono state confrontate in una valutazione statistica. La barra della scala = 100 Nm. Questa cifra è stata modificata da Axmann et al.19. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
estrazione di miRNA, trascrizione inversa e qPCR
L'estrazione di miRNA da lipoproteine o campioni di cellule di origine nativa/arricchita artificialmente è stata eseguita utilizzando un kit di estrazione di miRNA come mostrato nella Figura 5a. Con la presente, un ambiente privo di RNAsi era critico. Questo passo ha richiesto circa 1 h. la trascrizione inversa del campione di miRNA Estratto (Figura 5b) è stata eseguita utilizzando procedure biochimiche standard come descritto dal produttore. Questo passaggio ha richiesto circa 1,5 h. Infine, la quantità di cDNA ottenuta durante l'ultimo passaggio è stata determinata utilizzando qPCR (Figura 5C). Una curva standard, che riporta i valori di cq ottenuti al numero assoluto del filamento Mirna, ha prodotto il contenuto assoluto di Mirna del campione iniziale. Questo ha richiesto circa 2,5 h.
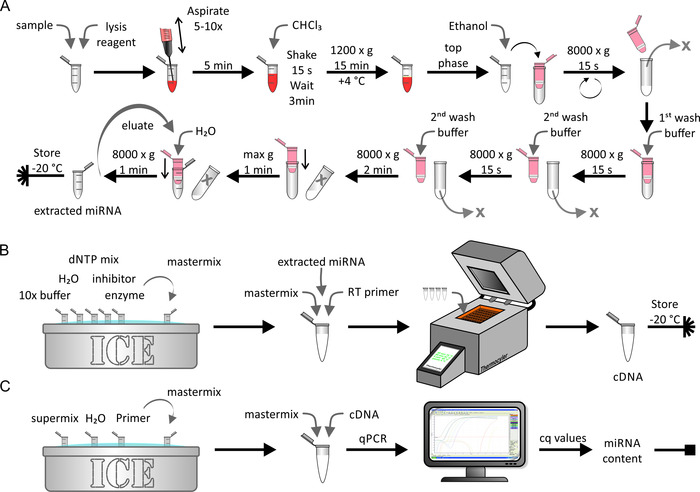
Figura 5: diagramma di flusso dell'estrazione di Mirna, trascrizione inversa e qPCR. A) estrazione di Mirna: miscelare il campione con il reagente di lisi e lizzare l'aspiratore mediante una siringa. Incubare per 5 min e aggiungere CHCl3. Agitare vigorosamente per 15 s e incubare per 3 min. Dopo la centrifugazione a 1.200 x g per 15 min a 4 ° c, raccogliere la fase superiore e mescolare con etanolo. Trasferire la soluzione a una colonna di spin (volume massimo < 700 μL) e centrifugare a 8.000 x g per 15 s. scartare l'eluente e ripetere l'ultimo passaggio con il resto della soluzione. Aggiungere il primo tampone di lavaggio e centrifugare a 8.000 x g per 15 s. gettare l'eluente, aggiungere il secondo tampone di lavaggio, e centrifugare a 8.000 x g per 15 s. Ripetere l'ultimo passo con un tempo di centrifugazione di 2 min. Asciugare ulteriormente la membrana tramite centrifugazione alla massima velocità per 1 min. elute il miRNA con H2o e centrifugare a 80.000 x g per 1 min. conservare il campione di Mirna Estratto a-20 ° c. (B) trascrizione inversa: scongelare il buffer 10x, H2O, dNTP mix, inibitore, e l'enzima sul ghiaccio e preparare il mix master. Aggiungere il miRNA Estratto dal pannello a al Master mix e il primer di trascrizione inversa ed eseguire la trascrizione inversa utilizzando una macchina termociclatrice. Conservare il campione cDNA a-20 ° c. (C) qPCR: scongelare il SuperMix, H2O, e primer su ghiaccio e preparare il mix master. Aggiungere il cDNA dal pannello B al Master mix ed eseguire qPCR. Analizzare i dati per ottenere i valori cq e calcolare il contenuto assoluto di Mirna del campione (vedere la Figura 6 e i risultati rappresentativi per i dettagli). Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Contenuto assoluto di miRNA e velocità di trasferimento
Il contenuto assoluto di miRNA di particelle di HDL e LDL arricchite con l'origine artificiale e arricchita artificialmente è stato calcolato dai valori cq dei campioni e da una curva standard del rispettivo Mirna, come mostrato nella Figura 6. La Figura 6a Mostra i dati calcolati dal software di analisi (con la normalizzazione dynamictube attivata [per la compensazione di diversi livelli di sfondo utilizzando il secondo derivato di ogni traccia di campionamento] e la correzione della pendenza del disturbo [normalizzazione al livello di rumore]). cq i valori delle curve standard sono stati determinati utilizzando la funzione di soglia di ricerca automatica del pacchetto software sul segnale di fluorescenza normalizzato misurato dalla macchina qPCR. Con la presente, il software ha massimizzato il valore R dell'adattamento della curva standard. Il livello di soglia è stato mantenuto costante per ogni specifica analisi del campione miRNA. Successivamente, i valori cq sono stati tracciati in funzione del numero di filamenti Mirna e è stata calcolata una linea di regressione. I valori cq di esempio sono stati determinati con lo stesso livello di soglia, come mostrato nella Figura 6b; le differenze di efficienza di reazione tra le diverse esecuzioni di qPCR sono state compensate automaticamente dal software utilizzando un campione di curva di calibrazione aggiuntivo incluso in ogni corsa. Utilizzando l'equazione della linea di regressione, è possibile calcolare la quantità sconosciuta di miRNA nell'esempio. Il numero di particelle di lipoproteina è stato stimato dalla concentrazione proteica iniziale e il suo peso molecolare medio (MWHDL ~ 250 kDa). Pertanto, non è stato assunto alcun contributo lipidico al peso molecolare, quindi il numero di filamenti di miRNA per particella di lipoproteina è stato leggermente sopravvalutato. Inoltre, è stato assunto un tasso di recupero del 100% di miRNA durante la fase di estrazione del miRNA. Inoltre, il contenuto di miRNA delle cellule prima e dopo l'incubazione con particelle di HDL è stato determinato e la velocità di trasferimento di miRNA è stata calcolata come mostrato nella Figura 6C.
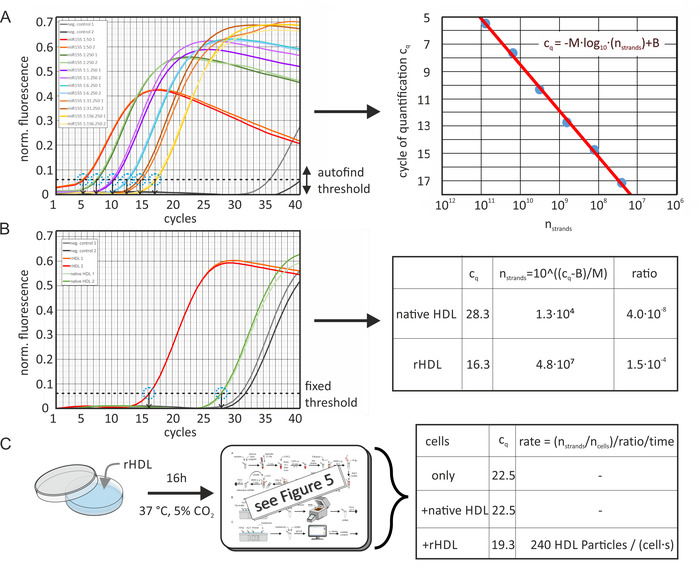
Figura 6: diagramma di flusso del calcolo del contenuto assoluto di Mirna e della velocità di trasferimento. A) curva standard per mir-155: un'aliquota mir-155 (100 μl, 10 μm) è stata diluita serialmente con acqua priva di RNA, come indicato. qPCR ha prodotto valori cq per ogni campione di diluizione seriale (misurato due volte) utilizzando la funzione di soglia di ricerca automatica del pacchetto software. Esperimenti di controllo negativo (senza l'aggiunta di miRNA) hanno prodotto valori cq di > 35. I punti dati dei valori cq come funzione del numero di filamenti miRNA per volume campione (calcolati dalla concentrazione iniziale e dalle diluizioni seriali) sono stati equipaggiati con l'equazione presentata (linea rossa, immagine a destra), ottenendo M =-3,36 e B = 42,12. L'efficienza della PCR determinata era 0,98. Le barre di errore sono state calcolate dai risultati delle ripetizioni sperimentali ed erano più piccole del diametro del cerchio dei punti dati. B) i valori cq delle particelle di HDL di origine nativa/arricchita artificialmente sono stati determinati con lo stesso livello di soglia determinato nel gruppo a e convertito nel numero di filamenti miRNA nel volume di campionamento qPCR. Il rapporto assoluto di miRNA del campione originale è stato calcolato dal numero (concentrazione) di particelle di HDL nel volume del campione (3,2 x 1011 particelle). C) i campioni cellulari (linea cellulare LDLA7-Srbi) sono stati incubati per 16 ore con particelle di HDL arricchite artificialmente (50 μg/ml) e analizzate in modo analogo. I valori cq determinati sono stati 22,5, 22,5 e 19,3 solo per le celle, per le cellule incubate con HDL nativo, o per le cellule incubate con la soluzione di particelle rhdl (entrambi 50 μg/ml), rispettivamente. Questi valori sono stati convertiti nel numero di filamenti miRNA come fatto nel pannello B. Il numero di filamenti di miRNA dopo l'incubazione (7,3 x 106) è stato corretto per sottrazione del numero di filamenti di Mirna prima dell'incubazione (8,6 x 105). Il risultato è stato diviso per il numero di cellule nel volume del campione (3.100), il miRNA-Particle-ratio (1,5 x 10-4), e il periodo di incubazione (16 h). Questo ha prodotto il tasso di trasferimento delle particelle di lipoproteina tramite l'assorbimento di miRNA (240 eventi di assorbimento delle particelle HDL per cella e secondo). Questa cifra è stata modificata da Axmann et al.19. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Array microfluidico multiwell
A causa di piccole rese di estrazione di miRNA, la trascrizione inversa del miRNA estratto è stata seguita da una fase di preamplificazione. Infine, qPCR, come mostrato in Figura 6, è stato eseguito. Per tutte le fasi, sono state utilizzate procedure biochimiche standard come descritto dal produttore. Qui viene mostrata una parte del profilo globale di miRNA sulle particelle di HDL dei pazienti uremici assunti per uno studio sull'influenza del CRF sull'efflusso di colesterolo dai macrofagi18 . In questo studio, la capacità di accettore del colesterolo di HDL o siero in — oltre ad altri — 17 giovani pazienti adulti uremici (CKD stadi 3-5) e 14 pazienti in emodialisi giovani adulti senza malattie associate e controlli corrispondenti è stato misurato. Per analizzare i dati, sono state utilizzate le impostazioni predefinite (valore CT massimo consentito: 40,0, inclusi i valori CT massimi nei calcoli e esclusi gli outlier tra i replicati). I valori Psono stati regolati utilizzando il tasso di falsa scoperta di Benjamini-Hochberg (correzione del verificarsi di falsi positivi), e come metodo di normalizzazione, è stata selezionata la normalizzazione globale , che trova i saggi comuni tra tutti campioni per usare la sua mediana CT per la normalizzazione. Nei risultati rappresentativi, sono raffigurati alcuni RQ di Mirna isolati da HDL di pazienti uremici (gli RQs dei controlli sono 1). Ovviamente, miR-122 e miR-224 sono molto espressi negli HDL di pazienti uremici. Tutto questo passo ha richiesto circa 1 giorno.
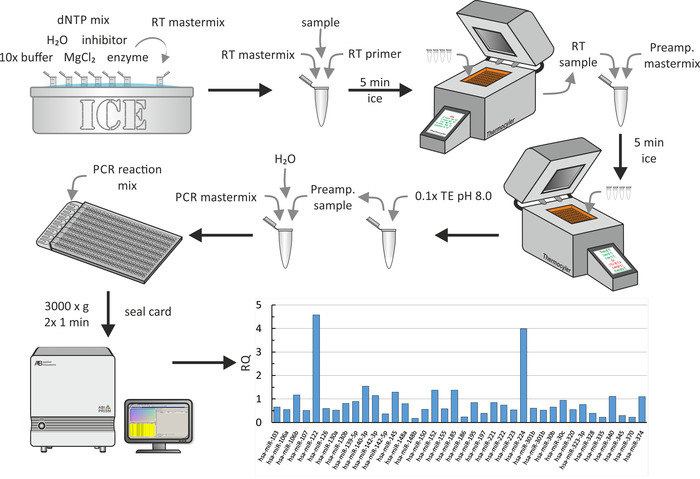
Figura 7: diagramma di flusso e risultati rappresentativi dell'Array microfluidico multiwell Program. Dopo l'estrazione del miRNA, come mostrato nella Figura 5a, mescolare il campione di Mirna con primer di trascrizione inversa e un mix master contenente 10x buffer, H2O, dNTP mix, inibitore, MgCl2, e enzima. Dopo l'incubazione su ghiaccio per 5 min, eseguire la trascrizione inversa utilizzando una macchina termociclatrice. Aggiungere il Master mix di preamplificazione, incubare per 5 min sul ghiaccio ed eseguire la preamplificazione utilizzando una macchina termociclatrice. Aggiungere 0,1 x TE (pH 8,0) e miscelare un'aliquota con miscela Master PCR e H2O. Pipettare la miscela di reazione PCR nella porta di riempimento dell'Array microfluidico multipozzetto e girare due volte a 3.000 x g per 1 min ciascuno. Eseguire qPCR utilizzando un sistema PCR e analizzare i dati per produrre valori RQ (qui, la figura mostra i valori RQ di particelle HDL di uremia pazienti in confronto ad un gruppo di controllo sano18). Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Discussione
Qui, l'isolamento delle frazioni di particelle di lipoproteina dal sangue umano e la determinazione del loro contenuto di miRNA individuale è descritta passo-passo. È fondamentale lavorare in un ambiente privo di RNAsi durante la manipolazione di miRNA isolati e sintetizzati — il miRNA incorporato in particelle è ovviamente riparato dalla degradazione enzimatica. Poiché il rapporto miRNA/particelle delle particelle di lipoproteina nativa è piuttosto basso, è necessario un arricchimento artificiale con miRNA per studiare l'assorbimento delle particelle di Holo nelle cellule. In tal modo, la ricostituzione delle particelle HDL come descritto in precedenza7 viene modificata per incorporare i filamenti di Mirna. Inoltre, la separazione della frazione lipidica e proteica durante questa procedura consente agli scienziati di esaminare i componenti associati ai lipidi e alle proteine della particella di lipoproteina19. Analogamente, la procedura di etichettatura delle particelle LDL è adattata. È interessante notare che l'aggiunta di Spermine — uno stabilizzatore naturale di nucleotidi — non ha influenzato il rapporto miRNA/particella. Va notato che, in linea di principio, il metodo consente l'inpiegatura di altre sostanze di miRNA all'interno di una particella di lipoproteina. Naturalmente, c'è un limite per quanto riguarda le dimensioni fisiche della sostanza in base alla dimensione complessiva di HDL (diametro: 5-12 Nm) e particelle LDL (diametro: 18-25 Nm).
Per quanto riguarda il controllo di qualità delle particelle lipoproteiche ricostituite/etichettate, HS-AFM è un metodo applicabile per caratterizzare le particelle HDL/LDL a livello di singola particella. Rispetto all'EM, consente tempi di preparazione più brevi e condizioni quasi fisiologiche (bagnato, temperatura ambiente).
Grazie alla sua intrinseca sensibilità e amplificazione, qPCR è il metodo di scelta per rilevare basse concentrazioni di miRNA. In alternativa, la microscopia a fluorescenza sensibile a singola molecola, che è in grado di rilevare anche singole molecole, non sarebbe adatta a causa delle basse concentrazioni, ad esempio, di filamenti di miRNA con etichetta fluorescente per particella. Così, il rapporto dei filamenti di miRNA per particella di lipoproteina nativa è stato trovato per essere 10-8. L'arricchimento artificiale aumenta il rapporto di un fattore di 10.000, che facilita la stima del tasso di assorbimento della lipoproteina cellulare (non viene rilevata alcuna differenza significativa utilizzando le particelle di lipoproteina nativa 19). L'elevata sensibilità della qPCR consente di determinare questo tasso di assorbimento misurando il numero di filamenti di miRNA dopo il tempo di incubazione e il rapporto miRNA/particella. Va notato che il valore calcolato ignora la degradazione cellulare e il rilascio di miRNA e, pertanto, rappresenta almeno un limite inferiore per il rapporto di assorbimento delle particelle di lipoproteina.
In futuro, il metodo può essere adattato per trasferire le sostanze farmaceutiche (in particolare anche quelle lipofiliche) nelle cellule e correlare il loro effetto biologico alla concentrazione intracellulare (determinata attraverso il tasso di assorbimento delle particelle di lipoproteina).
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Quest'opera è stata sostenuta dal progetto del Fondo scientifico austriaco P29110-B21, dal progetto "Hochschuljubiläumsstiftung der Stadt Wien zur Förderung der Wissenschaft" H-3065/2011, dal Fondo europeo per lo sviluppo regionale (EFRE, IWB2020), dallo stato federale di alta Austria, e la "terra OÖ Basisfinanzierung".
Materiali
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge | Beckman | Optima L-100 XP | Lipoprotein isolation |
| Ultracentrifuge rotor | Beckman | TI 55.2 | Lipoprotein isolation |
| Vacutainer (EDTA) | Becton Dickinson | 366643 | Lipoprotein isolation |
| Kaliumbromid | Sigma Aldrich | 2110 | Lipoprotein isolation |
| Ultracentrifuge tubes | Beckman | 342414 | Lipoprotein isolation |
| Ultracentrifuge tube sealer | Beckman | 342428 | Lipoprotein isolation |
| Sodiumchlorid | Carl Roth GmbH | P029.2 | Lipoprotein isolation |
| EDTA | Fisher Scientific | D/0650/50 | Lipoprotein isolation |
| Dialysis tubes, MWCO 12 - 14k | Spectra | 132700 | Lipoprotein isolation |
| synthetic miRNA | microSynth | - | synthetic miRNA |
| TRIS buffer pH 7 | Ambion | AM9850G | synthetic miRNA |
| TRIS buffer pH 8 | Ambion | AM9855G | synthetic miRNA |
| sterile tubes | Carl Roth GmbH | ENE8.1 | synthetic miRNA |
| Photometer | Eppendorf | Eppendorf Biophotometer | Bradford assay |
| Cuvette | Carl Roth GmbH | XK20 | Bradford assay |
| Coomassie G-250 Stain | BioRad GmbH | 161-0786 | Bradford assay |
| Sodiumchlorid | Carl Roth GmbH | 3957.1 | Delipitation |
| EDTA | Fluka Analytical | 03690-100ML | Delipitation |
| TRIS buffer pH 8.0 | Ambion | AM9855G | Delipitation |
| Ethanol 100% | AustrAlco | Ethanol Absolut 99.9% | Delipitation |
| Diethyl ether | Carl Roth GmbH | 3942.1 | Delipitation |
| Centrifuge | Thermo Scientific | Multifuge X3R | Delipitation |
| Chloroform | Carl Roth GmbH | 3313.1 | Reconstitution |
| Methanol | Carl Roth GmbH | 4627.1 | Reconstitution |
| Phosphatidylcholine | Sigma Aldrich GmbH | P3556-25mg | Reconstitution |
| Cholesterol oleate | Sigma Aldrich GmbH | C-9253-250mg | Reconstitution |
| cholesterol | Sigma Aldrich GmbH | C8667-500mg | Reconstitution |
| glass tubes | Carl Roth GmbH | K226.1 | Reconstitution |
| spermine | Sigma Aldrich GmbH | S3256-1G | Reconstitution |
| Thermomixer | Eppendorf | Thermomixer comfort | Reconstitution |
| Sodium deoxycholate | Sigma Aldrich GmbH | 3097-25G | Reconstitution |
| Magnetic stir bar | Carl Roth GmbH | 0955.2 | Reconstitution |
| 100µL micropipettes | Carl Roth GmbH | A762.1 | Reconstitution |
| PBS | Carl Roth GmbH | 9143.2 | Dialysis |
| Amberlite XAD-2 beads | Sigma Aldrich GmbH | 10357 | Dialysis |
| Slide-A-Lyzer casette (cut-off 20 kDa) | Thermo Scientific | 66003 | Dialysis |
| Syringe | Braun | 9161406V | Dialysis |
| Syringe needle | Braun | 465 76 83 | Dialysis |
| EGTA | Carl Roth GmbH | 3054.2 | Labeling of LDL particles |
| DMSO | life technologies | D12345 | Labeling of LDL particles |
| Atomic Force Microscope | RIBM | SS-NEX | Quality control of reconstituted/labeled lipoprotein particles |
| Muscovite Mica (V-1 Grade) | Christine Gröpl | G250-1/V1 | Quality control of reconstituted/labeled lipoprotein particles |
| AFM Cantilever | Nanoworld | USC-F1.2-k0.15 | Quality control of reconstituted/labeled lipoprotein particles |
| Gwyddion 2.49 | Czech Metrology Institute | http://gwyddion.net | Quality control of reconstituted/labeled lipoprotein particles |
| Chamber slides, Nunc Lab-Tek | VWR | 734-2122 | Cell culture |
| HBSS | Carl Roth GmbH | 9117.1 | Cell culture |
| Cell counter | Omni Life Science GmbH | Casy | Cell culture |
| miRNeasy Mini Kit | QIAGEN | 217004 | miRNA extraction |
| Centrifuge | Eppendorf | 5415R | miRNA extraction |
| 20G needle | Braun | Sterican 465 75 19 | miRNA extraction |
| 5ml syringe | Becton Dickinson | 309050 | miRNA extraction |
| PCR cabinet | Esco | PCR-3A1 | Reverse Transcription |
| TaqMan Reverse Transcription Kit | Thermo Scientific | 4366596 | Reverse Transcription |
| Rnase-free water | Carl Roth GmbH | T143 | Reverse Transcription |
| 0.2 ml tubes | Brand | 781305 | Reverse Transcription |
| Centrifuge | Carl Roth GmbH | Microcentrifuge AL | Reverse Transcription |
| Thermocycler | Sensoquest | Labcycler | Reverse Transcription |
| TaqMan Primer | Thermo Scientific | - | qPCR |
| iTaq Universal probe supermix | BioRad GmbH | 1725131 | qPCR |
| PCR machine | Corbett | Rotor-Gene RG-6000 | qPCR |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366596 | TaqMan Array |
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | TaqMan Array |
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 | TaqMan Array |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399933 | TaqMan Array |
| TaqMan Universal Master Mix II, no UNG | Applied Biosystems | 4440040 | TaqMan Array |
| TaqMan Advanced miRNA Human A Cards | Applied Biosystems | A31805 | TaqMan Array |
| Nuclease-free water | Thermo Scientific | R0581 | TaqMan Array |
| MgCl2 (25mM) | Thermo Scientific | R0971 | TaqMan Array |
| TE, pH 8.0 | Invitrogen | AM9849 | TaqMan Array |
| 7900HT Fast Real-Time PCR System | Applied Biosystems | 4351405 | TaqMan Array |
| TaqMan Array Card Sealer | Applied Biosystems | - | TaqMan Array |
Riferimenti
- Vickers, K. C., Palmisano, B. T., Shoucri, B. M., Shamburek, R. D., Remaley, A. T. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nature Cell Biology. 13 (4), 423-435 (2011).
- Tabet, F., et al. HDL-transferred microRNA-223 regulates ICAM-1 expression in endothelial cells. Nature Communications. 5, (2014).
- Wahid, F., Shehzad, A., Khan, T., Kim, Y. Y. MicroRNAs: Synthesis, mechanism, function, and recent clinical trials. Biochimica et Biophysica Acta - Molecular Cell Research. 1803 (11), 1231-1243 (2010).
- Carthew, R. W., Sontheimer, E. J. Origins and Mechanisms of miRNAs and siRNAs. Cell. 136 (4), 642-655 (2009).
- Fabian, M. R., Sonenberg, N., Filipowicz, W. Regulation of mRNA Translation and Stability by microRNAs. Annual Review of Biochemistry. 79 (1), 351-379 (2010).
- Filipowicz, W., Bhattacharyya, S. N., Sonenberg, N. Mechanisms of post-transcriptional regulation by microRNAs: Are the answers in sight?. Nature Reviews Genetics. 9 (2), 102-114 (2008).
- Jonas, A. Reconstitution of High-Density Lipoproteins. Methods in Enzymology. , 553-582 (1986).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Bustin, S. A., et al. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Patsch, J. R., Patsch, W. Zonal ultracentrifugation. Methods in Enzymology. 129 (1966), 3-26 (1986).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Yamamoto, D., et al. High-Speed Atomic Force Microscopy Techniques for Observing Dynamic Biomolecular Processes. Methods in Enzymology. 475, 541-564 (2010).
- Stangl, H., Cao, G., Wyne, K. L., Hobbs, H. H. Scavenger receptor, class B, type I-dependent stimulation of cholesterol esterification by high density lipoproteins, low density lipoproteins, and nonlipoprotein cholesterol. Journal of Biological Chemistry. 273 (47), 31002-31008 (1998).
- Hochberg, B. Controlling the False Discovery Rate: a Practical and Powerful Approach to Multiple Testing. Journal of the Royal Statistical Society. 57 (1), 289-300 (1995).
- Mestdagh, P., et al. A novel and universal method for microRNA RT-qPCR data normalization. Genome Biology. 10 (6), (2009).
- Schumaker, V. N., Puppione, D. L. 6] Sequential Flotation Ultracentrifugation. Methods in Enzymology. 128 (C), 155-170 (1986).
- Wagner, J., et al. Characterization of levels and cellular transfer of circulating lipoprotein-bound microRNAs. Arteriosclerosis, Thrombosis, and Vascular Biology. 33 (6), 1392-1400 (2013).
- Meier, S. M., et al. Effect of chronic kidney disease on macrophage cholesterol efflux. Life Sciences. 136, 1-6 (2015).
- Axmann, M., et al. Serum and Lipoprotein Particle miRNA Profile in Uremia Patients. Genes. 9 (11), 533 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon