Method Article
Una piattaforma di membrana modello per la ricostituzione delle dinamiche della membrana mitocondriale
In questo articolo
Riepilogo
La fusione mitocondriale è un'importante reazione omeostatica alla base delle dinamiche mitocondriali. Descritto qui è un sistema di ricostituzione in vitro per studiare la fusione mitocondriale interno-membrana che può risolvere il tethering della membrana, docking, emifusione, e l'apertura pore. Viene discussa la versatilità di questo approccio nell'esplorazione dei sistemi a membrana cellulare.
Abstract
La dinamica mitocondriale è essenziale per le diverse funzioni e risposte cellulari dell'organello. L'affollata, spazialmente complessa, membrana mitocondriale è un ambiente impegnativo per distinguere i fattori normativi. Il controllo sperimentale dei componenti proteici e lipididi può aiutare a rispondere a domande specifiche di regolazione. Tuttavia, la manipolazione quantitativa di questi fattori è difficile nei saggi cellulari. Per studiare il meccanismo molecolare della fusione mitocondri interno-membrana, abbiamo introdotto una piattaforma di ricostituzione in vitro che imita l'ambiente lipidico della membrana interna mitocondriale. Qui descriviamo passaggi dettagliati per la preparazione di bistrati lipididi e la ricostituzione delle proteine della membrana mitocondriale. La piattaforma ha permesso l'analisi degli intermedi nella fusione mitocondriale interno-membrana, e la cinetica per le singole transizioni, in modo quantitativo. Questo protocollo descrive la fabbricazione di bistrati con composizione lipicida asimmetrica e descrive considerazioni generali per la ricostituzione delle proteine transmembrane in un bistrato ammortizzato. Il metodo può essere applicato per studiare altri sistemi di membrana.
Introduzione
La compartimentazione delle membrane è un segno distintivo delle celluleeucariotiche 1 (Figura 1A). Le membrane biologiche sono sempre più riconosciute come più di un solvente bidimensionale e sono considerate come un ambiente che gioca un ruolo fondamentale nella regolazione della funzione proteica e dell'assemblaggio complesso macromolecolare2,3. I lipidi nativi sono ligand che regolano l'attività proteica dellamembrana 3,4. L'organizzazione spaziale della membrana e la capacità delle membrane di essere scolpite in forme diverse sono importanti proprietà fisiche per la selezione di nuovefunzioni 3,5.
Le piattaforme a membrana modello sono sistemi biomimetici che possono aiutarci a comprendere la struttura della membrana cellulare, la dinamica ela funzione 6,7,8. Le membrane modellotiche comprendono tipicamente una miscela lipidica di composizione ben definita, con proprietà biofisiche definite (rigidità, spessore ed elasticità). Accoppiate all'imaging a fluorescenza, le piattaforme di membrana modello consentono l'analisi quantitativa della struttura dellamembranae della funzione 9,10,11. Le strategie di ricostituzione dei bistrati di lipidi sono state utilizzate per studiare la fusione della membrana mediata da SNARE9,10, la fusione della membrana mediata dal DNA12e la fusione virale11,13. Un vantaggio di tali metodi è la possibilità di ottenere informazioni cinetiche per i passaggi intermedi che precedono un evento di reazione osservabile14.
La membrana plasmatica è stata ampiamente studiata utilizzando membrane modello. I bistrati con separazione di fase lipibile sono stati sviluppati per studiare le strutture di zattera lipidiche importanti nella segnalazionecellulare 11,15,16. Micromodelli lipididi planari17,18 sono stati utilizzati per indagare l'organizzazione dei recettori cellulari. Le membrane polimeriche o supportate da gel sono state utilizzate come sistemi biomimetici per studiare l'organizzazione membrana-citoscheletro, il partizionamento delle proteine della membrana durante la segnalazione cellulare e la migrazione ai contatticellulare 19.
I sistemi di membrana artificiale vengono applicati anche allo studio degli organelli subcellulari20. Gli organelli presentano morfologie caratteristiche che creano sotto-ambienti distinti. La rete endoplasmic reticulum (ER) è un esempio. Dopo la ricostituzione dei reticoloni in liposomi, le strutture a membrana tubolare con proprietà simili al ER cellulare sonoformate 21. L'aggiunta di atlastina, una proteina di fusione ER, può indurre tubù lipidi da liposomi a formare una rete20. Questo è un esempio di come i proteoliposomi possono fornire una visione funzionale della morfologia e della dinamica degli organelli.
La fusione e fissione della membrana mitocondriale sono essenziali per la salute della popolazione mitocondriale22,23,24,25. Un insieme di gtPases della famiglia di dynamina catalizza la fusione della membrana dei mitocondri. Mfn 1/2 catalizza la fusione della membrana esterna. Opa1 media la fusione della membranainterna 26 (Figura 1B). Opa1 ha due forme: una forma lunga (l-Opa1), transmembrana-ancorata alla membrana interna mitocondriale, e una forma breve 'solubile' (s-Opa1), presente nello spazio intermembrana. Il rapporto tra le due forme Opa1 è regolato dall'attività di due proteasi, Oma1 e Yme1L27,28,29,30. Importanti questioni nel regolamento Opa1 includono: come le due forme di Opa1, (breve e lungo) mediano la fusione della membrana e la loro interazionenormativa 28,29,31,32,33.
Qui descriviamo una strategia di ricostituzione applicata con successo per studiare la fusione mitocondriale interno-membrana che ha chiarito i ruoli di l- e s-Opa1 nella fusione interna-membrana. Abbiamo sviluppato una piattaforma che imita la membrana interna mitocondriale utilizzando un bistrato lipidico con tediche polimero e vesicles unilamellari da 200 nm. I vantaggi di un tether polimerico sotto il bistrato lipidico includono quanto segue. In primo luogo, conserva la proteina transmembrana ricostituita, che altrimenti potrebbe essere interrotta dalla vicinanza allo scivolo di vetro34. In secondo luogo, serve uno spesso strato d'acqua tra il bistrato lipidico e il substrato di vetro, che facilita gli studi di apertura dei pori9,e in terzo luogo la natura viscoelastica del polimero PEG consente cambiamenti di curvatura della membrana35. Abbiamo usato l'imaging a fluorescenza a tre colori per caratterizzare i passaggi nella fusione della membrana (Figura 1C-F).
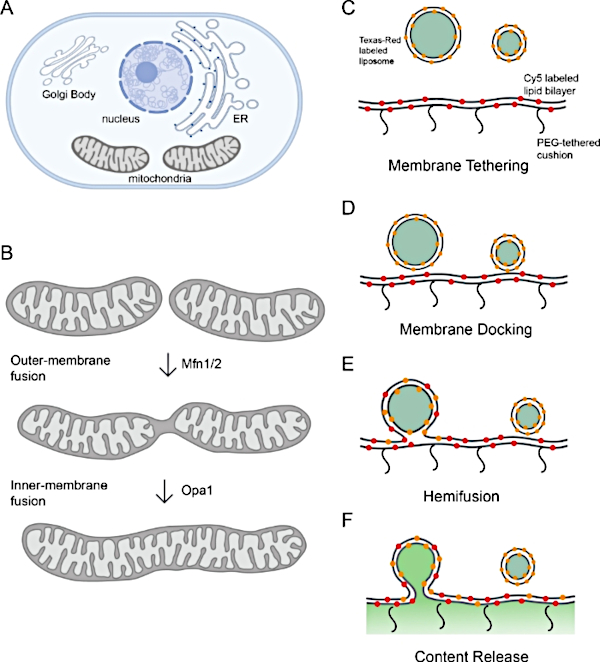
Figura 1: Monitoraggio della fusione della membrana mitocondriale.
(A) Gli organelli sono compartimenti cellulari della membrana. (B) Fasi sequenziali della fusione della membrana mitocondriale. La fusione della membrana esterna dei mitocondri è catalizzato da Mfn1 e/o Mfn2, mentre la fusione della membrana interna è mediata da Opa1. (C-F) Schematico della piattaforma di ricostituzione in vitro per studiare la fusione della membrana mitocondriale. La piattaforma comprende due parti: un proteoliposome e un bistratore lipidico polimerico, entrambi con l-Opa1 ricostituito. Le etichette fluorescenti, tra cui due diversi coloranti a membrana fluorescente e un marcatore di contenuto, aiutano a distinguere i passi durante la fusione della membrana. I due marcatori di membrana (Cy5-PE (rosso) e TexasRed PE (arancione) fanno una coppia FRET, che può riferire sull'attracco ravvicinato della membrana. La diffusione di TexasRed-PE che etichetta il proteoliposome è un indicatore di demixing lipidico (emifusione). Il rilascio del contenuto viene monitorato attraverso la dequenching del segnale calcein (mostrato in verde). Pannelli A e B creati utilizzando Biorender. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Preparazione delle miscele di lipidi
- Preparare una soluzione di stock di lipidi sciogliendo 1,2-dioleoyl-sn-glicero-3phosphocholine (DOPC), 1-palmitoyl-2-oleoyl-sn-glicero-3-phosphoethanolamine (POPE), L-fosphatidylinositol (Liver PI), cardiolipin, e 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-[methoxy(polietilene glicol)-2000] (18:1 PEG2000 PE) in cloroformio alla concentrazione di 25 mg/mL. Sciogliere il lipide coniugato colorante fluorescente (TexasRed DHPE e Cy5 DOPE) cloroformio ad una concentrazione di 1 mg/mL. Conservare la soluzione lipidico in fiale ambrata con focno resistente al cloroformio, ulteriormente sigillata con nastro di politetrafluoroetilene. La soluzione può essere conservata a -20 gradi centigradi per un massimo di 6 mesi.
- Creare soluzioni A e B.
- Mescolare il lipidi per preparare la soluzione A (concentrazione finale 1 mg/mL) che contiene DOPC (52,8 mol), POPE (20 mol%), fegato PI (7 mol%) cardiolipin (20 mol%) e 0,2 mol% fluoroforo.
- Fare la soluzione B (concentrazione finale 1 mg/mL) contenente DOPC (47,8 mol), POPE (20 mol%), PI epatico (7 mol%), cardiolipin (20 mol%) e DOPE-PEG2000 (5 mol%) e 0,2 mol% fluoroforo.
- Generare la miscela di lipidi aggiungendo il volume calcolato della soluzione di stoccaggio nelle fiale ambrate utilizzando una siringa di vetro. Abbinare il volume finale aggiungendo cloroformio in più nelle fiale.
NOTA: per FCS (spettroscopia di correlazione della fluorescenza), diminuire il rapporto tra il lipidi coniugato colorante a 0,002 mol% e sostituire il resto con DOPC.
2. Fabbricazione di bistrati lipididi
- Cuocere al microscopio coprire vetrini di vetro a 520 gradi centigradi per 30 min. Dopo la cottura, raffreddare i vetrini di copertura a temperatura ambiente.
- Pesare circa 10 g di idrossido di sodio e aggiungere a 500 mL di metanolo mescolando. Mescolare per 2 h, continuare ad aggiungere idrossido di sodio nella soluzione fino a quando i precipitati iniziano a mostrare. Assicurarsi di indossare PPE appropriato durante l'intero processo.
- Pulire i vetrini nella soluzione di sodio solfato al 10%; metanolo saturo di idrossido di sodio; e 50 mM di acido cloridrico, in sequenza (soripiazione del bagno in ogni condizione per 30 min). Pulire lo scivolo di vetro in acqua ultrapure per 10 min tra ogni condizione.
NOTA: Anche se altamente raccomandato per utilizzare soluzioni fresche per la preparazione del vetro, ogni soluzione può essere riutilizzata fino a 5x o entro 1 mese, a seconda di quale viene prima. Assicurarsi di mescolare-mescolare la soluzione prima di ogni utilizzo. - Conservare il vetro di copertura pulito sigillato in soluzione HCl fino a 2 settimane per garantire una buona qualità del bistrato. Se conservati in acqua ultrapure, utilizzare i vetrini entro una settimana.
- Pulire il politetrafluoroetilene trogolo del sistema di immersione Langmuir-Blodgett utilizzando cloroformio e acqua ultrapura fino a quando non si osserva alcuna bagnatura sulla depressione. Spruzzare cloroformio sulla superficie della depressione, pulire accuratamente con salviette di cellulosa 3x. Sciacquare con acqua ultrapura e rimuovere l'acqua tramite aspirazione. Ripetere 3x.
- Coprire la superficie della depressione con acqua ultrapura pulita.
- Prendere 2 pezzi di vetro di copertura trattato in superficie dalla soluzione di pulizia o dall'acqua ultrapura, e sciacquare lo scivolo di vetro con acqua ultrapura per circa 30 s.
- Posizionare il vetro di copertura in modo back-to-back. Utilizzare il morsetto del substrato per tenere i vetrini. Immergere lo scivolo di vetro sotto la superficie dell'acqua facendo clic manualmente su "dipper down" sul software di controllo Langmuir.
- Azzerare il bilanciamento della pellicola, diffondendo con attenzione la soluzione B goccia a goccia all'interfaccia aria-acqua (Figura 2A). Assicurarsi che i lipidi si diffondano solo all'interfaccia aria-acqua, senza cloroformio e goccioline di lipidi che affondano sul fondo della superficie del politetrafluoroetilene. In caso di incapacità di garantire che ciò crei un "canale" lipidico e prevenisse la formazione di monostrati.
- Smetti di aggiungere lipidi fino a quando la lettura dell'equilibrio della pellicola è di circa 15-20 mN/m, attendi 10-15 min. Avviare il controller della barriera per modificare la superficie facendo clic su "avvia esperimenti", fino a quando la lettura del bilanciamento della pellicola a 37 mN/m. Mantenere la pressione per 20-30 min (Figura 2B).
- Sollevare il vetro di copertura alla velocità di 22 mm/min mantenendo la tensione superficiale a 37 mN/m. Un monostrato lipidico con tethering polimerico verrà trasferito dall'interfaccia aria-acqua alla superficie del vetro di copertura attraverso il processo di immersione Blodgett (Figura 2C). Questo forma il volantino inferiore del bistrato lipidico.
- Pulire l'interfaccia aria-acqua per aspirazione, risciacquare la depressione con acqua ultrapura.
- Pulire uno scivolo di vetro con un solo pozzo (ad esempio, scivolo Shaefer) utilizzando cloroformio, etanolo e acqua ultrapura prima dell'uso. Impostare lo scivolo di vetro pulito sulla depressione con acqua ultrapura sotto lo strato d'acqua. Assicurarsi che il pozzo sia rivolto verso l'interfaccia aria-acqua e versare acqua ultrapure fresca fino a quando lo scivolo di vetro è completamente coperto. Ripetere il passaggio 2.8.
- Tenere il vetro di copertura con monostrato lipidico dal punto 2.4 utilizzando una ventre di silicio (assicurarsi che il lato monostrato sia lontano dalla coppa di aspirazione), spingere delicatamente il monostrato lipidico all'interfaccia aria-acqua, tenere il vetro di copertura per 2-3 s all'interfaccia, quindi spingere il vetro di copertura contro lo scivolo (Figura 2D). Togliere la diapositiva con una diapositiva di copertina.
NOTA: Il bistratore lipidico sarà tenuto sulla superficie del vetro di copertura rivolto verso l'area inserita tra i due vetrini(Figura 2E). - Portare il vetro di copertura con il bistrato ad un microscopio epifluorescenza. Immagine del bistratore lipidico. Se si osserva una distribuzione omogenea del colorante lipidico, fotobleach una piccola area del bilayer per 30 s, spegnere la sorgente luminosa per 30 s-1 min., quindi immagine di nuovo per osservare il recupero. Il bistrato lipidico mostrerà il recupero dalla fluorescenza.
NOTA: Le membrane con difetti o un cattivo recupero dalla fluorescenza non devono essere utilizzate per ulteriori esperimenti.
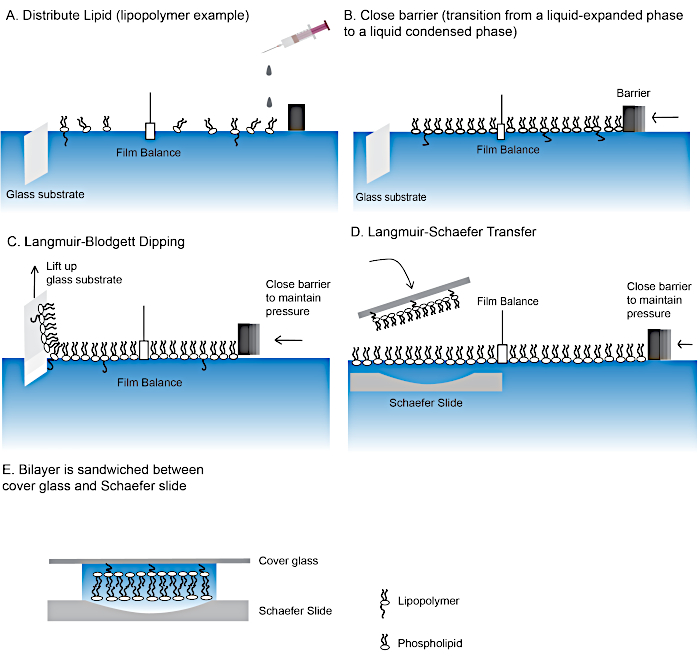
Figura 2: Passaggi per la creazione di un bistrato lipidico con legame polimerico.
Fasi di produzione di bistrati lipididi utilizzando le tecniche di immersione Langmuir-Blodgett (A-C) e Langmuir-Schaefer trasferimento (D). (E) Il "sandwich" finale contenente il bistrato lipidico. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
3. Ricostituzione delle proteine nel bistratore lipidico con legame con polimero
- Preparare un piatto di cristallizzazione contenente acqua ultrapura. Preparare un anello di immagine al microscopio pulito e posizionare sotto il piatto.
- Immergere il "sandwich" dello scivolo Schaefer e coprire il vetro che contiene il bistrato lipiduciato sotto l'acqua, separare delicatamente lo scivolo Di Schaefer e coprire il vetro, tenere lo scivolo di vetro di copertura dal basso, lontano dal lato bilayer, trasferire il vetro di copertura nell'anello dell'immagine, chiudere l'anello dell'immagine.
NOTA: Assicurarsi che il vetro di copertura con bistrato lipidico sia sempre in acqua e che l'anello sia ben sigillato. - Sostituire l'acqua ultrapura nell'anello immagine con il buffer Bis-Tris NaCl, assicurarsi che il bistrato lipidico non sia esposto ad alcuna bolla d'aria. Aggiungere 1,1 x 10-9 M n-Octyl-β-D-Glucopyranoside al bistrato lipidico. Aggiungere immediatamente la miscela di 1,2 x 10-9 M di DDM e 1,3 x10 -12 mol purificato l-Opa136 nell'anello dell'immagine. Incubare il campione su uno shaker a bassa velocità per 2 h (Figura 3).
NOTA: I detergenti possono variare a seconda delle proteine da ricostituire. - Distribuire 30 mg SM-2 Perline resina in 3 mL di tampone Bis-Tris e agitare prima di applicare. Utilizzare una pipetta di plastica per aggiungere 5-10 L di perline di resina SM-2 all'anello di immagine, incubare per 10 min, rimuovere le perline di resina mediante risciacquo. Il volume finale del buffer nell'anello dell'immagine è 1,5 mL.
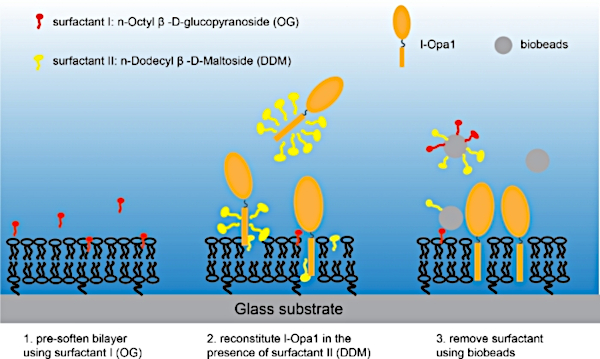
Figura 3: Procedura per ricostituire l-Opa1 in un bistrato lipidico con teoreria polimerica. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
4. Preparazione dei proteoliposomi
- Preparare 1 mg di miscela lipide A nella soluzione cloroformia. Evaporare il cloroformio sotto il flusso di azoto per 20 minuti e tenere sotto vuoto durante la notte e formare una pellicola lipidida.
- Preparare 50 mM calcein contenente tampone sciogliendo 15,56 g di calceina a 50 mL di soluzione NaOH da 1,5 mol, mescolare a temperatura ambiente fino a quando la calceina è completamente disciolta, ha aggiunto 12,5 mM Bis-Tris e acqua ultrapura al volume finale di 500 mL. Regolare il pH a 7,5.
- Sospendere la pellicola lipidida in calcetina contenente tampone, idratare completamente il lipidico riscaldando le sospensioni a 65 gradi centigradi per 20 minuti. 200 nm liposomi si formano attraverso l'estrusione utilizzando una membrana in policarbonato.
- Aggiungere 2 g di l-Opa1 in 0,5 M DDM a 0,2 mg di liposoma e incubare a 4 gradi centigradi per 1,5 h. Rimuovere il surfactant con dialisi utilizzando una cassetta di dialisi da 3,5 kDa contro 250 ml di 25 mM Bis-Tris, 150 mM NaCl e 50 mM calceina tampone a 4 gradi centigradi durante la notte, cambiando il buffer due volte.
- Rimuovere la calceina supplementare utilizzando una colonna di desalting PD-10.
5. Imaging e analisi dei dati
- Acquisire immagini TIRF utilizzando un obiettivo di immersione dell'olio 100x (N.A 1.4). Utilizzare laser a 543 nm e un laser da 488 nm per l'analisi di liposomi e proteoliposomi etichettati TexasRed-PE incapsulati con calceina. Utilizzare un laser a 633 nm per l'analisi di Cy5-PE incorporato nel bilayer lipidico planare.
- Allineare l'angolo TIRF utilizzando un bistrato lipidico per ottenere la massima emissione. La qualità del bistrato lipidico dopo la ricostituzione viene osservata utilizzando un obiettivo di olio 100x a 25 gradi centigradi. Il coefficiente di diffusione del bistratore fosfolopido e ricostituito viene determinato utilizzando FCS con un protocollo descritto altrove37.
- Aggiungere 10 L di 2 mg/mL proteoliposomes all'anello dell'immagine e impostare per 10 minuti prima dell'immagine. GTP, GMPPCP o PIL vengono aggiunti nell'anello di reazione con 1 mM MgCl2 e 1 mM di nucleotide.
- Per determinare l'influenza di s-Opa1 nella fusione della membrana, titrate s-Opa1 in un campione di proteoliposome/supportato bistrato contenente l-Opa1, e registrare gli eventi di fusione.
- L'imaging simultaneo di TexasRed-DHPE e calcein si ottiene attraverso un sistema di scissione dei fasci. Entrambi i laser da 488 nm e 543 nm vengono applicati contemporaneamente al campione come sorgenti di eccitazione farinostenibile. La luce di emissione viene quindi divisa utilizzando uno splitter di fascio da 560 nm. La luce di emissione divisa viene quindi filtrata da un filtro di 510 nm con una larghezza di banda di 42 nm e un filtro a 609 nm con una larghezza di banda di 40 nm. Il fascio filtrato viene proiettato su due aree adiacenti sul chip della fotocamera.
- Le emissioni fluorescenti vengono registrate simultaneamente tramite un filtro a 609 emissioni con una larghezza di banda di 40 nm e un filtro a 698 emissioni con una larghezza di banda di 70 nm. Il sistema al microscopio è dotato di una telecamera CMOS mantenuta a -10 gradi centigradi.
- L'identificazione delle particelle dei liposomi può essere eseguita utilizzando un algoritmo di riconoscimento delle particelle basato sulla gaussiana. La distribuzione e l'intensità delle particelle vengono analizzate canale per canale. Un segnale di bistrato lipidico viene utilizzato come maschera per isolare le particelle.
Risultati
La proteina transmembrana ricostituita si diffonde liberamente ed è distribuita omogeneamente nella membrana.
Le immagini di esempio di un bistrato lipidico e la sua fluidità lipidida convalidata dalla microscopia epifluorescenza sono illustrate nella Figura 4. La distribuzione dei lipidi nel bistrato prima e dopo la fotobleaching è illustrata nella Figura 4A,B. L'omogeneità del bistrato lipidico è stata mostrata utilizzando un microscopio di epifluorescenza prima e dopo la ricostituzione (Figura 4D,E). l-Opa1 ricostituito in bistrato lipidico è stato convalidato dalla spettroscopia di correlazione della fluorescenza (FCS). Usiamo lipidi coniugati coloranti per valutare la diffusione dei lipidi del bistrato. L'Opa1 ricostituito è stato etichettato con un anticorpo anti-Opa1 C-terminale con tag fluorescenti. La diffusione dei lipidi bistrato è stata misurata come 1,46 x 0,12 m2/s,mentre il coefficiente di diffusione dell'l-Opa1 ricostituito da bistrato era di 0,88 x 0,10 m2/s. L'intensità delle curve FCS indicate come 75% di l-Opa1 viene ricostituita nel bistrato lipidico(Figura 4G,H). Questi risultati suggeriscono che l-Opa1 si diffonde liberamente nel bistrato lipidico polimerico con il potenziale di auto-assemblarsi in complessi funzionali.
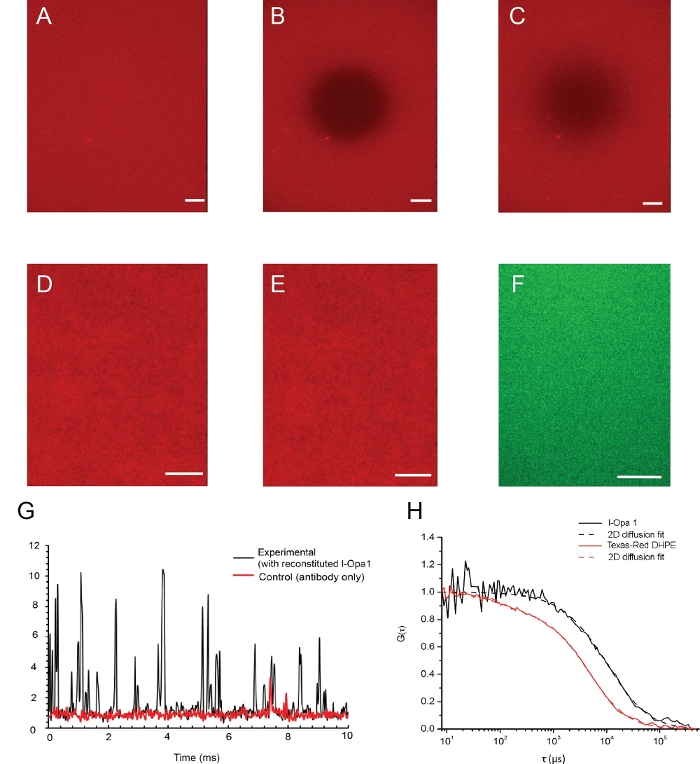
Figura 4: Distribuzione di lipidi e proteine ricostituita nella membrana del modello.
(A-C) Immagini di esempio di un bistrato lipidico e della sua fluidità lipidida convalidate dalla microscopia epifluorescenza. (A) Distribuzione di lipidi omogenei in bistrato prima del fotobleaching. (B) Istantanea immediatamente dopo la fotobleaching. (C) Bistrato immagine dopo il recupero della fluorescenza indica una buona fluidità lipidico della membrana dopo la ricostituzione. (D,E) Le immagini rappresentative della distribuzione dei lipidi prima (D) e dopo (E) l-Opa1 ricostituzione indicano che il processo di ricostituzione non ha creato difetti nel bistrato. Immagine rappresentativa TIRF di l-Opa1 etichettata con anticorpo coniugato Alexa 488 (F) che mostra una distribuzione omogenea di Opa1 al momento della ricostituzione. G. Rappresentante conteggi di fotoni grezzi del segnale l-Opa1 per spettroscopia di correlazione fluorescente. Nel controllo, nessun l-Opa1 è stato ricostituito nel bistrato, mentre l'anticorpo è stato aggiunto e risciacquato. La diffusione di l-Opa1 è significativamente più lenta dei lipidi nella membrana, coerente con la ricostituzione di successo della transmembrane l-Opa1 (H). Barra della scala: 10 m. Fare clic qui per visualizzare una versione più grande di questa figura.
Lo sbiancamento della fase di fluorescenza ha indicato una media di 2-3 copie di l-Opa1 ricostitute in un dato liposoma (Figura 5A,B). La distribuzione delle dimensioni di proteoliposoms ricostituito Opa1 è stata testata dopo la ricostituzione utilizzando DLS (Figura 5C). La ricostituzione di Opa1 in proteoliposoms è stata verificata anche utilizzando FCS. Il coefficiente di diffusione dell'anticorpo libero era di 164 x 22 m2/s; coefficiente di diffusione per liposomi etichettati con un colorante lipidico era di 2,22 x 0,33 m2/s, e il coefficiente di diffusione per i proteoliposomi l-Opa1 legato ad un anticorpo Anti-His etichettato TexasRed era 2,12 x 0,362/s.
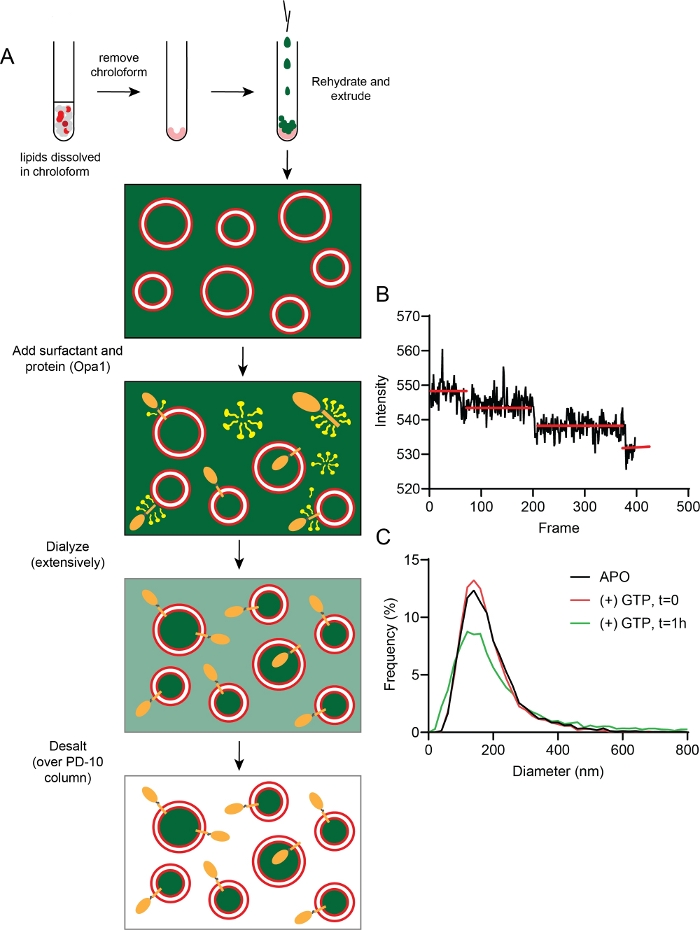
Figura 5: Fabbricazione e caratterizzazione dei proteoliposomi.
(A) Passi nella fabbricazione di proteoliposomi con calceina incapsulata e sconsigliata. (B) I dati rappresentativi dello sbiancamento fluorescente mostrano una media di 2-3 copie di l-Opa1 incorporate nel liposoma. (C) Distribuzione di dimensioni rappresentative di proteoliposomi (rosso) senza alcun nucleotide 1 h dopo l'incubazione GTP (verde). Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
Rilevamento del tethering a membrana, della demixing/emifusione dei lipidi e dell'apertura delle pore mediante microscopia fluorescente.
Il tethering a membrana viene monitorato osservando il segnale di TexasRed sulla superficie del bistrato lipidico utilizzando la microscopia TIRF (Figura 6A). Il comportamento della demixing dei lipidi di membrana (emifusione) è stato monitorato attraverso TexasRed mentre il colorante si diffonde dai liposomi al bistrato lipidico. Calcein dequenching aiuta a distinguere la piena formazione di pori di fusione dal solo demixing lipidico. Ciò consente il confronto tra le condizioni in cui le particelle si bloccano all'emifusione (Fig 6B) e le particelle che procedono alla fusione completa (Figura 6C).
Il tethering della membrana è indicato da un segnale lipidico stabile dai liposomi. La distanza può essere valutata in base al segnale FRET tra le etichette delle due membrane36. Il segnale di emirfusione non presenta alcun dequenching nel segnale calcein (Figura 6B, riga inferiore), ma un rapido decadimento del segnale TexasRed indica la diffusione del colorante nel bistrato lipidico (Figura 6B riga superiore). La fusione completa (con apertura dei pori) presenta sia il decadimento dei lipidi che il rilascio del contenuto (Figura 6C). L'intensità e l'intensità della calceina del TexasRed possono essere monitorate in modo dipendente dal tempo per fornire dettagli quantitativi per la cinetica della fusione dellamembrana 36.

Figura 6: Risultati rappresentativi che mostrano il tethering delle particelle (A, barra della scala 10 m), l'emifusione (B, barra della scala 0,5 m) e la fusione (C, barra di scala 0,5 m).
(A) Proteoliposomes legato a Opa1-ricostituito bilayer prima dell'aggiunta GTP. (B) Un esempio di emifusione. La riga superiore in B mostra il demixing dei lipidi (segnale TexasRed, rosso), la riga inferiore in B non mostra alcun rilascio di contenuto (segnale di calceina, verde) in queste condizioni. (C) Una traccia rappresentativa di proteoliposome fondersi con il bistrato lipidico. Rilascio del contenuto può essere osservato dalle immagini nella riga inferiore di mostrando dequenching di calcein (riga inferiore, verde). Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
I sistemi di membrana modello-membrana in vitro possono descrivere processi di membrana complessi in condizioni ben definite. Questi sistemi sono in grado di distinguere i componenti minimi necessari per processi molecolari complessi per rivelare meccanismimolecolari 6,15,20,38. Per le proteine della membrana, i liposomi e i bistrati supportati da planari sono sistemi di ricostituzione comuni. A differenza dei bistrati lipididi a supporto solido, il cuscino polimerico tra il substrato e la membrana supportata in bistrati polimeri consente la mobilità libera di grandi proteine della membrana e le proteine transmembrane per diffondere e assemblareliberamente 34. Queste caratteristiche ci hanno aiutato a studiare la cinetica della fusione della membrana interna deimitocondri 36.
Abbiamo preparato un bistrato lipidico polimerico utilizzando tecniche Langmuir-Blodgett/Langmuir-Schaefer (LB/LS). Questo ci permette di preparare un bistrato con componenti lipidi asimmetrici. Le membrane cellulari hanno una composizione asimmetrica del volantino e l'approccio LB/LS consente lo studio di tali bistrati. Con il trasferimento di Schaefer, un intero substrato di vetro può essere coperto da un bistrato lipidico. È fondamentale preparare una superficie pulita per la preparazione dei bilayer. Inoltre, ci vuole pratica per eseguire correttamente un trasferimento Schaefer. Il trasferimento infruttuoso di Schaefer può creare difetti indesiderati in un bilayer lipidico. In questo protocollo, la pressione aggiunta al bilanciamento della pellicola è applicabile per un bistrato contenente 20% cardiolipin. Per i bistrati con altri componenti, fare riferimento all'isotherm dell'area di pressione superficiale dei componenti chiave. Un metodo alternativo è il metodo Langmuir-Blodgett/vesicle fusion (LB/VF), in cui il monostrato lipidico inferiore viene trasferito dall'interfaccia aria-acqua di un trota Langmuir su un substrato pulito, quindi i liposomi si fondono alla parte superiore del monostrato lipido supportato e formano il bistratofinale 39. La ricostituzione delle proteine della membrana utilizzando il metodo LB/VF è più semplice di LB/LS, poiché la ricostituzione può essere eseguita attraverso la fusione di proteoliposomi. Tuttavia, la fusione della vescicola richiede l'aggiunta di liposomi in eccesso, che possono complicare lo studio degli eventi della membrana dipendenti dalle interazioni proteina-proteina dipendenti dalla concentrazione.
La ricostituzione efficace delle proteine transmembrane in bistrati lipididi polimeri e liposomi in un orientamento funzionale preferito è importante, ma difficile da applicare. I controlli sperimentali sono necessari per tenere conto di questo. Per i bistrati lipididi polimeri, è anche importante mantenere l'integrità del bistrato lipidico durante la ricostituzione. Le concentrazioni di surfactant devono essere mantenute relativamente basse per evitare lo scioglimento del bistrato lipidico, ma abbastanza elevate da prevenire la denaturazione della proteina di interesse37,40. Il metodo qui descritto è ideale per ricostituire le proteine della membrana per studi su una singola molecola, ma non è necessariamente scalabile per studi su larga scala. La scelta di Surfactant è un'altra considerazione importante. Spesso, il surfactant utilizzato per la purificazione e lo stoccaggio è un buon punto di partenza. La concentrazione massima di surfactant è di solito 200 volte meno del CMC36, in una gamma in cui il surfactant mantiene la stabilità delle proteine e impedisce l'aggregazione proteica, pur mantenendo l'integrità dellamembrana 36. Possono essere considerati cocktail contenenti 2 o 3 surfactant. Per la ricostituzione in liposomi, non è necessaria una bassa concentrazione di surfactant. Tuttavia, le concentrazioni di surfactant al di sotto della CMC sono preferibili per mantenere uniforme la distribuzione delle dimensioni e della morfologia per i liposomi. Per evitare perdite di colorante contenuto, è necessario dialyze contro un buffer contenente coloranti.
A differenza dei saggi di fusione a base di liposomi, la piattaforma che abbiamo stabilito fornisce un approccio per studiare la cinetica di ogni fase della fusione della membrana. Questo metodo fornisce la possibilità di studiare le proteine di fusione transmembrana in condizioni quasi autoctone. Le piattaforme di membrana modello possono essere applicate per studiare l'assemblaggio delle proteine della membrana e l'oligomerizzazione, la "scultura" della membrana e le interazioni proteina-lipidico delle proteine in ambienti subcellulari, come la membrana interna mitocondriale. Questo metodo permette anche l'esplorazione di importanti condizioni fisiologiche nell'interazione membrana-proteina, come l'asimmetria della composizione bistrato. Resta da definire il ruolo di un lipidico mitocondriale chiave, cardiolipin, nelle proprietà bistrati sia dei liposomi che dei bistrati supportati dai polimeri. Proprietà come la forza ionica, lo spessore della membrana, la rigidità della membrana, la curvatura della membrana e le proprietà di viscosità elastica della membrana possono influenzare la capacità delle proteine di assemblaggio in stati funzionali specifici. Studi futuri che applicano creativamente sistemi di membrana modello hanno il potenziale per scoprire nuovi aspetti dell'organizzazione e della funzione delle proteine della membrana.
Divulgazioni
Nessuno.
Riconoscimenti
Gli autori riconoscono il sostegno del Charles H. Hood Foundation Child Health Research Award e il generoso sostegno del Dipartimento di Biologia Molecolare del Massachusetts General Hospital.
Materiali
| Name | Company | Catalog Number | Comments |
| 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(Cyanine 5) | Avanti polar lipid | Cat #: 810335C1mg | membrane fluorescent markers |
| 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-[methoxy(polyethylene glycol)-2000] (ammonium salt) | Avanti Polar lipids | Cat #: 880130P | lipid molecules |
| 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol (sodium salt) | Avanti Polar lipids | Cat #: 710335P | lipid molecules |
| 18:1 (Δ9-Cis) PC (DOPC) | Avanti Polar lipids | Cat #: 850375P | lipid molecules |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphoethanolamine | Avanti Polar lipids | Cat #: 850757P | lipid molecules |
| Alexa Fluor 488 Antibody Labeling Kit | ThermoFisher Scientific | A20181 | |
| Amber vial with PTFE liner | Fisher scientific | 14-955-332 | sample vials to keep lipid solutions |
| Calcein | Sigma-Aldrich | Cat #: C0875; PubChem Substance ID: 24892279 | fluorescent dye |
| Chloroform | Fisher scientific | 298-500/ C295-4 | Fisher brand Chloroform is usually quite reliable for lipid works. |
| Concavity slide (1 well) | Electron Microscopy Science | 71878-05 | applied as Schaefer Slide |
| FCS analysis tool | Smith Lab, University of Akron | software tool | |
| Fiji /ImageJ | Fiji | SCR_002285 | software tool |
| Fisherbrand Cover Glasses: Circles | Fisher scientific | 12-545-102 | Cover glass for solid supported lipid bilayers, the item is now discontinued as authors prepared the manuscript. An alternative is Fisher brand premium cover glass with catalog number: 12-548-5M |
| GTP Disodium salt | SIGMA-ALDRICH INC | Cat #: 10106399001 | |
| Langmuir & Langmuir-Blodgett Trough | Biolin Scientifc | KN2002 | |
| L-α-lysophosphatidylinositol (Liver, Bovine) (sodium salt) | Avanti Polar lipids | Cat #: 850091P | lipid molecules |
| Mini Extruder | Avanti Polar lipids | 610020 | |
| n-Dodecyl-β-D-Maltopyranoside | Anatrace | Cat #: D310 25 GM | surfactant for reconstitution |
| n-Octyl-α-D-Glucopyranoside | Anatrace | Cat #: O311HA 25 GM | surfactant for reconstitution |
| PC Membranes 0.2μm | Avanti Polar Lipids | 610006 | |
| Rabbit Anti-Opa1 antibody | NOVUS BIOLOGICALS | Cat #: NBP2-59770 | antibody for Opa1 C-terminal detection |
| Slidebook | Intelligent imaging | RRID: SCR_014300 | software tool |
| Teflon threaded seal tape | Fisher Scientific | NC0636085 | taflon tape for sample storage |
| Texas Red 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine, Triethylammonium Salt (Texas Red DHPE) | ThermoFisher Scientific | Cat #: T1395MP | membrane fluorescent markers |
Riferimenti
- Sackmann, E., Lipowsky, R., Sackmann, E. Biological membranes architecture and function. Structure and Dynamics of Membranes. 1, 1-63 (1995).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118, 1099-1102 (2005).
- Schink, K. O., Tan, K. W., Stenmark, H. Phosphoinositides in Control of Membrane Dynamics. Annual Review of Cell and Developmental Biology. 32, 143-171 (2016).
- Gu, R. X., Ingolfsson, H. I., de Vries, A. H., Marrink, S. J., Tieleman, D. P. Ganglioside-Lipid and Ganglioside-Protein Interactions Revealed by Coarse-Grained and Atomistic Molecular Dynamics Simulations. Journal of Physical Chemistry B. 121 (15), 3262-3275 (2017).
- Schafer, D. A. Coupling actin dynamics and membrane dynamics during endocytosis. Current Opinion in Cell Biology. 14 (1), 76-81 (2002).
- Chan, Y. H., Boxer, S. G. Model membrane systems and their applications. Current Opinion in Chemical Biology. 11 (6), 581-587 (2007).
- Biswas, K. H., Groves, J. T. Hybrid Live Cell-Supported Membrane Interfaces for Signaling Studies. Annual Reviews in Biophysics. 48, 537-562 (2019).
- Pick, H., Alves, A. C., Vogel, H. Single-Vesicle Assays Using Liposomes and Cell-Derived Vesicles: From Modeling Complex Membrane Processes to Synthetic Biology and Biomedical Applications. Chemical Reviews. 118 (18), 8598-8654 (2018).
- Kiessling, V., Domanska, M. K., Tamm, L. K. Single SNARE-mediated vesicle fusion observed in vitro by polarized TIRFM. Biophysical Journal. 99 (12), 4047-4055 (2010).
- Kiessling, V., et al. Rapid fusion of synaptic vesicles with reconstituted target SNARE membranes. Biophysical Journal. 104 (9), 1950-1958 (2013).
- Yang, S. T., Kiessling, V., Tamm, L. K. Line tension at lipid phase boundaries as driving force for HIV fusion peptide-mediated fusion. Nature Communication. 7, 11401 (2016).
- Rawle, R. J., van Lengerich, B., Chung, M., Bendix, P. M., Boxer, S. G. Vesicle fusion observed by content transfer across a tethered lipid bilayer. Biophysical Journal. 101 (8), 37 (2011).
- Chao, L. H., Klein, D. E., Schmidt, A. G., Pena, J. M., Harrison, S. C. Sequential conformational rearrangements in flavivirus membrane fusion. Elife. 3, 04389 (2014).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Sezgin, E., Schwille, P. Model membrane platforms to study protein-membrane interactions. Molecular Membrane Biology. 29 (5), 144-154 (2012).
- Ge, Y., Gao, J., Jordan, R., Naumann, C. A. Changes in Cholesterol Level Alter Integrin Sequestration in Raft-Mimicking Lipid Mixtures. Biophysical Journal. 114 (1), 158-167 (2018).
- Dutta, D., Kam, L. C. Micropatterned, multicomponent supported lipid bilayers for cellular systems. Methods in Cell Biology. 120, 53-67 (2014).
- Groves, J., Ferrari, M., Desai, T., Bhatia, S. Supported Lipid Bilayers as Mimics for Cell Surfaces and as Tools in Biotechnology. BioMEMS and Biomedical Nanotechnology. , (2006).
- Shoaib, T., Nalam, P. C., He, Y., Chen, Y., Espinosa-Marzal, R. M. Assembly, Morphology, Diffusivity, and Indentation of Hydrogel-Supported Lipid Bilayers. Langmuir. 33 (28), 7105-7117 (2017).
- Wang, N., Rapoport, T. A. Reconstituting the reticular ER network - mechanistic implications and open questions. Journal of Cell Science. 132 (4), 227611 (2019).
- Powers, R. E., Wang, S., Liu, T. Y., Rapoport, T. A. Reconstitution of the tubular endoplasmic reticulum network with purified components. Nature. 543 (7644), 257-260 (2017).
- Chan, D. C. Fusion and fission: interlinked processes critical for mitochondrial health. Annual Reviews of Genetics. 46, 265-287 (2012).
- Nunnari, J., Suomalainen, A. Mitochondria: in sickness and in health. Cell. 148 (6), 1145-1159 (2012).
- Wai, T., Langer, T. Mitochondrial Dynamics and Metabolic Regulation. Trends in Endocrinology Metabolism. 27 (2), 105-117 (2016).
- Westermann, B. Mitochondrial fusion and fission in cell life and death. Nature Reviews Molecular Cell Biology. 11 (12), 872-884 (2010).
- Chen, H., et al. Mitofusins Mfn1 and Mfn2 coordinately regulate mitochondrial fusion and are essential for embryonic development. Journal of Cell Biology. 160 (2), 189-200 (2003).
- Ishihara, N., Fujita, Y., Oka, T., Mihara, K. Regulation of mitochondrial morphology through proteolytic cleavage of OPA1. EMBO Journal. 25 (13), 2966-2977 (2006).
- Anand, R., et al. The i-AAA protease YME1L and OMA1 cleave OPA1 to balance mitochondrial fusion and fission. Journal of Cell Biology. 204 (6), 919-929 (2014).
- Mishra, P., Carelli, V., Manfredi, G., Chan, D. C. Proteolytic cleavage of Opa1 stimulates mitochondrial inner membrane fusion and couples fusion to oxidative phosphorylation. Cell Metabolism. 19 (4), 630-641 (2014).
- Baker, M. J., et al. Stress-induced OMA1 activation and autocatalytic turnover regulate OPA1-dependent mitochondrial dynamics. EMBO Journal. 33 (6), 578-593 (2014).
- DeVay, R. M., et al. Coassembly of Mgm1 isoforms requires cardiolipin and mediates mitochondrial inner membrane fusion. Journal of Cell Biology. 186 (6), 793-803 (2009).
- Rainbolt, T. K., Lebeau, J., Puchades, C., Wiseman, R. L. Reciprocal Degradation of YME1L and OMA1 Adapts Mitochondrial Proteolytic Activity during Stress. Cell Reports. 14 (9), 2041-2049 (2016).
- Ban, T., et al. Molecular basis of selective mitochondrial fusion by heterotypic action between OPA1 and cardiolipin. Nature Cell Biology. 19 (7), 856-863 (2017).
- Tanaka, M., Sackmann, E. Polymer-supported membranes as models of the cell surface. Nature. 437 (7059), 656-663 (2005).
- Shilts, K., Naumann, C. A. Tunable cell-surface mimetics as engineered cell substrates. Biochimica Biophysica Acta Biomembrane. 1860 (10), 2076-2093 (2018).
- Ge, Y., et al. Two forms of Opa1 cooperate to complete fusion of the mitochondrial inner-membrane. Elife. 9, 50973 (2020).
- Ge, Y., Siegel, A. P., Jordan, R., Naumann, C. A. Ligand binding alters dimerization and sequestering of urokinase receptors in raft-mimicking lipid mixtures. Biophysical Journal. 107 (9), 2101-2111 (2014).
- Simons, K., Vaz, W. L. Model systems, lipid rafts, and cell membranes. Annual Review of Biophysics and Biomolecular Structure. 33, 269-295 (2004).
- Kiessling, V., Liang, B., Tamm, L. K. Reconstituting SNARE-mediated membrane fusion at the single liposome level. Methods in Cell Biology. 128, 339-363 (2015).
- Siegel, A. P., Kimble-Hill, A., Garg, S., Jordan, R., Naumann, C. A. Native ligands change integrin sequestering but not oligomerization in raft-mimicking lipid mixtures. Biophysics Journal. 101 (7), 1642-1650 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon