Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Bir Vaka Çalışması Olarak Tütün Geçici Protein Expression: Deneyler Yaklaşımı Tasarım Kullanma Karmaşık Sistemler Karakterizasyonu
Bu Makalede
Özet
Bu bitkilerde monoklonal antikorlar ve raportör proteinlerin geçici transgen ifadesi üzerindeki düzenleyici elemanlar, bitki büyüme ve gelişme parametreleri ve inkübasyon koşullarının etkisini belirlemek ve modellemek için kullanılabilecek deneyler yaklaşımın bir tasarımını tarif eder.
Özet
Bitkiler düşük maliyet, ölçeklenebilirlik ve güvenlik dahil biyofarmösetiklerinin üretimi için birden çok yarar sağlar. Geçici sentezleme, kısa geliştirme ve üretim kez ek bir avantaj sunar, ancak ekspresyon seviyeleri böylece iyi üretim uygulamaları bağlamında düzenleyici endişelere yol açan gruplar arasında önemli ölçüde değişebilir. Biz böyle partiler arasındaki ifade değişkenliğine ifadesi sırasında düzenleyici ifade yapı elemanları, bitki büyüme ve gelişme parametreleri ve inkübasyon koşulları gibi önemli faktörlerin etkisini belirlemek için deneyler (DOE) yaklaşımın bir tasarım kullanılmış. Bu model, anti-HIV monoklonal antikoru (2G12) ve bir flüoresan işaret proteini (DsRed) ifade eden bitkiler test edilmiştir. Biz modelin bazı özellikleri seçmek için gerekçe tartışmak ve potansiyel sınırlamaları belirlemek. Genel yaklaşım kolayca diğer sorunlara transfer edilebilir çünkü bu model ilkelerigeniş yeniden uygulanabilir: küçük modüller, optimum deney kombinasyonlar Yazılım destekli kurulum ve adım adım tasarım büyütme içine ilk sorunu bölerek bilgi-tabanlı parametre seçimi, karmaşıklığı azaltma. Bu nedenle, yöntem, sadece bitkilerde protein ekspresyonu karakterize etmek için değil, aynı zamanda mekanik bir açıklama eksik diğer karmaşık sistemlerin araştırılması için yararlıdır. Parametreler arasındaki birleştiricisi tarif akıllı denklem diğer sistemler için karmaşık mekanik modelleri oluşturmak için kullanılabilir.
Giriş
Bitkiler büyümeye ucuzdur çünkü bitkilerde biofarmasötik proteinlerin üretimi avantajlıdır, platformu sadece daha fazla büyüyen bitkiler tarafından büyütülüyor olabilir, ve insan patojenler 1,2 çoğaltmak mümkün değildir. DNA teslimat ve arıtılmış bir ürün teslimat noktası arasındaki süre yaşındaki az 2 ay 3 azaltılır, çünkü Agrobacterium tumefaciens ile yaprakların infiltrasyonu örneğin göre geçici sentezleme stratejileri, ek faydalar sağlar. Geçici sentezleme aynı zamanda zarar fonksiyon-mutantları tamamlamak için veya protein etkileşimlerini incelemek 4-6 etme kabiliyetleri açısından test etmek için genlerin fonksiyonel analiz için, örneğin kullanılır. Bununla birlikte, geçici ekspresyon düzeyi transjenik bitkilerde 7-9 ifade seviyelerinde daha büyük partiden partiye farklılıklar gösterir eğilimindedir. Bu biyofarmasötik üretim süreçleri geçici ifade wi göre olasılığını azaltıruyarlık bir kritik kalite nitelik ve risk değerlendirmesi 10 tabi olduğu için iyi üretim uygulamaları (GMP) kapsamında onaylanmış olacak. Böyle bir varyasyon da araştırmacılar araştırmak niyetinde herhangi etkileşimleri maskeleyebilir. Bu nedenle, bitkiler geçici ekspresyon düzeylerini etkileyen ve yüksek kaliteli bir nicel öngörü modeli oluşturmak için önemli faktörleri belirlemek için yola çıktı.
Tek-faktör-at-a-time (OFAT) yaklaşım, genellikle bir deney 11 sonucuna belirli parametreler (faktörler) etkisi (etkisi) (tepki) karakterize etmek için kullanılır. Bir soruşturma (deney) sırasında bireysel testler (ishal) (tasarım alanı) test edilir faktörler tarafından yayılmış potansiyel alan boyunca bir dize inci gibi hizalanmış olacaktır çünkü ancak bu suboptimaldir. Tasarım alanı ve deneyden elde edilen bilgilerin dolayısıyla derecesi kapsamıdüşük, Şekil 1A, 12 'de gösterildiği gibi. Şekil 1B 13 gösterildiği gibi Dahası, farklı faktörlerin (faktör etkileşimleri) arasında karşılıklı bağımlılık, kötü modeller ve / veya yanlış optima tahmin sonuçlanan gizli kalabilir.
Yukarıda tarif edilen dezavantajları birden fazla faktör iki çalışan 14 arasında değişmiştir olduğunu, yani bir denemenin çalışır tasarım alanı boyunca daha eşit bir şekilde dağılmış olduğu deneyde (DOE) yaklaşımı bir tasarım kullanılarak önlenebilir. Faktörler (faktöryel tasarımlar) ve yanıtları faktör etkilerinin kantitatif (yanıt yüzey yöntemleri, RSM s) 15 tarama karışımları için özel tasarımları vardır. Ayrıca, RSMS merkezi kompozit tasarım olarak fark edilebilir ama aynı zamanda ishal seçimi için farklı kriterler uygulayabilirsiniz özel yazılımı kullanılarak etkili bir şekilde gerçekleşebilir. Örneğin, bu şekilde adlandırılan D-optimality kriter IV-optimalite kriter tasarım alanı 15,16 boyunca düşük tahmin varyansını elde çalışır seçer oysa bu yüzden, elde edilen modelin katsayıları hatayı en aza indirmek için çalışır seçecektir. Biz burada açıklamak RSM bitkilerde geçici protein ifade hassas kantitatif verir, ancak kolayca (5-8 ~) çeşitli içeren herhangi bir sistem transfer edilebilir sayısal faktörler (örneğin sıcaklık, zaman, konsantrasyon) ve birkaç (~ 2 - 4) mekanik bir açıklama modeli kullanılamaz veya çok karmaşık kategorik faktörler (örneğin organizatörü, renkli) olan.
DoE yaklaşım tarımsal bilimler kökenli ama bu güvenilir veri elde etmek için gerekli çalışan sayısını azaltmak ve karmaşık işlemler için açıklayıcı modeller üretmek için yararlı olan herhangi bir duruma devredilemez çünkü diğer alanlara yayıldı. Bu da için "Rehberlik Doe dahil yol açmıştırSanayi, Beşeri İlaçların (İSK) 17 Ruhsatlandırma Teknik Gereksinimleri Uyumu Uluslararası Konferansı tarafından yayınlanan S8 (R2) İlaç Geliştirme ". DoE artık bilimsel araştırma ve sanayi 18 yaygın olarak kullanılmaktadır. Ancak, bakım sırasında alınmalıdır çoklu lineer regresyon modeli (baz model) için uygunsuz bir polinom derecesi seçerek çünkü planlama ve deney yürütme doğru tüm faktör etkilerini modellemek için ek çalışmalar için bir ihtiyaç tanıtabilirsiniz. Ayrıca, bozuk veya eksik veriler yanlış modeller ve kusurlu oluşturmak tahminler, ve hatta protokol ve tartışma bölümlerinde 18 anlatıldığı gibi herhangi bir model kurma girişimi engelleyebilir. protokol bölümünde, başlangıçta RSM-tabanlı bir deney için en önemli planlama adımları belirlemek ve daha sonra DOE dayalı tasarım açıklayacağız yazılım DesignExpert v8.1. Fakat benzer tasarımlar diğer yazılım includi ile inşa edilebilirng JMP, MODDE ve İSTATİKSEL. Deneysel prosedürler veri analizi ve değerlendirilmesi için talimatlar tarafından takip edilmektedir.
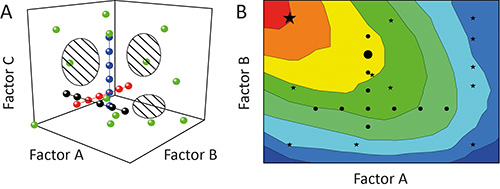
Şekil 1. OFAT ve DoE. A'nın karşılaştırılması. Bir deneyde (siyah, kırmızı ve mavi daireler) bir süre (OFAT) az bir faktör Sıralı varyasyon tasarım alanı (taralı bölgeler) düşük bir kapsama alanı sağlar. Buna karşılık, deney (DOE) stratejisi (yeşil daireler) tasarımını kullanarak aynı anda birden fazla faktörü varyasyon kapsamını ve elde edilen modeller. B böylece hassas artırır. Önyargılı tasarım alanı kapsama OFAT deneyler (siyah daireler) aynı zamanda, optimum işletme bölgeleri (kırmızı) belirlemek ve alt-optimal çözümleri (büyük siyah daire) tahmin başarısız demektir oysa DoE şires (siyah yıldız) tercih koşulları (büyük siyah yıldız) tanımlamak için daha olasıdır.
Access restricted. Please log in or start a trial to view this content.
Protokol
1.. Bir DoE Stratejisi Planlama
- Tasarım dahil ilgili faktörleri ve yanıtları belirlemek.
- Ölçüm için, bir ya da daha fazla yanıtları tanımlar. Burada, 2G12 ve DsRed ekspresyon düzeyleri, ilgili (sırası ile 10 ve 20 ug / ml) olarak en az bir fark tespit ve sistemin tahmin edilen standart sapma için yaklaşık bir değer (4 ve 8 ug de dahil olmak üzere, (ug / ml) kullanıldı / ml, sırasıyla) önceki deneylere dayalı.
- Mevcut literatürü kullanın, önceki deneylerde veya özel tarama tasarımları veri olan etkisi tepkiler üzerine sayısal olarak (Tablo 1) 7,8,19,20 önemli faktörler seçmek için (örneğin bir faktör tasarımı, giriş bölümüne bakınız).
- Faktör türlerini (sayısal veya kategorik) tahsis ve sayısal faktörler DoE soruşturma (Tablo 1) sırasında değiştirilebilir hangi aralıklar seçin.
- Numeri tespitc faktörler sürekli varyasyon uygulamak zordur.
- Belli bir seviyeye tam ayarlandı edilemez faktörleri sürekli varyasyon kullanmaktan kaçının. Örneğin İnkübasyon sıcaklığı genellikle sadece ± 2 ° C içinde kontrol edilebilir, örneğin 27.2 ° C, 25.9 ° C ve 29.3 ° C zorunluluk olarak değerlerini kullanarak, bu nedenle sürekli varyasyon kaçınılmalıdır.
- Bunun yerine, bu faktörlerin (Tablo 1) ayrık bir dizi düzeyi seçin. Seviyelerinin sayısı beklenen baz model (adım 1.2) ile eşleşmelidir. Merkezi kompozit tasarımları her zaman ayrı bir faktör seviyeleri var çünkü bu optimal tasarımlar için sadece önemli.
- , Bir kategorik faktör, nominal olup atama zımni hiçbir düzen (örneğin farklı üreticileri), veya sıra ve böylece ayrı bir sayısal parametre (bir bitki üzerinde örneğin farklı yapraklar) yani.
- Kategorik faktörlerin her iki tür için düzeylerini seçin.
- Yararlı bir temel modeli seçin.
- Ön deneyler ve literatüre dayanarak, her bir faktör ve tepki yanı sıra faktör etkileşimleri ve tepki arasındaki ilişkiyi tahmin. Örneğin, doğrusal (daha iyi), kuadratik (tek optimum veya doğrusal olmayan artış / azalış) veya küp (optimum çarpık).
- Ayrık sayısal faktörlerin seviyelerinin sayısı n faktörü ve tepki arasındaki ilişki polinom derecesi olmak üzere, n + 1 olduğundan emin olun. Örneğin, sıcaklık n = 2, tepkisi üzerinde bir ikinci dereceden bir etkiye sahip olması beklenmektedir ve bu nedenle en az üç sıcaklık seviyeleri kuadratik eden bir uyum elde etmek için araştırılmalıdır.
- Tahmin varyans belli bir eşiğin (alfa düzeyi) altında olması gereken tasarım alanı fraksiyonu (FDS) tanımlayın. FDS boyunca sağlam tahminler elde modellerini elde etmek için (tasarım alanının% 95'inden fazlasını kapsamaktadır)> 0.95 olması gerekirTüm tasarım uzay 21-23.
Not: 0.05 eşik% 5 anlamlılık düzeyinde (tip I hata) karşılık gelen ve tipik bir değerdir. Bu değeri artan bir DoE stratejisi için gerekli deney sayısını azaltacak ama aynı zamanda önemli bir etkisi cevapsız ve yanıtı üzerindeki etkileri tahminleri yanlış olacağını olacağı olasılığını artıracaktır.

Tablo 1. Varyasyon dahil tütün geçici protein ifadesini etkileyen faktörler kalın DoE. Faktörler sadece "farklı promotör / 5'UTRs kullanarak geçici ifadesi sırasında DsRed birikimi için bir tanımlayıcı model" altında açıklanan deneyler için tasarım dahil edildi sırasında aralıkları italik faktörler Sadece "Kuluçka optimize için tasarım dahil edildin koşulları ve "geçici ifade kullanılarak bitkilerde monoklonal antikorların üretimi için hasat planları.
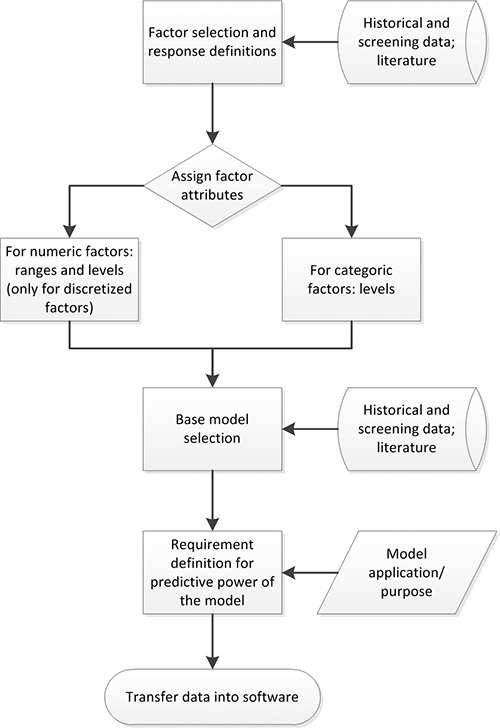
Şekil 2. DoE planlama süreci. Soruşturma kapsamında cevabında önemli bir etkisi olan faktörler mevcut verilere göre seçilir. Sonra faktör nitelikleri (örneğin sayısal), aralıkları ve düzeyleri atanır. Önceki bilgi ve deneyler uygun bir baz modeli tanımlamak için kullanılır. Akıllı güç gereksinimleri son model uygulama / amacına göre tanımlanır. Derlenen veriler daha sonra uygun DoE yazılım aktarılabilir.
2. DesignExpert bir RSM kurma
- DesignExpert başlatın ve "Yeni Tasarım" seçeneğini seçin. "Response Surface" penceresinde seçin ve# 34; Optimal "ve bölüm 1.1 'de seçilen sayısal ve kategorik faktörlerin) sayısını girin.
- Ilgili alanlara tüm faktörler için faktör adları, birimleri, türleri, alt türleri, düzeyleri / seviye sınırları ve düzeyleri için değerlerin sayısını girin.
- Sonraki sayfaya ve altında devam "Ara" seçenekleri "iyi" yanı sıra istenen "Optimalite" kriteri, burada "D-optimal" seçeneğini seçin.
- "Edit modeli ..." menüsünde, faktörler ve 1.2 bölümünde beklenen etkileşimleri) içeren modelini seçin. Emin değilseniz, burada belli bir dereceden için tüm faktörleri ve etkileşimleri, "kuadratik" kapsayan tam bir model seçin. (Tüm etkileşimleri içeren) tam bir modeli seçerek DOE için gerekli deney sayısını artırmak unutmayın.
- "Bloklar" sayısını seçin. Tüm bitkiler aynı partiden, bütün bakteriler aynı anda kültüre ve olmuştu çünkü burada, bir tek blok kullanıldıTüm enjeksiyonlar aynı operatör tarafından aynı gün yapılmıştır. Bitki birden fazla grup veya kullanılmış çeşitli operatörler Enjeksiyonu ise birden fazla blok kullanın.
- Tasarım Denge
- , Kategorik faktör düzeyleri arasında eşit deney çalışır dağıtmak amacıyla test edilir örneğin farklı destekleyicileri "kategorik denge zorla" etkinleştirin.
Not: Bu DesignExpert eniyilik algoritması tarafından seçilen noktalara bağlı olarak bir% değer olarak rapor edecek bir dereceye kadar tasarım optimalitesi azaltabilir. - Optimallık azalma en aza indirmek için uygulanan rastgele tohum algoritmasını kullanarak bir tasarımla birkaç kez yeniden hesaplayın.
Not: Optimallık kayıpları değişebilir 3-40% aynı giriş verilerini kullanarak, ama çoğaltır sayısı kategorik dengesini korumak için kullanılan değişen ~ dan.
- , Kategorik faktör düzeyleri arasında eşit deney çalışır dağıtmak amacıyla test edilir örneğin farklı destekleyicileri "kategorik denge zorla" etkinleştirin.
- Yazılım baz modeline dayalı "Model noktaları" sayısı için bir değer önerecektirBurada 70 çalışır. Burada her biri için "Model noktaları", 10 çalıştıran her bir sayısının>% 5 sağlamak için "uyum eksikliği tahmin etmek", "çoğaltır" ve sayısıdikkate ayarlayın.
- Yanıtların sayısını seçin ve isimlerini ve birimleri girin, bir sonraki sayfaya devam edin. Ek tepkileri de tasarım üzerinde herhangi bir olumsuz etkisi olmadan veri değerlendirme sırasında herhangi bir anda eklenebilir.
- DOE ve ishal için faktör düzeyleri hesaplanırken, algoritma başlatmak için Devam. Seçilen baz model belirli bir faktör için düzey sayısını uymuyorsa, bir bildirim görüntülenir ve hesaplama başlamaz. Bu durumda, ya da:
- Bu faktörü düzeylerinin sayısını artırın veya
- Düzeylerinin mevcut sayısına göre hesaplanır olamaz baz modelinde terimleri kaldırın.
- Örneğin, t faktörü "Kuluçka süresi" t (yani 2 d ve 5 d) için iki düzeyi varsao mevcut tasarım ve 2. seçilen terim t dahil karesel bir baz model, t üçüncü derecede (yani 8 d) eklemek veya modelinden faktör t 2 çıkarın ya.
- Hesaplama tamamlandıktan sonra görüntülenen optimal faktör kombinasyonlarını içeren tabloyu kaydedin.
- "Tasarım" düğüm "Değerlendirme" alt düğümünü seçin ve "Grafikler" sekmesine gidin.
- "Grafikler aracı" "FDS" seçin ve "FDS Grafik" kutusuna hata türü olarak "beklenen" seçin. Daha sonra, sırasıyla "d" ve "s" değerleri için DsRed için (burada 20 ve 8 ug / ml) halinde Kısım 1.1.1 'de tanımlanan sistemin tahmin edilen standart sapma için minimum bir fark tespit gibi yaklaşık bir değer) girin. Aynı zamanda uygulama için uygun alfa seviyesi (% olarak kabul edilebilir bir önemli bir etkiye eksik), tipik olarak 0.05 (% 5) girin.
- ) Hesaplanan FDS 1.3 bölümünde tanımlanan yüzdesini eşleşen emin olun (prediktif modeller için genellikle> 0.95). FDS arsa düz eğri tasarım alanı boyunca üniform öngörü doğruluğunu gösteren, tercih edilir.
- Bu durumda (burada bulduğumuz gibi Şekil 3A'da gösterildiği gibi, FDS% 1 idi) değilse, "geri" Tasarım "düğümüne gidin ve görev çubuğunda" Design Tools "seçeneğini seçin, ardından" Augment Tasarımı ... "ve seçim "Augment.
- Daha önce olduğu gibi aynı "Ara" ve "Optimalite" ölçütleri seçin. Ayrıca aynı "Edit modeli" ve "kategorik denge zorla" ayarlarını seçti. Tasarım güçlendirme (burada olduğu gibi) herhangi bir deneyden önce yapılır, sonra değiştirin "Block1" ayarı "bloğu içine çalışır koyun".
- "Çalışır" bölümünde eksikliğini tahmin etmek için en az% 5 "çoğaltır" ve "korumak, ek" Model noktaları "numarasını girinve uygun "toplam tasarım. Burada 100 ek çalışır ilave edildi ve hiçbir" çoğaltır "ve" uyum eksikliği tahmin etmek için "alındı.
- Hesaplama bittikten sonra, yukarıda tarif edildiği gibi yeniden gözden FDS grafik. FDS hala tatmin edici değilse, yukarıda tarif edildiği gibi, tasarım büyütme tekrarlayın. İşte FDS büyütme operasyonu sonrasında% 100 idi ve böylece daha fazla büyütme (Şekil 3B) ihtiyaç vardı.

FDS araziler 3. Karşılaştırılması Şekil. 90 çalışan oluşan bir. Bir DoE minimum fark tespit (20 ug / ml) ve Esti için değerler ile birlikte ikinci dereceden bir baz modelini kullanarak, tahmin standart hata için sadece% 1 arasında yetersiz bir FDS üretirSistemin (8 ug / ml). B standart sapması şeklindeki. 210 çalışan toplam DOE ve Büyütme tasarım alanı boyunca modelin üniforma hassasiyet gösteren% 100 FDS ve düz bir eğri elde etti.
3. Klonlama ve ifade analizi kasetler
- 5 ml LB ortamı içinde Escherichia coli yetiştirmek (10 g / L tripton, 5 g / L maya ekstresi, 170 mM NaCl, 50 mg / L ampisilin, LB-agar plakaları 15 g / L ağar dahil için) 6 için 37 ° C'de 160 rpm'de bir orbital çalkalayıcı üzerinde -8 saat veya gece boyunca karıştırılmıştır. DİKKAT: ampisilin zararlı bir maddedir. Bir pipet kullanarak plakaları üzerinde büyüyen koloniler sıvı kültür veya transfer hücre 50 ul ya da LB ortamı ile inoküle.
- PSO plazmid DNA saflaştırılması ve E. ppam (GenBank AY027531) 'in diğer türevleri için, E. coli K12 DH5a'ya, DNA saflaştırma kiti kılavuzunda 24 yönergeleri izleyin. DİKKAT: saflaştırma ko (ayrıntılar için kullanım kılavuzuna bakınız yukarıda) zararlı kimyasallar içerir. Plazmid dizisi Şekil 4 ve alıcı et al. 20 tarafından açıklanmıştır.
- NanoDrop cihazda 260 nm 'de bir 2 ul numunenin absorbansı ölçerek, ayrılmış malzeme içinde saflaştırılmış DNA konsantrasyonunun belirlenmesi.
- Kısıtlama endonükleaz (S) ile sindirme ile saflaştırılmış plazmid DNA kimliğini teyit edin.
- Benzersiz ve ayırt fragmanı desenler oluşturulur böylece plazmid sırasına göre res seçin. Bu tür tepkime hacminde, zaman, sıcaklık ve stabilizatör sığır serum albümin konsantrasyonu olarak sindirim koşulları için imalatçının tevsiyelerini izleyin. Hassasiyetine bağlı olarak, analiz aygıtı, sindirim başına plasmid DNA, 50-500 ng kullanın.
- Tris-borat-EDTA (TBE) tamponu (, pH 8.0 90 mM Tris, 90 mM borat, 2 mM EDTA) içinde agaroz kaynatılarak DNA fragmanlarının ayrılması için% 0.8-2.0 jeller hazırlayın. To büyük beklenen fragmanları, daha az agaroz jeli içinde kullanılmalıdır.
- 50 ul beş kat numune tampon maddesi 5 ul (5x SABU,% 0.1 (ağırlık / hacim) bromofenol mavisi,% 0.1 (w / v) Ksilen siyanol,% 10 (TBE içinde eritildi ağırlık / hacim) gliserol) ekleme DNA örneği yeniden işlenmiş ve ~ 40 dakika ya da fragmanların ayrılması berrak kadar elde edilir boyunca 100 V'de, agaroz jel elektroforezi ile parçalarını ayırmak. Her jel üzerinde, karşılaştırma için DNA merdiveni boyutu işaretleri içeren bir şerit, örneğin 2-3 ul 1-kb merdiven bulunmaktadır.
- Diğer üç 5'UTRs biriyle BDO'da omega 5'UTR değiştirin.
- ~ 60 dakika boyunca 37 ° C'de 20 Eco RI-HF birimleri ve NEBuffer 4 içinde Nco I-REs HF ile işlenerek-4 ug saflaştırılmış PSO'nun DNA'dan omega 5 'UTR sekansını bırakın. Bölümler 3.4.2 ve 3.4.3 'de tarif edildiği gibi O parçalarını ayırmak.
- İlave bir jel kullanılarak Agaroz jelden daha büyük olan parçası (pS "omurga") izoleüretici kılavuzu 25 ve bölüm 3.3 'de tarif edildiği gibi, saflaştırılmış DNA'nın konsantrasyonunu belirlemek) göre kit şeklinde gösterilmektedir. DİKKAT: jel ekstraksiyon kiti zararlı kimyasallar içerir, ayrıntılar için kullanım kılavuzuna bakın.
- ~ 60 dakika boyunca 37 ° C'de 20 Eco RI-HF birimleri ve NEBuffer 4 içinde Nco I-HF REs her vektörün ~ 10 ug muamele edilmesiyle uygun bir verici vektörlerden CHS, CHS-LPH ve TL 5'UTRs izole edin. Daha sonra bölümler 3.4.2 ve 3.4.3 'de tarif edildiği gibi parçalarını ayırmak ve bölüm 3.5.2' de tarif edildiği gibi daha küçük 5 'UTR fragmanı-ihtiva-eden saflaştırılması.
- Üreticinin tavsiyelerine göre 26, 5 dakika boyunca 25 ° C 'de izole edilmiş, saflaştırılmış doğrusal pS vektörüne ayrı ayrı parçalar halinde izole edilmiş, saflaştırılmış 5'UTRs Arter. ~ 50 ng vektör DNA ve 20 ul bir toplam hacim içinde 5 'UTR DNA üç kat molar miktarda kullanılmalıdır.
- Th ile dönüştürülmesi E coli hücrelerie yeniden birleştirici plazmitler, 26,27.
- Ekle ~ 10 ng ligasyon karışımı (~ 4-5 ul) (Bölüm 3.5.4) 50 ul rbcL yetkili E. coli ve hafifçe karıştırın ve daha sonra buz üzerinde 30-60 dakika boyunca inkübe edin. 5-30 dakika boyunca buz üzerinde 42 ° C ve soğuk 1.5 dakika boyunca ısı şoku.
- Antibiyotik içermeyen LB ortamı içinde 950 ul ilave edin ve 37 ° C'de 1 saat ve 160 rpm'de inkübe edilir. Transformantların seçimi için amfisilin içeren LB agar levhaları üzerinde kültür içerisinde 50 ile 100 ul yayılmış ve 37 ° C de ~ 16-20 saat boyunca inkübe
- Ampisilin ihtiva eden LB ortamı içinde 5 ml'lik alikota Bölüm 3.1 'de tarif edilen her bir promotör-5' UTR bağlanmasını temsil eden 5-10 ayrı koloniler inoküle. Daha sonra plazmid DNA saflaştırılması ve bölümlere 3,2-3,4 tarif edildiği gibi kimliğini teyit etmektedir.
- NEBuff in Asc I ve Eco RI Res kullanan dört 5'UTR plazmidlerin her nos promoter ile 35SS promotörünün yerinier bölümler 3.5 ve 3.6 'da 1 saat boyunca 37 ° C' de tarif edildiği gibi 4.
- A. içine sekiz edilen plazmidlerin her biri tanıtılması elektroporasyon 28 tarafından pMP90RK: GV3101 ırkına tumefaciens.
- 50 ul yetkin A'ya saflaştırılmış plazmid DNA ~ 500 ng ekleme buz üzerinde hücreleri Tumefaciens. Yavaşça karıştırın ve önceden soğutulmuş bir 0.2 cm elektroporasyon küvetine içine aktarın. Karışım, küvet altındaki ve kabarcıkları içermeyen emin olun.
- Pulse hücreler 5 msn 2.5 kV ve nabız yoğunluğunu ve süresini onaylayın.
- DNA numunesi ya da yetkili A yanlış hazırlanmasında, yüksek tuz konsantrasyonlarını önlemek Bu dönüşüm etkinliğini büyük ölçüde azaltarak, hücre süspansiyonu anlık buharlaşmasını neden olan yüksek iyon akımları neden olabilir, çünkü hücreler tumefaciens.
- Antibiyotikler olmadan YEB ortamının 950 ul (5 g / L sığır eti ekstresi, 1 g / L maya ekstresi, 5 g / L pepton eklemee, 5 g / L sukroz, 2 mM MgSO 4, pH 7.0), yavaşça karıştırın ve steril bir 1.5 ml reaksiyon tüpü içine hemen transfer. 26-28 ° C ve 160 rpm'de 2-4 saat boyunca inkübe edin.
- (50 mg / L ihtiva eden antibiyotik YEB agar plakaları üzerine 1-2 ul yayılmış transformantların seçimi için karbenisilin, 25 mg / L kanamisin, 25 mg / L rifampisin). DİKKAT: rifampisin toksik bir maddedir.
- Her bir promotör-5 'UTR bağlanmasını temsil eden ayrı koloniden hücrelerle antibiyotik ihtiva eden ortam içinde üç YEB 5 ml'lik numuneler aşılamak ve 26-28 ° C ve 160 rpm'de 48-72 saat boyunca inkübe edilir.
- A. başarısını teyit tumefaciens dönüşüm.
- Aktarım bölüm 3.8.6, her bir alikot 2 ul PCR master 48 ul alikotları (her biri 10 uM primer stoklar 2 ul (her bir primerden 400 nM nihai konsantrasyon), ayırmak1 ul 10 mM dNTP karışımı (her biri deoksinükleosit trifosfattan 200 uM son konsantrasyon) 5 ul 10 misli 0.75 ul), High Fidelity enzim karışımı aç ve 39,25 ul steril damıtılmış su, 15 mM MgCl2 ile High Fidelity tampon genişlet.
- Uygun primerler kullanılarak her bir plazmid ekspresyon kaseti (FWD yükseltin: 5'-CCT CAG GAA CAA GAG TAC-3 ', promoterden 1026 nükleotidini bağlanması; REV: 5'-CCA AAG CGA AAC CAC GTA-3', bağlayıcı PCR, uygun koşullar altında), 35S poliadenilasyon sitesi içinde. Burada, 94 ° C, 15 saniye boyunca 94 ° C 30 döngü denatürasyon, ardından başlangıç denatürasyon için kullanıldı, 30 saniye ve 120 saniye için 72 ° C uzatma için 51 ° C tavlama, ve 8 için 72 ° C'de son uzatma adımı min.
- Bölüm 3.4.3 'de tarif edildiği gibi agaroz jel elektroforezi ile PCR ürünlerinin boyutunu belirler.
- A. hazırlayın tarafından gliserol stokları tumefaciensA. 500 ul ile 500 ul% 50 karıştırma (v / v) steril gliserol dönüşüm (bölüm 3.9.3) teyit edildiği için bölüm 3.8.6 den kültürleri Tumefaciens. Bundan başka, kullanılıncaya kadar -80 ° C'de gliserol stokları saklayın.
- RNAfold Web sunucusu 29 ile farklı mRNA'ların katlama enerjisini hesaplamak ve kodlama bölgesinin ilk 50 bp kadar transkripsiyon başlangıç bölgesinden oluşan bir nükleotid sekansı içerir.
- "Algoritmalar ve temel seçenekleri Fold" bölümünde "minimum serbest enerji (MFE) ve bölüm işlevi" ve seçenekleri "izole baz çifti kaçının" seçeneğini seçin.
- "Gelişmiş katlama seçenekleri" "her durumda bir sarmalın her iki tarafında enerjilerini sarkan" seçin, "RNA parametreleri (Turner modeli, 2004)" ve 25 ° C "belirli bir sıcaklıkta (C) enerji parametrelerini rescale" değiştirmek
- "Çıkış seçenekleri" bölümündeki tüm seçenekleri seçin. karşılaştırma değerleri "topluluk içinde MFE yapının frekans" "termodinamik topluluk serbest enerji" ve ayıklayın.
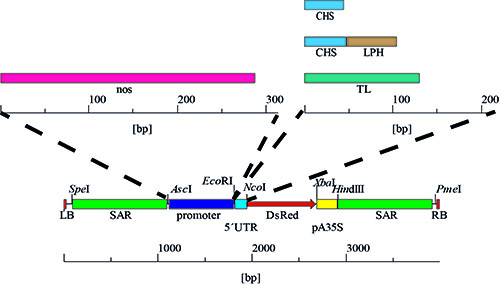
Şekil 4,. Promoter ve 5 'UTR Ekspresyon kasetleri nos dizisi dört ek varyantları elde ve bu hızlandırıcının yerine ardından CaMV promoteri 35SS ile dört kombinasyon ile sonuçlanan, 5'UTR aşamalı değişimi ile elde edilmiştir. Varyantları Sekiz farklı promotör / 5 'UTR kombinasyonları toplam.
4. Bitki Yetiştirme
- Gübrenin% 0,1 'lik çözelti hazırlayın FertyPH 5.9 'de deiyonize su içinde 2 Mega.
- Kalıntı kimyasal kaldırmak ve son olarak suni gübre ile denge sağlaması için deiyonize su ile aşırı yıkama ile 10 x 10 x 8 cm kaya yünü blok hazırlayın.
- Gübre uzak tohumlarını yıkama önlemek için dikkatli olmak kısa bir floş ardından her taşyünü blokta 1-2 tütün tohum koyarak tohum tütün bitkileri.
- Çimlenme ve 25/22 ° C gündüz / gece sıcaklık,% 70 nispi nem ve 16 saatlik aydınlık periyoduyla bir serada 42 gün için tütün bitkileri yetiştirmek (180 mmol sek -1 m -2, λ = 400-700 nm) . Bu fotoperiyodunda sırasında, topraksız kültürde 15 dakika her saat için gübre ile sulamak.
5. Geçici Protein Expression
- A. hazırlayın yapraklar içine enjeksiyon için tumefaciens.
- % 1 A ile antibiyotik ihtiva eden ortam YEB 5-50 ml inoküle Cryo-stok kültür Tumefaciens ve OD 600nm 5.0 (~ 48-72 saat hacmi ve damar türüne bağlı olarak) ulaşır.
- A. seyreltin Su ve enjeksiyon için gerekli OD 600Nm maç için 2 kat infiltrasyon ortamı (4.3 g / L, Murashige ve Skoog tuzları (pH 5.6), 5 g / L sükroz, 1.8 g / L glukoz, 100 mM asetosiringon) ile tumefaciens kültürü. Hemen enjeksiyondan önce OD 600Nm onaylayın. Not: ~ 1.43 ± 0.12 x 10 9 koloni oluşturan birim ml başına 1.0 karşılık gelir OD 600Nm.
- A. enjekte yapraklara süspansiyon tumefaciens.
- Seçin ve (örneğin DoE stratejiye göre) tedavi edilecek bunun noncotyledon yaprakları ve pozisyonları etiket.
- Farklı A. enjekte etmeyin Aynı interkostal alana çözüm tumefaciens. Bunun yerine, orta damar ekseninin her iki tarafında karşılıklı bölümleri kullanın. Ikiden fazla farklı çözümler aynı pozisyonda enjekte edilmesi gerekiyorsa bir additiona kullanımıl bitki.
- İyice A. sallamak 1-ml'lik şırınga içine seyreltme ve aspire hazırlanıyor beri yerleşmiş olabilecek hücrelerin tekrar süspansiyon tumafaciens çözüm.
- Yavaşça A. akışını kolaylaştırmak için benzer bir pipet ya da enjeksiyon için amaçlanan konumunda epidermis çizik çözüm Tumefaciens. Bunu yaparken yaprak bıçak kırılması kaçının.
- Muamele edilir ve yavaşça, yaprağın alt tarafı üzerine (ekteki herhangi bir iğne ile birlikte) çıkış itmek üzere interkostal alana karşı namlusuna temas yaprak bıçak dik şırıngayı tutun. Kayması veya çatlaması gelen yaprak bıçak önlemek için yavaşça aynı anda yaprak üst tarafı basın.
- Yavaşça şırınga pistonu aşağı doğru itin. A. koyu yeşil ve nemli görünen tedavi alanlarda belirtildiği gibi turnefaciens çözüm yaprak bıçak içindeki hücrelerarası boşluk girecek. Bütün intercosta kadar çeşitli pozisyonlarda bu işlemi tekrarlayınl alan A ile sızmış tumafaciens. Ardından bir sonraki interkostal alanı ile devam ediyor.
- Emin şırınga yaprak dik kalır olun. Şırıngayı Yatırma bakteriyel süspansiyon, yüksek basınç altında hamle neden olur.
- Eğer farklı A. tumefaciens çözeltiler (örneğin, farklı promotörleri test etmek için) kullanılır, sonraki çözümü uygulamadan önce benzer bir kağıt havlu ya da kullanarak ilk enjeksiyondan yaprağın alt tarafı üzerinde kalan herhangi bir fazla uzaklaştırın.
- Bitki ve örnekleme sonrası infiltrasyonu inkübasyon.
- Muamele edilen bitkiler için Phytotron'da hazırlanması ~ DOE için gerekli olan seviyelerde sıcaklık ve nem dengelenmesini sağlamak için enjeksiyondan önce 24 saat.
- Enjeksiyondan sonra, Phytotron'da içine bitkiler aktarmak ve DOE belirlenir Kuluçka süresinin sonuna kadar su ile sulama için yeterince büyük bir tepsi içine yerleştirin.
- 16 saatlik bir phot kurmakoperiod 0,7 m 2 başına altı Osram soğuk beyaz 36 W floresan tüpler kullanılarak (75 mmol sek -1 m -2; λ = 400-700 nm). 0.7 m 2 başına altı bitki sayısını sınırlayarak birbirinden gölgeleme gelen bitkiler önleyin.
- Numune almadan önce, doğru interkostal alan 5.2.1 ve DoE planında eklenen etiketi karşılaştırarak seçildi emin olun.
- DOE tarafından belirtilen konumlarda ve zaman zaman tedavi interkostal alanlardan 4-5 yaprak diskleri kaldırmak için bir mantar delici kullanın. Örnekleme sırasında bitkinin bütün yaprak çıkarmayın. Rüptürü önlemek için diskler çıkarırken bir el kağıt havlu ile yaprak stabilize.
- Her numunenin kütlesinin belirlenmesi ve numune adı ve kütle ile etiketlenmiş 1.5 ml'lik plastik bir reaksiyon tüpü içine koyun. Protein miktarının daha önce -20 ° C veya -80 ° C 'de örnekleri saklayın. Işlem numune istikrar bağlı olarak birkaç ay boyunca bu aşamada durdurulmuş olabilirve depolama sıcaklığı.
6. Protein miktar tayini
- Yaprak disk örneklerinden ekstrakte proteinleri.
- Numune kütlesinin mg başına ve hiçbir büyük fragmanları kalmayıncaya kadar bir elektrik havan tokmağı kullanılarak reaksiyon tüpünde yaprak diskleri öğütmek, (pH 8.0 50 mM sodyum fosfat, 500 mM sodyum klorid) ekstraksiyon tamponu 3 ml ekle. Örnek aşırı ısınmasını önlemek.
- 4 ° C'de 20 dakika boyunca 16,000 x g'de iki numune santrifüj ile dağılmış katı maddeler uzaklaştırılmış Pelet bozmadan her adımdan sonra temiz bir 1.5 ml reaksiyon tüpüne süpernatantı aktarın.
- Santrifüj işleminden sonra, işlem tesis, örnek stabilite ve depolama sıcaklığına bağlı olarak, birkaç ay boyunca -20 ° C veya -80 ° C'de dondurularak özler durdurulmuş olabilir. , Bir dondurma-çözme döngüsü, hedef protein (ler) in konsantrasyonu etkilemez emin olun.
- DsRed floresan ölçün.
- 96 oyuklu siyah yarı alanlı plakalar (kuyu başına 50 ul ekstre) her bir örnek üç teknik çoğaltır hazırlayın. Pipetleme sırasında kabarcıkların oluşumunu önlemek.
- Bir astar referans eğri oluşturmak için 96 oyuklu plaka başına DsRed bir standart altı seyreltilerde (0, 25, 75, 125, 175 ve 225 ug / ml) bir dizi kullanın. PBS içinde dilüe ve 3 ay içinde kullanım için 4 ° C'de saklayın.
- 530/25 nm uyarım ve 590/35 nm emisyon filtreleri ile donatılmış bir 96 çukurlu levha okuyucusuna yerleştirilir, ardışık iki floresan ölçün.
- Her numune için, üzerinde iki floresan okur ve üç teknik çoğaltır ortalama ve 0 ug / ml ihtiva eden DsRed boş kontrolü için kaydedilen değer çıkarın. Ayrıca standart dilüsyonlarının okur bu değerin çıkartılması ve (ordinatlarının kökeni ile) bir referans eğrisi veren bir lineer regresyon için bu boş-düzeltilmiş değerleri kullanın.
- F dönüştürmek için referans eğrisinin eğimini kullanınDsRed konsantrasyonları içine numune için ölçülen luorescence. Gerekirse, okuma standartların konsantrasyonu aralığı içinde kalan ve böylece örnekleri sulandırmak ve sonraki hesaplamalarda bu seyreltme faktörü göz önünde bulundurun.
- 2G12 konsantrasyonunu belirleyin.
- Bir akış hücresinin aktive edilmiş protein A yüzeyine birleştirilmesi ile antikor konsantrasyonunun ölçümü için bir yüzey plasmon rezonans (SPR) cihazı hazırlayın. Protein A 30,31 bağlanması olmaksızın yüzey etkisiz hale getirerek bir referans olarak bir akış hücresi kullanın.
- Bitki SPR çalışan tamponu (10 mM HEPES pH 7.4, 3 mM EDTA, 150 mM NaCl,% 0.05 v / v Tween-20) içinde 1:20 oranında seyreltilir ve özü üç Protein A antikor bağlanması tepki ünitesi (RU) ölçülmesi 90 ul enjeksiyon (30 ul / dk 'da 180 saniye) sonunda, her numunenin teknik çoğaltır. Deney fl ölçülen RU gelen referans akış hücresi (no Protein A) ölçülen RU çıkarmaow hücre (Protein A yüzey).
- Her 10-15 örnekleri sonra, 585 ng / ml 2G12 standart ölçün ve yukarıda tarif edildiği gibi, referans akış hücresi olarak ölçülen değerini çıkarın. Ordinatlarının kökeni ile doğrusal bir referans eğri hesaplamak için bu standartların ortalama RU kullanın.
- 1:20 seyreltme ve referans eğrisinin eğimine göre numunelerdeki 2G12 konsantrasyonları hesaplayın.
- Bozabilir ölçüm referans hücreye güçlü spesifik olmayan bağlanma için kontrol edin. Ayrıca daha fazla varyasyon Protein A yüzey yaşlanmasını yansıtan 2G12 standartları, analiz boyunca yaklaşık sabit değerleri (<% 5 değişkenlik) korumak olmadığını kontrol edin.
7. Veri Analizi ve Değerlendirme
- (Ii) sıra dışı sonuçların, örneğin faktör, kombinasyon geni; el ile (i) aşırı değerleri gösteren (yüksek veya düşük) ölçüm hataları için sentezleme deneylerinde gözlemlenen tepkiler analizveri değişimini gösteren beklenmeyen tepkiler oranı ve (iii) değerleri eksik.
Not: hatalı verilerine dayalı bir model bina engellemek için bu eylemleri kullanın. - DesignExpert ve "Tasarım" düğüm haline analiz yanıt verileri (burada protein konsantrasyonları) aktarın. Yanıtı verileri doğru gelen faktör ayarlarına atandığından emin olun. Yönleri aşağıda ele kapsar DesignExpert yazılım mevcuttur geniş bir yardım bölümü var.
- "Analiz" düğümünde, analiz edilecek yanıtı seçin ve başlangıçta dönüşüm sekmesinde "hiçbiri" seçeneğini seçin.
Not: bir transformasyon 10 daha büyük yanıtın dakika / max oranları için tavsiye edilir. En yararlı dönüşüm aşağıda açıklanan "Tanı aracı" (bölüm 7.8) ve "Tanı" bölümünde "Tanı" sekmesinde Box-Cox-grafiğinden elde edilebilir. Bir Log 10 dönüşüm genellikle uygun. - soruşturma kapsamında sisteme (örneğin iki-faktör etkileşimleri, kuadratik etkileri) için önemli olan faktörler hakkında genel bilgi sağlar "Fit özeti" sekmesine, devam edin. Yazılım önemi dayanan bir başlangıç modeli önerir.
- "Model" sekmesinden, bir başlangıç modeli "Fit özeti" sonuçlarına dayalı önceden seçilir. Bu modeli düzenlemek için otomatik modunu kullanın:
- Önerilen model "2FI" (iki faktör etkileşim) ise o zaman "kuadratik" seçmek için, bir düzen önerilen modelin üzerinde bir "Süreç düzeni" seçin, örn.
- Tekrarlanarak başlangıçta 0.100 olmalıdır "Alpha out" değere göre "ANOVA" sekmesine geçmeden sonra modelden anlamsız koşullarını kaldırmak hangi, "Seçim" alanında "Geri" seçiniz.
- Yazılımı tarafından sorulursa bu necessar, çünkü her zaman otomatik modeli hiyerarşiyi düzeltmekgüvenilir modeller üretmek 32,33 y.
- "ANOVA" sekmesinde, önerilen model ve dahil faktörleri araştırmak. Gerekirse, elle "Seçim" değiştirerek geri "Model" sekmesine geçerek (% 5 anlamlılık düzeyine karşılık gelen, burada 0.05), önceden tanımlanmış bir eşiğin üzerinde p-değerleri ile herhangi bir faktör kaldırmak veya olası mekanik dikkate dayalı olanlar "Kılavuz" ve modeli uygun faktörleri ortadan kaldırır.
- Geri "ANOVA" sekmesinde, "Model" (bu önemi gösterir çünkü düşük bir değer arzu edilir) ve "eksikliği-of-fit" (yüksek değeri p-değeri açısından tazelenmiş modeli değerlendirmek arzu edilir Bu anlamlılık yokluğu) hem de "R-kare" için değerlere işaret etmektedir, çünkü (> 0,80 değerleri üç değerleri için tercih edilir), "R-kare Arındırılmış" ve "R-kare tahmin".
- Fo bu değerleri karşılaştırmakçeşitli seviyelerinde faktörler hariç / dahil olmak üzere farklı modelleri r.
Not: "Tasarım" düğüm tepki sütun çoğaltmak ve ayrı ayrı her bir analiz yapmak, ya da böyle bir tablo gibi başka bir program içine bütün "ANOVA" tablo ihracat için yararlı olabilir.
- Fo bu değerleri karşılaştırmakçeşitli seviyelerinde faktörler hariç / dahil olmak üzere farklı modelleri r.
- Modelin kalitesini teyit ve "Tanı Aracı" tüm sekmeleri ("Tanı" ve "Etki" bölümü) inceleyerek model üzerinde güçlü bir etkiye sahip kümesi içinde potansiyel aykırı algılamak için "Tanı" sekmesine devam .
- "Tanı araç" "Tanı" bölümünde, aşağıdaki işlemleri gerçekleştirin:
- Emin noktaları rastgele olarak sınırları içinde dağınık grafiği "Artıklar vs Öngörülen". Burada, zikzak şeklinde bir dağıtım veri dönüşümü için bir ihtiyaç gösterir.
- Emin noktaları rastgele olarak sınırları içinde dağılmışGrafiği "Residuals vs Öngörülen". Burada, zikzak şeklinde bir dağıtım veri dönüşümü için bir ihtiyaç gösterir.
- Araştırmak "Residuals vadede vs" grafik dağılım noktaları rastgele sınırlar içinde olup olmadığını. Bir desen (örn. "merdiven") modeli üzerindeki nüfuzunu dengelemek için kullanılabilir veri ve blok binada bir eğilim gösterir.
- Komplo "Gerçek vs Öngörülen" in ideal bir model belirten düz bir diyagonal çizgi arayın. Çapraz çapında veri saçılma daha fazla, daha az hassas bir model.
- Veri dönüşümü belirten mavi (en iyi) ve yeşil Box-Cox-arsa (gerçek) dikey çizgiler bir örtüşme için kontrol edin. Yeşil hat (kırmızı dikey çizgilerle belirtildiği aralığı dışında) mavi uyuşmuyor ise arka bölüme 7.3 gidin ve Box-Cox-arsa önerdiği veri dönüşümü seçerek model kurma yuvarlak geliştirmek. Yine yeniden çoğaltılmasıyanıt alınamaması sütun farklı modeller karşılaştırmak yararlı olabilir.
- "Residuals Factor vs" listelerinde (her model faktör için bir grafik var) olarak puan sınırları içinde rasgele dağılım emin olun. Veri dağıtım eğrilik modeldeki eksik bir faktör olduğunu gösterir.
- "Tanı araç" "Etkisi" bölümünde, "Kaldıraç", "DFFITS" ve "DFBETAS" araziler ve eşit düşük dağıtım olmadan, emin "Dışarıdan studentized artıkların" de sınırlar dahilinde verilerin rastgele saçılması var olun "Aşçı Mesafe" arsa aşırı değerleri.
- "Tanı araç" "Tanı" bölümünde, aşağıdaki işlemleri gerçekleştirin:
- "Model grafikleri" sekmesinde, değerlendirildi modeli görselleştirmek. Sayısal faktörlerin sınırlı sayıda (örneğin 3) yüzey tepki ("3D yüzey") temsil elle optima / özelliklerini değerlendirmek için yararlıdır.
Not: yanıt yüzeyleri sadece iki fac etkisini göstermektedirsoruşturma kapsamında yanıtı üzerine monte edin. Yanıtta herhangi bir ek faktörün etkisi "Faktörler aracı" penceresinde değerini (sayısal faktörler) veya seviye (kategorik faktörler) değiştirerek ortaya çıkar. Alternatif olarak, faktörler "Faktörler aracı" penceresinde sağ tıklayarak ve istenen bağımsız değişken ekseni seçerek arsa eksenine atanabilir.- Faktör düzeyleri işlemek ve "Faktörler aracı" kullanarak grafik koordinatIara onları atayın.
- "Dosya" sekmesinde "komutunu ... Dosyaya Aktar grafiği" kullanarak ihracat grafikleri.
- Yoluyla (örneğin sınırları ve ağırlıkları) da bazı kısıtlamalar uygulanabilir hangi model faktörlere bağlı olarak (aralık, maksimize, minimize) sayısal tepki optimize etmek "Optimizasyon" düğüm "Sayısal" alt düğümü kullanın "Kriterler" sekmesi.
- "Çözümler" hesaplamak ve sayısal çözümleri incelemek"Kriterler" sekmesinde sağlanan girişine göre sekme.
- Yüksek veya düşük tepki değerleri ile ilişkili faktör ayarlarını açığa ileri analiz (örn. histogramlar) için diğer yazılım (örneğin elektronik tablolar) için bu çözümleri ihracat.
Not: Birden fazla üç sayısal faktörler incelendi ve 3D gösterimi zordur eğer bu yararlıdır.
- Özel faktör ayarları için yanıtı tahmin etmek "Nokta tahmin" alt düğümü kullanın.
- Tüm ayarları (burada tüm promotör ve 5'UTR kombinasyonları yanı sıra tüm yaprakları ve inkübasyon süreleri veya yaprak pozisyonları ve inkübasyon sıcaklıkları) değerlendirilmesi için bu tahmini kullanın.
- Tahmin edilen değerler verme ve belirli bir zamanda özel bir promotör-5 'UTR kombinasyonu için ifade hesaplanarak, örneğin tütün bitkilerinde geçici ifade tanımlamak için kullanılabilecek bir veri kümesinden, bunları hazırlanması tüm yaprak pozisyonlar ya da akro üzerinden ortalaması, bir bitkinin tüm yaprakları ss.
- Spesifik ekspresyon seviyesini (-1 ug g) ya da spesifik ifade oranı (mg g-1 hr-1) elde etmek için farklı yaprakların kitleler göre ifade verileri normalize.
- Alternatif olarak, bir elektronik tabloya "ANOVA" sekmesinin altındaki "Nihai denklemi" katsayılarını ihracat ve aynı veri dizisi elde etmek için faktör ayarları bir dizi ile çarpın.
Not: monte modeli denklem ekstrapolasyonuna için uygun değildir, çünkü bu dizi sadece ilk tasarım alanı içinde faktör değerleri içerebilir.
- Büyük ilgi modeli noktaları için bir onay deney yapmak.
- "Nokta tahmin" (bölüm 7.11) için seçilen şartlar altında geçici protein ifadesi gerçekleştirin. Örneğin aynı sıcaklıkları ve yaprakları vb kullanabilirsiniz
- Bu geçici ifade e protein konsantrasyonlarını belirlemekolarak (bölüm 6) Yukarıda tarif ve modelin öngördüğü değerler ile karşılaştırmak Xperiment.
- Onay deneyde ortalama protein konsantrasyonu nokta tahmini sırasında elde edilen yüzey tepki modelin tahmin aralığı içinde olup olmadığını kontrol edin. Bir maç modelin öngörü gücü doğruluyor. Bir uyuşmazlığı düşük modeli kaliteli ve ek çalışır gerekli olabilir gösterir.
- Not: Bitki parti-parti değişkenlik tahmin aralığı genişletir ve bitkilerin farklı bir parti ile yapılan deneyler onay devalüe olabilir. Bununla birlikte, bir model oluşturmak için kullanılır ama örnekler modelde içerdiği gibi aynı partiden bitki kökenli değildi örnekler olarak etkili kontrol hizmet edebilir.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Farklı destekleyiciler ve 5'UTRs kullanarak geçici ifadesi sırasında DsRed birikimi için bir açıklayıcı bir model
Yaprak ekstreleri içinde DsRed floresan rekombinant protein ekspresyon seviyesini gösterir ve bu nedenle DoE stratejisinde tepki olarak kullanılmıştır kullanılmıştır. Alakalı olarak en az algılanabilir bir fark 20 ug / ml oldu ve sistemin tahmin edilen standart sapma ilk deneylere göre 8 ug / ml idi. Geçici ifade modele dahil faktörler...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Kaynaklar genellikle kıt ve pahalı olduğu için her deneme dikkatli bir planlama gerektirir. Planlama aşamasında (örneğin tüm önemli faktör etkileşimleri kapsamaz bir baz model seçerek) esnasında hataları büyük ölçüde çıkan modellerin tahmin gücünü azaltmak ve böylece tüm deney devalüasyon olabilir, çünkü bu DoE stratejileri için özellikle doğrudur. Ancak, bu hataları kolayca temel prosedürleri takip ederek önlenebilir.
DoE planlama sırasında Husus...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yayın ücreti kısmen el yazması veya içeriğinin herhangi bir sorumlu hazırlanmasında dahil rol almayan şirketlerin Statease, Inc (ABD) ve Statcon (Almanya), sponsor oldu.
Teşekkürler
Yazarlar bu çalışmada kullanılan tütün bitkileri yetiştirilmesi için ppam bitki ekspresyon vektörü ve İbrahim Al Amed sağlamak için Dr Thomas Rademacher minnettarız. Biz el yazması düzenleme ile yaptığı yardım için Dr Richard M. Twyman teşekkür etmek istiyorum. Bu çalışma kısmen Avrupa Araştırma Konseyi İleri Hibe "Future-Pharma" tarafından finanse edildi, öneri sayısı 269.110 ve Fraunhofer Zukunftsstiftung (Fraunhofer Gelecek Vakfı).
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Referanslar
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. Design and Analysis of Experiments. , John Wiley & Sons Inc. (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , Wiley. (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- FDA. , U.S. Department of Health and Human Services. Rockville, MD, USA. (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. DOE Simplified: Practical Tools for Effective Experimentation. , Productivity Inc. (2000).
- Anderson, M. J., Whitcomb, P. J. Response Surface Methods Simplified. , Productivity Press. (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- Plasmid DNA purification User manual. , MACHEREY-NAGEL. Düren. (2012).
- PCR clean-up Gel extraction User manual. , MACHEREY-NAGEL. Düren. (2012).
- Quick Ligation Protocol. 4, NewEnglandBiolabs. Ipswich. (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır