Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Charakterisierung komplexer Systeme Mit dem Design of Experiments Ansatz: Transient Protein Expression in Tabak als Case Study
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Anordnung von Experimenten Ansatz, der verwendet werden kann, um festzustellen und das Modell des Einflusses der Transregulationselemente, Pflanzenwachstum und Entwicklung Parameter und Inkubationsbedingungen für die vorübergehende Expression von monoklonalen Antikörpern und Reporter-Proteine in Pflanzen auf.
Zusammenfassung
Pflanzen bieten mehrere Vorteile für die Produktion von Biopharmazeutika mit niedrigen Kosten, Skalierbarkeit und Sicherheit. Die transiente Expression bietet den zusätzlichen Vorteil der kurzen Entwicklungs-und Produktionszeiten, sondern Expressionsniveaus erheblich zwischen den Chargen so was zu rechtlichen Bedenken im Rahmen der guten Herstellungspraxis variieren. Wir verwendeten eine Versuchsplanung (DOE) Ansatz, die Auswirkungen der wichtigsten Faktoren wie regulatorische Elemente in dem Expressionskonstrukt, das Pflanzenwachstum und Entwicklung Parametern und den Inkubationsbedingungen während der Expression, auf die Variabilität der Expression zwischen den Chargen zu bestimmen. Wir untersuchten Pflanzen ein Modell Anti-HIV-Antikörper (2G12) und einen fluoreszierenden Marker-Protein (DsRed) ausdrückt. Wir diskutieren die Gründe für die Auswahl bestimmter Eigenschaften des Modells zu identifizieren und ihr Potenzial Einschränkungen. Das allgemeine Konzept kann leicht auf andere Probleme übertragen werden, da die Prinzipien des Modells einwieder breit anwendbar: wissensbasierte Parameterauswahl, Komplexitätsreduktion durch Aufteilung der anfängliche Problem in kleinere Module, Software-geführte Installation der optimalen Experiment Kombinationen und schrittweise Design Augmentation. Daher ist die Methode nicht nur nützlich für die Charakterisierung der Proteinexpression in Pflanzen, sondern auch für die Untersuchung von anderen komplexen Systemen fehlt eine mechanistische Beschreibung. Die Vorhersagegleichungen zur Beschreibung der Interkonnektivität zwischen Parameter können verwendet werden, um mechanistische Modelle für andere komplexe Systeme aufgebaut werden.
Einleitung
Die Produktion von biopharmazeutischen Proteinen in Pflanzen ist vorteilhaft, da Pflanzen kostengünstig zu wachsen, kann die Plattform sich nur durch den Anbau von Pflanzen mehr skaliert werden, und menschliche Krankheitserreger sind nicht in der Lage zu 1,2 replizieren. Transiente Expressionsstrategien zum Beispiel auf Basis der Infiltration von Blättern mit Agrobacterium tumefaciens bietet zusätzliche Vorteile, da die Zeit zwischen dem Punkt der DNA-Zufuhr und die Abgabe eines gereinigten Produktes von Jahren auf weniger als 2 Monate 3 reduziert. Transiente Expression ist auch für die funktionelle Analyse, z. B. verwendet werden, um Gene, die für ihre Fähigkeit zur Ergänzung Verlust-Funktion Mutanten oder Protein-Wechselwirkungen zu untersuchen 4-6 testen. Allerdings neigen transiente Expressionsniveaus größere Charge zu Charge Variation als Expressionsniveaus in transgenen Pflanzen 7-9 zeigen. Dies verringert die Wahrscheinlichkeit, dass biopharmazeutische Fertigungsprozesse basierend auf transiente Expression wiwerde im Rahmen der guten Herstellungspraxis (GMP) genehmigt werden, weil die Reproduzierbarkeit ist ein kritischer Qualitätsmerkmal und unterliegt Risikobewertung 10. Solche Variation können auch alle Interaktionen, die Forscher wollen untersuchen, zu maskieren. Deshalb machten wir uns auf die wichtigsten Faktoren, die transiente Expression in Pflanzen zu beeinflussen und eine hochwertige quantitative Vorhersagemodell zu bauen identifizieren.
Das Ein-Faktor-at-a-time (OFAT)-Ansatz wird oft verwendet, um die Auswirkungen (Wirkung) bestimmter Parameter (Faktoren) auf das Ergebnis (Antwort) eines Experiments 11 charakterisieren. Aber das ist nicht optimal, weil die einzelnen Tests (läuft) während einer Untersuchung (Experiment) werden wie Perlen auf einer Schnur durch die Potentialfläche durch die Faktoren, die getestet (design space) werden aufgespannt ausgerichtet werden. Die Abdeckung des Bauraums und somit den Grad der Informationen aus dem Experiment abgeleitetenniedrig, wie in Fig. 1A 12 gezeigt. Ferner können Abhängigkeiten zwischen verschiedenen Faktoren (Faktor-Wechselwirkungen) verborgen bleiben, was zu schlechter Modelle und / oder der Vorhersage falsch Optima, wie in 1B 13 gezeigt.
Die oben beschriebenen Nachteile können durch Verwendung eines Versuchsplanung (DOE) Methode, bei der die Durchläufe eines Experiments sind mehr gleichmäßig über den Entwurfsraum verstreut, was bedeutet, dass mehr als ein Faktor zwischen zwei Läufen 14 variiert vermieden werden. Es gibt spezielle Ausführungen für Mischungen, Screening Faktoren (Faktoren-Modelle) und die Quantifizierung der Auswirkungen auf Faktor Antworten (Response Surface Methoden, RSM s) 15. Weiterhin kann RSMs als zentrale Verbund Designs realisiert werden, sondern kann auch wirksam durch Verwendung spezieller Software, die Kriterien für die Auswahl der Läufe gelten erzielt werden. Beispielsweise die sogenannten D-optimality Kriterium läuft so zu wählen, um den Fehler in den Koeffizienten des resultierenden Modells zu minimieren, während die IV-Optimalitätskriterium wählt Läufe, die die niedrigste Vorhersage Varianz in der gesamten Entwurfsraum 15,16 zu erreichen. Der RSM beschreiben wir hier ermöglicht die genaue Quantifizierung von transienten Proteinexpression in Pflanzen, aber es kann problemlos an jedes System mit mehreren (~ 5-8) übertragen werden numerische Faktoren (zB Temperatur, Zeit, Konzentration) und ein paar (~ 2 - 4) categoric Faktoren (z. B. Promotor, Farbe), in dem eine mechanistische Beschreibung ist nicht verfügbar oder zu komplex, zu modellieren.
Die DoE-Ansatz entstand in den Agrarwissenschaften, hat aber auf andere Bereiche ausgebreitet, weil es zu jeder Situation, wo es sinnvoll ist, die Anzahl der Durchläufe notwendig, um verlässliche Daten zu erhalten, verringern und Beschreibungsmodelle für komplexe Prozesse übertragbar. Dies wiederum hat zur Aufnahme von DoE in der "Anleitung für LED-Industrie, Q8 (R2) Pharmaceutical Development "von der Internationalen Konferenz zur Harmonisierung der technischen Anforderungen an die für die Registrierung von Humanarzneimitteln (ICH) 17 veröffentlicht. DoE wird heute weitgehend in der wissenschaftlichen Forschung und der Industrie 18 verwendet. Allerdings muss darauf während genommen werden die Planung und Durchführung des Experiments, weil die Auswahl eines falschen Polynomgrad für die Multiple-linearen Regressionsmodell (Basismodell) eine Notwendigkeit für zusätzliche Fahrten, um alle Effekte richtig zu modellieren Faktor einzuführen. Außerdem beschädigte oder fehlende Daten zu erzeugen falsche Modelle und fehlerhaft Vorhersagen, und kann sogar verhindern, dass Modellbildung Versuch wie in den Protokoll-und Diskussionsabschnitte 18 beschrieben. In der Protokollabschnitt, werden wir zunächst die wichtigsten Planungsschritte für eine RSM-basierten Versuchs und dann erklären, das Design auf der Grundlage des DoE Software DesignExpert v8.1. Aber ähnliche Designs lassen sich mit anderen Software-includi gebaut werdenng JMP, Modde und STATISTICA. Die experimentellen Verfahren werden von Anweisungen für die Datenanalyse und Auswertung folgt.
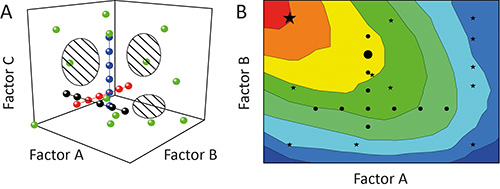
Fig. 1 ist. Vergleich von OFAT und DoE. A. Sequential Variation eines Faktors zu einer Zeit (OFAT) in einem Experiment (schwarz, rot und blau Kreise) erreicht eine geringe Abdeckung der Designraum (schraffierte Regionen). Im Gegensatz dazu die Variation von mehr als einem Faktor zu einem Zeitpunkt unter Verwendung der Versuchsplanung (DOE) Strategie (grüne Kreise) erhöht die Reichweite und damit die Genauigkeit der resultierenden Modelle. B. Das vorgespannte Design Raumabdeckung bedeutet, dass OFAT Experimente (schwarze Kreise) kann auch fehlschlagen, um optimale Betriebsbereiche (rot) zu erkennen und vorherzusagen, suboptimale Lösungen (großer schwarzer Kreis), während DoE strategisches (schwarze Sterne) sind eher zu bevorzugten Bedingungen (große schwarze Sterne) zu identifizieren.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Planen Sie eine DoE-Strategie
- Identifizieren relevanten Faktoren und Antworten für die Aufnahme in den Entwurf.
- Definieren Sie eine oder mehrere Antworten für die Messung. Hier wurden 2G12 und DsRed-Expressionsniveaus verwendet (ug / ml), einschließlich der minimalen nachweisbaren Unterschied als relevant (10 und 20 ug / ml) betrachtet und ein Näherungswert für die geschätzte Standardabweichung des Systems (4 und 8 ug / ml) auf der Grundlage früherer Experimente.
- Verwenden Sie die verfügbaren Literatur, Daten aus früheren Experimenten oder Fach Screening-Designs (z. B. einem faktoriellen Design finden Sie in der Einleitung), um signifikante Faktoren, deren Einfluss auf die Antworten quantifiziert werden (Tabelle 1) 7,8,19,20 auswählen.
- Ordnen Sie die Faktor-Typen (numerisch oder kategorische) und wählen Sie Bereiche, innerhalb derer die numerischen Faktoren werden während des DoE Untersuchung (Tabelle 1) variiert werden.
- Identifizieren numeric Faktoren für die kontinuierliche Änderung ist schwierig zu implementieren.
- Vermeiden, mit kontinuierlicher Variation für Faktoren, die nicht exakt auf einen bestimmten Wert eingestellt werden kann. Z. B. Inkubationstemperatur kann in der Regel nur innerhalb von ± 2 ° C gesteuert werden, damit eine kontinuierliche Variation unter Verwendung von Werten wie beispielsweise 27,2 ° C, 25,9 ° C und 29,3 ° C darf vermieden werden.
- Wählen Sie stattdessen eine Reihe von diskreten Niveaus für diese Faktoren (Tabelle 1). Die Anzahl der Ebenen sollten den erwarteten Basismodell (Schritt 1.2) entsprechen. Dies ist nur für optimale Designs wichtig, weil zentrale Verbund Designs haben immer diskreten Faktorstufen.
- Weisen Sie, ob eine kategorische Faktor ist nominal, dh keine stillschweigende Bestellung (zB verschiedener Hersteller) oder Ordnungs und damit einen diskreten numerischen Parameter (z. B. verschiedene Blätter an einer Pflanze).
- Wählen Sie die Ebenen für beide Arten von kategorischen Faktoren.
- Wählen Sie eine nützliche Basis-Modell.
- Auf Basis der vorläufigen Experimente und Literatur, rechnen die Beziehung zwischen einzelnen Faktoren und der Reaktion sowie die Wechselwirkungen zwischen Faktoren und der Antwort. Zum Beispiel, linear (je mehr, desto besser), quadratisch (Einzel optimale oder nichtlineare Zunahme / Abnahme) oder kubische (verzerrt Optimum).
- Stellen Sie sicher, dass die Anzahl der Ebenen für diskrete numerische Faktoren ist n + 1, wobei n das Polynom Grad der Beziehung zwischen Faktor und Reaktion. Zum Beispiel wird, wenn die Temperatur erwartet, dass eine quadratische Wirkung auf die Reaktion haben, n = 2 und damit mindestens drei Temperaturniveaus sollten untersucht, um eine Anpassung an die quadratischen Effekt zu erzielen.
- Definieren Sie den Anteil der Designraum (FDS), für die die Vorhersage Varianz sollte unter einem bestimmten Schwellenwert (Alpha-Ebene) sein. Die FDS sollte> 0,95 sein (decken mehr als 95% der Design-Raum), um Modelle, die robust Vorhersagen in ganz ergeben zu erreichender gesamte Design-Raum 21-23.
Hinweis: Ein Schwellenwert von 0,05 entspricht einem Signifikanzniveau von 5% (Typ-I-Fehler) und ist ein typischer Wert. Eine Erhöhung dieses Wertes wird die Anzahl der Versuche für eine DoE-Strategie zu reduzieren, aber gleichzeitig die Wahrscheinlichkeit erhöhen, dass signifikante Effekte werden verfehlt werden und dass die Schätzungen ihrer Auswirkungen auf die Antwort falsch sein.

Tabelle 1. Faktoren transiente Proteinexpression in Tabak einschließlich der Veränderung beeinflussen reicht während DoE. Faktoren fett wurden nur im Design für die als "Ein beschreibe Modell für DsRed Akkumulation während transiente Expression mit verschiedenen Promotor / 5'UTRs" beschriebenen Experimente enthalten wohin Faktoren in Kursivschrift wurden nur in der Ausführung für "Optimierung incubatio enthaltenn Bedingungen und Erntesysteme für die Herstellung von monoklonalen Antikörpern in Pflanzen mit transiente Expression ".
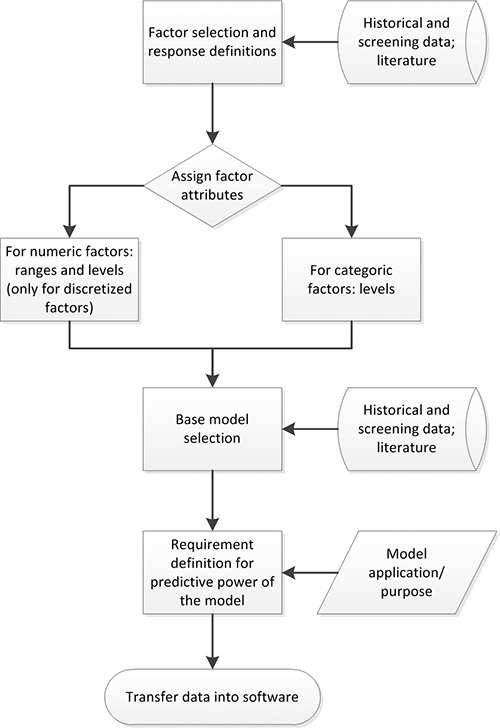
2. DoE Planungsprozesses. Faktoren, die erhebliche Auswirkungen auf die Reaktion untersucht werden auf der Grundlage der verfügbaren Daten ausgewählt. Dann Faktor Attribute (zB numerisch), Bereiche und Ebenen zugeordnet sind. Vorkenntnisse und Experimente verwendet werden, um eine geeignete Basis-Modell zu definieren. Die Vorhersagekraft Anforderungen werden auf der Grundlage der Anwendung / Zweck des endgültigen Modells definiert. Die erhobenen Daten können dann in entsprechende DoE-Software übertragen werden.
2. Einrichten einer RSM in DesignExpert
- Starten DesignExpert und wählen Sie "New Design". Im Fenster "Response Surface", wählen Sie &# 34; Optimal "und geben Sie die Anzahl der numerischen und kategorischen Faktoren in Abschnitt 1.1 ausgewählt).
- Geben Sie Faktor Namen, Einheiten, Typen, Untertypen, Anzahl der Ebenen / Niveau Grenzen und Werte für die Stufen für alle Faktoren in die entsprechenden Felder ein.
- Weiter zur nächsten Seite und unter "Suchen" Optionen "Best" sowie die gewünschte "Optimalität"-Kriterium, hier "D-optimal".
- Im Menü "Bearbeiten ... Modell", wählen Sie das Modell, das die Faktoren und Wechselwirkungen in Abschnitt 1.2 voraussichtlich) enthält. Wenn Sie nicht sicher, wählen Sie eine vollständige Modell, das alle Faktoren und Wechselwirkungen für eine bestimmte Polynomgrad, hier "Quadratisch" umfasst. Beachten Sie, dass die Auswahl eines voll-Modell (mit allen Interaktionen) können die Anzahl der Versuche für die DoE erforderlich erhöhen.
- Wählen Sie die Anzahl der "Bausteine". Hier wurde ein einzelner Block verwendet, da alle Pflanzen wurden aus der gleichen Charge, alle Bakterien gleichzeitig kultiviert worden war, undAlle Injektionen wurden am selben Tag vom selben Betreiber durchgeführt. Verwenden Sie mehr als einen Block, wenn mehr als eine Charge von Pflanzen verwendet wird oder mehrere Betreiber die Injektionen zu behandeln.
- Balance das Design
- Aktivieren Sie "Force kategorisch Balance", um die Läufe des Experiments gleichmäßig unter den kategorischen Faktorstufen zu verteilen, z. B. verschiedene Promotoren, die getestet werden.
Hinweis: Dies kann die Optimalität der Konstruktion zu einem Grad, DesignExpert wird als%-Wert je nach den von der Optimalität Algorithmus ausgewählt Punkte berichten reduzieren. - Neuberechnung der Design mehrmals mit Hilfe der implementierten Zufallsgenerator Algorithmus, um die Reduzierung der Optimalität zu minimieren.
Hinweis: Verluste in Optima kann von ~ 3-40% unter Verwendung derselben Eingangsdaten, aber die Änderung der Anzahl von Wiederholungen verwendet werden, um categoric Gleichgewicht zu halten.
- Aktivieren Sie "Force kategorisch Balance", um die Läufe des Experiments gleichmäßig unter den kategorischen Faktorstufen zu verteilen, z. B. verschiedene Promotoren, die getestet werden.
- Die Software wird ein Wert für die Anzahl der "Model-Punkte" auf der Grundlage der Basis-Modell vorschlagen, Hier 70 läuft. Stellen Sie die Anzahl der Durchläufe für "Replikate" und "Um den Mangel an Passform zu schätzen", um sicherzustellen, dass es> 5% der Anzahl der "Model-Punkte" für jeden, hier jeweils 10 läuft.
- Weiter zur nächsten Seite wählen Sie die Anzahl der Antworten und geben ihre Namen und Einheiten. Weitere Antworten kann auch jederzeit während der Datenauswertung ohne negative Auswirkungen auf das Design hinzugefügt werden.
- Weiter, um den Algorithmus zu starten, die Berechnung der Faktorstufen für die Läufe des DoE. Wenn das ausgewählte Basismodell ist die Anzahl der Ebenen für einen bestimmten Faktor nicht übereinstimmen, wird eine Benachrichtigung angezeigt, und die Berechnung wird nicht gestartet. In diesem Fall entweder:
- Erhöhen Sie die Anzahl der Ebenen für diesen Faktor, oder
- Deaktivieren Sie die Bedingungen im Basismodell, das nicht auf der Grundlage der aktuellen Anzahl der Ebenen berechnet werden kann.
- Beispielsweise wenn es zwei Ebenen für den Faktor "Inkubationszeit" t (dh 2 d und 5 d) in ter aktuelle Design und eine quadratische Basismodell mit der Bezeichnung t 2 gewählt wurde, fügen Sie entweder eine dritte Ebene für t (dh 8 d) oder entfernen Sie den Faktor t 2 aus dem Modell.
- Speichern Sie die Tabelle, die optimalen Faktorkombinationen, die angezeigt wird, wenn die Berechnung abgeschlossen ist enthalten.
- In der "Design"-Knoten wählen Sie die "Auswertung" Unterknoten und gehen Sie auf die Registerkarte "Diagramme".
- Im "Graphs Werkzeug" Auswahl "FDS" und in der "FDS Graph"-Box wählen Sie "Pred" als Fehlertyp. Geben Sie dann die Mindest nachweisbaren Unterschied sowie den ungefähren Wert für die geschätzte Standardabweichung der in Abschnitt 1.1.1 definiert System) als Werte für "d" und "s" bzw. (hier 20 und 8 pg / ml für DsRed). Geben Sie außerdem das Alpha-Niveau (akzeptable% fehlt eine signifikante Wirkung) geeignet für die Anwendung, in der Regel 0,05 (5%).
- Stellen Sie sicher, dass die berechnete FDS entspricht dem in Abschnitt 1.3 definierten Prozentsatz) (typischerweise> 0,95 für Vorhersagemodelle). Eine flache Kurve in der FDS Grundstück ist vorzuziehen, was gleichmäßige Vorhersagegenauigkeit der gesamten Design-Raum.
- Wenn dies nicht der Fall (wie hier, war FDS 1%, wie in 3A gezeigt), gehen Sie zurück zu den "Design"-Knoten und in der Taskleiste "Design Tools", dann "Augment Entwurf ..." und wählen Sie " Ergänzen ".
- Wählen Sie die gleichen "Suche" und "Optimalität" Kriterien wie zuvor. Außerdem wählte die gleiche "Edit-Modell" und Einstellungen "Force kategorisch balance". Wenn das Design Vergrößerung wird vor jedem Experiment (wie hier geschehen) durchgeführt wird, dann ändern Sie das "Setzen Sie läuft in den Block" auf "Block1".
- In der Rubrik "Runs" geben Sie die Anzahl der zusätzlichen "Model-Punkte", die Aufrechterhaltung von mindestens 5% "Replikate" und "To Mangel schätzenvon fit "in der Gesamtdesign. Dabei wurden 100 zusätzliche Fahrten aufgenommen und keine" Replikate "und" fehlender Passform schätzen "enthalten waren.
- Sobald die Berechnung abgeschlossen ist, überdenken die FDS Graphen wie oben beschrieben. Wenn der FDS ist immer noch nicht zufriedenstellend ist, wiederholen Sie den Entwurf Vergrößerung wie oben beschrieben. Hier war die FDS 100% nach Vergrößerung und damit keine weitere Verstärkung erforderlich war (3B).

Abbildung 3. Vergleich FDS Parzellen. A. A DoE, bestehend aus 90 Läufen produziert eine unzureichende FDS von nur 1% für die Standardfehler der Vorhersage, unter Verwendung einer quadratischen Basis-Modell in Kombination mit den Werten für die Mindest nachweisbaren Unterschied (20 ug / ml) und Schätzungengepaart Standardabweichung des Systems (8 &mgr; g / ml). B. Augmentation des DoE zu insgesamt 210 Pisten erreicht eine 100% FDS und einen Flach Kurve, die gleichbleibende Präzision des Modells in der gesamten Design-Raum.
3. Klonierung und Analyse der Expressionskassetten
- Kultivieren von Escherichia coli in 5 ml LB-Medium (10 g / l Trypton, 5 g / l Hefeextrakt, 170 mM NaCl, 50 mg / l Ampicillin, für LB-Agar-Platten wurden um 15 g / l Agar) bei 37 ° C für 6 -8 Stunden oder über Nacht auf einem Schüttler bei 160 Umdrehungen pro Minute. ACHTUNG: Ampicillin ist eine schädliche Substanz. Impfen der LB-Medium mit entweder 50 ul Flüssigkultur oder die Übertragung von Zellen Kolonien auf Platten mit einer Pipettenspitze gewachsen.
- Für die Reinigung von Plasmid-DNA PSO und andere Derivate von pPAM (GenBank AY027531) aus E. coli K12 Stamm DH5a, folgen Sie den Anweisungen in der DNA-Reinigung-Kit Handbuch 24. ACHTUNG: die Reinigung kes enthält schädliche Chemikalien (siehe oben Handbuch). Die Plasmid-Sequenz ist in Fig. 4 und 20 abgeschlossene et al. Beschrieben.
- Bestimmung der DNA-Konzentration in dem Eluat durch Messung der Extinktion einer 2 ul Probe bei 260 nm in einem ND-Gerät.
- Bestätigen der Identität der gereinigten Plasmid-DNA durch Verdauung mit Restriktionsendonukleasen (RE).
- Wählen Sie die REs nach der Plasmid-Sequenz, so dass einzigartige und unterscheidbare Fragmentmuster erzeugt werden. Die Empfehlungen des Herstellers für den Aufschlußbedingungen, wie Reaktionsvolumen, Zeit, Temperatur und Konzentration der Stabilisator Rinderserumalbumin. Je nach Empfindlichkeit der Analyseeinrichtung verwenden 50-500 ng der Plasmid-DNA pro Verdauung.
- Vorbereitung 0,8-2,0% Gele für die Trennung von DNA-Fragmenten durch Agarose in kochendem Tris-Borat-EDTA (TBE)-Puffer (90 mM Tris, 90 mM Borat, 2 mM EDTA, pH 8,0). Ter größer die erwarteten Fragmente sollten weniger Agarose in dem Gel verwendet werden.
- Werden 5 ul fünffache Probenpuffer (5x SABU, 0,1% (w / v) Bromphenolblau, 0,1% (G / V) Xylolcyanol, 10% (w / v) Glycerin in TBE gelöst) und 50 ul der RE-behandelten DNA-Probe zu trennen und die Fragmente durch Agarose-Gel-Elektrophorese bei 100 V für ~ 40 min, oder bis eine klare Trennung der Fragmente erreicht wird. Auf jedem Gel, gehören eine Spur, die DNA-Leiter Größenmarker für den Vergleich, z. B. 2-3 ul 1-kb-Leiter.
- Ersetzen Sie die Omega-5'-UTR in PSO mit einem der anderen drei 5'UTRs.
- Lassen Sie die Omega-5'-UTR-Sequenz von ~ 4 ug gereinigte PSO DNA durch Behandlung mit 20 Einheiten EcoRI-HF-und NcoI-HF REs in NEBuffer 4 bei 37 ° C ~ 60 min. Dann trennen Sie die Fragmente wie in den Abschnitten 3.4.2 und 3.4.3 beschrieben.
- Das größere Fragment zu isolieren (PS "Rückgrat") aus dem Agarosegel unter Verwendung eines Gels Extraction Kit nach der Anleitung des Herstellers 25 und bestimmen die Konzentration der, wie in Abschnitt 3.3 beschrieben gereinigten DNA). ACHTUNG: die Gel-Extraktions-Kit enthält schädliche Chemikalien finden Sie im Handbuch.
- Isolieren Sie die CHS CHS-LPH und TL 5'UTRs von geeigneten Spender-Vektoren durch die Behandlung ~ 10 ug jeden Vektor mit 20 Einheiten Eco RI-HF-und NcoI-HF REs in NEBuffer 4 bei 37 ° C ~ 60 min. Dann trennen Sie die Fragmente wie in den Abschnitten 3.4.2 und 3.4.3 beschrieben und reinigen die kleineren 5'-UTR-haltigen Fragment, wie in Abschnitt 3.5.2 beschrieben.
- Ligieren des isolierten gereinigten 5'UTRs in separaten Aliquots des isolierten gereinigten linearen pS Vektor bei 25 ° C für 5 min nach Angaben des Herstellers 26. Verwenden ~ 50 ng Vektor-DNA und ein dreifacher molarer Überschuss an 5'-UTR-DNA in einem Gesamtvolumen von 20 ul.
- Transformation E. coli-Zellen mit thE rekombinanten Plasmide 26,27.
- In ~ 10 ng (~ 4-5 ul) Ligationsansatz (siehe Abschnitt 3.5.4) auf 50 ul RbCI kompetente E. coli und vorsichtig mischen und dann für 30-60 Minuten auf Eis inkubiert. Hitzeschock für 1,5 min bei 42 ° C und kalt auf Eis für 5-30 min.
- Add 950 ul antibiotikafreien LB-Medium und Inkubation für 1 h bei 37 ° C und 160 Upm. Für die Selektion von Transformanten, verteilt 50 und 100 &mgr; l der Kultur auf LB-Agarplatten, die Ampicillin enthalten und Inkubation für ~ 16-20 Stunden bei 37 ° C.
- Impfen 10.05 einzelnen Kolonien, die jeden in Abschnitt 3.1 in 5-ml-Aliquots von LB-Medium Ampicillin enthält beschriebenen Promotor-5'-UTR-Ligation. Dann reinigen die Plasmid-DNA und bestätigen ihre Identität wie in den Abschnitten 3.2 bis 3.4 beschrieben.
- Ersetzen Sie die 35SS-Promotors mit dem nos-Promotor in jeder der vier 5'-UTR-Plasmide mit Asc I und Eco RI REs in NEBuffER 4 bei 37 ° C für 1 h, wie in den Abschnitten 3.5 und 3.6 beschrieben.
- Einführung jedes der acht erhaltenen Plasmide in A. GV3101: pMP90RK durch Elektroporation 28.
- In ~ 500 ng gereinigte Plasmid-DNA zu 50 ul zuständigen A. tumefaciens-Zellen auf Eis. Vorsichtig mischen und in ein vorgekühltes übertragen 0,2 cm Elektroporationsküvette. Stellen Sie sicher, dass das Gemisch am Boden der Küvette und keine Luftblasen enthalten.
- Impuls Zellen bei 2,5 kV für 5 ms und bestätigen die Intensität und Dauer des Impulses.
- Vermeiden Sie erhöhten Salzkonzentrationen in der DNA-Probe oder falsche Vorbereitung des zuständigen A. tumefaciens-Zellen, da diese bei hohen Ionenströme verursacht sofortigen Verdampfung der Zellsuspension führen Transformationseffizienz stark verringert.
- In 950 ul YEB-Medium ohne Antibiotika (5 g / L Rindfleischextrakt, 1 g / l Hefeextrakt, 5 g / l Peptone, 5 g / l Saccharose, 2 mM MgSO4, pH 7,0), vorsichtig mischen und sofort übertragen, in ein steriles 1,5 ml Reaktionsgefäß. Inkubation für 2-4 h bei 26-28 ° C und 160 Upm.
- Verbreiten Sie 1-2 ul YEB auf Agar-Platten mit Antibiotika (50 mg / L Carbenicillin, 25 mg / l Kanamycin, 25 mg / l Rifampicin) für die Selektion von Transformanten. VORSICHT: Rifampicin ist eine toxische Substanz ist.
- Impfen drei 5-ml-Aliquots YEB Medium mit Antibiotika mit Zellen aus einzelnen Kolonien, die jeden Promoter-5'-UTR-Ligation und Inkubation für 48-72 Stunden bei 26-28 ° C und 160 Umdrehungen pro Minute.
- Bestätigen Sie den Erfolg von A. tumefaciens-Transformation.
- Transfer 2 ul jedes Aliquot von Abschnitt 3.8.6 zu 48 ul Aliquots der PCR-Mastermix (2 ul jeder 10 uM Primer Lager (400 nM Endkonzentration von jedem Primer), trennen1 ul 10 mM dNTP-Mix (200 uM Endkonzentration jedes Desoxynucleosidtriphosphat), 5 ul 10-fach Expand High Fidelity-Puffer mit 15 mM MgCl 2, 0,75 ul Expand High Fidelity Enzym-Mix und 39.25 ul sterilem destilliertem Wasser).
- Amplifikation der Expressionskassette von jedem Plasmid unter Verwendung von geeigneten Primern (FWD: 5'-CCT CAG GAA GAG CAA TAC-3 ', Bindungs 1026 Nukleotide stromaufwärts des Promotors, REV: 5'-CCA AAG CGA GTA CAC AAC-3', Bindungs innerhalb der 35S Polyadenylierungssignal) unter geeigneten PCR-Bedingungen. Hier 94 ° C wurde für die anfängliche Denaturierung, gefolgt von 30 Zyklen von 94 ° C Denaturierung für 15 Sekunden verwendet wird, 51 ° C Annealing für 30 sec und 72 ° C Elongation für 120 sec und einer abschließenden Verlängerungsschritt bei 72 ° C für 8 Minute
- Bestimmung der Größe der PCR-Produkte mittels Agarose-Gelelektrophorese, wie in Abschnitt 3.4.3 beschrieben.
- Bereiten A. tumefaciens Glycerin Aktien vonMischen von 500 &mgr; l 50% (v / v) steriles Glycerin mit 500 ul A. tumefaciens Kulturen aus Abschnitt 3.8.6, für die Transformation wurde (Abschnitt 3.9.3) bestätigt. Speichern die Glycerinstammkulturen bei -80 ° C bis zur weiteren Verwendung.
- Berechnung der Faltungsenergien der verschiedenen mRNAs mit der RNAfold Webserver 29 und umfassen die Nukleotidsequenz von der Transkriptionsstartstelle bis zu den ersten 50 bp der codierenden Region.
- In der Rubrik "Fold Algorithmen und grundlegende Optionen" wählen Sie die "minimale freie Energie (MFE) und Partitionsfunktion" und "Vermeidung von isolierten Basenpaare"-Optionen.
- In der "Advanced Klapp-Optionen" wählen Sie "baumelt Energien auf beiden Seiten einer Helix in jedem Fall", "RNA-Parameter (Turner-Modell, 2004)" und ändern "neu skalieren Energieparameter zu bestimmten Temperatur (C)" bis 25 ° C
- In der Rubrik "Ausgabeoptionen" wählen Sie alle Optionen.
- Von der "Ergebnisse für thermodynamische Ensemble-Vorhersage" des Ausgabe-Datei, entpacken Sie die "freie Energie des thermodynamischen Ensemble" und die "Frequenz der MFE-Struktur in den Ensembles" Werte für den Vergleich.
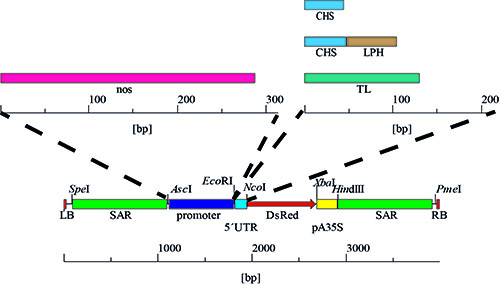
4. Promotor und 5'-UTR-Varianten. Die Expressionskassetten wurden durch stufenweise Austausch der 5'-UTR erzeugt, was vier Kombinationen mit 35SS CaMV-Promotor, gefolgt von dem Austausch des Promotors mit dem nos-Sequenz ergab vier weitere Varianten und insgesamt acht unterschiedliche Promotor / 5'UTR Kombinationen.
4. Pflanzenbau
- Vorbereitung einer 0,1% igen Lösung des Düngemittels Ferty2 Mega in deionisiertem Wasser bei pH 5,9.
- Bereiten 10 x 10 x 8 cm Steinwollblöcke durch umfangreiche Spülung mit VE-Wasser, um restliche Chemikalien zu entfernen und schließlich ins Gleichgewicht mit Dünger.
- Seed Tabakpflanzen, indem 1-2 Tabaksamen auf jeder Wollblock, gefolgt von einem kurzen bündig mit Dünger vorsichtig, um zu vermeiden Waschen der Samen entfernt.
- Keimen und pflegen die Tabakpflanzen 42 Tage im Gewächshaus bei 25/22 ° C Tag / Nachttemperatur, 70% relativer Luftfeuchtigkeit und einem 16-Stunden-Photoperiode (180 mmol m -2 s -1; λ = 400-700 nm) . Während dieser Photoperiode, bewässern mit Dünger für 15 Minuten jede Stunde in Hydrokultur.
5. Transient Protein Expression
- Bereiten A. tumefaciens für die Injektion in Blättern.
- Impfen 5-50 ml YEB Medium mit Antibiotika mit 1% A. tumefaciens Kryo-Stammkultur und Inkubation bei 27 ° C, bis die OD 600 nm erreicht 5,0 (~ 48-72 h je nach Volumen und Schiffstyp).
- Verdünnen Sie die A. tumefaciens-Kultur mit Wasser und 2-fach Infiltrationsmedium (4,3 g / l Murashige-Skoog-Salzen (pH 5,6), 5 g / l Saccharose, 1,8 g / l Glucose, 100 mM Acetosyringon), um die OD 600 nm für die Injektion erforderlichen entsprechen. Bestätigen Sie die OD 600nm kurz vor der Injektion. Hinweis: Ein OD 600 nm von 1,0 entspricht ~ 1,43 ± 0,12 x 10 9 koloniebildenden Einheiten pro ml.
- Spritzen Sie die A. tumefaciens-Suspension in Blättern.
- Wählen und beschriften Sie die noncotyledon Blätter und Positionen darauf zu (z. B. nach einer Strategie DoE) behandelt werden.
- Sie verschiedene A. injizieren tumefaciens-Lösungen in der gleichen Zwischenrippenfeld. Verwenden Sie stattdessen gegenüberliegenden Abschnitten auf jeder Seite der Mittelnerv-Achse. Wenn mehr als zwei unterschiedliche Lösungen müssen an der gleichen Position eingespritzt werden verwendet eine additional Anlage.
- A. gründlich schütteln tumefaciens-Lösung, um alle Zellen, die seit der Herstellung der Verdünnung und saugen in eine 1-ml-Spritze abgelagert haben resuspendieren.
- Leicht zerkratzen die Epidermis an der Stelle für die Injektion mit einer Pipettenspitze oder ähnliches, um den Zustrom der A. erleichtern soll tumefaciens Lösung. Vermeiden Zerreißen der Blattspreite während dies zu tun.
- Halten Sie die Spritze senkrecht zur Blatt Klinge berühren den Lauf gegen die Zwischenrippenfeld behandelt werden und schieben den Auslass (ohne Nadel befestigt) auf der Unterseite des Blattes. Drücken Sie die Oberseite des Blattes sanft zugleich die Blattspreite Verschieben oder Brechen zu verhindern.
- Drücken Sie vorsichtig den Spritzenkolben. Die A. tumefaciens-Lösung wird die interzellulären Räume innerhalb der Blattspreite eingeben, wie durch die behandelten Bereiche dunkler grün und feucht erscheinen angezeigt. Wiederholen Sie diesen Vorgang an mehreren Positionen, bis die ganze intercostal Feld mit A. infiltriert tumefaciens. Dann mit dem nächsten Zwischenrippen Bereich fortzusetzen.
- Stellen Sie sicher, dass die Spritze senkrecht zur Blatt bleibt. Kippen Sie die Spritze bewirkt, dass der Bakteriensuspension unter hohem Druck herausspritzen.
- Wenn verschiedene A. tumefaciens-Lösungen verwendet werden (z. B. auf verschiedene Promotoren zu testen), entfernen Sie alle verbleibenden auf der Unterseite des Blattes von der ersten Injektion mit einem Papiertuch oder ähnliches, bevor die nächste Lösung überschüssige Lösung.
- Post-Infiltration Inkubation von Pflanzen und Probenahme.
- Bereiten Sie eine Phytotron für den behandelten Pflanzen ~ 24 Stunden vor der Injektion auf Temperatur-und Feuchtigkeitsausgleich bei den für die DoE erforderliche Niveau zu ermöglichen.
- Nach der Injektion in die Pflanzen übertragen Phytotron und legen Sie sie in Schalen groß genug für die Bewässerung mit Wasser, bis zum Ende der Inkubationszeit von der DoE bestimmt.
- Stellen Sie eine 16 Stunden photoperiod mit sechs Osram cool white 36 W Leuchtstoffröhren je 0,7 m 2 (75 mmol m -2 s -1; λ = 400-700 nm). Verhindern, dass die Pflanzen aus einander Beschattung durch die Begrenzung der Anzahl der Pflanzen auf sechs pro 0,7 m 2.
- Vor der Probenahme, sicherzustellen, dass die richtigen Zwischenrippenfeld wurde durch Vergleich der Label in Abschnitt 5.2.1 und der DoE-Plan aufgenommen ausgewählt.
- Verwenden Sie einen Korkbohrer auf 4-5 Blattscheiben von den behandelten Zwischenrippenfelder an den von der DoE angegebenen Positionen und Zeiten, zu entfernen. Sie das ganze Blatt von der Pflanze während der Probenahme nicht entfernen. Stabilisierung des Blattes mit einer Handpapierhandtuch beim Entfernen der Scheiben zu Bruch zu verhindern.
- Bestimmen Sie die Masse von jeder Probe und legen Sie sie in einem 1,5 ml Reaktionskunststoffrohr mit Beispielnamen und Masse gekennzeichnet. Lagern Sie die Proben bei -20 ° C oder -80 ° C vor der Proteinquantifizierung. Das Verfahren kann an dieser Stelle für mehrere Monate abhängig von der Probenstabilität pausiertund die Lagertemperatur.
6. Proteinquantifizierung
- Extrahieren von Proteinen aus den Blattscheibenproben.
- 3 ml Extraktionspuffer (50 mM Natriumphosphat, 500 mM Natriumchlorid, pH 8,0) pro mg Probenmasse und Schleifblattscheiben im Reaktionsrohr mit einer elektrischen Stößel, bis keine großen Fragmente bleiben. Überhitzung der Probe zu vermeiden.
- Entfernen dispergierten Feststoffe zweimal Zentrifugieren der Probe bei 16.000 × g für 20 min bei 4 ° C. Den Überstand in ein sauberes 1,5 ml Reaktionsgefäß nach jedem Schritt, ohne das Pellet zu stören.
- Nach der Zentrifugation kann der Prozess durch Einfrieren in Abhängigkeit von der Probenstabilität und Lagertemperatur die Pflanzenextrakte bei -20 ° C oder -80 ° C für mehrere Monate pausiert. Zu bestätigen, daß ein Gefrier-Auftau-Zyklus beeinträchtigt nicht die Konzentration des Zielprotein (en).
- Messen Sie die DsRed-Fluoreszenz.
- Bereiten Sie drei technische Replikate jeder Probe in schwarzen 96-Well-Halbplatten Bereich (50 ul Extrakt pro Vertiefung). Vermeiden Sie die Bildung von Blasen beim Pipettieren.
- Verwenden Sie einen Satz von sechs Verdünnungen (0, 25, 75, 125, 175 und 225 pg / ml) einer DsRed-Standard pro 96-Well-Platte, eine Auskleidung Referenzkurve zu erzeugen. Bereiten Sie die Verdünnungen in PBS und speichern sie bei 4 ° C für den Einsatz innerhalb von 3 Monaten.
- Messung der Fluoreszenz zweimal nacheinander in einer 96-Well Plattenlesegerät mit 530/25 nm-Anregungs-und 590/35 nm-Emissionsfiltern ausgestattet.
- Für jede Probe, die Fluoreszenz im Durchschnitt über die zwei liest und die drei technischen Wiederholungen und subtrahieren den Wert für die Blindkontrolle mit 0 pg / ml DsRed aufgezeichnet. Auch subtrahieren diesen Wert aus der liest der Standardverdünnungen und diese leer korrigierten Werte für eine lineare Regression was eine Referenzkurve (durch den Ursprung des Koordinaten).
- Verwenden Sie die Steigung der Referenzkurve, die f konvertierenluorescence für die Proben in DsRed-Konzentrationen gemessen. Falls erforderlich, die Proben zu verdünnen, so dass die Anzeige innerhalb des Konzentrationsintervall der Standards fällt und betrachten diesen Verdünnungsfaktor in nachfolgenden Berechnungen.
- Bestimmen Sie die Konzentration von 2G12.
- Vorbereitung einer Oberflächenplasmonresonanz (SPR)-Vorrichtung für die Messung der Antikörperkonzentration durch Kuppeln von Protein A an die aktivierte Oberfläche einer Durchflusszelle. Verwenden Sie ein anderes Flusszelle durch Inaktivierung des Referenzoberfläche ohne Kopplung von Protein A 30,31.
- Verdünnte Pflanzenextrakte 1:20 SPR-Laufpuffer (10 mM HEPES pH 7,4, 3 mM EDTA, 150 mM NaCl, 0,05% v / v Tween-20) und messen die Antworteinheiten (RU) der Antikörperbindung an Protein A in drei technische Wiederholungen jeder Probe am Ende einer 90 ul-Injektion (180 sec bei 30 ul / min). Subtrahieren Sie die RU gemessen am Referenzflusszelle (kein Protein A) von der RU in der experimentellen fl gemessenow Zelle (Protein A-Oberfläche).
- Messen Sie 585 ng / ml 2G12 Standard nach jeweils 10 bis 15 Proben und subtrahieren die in den Referenzdurchflusszellen, wie oben beschrieben gemessen wird. Verwenden Sie die durchschnittliche RU dieser Standards, um eine lineare Bezugskurve durch den Ursprung des Koordinaten zu berechnen.
- Berechnung der 2G12-Konzentrationen in den Proben auf der Basis der 1:20-Verdünnung und der Steigung der Referenzkurve.
- Überprüfen Sie für eine starke unspezifische Bindung an die Referenzzelle, die korrupten kann die Messung. Überprüfen Sie auch, ob die Standards zu halten 2G12 etwa konstante Werte (<5% Abweichung) während Analyse, wie größere Variation spiegelt Alterung der Protein-A-Oberfläche.
7. Datenanalyse und Auswertung
- Manuell zu analysieren, die in den Expressionsversuche für (i) Extremwerte (hoch oder niedrig), die Messfehler beobachteten Reaktionen, (ii) ungewöhnliche Ergebnisse, z. B. Faktorkombinationen, die Gen-bewerten unerwartete Antworten anzeigt Datenaustausch, und (iii) fehlende Werte.
Hinweis: Verwenden Sie diese Aktionen auf Modellbildung zu verhindern, basierend auf fehlerhaften Daten. - Übertragen Sie die Antwortdaten analysiert (hier werden die Proteinkonzentrationen) in das "Design"-Knoten DesignExpert. Stellen Sie sicher, dass die Antwortdaten korrekt zu den entsprechenden Faktor Einstellungen zugewiesen. Es gibt eine umfangreiche Hilfe-Sektion in der DesignExpert Software, die angesprochenen Aspekte unter Cover vorhanden.
- In der "Analyse"-Knoten, wählen Sie die Antwort zu analysieren und wählen Sie zunächst "keine" in der Registerkarte Transformation.
Hinweis: eine Transformation für Min / max-Verhältnisse der Reaktion größer als 10 empfohlen. Die nützlichsten Transformation kann aus der Box-Cox-Plot in der Registerkarte "Diagnose" im Abschnitt "Diagnose" der "Diagnose-Tool" beschrieben (Abschnitt 7.8) erhalten werden. Ein Log 10 Transformation ist oft sinnvoll. - Fahren Sie mit dem "Fit Zusammenfassung"-Registerkarte, die allgemeine Informationen über Faktoren, die für das untersuchte System (z. B. Zwei-Faktor-Wechselwirkungen, quadratische Effekte) sind bietet. Die Software wird eine erste Modell, das auf seine Bedeutung vor.
- In der Registerkarte "Modell" wird ein erstes Modell auf der Basis der "Fit Sicht" Ergebnisse vorgewählt. Verwenden Sie den automatischen Modus, um dieses Modell zu bearbeiten:
- Wählen Sie eine "Prozessordnung", die eine Bestellung über das vorgeschlagene Modell ist, z. B. wenn das vorgeschlagene Modell ist "2FI" (Zwei-Faktor-Interaktion) und wählen Sie dann "Quadratisch".
- Wählen Sie "Rückwärts" im Feld "Auswahl", die iterativ zu entfernen wird nicht signifikante Begriffe aus dem Modell nach hend auf die "ANOVA"-Reiter auf der Grundlage der "Alpha out" Wert, der sollte sich zunächst 0.100 sein.
- Wenn von der Software aufgefordert, immer automatisch korrigieren Sie die Modellhierarchie, denn dies ist notwendigerweisey, zuverlässige Modelle 32,33 generieren.
- In der Registerkarte "ANOVA", untersuchen die vorgeschlagene Modell und die enthaltenen Faktoren. Wenn nötig, alle Faktoren mit p-Werte über einer vorgegebenen Schwelle manuell entfernen (hier 0,05, was einer 5%-Niveau) oder solche, die wahrscheinlich auf mechanistische Überlegung, durch Zurückschalten auf der Registerkarte "Modell" ändern "Auswahl" auf "Manuell" und die Beseitigung der entsprechenden Faktoren aus dem Modell.
- Zurück in der Registerkarte "ANOVA" bewerten aktualisiert Modell in Bezug auf die p-Wert der "Model" (ein niedriger Wert ist wünschenswert, da dies zeigt Bedeutung) und dem "Lack-of-fit" (ein hoher Wert ist wünschenswert weil dies zeigt Mangel an Bedeutung) sowie die Werte für "R-Quadrat", "Bereinigt R-squared" und "Vorhergesagte R-Quadrat" (Werte> 0,80 sind wünschenswert für alle drei Werte).
- Vergleichen Sie diese Werte for verschiedene Modelle mit / ohne an mehreren Faktoren Signifikanzniveaus.
Hinweis: Es kann hilfreich sein, um die Reaktion Spalte in der "Design"-Knoten duplizieren und individuell durchführen jede Analyse oder die ganze "ANOVA"-Tabelle in ein anderes Programm zu exportieren, wie eine Tabellenkalkulation.
- Vergleichen Sie diese Werte for verschiedene Modelle mit / ohne an mehreren Faktoren Signifikanzniveaus.
- Fahren Sie auf die Registerkarte "Diagnose", um die Qualität des Modells zu bestätigen und erkennen potentielle Ausreißer in der Datenmenge, die einen starken Einfluss auf das Modell durch die Untersuchung alle Registerkarten in der "Diagnose Tool" (Abschnitt "Einfluss", "Diagnose" und) haben .
- In der Rubrik "Diagnose" der "Diagnose-Tool", die folgenden Aktionen:
- Achten Sie darauf, die Punkte werden zufällig innerhalb der Grenzen in der Streu "Residuen gegen vorhergesagte" Diagramm. Hier wird eine winkelförmige Verteilung auf eine erforderliche Datenumwandlung.
- Achten Sie darauf, die Punkte werden zufällig innerhalb der Grenzen in die Streu"Residuen gegen vorhergesagte" Diagramm. Hier wird eine winkelförmige Verteilung auf eine erforderliche Datenumwandlung.
- Untersuchen Sie, ob die Punkte in der "Residuen vs run" Streudiagramm zufällig innerhalb der Grenzen. Ein Muster (zB "Treppe") gibt einen Trend in der Daten-und Gebäudeblock kann verwendet werden, um ihren Einfluss auf das Modell entgegenzuwirken.
- Suchen Sie nach einer gerade diagonale Linie, die die ideale Modell in der "Vorhergesagte und tatsächliche" Handlung. Desto größer ist die Streuung der Daten auf der Diagonalen, desto ungenauer die Modell.
- Prüfen Sie, ob eine Überlappung der blauen (am besten) und Grün (Ist) vertikale Linien in der Box-Cox-Plot, der die Datentransformation. Wenn die grüne Linie ist die blaue (außerhalb des Intervalls durch die roten vertikalen Linien) nicht überein gehen Sie zurück zu Abschnitt 7.3 sowie Modellbildung Runde verbessern, indem die Auswahl der Datentransformation von der Box-Cox-Plot vorgeschlagen. Auch die Verdoppelung der WiederReaktion Spalte kann sinnvoll sein, verschiedene Modelle zu vergleichen.
- Stellen Sie sicher, dass die Punkte in den "Residuen vs Factor"-Charts (es gibt ein Diagramm für jedes Modell Faktor) streuen zufällig innerhalb der Grenzen. Krümmung in der Datenverteilung kann ein Faktor fehlt im Modell zeigen.
- In dem Abschnitt "Einfluss" der "Diagnose-Tool", stellen Sie sicher, dass zufällige Streuung der Daten innerhalb der Grenzen in den "Student-Residuen Äußerlich", "Leverage", "DFFITS" und "DfBetas" Grundstücke und gleichmäßig niedrige Verteilung ohne Extremwerte in der "Cooks Distance" Handlung.
- In der Rubrik "Diagnose" der "Diagnose-Tool", die folgenden Aktionen:
- In der Registerkarte "Modellgraphen", visualisieren die ausgewertet Modell. Für eine begrenzte Anzahl von numerischen Faktoren (z. B. 3) die Antwortoberfläche ("3D-Oberfläche") Darstellung ist nützlich, um optima / Merkmale manuell zu beurteilen.
Hinweis: Antwortflächen nur die Auswirkungen der beiden Faktoren zu veranschaulichentoren auf die Reaktion untersucht. Die Auswirkungen von zusätzlichen Faktor auf die Reaktion wird durch die Änderung seines Wertes (numerische Faktoren) oder Ebene (kategorische Faktoren) im Fenster "Werkzeug Factors" enthüllt. Alternativ können Faktoren für die Handlung Achse durch Rechtsklick in das Fenster "Faktoren Tool" und wählen Sie die gewünschte unabhängige Variable Achse zugeordnet werden.- Bearbeiten Sie die Faktorstufen und weisen Sie auf die Grafik mit Hilfe der Koordinaten "Faktoren-Tool".
- Export Graphen mit der "Export Graph in Datei ..." in der Registerkarte "Datei"-Befehl.
- Verwenden Sie die "Numerical" Unterknoten in der "Optimierung" Knoten, um die Antwort numerisch über die Optimierung (minimieren, maximieren, in-Bereich) in Abhängigkeit von den Faktoren-Modell, an dem wiederum bestimmte Einschränkungen angewendet werden können (z. B. Grenzen und Gewichte) Registerkarte "Kriterien".
- Berechnen und untersuchen numerische Lösungen in den "Solutions"Registerkarte basierend auf der Eingabe in der Registerkarte "Kriterien" zur Verfügung gestellt.
- Exportieren Sie diese Lösungen auf andere Software (z. B. Tabellen) für weitere Analysen (zB Histogramme) enthüllt die Faktoreinstellungen mit hoher oder niedriger Antwortwerte verbunden.
Hinweis: Dies ist hilfreich, wenn mehr als drei numerischen Faktoren untersucht und 3D-Darstellung ist schwierig.
- Verwenden Sie den "Point Vorhersage" Unterknoten, die Antwort für bestimmte Faktor-Einstellungen vorherzusagen.
- Mit dieser Vorhersage für alle Einstellungen ausgewertet werden (hier alle Promotor und 5'-UTR-Kombinationen sowie alle Blätter und Inkubationszeiten oder Blattpositionen und Inkubation Temperaturen).
- Exportieren der vorhergesagten Werte und kompiliert sie an eine Datenreihe, die verwendet werden können, um die transiente Expression in Tabakpflanzen beschrieben, beispielsweise durch Berechnen des Ausdrucks für eine spezifische Promotor-5'-UTR-Kombination zu einem bestimmten Zeitpunkt gemittelt über alle Lamellen-Positionen oder across alle Blätter einer Pflanze.
- Normalisieren der Expressionsdaten, basierend auf den Massen der verschiedenen Blättern, um die spezifische Expressionsniveau (g g -1) oder spezifische Expressionsrate (g g -1 h -1) zu erhalten.
- Alternativ exportieren Sie die Koeffizienten der "Final Gleichung" aus dem unteren Bereich der Registerkarte "ANOVA" in eine Tabellenkalkulation und multiplizieren Sie sie mit einer Reihe von Einstellungen Faktor, um die gleiche Datenfeld ergeben.
Hinweis: Dieses Feld kann nur Werte enthalten Faktor innerhalb der ersten Design-Raum, weil das angepasste Modell Gleichung nicht zur Extrapolation geeignet.
- Führen Sie eine Bestätigungsexperiment für Modell Punkte, die von großem Interesse sind.
- Führen transiente Proteinexpression unter Bedingungen für die "Point Vorhersage" (Abschnitt 7.11) ausgewählt. ZB die gleichen Temperaturen und Blätter usw.
- Bestimmen Sie die Proteinkonzentrationen für diese vorübergehende Expression eXperiment wie oben beschrieben (Abschnitt 6) und vergleichen sie mit den Werten, die durch das Modell vorhergesagt.
- Prüfen Sie, ob der durchschnittliche Proteinkonzentration in der Bestätigungs-Experiment innerhalb der Response-Surface-Modell der Vorhersageintervall während Punkt erhalten Vorhersage fällt. Eine Übereinstimmung bestätigt die Vorhersagekraft des Modells. Eine Abweichung zeigt eine geringe Modellqualität und zusätzliche Fahrten erforderlich werden kann.
- Hinweis: Anlage von Charge zu Charge Variabilität erweitert das Vorhersageintervall und Bestätigung können Experimente mit einer anderen Charge von Pflanzen durchgeführt abzuwerten. Allerdings können Proben, die nicht verwendet wurden, um das Modell zu generieren, aber das aus der gleichen Pflanze Batch entstand als die Proben im Modell enthalten sind, wie wirksame Kontrollen zu dienen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Ein Beschreibungsmodell für DsRed Akkumulation während transiente Expression mit verschiedenen Promotoren und 5'UTRs
DsRed-Fluoreszenz in Blattextrakten wurde verwendet, um das Expressionsniveau des rekombinanten Proteins zu zeigen und damit als Antwort auf das SVM-Strategie verwendet. Die minimale nachweisbare Differenz wir als relevant war 20 ug / ml und die geschätzte Standardabweichung des Systems war 8 ug / ml bezogen auf den anfänglichen Experimenten. Faktoren,...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Jedes Experiment erfordert eine sorgfältige Planung, da die Ressourcen sind oft knapp und teuer. Dies gilt insbesondere für DoE Strategien, weil Fehler in der Planungsphase (z. B. Auswahl eines Basismodell, die nicht alle wesentlichen Faktor Wechselwirkungen einschließen) im wesentlichen die Vorhersagekraft der resultierenden Modelle verringern und damit entwerten das gesamte Experiment. Allerdings können diese Fehler leicht durch folgenden grundlegenden Verfahren vermieden werden.
...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Veröffentlichungsgebühr wurde teilweise von den Unternehmen Statease, Inc. (USA) und Statcon (Deutschland), die bei der Herstellung des Manuskripts oder für ihren Inhalt verantwortlich nicht in den beteiligten beteiligt waren gesponsert.
Danksagungen
Die Autoren danken Herrn Dr. Thomas Rademacher für die Bereitstellung der pPAM Pflanzenexpressionsvektor und Ibrahim Al Amedi zur Kultivierung der Tabakpflanzen in dieser Studie verwendet. Wir möchten Dr. Richard M. Twyman für seine Unterstützung bei der Bearbeitung des Manuskripts danken. Diese Arbeit wurde zum Teil durch den European Research Council Advanced Grant "Future-Pharma" gefördert, Vorschlagsnummer 269110 und der Fraunhofer-Zukunftsstiftung (Fraunhofer-Zukunftsstiftung).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Referenzen
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. Design and Analysis of Experiments. , John Wiley & Sons Inc. (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , Wiley. (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- FDA. , U.S. Department of Health and Human Services. Rockville, MD, USA. (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. DOE Simplified: Practical Tools for Effective Experimentation. , Productivity Inc. (2000).
- Anderson, M. J., Whitcomb, P. J. Response Surface Methods Simplified. , Productivity Press. (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- Plasmid DNA purification User manual. , MACHEREY-NAGEL. Düren. (2012).
- PCR clean-up Gel extraction User manual. , MACHEREY-NAGEL. Düren. (2012).
- Quick Ligation Protocol. 4, NewEnglandBiolabs. Ipswich. (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten