Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
HIV-1 ile infekte Canlı İlköğretim İnsan Makrofagların Ölçülen fagosom Göç ve Hız
Bu Makalede
Özet
We describe a method to measure the velocity of phagosomes moving towards the cell center in living cells infected with or without the human immunodeficiency virus (HIV) type 1, using spinning disk confocal fluorescence microscopy to identify fluorescent infected cells and bright field microscopy to detect phagosomes.
Özet
Macrophages are phagocytic cells that play a major role at the crossroads between innate and specific immunity. They can be infected by the human immunodeficiency virus (HIV)-1 and because of their resistance to its cytopathic effects they can be considered to be persistent viral reservoirs. In addition, HIV-infected macrophages exhibit defective functions that contribute to the development of opportunistic diseases.
The exact mechanism by which HIV-1 impairs the phagocytic response of macrophages was unknown. We had previously shown that the uptake of various particulate material by macrophages was inhibited when they were infected with HIV-1. This inhibition was only partial and phagosomes did form within HIV-infected macrophages. Therefore, we focused on analyzing the fate of these phagosomes. Phagosome maturation is accompanied by migration of these compartments towards the cell center, where they fuse with lysosomes, generating phagolysosomes, responsible for degradation of the ingested material. We used IgG-opsonized Sheep Red Blood Cells as a target for phagocytosis. To measure the speed of centripetal movement of phagosomes in individual HIV-infected macrophages, we used a combination of bright field and fluorescence confocal microscopy. We established a method to calculate the distance of phagosomes towards the nucleus, and then to calculate the velocity of the phagosomes. HIV-infected cells were identified thanks to a GFP-expressing virus, but the method is applicable to non-infected cells or any type of infection or treatment.
Giriş
Macrophages play a major role in the innate immune system and in homeostasis. They are professional phagocytes that internalize and eliminate pathogens and debris by a process called phagocytosis 1,2. The phagosome, the closed compartment that forms after the engulfment of particulate material, undergoes a series of fusion and fission events with endocytic compartments, leading to a degradative compartment called the phagolysosome. This compartment has an acidic pH, due to the acquisition of proton-pumping v-ATPases, contains hydrolytic enzymes and is enriched in lysosomal-associated membrane proteins (LAMPs). The maturation of phagosomes is accompanied by their migration on microtubules 3,4 towards the cell center to reach a perinuclear location where lysosomes are accumulated.
Many pathogens have been reported to hijack phagosome maturation, including bacteria with intracellular lifestyles that modify the vacuolar environment where they reside 5. The Human Immunodeficiency Virus (HIV)-1 targets macrophages in addition to T cells. As macrophages are resistant to the cytopathic effects of the virus, unlike T cells, they can be considered as a reservoir for the virus. In addition, macrophages infected with HIV-1 show defective phagocytic functions and contribute to the emergence of opportunistic diseases. In particular, severe invasive non-typhoidal Salmonella disease caused by Salmonella Typhimurium ST313 has been prevalent for the last three decades in sub-Saharan African children or adults infected with HIV 6. It has been estimated that the risk of developing tuberculosis is more than 20 times greater in people living with HIV than among those without HIV infection.
For all these reasons, it is important to better define the molecular mechanisms underlying the phagocytic defects in HIV-infected macrophages. We have shown that the uptake of particulate material, opsonized particles, bacteria or fungi, was inhibited in HIV-infected macrophages 7. Given that this inhibition is partial, we then set out to analyze the fate of the internalized phagosomes in HIV infected human macrophages 8. Because phagosome maturation is tightly connected with migration to the cell center and fusion with lysosomes, a defect in phagosomal maturation can be due to modifications of the trafficking modalities in the infected cell. The method described here uses IgG-opsonized Sheep Red Blood Cells (IgG-SRBCs) as a model to target receptor-mediated phagocytosis and in particular receptors for the Fc portion of immunoglobulins (FcR). These particles are easier to image in bright field (BF) than latex beads because extracellular and intracellular SRBCs show different refraction properties 9. To measure the velocity of phagosomes moving towards the nucleus in HIV-infected macrophages, we used a fluorescent virus 10 and set up a simple manual tracking method that is described here. The method does not require advanced programming and simply uses ImageJ. It is amenable to adherent cells and any type of particle or pathogen that can be visualized with bright field microscopy or with fluorescent imaging.
Protokol
protokol, ulusal ve uluslararası mevzuata ve yerel yönetmeliklere tam uyum içinde yapılmalıdır. araştırma amaçlı kan bağışı için rızalarını verdi sağlıklı donörlerden kan Kurumları anlaşma imzaladı hangi ile Kan Transfüzyon Merkezleri elde edilmiştir. İnsan kanı kullanılması özel bir koruma alınmalıdır. HIV-1 ile yapılan deneyler 3 veya 2 (BSL-3 veya 2) laboratuvar, yerel mevzuata uygun bir biyogüvenlik seviyesinde yapılmalıdır.
Yapışma yoğunluk gradyan santrifüjü ve seçim İnsan monosit türevli makrofajlar (hMDMs) hazırlanması 1.
- Sağlıklı vericilerden (9 mi) taze kan başla. 70 ml'lik bir son hacim elde etmek için Ca + 2 ve Mg + 2 olmaksızın, steril 1 x fosfat tamponlu tuzlu su (PBS) ile taze kan tüm hacmi seyreltilir ve hafifçe iki 50 ml konik tüp içine seyreltilmiş kan eklemek (tüp başına 35 mi) bir 15 ml üstündekieutral, yüksek her bir tüp içinde daha önce çözelti içinde, yüksek kütle, hidrofilik polisakarit dallanmış.
- fren olmaksızın 20 ° C de 20 dakika boyunca 537 x g'de santrifüje kan her iki tüp. Daha sonra ara yüzeyde parçalı hücre halkasında bulunan periferal kan mononükleer hücreleri (PBMC'ler) toplamak ve Ca + 2 ve Mg + 2 olmadan 1x 15 ml PBS içeren yeni bir 50 mL tüp içine transfer edin.
- Santrifüj 20 ° C'de 5 dakika boyunca 218 x g'de hücreleri Ca + 2 ve Mg olmayan 1x PBS 45 ml pelet 2+.
- Santrifüj 20 ° C'de 5 dakika boyunca 218 xg'de hücreleri, Ca + 2 ve Mg + 2 olmadan 1x PBS, 10 ml pelet tekrar süspansiyon ve Tripan Mavisi 1/200 nihai seyreltme seyreltilerek hücreleri sayın.
- 20 ° C'de 5 dakika boyunca 218 x g'de hücreleri santrifüj ve 1640 ortamında 2 mM L-glutamin ve 100 ug / ml Penic ile desteklenmiş RPMI (Roswell Park Memorial Institute) peletillin-streptomisin, 6 gözlü bir plakanın her bir gözünde ortam 2 ml oyuk başına 7 x 10 6 PBMC'ler için.
- 2 saat boyunca% 5 CO2 ile 37 ° C 'de inkübe edin. 2 saat sonra monositler plastik yapışmış olur.
- Taze izole edilmiş monositlerin, hMDMs içine ayırt orta aspire ile takviye edilmiş 2 ml hMDM ortamı (RPMI 1640, fetal buzağı serumu (FCS decomplemented% 10), 2 mM L-glutamin ve% 1 penisilin-streptomisin) ile değiştirmek için izin vermek için rekombinant insan makrofaj koloni uyarıcı faktör, 10 ng / ml'lik bir son konsantrasyonda (RHM-CSF) vardır.
- 11 gün sonra (Şekil 1),% 5 CO2 ile 37 ° C 'de inkübe hücreleri.
- 11. günde de orta kaldırmak ve oyuk başına soğuk hMDM ortam, 2 ml ile 2 kez yıkanır. Sonraki soğuk 1 x PBS, 1 ml her bir çukura 2 kez yıkayın.
- (2 mM ethylenediaminetetraacetatic asit ile, soğuk 1 x PBS, 1 ml her bir 1 kez yıkama, tam olarak farklılaşmış hMDMs ayırmak içinEDTA) ve 4 ° C'de 15-60 dakika boyunca oyuk başına 2 mM EDTA, soğuk 1 x PBS, 2 ml hücreleri inkübe edin.
NOT: Hücreler var ayırmak için alternatif yöntemleri (örneğin, tripsin veya tripsin benzeri faaliyetler) iyi sonuçlar 11 verebilir. - (Yukarı ve aşağı kuyuda hafifçe pipetleme tamamlandı) ayrıldıktan sonra, hücreleri toplamak ve soğuk hMDM orta 10 ml içeren buz üzerinde bir 50 ml tüp koyun.
- Santrifüj 20 ° C'de 5 dakika boyunca 218 x g'de hücreler soğuk hMDM ortamı 10 ml pelet tekrar süspansiyon ve Tripan Mavisi 1/20 seyreltilerek hücreleri sayın.
- 35 mm mikroskopi dereceli cam alt tabak başına 1 x 10 6 hMDMs hücreleri tohum ve 1 gün için% 5 CO2 37 ° C'de inkübe edin.
HIV-1 viral Hisse Senedi 2. Üretim ve Niceleme
Not: NLR4.3 HIV-1 Gag-iGFP (yeşil floresan protein) M. SCHINDL bir R5-tropik zarf, hediye taşıyanER 10 makrofajlan enfekte etmek ve gerçek zamanlı olarak enfekte olan hücreleri görmek için kullanılmaktadır.
- Ticari bir transfeksiyon reaktifi kullanılarak karşılık gelen proviral DNA 6 ug insan embriyonik böbrek 293T hücreleri (2 x 10 6, 100 mm çanak) transfeksiyonu ile viral stoklar üretir.
- hücreler ve sayılmasına yönelik bir beta-galaktosidaz renk ardından viral stoklar seri dilüsyonları kullanılarak (HIV-1 LTR kontrolü altında beta-galaktosidaz genini taşıyan) HeLa tzm-BL göstergesi hücreleri kullanılarak virüs stoklarının enfeksiyon niceliğini mavi hücreler 12.
HIV-1 ile hMDMs 3. Enfeksiyon
- hMDMs (1.13 kaplanmıştır hücreler) enfeksiyon (MOI) bir çokluğunda hMDM ortamının 0.02-0.03 1 ml virüs ekleyin. Kuyular hMDM ortamının sadece 1 ml ilave kontrol ve 2 gün (Şekil 1),% 5 CO2 ile 37 ° C 'de yemekler kuluçkalanması.
- günde 2 hMDM Med hücreleri yıkayın3 kez amonyum ve tabak başına taze hMDM ortamının 1 ml. 6 gün (Şekil 1),% 5 CO2 ile 37 ° C 'de inkübe hücreleri.
Koyu kırmızı kan hücrelerine 4. Opsonizasyon
- Tabak başına 7 x 10 6 SRBClere hazırlanması için, 4 dakika boyunca 600 x g'de santrifüj ile 1x PBS içinde% 0.1 Sığır Serum Albumin (BSA) ihtiva eden çözeltinin 100 ul SRBClere iki kez yıkayın.
- SRBClere 5 ul başına alt aglütinasyon konsantrasyonda tavşan IgG, anti-SRBClere PBS / BSA% 0.1 1 x 500 ul yıkandı SRBClere yeniden süspanse edin ve 30 dakika boyunca oda sıcaklığında dönme ile inkübe edilir.
Not: anti-SRBC'ler IgG alt aglütinasyon konsantrasyonunu belirlemek 96 oyuklu bir plaka içerisinde 20 ul 1 / 25.600 1/50 arasında (13.1 mg / ml stok konsantrasyonunda) IgG seri dilüsyonları hazırlanması. Her oyuktaki 20 ul 2 x 10 6 SRBClere ekleyin ve birkaç saat boyunca, karanlık bir odada tabak koydu. alt aglütinasyon konsantrasyonudurhemen önce iyice aglütinasyon (IgG + SRBC'ler bir ağ oluşturan) ile kuyu seyreltme. - Dönme sonra 4 dakika boyunca 600 x g'de IgG opsonize-SRBClere santrifüj ve 4 dakika için 600 x g'de santrifüj ile 1X PBS / BSA% 0.1, 100 ul ile yıkayın.
- 2 mM L-glutamin ve% 1 penisilin-streptomisin (1 ml / kutu) ile desteklenmiş önceden ısıtılmış fenol kırmızısız RPMI ortamında IgG opsonize-SRBClere yeniden süspanse edin.
5. Canlı Hücre video Mikroskopi Fagositoz Assay
- Bu CO2 nemlendirme su ile bir şişe içinden geçen 37 ° C'de bir ısıtma odası ile donatılmış bir döner disk sistemi gibi bir konfokal görüntüleme sistemi kullanın.
- fagositoz testinde başlangıcından önce 37 ° C 'de mikroskop aşamasında için deney öncesinde ısıtma odasına açın. mikroskop ve bilgisayarı açın ve görüntüleme yazılımı yükleyin.
- Böyle tarama hızı, Magnificat gibi görüntüleme ayarlarını optimizeiyon, çözünürlük, vs 60 ila 120 dakika arasında her dakika sahada başına ve görüntü tek bir karede en az bir hücre var.
NOT: Burada, örnek 63X lens ile 60 dakika boyunca bir kare her dakika görüntülü. - Mikroskop sahnede görüntüleme çanak yerleştirin. Odağı ayarlamak ve konum alanına sadece bir bütün HIV-1 ile enfekte makrofaj bulmak için. kullanılan görüntüleme sistemi ve prob göre uygun uyarma / emisyon ayarlarını kullanın. Fagozomların gözlemlemek için parlak bir alan (BF) kanalı ekleyin (Şekil 1ii). iletilen ışık ve maruz kalma süresi yüzdesini ayarlayarak farklı kanal görüntülerin görünümünü optimize edin.
NOT: Burada, NLR4.3 HIV-1 Gag-iGFP lazer% 20 (Şekil 1i) ile zaman maruz kalma 50 ms ile 491 nm lazer kullanarak heyecanlıydı. - Görüntüleme tabağı çıkarın ve çanağın 7 x 10 6 SRBC'ler / ml (Şekil 1) SRBC süspansiyonu 1 ml.
- 500 xg'de santrifüjOda sıcaklığında 2 dakika, fagositozu senkronize bu santrifüj sonunda zamanını kaydetmek ve sahneye çanak dönün.
- en az 1 saat süreyle her dakika - (genellikle 20 uçakları 0.3 mikron adım mesafe ile hücre kalınlığı boyunca) Z-yığınlarının içinde odaklama ve çekim GFP ve BF görüntüleri optimize. kullanılan görüntüleme sisteminin yerel dosya formatında zaman atlamalı video kaydetmek.
NOT: Burada, zaman atlamalı filmleri yerli formatında kaydedilen, * .stk dosyaları.
Time-lapse Filmler 6. Analizi
- video düzenleme için, menü "Uygulamalar" aşağı ve video düzenleme yazılımı sekmesi "Değerlendirme Çok Boyutlu Veriler" konulu damla tıklayın.
- Dosyayı açmak için, ardından "Dizin Seç" "Select Bankası Dosyası" üzerine tıklayın. "Veri setleri" kutusuna, (Kara Delikler formatında) analiz ve "Görünüm" tıklayın edinimi seçin. veri sütunu An zamanla bir tablo temsil edilmektedirdoğrultusunda d Z-planı.
- Z-projeksiyon (Şekil 2, sol panel) enfeksiyonu temsil etmek, "Dalgaboyları" kutusuna 491 nm dalga boyu seçmek ve "tüm uçakları" ile "Z projeksiyon" sekmesine tıklayın.
- (Bir zaman dizisini analiz "Dalgaboyları" kutusuna BF dalga boyunu seçin ve dış SRBClere (Şekil 2, kırmızı ok ucu), iç SRBC'ler (Şekil 2, kırmızı daire) ve çekirdeği ayırt etmek için Z-ekseni üzerinde optimum düzlemi seçmek için Şekil 2, mavi daire).
- video montajı kaydetmek için, "Seçim [X]" sekmesini ve ardından "Load Image (lar)" üzerine tıklayın. Son olarak, .tif formatta yüklü görüntüleri kaydetmek ve ImageJ yazılım sonraki açın.
- Çekirdeğin konumunu ve BF kanalda gözlenen farklı fagozomların (Şekil 3) ölçmek için ImageJ yazılım üzerinde eklenti "Manuel İzleme" kullanın.
- DowImageJ sitesinde "Manuel İzleme" eklentisi nload. ImageJ üzerinde (Şekil 3, aşama 1 ve 2) eklentisi ve görüntü dizisi analiz edilecek açın.
- Böyle komşu kareler arasındaki süreyi temsil eden "Zaman Aralığı", ve piksel başına mesafeyi temsil eden "x / y kalibrasyon" (Şekil 3, adım 3) gibi ayarları girin.
NOT: 63X zoom ve 6.45 x 6.45 mikron 2 piksel boyutunda bir kamera kullanıldığı için Burada, her kare ve 0.205 mikron x / y kalibrasyon arasında 1 dakikalık bir zaman aralığı ile görüntü dizisi kaydedin. - Izleme başlatmak için, (Şekil 3, adım 4) "Add track" üzerine tıklayın ve içselleştirildiği zaman ilk kez bir SRBC merkezinde tıklayın. Bir sonraki kare otomatik olarak görüntülenir.
- Süre içinde farklı pozisyonlarda (Şekil 3, kırmızı kutu içinde adım 6) sahip tüm karelerde SRBC merkeze tıklayın devam edin.
- kendi merkezinde tıklayarak bütün karelerde konumunu var çekirdeği izlemek için başlayın. Sonraki zaman SRBClere fonksiyonu farklı onların içselleştirme (kare), en tek tek (kendi merkezine tıklayarak) fagozomların izleyebilirsiniz. Kolaylık BF kanalda SRBC görmek için, izleme (Şekil 3, adım 5) sırasında "Parlaklık ve kontrast" penceresini kullanın.
- Her SRBC izleme arasında, yeni bir parça var (Şekil 3, adım 4) "Add track" üzerine tıklayın. Izleme sayısı ikinci sütunda değişmiş olacak, sonuç tablonun "Parça n °" (Şekil 3, adım 6).
- Analizin bir sonraki adıma devam etmek için bir e-tablodaki verileri kaydetmek.
- (Çekirdek ve SRBClere içselleştirilmesi sonra ilk 5 dakika boyunca fagozomların hızı doğru SRBClere içeren fagozomların bir seyahat mesafe hesaplamak için Fi tablo yazılımını kullanınşekil 4).
- tablo tablo ve yeni bir tablo dosyasını açın. Bu yeni Aşağıdaki parametreler sadece dosyaya, zaman, x-içine aktarın ve çekirdek ve tüm SRBClere (Şekil 4A) koordinatı y.
- Sadece kendi koordinatlar ile SRBClere ve çekirdek arasındaki mesafeyi hesaplamak XY koordinat düzleminde (Şekil 4B) çekirdeği ve SRBC dikkate alın.
Not: iki nokta arasındaki mesafe onları bağlayan yolun uzunluğudur. düzlemde, SRBC ve çekirdek arasındaki mesafe Pisagor teoremi ile verilir.- C (nukleus ve SRBC arasındaki mesafe) hipotenüs ve a uzunluğunu gösterir b diğer iki tarafın uzunluklarını göstermek durumunda, Pisagor denklemi olarak Pisagor teoremini ifade:

Not: Aynı zamanda, Ortonormal üzerinde yatay mesafe(x çekirdeği -x SRBC) ve dikey mesafe b (SRBC -y y çekirdeği) 'dir. - Böylece, çekirdek ve SRBC (Şekil 4A, mor kutu) ile arasındaki mesafeyi hesaplamak:
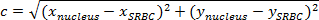
NOT: mikron mesafe olması x / y kalibrasyon (piksel) elde mesafe değeri çarpın. Burada, x / y kalibrasyon 0.205 mm. - Her zaman, çekirdek ve başlangıç mesafesi (Şekil 4C) için SRBC arasındaki mesafeyi çıkarın.
- İlk 5 dakika için zamana karşı ölçüm çizilir. Arsa doğrusal eğilim çizgisi (Şekil 5A) uygulayın ve SRBC içselleştirme sonra ilk 5 dakika boyunca çekirdeğin doğru fagosom hızını temsil doğrusal trend çizgisinin eğimini belirler.
- ortalama ve istatistiki hatayı hesaplamak için veri harmanlamakhız değerleri uygun yazılımı (Şekil 5B) kullanılarak uygun bir biçimde bunları çizmek ve.
- C (nukleus ve SRBC arasındaki mesafe) hipotenüs ve a uzunluğunu gösterir b diğer iki tarafın uzunluklarını göstermek durumunda, Pisagor denklemi olarak Pisagor teoremini ifade:
Sonuçlar
HIV-1 ile enfekte edilmiş ve enfekte olmayan hMDMs ile fagositozu FcR aracılı modeli hedefleri olarak IgG opsonize SRBClere (Şekil 1) kullanılarak burada tarif edilmektedir. Bu protokol kritik adım hMDMs hazırlanması ve HIV-1 enfeksiyonu bulunmaktadır. Gerçekten de, farklı makrofaj verim ve kalite donörler arasında değişir, ve% 10-40 aralığında verimliliği ile enfeksiyon oranı. Buna ek olarak, IgG-opsonize-SRBClere hazırlanması Bu, tanıma neden ve ...
Tartışmalar
Bu teknik pek çok kritik adımlar vardır. enfeksiyon yüzdesi donör bağımlı olduğundan İlk olarak, HİV-1 ile hMDMs hazırlanması ve enfeksiyon önemlidir. Potansiyel in vivo virüs tarafından karşılaşılan makrofajlar durumu iyi şimdiye kadar karakterize edilmemiştir, çünkü notun, biz, enfeksiyon öncesinde in vitro polarize olmayan makrofajlar kullanmaya karar verdik. Bu makrofajlar de M1 veya M2 olduğunu gösteren çok sayıda yüzey markerlerinin ifadesiyle kontrol ve CD4 ve CCR5...
Açıklamalar
The authors have nothing to disclose.
Teşekkürler
We thank Dr Jamil Jubrail for reading the manuscript. This work was supported by grants from CNRS, Inserm, Université Paris Descartes, Agence Nationale de la Recherche (2011 BSV3 025 02), Fondation pour la Recherche Médicale (FRM DEQ20130326518 including a doctoral fellowship for GLB) and Agence Nationale de Recherche sur le SIDA et les hépatites virales (ANRS, including a post-doctoral fellowship for CD) to FN. A. Dumas was supported by doctoral fellowships from Université Paris Descartes and Sidaction.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Falcon 100mm TC-Treated Cell Culture Dish | Corning | 353003 | For viral production |
| Glass Bottom Dishes 35 mm uncoated 1.5 | MatTek corporation | P35G-1.5-14-C Case | For acquisition |
| Falcon Tissue Culture Plates 6-well | Thermo Fischer Scientific | Corning. Inc. 353934 | For human monocyte-derived macrophages |
| Ficoll-Plaque PLUS | Dominique Dutscher | 17-1440-03 | a neutral, highly branched, high-mass, hydrophilic polysaccharide in solution for density centrifugation |
| DPBS, no calcium, no magnesium | Thermo Fischer Scientific | 14190-094 | Room temperature |
| Dulbecco's Modified Eagle Medium (DMEM) 1X, liquid (High Glucose) | GIBCO, Molecular probes | 31966-021 | Conserved at 4°C ; for HEK cells culture |
| RPMI 1640 medium GLUTAMAX Supplement | Life technologies | 61870-010 | Conserved at 4°C; for hMDMs culture |
| Fœtal Calf Serum (FCS) | Eurobio | CVFSVF0001 | Conserved at -20°C ; decomplemented |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fischer Scientific | 15140-122 | Conserved at -20°C ; for hMDMs culture |
| RPMI 1640 medium, no phenol red (10x500 ml) | Life technologies | 11835-105 | Warm in 37°C water bath before use ; for phagocytosis assay |
| FuGENE6 Transfection Reagent | Promega | E2692 | Conserved at 4°C ; for viral production |
| Sheep red blood cells (SRBCs) | Eurobio | DSGMTN00-0Q | Conserved in Alsever buffer at 4°C before use |
| Anti-sheep red blood cells IgG | MP Biomedicals | 55806 | Conserved at 4°C |
| Bovine Serum Albumin heat shock fraction, pH 7, ≥98% | Sigma | A7906 | Conserved at -20°C |
| Inverted microscope DMI600 | Leica | ||
| Confocal Spinning Disk Unit CSU-X1M1 | Yokogawa | ||
| 491 nm 50mW laser | COBOLT CALYPSO | ||
| HCX PL APO CS Objectif | Leica | Objective lens ; Magnification 100x ; Numerical aperture 1.40 ; Immersion oil | |
| CoolSnap HQ2 (FireWire) Camera | Photometrics | Pixel size 6.45 x 6.45 µm ; Definition 1392 x 1040 ; Encoding the image in 14 Bit | |
| Metamorph 7.7.5 software | Molecular Devices | For the control of the microscope | |
| GraphPad Prism software | For the statistics analysis |
Referanslar
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annu. Rev. Pathol. 7, 61-98 (2012).
- Niedergang, F. . Encyclopedia of Cell Biology. 2, 751-757 (2016).
- Blocker, A., Griffiths, G., Olivo, J. C., Hyman, A. A., Severin, F. F. A role for microtubule dynamics in phagosome movement. J Cell Sci. 111 (Pt 3), 303-312 (1998).
- Blocker, A., et al. Molecular requirements for bi-directional movement of phagosomes along microtubules. J Cell Biol. 137, 113-129 (1997).
- Flannagan, R. S., Cosio, G., Grinstein, S. Antimicrobial mechanisms of phagocytes and bacterial evasion strategies. Nat Rev Microbiol. 7, 355-366 (2009).
- Feasey, N. A., Dougan, G., Kingsley, R. A., Heyderman, R. S., Gordon, M. A. Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa. Lancet. 379, 2489-2499 (2012).
- Mazzolini, J., et al. Inhibition of phagocytosis in HIV-1-infected macrophages relies on Nef-dependent alteration of focal delivery of recycling compartments. Blood. 115, 4226-4236 (2010).
- Dumas, A., et al. The HIV-1 protein Vpr impairs phagosome maturation by controlling microtubule-dependent trafficking. J Cell Biol. 211, 359-372 (2015).
- Greenberg, S., el Khoury, J., Kaplan, E., Silverstein, S. C. A fluorescence technique to distinguish attached from ingested erythrocytes and zymosan particles in phagocytosing macrophages. J. Immunol. Methods. 139, 115-122 (1991).
- Koppensteiner, H., Banning, C., Schneider, C., Hohenberg, H., Schindler, M. Macrophage internal HIV-1 is protected from neutralizing antibodies. J Virol. 86, 2826-2836 (2012).
- Gartner, S. The macrophage and HIV: basic concepts and methodologies. Methods Mol Biol. 1087, 207-220 (2014).
- Wei, X., et al. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob Agents Chemother. 46, 1896-1905 (2002).
- Harrison, R. E., Bucci, C., Vieira, O. V., Schroer, T. A., Grinstein, S. Phagosomes fuse with late endosomes and/or lysosomes by extension of membrane protrusions along microtubules: role of Rab7 and RILP. Mol Cell Biol. 23, 6494-6506 (2003).
- Toyohara, A., Inaba, K. Transport of phagosomes in mouse peritoneal macrophages. J Cell Sci. 94 (Pt 1), 143-153 (1989).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır