Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Floresan Sinyallerini Etkin Bir Şekilde Ayırt Etmek Için Uyarma-Tarama hiperspektral görüntüleme mikroskobu
Bu Makalede
Özet
Spektral görüntüleme, birden fazla floresan sinyalin tek bir örnekte tanımlanması ve ayrılması için güvenilir bir çözüm haline gelmiştir ve ilgi sinyallerini arka plandan veya otofloresanstan kolayca ayırt edebilir. Uyarma-tarama hiperspektral görüntüleme aynı anda sinyal-gürültü oranını artırırken gerekli görüntü edinme süresini azaltarak bu tekniği geliştirir.
Özet
Çeşitli teknikler, fenomenleri tanımlamak veya incelemek veya işlevleri açıklamak için floresan sinyallerinin algılanmasına dayanır. Bu floresan sinyallerinin ayrılması, floresan kaynaklarının birbirlerinden, arka plan sinyalleri ve otofloresanlardan ayrılabildiği hiperspektral görüntülemenin ortaya çıkmasına kadar hantal olduğu kanıtlanmıştır (spektral bilgileri ne kadar dır? imzalar). Ancak, geleneksel, emisyon taramalı hiperspektral görüntüleme, hem uyarma hem de emisyon ışığının gerekli filtrelenmesi nedeniyle yavaş edinim süreleri ve düşük sinyal-gürültü oranlarından muzdariptir. Daha önce uyarma taramalı hiperspektral görüntülemenin gerekli kazanım süresini kısaltırken aynı zamanda elde edilen verilerin sinyal-gürültü oranını artırdığı gösterilmiştir. Ticari olarak kullanılabilen ekipmanı kullanan bu protokol, tek bir örnekteki birkaç floresan kaynaktan gelen sinyallerin ayrılması için uyarma taramalı hiperspektral görüntüleme mikroskobu sisteminin nasıl monte edilebildiğini, kalibre edilebildiğini ve kullanılacağını açıklar. Hücrelerin ve dokuların mikroskobik görüntülemesi için son derece geçerli olmakla birlikte, bu teknik, uyarma dalga boylarını değiştirmek mümkün olan floresan kullanan her türlü deney için de yararlı olabilir: kimyasal görüntüleme, çevre uygulamaları, göz bakımı, gıda bilimi, adli tıp, tıp bilimi ve mineraloji.
Giriş
Spektral görüntüleme çeşitli şekillerde yapılabilir ve çeşitliterimler1,2,3,4ile adlandırılır. Genel olarak, spektral görüntüleme en az iki mekansal boyut ve bir spektral boyutta elde edilen verileri ifade eder. Multispektral ve hiperspektral görüntüleme en sık dalga boyu bantlarının sayısı veya spektral bantların bitişik olup olmadığı ile ayırt edilir1. Bu uygulama için hiperspektral veriler, uyarma için kullanılan her bandpass filtresinin yarım maksimum (FWHM) tam genişliğinin yarısından az olmayan merkez dalga boylarının aralıkları ile elde edilen bitişik dalga boyu bantları ile elde edilen spektral veriler olarak tanımlanır (örn. 5 nm 14-20 nm bant genişlikli bandpass filtreler iã§in merkez dalga boyu sÃ1/4releri). Veri bantlarının bitişik yapısı, veri kümesinin aşırı örneklemesine olanak sağlayarak, spektral etki alanını örneklemede Nyquist ölçütlerinin karşılanmasını sağlar.
Hiperspektral görüntüleme NASA tarafından 1970'li ve 1980'li yıllarda ilk Landsat uydusu 5 ile birlikte geliştirilmiştir5,6. Birkaç bitişik spektral banttan veri toplamak, her pikselin parlak spektrumunun oluşmasına olanak sağladı. Tek tek bileşenlerin parlaklık spektrumunun tanımlanması ve tanımlanması, yüzey malzemelerinin karakteristik spektrumları tarafından algılanmasının yanı sıra, sinyaldeki değişimler gibi müdahale eden sinyallerin de atmosferik koşullar. Karakteristik spektrumlarını kullanarak malzemelerin saptanması kavramı 1996 yılında Schröck ve ark. beş farklı floroforve bilinen spektrumların kombinasyonlarını kullanarak etiketli kromozomları bir süreç olarak adlandırıldığında biyolojik sistemlere uygulanmıştır. spektral karyotipleme7. Bu teknik 2000 yılında Tsurui ve ark. tarafından doku örneklerinin floresan görüntülemesi için, yedi floresan boya ve tekil değer ayrışması kullanılarak, her pikselin referanstaki doğrusal spektrum kombinasyonlarına spektral ayrıştırma elde edilmesi için ayrıntılı olarak geliştirilmiştir. kütüphane8. Uzaktan algılama benzerlerine benzer şekilde, bilinen her floroforun katkısı hiperspektral görüntüden hesaplanabilir, her florofor spektrumunun priori bilgileri göz önüne alındığında.
Hiperspektral görüntüleme de tarım alanlarında kullanılmıştır9, astronomi10, biyotıp11, kimyasal görüntüleme12, çevresel uygulamalar13, göz bakımı14, gıda bilimi15, adli tıp16,17, tıp bilimi18, mineraloji19, ve gözetim20. Mevcut floresan mikroskop hiperspektral görüntüleme sistemlerinin önemli bir sınırlamastandart hiperspektral görüntüleme teknolojisi dar bantlarda floresan sinyalleri izole 1) ilk örnek uyarma kontrol etmek için uyarma ışığı filtreleme, daha sonra 2) daha fazla filtreleme daha sonra matematiksel olarak 21 ayrılabilir dar bantlarhalinde floresan emisyon ayırmak için yayılan ışık . Hem uyarma aydınlatmasını hem de yayılan floresanları filtrelemek, mevcut sinyal miktarını azaltır, bu da sinyal-gürültü oranını düşürür ve uzun edinme süreleri gerektirir. Düşük sinyal ve uzun edinme süreleri, hiperspektral görüntülemenin tanı aracı olarak uygulanabilirliğini sınırlar.
Hiperspektral görüntülemekullanan ancak mevcut sinyali artıran bir görüntüleme yöntemi geliştirilmiştir, böylece gerekli edinme süresini kısaltır21,22. Uyarma taramalı hiperspektral görüntüleme adı verilen bu yeni yöntem, uyarma dalga boyunu değiştirerek ve geniş bir yayılan ışık aralayarak spektral görüntü verilerini elde eder. Daha önce bu tekniğin emisyon tarama teknikleri21,22ile karşılaştırıldığında sinyal-gürültü oranı büyüklük artışları siparişleri verimleri gösterilmiştir. Sinyal-gürültü oranındaki artış büyük ölçüde tespit edilen geniş bantgeçişinden (~600 nm) kaynaklanırken, özgüllük floresan emisyon yerine sadece uyarma ışığının filtrelanmasıyla sağlanır. Bu, yayılan tüm ışığın (her uyarma dalga boyuiçin) dedektör21'e ulaşmasını sağlar. Ayrıca, bu teknik eksojen etiketlerden otofloresans ayırt etmek için kullanılabilir. Ayrıca, artan tespit edilebilir sinyal nedeniyle edinme süresini kısaltma yeteneği fotobeyazrlama tehlikesini azaltır yanı sıra spektral video görüntüleme için kabul edilebilir bir edinim hızında spektral taramaları sağlar.
Bu protokolün amacı, hiperspektral görüntüleme mikroskobu için bir veri toplama rehberi olarak hizmet vermektir. Buna ek olarak, ışık yolunu ve donanımı anlamanın yolunu anlamaya yardımcı olan açıklamalar da dahildir. Ayrıca açıklanan bir uyarma tarama hiperspektral görüntüleme mikroskobu için açık kaynak yazılım uygulamasıdır. Son olarak, sistemin NIST tarafından izlenebilir bir standarda göre nasıl kalibre edilemeyeceği, doğru sonuçlar için yazılım ve donanım ayarlarını ayarlama ve algılanan sinyali tek tek bileşenlerin katkılarına karıştırması için açıklamalar sağlanır.
Protokol
1. Cihaz kurulumu
- Işık kaynağı: yüksek güç çıkışı ve yüksek kolimasyon (bu çalışmalar için 300 W Xe ark lambası kullanılmıştır) ile geniş bant spektral ışık kaynağı seçin.
- Deklanşör (isteğe bağlı): hızlandırılmış görüntüleme için fotobeyaztma işlemini azaltmak için optik yola bir deklanşör ekleyin.
- Ayarlanabilir filtre sistemi: istenilen dalga boyu ayarlanabilir uyarma aralığını (örneğin, 360-485 nm) etkinleştirmek için mekanik bir ayar tertibatı ve ince film ayarlanabilir filtre (TFTF) seti içerir.
- Mikroskop: motorlu dikroik filtre tareti ve denetleyicisi içeren ters floresan mikroskobu kullanın.
- Floresan filtre küpü: uzun pas floresan filtre küpü monte edin. En iyi sonuçlar için, emisyon ışığından uyarma nın en iyi şekilde ayrılmasını sağlamak için aynı dalga boyunda bir dikroik ayna ve uzun pas emisyon filtresi seçin (örn. 495 nm).
- Amaç: deney için kullanılan dalga boyu aralığı üzerinde tek tip odak sağlamak için uygun bir apokromatik hedef kullanın (bu çalışma için 60x su hedefi kullanılmıştır).
- Otomatik sahne aşaması (isteğe bağlı): görüntü dikişi yoluyla birden çok görüş alanının ve/veya çok büyük alanların hızlı örneklemesi için otomatik bir aşama kullanın. Hedefi ve kamera ile sahne kalibre.
- Kamera: deneyin mekansal çözünürlük, hassasiyet ve gürültü gereksinimlerini karşılamak için uygun bir kamera seçin (bu sistem yüksek hassasiyetli sCMOS kamera kullandı).
2. Satın alma yazılımı
- Micro-Manager gibi her donanım bileşeninin bağımsız denetimine izin veren bir yazılım paketi seçin.
- Yapılandırma dosyasının oluşturulması: yapılandırma dosyası, Micro-Manager'ın sistemin donanımını çalıştırmak için yükleyeceği kaydedilmiş bir araç ve yönerge ler kümesidir. Yapılandırma dosyası oluşturmak için Araçlar > Donanım YapılandırmaSihirbazı'nı tıklatın.
- 1. adımda, devam etmek için yeni yapılandırma oluştur > Sonraki'ni tıklatın. Kullanıcının zaten kullanılabilir bir yapılandırmavarsa, varolan yapılandırmayı değiştirmeyi veya keşfetmeyi seçebileceğini unutmayın.
- 2. adımda, mikroskop, lamba, sahne, deklanşör ve kamera gibi Micro-Manager aracılığıyla kontrol edilebilecek aygıtları bulmak için Kullanılabilir Cihazlar listesine göz atın. Vurgulandığında Ekle'yi tıklatın... cihazı yüklenen aygıtlar listesine eklemek için düğmeye basın. Bu yeni bir pencere açar.
- Etiket kutusuna aygıtın etiketini belirleyin. (örn. sCMOS kamera)
- Değeraltında, her aygıt için uygun COM bağlantı noktasını seçin. Bu, aygıtın özelliklerilistesinin pencerenin alt kısmında görünmesine neden olur.
- Aygıt özelliklerini varsayılan ayarlarına bırakın. Her aygıt özelliği için özel değerlerin isteneildiği gibi girilebileceğini unutmayın.
- Her aygıt eklendiğinde, bir sonraki adıma devam etmek için İleri düğmesini tıklatın. Herhangi bir aygıt dışarıda bırakılırsa veya değiştirilmesi gerekiyorsa, yapılandırma dosyası nın daha sonra adım 2.1.1.1'de açıklandığı şekilde değiştirilebileceğini unutmayın.
- 3. adımda, varsayılan kamerayı, deklanşöre ve odak aşamasını seçmek için açılır menüleri kullanın. Otomatik deklanşör isteniyorsa, odak aşaması açılır listesinin altındaki Onay kutusunu tıklatın. Z-sahnenin odak yönü biliniyorsa, Aşama odak yönü (gelişmiş)altında açılan listeden seçin. Aksi takdirde, varsayılanı Bilinmeyenolarak bırakın.
- 4. adımda, Lamba, sahne ve deklanşör için 100 ms gecikmesi gibi otomatik cihazlarla ilişkili gecikmeleri ayarlamak için Gecikme [ms] başlığı altındaki kutuları çift tıklatın ve mekanik ayar tertibatına komutlar için 250 ms gecikme filtreler arasında mekanik geçiş süresi.
- Adım 5'te, durum aygıtları için etiket atayın. Uyarma taramalı mikroskop sisteminin yapılandırması, her floresan filtre küpünü tanımlamak için filtre bloğundaki etiketleri kullanır (örneğin, durum 0, parlakalan görüntüleme sırasında kullanılan boş filtre küpü; durum 3 etiket 495 nm ve özel 495 nm dikroik filtre küpü 495 nm uyarma ve emisyon ışığı ayırmak için kullanılır karşılık gelir. Etiketler, her uyarma dalga boyu için anahtarlama komutlarını depolamak için mekanik ayar tertibatında da kullanılır (örneğin, durum 0 mekanik ayar tertibatı için 340 nm uyarma dalga boyu ayar komutuna karşılık gelir).
- 6. adımda, yapılandırma dosyası için konum ve dosya adını kaydetmek için Yapılandırma dosya kutusunun yanındaki Gözat düğmesini tıklatın. Yapılandırma dosyasını kaydetmek için bitiş'i tıklatın.
- Başlangıç grupları ve hazır ayarlar: Micro-Manager, donanımın bir alt kümesini etkinleştirmek veya değiştirmek için her yapılandırma dosyasında farklı grupları depolayabilir. Örneğin, bir "sistem başlatma" grubu ve önceden ayarlanmış bir kombinasyon, kamera için binning boyutu ve okuma oranları gibi varsayılan ayarları saklar.
- Grup alt bölümündeki ana pencerede +'yı tıklatarak bir grup (örneğin, Sistem)oluşturun.
- Grupta herhangi bir kullanım kontrol edin? kümeedilmesi için varsayılan bir değer gerektirecek grup içindeki kutular (örneğin, ilişkili varsayılan kamerada binning).
- Önceden ayarlanmış alt bölümdeki ana pencerede +'yı tıklatarak yeni bir önceden ayar (örneğin, "Başlangıç") oluşturun.
- Adım 2'de işaretlenen her kutu burada önceden ayarlanmış bir seçenek olarak görünür. Denetlenen her kutu için yüklenecek varsayılan bir değer seçin.
- Uyarma dalga boyları için grup ve ön ayarlar: Micro-Manager her uyarma dalga boyunu kendi dalga boyu anahtarlama komutu ile kendi kanalı olarak ele alır; bu nedenle, her biri kendi önceden ayarlanmış olarak kaydedilmelidir.
- İstenilen uyarma dalga boylarını içeren yeni bir grup oluşturun (örn. "495 nm dichroik")
- Grupta Kullanım'ı kontrol edin?
- Her uyarma dalga boyu için yeni bir önceden ayar oluşturun (örneğin, "340 nm").
- Her önceden ayarlanmış özellik adını ve önceden ayarlanmış değeri (örneğin, özellik adı: "Etiket"; önceden ayarlanmış değer: "340 nm") atayın.
- Çok boyutlu edinme aracı: tıklayınMulti-D Acq.Micro-Manager penceresinin sol üst kısmında tek bir düğmeyi tıklatarak her uyarma dalga boyunda örnek bir görüntü yakalamak için kullanmak için özelleştirilebilir bir araç açmak için yakın. Edinmeyi tam olarak istediğiniz gibi oluşturmak için çeşitli özelleştirme seçenekleri vardır.
- Zaman puanları (bu örnekte kullanılmaz): zaman atlamalı bileşeni olmayan çalışmalarda kutuyu işaretsiz bırakın. Zaman atlamalı çalışmalar isteniyorsa, bu kutuyu işaretleyin. İşaretlenirse, edinme ayarlarının geri kalanını Numara kutusuna gerçekleştirmek için kaç kez girin. Aralık kutusuna süre girerek ardışık satın almalar arasındaki süreyi ayarlayın ve uygun birimi (milisaniye, saniye veya dakika; genellikle saniye) seçin.
- Birden çok pozisyon (XY; bu örnekte kullanılmaz): tek bir XY pozisyonunun çalışmaları için kutuyu işaretsiz bırakın. Birden çok XY konumu isteniyorsa, kutuyu işaretleyin ve ayrı bir pencere açmak için konum listesini edin düğmesini tıklatın. Sahneyi istenilen her konuma taşıyın ve bu konumu yazılımda kaydetmek için İşaretle düğmesini tıklatın. İstenilen tüm XY pozisyonları işaretlenene kadar tekrarlayın. Devam etmek için bu pencereyi kapatın.
- Z yığınları (slaytlar; bu örnekte kullanılmaz): Tek bir Z konumunun çalışmaları için kutuyu işaretsiz bırakın. Birden çok Z pozisyonu isteniyorsa, kutuyu işaretleyin. Sahneyi istenilen başlangıç Z konumuna taşıyın ve Z başlat kutusunun yanındaki Ayarla düğmesini tıklatın. Sahneyi istenilen bitişz konumuna taşıyın ve Z sonu kutusunun yanındaki Ayarla düğmesini tıklatın. Z-adım kutusuna istediğiniz adım boyutunu (mikronlarda) girin.
- Kanallar: Kanallar kutusunun işaretli olduğundan emin olun. Burada, kanallar bireysel uyarma dalga boylarına verilen adlardır. Kullanılabilir "Kanallar", 2.1.2 ve 2.1.3 adımlarında açıklanan "Gruplar"a karşılık gelir.
- Grup: kullanılabilir uyarma dalga boylarını seçmek için bir grup seçmek için açılır listeyi tıklatın (örn. 495 nm dikroik).
- Her kanal için satın almalar arasında deklanşör açık tutmak için Deklanşör Açık tut kutusunu tıklatın. Bu kutu işaretlenirse deklanşör, ana pencerede otomatik panjur seçeneğinin de seçildiğini varsayarak, deklanşöre ardışık spektral taramalar arasında kapanmaya devam edeceğini unutmayın.
- Satın alma listesine kanal eklemek için Yeni düğmesini tıklatın. Kaldır düğmesiartık istenmemiş kanalları kaldırmak için kullanılabilir ve seçili herhangi bir kanalı yeniden sıralamak için Yukarı ve Aşağı kullanılabilir.
- Spektral tararakta istenilen her dalga boyu için Kullanım? onay kutusunun seçildiğinden emin olun.
- Yapılandırma: Configuration altında açılan listeye tıklayın ve istenilen spektral aralıktaki ilk dalga boyunu seçin (örn. 340 nm).
- Pozlama: Pozlama altındaki kutuyu çift tıklatın ve seçilen dalga boyu için istenen pozlama süresini girin (örn. 100 ms için "100"). Uygun pozlama sürelerini seçmek için öneriler için bölüm 5'e ("Veri Toplama") bakın.
- Z-ofset (spektral tama geçerli değildir): spektral bir tama yaparken bu kutuyu boş (0'da) bırakın.
- Z-stack: Z-stack performans ise spektral aralıktaki her dalga boyu için bu kutunun işaretli olduğundan emin olun.
- Skip Fr. (spektral tbmkte geçerli değildir): spektral tbmktür yaparken bu kutuyu boş (0'da) bırakın. Bir veya birkaç uyarma dalga boylarının diğerlerinden önemli ölçüde daha güçlü olması veya bireysel uyarma dalga boylarının özellikle fototoksik olması durumunda kareleri seçici olarak atlamanın yararlı olabileceğini unutmayın.
- Renk: Spektral talan yaparken bu kutuyu boş bırakın. Renklerin veri görselleştirme amacıyla tek tek dalga boyu bantları için seçilebileni, ancak rengin sonraki spektral karıştırma için uygun olmadığını unutmayın.
- Verilen spektral tarama aralığı içinde istenilen her uyarma dalga boyu eklenene kadar bu adımları tekrarlayın.
- Edinme sırası: Yukarıdaki seçeneklerin (2.1.4.1-2.1.4.5) hangi sırada gerçekleştirileceğini seçmek için açılır listeye tıklayın. Burada gösterilen gibi spektral taramalar için, satın alma sırası sadece Kanal. Çok Boyutlu Edinme aracı [zaman noktaları, Çoklu Konumlar (XY) ve Z yığınları (dilimler)] içinde ek kutular işaretlenirken ek seçeneklerin göründüğünü ve önce konum veya zaman seçimine izin verdiğini ve ardından dilim veya Kanal.
- Otomatik netleme (bu örnekte kullanılmaz): Birden Çok Pozisyon (XY) seçilirse, birkaç farklı otomatik netleme stili kullanılabilir. Seçenekler düğmesini tıklatın ve kapat düğmesinin yanındaki açılır listeden bir otomatik netleme yöntemi seçin.
- Özet: listelenen bilgilerin beklenen edinme ayarlarıyla eşleştiğinden emin olmak için zaman puanı, XY pozisyonları, Z dilimleri, kanallar, toplam görüntü sayısı, toplam bellek, tetkik süresi ve edinme siparişinin özeti için bu pencereyi gözden geçirin.
- Görüntüleri kaydet: Çok Boyutlu Edinme aracının kullanımı yla toplanan verileri kaydetmek için bu kutunun işaretli olduğundan emin olun.
- Tıklayın ... dosyaların kaydedileceği bir dizin kökü seçmek için Dizin kökünü yanındaki düğme. Dizin kökünü, deneyin ilgili ayrıntılarını açıklayan bir moda olarak adlandırın (örneğin, GCaMP Airway Düz Kas Hücreleri).
- Geçerli görüntü edinimini açıklayan Ad ekinin yanındaki kutuya bir ad girin (örn. FOV1_100ms_60X_495nmDichroicFilter). Görünüm alanını ve pozlama süresini tanımlayan bir ad önekinin yanı sıra kullanılan nesne ve dikroik filtre gibi diğer ilgili bilgileri oluşturman tavsiye edilir. Bu ayarlar kullanılarak çekilen ilk görüntü yığınının ad önekinden sonra "_1" ile kaydolacağını unutmayın. Aynı dizin kökü ve ad öneki ile alınan sonraki yığın "_n" ile kaydedilir ve dizinde bu adla "n" bir yığının kaç kez alındığı kaydedilir.
- Her uyarma dalga boyunda oluşturulan görüntünün kaydedilmesini sağlamak için Ayrı görüntü dosyalarını tıklatın.
- Kaydet: Bu edinme ayarlarını kolay gelecekte kullanmak üzere kaydetmek için Çok Boyutlu Edinme aracının sağ üst kısmındaki "Olarak Kaydet" düğmesini tıklatın. Dizin kökü ve ad öneklerinin de kaydolacağını unutmayın.
- Edinme: son olarak, Edinme tıklayın! düğmesini yukarıda seçilen edinme ayarlarına göre görüntüleri edinmeye başlamak için.
- Yapılandırma dosyasının oluşturulması: yapılandırma dosyası, Micro-Manager'ın sistemin donanımını çalıştırmak için yükleyeceği kaydedilmiş bir araç ve yönerge ler kümesidir. Yapılandırma dosyası oluşturmak için Araçlar > Donanım YapılandırmaSihirbazı'nı tıklatın.
3. Spektral yanıt düzeltme (isteğe bağlı):
- Spektral çıkış düzeltmesi, sistemin spektral yanıtını nist izlenebilir lamba veya diğer NIST izlenebilir standart veya alet gibi bilinen bir standarda kalibre etmek için gerçekleştirilebilir. Sonuçlar diğer spektral görüntüleme cihazları, spektrometreler veya farklı laboratuvarlar arasında karşılaştırıldığında bu adım özellikle önemlidir. Bu işlem daha önce ayrıntılı olarak bildirilmiştir21,23.
- Örnek aşamasında ölçülen aydınlatmanın spektroradyometrik gücünü elde etmek için NIST izlenebilir Bir ışık kaynağına (LS-1-CAL-INT, Ocean Optics gibi) veya diğer NIST izlenebilir standartta kalibre edilmiş bir spektrometre kullanın. Işık kaynağı ayarı, dikroik ayna ve objektif her kombinasyonu için ayrı bir düzeltme gerçekleştirin. Mikroskop hedefinden geniş açı aydınlatmayı doğru bir şekilde ölçmek için spektrometreye bağlı entegre bir küre kullanın.
- Spektroradyometrik verileri ışıklı dalga boylarına entegre etmek için yamuk kuralı gibi bir tümleştirme yöntemi kullanın. Orta dalga boyu etrafında ortalanmış entegrasyon için 40 nm bant genişliği, nominal bant genişliği 14-20 nm tam genişliğe sahip çoğu filtre için yarım maksimum (FWHM) yeterlidir. Entegre değer, her uyarma dalga boyu bandındaki spektral aydınlatmanın yoğunluğunu temsil eder.
- Spektral aydınlatma yoğunluğu profilinin görüntülenmesi için her dalga boyu bandının tümleşik yoğunluğunu uyarma merkezi dalga boyu işlevi olarak çizin.
- En düşük entegre yoğunluğa sahip dalga boyu bandını belirleyin.
- Dalga boyuna bağlı düzeltme faktörü oluşturmak için en düşük entegre yoğunluğu her dalga boyu bandının entegre yoğunluğuna bölerek spektral aydınlatma yoğunluğu profilini normalleştirin.
4. Örnek hazırlama
- Bir "boş" örnek hazırlayın (örneğin, hücre odasına cam bir kapak kaydırın ve 1 mL arabellek ekleyin). Bu arabellek daha önce24açıklanmıştır.
- Herhangi bir örnek otofloresans belirlemek için etiketlenmemiş bir örnek hazırlayın (örneğin, etiketsiz hava yolu düz kas hücreleri içeren bir cam kapak kaymayerleştirin25,hücre odasına26 ve tampon 1 mL ekleyin).
- Denemede kullanılan her floresan etiket için ayrı, tek etiketli bir örnek hazırlayın:
- 100 nM konsantrasyonu elde etmek için tampon seyreltilmiş mitokondriyal etiket ekleyin. Hava yolu düz kas hücreleri içeren coverslip bu tampon ekleyin. 20-25 °C'de 20 dk kuluçka. Kapak kaymasını hücre odasına taşıyın ve 1 mL arabellek ekleyin. Mitokondriyal etiketin en uygun konsantrasyonunun üretici, ilişkili renk ve istenilen özgüllük gibi faktörlere göre değişeceğini unutmayın.
- GCaMP probu27 ile transfected hava yolu düz kas hücreleri içeren bir coverslip hücre odasına taşımak ve tampon 1 mL ekleyin.
- İstenilen floresan etiketlerin karışımını içeren bir veya daha fazla deneysel numune hazırlayın.
- GCaMP probu ile transfected hava yolu düz kas hücreleri içeren bir coverslip tampon (100 nM mitokondriyal etiket 1 00 00 nM konsantrasyonu) ekleyin ve 20-25 °C'de 20 dakika kuluçka. Kapak kaymasını hücre odasına taşıyın ve 1 mL arabellek ekleyin.
5. Veri toplama:
- Uygun dikroik ışın bölücü (örneğin, 495 nm dikroik filtre küpü), objektif (örn. 60x su hedefi) ve kameranın (sCMOS kamera) seçilip seçilmediğini kontrol edin.
- Örneği sahneye yükleyin.
- Pozlama [ms] yanındaki kutuyu çift tıklatın ve pozlama süresini 100 ms olarak ayarlamak için "100" yazın.
- İlk örnek görüntüleme için Micro-Manager ana penceresinin açılır menüsünden 475 nm seçin. 475 nm'nin örnek görüntüleme veya görüntü yığını boyunca aşırı doygunluk olup olmayacağını belirlemek için en uygun dalga boyu olmayabileceğini unutmayın.
- Örneği görüntülemek için Canlı'yı tıklatın.
- En küçük ve maksimum değerleri anlamlı görsel aralıklara getirmek için pencerenin altındaki histogramın yanındaki otomatik yoğunluk izleme aralığı düğmesi Otomatik'i tıklatın.
- Numuneye odaklanmak için mikroskobun odak tonlarını kullanın. Genellikle odaklama yardımcı olmak için örnek kenarını bulmak için yararlıdır. Görüntüdeki kenar özellikleri keskin göründüğünde örnek odaklanır. Floresan görüntüsünü görüntülemeye yardımcı olmak için odaklama sırasında Otomatik düğmesini birkaç kez tıklatmanız gerekebileceğini unutmayın. Ayrıca, mikroskop iletim modundaysa, bazı kullanıcılar örneğe odaklanmayı daha kolay bulabilir.
- İletim modunda, güvenlik için, öncelikle iletim görüntüleme için ışık yolunu ayarlamadan önce spektral ışık kaynağının göz merceğe ışık iletmediğini sağlayın. Ayrıca iletim ve floresan görüntülerin odak arasında küçük sapmalar olabileceğini unutmayın. Kabul edilebilir bir odak elde edildiğinde, spektral floresan görüntüleme için ışık yolunu yeniden yapılandırın.
- Multi-DAcq'yi tıklatın. çok boyutlu edinme aracını açmak için pencerenin sol üst kısmına yakın düğme (bölüm 2'de açıklanan ve yapılandırılmış).
- Çok Boyutlu Edinme aracı penceresinin sağ üst kısmındaki Load... düğmesine tıklayarak ve uyarma ayarlarına yönlendirerek spektral edinim için uygun bir spektral aralık seçin (örn. 495 nm dikroik filtre için 360-485 nm daha önce kaydedilmiştir (adım 2.1.4.10). Uygun spektral aralıklarla ilgili bilgi için tartışmaya bakın.
- Arka plan ve gürültü çıkarma için kullanılacak floresan veri içermeyen bir arka plan/boş görüntü edinin. Bu, boş bir örnek (adım 4.1) kullanılarak veya deneysel bir örneğin boş bir bölgesine yönlendirilerek gerçekleştirilebilir. Adım 5.6'da açıklandığı gibi, bu en kolay şekilde örnek kenarını bularak elde edilir ve böylece kenar görüş alanı içinde ortalanır.
- Edinme ayarları onaylandıktan ve arka plan bölgesi görünür olduğunda, daha sonra çıkarma için kullanılacak arka plan ve gürültü içeren bir spektral görüntü yığını elde etmek için Satın Al! düğmesini tıklatın. Örneklerin numunenin kenarından uzakta daha yoğun floresan olması muhtemeldir. "En parlak" bölgeleri bulmak ve uygun edinme sürelerini belirlemek ve aşırı pozlamayı önlemek için birkaç test görüntü yığını gerçekleştirmek için örnek hakkında hareket etmek tavsiye edilebilir.
- Dalga boylarının hiçbir kombinasyonunun ve pozlama sürelerinin aşırı pozlamayla sonuçlanmadığından emin olmak için, numunenin yoğun floresangibi görünen bir bölgesi üzerinde tek bir görüntü yığını alın.
- Hiçbir dalga boylarının aşırı pozlanmış piksel içermediğini doğrulamak için ImageJ'i kullanın.
- ImageJ'de Dosya > Görüntü Sırası 'nı tıklatın ve adım 5.10'da alınan spektral görüntüleri içeren klasöre gidin. Bu yeni bir pencere açar. Görüntü Sayısı'nın yanındaki kutuyu tıklatın ve spektral taramada yer alan dalga boylarının sayısını girin (örn. 26). Başlangıç görüntüsünü ve Artış'ı "1" olarak bırakın. Devam etmek için Tamam düğmesini tıklatın.
- ImageJ'in ölçüm işlevini kullanmak için klavyedeki M tuşuna basın. Max altında listelenen numaranın kameranın üst algılama sınırı olmadığından emin olun (örn. 65.535). Spektral tarayan her görüntü için tekrarlayın. Kameranın algılama limitinin kameranın kendisine bağlı olduğunu unutmayın. Üst sınır MicroManager ana penceresinde histogramın sağ üst kısmında görüntülenir. Alternatif olarak, üst sınır ImageJ'de bir görüntü açılarak ve Görüntü > Ayarla > Parlaklık/Kontrast'ayönlendirilerek belirlenebilir. Açılan pencerede Ayarla düğmesini tıklatın, maksimum görüntülenendeğerin yanındaki boş akalına aşırı büyük bir değer (örneğin, 999.999) girin ve devam etmek için Tamam'ı tıklatın. Yeni histogramda görüntülenen maksimum değer kameranın üst algılama sınırı olmalıdır.
- Herhangi bir görüntü kameranın üst algılama limitini içeriyorsa (örn. 65.535), spektral aralıktaki maksimum sinyalin kameranın dinamik aralığını aşmadığından emin olmak için spektral tarayın pozlama süresini ayarlayın.
- Herhangi bir otofloresans belirlemek için etiketlenmemiş örneklerden spektral görüntü verileri elde edin. Denemenin geri kalanı olarak aynı dalga boyu aralığı ve kamera ayarlarını kullanarak etiketlenmemiş numunelerin tarar'ını edinin (örneğin, dalga boyu 100 ms'de 360-485 nm).
- Spektral kitaplığı oluşturmak için spektral denetim olarak kullanmak üzere tek etiketli örneklerden spektral görüntü verileri elde edin. Aynı dalga boyu aralığını, pozlama süresini ve kamera ayarlarını kullanarak her etiket için taramaya devam edin. Bazı örneklerin minimum gürültü katkısıyla doğru etiket spektrum algılamasına izin vermek için daha uzun bir edinme süresi gerekebileceğini unutmayın.
- Deneysel bir örnek üzerinde ölçümler yapın (örneğin, GCaMP probu ve mitokondriyal etiketli insan hava yolu düz kas hücreleri).
- Deney örneğini içeren bir slayt veya kapak fişi mikroskoba yerleştirin.
- Dokuların uygun şekilde etiketlenmiş hücreleri ile bir görünüm alanı seçin.
- Yukarıda açıklandığı gibi, istenilen edinme ayarlarını kullanarak spektral görüntü verilerini edinin.
6. Görüntü analizi
- Görüntüleri düz bir spektral yanıta doğru.
- Bölüm 5.9'da elde edilen arka plan spektrumu çıkarın ve bölüm 3.1.5'te belirlenen düzeltme katsayısı ile çarpın. Bu basit bir MATLAB komut dosyası veya ImageJ yordamı ile yapılabilir. Güney Alabama Üniversitesi Biyogörüntüleme Kaynakları web sitesinde bir çıkarma/düzeltme MATLAB kodu (bu örnekte kullanılır) mevcuttur.
- Çıkarma/düzeltme kodunu MATLAB'a yükleyin.
- EDITOR sekmesinde Çalıştır'ı tıklatın. Bu, Bsq Düzeltme düğmesi içeren yeni bir pencere açar. Çalışma dizini, arka plan dosyası, düzeltme faktörü dosyası ve düzeltilmemiş tiff klasörü seçeneklerini içeren başka bir pencereyi açmak için Bsq Düzeltme düğmesini tıklatın.
- Gözat'ı kullanın... sonraki pencerelerin açılacağı dosya yolunu seçmek için Çalışma Dizini yanındaki düğme. Kaydedilen arka plan spektrumu içeren klasöre gidin (.dat biçiminde).
- Gözat'ı kullanın... adım 6.1.1.2.1'de seçilen dizini açmak ve istenen arka plan dosyasını seçmek için Arka Plan Dosyası (.dat) yanındaki düğmeyi (.dat formatında). Devam etmek için Aç düğmesini tıklatın.
- Düzeltme faktörü (.dat) yanındaki Gözat... düğmesini kullanarak bir düzeltme faktörü seçin. Düzeltme faktörü için dizin, üst MATLAB kodunun konumuna varsayılan olarak verilir. Gerekirse, düzeltme faktörü dosyasını içeren alt klasöre gidin (.dat biçiminde). Uygun düzeltme faktörü dosyasını vurgulayın ve devam etmek için Aç düğmesini tıklatın.
- 6.1.1.2.1 adımda seçilen dizini açmak için Düzeltilmemiş Tiff Klasörü yanındaki Gözat... düğmesini kullanın ve arka plan çıkarma ve görüntü düzeltme için klasörü seçin (bu genellikle hemen ham içeren Pos0 klasörüdür spektral görüntüler). Devam etmek için Aç düğmesini tıklatın.
- Spektral Düzeltme butonuna tıklayın. İşlem tamamlandıktan sonra "Bsq Düzeltme Tamamlandı" iletisiyle bir pencere açılır. Devam etmek için Tamam düğmesini tıklatın. Düzeltme görüntüleri, orijinal ham spektral görüntü klasörü ve ek bir "_Düzeltilmiş" (örneğin, Pos0_Düzeltilmiş) ile birlikte yeni bir klasörde depolanır.
- Bölüm 5.9'da elde edilen arka plan spektrumu çıkarın ve bölüm 3.1.5'te belirlenen düzeltme katsayısı ile çarpın. Bu basit bir MATLAB komut dosyası veya ImageJ yordamı ile yapılabilir. Güney Alabama Üniversitesi Biyogörüntüleme Kaynakları web sitesinde bir çıkarma/düzeltme MATLAB kodu (bu örnekte kullanılır) mevcuttur.
- Spektral kitaplığı oluşturun. Çeşitli yazılım paketleri ImageJ de dahil olmak üzere görüntülerden spektral veri ayıklamak için kullanılabilir. En iyi sonuçlar için, hangi dalga boyu bandının en yoğun değere sahip olduğunu belirleyerek ve her dalga boyu bandındaki ölçümü bu maksimum değere bölerek her bir son üyeyi en yüksek yoğunluktaki dalga boyu bandına normalleştirin. Ayrıca otofloresan numuneler içinde oluşturulan tek etiketli kontroller ek değişiklikler 28,29,30,31,32gerekir unutulmamalıdır. Ayrıntılar için adım 6.2.4 ve tartışma ya da bkz.
- ImageJ'de Dosya > İçe Aktar > GörüntüSırası'nı tıklatın. Bu yeni bir pencere açar. Düzeltilmiş spektral görüntüleri içeren klasöre gidin. Yeni bir pencere açmak için klasördeki herhangi bir resim dosyasına çift tıklayın. Görüntü Sayısı'nın yanındaki kutuyu tıklatın ve spektral taramada yer alan dalga boylarının sayısını girin (örn. 26). Başlangıç görüntüsünü ve Artış'ı "1" olarak bırakın. Devam etmek için Tamam düğmesini tıklatın.
- ImageJ ana penceresinde, Çokgen seçim simgesini tıklatın ve ardından floresan verileri içeren bir bölgenin etrafında çokgen oluşturmak için görüntüye tıklayarak fare işaretçisini kullanın. İlk dalga boyunda çok az floresan verisi olabileceğini unutmayın. Floresan verileri içeren bölgeleri daha iyi görselleştirmek için başka bir dalga boyu görüntüsüne gidin ve/veya parlaklık/kontrast aracını (Resim> Ayarla > Parlaklık/Kontrast >Otomatik) kullanın.
- Çizilen çokgenile, Resim > Yığınlar > Plot Z ekseniProfili'ni tıklatarak Z ekseni profilini oluşturun. Bu yeni bir pencere açar. Spektral verileri .csv dosyası olarak kaydetmek için Kaydet'i tıklatın.
- Otomatik floresan kontaminasyonundan yoksun saf etiketlere sahip uygun bir spektral kitaplık sağlamak için bir çıkarma gerçekleştirin. Tek etiketli ve floresan karışımından saf bir spektrum ayırmak için aşağıdaki denklem 29'u kullanın:
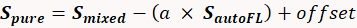 (1)
(1)
Nerede: Ssaf etiketsaf spektrum, Skarışık örnek otofloresan ile kontamine ölçülen spektrum, S autoFL ölçülen otofloresan spektrumu, "a" bir skaler olduğunu otofloresan spektrumunun çarpanı (0 ile 1 arasında; örneğin 0,4) ve "ofset" bir ofsettir (genellikle 0). Otofloresanlığın belirgin katkısı için sıfıra yakın bir değer elde edilene kadar "a" terimini değiştirin. Ek bilgi çeşitli yayınlar da bulunabilir28,29,30,31,32.
- Spektral görüntü verilerini karıştırın. Karıştırmayan adım, her floresan etiket için bir bolluk görüntüsü oluşturur, burada bolluk ilgili etiketten görüntüdeki göreli floresan sinyali miktarıdır. Birkaç unmixing algoritmaları ImageJ33,34,35ile kullanılabilir. Ayrıca, güney Alabama Üniversitesi Biyogörüntüleme Kaynakları web sitesinde spektral unmixing MATLAB kodu (bu örnekte kullanılır) mevcuttur. Spektral unmixing daha kapsamlı bir bakışmevcuttur 36.
- Spektral karıştırma kodunu MATLAB'a yükleyin.
- Wavelength.mat ve Library.mat dosyalarını deneysel koşullara karşılık gelecek şekilde (örneğin, "Dalga boyu" 5'lik artışlarla 360-485 olarak) edin.
NOT: Bu dalga boyları için karşılık gelen değerler her floresan etiket (ve otofloresan) için "Kitaplık"a yüklenmelidir. Endmember_Name etiketleri "Kitaplık" sütun değerleriyle aynı sırada listelemelidir. Library.mat dosyasının hem "Kitaplık" hem de "Endmember_Name" değişkenlerini içermesi gerektiğini unutmayın. - EDITOR sekmesinde Çalıştır'ı tıklatın. Bu, kullanıcının bir dizin seçmesine izin veren yeni bir pencere açar. Karıştırılmamış spektral görüntüleri içeren klasöre gidin. Seçildikten sonra, bu yeni bir pencere açar.
- Ortaya çıkan boşluklarda, görüntü adını (örneğin, GCaMP ve Mito FOV1 ile HASMC), dalga boyu bantlarının sayısını (örneğin, 26), zaman noktası sayısını (örneğin, 1) ve fret ölçümünün kendi boşluklarında istenip istenmediğini (örn. n) girin. FRET için "n" seçilirse, her iki Son Üye boş boş bırakın. Yeni bir pencere açacak devam etmek için Tamam'a basın.
- Wavelength.mat dosyasını içeren klasöre naviagate. Seçildikten sonra, yeni bir pencere açacak devam etmek için Aç'ı tıklatın.
- Library.mat dosyasını içeren klasöre gidin. Seçildikten sonra, spektral görüntüleri içeren dizinin içindeki yeni bir "Karışık Olmayan" klasöründe karışık olmayan görüntüleri kaydedecek olan devam etmek için Aç'ı tıklatın.
- Kalite için karışık spektral görüntüleri inceleyin.
- Saf bileşenlerin dağılımını görsel olarak incelemek için her karışık olmayan görüntüyü açın.
- Hata görüntüsünün büyüklüğünü karıştırılmamış görüntülerinkilerle karşılaştırın (adım 5.11'e benzer şekilde, bu ImageJ'in ölçü işlevi aracılığıyla yapılabilir). Hata görüntüsünün büyüklüğü karışık olmayan bolluk görüntülerine benzerse, spektral görüntü verilerinde spektral kitaplık ve doğrusal spektral karıştırma işlemi tarafından doğru bir şekilde hesaba katılmayan sinyaller olması olasıdır.
- Tanımlanamayan artık yapılar olup olmadığını görmek için hata görüntüsünü karışık olmayan görüntülerle karşılaştırın.
- Saf bileşenlerin dağılımını görsel olarak incelemek için her karışık olmayan görüntüyü açın.
Sonuçlar
Bu protokolden birkaç önemli adım, hem doğru hem de görüntüleme ve spektral eserlerden yoksun veri toplanmasını sağlamak için gereklidir. Bu adımları atlamak, önemli görünen ancak başka bir spektral görüntüleme sistemiyle doğrulanamayan veya çoğaltılamayan verilere neden olabilir ve böylece söz konusu verilerle elde edilen sonuçlar etkin bir şekilde geçersiz kılınabilir. Bu önemli adımlar arasında baş uygun spektral çıkış düzeltme (bölüm 3). Düze...
Tartışmalar
Uyarma taramalı hiperspektral görüntüleme kurulumunun en iyi kullanımı ışık yolunun inşası ile başlar. Özellikle, ışık kaynağı seçimi, filtreler (tunable ve dichroic), filtre anahtarlama yöntemi ve kamera mevcut spektral aralığı, olası tarayın hızı, dedektör hassasiyeti ve mekansal örnekleme belirler. Cıva ark lambaları birçok uyarma dalga boyu zirveleri sunuyoruz ama düz bir spektral çıkış sağlamaz ve bir NIST izlenebilir yanıt geri spektral görüntü verilerini düzeltmek için ?...
Açıklamalar
Dr. Leavesley ve Rich, spektral görüntüleme teknolojisini ticarileştirmek için kurulan spectracyte LLC şirketinin finansal çıkarlarını ifşa etmektedir.
Teşekkürler
Yazarlar NSF 1725937, NIH P01HL066299, NIH R01HL058506, NIH S10OD020149, NIH UL1 TR001417, NIH R01HL137030, AHA 18PRE34060163 ve İbrahim Mitchell Araştırma Fonu'ndan destek almak istiyorum.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Airway Smooth Muscle Cells | National Disease Research Interchange (NDRI) | Isolated from human lung tissues obtained from NDRI | Highly autofluorescent, calcium sensitive cells |
| Automated Shutter | Thorlabs Inc. | SHB1 | Remote-controllable shutter to minimize photobleaching |
| Automated Stage | Prior Scientific | H177P1T4 | Remote-controllable stage for automated multiple field of view or stitched image collection. |
| Automated Stage Controller (XY) | Prior Scientific | Proscan III (H31XYZE-US) | For interfacing automated stage with computer and joystick |
| Buffer | Made in-house | Made in-house | 145 mM NaCl, 4 mM KCl, 20 mM HEPES, 10 mM D-glucose, 1 mM MgCl2, and 1mM CaCl2, at pH 7.3 |
| Cell Chamber | ThermoFisher Scientific | Attofluor Cell Chamber, A7816 | Coverslip holder composed of surgical stainless steel and a rubber O-ring to seal in media and prevent sample and/or objective contamination |
| Excitation Filters | Semrock Inc. | TBP01-378/16 | Center wavelength range (340-378 nm), Bandwidth (Minimum 16 nm, nominal FWHM 20 nm), Refractive index (1.88) |
| Semrock Inc. | TBP01-402/16 | Center wavelength range (360-400 nm), Bandwidth (Minimum 16 nm, nominal FWHM 20 nm), Refractive index (1.8) | |
| Semrock Inc. | TBP01-449/15 | Center wavelength range (400-448.8 nm), Bandwidth (Minimum 15 nm, nominal FWHM 20 nm), Refractive index (1.8) | |
| Semrock Inc. | TBP01-501/15 | Center wavelength range (448.8-501.5 nm), Bandwidth (Minimum 15 nm, nominal FWHM 20 nm), Refractive index (1.84) | |
| Semrock Inc. | TBP01-561/14 | Center wavelength range (501.5-561 nm), Bandwidth (Minimum 14 nm, nominal FWHM 20 nm), Refractive index (1.83) | |
| Fluorescence Filter Cube Dichroic Beamsplitter | Semrock Inc. | FF495-Di03 | Separates excitation and emission light at 495 nm (>98% reflection between 350-488 nm, >93% transmission between 502-950 nm), Filter effective index (1.78) |
| Fluorescence Filter Cube Longpass Filter | Semrock Inc. | FF01 496/LP-25 | Allows passage of light longer than 496 nm ( >93% average transmission between 503.2-1100 nm), Refractive index (1.86) |
| GCaMP Probe | Addgene | G-CaMP3; Plasmid #22692 | A single-wavelength GCaMP2-based genetically encoded calcium indicator |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used for accurate measurement of wide-angle illumination |
| Inverted Fluorescence Microscope | Nikon Instruments | TE2000 | Inverted microscopes allow direct excitation of sample without the need to penetrate layers of media and/or tissue. |
| Mitotracker Green FM | ThermoFisher Scientific | M7514 | Labels mitochondria |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| NIST-Traceable Fluorescein | ThermoFisher Scientific | F36915 | For verifying appropriate spectral response of the system |
| NucBlue | ThermoFisher Scientific | R37605 | Labels cell nuclei |
| Objective (10X) | Nikon Instruments | Plan Apo λ 10X/0.45 ∞/0.17 MRD00105 | Useful for large fields of view |
| Objective (20X) | Nikon Instruments | Plan Apo λ 20X/0.75 ∞/0.17 MRD00205 | Most often used for tissue samples |
| Objective (60X) | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | Most often used for cell samples |
| sCMOS Camera | Photometrics | Prime 95B (Rev A8-062802018) | For acquiring high-sensitivity digital images |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral output of excitation-scanning spectral system |
| Tunable Filter Changer | Sutter Instrument | Lambda VF-5 | Motorized unit for automated excitation filter tuning/switching |
| Xenon Arc Lamp | Sunoptic Technologies | Titan 300HP Lightsource | Light source with relatively uniform spectral output |
Referanslar
- Hagen, N. A., Kudenov, M. W. Review of snapshot spectral imaging technologies. Optical Engineering. 52 (9), 90901 (2013).
- Li, Q., He, X., Wang, Y., Liu, H., Xu, D., Guo, F. Review of spectral imaging technology in biomedical engineering: achievements and challenges. Journal of Biomedical Optics. 18 (10), 100901 (2013).
- Lu, G., Fei, B. Medical hyperspectral imaging: a review. Journal of Biomedical Optics. 19 (1), 10901 (2014).
- Mehta, N., Shaik, S., Devireddy, R., Gartia, M. R. Single-Cell Analysis Using Hyperspectral Imaging Modalities. Journal of Biomechanical Engineering. 140 (2), 20802 (2018).
- Goetz, A. F. H. Three decades of hyperspectral remote sensing of the Earth: A personal view. Remote Sensing of Environment. 113, S5-S6 (2009).
- Goetz, A. F. Measuring the Earth from Above: 30 Years(and Counting) of Hyperspectral Imaging. Photonics Spectra. 45 (6), 42-47 (2011).
- Schröck, E. Multicolor spectral karyotyping of human chromosomes. Science. 273 (5274), 494-497 (1996).
- Tsurui, H. Seven-color fluorescence imaging of tissue samples based on Fourier spectroscopy and singular value decomposition. Journal of Histochemistry & Cytochemistry. 48 (5), 653-662 (2000).
- Lu, R., Chen, Y. R. Hyperspectral imaging for safety inspection of food and agricultural products. SPIE. 3544, 121-134 (1999).
- Hege, E. K., O'Connell, D., Johnson, W., Basty, S., Dereniak, E. L. Hyperspectral imaging for astronomy and space surviellance. SPIE. 5159, 380-392 (2004).
- Vo-Dinh, T. A hyperspectral imaging system for in vivo optical diagnostics. IEEE Engineering in Medicine and Biology Magazine. 23 (5), 40-49 (2004).
- Dorrepaal, R. M., Gowen, A. A. Identification of Magnesium Oxychloride Cement Biomaterial Heterogeneity using Raman Chemical Mapping and NIR Hyperspectral Chemical Imaging. Scientific Reports. 8 (1), 13034 (2018).
- Swayze, G. A. Using imaging spectroscopy to map acidic mine waste. Environmental Science & Technology. 34 (1), 47-54 (2000).
- Khoobehi, B., Beach, J. M., Kawano, H. Hyperspectral imaging for measurement of oxygen saturation in the optic nerve head. Investigative Ophthalmology & Visual Science. 45 (5), 1464-1472 (2004).
- Gowen, A., O'Donnell, C., Cullen, P., Downey, G., Frias, J. Hyperspectral imaging-an emerging process analytical tool for food quality and safety control. Trends in Food Science & Technology. 18 (12), 590-598 (2007).
- Edelman, G., van Leeuwen, T. G., Aalders, M. C. Hyperspectral imaging for the age estimation of blood stains at the crime scene. Forensic Science International. 223 (1), 72-77 (2012).
- Edelman, G., Gaston, E., Van Leeuwen, T., Cullen, P., Aalders, M. Hyperspectral imaging for non-contact analysis of forensic traces. Forensic Science International. 223 (1), 28-39 (2012).
- Markgraf, W., Feistel, P., Thiele, C., Malberg, H. Algorithms for mapping kidney tissue oxygenation during normothermic machine perfusion using hyperspectral imaging. Biomedical Engineering/Biomedizinische Technik. 63 (5), 557-566 (2018).
- Boubanga-Tombet, S. Thermal Infrared Hyperspectral Imaging for Mineralogy Mapping of a Mine Face. Remote sensing. 10 (10), 1518 (2018).
- Yuen, P. W., Richardson, M. An introduction to hyperspectral imaging and its application for security, surveillance and target acquisition. The Imaging Science Journal. 58 (5), 241-253 (2010).
- Favreau, P. F. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19 (4), 046010-046010 (2014).
- Favreau, P. Thin-film tunable filters for hyperspectral fluorescence microscopy. Journal of biomedical optics. 19 (1), 011017-011017 (2014).
- Leavesley, S. J. Hyperspectral imaging microscopy for identification and quantitative analysis of fluorescently-labeled cells in highly autofluorescent tissue. Journal of Biophotonics. 5 (1), 67-84 (2012).
- Annamdevula, N. S. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Deshpande, D. A., Walseth, T. F., Panettieri, R. A., Kannan, M. S. CD38/cyclic ADP-ribose-mediated Ca2+ signaling contributes to airway smooth muscle hyper-responsiveness. The FASEB Journal. 17 (3), 452-454 (2003).
- Deshpande, D. A. Modulation of calcium signaling by interleukin-13 in human airway smooth muscle: role of CD38/cyclic adenosine diphosphate ribose pathway. American Journal of Respiratory Cell and Molecular Biology. 31 (1), 36-42 (2004).
- Guo, M. Cytokines regulate β-2-adrenergic receptor responsiveness in airway smooth muscle via multiple PKA-and EP2 receptor-dependent mechanisms. Biochemistry. 44 (42), 13771-13782 (2005).
- Mansfield, J. R., Gossage, K. W., Hoyt, C. C., Levenson, R. M. Autofluorescence removal, multiplexing, and automated analysis methods for in-vivo fluorescence imaging. Journal of Biomedical Optics. 10 (4), 41207 (2005).
- Mansfield, J. R., Hoyt, C., Levenson, R. M. Visualization of microscopy-based spectral imaging data from multi-label tissue sections. Current Protocols in Molecular Biology. 84 (1), 14-19 (2008).
- Bouchard, M. B. Recent advances in catheter-based optical coherence tomography (OCT) have provided the necessary resolution and acquisition speed for high-quality intravascular imaging. Complications associated with clearing blood from the vessel of a living patient have. Journal of Biomedical Optics. 12 (5), 51601 (2007).
- Mansfield, J. R. Distinguished photons: a review of in vivo spectral fluorescence imaging in small animals. Current Pharmaceutical Biotechnology. 11 (6), 628-638 (2010).
- Levenson, R. M., Mansfield, J. R. Multispectral imaging in biology and medicine: slices of life. Cytometry Part A. 69 (8), 748-758 (2006).
- Gammon, S. T., Leevy, W. M., Gross, S., Gokel, G. W., Piwnica-Worms, D. Spectral unmixing of multicolored bioluminescence emitted from heterogeneous biological sources. Analytical Chemistry. 78 (5), 1520-1527 (2006).
- . . Spectral Unmixing Plugins. , (2006).
- . . Spectral Unmixing of Bioluminescence Signals. , (2006).
- Keshava, N., Mustard, J. F. Spectral unmixing. IEEE Signal Processing Magazine. 19 (1), 44-57 (2002).
- Deal, J. Identifying molecular contributors to autofluorescence of neoplastic and normal colon sections using excitation-scanning hyperspectral imaging. Journal of Biomedical Optics. 23 (12), (2018).
- Microscopy Key, . Microscopy: Key Considerations for Nonlaser Light Sources | Features. BioPhotonics. , (2016).
- Chiu, L., Su, L., Reichelt, S., Amos, W. Use of a white light supercontinuum laser for confocal interference-reflection microscopy. Journal of Microscopy. 246 (2), 153-159 (2012).
- . . Choosing the best light source for your fluorescence experiment. , (2019).
- Beier, H. T., Ibey, B. L. Experimental comparison of the high-speed imaging performance of an EM-CCD and sCMOS camera in a dynamic live-cell imaging test case. PLoS ONE. 9 (1), e84614 (2014).
- Tutt, J. Comparison of EM-CCD and scientific CMOS based camera systems for high resolution X-ray imaging and tomography applications. Journal of Instrumentation. 9 (6), P06017 (2014).
- Coates, C. New sCMOS vs. current microscopy cameras. Biophotonics International. 18 (5), 24-27 (2011).
- Neher, R., Neher, E. Optimizing imaging parameters for the separation of multiple labels in a fluorescence image. Journal of Microscopy. 213 (1), 46-62 (2004).
- Deal, J. Hyperspectral imaging fluorescence excitation scanning spectral characteristics of remodeled mouse arteries. SPIE. 10890, 108902M (2019).
- Deal, J., Rich, T. C., Leavesley, S. J. Optimizing channel selection for excitation-scanning hyperspectral imaging. SPIE. , 108811B (2019).
- Biehlmaier, O., Hehl, J., Csucs, G. Acquisition speed comparison of microscope software programs. Microscopy Research and Technique. 74 (6), 539-545 (2011).
- . . Comparison with other microscopy software - Micro-manager. , (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır