Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Возбуждение-сканирование гиперспектральной микроскопии изображений для эффективной дискриминации сигналов флуоресценции
В этой статье
Резюме
Спектральная визуализация стала надежным решением для идентификации и разделения множественных сигналов флуоресценции в одном образце и может легко отличить сигналы интереса от фона или автофлюоресценции. Экзитс-сканирующая гиперспектральная визуализация улучшает эту технику, уменьшая необходимое время приобретения изображения, одновременно увеличивая соотношение сигнала к шуму.
Аннотация
Некоторые методы полагаются на обнаружение сигналов флуоресценции для выявления или изучения явлений или для выяснения функций. Разделение этих сигналов флуоресценции было доказано громоздким до появления гиперспектральной визуализации, в которой источники флуоресценции могут быть отделены друг от друга, а также от фоновых сигналов и автофлуоресценции (с учетом знаний об их спектральной подписей). Тем не менее, традиционные, выбросов сканирования гиперспектральной визуализации страдает от медленного времени приобретения и низкие соотношения сигнала к шуму из-за необходимой фильтрации как возбуждение и выброс света. Ранее было показано, что гиперспектральная визуализация, сканирующая возбуждение, сокращает необходимое время приобретения, одновременно увеличивая соотношение сигнала к шуму приобретенных данных. Используя коммерчески доступное оборудование, этот протокол описывает, как собирать, калибровать и использовать систему микроскопии гиперспектральной визуализации, сканирующую возбуждение, для отделения сигналов от нескольких источников флуоресценции в одном образце. Хотя этот метод очень применим к микроскопической визуализации клеток и тканей, он также может быть полезен для любого типа эксперимента с использованием флуоресценции, в которой можно изменять длины волн возбуждения, включая, но не ограничиваясь: химическую визуализацию, экологических приложений, ухода за глазами, пищевой науки, судебной науки, медицинской науки и минералогии.
Введение
Спектральная визуализация может быть выполнена различными способамии упоминается несколькими терминами 1,2,3,4. В целом спектральная визуализация относится к данным, полученным по крайней мере в двух пространственных измерениях и одном спектральном измерении. Многоспектральные и гиперспектральные изображения чаще всего различаются по количеству полос длин волн или же спектральные полосы смежные1. Для этого приложения гиперспектральные данные определяются как спектральные данные, полученные с смежными полосами длины волны, достигнутые путем интервала длины центральной волны не менее половины полной ширины на половину максимум (FWHM) каждого фильтра bandpass, используемого для возбуждения (т.е. 5 нм интервал длины центральной волны для фильтров полоспаса с пропускной способностью 14-20 нм). Смежный характер диапазонов данных позволяет перебирать набор данных, гарантируя, что критерии Nyquist удовлетворяются при выборке спектрального домена.
Гиперспектральная визуализация была разработана НАСА в 1970-х и 1980-х годах в сочетании с первым спутником Landsat5,6. Сбор данных из нескольких смежных спектральных полос позволил понять спектр сияния каждого пикселя. Выявление и определение спектра сияния отдельных компонентов позволило не только обнаружить поверхностные материалы по их характерным спектрам, но и позволило удалить промежуточные сигналы, такие как вариации сигнала из-за атмосферных условий. Концепция обнаружения материалов с использованием их характерных спектров была применена к биологическим системам в 1996 году, когда Шрёк и др. использовали комбинации пяти различных фторофоров и их известных спектров, чтобы различать помеченные хромосомы в процессе, называемом спектральный кариотипирование7. Этот метод был разработан в 2000 году Tsurui et al. для флуоресценции изображений образцов тканей, используя семь флуоресцентных красителей и разложение единственного значения для достижения спектрального разделения каждого пикселя на линейные комбинации спектров в справке библиотека8. Подобно их коллегам дистанционного зондирования, вклад каждого известного фторфора может быть рассчитан на основе гиперспектрального изображения, учитывая априори информацию спектра каждого фторофра.
Гиперспектральная визуализация также была использована в областях сельского хозяйства9, астрономия10, биомедицина11, химическая визуализация12, экологические приложения13, уход за глазами14, пищевая наука15, судебно-медицинской экспертизы16,17, медицинской науки18, минералогии19, и наблюдения20. Ключевым ограничением текущего флуоресцентного микроскопа гиперспектральных систем визуализации является то, что стандартная технология гиперспектральной визуализации изолирует сигналы флуоресценции в узких диапазонах на 1) сначала фильтрации возбуждания света для контроля образца возбуждения, то 2) дальнейшая фильтрация излучаемого света, чтобы отделить излучение флуоресценции в узкие полосы, которые позже могут быть отделены математически21. Фильтрация как возбуждения освещения и испускаемых флуоресценции уменьшает количество доступного сигнала, что снижает соотношение сигнала к шуму и требует длительного времени приобретения. Низкий сигнал и длительное время приобретения ограничивают применимость гиперспектральной визуализации в качестве диагностического инструмента.
Была разработана методика визуализации, которая использует гиперспектральную визуализацию, но повышает доступный сигнал, тем самым уменьшая необходимое время приобретения21,22. Эта новая модальность, называемая гиперспектральной визуализацией, сканирующей возбуждающие возбуждение, приобретает спектральные данные изображений, изменяя длину волны возбуждения и собирая широкий спектр излучаемого света. Ранее было показано, что этот метод дает порядки величины увеличивается в сигнал к шуму соотношение по сравнению с методами сканирования выбросов21,22. Увеличение соотношения сигнала к шуму в значительной степени обусловлено широкой полосой (600 нм) обнаруженного светового света, в то время как специфичность обеспечивается путем фильтрации только света возбуждения вместо флуоресценционного излучения. Это позволяет всем излучаемым светом (для каждой длины волны возбуждения) достичь детектора21. Кроме того, этот метод может быть использован для дискриминирования автофлюоресценции от экзогенных меток. Кроме того, способность сократить время приобретения за счет увеличения обнаруживаемого сигнала снижает опасность фотоотбеления, а также позволяет спектрального сканирования со скоростью приобретения, что является приемлемым для спектральной видеосъемки.
Цель этого протокола заключается в том, чтобы служить в качестве руководства по сбору данных для экзит-сканирующей микроскопии гиперспектральной визуализации. Кроме того, включены описания, которые помогают понять световой путь и аппаратное обеспечение. Также описана реализация программного обеспечения с открытым исходным кодом для микроскопа гиперспектральной визуализации, сканирующего возбуждение. Наконец, приводятся описания того, как откалибровать систему в стандарт NIST- traceable, настроить настройки программного и аппаратного обеспечения для точных результатов и не смешивать обнаруженный сигнал в вклады отдельных компонентов.
протокол
1. Настройка устройства
- Источник света: выберите широкодиапазонный спектральный источник света с высокой мощностью и высокой коллимацией (для этих исследований использовалась дуговая лампа 300 Вт Xe).
- Затвор (необязательно): добавьте затвор к оптическому пути, чтобы уменьшить фотоотбеление для замедленного изображения.
- Tunable фильтровальная система: включите механическую сборку тюнинга и тонкопленочный настраиваемый фильтр (TFTF), позволяющий обеспечить желаемый диапазон возбуждения, регулируемый длиной волны (например, 360-485 нм).
- Микроскоп: используйте перевернутый флуоресценционный микроскоп, включающий моторизованную дихротический фильтр башни и контроллера.
- Куб флуоресцентного фильтра: соберите длиннопроходной кубик флуоресценции фильтра. Для достижения наилучших результатов выберите дихроическое зеркало и фильтр для длительного прохода на одной волне для достижения оптимального отделения возбуждения от излучения (например, 495 нм).
- Цель: использовать соответствующую апохроматической цели для обеспечения равномерного внимания на диапазоне длин волн, используемых для эксперимента (60x цель воды была использована для этого исследования).
- Автоматизированная стадия (необязательно): использовать автоматизированный этап для быстрой выборки нескольких полей зрения и/или очень больших полей через прошивание изображений. Калибровать сцену с целью и камерой.
- Камера: выберите соответствующую камеру для достижения пространственного разрешения, чувствительности и шумовых требований эксперимента (эта система использовала камеру sCMOS с высокой чувствительностью).
2. Программное обеспечение для приобретения
- Выберите программный пакет, позволяющий осуществлять независимый контроль над каждым аппаратным компонентом, например Micro-Manager.
- Создание файла конфигурации: файл конфигурации представляет собой сохраненный набор инструментов и инструкций, которые Micro-Manager будет загружать для работы оборудования системы. Чтобы создать файл конфигурации, нажмите Инструменты
- В шаге 1, нажмите Создать новую конфигурацию Обратите внимание, что пользователь может изменить или изучить существующую конфигурацию, если она уже доступна.
- В шаге 2 просмотрите список доступных устройств, чтобы найти устройства для управления с помощью Micro-Manager, таких как микроскоп, лампа, сцена, затвор и камера. При выделении щелкните Добавить... кнопка для добавления устройства в список установленных устройств. Это откроет новое окно.
- Обозначьте этикетку устройства в поле Label. (например, камера SCMOS)
- Под значением выберите подходящий порт COM для каждого устройства. Это приведет к тому, что в нижней части окна появится список свойств устройств.
- Оставьте свойства устройства в настройках по умолчанию. Обратите внимание, что пользовательские значения для каждого свойства устройства могут быть введены по желанию.
- При добавлении каждого устройства нажмите кнопку Next, чтобы продолжить следующий шаг. Обратите внимание, что если какие-либо устройства не будут исключены или нуждаются в изменении, файл конфигурации может быть изменен позже, как описано в шаге 2.1.1.1.
- В шаге 3 используйте выпадающие меню, чтобы выбрать камеру по умолчанию, затвор и этап фокусировки. Если автозатвор нонпов, щелкните чек-бокс ниже списка выпадающих на стадию фокуса. Если известно направление фокусировки этапа, выберите его из списка выпадающих направлений в соответствии с направлениями фокусировки этапов (продвинутые). В противном случае оставьте значение по умолчанию как неизвестное.
- В шаге 4, дважды щелкните коробки под задержкой »мс» заголовок установить задержки, связанные с автоматизированными устройствами, такими как задержки 100 мс для лампы, стадии и затвора, и 250 мс задержки для команд на механическую сборку настройки для учета механическое время переключения между фильтрами.
- В шаге 5 назначаем метки для государственных устройств. Конфигурация для системы микроскопа с сканированием возбуждения использует метки в блоке фильтра для идентификации каждого куба флуоресценции фильтра (например, состояние 0 соответствует метке Bright, пустой куб фильтра, используемый при визуализации яркого поля; в то время как состояние 3 соответствует этикетке 495 нм и обычай 495 нм дихроический фильтр куб, используемый для разделения возбуждения и выбросов света на 495 нм). Этикетки также используются в механической сборке настройки для хранения команд переключения для каждой длины волн возбуждения (например, состояние 0 соответствует команде настройки длины возбуждения 340 нм для механической сборки тюнинга).
- На шестом шаге нажмите кнопку «Просмотр» рядом с файлом Configuration, чтобы выбрать место сохранения и имя файла для файла конфигурации. Нажмите на финиш, чтобы сохранить файл конфигурации.
- Группы стартапов и пресеты: Micro-Manager может хранить различные группы в каждом файле конфигурации для активации или изменения подмножества оборудования. Например, группа "системный запуск" и предустановленная комбинация будут хранить настройки по умолчанию, такие как размер связывания и скорость считывания для камеры.
- Создайте группу (например, System),нажав на основное окно в подразделе Группы.
- Проверить любое использование в группе? коробки внутри группы, которые потребуют установки значения по умолчанию (например, связывание в связанной камере по умолчанию).
- Создайте новую пресет-установку (например, "Startup"), нажав на основное окно в подразделе Preset.
- Каждая коробка, проверенная в шаге 2, будет отображаться как предустановленная опция здесь. Выберите значение по умолчанию для загрузки для каждого проверенного окна.
- Группа и пресеты для волн возбуждения: Micro-Manager рассматривает каждую длину волн возбуждения как свой собственный канал с его собственной командой переключения длины волны; поэтому каждый из них должен быть сохранен как свой собственный предустановленный.
- Создайте новую группу, содержащую набор желаемых длин волн возбуждения (например, дихроик 495 нм)
- Проверьте использование в группе? коробка под названием Label, соответствующая устройству VF-5.
- Создайте новый предустановленный набор, названный для каждой длины волн возбуждения (например, "340 нм").
- Присвоить каждому предустановленное имя свойства и предустановленное значение (например, имя свойства: "Label"; предустановленное значение: "340 нм").
- Многомерный инструмент приобретения: нажмитеMulti-D Acq.в верхнем левом верхнем левом окне Micro-Manager, чтобы открыть настраиваемый инструмент для захвата изображения образца на каждой длине волн возбуждения одним нажатием кнопки. Есть несколько вариантов настройки, чтобы построить приобретение точно по желанию.
- Тайм-пойнты (не используемые в данном примере): для исследований, не имеющих компонента замедленного времени, оставьте коробку без контроля. Если пожелается замедленное исследование, проверьте эту коробку. Если проверено, введите количество раз, чтобы выполнить остальные настройки приобретения в поле Номера. Установите время между последовательными приобретениями, введя продолжительность в поле Interval и выберите подходящий блок (миллисекунды, секунды или минуты; обычно секунды).
- Несколько позиций (XY; не используется в этом примере): для исследования одного положения XY, оставьте коробку без проверки. Если несколько позиций XY являются желательными, проверьте поле и нажмите кнопку списка позиции Edit, чтобы открыть отдельное окно. Переместите сцену в нужное место и нажмите кнопку «Знак», чтобы сохранить эту позицию в программном обеспечении. Повторяйте до тех пор, пока не будут отмечены все желаемые позиции XY. Закройте это окно, чтобы продолжить.
- Стеки (слайды; не используются в данном примере): для изучения одного положения, оставьте коробку без контроля. Если несколько позиций являются желательными, проверьте поле. Переместите сцену в нужное начало и нажмите кнопку Set рядом с окном «старт». Переместите сцену в желаемое положение окончание и нажмите кнопку Set рядом с коробкой с конечным результатом. Введите желаемый размер шага (в микронах) в поле для шага.
- Каналы: убедитесь, что коробка каналов проверена. Здесь каналы — это имена, присвоенные отдельным длинам волн возбуждения. Имеющиеся "Каналы" соответствуют "Группам", описанным в шагах 2.1.2 и 2.1.3.
- Группа: нажмите на выпадающий список, чтобы выбрать группу, из которой можно выбрать доступные длины волн возбуждения (например, дихроик 495 нм).
- Нажмите на поле Keep Shutter Open, чтобы держать затвор открытым между приобретениями для каждого канала. Обратите внимание, что затвор будет по-прежнему закрыт между последовательными спектральными сканированиями, если это поле проверено, предполагая, что опция автозатвора также выбрана в главном окне.
- Нажмите кнопку «Новая», чтобы добавить каналы в список приобретений. Обратите внимание, что кнопка Удалить можно использовать для удаления любых каналов, которые больше не нужны, и что Up and Down может быть использован для повторного заказа любого выбранного канала.
- Убедитесь, что флажок Use? выбран для каждой нужной длины волны в спектральном сканировании.
- Конфигурация: щелкните список выпадающих вниз под конфигурацией и выберите первую длину волны в нужном спектральном диапазоне (например, 340 нм).
- Экспозиция: дважды щелкните поле под экспозицией и введивите желаемое время экспозиции для выбранной длины волны (например, "100" на 100 мс). См. раздел 5 ("Приобретение данных") для предложений по выбору подходящего времени экспозиции.
- Смещение (не применимо при спектральном сканировании): оставьте эту коробку пустой (при 0) при выполнении спектрального сканирования.
- - стек: убедитесь, что эта коробка проверяется на каждую длину волны в спектральном диапазоне при выполнении стека.
- Пропустить fr. (не применимо в спектральном сканировании): оставьте эту коробку пустой (на 0) при выполнении спектрального сканирования. Обратите внимание, что это может быть полезно выборочно пропустить кадры в случае, если один или несколько волн возбуждения длины значительно более мощным, чем другие, или если отдельные длины волн возбуждения оказаться особенно фототоксичные.
- Цвет: оставьте эту коробку пустой при выполнении спектрального сканирования. Обратите внимание, что цвета могут быть выбраны для отдельных полос длинволн для визуализации данных, но цвет не подходит для последующего спектрального несмешивания.
- Повторяйте эти шаги до тех пор, пока не будет добавлена каждая желаемая длина волн возбуждения в пределах заданного спектрального диапазона сканирования.
- Заказ на приобретение: нажмите на выпадающий список, чтобы выбрать, в каком порядке будут выполнены вышеуказанные варианты (2.1.1.1-2.1.4.5). Для спектральных сканирований, таких как показано здесь, порядок приобретения просто канал. Обратите внимание, что дополнительные опции появляются по мере того, как дополнительные коробки проверяются в инструменте Multi-Dimensional Acquisition (Timepoints, Multiple Positions (XY) и «стеки» (срезы)» и позволяют выбирать позицию или время, за которым следуют либо срезы, либо Канал.
- Автофокус (не используется в этом примере): Если выбрано несколько позиций (XY), доступно несколько различных стилей автофокусировки. Нажмите кнопку Параметры и выберите метод автофокусировки из списка выпадающих рядом с кнопкой "закрыть".
- Резюме: просмотрите это окно для сводки количества тайм-пойнтов, позиций XY, срезов, каналов, общего количества изображений, общей памяти, продолжительности сканирования и порядка приобретения, чтобы обеспечить ожидаемые параметры получения информации.
- Сохранить изображения: убедитесь, что это поле проверяется для сохранения данных, собранных с помощью многомерного инструмента приобретения.
- Нажмите на ... кнопка рядом с корнем каталога, чтобы выбрать корень каталога, в котором будут сохранены файлы. Назовите корень каталога таким образом, который описывает соответствующие детали эксперимента (например, GCaMP Airway Smooth Muscle Cells).
- Введите имя в поле рядом с приставкой Name, описывающим текущее приобретение изображения (например, FOV1-100ms-60X-495nmDichroicFilter). Целесообразно создать приставку имени, которая определяет поле зрения и время экспозиции, а также другую соответствующую информацию, такую как объект и используемый дихротический фильтр. Обратите внимание, что первый стек изображений, сделанный с помощью этих настроек, будет сохранен с помощью "No1" после приставки имени. Любой последующий стек, взятый с тем же корнем каталога и приставкой имени, будет сохранен с помощью "N", в котором "n" - это количество раз, когда стек был взят с этим именем в каталоге.
- Нажмите Отдельные файлы изображений, чтобы гарантировать сохранение изображения, генерируемого на каждой длине волн возбуждения.
- Сохранить как: нажмите кнопку Сохранить как... Обратите внимание, что корень каталога и префиксы имен также будут сохранены.
- Приобретите: наконец, нажмите На Acquire! кнопка, чтобы начать приобретать изображения в соответствии с настройками приобретения, выбранными выше.
- Создание файла конфигурации: файл конфигурации представляет собой сохраненный набор инструментов и инструкций, которые Micro-Manager будет загружать для работы оборудования системы. Чтобы создать файл конфигурации, нажмите Инструменты
3. Коррекция спектрального ответа (необязательно):
- Спектральная коррекция вывода может быть выполнена для калибровки спектральной реакции системы на известный стандарт, такой как лампа NIST-traceable или другой стандарт или инструмент NIST-traceable. Этот шаг особенно важен, если результаты сравниваются с другими спектральными приборами, спектрометрами или между различными лабораториями. Этот процесс был подробно сообщен ранее21,23.
- Используйте спектрометр, откалиброванный к источнику NIST-traceable light source (например, LS-1-CAL-INT, Ocean Optics) или другому стандарту NIST-traceable для приобретения спектрорадиометрической мощности освещения, измеряемого на этапе выборки. Выполните отдельную коррекцию для каждой комбинации параметров источника света, дихроического зеркала и объективности. Используйте интегрирующую сферу в сочетании с спектрометром для точного измерения широкоугольного освещения от цели микроскопа.
- Используйте метод интеграции, такой как трапециевидное правило, для интеграции спектрорадиометрических данных по освещенным длинам волн. Пропускная способность 40 нм для интеграции, сосредоточенной вокруг центральной длины волны, достаточна для большинства фильтров, которые имеют номинальную пропускную способность между 14-20 нм полной шириной на половину максимума (FWHM). Интегрированное значение представляет интенсивность спектрального освещения на каждой полосе длины волн возбуждения.
- Участок интегрированной интенсивности каждой полосы длины волны в качестве функции возбуждания центр длины волны, чтобы визуализация спектрального профиля интенсивности освещения.
- Определите полосу длины волны с наименьшей интегрированной интенсивностью.
- Нормализовать профиль интенсивности спектрального освещения, разделив наименьшую интегрированную интенсивность интегрированной интенсивностью каждой полосы длины волны для генерации фактора коррекции, зависящей от длины волны.
4. Подготовка образцов
- Подготовьте «пустой» образец (например, поместите стеклянный покрывало в камеру ячейки и добавьте 1 мл буфера). Этот буфер был описан ранее24.
- Подготовьте немаркированный образец для определения любого образца автофлюоресценции (например, поместите стеклянный покрывало, содержащее немаркированные клетки мышц25,26 в камере клеток и добавьте 1 мл буфера).
- Подготовьте отдельный образец с одной маркировкой для каждой флуоресцентной метки, используемой в эксперименте, следующим образом:
- Добавьте разбавленную митохондриальную этикетку в буфер для достижения концентрации 100 НМ. Добавьте этот буфер к крышке, содержащей гладкие мышечные клетки дыхательных путей. Инкубировать в течение 20 мин при 20-25 градусах Цельсия. Переместите coverslip в камеру ячейки и добавьте 1 мл буфера. Обратите внимание, что оптимальная концентрация митохондриальной этикетки будет варьироваться в зависимости от таких факторов, как производитель, связанный цвет и желаемая специфичность.
- Переместите крышку, содержащую гладкие мышечные клетки дыхательных путей, трансфицируемые зондом GCaMP27, в камеру клетки и добавьте 1 мл буфера.
- Подготовьте один или несколько экспериментальных образцов, содержащих смесь желаемых флуоресцентных этикеток.
- Добавьте 1 мл буфера (100 нм концентрации митохондриальной этикетки) к крышке, содержащей гладкие мышечные клетки дыхательных путей, трансфицированные зондом GCaMP, и инкубировать в течение 20 мин при 20-25 градусах Цельсия. Переместите coverslip в камеру ячейки и добавьте 1 мл буфера.
5. Получение данных:
- Убедитесь, что выбран ынатворный луч (например, куб дихроического фильтра 495 нм), объективный (например, 60-кратная цель воды) и камера (камера sCMOS).
- Загрузите образец на сцену.
- Дважды щелкните поле рядом с экспозицией и введите "100", чтобы установить время экспозиции на уровне 100 мс. Обратите внимание, что время экспозиции, возможно, потребуется увеличить или уменьшить в зависимости от интенсивности флуоресценции образца.
- Выберите 475 нм из выпадающего меню главного окна Micro-Manager для первоначального просмотра образца. Обратите внимание, что 475 нм может быть не оптимальной длиной волны для просмотра образца или определения того, будет ли перенасыщение происходит ьизм на протяжении всего стека изображений.
- Нажмите Live, чтобы просмотреть образец.
- Нажмите кнопку автоматического обзора интенсивности Авто рядом с гистограммой в нижней части окна, чтобы принести минимальные и максимальные значения в значимые визуальные диапазоны.
- Используйте фокусные ручки микроскопа, чтобы сосредоточиться на образце. Это часто полезно, чтобы найти край образца, чтобы помочь в фокусировке. Образец будет сфокусирован, когда элементы края изображения будут казаться острыми. Обратите внимание, что это может быть необходимо нажать кнопку Auto несколько раз во время фокусировки, чтобы помочь в просмотре флуоресценции изображения. Кроме того, некоторым пользователям может быть легче сосредоточиться на образце, если микроскоп находится в режиме передачи.
- Если фокусировка в режиме передачи, для безопасности, сначала убедитесь, что спектральный источник света не передает свет на окуи, прежде чем регулировать световой путь для передачи изображений. Также обратите внимание, что могут быть небольшие отклонения между фокусом передачи и флуоресценции изображений. Когда достигнута приемлемая направленность, перенастроить световой путь для спектральной флуоресценции.
- Нажмите на Multi-D Acq. кнопка в верхнем левом верхнем цвете окна для открытия инструмента многомерного приобретения (описанного и настроенного в разделе 2).
- Выберите подходящий спектральный диапазон для спектрального приобретения (например, 360-485 нм для дихроического фильтра 495 нм), нажав кнопку Load... в правом верхнем углу окна инструмента Multi-Dimensional Acquisition и ориентируясь на настройки возбуждения сохранены ранее (в шаге 2.1.4.10). Смотрите обсуждение информации о соответствующих спектральных диапазонах.
- Приобретите фоновое/пустое изображение, которое не содержит данных флуоресценции для использования для фонового и шумового вычитания. Это может быть выполнено с помощью пустого образца (шаг 4.1) или путем навигации по пустой области экспериментального образца. Как описано в шаге 5.6, это наиболее легко достигается путем нахождения края образца, а затем позиционирования его так, что край по центру в поле зрения.
- После подтверждения параметров приобретения и видимой фоновой области нажмите кнопку Acquire!, чтобы приобрести спектральный стек изображения, содержащий фон и шум для последующего использования для вычитания. Следует отметить, что образцы, вероятно, имеют более интенсивную флуоресценцию вдали от края образца. Возможно, целесообразно переместить образец, чтобы найти "самые яркие" регионы и выполнить несколько тестовых стеков изображений, чтобы определить подходящее время приобретения и избежать передержки.
- Возьмите один стек изображения на области образца, который, как представляется, имеют интенсивную флуоресценцию, чтобы гарантировать, что никакое сочетание длин волн и что время экспозиции приводит к чрезмерной экспозиции.
- Используйте ImageJ, чтобы подтвердить, что длины волн не содержат переэкспонированных пикселей.
- В ImageJ, нажмите файл (ru) импортное изображение и перейдите к папке, содержащей спектральные изображения, сделанные в шаге 5.10. Это откроет новое окно. Нажмите на поле рядом с числом изображений и введите количество длин волн, включенных в спектральный скан (например, 26). Оставьте Исходное изображение и приращение как "1". Нажмите кнопку OK, чтобы продолжить.
- Нажмите M на клавиатуре, чтобы использовать функцию измерения ImageJ. Убедитесь, что номер, указанный под Максом, не является верхним пределом обнаружения камеры (например, 65 535). Повторите для каждого изображения в спектральном сканировании. Обратите внимание, что предел обнаружения камеры зависит от самой камеры. Верхний предел отображается в верхней правой части гистограммы в главном окне MicroManager. Кроме того, верхний предел может быть определена путем открытия изображения в ImageJ и навигации по изображению Нажмите кнопку Set в полученном всплывающем окне, введите чрезмерно большое значение (например, 999,999) в пустой рядом с максимальным отображаемым значением,и нажмите OK, чтобы продолжить. Максимальное значение, отображаемые в новой гистограмме, должно быть верхним пределом обнаружения камеры.
- Если какие-либо изображения содержали верхний предел обнаружения камеры (например, 65 535), отрегулируйте время экспозиции спектрального сканирования, чтобы обеспечить, чтобы максимальный сигнал во всем спектральном диапазоне не превышал динамический диапазон камеры.
- Приобретение спектральных данных изображений из немаркированных образцов для определения любой аутофлуоресценции. Приобретите сканирование немаркированных образцов, используя одинаковый диапазон длин волн и настройки камеры в качестве оставшейся части эксперимента (например, 360-485 нм при 100 мс на длину волны).
- Приобретение спектральных изображений из одномаркированных образцов для использования в качестве спектрального элемента управления для создания спектральной библиотеки. Выполните сканирование для каждой метки, используя одинаковый диапазон длин волн, время экспозиции и настройки камеры. Обратите внимание, что для некоторых образцов может потребоваться более длительное время приобретения, чтобы обеспечить точное обнаружение спектра меток с минимальным вкладом шума.
- Выполняйте измерения на экспериментальном образце (например, гладкие мышечные клетки дыхательных путей человека с зондом GCaMP и митохондриальной этикеткой).
- Поместите слайд или крышку, содержащую экспериментальный образец, на микроскоп.
- Выберите поле зрения с соответствующим образом помечены клетки тканей.
- Приобретите данные спектрального изображения, используя желаемые настройки приобретения, как описано выше.
6. Анализ изображений
- Правильные изображения с плоской спектральной реакцией.
- Вычесть фоновый спектр, полученный в разделе 5.9, и умножить на коэффициент коррекции, определенный в разделе 3.1.5. Это можно сделать с помощью простого скрипта MATLAB или процедуры ImageJ. Код subtraction/коррекции MATLAB (используется в этом примере) доступен на веб-сайте Университета южной Алабамы Bioimaging Resources.
- Загрузите код вычитания/коррекции в MATLAB.
- Во вкладке EDITOR нажмите Run. Это откроет новое окно, содержащее кнопку коррекции Bsq. Нажмите кнопку Коррекция Bsq, чтобы открыть другое окно, содержащее выбор для рабочего каталога, фонового файла, файла фактора коррекции и неисправленной папки tiff.
- Используйте просмотр... кнопка рядом с рабочим каталогом, чтобы выбрать путь файла, где будут открыты последующие окна. Перейдите к папке, содержащей сохраненный фоновый спектр (в формате .dat).
- Используйте просмотр... кнопка рядом с фоновым файлом (.dat), чтобы открыть каталог, выбранный в шаге 6.1.1.2.1 и выбрать нужный фоновый файл (в формате .dat). Нажмите кнопку "Открытая", чтобы продолжить.
- Используйте кнопку «Обзор» рядом с фактором коррекции (.dat), чтобы выбрать коэффициент коррекции. Каталог для фактора коррекции по умолчанию влияет на расположение родительского кода MATLAB. При необходимости перейдите к субфолдеру, содержащему файл фактора коррекции (в формате .dat). Выделите правильный файл фактора коррекции и нажмите кнопку Open для продолжения.
- Используйте кнопку Browse... рядом с Неисправленым Tiff Folder, чтобы открыть каталог, выбранный в шаге 6.1.1.2.1 и выбрать папку для фонового вычитания и коррекции изображения (обычно это папка Pos0, которая сразу же содержит сырье спектральных изображений). Нажмите кнопку "Открытая", чтобы продолжить.
- Нажмите кнопку «Нажмите на спектральную коррекцию». После завершения обработки откроется окно с сообщением "Bsq Коррекция завершена". Нажмите кнопку OK, чтобы продолжить. Изображения коррекции хранятся в новой папке с именем с исходной папкой исходного спектрального изображения и дополнительной «Исправленной» (например, Pos0-Corrected).
- Вычесть фоновый спектр, полученный в разделе 5.9, и умножить на коэффициент коррекции, определенный в разделе 3.1.5. Это можно сделать с помощью простого скрипта MATLAB или процедуры ImageJ. Код subtraction/коррекции MATLAB (используется в этом примере) доступен на веб-сайте Университета южной Алабамы Bioimaging Resources.
- Создайте спектральную библиотеку. Несколько пакетов программного обеспечения могут быть использованы для извлечения спектральных данных из изображений, включая ImageJ. Для достижения наилучших результатов, нормализовать каждый endmember к диапазону длины волны наибольшей интенсивности, определяя, какая полоса длины волны имеет наиболее интенсивное значение и деления измерения на каждой полосе длины волны, что максимальное значение. Следует также отметить, что одномаркировка управления, генерируемого в автофлуоресцентных образцах, потребует дополнительных модификаций28,29,30,31,32. Смотрите шаг 6.2.4 и обсуждение для деталей.
- В ImageJ, нажмите файл (gt; Импорт) Это откроет новое окно. Перейдите к папке, содержащей исправленные спектральные изображения. Дважды щелкните любой файл изображения в папке, чтобы открыть новое окно. Нажмите на поле рядом с числом изображений и введите количество длин волн, включенных в спектральный скан (например, 26). Оставьте Исходное изображение и приращение как "1". Нажмите кнопку OK, чтобы продолжить.
- В главном окне ImageJ щелкните значок выбора полигона, а затем используйте указатель мыши, нажав на изображение, чтобы создать полигон вокруг области, содержащей данные флуоресценции. Обратите внимание, что в начальной длине волны практически не может быть данных о флуоресценции. Перейдите к другому изображению длины волны и / или использовать яркость / контрастный инструмент (Изображение йgt; Отрегулируйте йgt; Яркость / Контрастный йgt; Авто), чтобы лучше визуализировать регионов, содержащих данные флуоресценции, представляющие интерес.
- С полигоном обращается, генерировать профиль оси, нажав на изображение Это откроет новое окно. Нажмите Сохранить, чтобы сохранить спектральные данные в виде файла .csv.
- Выполните вычитание для обеспечения надлежащей спектральной библиотеки с чистыми этикетками, лишенными загрязнения автофлюоресценции. Используйте следующее уравнение29, чтобы изолировать чистый спектр от смеси одной метки и флуоресценции:
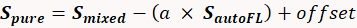 (1)
(1)
Где: S чистый является чистый спектр этикетки, Sсмешанный является измеренный спектр образца загрязненных автофлюоресценции, S autoFL является измеренный спектр автофлюоресценции, "а" является масштабирование множитель спектра автофлюоресценции (между 0 и 1; например, 0,4), и "смещение" является смещением (обычно 0). Варите термин "а" до достижения почти нулевого значения за очевидный вклад автофлюоресценции. Дополнительную информацию можно найти в нескольких публикациях28,29,30,31,32.
- Не смешивайте данные спектрального изображения. Несмешивающий шаг будет генерировать изображение изобилия для каждого флуоресцентного ярлыка, где изобилие количество относительного сигнала флуоресценции на изображении от соответствующей этикетки. Несколько алгоритмов unmixing доступны для использования с ImageJ33,34,35. Кроме того, спектральный несмешивающий код MATLAB (используется в этом примере) доступен на веб-сайте Университета южной Алабамы Bioimaging Resources. Более полный обзор спектрального unmixing доступен36.
- Загрузите спектральный код unmixing в MATLAB.
- Редактизировать файлы Wavelength.mat и Library.mat в соответствии с экспериментальными условиями (например, "Длина волны" как 360-485 с шагом в пять).
ПРИМЕЧАНИЕ: Соответствующие значения для этих длин волн должны быть загружены в "Библиотека" для каждой флуоресцентной метки (и автофлюоресценции). Имя endmember должно перечислять метки в том же порядке, что и значения столбца "Библиотека". Обратите внимание, что файл Library.mat должен содержать как переменные "Библиотека" и "Endmember-Name". - Во вкладке EDITOR нажмите Run. Это откроет новое окно, позволяющее пользователю выбрать каталог. Перейдите к папке, содержащей спектральные изображения, чтобы быть unmixed. После выбора, это откроет новое окно.
- В полученных пробелах введите имя изображения (например, HASMC с GCaMP и Mito FOV1), количество полос длин волн (например, 26), количество таймтов (например, 1) и желательно ли измерение FRET (например, n) в соответствующих заготовках. Если "n" выбран для FRET, оставьте оба пробела End-Member пустыми. Нажмите OK, чтобы продолжить, который откроет новое окно.
- Naviagate в папку, содержащую файл Wavelength.mat. После выбора нажмите Open, чтобы продолжить, что откроет новое окно.
- Перейдите к папке, содержащей файл Library.mat. После выбора нажмите Open, чтобы продолжить, что сохранит несмешанные изображения в новой папке "Unmixed" внутри каталога, содержащей спектральные изображения.
- Осмотрите несмешанные спектральные изображения на качество.
- Откройте каждое несмешанное изображение, чтобы визуально проверить распределение чистых компонентов.
- Сравните величину изображения ошибки с изображением несмешанных изображений (по аналогии с шагом 5.11, это может быть сделано с помощью функции измерения ImageJ). Если величина изображения ошибки по величине схожа с изображениями несмешанного изобилия, то вполне вероятно, что в данных спектрального изображения есть сигналы, которые точно не учитываются спектральной библиотекой и линейным спектральным процессом несмешивания.
- Сравните изображение ошибки с несмешанными изображениями, чтобы увидеть, есть ли неопознанные остаточные структуры.
- Откройте каждое несмешанное изображение, чтобы визуально проверить распределение чистых компонентов.
Результаты
Несколько важных шагов из этого протокола необходимы для обеспечения сбора данных, которые являются точными и лишенными изображений и спектральных артефактов. Пропуск этих шагов может привести к тому, что данные будут значительными, но не могут быть проверены или вос...
Обсуждение
Оптимальное использование гиперспектральной визуализации, сканирующего возбуждение, начинается со построения световой траектории. В частности, выбор источника света, фильтров (невозможных и дихронических), метода переключения фильтров и камеры определяют доступный спектральный диа...
Раскрытие информации
Доктора Ливзли и Рич раскрывают финансовую заинтересованность в стартап-компании SpectraCyte LLC, основанной для коммерциализации технологии спектральной визуализации.
Благодарности
Авторы хотели бы отметить поддержку со стороны NSF 1725937, NIH P01HL066299, NIH R01HL058506, NIH S10OD020149, NIH UL1 TR001417, NIH R01HL137030, AHA 18PRE3406016.
Материалы
| Name | Company | Catalog Number | Comments |
| Airway Smooth Muscle Cells | National Disease Research Interchange (NDRI) | Isolated from human lung tissues obtained from NDRI | Highly autofluorescent, calcium sensitive cells |
| Automated Shutter | Thorlabs Inc. | SHB1 | Remote-controllable shutter to minimize photobleaching |
| Automated Stage | Prior Scientific | H177P1T4 | Remote-controllable stage for automated multiple field of view or stitched image collection. |
| Automated Stage Controller (XY) | Prior Scientific | Proscan III (H31XYZE-US) | For interfacing automated stage with computer and joystick |
| Buffer | Made in-house | Made in-house | 145 mM NaCl, 4 mM KCl, 20 mM HEPES, 10 mM D-glucose, 1 mM MgCl2, and 1mM CaCl2, at pH 7.3 |
| Cell Chamber | ThermoFisher Scientific | Attofluor Cell Chamber, A7816 | Coverslip holder composed of surgical stainless steel and a rubber O-ring to seal in media and prevent sample and/or objective contamination |
| Excitation Filters | Semrock Inc. | TBP01-378/16 | Center wavelength range (340-378 nm), Bandwidth (Minimum 16 nm, nominal FWHM 20 nm), Refractive index (1.88) |
| Semrock Inc. | TBP01-402/16 | Center wavelength range (360-400 nm), Bandwidth (Minimum 16 nm, nominal FWHM 20 nm), Refractive index (1.8) | |
| Semrock Inc. | TBP01-449/15 | Center wavelength range (400-448.8 nm), Bandwidth (Minimum 15 nm, nominal FWHM 20 nm), Refractive index (1.8) | |
| Semrock Inc. | TBP01-501/15 | Center wavelength range (448.8-501.5 nm), Bandwidth (Minimum 15 nm, nominal FWHM 20 nm), Refractive index (1.84) | |
| Semrock Inc. | TBP01-561/14 | Center wavelength range (501.5-561 nm), Bandwidth (Minimum 14 nm, nominal FWHM 20 nm), Refractive index (1.83) | |
| Fluorescence Filter Cube Dichroic Beamsplitter | Semrock Inc. | FF495-Di03 | Separates excitation and emission light at 495 nm (>98% reflection between 350-488 nm, >93% transmission between 502-950 nm), Filter effective index (1.78) |
| Fluorescence Filter Cube Longpass Filter | Semrock Inc. | FF01 496/LP-25 | Allows passage of light longer than 496 nm ( >93% average transmission between 503.2-1100 nm), Refractive index (1.86) |
| GCaMP Probe | Addgene | G-CaMP3; Plasmid #22692 | A single-wavelength GCaMP2-based genetically encoded calcium indicator |
| Integrating Sphere | Ocean Optics | FOIS-1 | Used for accurate measurement of wide-angle illumination |
| Inverted Fluorescence Microscope | Nikon Instruments | TE2000 | Inverted microscopes allow direct excitation of sample without the need to penetrate layers of media and/or tissue. |
| Mitotracker Green FM | ThermoFisher Scientific | M7514 | Labels mitochondria |
| NIST-Traceable Calibration Lamp | Ocean Optics | LS-1-CAL-INT | A lamp with a known spectrum for use as a standard |
| NIST-Traceable Fluorescein | ThermoFisher Scientific | F36915 | For verifying appropriate spectral response of the system |
| NucBlue | ThermoFisher Scientific | R37605 | Labels cell nuclei |
| Objective (10X) | Nikon Instruments | Plan Apo λ 10X/0.45 ∞/0.17 MRD00105 | Useful for large fields of view |
| Objective (20X) | Nikon Instruments | Plan Apo λ 20X/0.75 ∞/0.17 MRD00205 | Most often used for tissue samples |
| Objective (60X) | Nikon Instruments | Plan Apo VC 60X/1.2 WI ∞/0.15-0.18 WD 0.27 | Most often used for cell samples |
| sCMOS Camera | Photometrics | Prime 95B (Rev A8-062802018) | For acquiring high-sensitivity digital images |
| Spectrometer | Ocean Optics | QE65000 | Used to measure spectral output of excitation-scanning spectral system |
| Tunable Filter Changer | Sutter Instrument | Lambda VF-5 | Motorized unit for automated excitation filter tuning/switching |
| Xenon Arc Lamp | Sunoptic Technologies | Titan 300HP Lightsource | Light source with relatively uniform spectral output |
Ссылки
- Hagen, N. A., Kudenov, M. W. Review of snapshot spectral imaging technologies. Optical Engineering. 52 (9), 90901 (2013).
- Li, Q., He, X., Wang, Y., Liu, H., Xu, D., Guo, F. Review of spectral imaging technology in biomedical engineering: achievements and challenges. Journal of Biomedical Optics. 18 (10), 100901 (2013).
- Lu, G., Fei, B. Medical hyperspectral imaging: a review. Journal of Biomedical Optics. 19 (1), 10901 (2014).
- Mehta, N., Shaik, S., Devireddy, R., Gartia, M. R. Single-Cell Analysis Using Hyperspectral Imaging Modalities. Journal of Biomechanical Engineering. 140 (2), 20802 (2018).
- Goetz, A. F. H. Three decades of hyperspectral remote sensing of the Earth: A personal view. Remote Sensing of Environment. 113, S5-S6 (2009).
- Goetz, A. F. Measuring the Earth from Above: 30 Years(and Counting) of Hyperspectral Imaging. Photonics Spectra. 45 (6), 42-47 (2011).
- Schröck, E. Multicolor spectral karyotyping of human chromosomes. Science. 273 (5274), 494-497 (1996).
- Tsurui, H. Seven-color fluorescence imaging of tissue samples based on Fourier spectroscopy and singular value decomposition. Journal of Histochemistry & Cytochemistry. 48 (5), 653-662 (2000).
- Lu, R., Chen, Y. R. Hyperspectral imaging for safety inspection of food and agricultural products. SPIE. 3544, 121-134 (1999).
- Hege, E. K., O'Connell, D., Johnson, W., Basty, S., Dereniak, E. L. Hyperspectral imaging for astronomy and space surviellance. SPIE. 5159, 380-392 (2004).
- Vo-Dinh, T. A hyperspectral imaging system for in vivo optical diagnostics. IEEE Engineering in Medicine and Biology Magazine. 23 (5), 40-49 (2004).
- Dorrepaal, R. M., Gowen, A. A. Identification of Magnesium Oxychloride Cement Biomaterial Heterogeneity using Raman Chemical Mapping and NIR Hyperspectral Chemical Imaging. Scientific Reports. 8 (1), 13034 (2018).
- Swayze, G. A. Using imaging spectroscopy to map acidic mine waste. Environmental Science & Technology. 34 (1), 47-54 (2000).
- Khoobehi, B., Beach, J. M., Kawano, H. Hyperspectral imaging for measurement of oxygen saturation in the optic nerve head. Investigative Ophthalmology & Visual Science. 45 (5), 1464-1472 (2004).
- Gowen, A., O'Donnell, C., Cullen, P., Downey, G., Frias, J. Hyperspectral imaging-an emerging process analytical tool for food quality and safety control. Trends in Food Science & Technology. 18 (12), 590-598 (2007).
- Edelman, G., van Leeuwen, T. G., Aalders, M. C. Hyperspectral imaging for the age estimation of blood stains at the crime scene. Forensic Science International. 223 (1), 72-77 (2012).
- Edelman, G., Gaston, E., Van Leeuwen, T., Cullen, P., Aalders, M. Hyperspectral imaging for non-contact analysis of forensic traces. Forensic Science International. 223 (1), 28-39 (2012).
- Markgraf, W., Feistel, P., Thiele, C., Malberg, H. Algorithms for mapping kidney tissue oxygenation during normothermic machine perfusion using hyperspectral imaging. Biomedical Engineering/Biomedizinische Technik. 63 (5), 557-566 (2018).
- Boubanga-Tombet, S. Thermal Infrared Hyperspectral Imaging for Mineralogy Mapping of a Mine Face. Remote sensing. 10 (10), 1518 (2018).
- Yuen, P. W., Richardson, M. An introduction to hyperspectral imaging and its application for security, surveillance and target acquisition. The Imaging Science Journal. 58 (5), 241-253 (2010).
- Favreau, P. F. Excitation-scanning hyperspectral imaging microscope. Journal of Biomedical Optics. 19 (4), 046010-046010 (2014).
- Favreau, P. Thin-film tunable filters for hyperspectral fluorescence microscopy. Journal of biomedical optics. 19 (1), 011017-011017 (2014).
- Leavesley, S. J. Hyperspectral imaging microscopy for identification and quantitative analysis of fluorescently-labeled cells in highly autofluorescent tissue. Journal of Biophotonics. 5 (1), 67-84 (2012).
- Annamdevula, N. S. Spectral imaging of FRET-based sensors reveals sustained cAMP gradients in three spatial dimensions. Cytometry Part A. 93 (10), 1029-1038 (2018).
- Deshpande, D. A., Walseth, T. F., Panettieri, R. A., Kannan, M. S. CD38/cyclic ADP-ribose-mediated Ca2+ signaling contributes to airway smooth muscle hyper-responsiveness. The FASEB Journal. 17 (3), 452-454 (2003).
- Deshpande, D. A. Modulation of calcium signaling by interleukin-13 in human airway smooth muscle: role of CD38/cyclic adenosine diphosphate ribose pathway. American Journal of Respiratory Cell and Molecular Biology. 31 (1), 36-42 (2004).
- Guo, M. Cytokines regulate β-2-adrenergic receptor responsiveness in airway smooth muscle via multiple PKA-and EP2 receptor-dependent mechanisms. Biochemistry. 44 (42), 13771-13782 (2005).
- Mansfield, J. R., Gossage, K. W., Hoyt, C. C., Levenson, R. M. Autofluorescence removal, multiplexing, and automated analysis methods for in-vivo fluorescence imaging. Journal of Biomedical Optics. 10 (4), 41207 (2005).
- Mansfield, J. R., Hoyt, C., Levenson, R. M. Visualization of microscopy-based spectral imaging data from multi-label tissue sections. Current Protocols in Molecular Biology. 84 (1), 14-19 (2008).
- Bouchard, M. B. Recent advances in catheter-based optical coherence tomography (OCT) have provided the necessary resolution and acquisition speed for high-quality intravascular imaging. Complications associated with clearing blood from the vessel of a living patient have. Journal of Biomedical Optics. 12 (5), 51601 (2007).
- Mansfield, J. R. Distinguished photons: a review of in vivo spectral fluorescence imaging in small animals. Current Pharmaceutical Biotechnology. 11 (6), 628-638 (2010).
- Levenson, R. M., Mansfield, J. R. Multispectral imaging in biology and medicine: slices of life. Cytometry Part A. 69 (8), 748-758 (2006).
- Gammon, S. T., Leevy, W. M., Gross, S., Gokel, G. W., Piwnica-Worms, D. Spectral unmixing of multicolored bioluminescence emitted from heterogeneous biological sources. Analytical Chemistry. 78 (5), 1520-1527 (2006).
- . . Spectral Unmixing Plugins. , (2006).
- . . Spectral Unmixing of Bioluminescence Signals. , (2006).
- Keshava, N., Mustard, J. F. Spectral unmixing. IEEE Signal Processing Magazine. 19 (1), 44-57 (2002).
- Deal, J. Identifying molecular contributors to autofluorescence of neoplastic and normal colon sections using excitation-scanning hyperspectral imaging. Journal of Biomedical Optics. 23 (12), (2018).
- Microscopy Key, . Microscopy: Key Considerations for Nonlaser Light Sources | Features. BioPhotonics. , (2016).
- Chiu, L., Su, L., Reichelt, S., Amos, W. Use of a white light supercontinuum laser for confocal interference-reflection microscopy. Journal of Microscopy. 246 (2), 153-159 (2012).
- . . Choosing the best light source for your fluorescence experiment. , (2019).
- Beier, H. T., Ibey, B. L. Experimental comparison of the high-speed imaging performance of an EM-CCD and sCMOS camera in a dynamic live-cell imaging test case. PLoS ONE. 9 (1), e84614 (2014).
- Tutt, J. Comparison of EM-CCD and scientific CMOS based camera systems for high resolution X-ray imaging and tomography applications. Journal of Instrumentation. 9 (6), P06017 (2014).
- Coates, C. New sCMOS vs. current microscopy cameras. Biophotonics International. 18 (5), 24-27 (2011).
- Neher, R., Neher, E. Optimizing imaging parameters for the separation of multiple labels in a fluorescence image. Journal of Microscopy. 213 (1), 46-62 (2004).
- Deal, J. Hyperspectral imaging fluorescence excitation scanning spectral characteristics of remodeled mouse arteries. SPIE. 10890, 108902M (2019).
- Deal, J., Rich, T. C., Leavesley, S. J. Optimizing channel selection for excitation-scanning hyperspectral imaging. SPIE. , 108811B (2019).
- Biehlmaier, O., Hehl, J., Csucs, G. Acquisition speed comparison of microscope software programs. Microscopy Research and Technique. 74 (6), 539-545 (2011).
- . . Comparison with other microscopy software - Micro-manager. , (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены