Method Article
كفاءة نوكليك حمض استخراج و16S الريباسي جين تسلسل للبكتيريا الجماعة توصيف
In This Article
Summary
وصفنا وسيلة فعالة وقوية، وفعالة من حيث التكلفة لاستخراج الحمض النووي من مسحات لتوصيف المجتمعات البكتيرية باستخدام 16S الريباسي amplicon الجينات التسلسل. يسمح طريقة لنهج المعالجة المشترك لأنواع عينة متعددة ويستوعب عددا من العمليات التحليلية المصب.
Abstract
هناك إدراك متزايد لدور المجتمعات الميكروبية كما جهري الصحية الحرجة الإنسان والمرض. سمحت تقنيات التسلسل عالية الإنتاجية لتوصيف السريع والفعال للمجتمعات البكتيرية باستخدام 16S الريباسي التسلسل الجيني من مجموعة متنوعة من المصادر. على الرغم من الأدوات المتاحة بسهولة ل16S الريباسي تحليل تسلسل لها سير العمل الحسابية الموحدة، وتجهيز العينات لاستخراج الحمض النووي لا يزال يشكل مصدر استمرار التباين بين الدراسات. نحن هنا وصف وسيلة فعالة وقوية، وفعالة من حيث التكلفة لاستخراج الحمض النووي من مسحات. نحن أيضا رسم طرق المصب لتسلسل الجينات 16S الريباسي، بما في ذلك الجيل من المكتبات التسلسل، ومراقبة جودة البيانات، وتحليل تسلسل. سير العمل يمكن أن تستوعب عدة أنواع العينات، بما في ذلك البراز ومسحات تم جمعها من مجموعة متنوعة من المواقع التشريحية والأنواع المضيفة. بالإضافة إلى ذلك، تعافى DNA و RNA يمكن فصلها واستخدامهالتطبيقات أخرى، بما في ذلك التسلسل الجينوم كله أو RNA وما يليها. الطريقة الموصوفة يسمح لنهج المعالجة المشترك لأنواع عينة متعددة ويستوعب تحليل المصب من المعلومات الجينومية، metagenomic والنسخي.
Introduction
واستعمرت السفلي الإنسان الجهاز التناسلي والجهاز الهضمي والجهاز التنفسي، والجلد من قبل المجتمعات البكتيرية المعقدة التي تعتبر بالغة الأهمية للحفاظ على توازن الأنسجة ودعم صحة المضيف 1. على سبيل المثال، بعض العصيات اللبنية خلق بيئة غير مضيافة لمسببات الأمراض التي تزيد من حامضية المهبل قبو، وإنتاج المؤثرات المضادة للميكروبات وتحوير المضيف المحلي الحصانة 2-4. زاد التقدير المتزايد لأهمية وmicrobiome البكتيرية أيضا مصلحة في وصف المجتمعات البكتيرية في العديد من السياقات السريرية. نحن هنا تصف طريقة لتحديد تكوين microbiome البكتيرية من مسحات التناسلية. بروتوكول يمكن تعديلها بسهولة لعينات البراز ومسحات جمعت من مواقع تشريحية أخرى والأنواع المضيفة الأخرى.
بسبب القيود المتأصلة في عدد العينات التي يمكن جمعها وتخزينها من عشيق معينذ مشارك، وقد تم تصميم هذا البروتوكول لاستخراج الحمض النووي، RNA، وربما حتى البروتين من مسحة واحدة باستخدام تكييفها الفينول كلوروفورم مقرها حبة الضرب طريقة 5،6. مزيج من التعطيل الفعلي لجدران الخلايا البكتيرية مع حبة الضرب وتعطيل الكيميائية مع المنظفات يسمح تحلل سريع من البكتيريا إيجابية الجرام، سالبة الجرام، وحمض السرعة دون خطوات عملية الهضم الأنزيمية إضافية. للحصول على الحمض النووي الريبي جودة عالية، فمن المستحسن استخدام مسحات الجافة التي تم الاحتفاظ بها عند أو أقل من 4 درجات مئوية بعد جمع فورا وخلال النقل إلى المختبر (إن وجدت)، وتخزينها على المدى الطويل في -80 درجة مئوية.
لتحديد microbiome البكتيرية في عينة معينة، هذا الإجراء يستخدم 16S الريباسي amplicon الجينات التسلسل، والذي هو حاليا الوسيلة الأكثر فعالية من حيث التكلفة لتعيين شامل التصنيف البكتيرية وأداء الكمي النسبي. وتشمل طرق بديلة استهدف QPCR 7، microarr مخصصةآيس 8، وكامل تسلسل الجينوم 9. الجينات 16S الريباسي يحتوي على تسعة منطقة التغير الزائد، وليس هناك توافق في الآراء بشأن المنطقة V المثلى لتسلسل للدراسات microbiome المهبل. نحن هنا استخدام مجموعة التمهيدي 515F / 806R وبناء على خط الانابيب الذي صممه Caporaso وآخرون. 10-12. Caporaso الصورة وآخرون. 515F / 806R مجموعة التمهيدي يمكن مضاعفة مئات العينات على المدى تسلسل واحد نظرا لتوافر الآلاف من الاشعال barcoded التحقق من صحة والتوافق مع منصات التسلسل البورشيد. وخلافا للوضع 27F / 338R التمهيدي مشروع Microbiome الإنسان البالغ عددهم 13، 515F / 806R أيضا يزيد من فعالية Bifidobacteriaceae ويلتقط بالتالي بدقة الغاردنريلة المهبلية، وهو عضو مهم في المجتمع الميكروبي المهبل لدى بعض النساء. بدلا من ذلك، تم استخدام زوج التمهيدي 338F / 806R بنجاح لسلسلة البايرو عينات مهبلية 14 وزوج التمهيدي 515F / 926R ديه RECEأصبح ntly متاحة للجيل المقبل من تسلسل 12.
وأخيرا، يقدم هذا البروتوكول التعليمات الأساسية لأداء 16S تحليل amplicon باستخدام رؤى الكمي في إيكولوجيا الجرثومية (QIIME) مجموعة من البرامج (15). نجاح تنفيذ الأوامر QIIME الموصوفة هنا غلة جدول يحتوي على تركيزات التصنيفية البكتيرية لكل عينة. العديد من خطوات إضافية لمراقبة الجودة وطرق تصنيف التصنيفية، وخطوات تحليل يمكن إدراجها في التحليل، كما هو موضح في التفاصيل على موقع QIIME (http://qiime.org/index.html). إذا كان سيتم إجراء تحليل على جهاز كمبيوتر أبل، وحزمة MacQIIME 16 على سهولة التركيب من QIIME وتبعياته. وتشمل حزم البرامج البديلة ل16S الريباسي تحليل تسلسل الجينات Mothur 17 و 18 UPARSE.
Protocol
وتمت الموافقة على بروتوكول الدراسة وفقا للمبادئ التوجيهية للجنة بحوث الطبية الحيوية الأخلاق من جامعة كوازولو ناتال (ديربان، جنوب أفريقيا) ومجلس مستشفى ماساتشوستس العام المراجعة المؤسسية (2012P001812 / MGH، بوسطن، MA).
1. استخراج إجمالي الأحماض النووية من عنقي مهبلي مسحات
ملاحظة: إجراء الاستخراج الحمض النووي في مجموعات من 16 عينة أو أقل. البروتوكول كما هو مكتوب أدناه يفترض تتم معالجة العينات في مجموعات من 12. إذا كان أداء جولات متعددة من الاستخراج، متسلسل ترقيم دفعات استخراج وتسجيل رقم الدفعة استخراج كل عينة وكذلك معلومات العينة الأخرى (بما في ذلك البيانات الفوقية مثل المشارك رقم الهوية، العمر ، تاريخ / وقت جمع مسحة، نوع وسائل منع الحمل الهرمونية، التي تنتقل عن طريق الاتصال الجنسي نتائج الاختبارات العدوى، وما إلى ذلك) في الجدول 1.
- إعداد الكواشف وغطاء الدخان
- إعداد عازلة مكونة من 200 ملي كلوريد الصوديوم (كلوريد الصوديوم)، و 200 ملي تريس، و 20 ملم حمض الإيديتيك (EDTA) في 100 مل من الماء nuclease خالية. تصفية تعقيم الحل عن طريق تمرير من خلال مرشح 0.22 ميكرون. هدئ قسامة 10 مل من العازلة على الجليد الرطب.
- ضبط درجة الحموضة من الفينول: كلوروفورم: كحول أيزو أميلي (IAA) (25: 24: 1) إلى الرقم الهيدروجيني 7.9 وذلك بإضافة 65 ميكرولتر من العازلة قلوية تريس لكل 1 مل الفينول، والهز الخليط لمدة 2 دقيقة، والسماح للمرحلتين ل منفصلة إما طبيعيا أو بواسطة الطرد المركزي في 10000 x ج لمدة 5 دقائق على RT.
تحذير: الفينول سامة إذا ابتلع، في حالة استنشاقه، أو عن طريق ملامسة الجلد والعينين. لا تتنفس الدخان. ارتداء قفازات منيعة، ونظارات السلامة مع الجانب الدروع، ومعطف المختبر. - تصفية تعقيم 25 مل من 20٪ كبريتات الصوديوم دوديسيل (SDS) من خلال مرشح 0.22 ميكرون. جعل 5 مل aliquots من الحزب الديمقراطي الصربي تعقيمها.
- هدئ قسامة 10 مل من الأيسوبروبانول في -20 درجة مئوية.
- إعداد أنبوب حبة الضرب ليالي كلمكتب شؤون المرأة لتتم معالجتها وزنها عن طريق 0.3 غرام من الخرز الزجاجي في العقيمة أنبوب مل 2 التي هي مناسبة لالخافق حبة.
- الحصول على مسحات طريق أخذ عينات من الجزء المهبلي من العنق مع مسحة ماصة معقمة. مباشرة بعد جمع ووضع مسحة إلى cryovial فارغة وعقيمة، تخزينها في 4 درجة مئوية لمدة 1-4 ساعة أثناء النقل إلى المختبر، وتخزينها لعدة أشهر في -80 درجة مئوية. نقل مسحات (الواردة في أنابيب الفردية) التي سيتم تجهيزها لالجليد الرطب.
- إعداد مجلس الوزراء السلامة البيولوجية (BSC). استخدام BSC مع "كشتبان" متصلا العادم بناء لضمان إزالة المناسبة من المواد الكيميائية المتطايرة.
- إزالة جميع المواد من غطاء محرك السيارة.
- تنظيف جميع الأسطح من غطاء محرك السيارة مع التبييض، تليها المطهر الذي يزيل RNases، DNases، والحمض النووي من السطوح. جلبت تنظيف جميع البنود اللاحقة في غطاء محرك السيارة استخدام التبييض تليها مطهر حمض النووي، بما في ذلك القفازات. استخدام ريبونوكلياز / الدناز خالية من الكواشف جديدة، مثل صنصائح ipette، كلما كان ذلك ممكنا.
- الشريط لتعقيم حقيبة واقية كيماوية إلى الجزء الخلفي من غطاء محرك السيارة. يجب وضع كل النفايات الجافة تحتوي على الفينول أو الكلوروفورم في هذه الحقيبة للتخلص السليم.
- ضع زجاجة معقمة في غطاء محرك السيارة لجمع النفايات السائلة التي تحتوي على الفينول أو الكلوروفورم.
- استخراج الفينول كلوروفورم.
- في غطاء محرك السيارة، إلى كل أنبوب الضرب حبة، إضافة 500 ميكرولتر من العازلة (من الخطوة 1.1.1) و 210 ميكرولتر من 20٪ كبريتات الصوديوم دوديسيل، و 500 ميكرولتر من الفينول: الكلوروفورم: IAA (25: 24: 1، ودرجة الحموضة 7.9 ).
- نقل مسحة من القارورة النقل في أنبوب حبة الضرب باستخدام زوج جديد من ملقط معقم. فرك جيدا الرأس مسحة على الجدران الداخلية للأنبوب حبة الضرب لا يقل عن 30 ثانية. إعادة رسملة العينة عند الانتهاء. إذا كان أداء الاستخراج من مسحات متعددة، تغيير القفازات بين كل عينة.
- هدئ العينة على الجليد لمدة 10 دقيقة على الأقل. إزالة مسحة منحبة ضرب أنبوب من خلال عقد مقبض مسحة مع ملاقط معقمة أثناء الضغط على رئيس مسحة ضد جدار الأنبوب الداخلي باستخدام تلميح P200 نظيفة. تجاهل مسحات في كيس النفايات الكيميائية الجافة. ملاحظة: إن "ممسحة" عمل (الضغط على رئيس مسحة) تحرير السائل من مسحة ماصة وزيادة الانتعاش الحمض النووي.
- وضع أنبوب حبة الضرب في الخافق حبة والتجانس لمدة 2 دقيقة على 4 درجات مئوية.
- أجهزة الطرد المركزي في أنبوب الضرب حبة لمدة 3 دقائق في 6000 x ج و 4 درجات مئوية لتكوير الحطام وفصل المراحل المائية والفينول.
- نقل المرحلة المائية (~ 500-600 ميكرولتر) إلى أنبوب 1.5 مل العقيمة. إضافة حجم مساو من الفينول: الكلوروفورم: IAA. مزيج من قبل انقلاب وvortexing لوجيزة.
- أنبوب الطرد المركزي لمدة 5 دقائق في 16000 x ج و 4 درجات مئوية.
- نقل المرحلة المائية لالعقيمة أنبوب 1.5 مل جديد. كن متحفظا ولا نقل المواد من طبقة الطور البيني أو الفينول الكامنةمرحلة. لاحظ حجم المرحلة المائية المنقولة. حفظ مرحلة الفينول لعزل بروتين في المستقبل.
- إضافة 0.8 حجم الأيزوبروبانول و 0.1 حجم 3M خلات الصوديوم (5.5 درجة الحموضة). مزيج دقيق من قبل قلب وvortexing لفترة وجيزة.
- ترسيب الحمض النووي التي تقشعر لها الأبدان أنبوب في -20 درجة مئوية لمدة لا تقل عن 2 ساعة (حتى O / N).
- هطول الأيسوبروبانول وغسل الإيثانول
- أنبوب الطرد المركزي لمدة 30 دقيقة في ما يقرب من 16000 x ج و 4 درجات مئوية. بعناية استخدام ماصة لإزالة طاف، وترك بيليه سليمة.
- إضافة 500 ميكرولتر من الإيثانول بنسبة 100٪. طرد بيليه مع vortexing لطيف أو pipetting لدون لمس بيليه. أجهزة الطرد المركزي لمدة 5 دقائق في 16000 x ج و 4 درجات مئوية.
- بعناية تجاهل طاف الإيثانول. استخدام الماصة P10 لإزالة أكبر قدر من الايثانول ممكن من دون إزعاج بيليه.
- الهواء الجاف بيليه في RT لمدة 15 دقيقة.
- Resuspend وبيليه في 20 ميكرولتر من شltra نقي 0.1X عازلة تريس، EDTA. السماح للعينة البرد على الجليد لمدة 10 دقيقة وماصة مرارا وتكرارا لضمان إعادة تعليق كامل. إذا لم يكن بيليه تذوب، ونقل الأنبوب إلى كتلة C حرارة 40 درجة لمدة تصل إلى 10 دقيقة للمساعدة في حل.
- قياس تركيز الحمض النووي باستخدام معمل 19.
- إذا رغبت في ذلك، والحمض النووي منفصلة من الحمض النووي الريبي باستخدام طقم تنظيف العمود، وبعد بروتوكول الشركة المصنعة 20.
- تخزين الحمض النووي في -80 درجة مئوية أو متابعة.
2. PCR التضخيم من 16S الريباسي جين V4 Hypervariable منطقة
ملاحظة: إجراء التضخيم PCR في مجموعات من 12 عينة أو أقل لتقليل مخاطر التلوث والخطأ البشري. إذا كان أداء جولات متعددة من التضخيم، متسلسل ترقيم دفعات التضخيم وتسجيل عدد التضخيم دفعة كل عينة في الجدول 1.
- إعداد سو الكواشف وPCR غطاء محرك السيارة
- إضافة PCR التضخيم المعلومات الواردة في الجدول رقم 1، والذي سيكون بمثابة أساس ملف التعيين في مرحلة تحليل تسلسل.
- إزالة جميع المواد من غطاء PCR وتنظيف الأسطح الداخلية تماما مع التبييض تليها المطهر الذي يزيل RNases، DNases، والحمض النووي. تأكد من تطهير كل كاشف وقطعة من المعدات (على سبيل المثال، الماصات) قبل وضعها في غطاء محرك السيارة. ارتداء قفازات جديدة تنظيفها مع مطهر حمض النووي قبل العمل في غطاء محرك السيارة.
- إذا لزم الأمر، وتمييع القالب الحمض النووي إلى 50-100 نانوغرام / ميكرولتر باستخدام المياه خالية من الحمض النووي وnuclease خالية.
- قسامات ذوبان الجليد من 5X عالية الدقة (HF) العازلة، dNTPs، والاشعال في غطاء محرك السيارة PCR نظيفة. بلطف دوامة وأجهزة الطرد المركزي عن حلول بعد ذوبان الجليد. للحد من تجميد أذاب دورات وخطر التلوث الأوراق المالية، وإعداد قسامات من 5X HF العازلة، dNTPs، والاشعال.
- ضعالفوق نظيفة رف برودة لأنابيب microcentrifuge ولوحة برودة PCR في غطاء محرك السيارة.
- للتفاعل PCR، وإعداد مزيج الرئيسي من خلال الجمع بين 15.5 ميكرولتر من نقي جدا المياه، 5 ميكرولتر من العازلة 5X HF، 0.5 ميكرولتر من dNTPs، 0.5 ميكرولتر من 515F التمهيدي إلى الأمام، 0.75 ميكرولتر من 3٪ DMSO، و 0.25 ميكرولتر من البلمرة ل كل رد فعل. تجميع جميع مكونات رد فعل في برودة وإضافة البلمرة الماضي. مزيج دقيق من قبل pipetting. إضافة عينتين إضافية لعدد رد فعل عند إعداد مزيج الرئيسي، لحساب الخطأ pipetting ل.
- إعداد تفاعل PCR:
ملاحظة: إجراء التكبير في ثلاث نسخ، وهذا يعني تضخيم كل عينة في ثلاثة 25 ميكرولتر ردود فعل منفصلة. تشغيل التحكم في المياه لم قالب مع كل زوج التمهيدي. العمل بسرعة ولكن بعناية، وتجنب إدخال أي تلوث.- تسمية مهبط 8 جيدا مع قبعات الفردية والمكان إلى برودة PCR.
- ماصة 90 ميكرولتر من مزيج الرئيسي في أولحسنا.
- إضافة 2 ميكرولتر من التمهيدي العكسي (التكميلي ملف 1). مما لا شك فيه أن نلاحظ بعناية الباركود التمهيدي العكسي المستخدمة مع كل عينة في الجدول 1.
- تخلط جيدا ونقل 23 ميكرولتر من مزيج الرئيسي إلى البئر الرابع (سيطرة أي قالب).
- إضافة 2 ميكرولتر من الماء إلى البئر الرابعة.
- إضافة 6 ميكرولتر من العينة المناسبة لأول بئر. تخلط جيدا ونقل 25 ميكروليتر إلى البئر الثانية. نصائح التغيير ونقل 25 ميكرولتر آخر من البئر الأول إلى الثالث أيضا. تتويج بحزم كل جيدا، والتأكد من عدم لمس داخل الآبار أو غطاء في هذه العملية.
- كرر لكل عينة.
- أداء PCR التضخيم
- نقل أنابيب الشريط لthermocycler وتشغيل البرنامج التالي: 30 ثانية في 98 درجة مئوية، تليها 30 دورات من 10 ثانية في 98 درجة مئوية، و 30 ثانية في 57 درجة مئوية، و 12 ثانية في 72 درجة مئوية، تليها عقد 10 دقيقة في 72 درجة مئوية والأخير عقد لر 4 درجة مئوية.
- قم بالخطوات التالية على مقاعد البدلاء المختبر نظيفة. تدور بسرعة أنابيب لجمع السائل من الجدران. الجمع بين ردود الفعل PCR ثلاث نسخ من كل عينة، مع إجمالي حجم 75 ميكرولتر، في أنبوب المسمى العقيمة. كما نقل 25 ميكرولتر من كل سيطرة أي قالب في أنبوب معقم منفصل. لا تجمع بين amplicons من عينات مختلفة حتى الان.
- التحقق من نجاح التضخيم PCR من العينات عن طريق الكهربائي للهلام.
- إعداد agarose هلام 1.5٪ (1.5 غرام من مسحوق الاغاروز في 100 مل من العازلة 1X تاي) مع آبار كافية لاجراء كل amplicon، التحكم في المياه، وسلم 21.
- في حين أن يصلب هلام (حوالي 30 دقيقة)، وإعداد نموذج لالكهربائي: إضافة 1 ميكرولتر من صبغ 6X تحميل لأنبوب جديد، وصفت. إلى أن أنبوب، إضافة 5 ميكرولتر من amplicon ومزيج من قبل pipetting.
- وعندما وضعت جل، وإزالة أمشاط، ووضع جل في الخزان الكهربائي، وملء خزان العازلة مع 1X تاي.
- لأول بئر، إضافة 5 ميكرولتر من سلم ال DNA.
- تحميل 5 ميكرولتر من amplicon عينة إلى أخرى بشكل جيد. تحميل 5 ميكرولتر من amplicon عدم القالب لبئر منفصل. مواصلة حسب الحاجة لكل عينة.
- عندما تم تحميل جميع العينات، حرك غطاء خزان في مكان وتشغيل مصدر الطاقة إلى 120 V. السماح للهلام لخوض 30 - 60 دقيقة.
- عرض هلام تحت ضوء الأشعة فوق البنفسجية.
- تحقق من التضخيم الناجح لكل عينة بالإشارة إلى فرقة قوية واحدة نحو 380 نقطة أساس. إذا كان هناك فرقة مزدوجة، وإعادة تضخيم عينة مع الباركود العكسي مختلفة (الخطوة 2.3). إذا لم يكن هناك فرقة في كل شيء، وإعادة تضخيم عينة باستخدام نفس الباركود العكسي أو الباركود العكسي جديد (الخطوة 2.3). إذا إعادة التضخيم غير ناجحة، قد تكون مثبطات PCR في العينة، وفي هذه الحالة، إجراء تنظيف الحمض النووي القائم على عمود لإزالة مثبطات PCR.
ملاحظة: التضخيم الناجح قد لا يكون ممكنا إذا كان تركيز الحمض النووي للبكتيريا في أوعينة iginal هي كافية (<5 نانوغرام / ميكرولتر). - التحقق من عدم وجود تلوث كاشف بالإشارة إلى عدم وجود الفرقة في السيطرة على أي قالب.
- تحقق من التضخيم الناجح لكل عينة بالإشارة إلى فرقة قوية واحدة نحو 380 نقطة أساس. إذا كان هناك فرقة مزدوجة، وإعادة تضخيم عينة مع الباركود العكسي مختلفة (الخطوة 2.3). إذا لم يكن هناك فرقة في كل شيء، وإعادة تضخيم عينة باستخدام نفس الباركود العكسي أو الباركود العكسي جديد (الخطوة 2.3). إذا إعادة التضخيم غير ناجحة، قد تكون مثبطات PCR في العينة، وفي هذه الحالة، إجراء تنظيف الحمض النووي القائم على عمود لإزالة مثبطات PCR.
- تخزين 70 ميكرولتر المتبقية من amplicon في -20 درجة مئوية. تجاهل 20 ميكرولتر المتبقية من سيطرة أي قالب، على افتراض أنها لم تثمر عن الفرقة.
3. مكتبة تجميع وتسلسلها الإنتاجية العالية
- إنشاء تجمع amplicon من خلال الجمع بين حجم مساو (2-5 ميكرولتر) من كل amplicon في أنبوب معقم واحد. إذا كانت الفرقة من عينة بدت ضعيفة بشكل خاص، إضافة مرتين حجم نسبة إلى بقية العينات.
- إزالة الاشعال PCR من تجمع amplicon استخدام عدة PCR التنظيف، باتباع إرشادات الشركة المصنعة ل22. إجراء تنظيف مع أعمدة متعددة إذا كان حجم تجمع amplicon أكثر من 100 ميكرولتر. ملاحظة: كل عمود لديه قدرة 100 ميكرولتر.
- تخزين مكتبة في -20 & #176؛ ج أو انتقل إلى الخطوة التالية.
- إذا كان ذلك ممكنا، والجمع بين برك amplicon خالية من التمهيدي إلى إنشاء مكتبة النهائية. تحديد تركيز الحمض النووي من المكتبة باستخدام معمل أو نظام فلوروميتريك 23. وهناك نسبة 260/280 بين 1،8-2،0 هي مؤشر على الحمض النووي محض.
- تمييع المكتبة إلى 20 نانومتر. التأكد من نوعية المكتبة من خلال وضع تصور لفرقة واحدة حوالي 400 شركة بريتيش بتروليوم باستخدام أداة الكهربائي. تأكيد تركيز المكتبة باستخدام نظام فلوروميتريك 23.
- إجراء النهائي 01:10 التخفيف في الماء لتخفيف المكتبة إلى 2 نانومتر. ثم، تخزين مكتبة عند 20 درجة مئوية إلى أجل غير مسمى.
- إرسال قسامة من مكتبة النهائية مع الاشعال التسلسل المطلوبة الثلاث (اقرأ 1، اقرأ 2، ومؤشر، وانظر جداول المواد / المعدات) لتكون متسلسلة على التسلسل البورشيد. إذا تم المضاعفة أقل من 300 عينة لتسلسل، استخدم نهاية واحدة 300 نقطة أساس المدى مع وجود مؤشر 12 نقطة أساس قراءة سغ MiSeq، مع تركيز مكتبة النهائي من 5:00 والتشويه والتحريف PhiX ارتفاع في 10٪. انظر المواد التكميلية Caporaso وآخرون. ISME J، 2012 10 للحصول على تعليمات مفصلة التسلسل.
تحليل 4. تسلسل
ملاحظة: المبينة هنا هي خط أنابيب الأساسي لتحليل تسلسل باستخدام حزمة برامج QIIME 1.8.0. لالبساطة، تفترض الأوامر بشرط أن يكون ملف تعيين يسمى mapping.txt، قراءة مؤشر 12 نقطة أساس يسمى ملف index.fastq، والتسلسل 300 نقطة أساس قراءة يسمى ملف sequences.fastq. تثبيت QIIME أو MacQIIME 16 و تآلف مع أساسيات يونيكس لتنفيذ هذه الأوامر. قراءة دليل كامل لQIIME في:
- استكمال ملف التعيين للتجربة (الجدول 1). تشمل التعريف قدر الإمكان. ملاحظة التي تم استخراج عينات أو تضخيمها في نفس الدفعة، لتحديد ما إذا كانت هناك آثار دفعة واحدة. حفظ ملف تعيين كملف نصي، على سبيل المثال، mapping.txt. تحقق من صحة تنسيق ملف التعيين عن طريق تنفيذ الأمر التالي: validate_mapping_file.py -m mapping.txt -o mapping_output
- التحقق من جودة تسلسل يقرأ باستخدام عالية الإنتاجية تسلسل جودة البيانات التحقق من البرنامج، مثل FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/). يوضح الشكل 5 في تسلسل قاعدة الجودة ما يمكن توقعه من مسيرته الناجحة.
ملاحظة: المنظم يعين كل قاعدة النوكليوتيدات على درجة جودة PHRED، والتي تتطابق مع احتمال أن القاعدة قد سمى خطأ. والنتيجة PHRED جودة من 10 تشير إلى أن هناك فرصة 10٪ أن النوكليوتيدات وقد تم بشكل غير صحيح تعيينالطبعه، 20 يشير إلى وجود فرصة بنسبة 1٪، 30 يشير إلى وجود فرصة بنسبة 0.1٪، و 40 (أعلى درجة ممكنة) يشير إلى 0.01٪ فرصة 24 ساعة. - باستخدام ملف تعيين كمفتاح، demultiplex، جودة تصفية البيانات التسلسل، وحفظ النتائج إلى مجلد (في هذه الحالة، ودعا "sl_out") من خلال تنفيذ هذا الأمر 25: split_libraries_fastq.py --rev_comp_mapping_barcodes -i sequences.fastq -o sl_out / -b index.fastq -m mapping.txt -q 29
ملاحظة: العلم ف يدل على أقصى درجة غير مقبولة PHRED جودة، على سبيل المثال، "-q 29" بتصفية أي تسلسل مع عشرات PHRED أقل من 30، وضمان 99.9٪ من الدقة من المكالمات قاعدة. - باستخدام التشغيلية الوحدة التصنيفية (اتو) قاعدة بيانات مرجعية Greengenes 16S 26 (http://qiime.org/home_static/dataFiles.html)، نفذ مرجعية مفتوحة OTU اختيار من تنفيذ هذا الأمر 27: pick_open_reference_otus.py -i sl_out / seqs. سانا -r 97_otus.fasta -o ucrss / -s 0.1
ملاحظة: يشير العلم -sجزء من متواليات التي فشلت في محاذاة إلى قاعدة البيانات المرجعية التي سيتم تضمينها في التجميع من جديد. "-s 0.1" يضم 10٪ من تسلسل فشلت في تجميع من جديد. استخدام العلم -a لتتم بشكل مواز لعملية اختيار OTU وتقليل وقت المعالجة من أيام إلى ساعات إذا كانت النوى المتعددة المتاحة. - إنشاء جدول فرة التصنيفية سهلة الاستخدام عن طريق دمج OTUs على مستوى الأنواع من تنفيذ هذا الأمر 28: summarize_taxa.py -i ucress / otu_table_mc2.biom -o summarized_otuSpecies / -L 7
ملاحظة: الجدول الناتج يمكن مشاهدتها بسهولة في أي برنامج جداول البيانات. لاحظ أن 16S الريباسي التسلسل لا يوفر موثوق قرار على مستوى الأنواع. - تحديد التنوع البيئي داخل كل عينة عن طريق حساب عدة مقاييس التنوع ألفا مع alpha_diversity.py النصي QIIME. ثم، تحديد التنوع بين أزواج من العينات باستخدام البرنامج النصي beta_diversity.py QIIME.
- فيماualize البيانات، على سبيل المثال، باستخدام مبدأ الإمبراطور 29 ينسق مؤامرة أو خريطة التمثيل اللوني.
- أداء مقارنات إحصائية رسمية للفئات ملف التعيين، على سبيل المثال، مع النصي compare_catagories.py QIIME البالغ عددهم 30.
ملاحظة: يستخدم هذا الأمر المدمج في "validate_mapping_file.py" النصي QIIME أن يجعل مجلد جديد، يسمى "mapping_output"، التي تحتوي على ملف .html تشير إلى أخطاء ملف التعيين، إن وجدت.
النتائج
لمحة عامة عن بروتوكول، والتي تمكن من تحديد الكميات المتوفرة البكتيرية النسبية من مسحة باستخدام 16S الريباسي التسلسل الجيني، ويظهر في الشكل وقد تم تحسين بروتوكول 1. ولمسحات مهبلية الإنسان، ولكن يمكن أن تتكيف بسهولة لمعظم مواقع أخذ العينات المخاطية والمضيفين الآخرين. ويوضح الشكل 2 الحمض النووي ذات جودة عالية والحمض النووي الريبي التي يمكن أن تكون معزولة باستخدام بروتوكول حبة الضرب الشكل 3 يوضح التضخيم PCR ناجحة من 12 عينة، حيث كل التضخيم مع عينة أسفرت عن فرقة قوية واحدة من الصحيح حجم وكل التحكم في المياه لم تسفر عن الفرقة الشكل 4 يوضح الكمي لتجمع مكتبة النهائي قبل التسلسل. ويبين الشكل 5 لمحة تسلسل جودة نموذجي بعد نهاية واحدة 300 نقطة أساس MiSeq التشغيل.
"SRC =" / ملفات / ftp_upload / 53939 / 53939fig1.jpg "/>
الشكل 1. تخطيطي لمحة عامة عن البروتوكول. أولا، يتم استخراج الحمض النووي من مسحة من حبة الضرب في حل مخزنة تحتوي على الفينول، والكلوروفورم، وأيزو أميلي الكحول. ثم يتم تضخيمه منطقة متغيرة 4 من الجينات 16S الريباسي من الحمض النووي الناتجة باستخدام PCR. amplicons PCR من ما يصل الى مئات العينات ثم يتم الجمع بين والتسلسل على تشغيل واحد. تتم مطابقة تسلسل مما أدى إلى قاعدة بيانات مرجعية لتحديد الكميات المتوفرة البكتيرية النسبية. لا يمكن أن يؤديها البروتوكول بكامله في ما يقرب من ثلاثة أيام. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
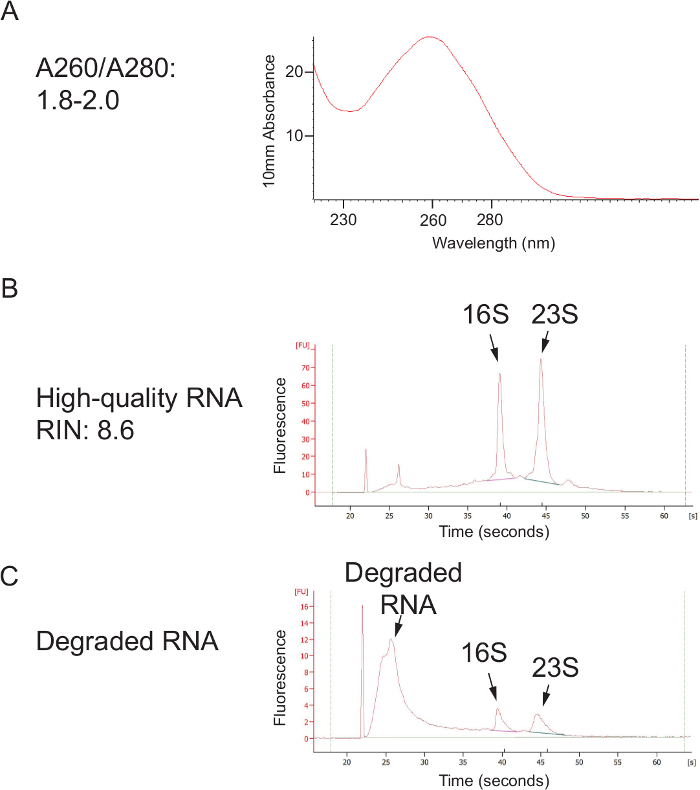
الشكل 2. ذات جودة عالية نوكليك حمض المستخرجة باستخدام الفينول: كلوروفورم حبة الضرب Method. (أ) نوعية الحمض النووي، وفقا لتقييم باستخدام مقياس الطيف. نسبة A260 / A280 بين 1.8 و 2.0 تشير الحمض النووي النقي الذي لا الملوثة مع الفينول أو البروتين. (ب) بعد عمود تنظيف، يمكن هذا البروتوكول تسفر عالية الجودة RNA، وأشار قوية قمم 16S و 23S الريباسي. يمكن أن يحدث (C) تدهور RNA إذا لم يتم التقيد بهذا عينة البرد بعد جمع (أثناء النقل والتخزين) أو إذا RNases موجودة أثناء معالجة. الرجاء النقر هنا لمشاهدة نسخة أكبر من هذا الرقم.
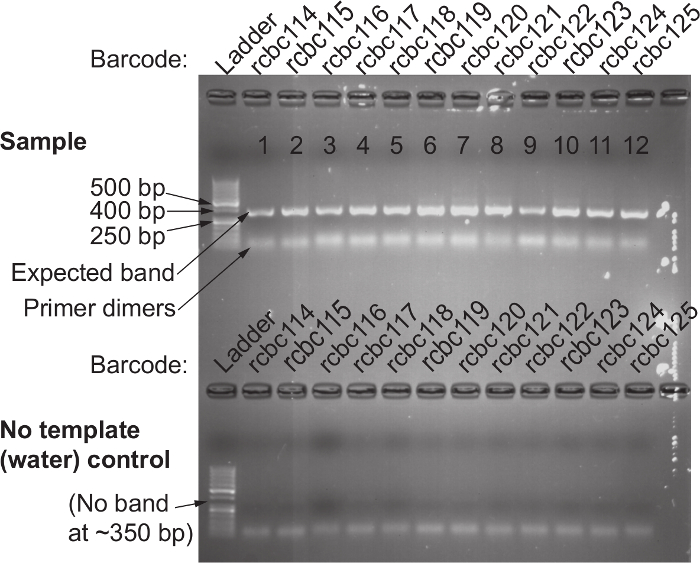
الشكل 3. تأكيد نجاح 16S الريباسي جين التضخيم باستخدام 515F وBarcoded 806R التمهيدي تعيين. يستخدم العلوي) جل الكهربائي لتأكيد وجود فرقة واحدة نحو 380 زوج قاعدي طن كل العينة التي تم تضخيم مع القالب. عدم وجود فرقة يشير التضخيم ناجحة. هذا عادة ما يكون بسبب خطأ بشري وتفاعل PCR من ذلك ينبغي تكرار العينة. أسفل) لا قالب (تشغيل المياه) ضوابط بالتوازي مع نفس الزوج التمهيدي لا ينبغي أن يكون عصابة الحاضر. وجود الفرقة في التحكم في المياه يشير الكواشف الملوثة. تجاهل الكواشف التي قد تكون ملوثة وإعادة تفعل التكبير PCR من كل من السيطرة قالب والماء لهذا الزوج التمهيدي. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
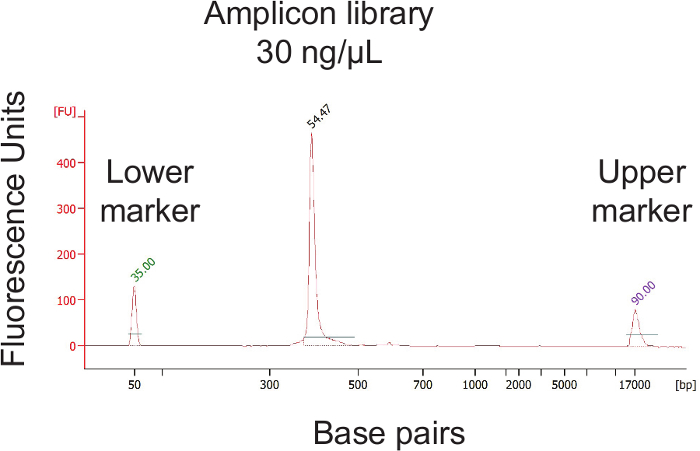
الشكل 4. الكمي لتركيز النهائي مكتبة حمام والتحقق من صحة مكتبة الحجم بعد تجميع amplicons عينة الفردية، اليجب تحديد تركيز (ه) من تجمع مكتبة النهائي. بركة مكتبة ثم يجب إجراء مزيد من المخفف لتحقيق تركيز نانومتر (2). الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
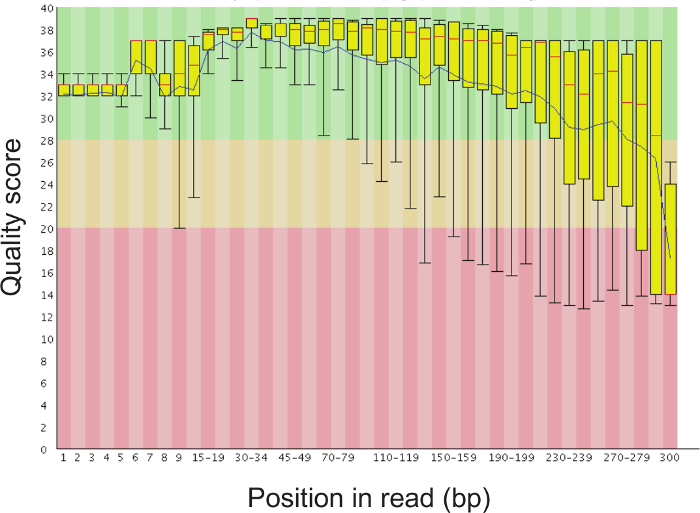
الرقم 5. ممثل بار قطعة نقاط الجودة تسلسل في كل موقف للقراءة. فمن الطبيعي للجودة تسلسل لإسقاط بعد 200 زوج قاعدي، ولكن ينبغي أن تظل متوسط درجة جودة أعلى 30. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
| #SampleID | الباركود تسلسل | LinkerPrimer تسلسل | rcbcPrimer | SampleType | استخلاص دفعة | توسيع طبق | وصف | |||
| ويمكن الاطلاع على #An ملف سبيل المثال رسم الخرائط في: http://qiime.org/_static/Examples/File_ صيغ / مثال_ رسم الخرائط_ File.txt | ||||||||||
| AG2350 | TCCCTTGTCTCC | CCGGACTACHVGGGTWTCTAAT | rcbc000 | عنقي مسح | 1 | ا | ||||
الجدول 1. رسم الخرائط ملف قالب. إنشاءتينغ ملف رسم دقيق وشامل هو أمر حاسم لتنفيذ بروتوكول بنجاح. لم يكن مطلوبا من ملف التعيين فقط لتنفيذ QIIME، ولكنه يمكن أيضا الباحث إلى الحفاظ على الصلة بين الباركود العينة والبيانات الوصفية، لتحليل البيانات عن أي تحيزات منهجية (على سبيل المثال، دفعة إلى دفعة الاختلاف)، وتحديد الارتباطات مثيرة للاهتمام بين البيانات الوصفية والسكان البكتيرية. ويرد ملف تعيين البدائي، ولكن يتم تشجيع المستخدمين على إضافة العديد من الأعمدة التي تحتوي على بيانات التعريف وقت ممكن. أمثلة على بيانات تعريف إضافية لمسحة المهبل تشمل المشارك العمر، تاريخ / وقت جمع مسحة، الهرموني نوع وسائل منع الحمل (إن وجدت)، التي تنتقل بالاتصال الجنسي العدوى اختبار النتائج، الخ
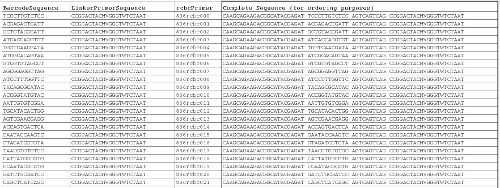
خلفية ملف 1. قائمة Barcoded عكسي متواليات التمهيدي 10 . الأعمدة الثلاثة الأولى يمكن استخدامها لاستكمال ملف التعيين، ويوفر العمود الأخير في تسلسل التمهيدي كامل لترتيب الأغراض. يرجى النقر هنا لتحميل هذا الملف.
Discussion
نحن هنا وصف بروتوكول لتحديد وتوصيف وفرة البكتيرية النسبية داخل مسحة مهبلية الإنسان. يمكن بسهولة أن تتكيف هذا البروتوكول بالنسبة للأنواع الأخرى عينة، مثل البراز ومسحات من مواقع أخرى من الجسم، والعينات التي تم جمعها من مجموعة واسعة من المصادر. استخراج الحمض النووي التي كتبها حبة الضرب في حل مخزنة من الفينول وكلوروفورم يسمح لعزل كل من الحمض النووي والحمض النووي الريبي، وهو أمر مهم بشكل خاص عند العمل مع العينات الثمينة التي تم جمعها من خلال الدراسات السريرية. الحمض النووي للبكتيريا معزولة ممتاز لتحديد التصنيف البكتيرية والتجمع الجيني، في حين جمع في وقت واحد من الحمض النووي الريبي يوفر فرصة لتحديد بكتيريا وظيفية، المضيفة، والمساهمات الفيروسية من خلال الحمض النووي الريبي يليها. يستخدم بروتوكول صفها على التحقق من صحة مجموعة التمهيدي من خطوة واحدة التي تم نشرها بنجاح على مجموعة واسعة من أنواع العينات، بما في ذلك الإنسان، والكلاب، والعينات البيئية 10 . توفر الآلاف من الاشعال barcoded يمكن مضاعفة العينات وفورات هائلة في التكاليف التسلسل. التكلفة الكاملة (بما في ذلك جميع المواد الكيميائية، تشغيل تسلسل واحد، والاشعال ولكن ليس المعدات) حوالي 20 $ لكل عينة عندما يتداخل 200 عينة. بالإضافة إلى ذلك، هناك استنساخ عالية جدا عند معالجة مسحات متعددة من نفس الموقع عينة بشكل مستقل من خلال خط أنابيب بأكمله. وعموما، فإن تكلفة بروتوكول يتسم بالكفاءة والمرونة، يمكن الاعتماد عليها، وتكرار.
والنووي جزء استخلاص الحمض من هذا البروتوكول هو محدود من احتياطات السلامة اللازمة عند التعامل مع الفينول والكلوروفورم، وتحديات أتمتة خط أنابيب إلى الإنتاجية العالية، وشكل لوحة 96-جيدا. بالإضافة إلى ذلك، حبة الضرب قوية تستخدم لمقص تحلل الميكانيكية الحمض النووي البكتيري إلى ما يقرب من 6 أجزاء كيلو قاعدة. إذا كانت هناك حاجة شظايا الحمض النووي أطول لتطبيقات المصب، مدة حبة الضرب الصورةيمكن اختصار hould معها. القيود المفروضة على جزء تحديد البكتيريا من هذا البروتوكول هي ملازمة لأي الطريقة التي تعتمد على التسلسل الجيني 16S الريباسي. 16S الريباسي التسلسل مثالية لتحديد البكتيريا إلى مستوى جنس وحتى الأنواع، ولكن نادرا ما توفر لهم تحديد مستوى الضغط. في حين أن المنطقة المتغيرة V4 من الجينات 16S الريباسي توفر التمييز القوي بين معظم الأنواع البكتيرية 11، قد تحتاج إلى استخدامها لتحديد بالضبط أنواع معينة، مثل crispatus الملبنة طرق حسابية إضافية مثل Oligotyping 31. أخيرا، معلومات عن القدرات الوظيفية البكتيرية دقيقة ضمن عينة معينة لا يمكن تحديده من خلال 16S الريباسي التسلسل الجيني وحده، رغم أن هذا البروتوكول يتيح استخراج الحمض النووي الجينوم بأكمله والحمض النووي الريبي التي يمكن استخدامها لتحقيق هذا الغرض.
الخطوة الأكثر أهمية لضمان نجاح مع هذا البروتوكول هو رعاية كبيرة لمنع التلوث الدرجي جمع العينات، واستخراج الحمض النووي، وPCR التضخيم. ضمان العقم عند جمع العينات من خلال ارتداء قفازات نظيفة واستخدام مسحات معقمة والأنابيب، ومقص. لتقييم لتلوث المواد جمع، وجمع مسحات سيطرة سلبية عن طريق وضع مسحة غير المستخدمة إضافية مباشرة في أنابيب النقل في وقت أخذ العينات. في المختبر، وتنفيذ كافة الخطوات قبل التضخيم في غطاء معقمة تحتوي على المواد فقط تطهير واستخدام درجة الجزيئي الوحيد، الكواشف خالية من الحمض النووي. خلال استخلاص الحمض النووي، ومنع انتقال التلوث باستخدام ملقط معقم جديدة وقفازات جديدة مع كل عينة، وحفظ جميع أنابيب مغلقة إلا في الاستخدام. تجهيز مسحات غير المستخدمة بالتوازي يضمن عقم كل من جمع العينات واستخلاص الحمض النووي. يجب مسحات غير المستخدمة لا تسفر عن بيليه بعد هطول الأمطار الأيسوبروبانول وغسل الايثانول. إذا لم تظهر بيليه، نفذ 16S الريباسي التضخيم الجيني لتحديد مصدر محتمل للالتلوث (على سبيل المثال، فإن وجود بكتيريا المكورات العنقودية أو تشير إلى تلوث الجلد). بالإضافة إلى ذلك، نفذ التكبير PCR مع عدم وجود ردود فعل تحكم قالب بالتوازي مع ضمان الكواشف PCR وردود الفعل لم تكن ملوثة. إذا ظهرت الفرقة في السيطرة على أي قالب، تجاهل الكواشف وتكرار التضخيم مع الكواشف جديدة. سوف اتخاذ هذه الاحتياطات ضمان تسلسل الناجح للبكتيريا من الفائدة.
الخطوة التضخيم PCR تميل إلى تتطلب معظم استكشاف الأخطاء وإصلاحها. تضخيم في مجموعات من اثني عشر عينات يوفر التوازن بين الكفاءة والاتساق. الغياب الكامل للفرق في جميع العينات في مجموعة التضخيم نظرا إلى وجود فشل منهجي، على سبيل المثال، وينسى لإضافة كاشف أو برمجة thermocycler بشكل غير صحيح. ومن المقرر عادة إلى خطأ بشري لعدم وجود فرقة من بعض العينات، والتكبير ينبغي إعادة صالامم المتحدة مع نفس المزاوجة بين العينة والتمهيدي عكسي. في حالة استمرار عدم وجود الفرقة، وعينة يمكن إعادة تضخيم باستخدام التمهيدي العكسي مع الباركود مختلفة. فشل التضخيم المتكررة مع الاشعال عكس متعددة قد تشير إلى وجود المانع في العينة. في هذه الحالة، وتنظيف الحمض النووي مع عمود في كثير من الأحيان إزالة مثبطات دون تغيير ملحوظ فرة البكتيرية النسبية. إذا نطاقات متعددة تنتج بعد التضخيم، وإعادة تضخيم عينة مع مختلف الباركود التمهيدي عكسي.
بالإضافة إلى منع التلوث البيئي وضمان التضخيم من منتج معين واحد، ويعتمد التسلسل الناجح على الرعاية عند إعداد تجمع المكتبة. والهدف هو الجمع بين كميات متساوي المولية من amplicons كل عينة لضمان تقريبا نفس العدد من التسلسل يقرأ في العينة. إذا كان تركيز الحمض النووي قبل التضخيم قابلة للمقارنة، ببساطة بإضافة كميات متساوية من كل ساamplicons mple هي كافية عند إنشاء تجمع المكتبة. ومع ذلك، إذا كان تركيز الحمض النووي هي مختلفة إلى حد كبير، وأضاف في حجم مساو، على عينة مع تركيز منخفض الحمض النووي سوف تكون ممثلة تمثيلا ضعيفا مع عدد قليل من يقرأ. في هذه الحالة، فمن الممكن لإضافة عدد أكبر من amplicons من العينة تركيز منخفض على أساس الكثافة النسبية للفرقة هلام. بدلا من ذلك، فمن الممكن لإزالة أكثر صرامة الاشعال من amplicons الفردية وقياس تركيز amplicon عينة الفردية في استخدام فلوروميتريك عدة dsDNA الكمي، وعلى وجه التحديد تجمع كميات متساوي المولية من كل عينة.
مرة واحدة يتم إنشاء تجمع amplicon متوازن، يصبح من المهم جدا قياس بعناية تركيز على حمام السباحة و. تخفيف دقيق لاحق وارتفاع في مع PhiX لزيادة تعقيد قراءة هو أمر حاسم لتحقيق نتائج التسلسل الأمثل. التعاقب الإنتاجية العالية التي تستخدم sequenالوراثة عن طريق التخليق حساسون جدا لكثافة الكتلة على الخلية التدفق. تحميل تجمع المكتبات التي يتركز جدا سيؤدي في overclustering، مع انخفاض درجات الجودة، وانخفاض إخراج البيانات، وdemultiplexing دقيقة 32. تحميل تجمع المكتبات التي يتم تمييع جدا سيؤدي أيضا في إخراج البيانات منخفضة. بعناية قياس تجمع مكتبة قبل التسلسل وضمان أفضل النتائج.
يوفر 16S الريباسي التسلسل الجيني لتقييم شامل من البكتيريا الموجودة في عينة معينة، وخطوة أولى في غاية الأهمية في توليد الفرضية. وجود مجموعة غنية من البيانات الوصفية مزيد تمكن الباحث لاختبار الارتباط بين سيما الأنواع البكتيرية والعوامل البيولوجية الهامة. وعلاوة على ذلك، يمكن استخدام نفس 16S المعلومات للاستدلال على وظائف البكتيرية باستخدام أدوات مثل PICRUSt 33. والهدف النهائي هو استخدام 16S توصيف لتحديد الاتحادات الرواية التي يمكن أن تكونمزيد من الاختبارات والتحقق من صحتها في نظم نموذج، إضافة إلى فهمنا متزايد من تأثير microbiome البكتيريا على صحة الإنسان والمرض.
Disclosures
The authors have nothing to disclose.
Acknowledgements
ونود أن نشكر إليزابيث بيرن، ديفيد Gootenberg، وكريستينا Gosmann لردود الفعل الحاسم على البروتوكول. ميغان Baldridge، سكوت هاندلي، سيندي موناكو، وجايسون نورمان لتوجيه إعداد العينات والمظاهرات. ويندي غاريت، كورتيس Huttenhower، اقفز العذراء، وبروس ووكر للحصول على المشورة البروتوكول ومناقشات مثمرة. وجيسيكا Hoisington لوبيز لدعم التسلسل. وأيد هذا العمل من قبل مؤسسة بيل وميليندا وNIAID (1R01AI111918). تلقى DSK دعم إضافي من صندوق ويلكوم بوروز. وأيد [منا من قبل عدد جائزة T32GM007753 من NIGMS، وبول وديزي سوروس زمالة. المحتوى هو فقط من مسؤولية الكتاب ولا تمثل بالضرورة وجهة النظر الرسمية للNIGMS أو المعاهد الوطنية للصحة.
Materials
| Name | Company | Catalog Number | Comments |
| Equipment: | |||
| Mini-Beadbeater-16 | BioSpec | 607 | |
| PCR workstation | Any PCR hood can be used, e.g., the AirClean 600. | ||
| Thermocycler | Any thermal cycler with a heated lid can be used, e.g., MJ Research PTC-200. | ||
| Electrophoresis system | Any electrophoresis system can be used, e.g. the Thermo Scientific Owl EasyCast B1 Mini Gel Electrophoresis system. | ||
| Nanodrop | Thermo Scientific | 2000C | Any other DNA quantification method will be sufficient |
| Bioanalyzer | Agilent | 2100 | An alternative is the Agilent 2200 TapeStation Instrument. Not absolutely necessary but very helpful. |
| MiSeq or HiSeq | Illumina | ||
| Name | Company | Catalog Number | Comments |
| Materials: | |||
| Catch-All Sample Collection swab | Epibio | QEC89100 | Other swabs can be used but the Catch-All swab is recommended by the Human Microbiome Project. |
| ELIMINase | Fisher | 04-355-31 | |
| SteriFlip 50 ml filtration device (0.22 µm) | EMD Millipore | SCGP00525 | |
| 0.1 mm glass beads | BioSpec | 11079101 | |
| 2 ml screw-cap tubes | Sarstedt | 72.694.006 | For bead beating |
| UltraPure 5M NaCl | Life Technologies | 24740-011 | Molecular Biology Grade |
| 1 M Tris-HCl | Ambion (Invitrogen) | AM9856 | Molecular Biology Grade |
| 0.5 M EDTA | Ambion (Invitrogen) | AM9260G | Molecular Biology Grade |
| Sodium Dodecyl Sulfate, 20% Solution | Fisher | BP1311-200 | Molecular Biology Grade |
| UltraPure DNase/RNase-free distilled water | Ambion | 10977-015 | Molecular Biology Grade, for buffer preparation |
| 2-Propanol BioReagent, for molecular biology, ≥99.5% | Sigma | I9516-500ML | Molecular Biology Grade |
| Phenol:Chloroform:IAA, 25:24:1 | Invitrogen | AM9730 | Warning: Toxic |
| 3 M Sodium Acetate, pH 5.5 | Life Technologies | AM9740 | Molecular Biology Grade |
| Disposable sterile polystyrene forceps, PS | Cole Parmer | EW-06443-20 | |
| 1.5 ml, clear, PCR clean tubes | Eppendorf | 22364120 | |
| PCR grade water | MoBio | 17000-11 | For PCR |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| dNTP mix | Sigma | D7295-0.5mL | |
| 0.2 ml PCR 8-tube with attached clear flat caps, natural | USA Scientific | 1492-3900 | Any 8-tube strips that are DNase, RNase, DNA, and PCR inhibitor free will work |
| Agarose | BioExpress | E-3121-25 | |
| 50x TAE buffer | Lonza | 51216 | |
| DNA gel stain | Invitrogen | S33102 | |
| 6x DNA Loading Dye | Thermo (Fisher) | R0611 | |
| 50 bp GeneRuler Ladder | Thermo (Fisher) | SM0373 | |
| AllPrep DNA/RNA kit | Qiagen | 80284 | |
| UltraClean PCR Clean-up Kit | MoBio | 12500-100 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Thermo Fisher Scientific | P11496 | An alternative is Qubit Fluorometric Quantification (Life Technologies) |
| Name | Company | Catalog Number | Comments |
| Primers: | |||
| 515F (forward primer) 5'-AATGATACGGCGACCACCGAG ATCTACACTATGGTAATTGT GTGCCAGCMGCCGCGGTAA-3' | Order at 100 nmole; Purification: Standard Desalting. Resuspend at 100 µM. **Critical: primers must be resuspended with MoBio PCR Grade Water (see above) in a hood to avoid contamination.** | ||
| Reverse primers, see the Supplemental Code File and: ftp://ftp.metagenomics.anl.gov/data/misc/EMP/SupplementaryFile1_barcoded _primers_515F_806R.txt | IDT is recommended | If ordering large sets of primers, order as a 96-well plate at the 100 nmole scale. Resuspend at 100 μM. Full directions for primer ordering and resuspension at http://www.earthmicrobiome.org/files/2013/04/EMP_primer_ordering_and _resuspension.doc. **Critical: primers must be resuspended with MoBio PCR Grade Water (see above) in a hood to avoid contamination.** | |
| Read 1 Sequencing Primer 5'-TAT GGT AAT TGT GTG CCA GCM GCC GCG GTA A-3' | 25 nmole; Purification: Standard Desalting. Resuspend at 100 µM. | ||
| Read 2 Sequencing Primer 5'-AGT CAG TCA GCC GGA CTA CHV GGG TWT CTA AT-3' | 26 nmole; Purification: Standard Desalting. Resuspend at 100 µM. | ||
| Index Sequencing Primer 5'-ATT AGA WAC CCB DGT AGT CCG GCT GAC TGA CT-3' | 27 nmole; Purification: Standard Desalting. Resuspend at 100 µM. | ||
| PhiX Control v3 | Illumina | FC-110-3001 | Required if performing the sequencing in-house. If the sequencing will be performed by a third-party sequencing center, they will already have PhiX. |
References
- Huttenhower, C. Structure, function and diversity of the healthy human microbiome. Nature. 486, 207-214 (2012).
- O'Hanlon, D. E., Moench, T. R., Cone, R. A. Vaginal pH and microbicidal lactic acid when lactobacilli dominate the microbiota. PloS one. 8, e80074(2013).
- Aldunate, M. Vaginal concentrations of lactic acid potently inactivate HIV. The Journal of antimicrobial chemotherapy. 68, 2015-2025 (2013).
- Anahtar, M. N., et al. Cervicovaginal bacteria are a major modulator of host inflammatory responses in the female genital tract. Immunity. 42, 965-976 (2015).
- Reyes, A., Wu, M., McNulty, N. P., Rohwer, F. L., Gordon, J. I. Gnotobiotic mouse model of phage-bacterial host dynamics in the human gut. Proceedings of the National Academy of Sciences of the United States of America. 110, 20236-20241 (2013).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical biochemistry. 162, 156-159 (1987).
- Srinivasan, S., et al. Temporal variability of human vaginal bacteria and relationship with bacterial vaginosis. PloS one. 5, e10197(2010).
- Dols, J. A., et al. Microarray-based identification of clinically relevant vaginal bacteria in relation to bacterial vaginosis. American journal of obstetrics and gynecology. 204, 301-307 (2011).
- Segata, N., et al. Metagenomic microbial community profiling using unique clade-specific marker genes. Nature methods. 9, 811-814 (2012).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. The ISME journal. 6, 1621-1624 (2012).
- Caporaso, J. G., et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America. 108, Suppl 1 4516-4522 (2011).
- 16S rRNA Amplification Protocol. Earthmicrobiome Project. , Available from: www.earthmicrobiome.org (2015).
- Ravel, J., et al. Vaginal microbiome of reproductive-age women. Proceedings of the National Academy of Sciences of the United States of America. 108, Suppl 1 4680-4687 (2011).
- Srinivasan, S., et al. Bacterial communities in women with bacterial vaginosis: high resolution phylogenetic analyses reveal relationships of microbiota to clinical criteria. PloS one. 7, e37818(2012).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature methods. 7, 335-336 (2010).
- Werner, J. MacQIIME. , Available from: http://www.wernerlab.org/software/macqiime (2015).
- Schloss, P. D., et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and environmental microbiology. 75, 7537-7541 (2009).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature. 10, 996-998 (2013).
- NanoDrop 2000/2000c Spectrophotometer, V1.0 User Manual. Thermo Fisher Scientific. , Available at: http://www.thermoscientific.com/content/dam/tfs/ATG/CAD/CAD Documents/Product Manuals & Specifications/Molecular Spectroscopy/UV Visible Spectrophotometers/Spectrophotometer Systems/NanoDrop/NanoDrop-2000-User-Manual-EN.pdf (2009).
- AllPrep DNA/RNA Mini Kit. Qiagen. , Available from: https://http://www.qiagen.com/us/shop/sample-technologies/rna-sample-technologies/dna-rna-protein/allprep-dnarna-mini-kit/ - orderinginformation (2015).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of visualized experiments. , (2012).
- UltraClean PCR Clean-Up Kit Instruction Manual. MoBio. , Available from: http://www.mobio.com/images/custom/file/12500(1).pdf (2013).
- Quant-iT PicoGreen dsDNA Reagent. Invitrogen. , Available at: https://tools.thermofisher.com/content/sfs/manuals/mp07581.pdf (2008).
- Quality Scores for Next-Generation Sequencing. Illumina. , Available from: http://www.illumina.com/documents/products/technotes/technote_Q-Scores.pdf (2011).
- split_libraries_fastq.py. QIIME. , Available from: http://qiime.org/scripts/split_libraries_fastq.html (2015).
- DeSantis, T. Z., et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Applied and environmental microbiology. 72, 5069-5072 (2006).
- pick_open_reference_otus.py. QIIME. , Available from: http://qiime.org/scripts/pick_open_reference_otus.html (2015).
- summarize_taxa_otus.py. QIIME. , Available from: http://qiime.org/scripts/summarize_taxa.html (2015).
- Vazquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: a tool for visualizing high-throughput microbial community data. GigaScience. 2, 16(2013).
- Comparing categories. QIIME. , Available from: http://qiime.org/tutorials/category_comparison.html (2015).
- Eren, A. M., et al. Exploring the diversity of Gardnerella vaginalis in the genitourinary tract microbiota of monogamous couples through subtle nucleotide variation. PloS one. 6, e26732(2011).
- Diagnosing and preventing flow cell overclustering on the MiSeq system. Illumina. , Available from: http://support.illumina.com/content/dam/illumina-marketing/documents/products/other/miseq-overclustering-primer-770-2014-038.pdf (2015).
- Langille, M. G., et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature biotechnology. 31, 814-821 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved