Method Article
高效的核酸提取和16S rRNA基因序列的细菌群落特征
摘要
我们描述了从拭子用16S rRNA基因扩增子测序细菌群落特征提取核酸的高效,可靠和成本有效的方法。该方法允许对多种样品类型共同的处理方法,并容纳许多下游分析流程。
摘要
有微生物群落为人类健康与疾病的关键调制器的作用日益升值。高通量测序技术已经允许用于使用16S rRNA基因测序从各种来源的细菌群落的快速和有效的表征。虽然的16S rRNA序列分析现成的工具都有标准化的计算工作流程,提取DNA样本的处理仍然是变异的不同研究的持续来源。在这里,我们描述了从拭子中提取核酸的高效,可靠和成本有效的方法。我们也对划定的16S rRNA基因测序下游方法,包括新一代测序库,数据质量控制,及序列分析。工作流可以容纳多个样品类型,包括粪便和各种解剖位置和宿主物种的采集拭子。此外,回收的DNA和RNA可以分离并用于对于其它应用,包括全基因组测序或RNA-起。所描述的方法允许对多种样品类型共同的处理方法,可容纳的基因组,宏基因组和转录信息下游分析。
引言
人类的下生殖道,胃肠系统,呼吸道和皮肤是由维持组织稳态和配套主机1的健康至关重要复杂的细菌群落定植。例如,某些乳酸菌通过酸化阴道穹窿,产生抗菌效应和调制本地宿主免疫2-4创建病原体荒凉环境。对于细菌微生物的重要性日益升值也增加了许多临床表征环境细菌群落的兴趣。在这里,我们描述了一种方法,以确定从生殖器拭子细菌微生物的组合物。该协议可以很容易地修改从其他解剖位置和其他的宿主动物的粪便样品和棉签。
由于能够收集和存储从给定螺栓中的样本数量的固有限制ý参与者,该协议的目的是用一个适于苯酚-氯仿基于珠子的打浆方法-5,6-提取DNA,RNA,甚至潜在蛋白由单一拭子。与珠打浆和化学破坏用清洁剂细菌细胞壁的物理破坏的组合允许在不附加酶消化步骤革兰氏阳性,革兰氏阴性和抗酸细菌的迅速溶解。获得高品质的RNA,它建议使用保持在或低于4°即干拭子Ç采集后立即并运送至实验室(如果适用的话),并且存储长期在-80℃中。
要确定一个给定的样本中细菌的微生物,该程序利用16S rRNA基因扩增子测序,这是目前最具成本效益的手段,全面分配细菌分类,并执行相对定量。替代方法包括定量PCR针对性7,自定义microarrAYS 8和全基因组测序9。 16S rRNA基因包含九个高变区,并且没有关于最佳V区测序阴道微生物的研究一致。这里我们使用515F / 806R引物,并建立对管道通过Caporaso 等 10-12设计。 Caporaso的等 515F / 806R引物组能够数百个样品在单个测序运行的多路复用由于十万验证条形码引物和与Illumina的测序平台的兼容性的可用性。不同于人类微生物组项目的27F / 338R引物对13,515F / 806R也有效地放大Bifidobacteriaceae,从而准确地捕捉阴道加德纳菌 ,一些女性阴道微生物群落的重要成员。或者,338F / 806R引物对已被成功地用于阴道样品14和515F / 926R引物对具有RECE的焦磷酸测序下一代测序12 ntly变得可用。
最后,该协议提供了基本的说明来使用定量洞察微生物生态学(QIIME)软件包15进行16S扩增分析。成功地实施此处描述的QIIME命令产生包含每个样品细菌分类学丰度的表。许多额外的质量控制步骤,分类学分类方法,以及分析步骤可以并入分析,作为QIIME网站上详细描述(http://qiime.org/index.html)。如果分析将在苹果电脑上执行,该MacQIIME包16提供了易于安装QIIME及其依赖的。为16S rRNA基因序列分析替代软件包包括Mothur 17和18 UPARSE。
研究方案
本研究方案被批准,随后夸祖鲁 - 纳塔尔大学的生物医学研究伦理委员会(德班,南非)和马萨诸塞州总医院的机构审查委员会(2012P001812 / MGH,波士顿,MA)的指导方针。
1.宫颈阴道拭子总核酸的提取
注:套16个样本个或更少的执行核酸提取。如下面所写的协议假定样品在套12处理。如果在执行提取的多轮,连续编号的提取批次和记录每个样品的萃取批号以及其它样品信息(包括元数据,例如参与者的ID号,年龄,拭子采集,激素避孕类型,感染性传播疾病的检测结果等 ) 表1中的日期/时间。
- 试剂和通风柜制备
- 准备由200mM的氯化钠(NaCl),200毫摩尔Tris,并在100毫升的不含核酸酶的水的20mM乙二胺四乙酸(EDTA)的缓冲器。过滤-消毒使其通过0.22μm滤器的溶液。冷却的湿冰上10ml缓冲液的等分试样。
- 至pH 7.9,加入65微升每1ml苯酚的Tris碱缓冲液中,进行2分钟,振荡混合物,并允许两个阶段(1 25:24),调整苯酚的pH值::氯仿异戊醇(IAA)分离天然或离心以10,000 xg离心在RT 5分钟。
注意:如果吞咽,吸入或与皮肤和眼睛接触苯酚是有毒的。不要吸入烟雾。戴着防渗透手套,防护眼镜带侧护罩和白大褂。 - 过滤-消毒通过0.22微米的过滤器25毫升20%十二烷基硫酸钠(SDS)的。使每份5毫升消毒SDS的。
- 在-20℃冷却的异丙醇10ml的等分试样。
- 准备珠磨管每个sWAB到通过称出将0.3g玻璃珠成无菌的ml管是适合的珠打浆机进行处理。
- 通过用无菌吸收性拭子采样宫颈获得拭子。立即收集后,将拭子放入一个空的无菌离心管,储存在4℃下进行1至4小时运输到实验室中,并存储在-80℃下几个月。转移拭子(包含单独的管内),以进行处理,以湿冰上。
- 准备生物安全柜(BSC)。使用在BSC与一个"顶针"连接到建筑物排气,以确保适当除去的挥发性化学物质。
- 从引擎盖删除所有材料。
- 清洁用漂白剂罩,随后是去污剂,去除RNA酶,DNA酶和DNA从表面上的所有表面。清洁所有后续项目带进使用漂白后跟一个核酸去污剂,包括手套罩。使用新鲜的RNase /无DNase的试剂,如对ipette提示,只要有可能。
- 带的灭菌化学生物危害袋机罩的后方。含苯酚或氯仿所有的干垃圾应放入这个袋子妥善处置。
- 将无菌瓶插入罩,收集含有苯酚或氯仿废液。
- 酚氯仿提取。
- 在引擎盖,向每个珠击管中,加入500μl的缓冲液(来自步骤1.1.1),210微升20%的十二烷基硫酸钠,和500微升酚:氯仿:IAA(25:24:1,pH值7.9 )。
- 传送从传送小瓶拭子成使用一对新的无菌镊子的珠击管。彻底摩擦珠打浆管的内壁的拭子头部为至少30秒。重新盖完成后的样本。如果从多个拭子执行提取,改变每个样品之间手套。
- 冰浴样品至少10分钟。取下拭子珠握住手柄棉签用无菌镊子,同时用干净的P200尖按压棉签头靠在内部管壁跳动管。丢弃在干燥的化学废物袋中的棉签。注:"刮板"操作(按压拭子头)将从吸湿拭子释放液体,并增加核酸回收。
- 放置珠击管进入珠打浆机,并在4℃下均化2分钟。
- 离心机在6000 xg离心3分钟,4℃下的珠击管以沉淀碎片,并分离水相和苯酚相。
- 水相转移(〜500 - 600微升)到无菌1.5毫升管。添加酚等体积:氯仿:IAA。通过反演和短暂的震荡混匀。
- 离心反应管为16,000 xg离心5分钟,4℃。
- 将水相转移至新的无菌1.5毫升管中。是保守的,并且不从相间层或底层苯酚转移材料相。注意转印水相的体积。保存以供将来蛋白分离苯酚阶段。
- 添加0.8体积的异丙醇和0.1体积的3M乙酸钠(pH 5.5)。通过反演和简要涡旋调匀。
- 通过在-20℃下冷却该管,至少2小时(最多O / N)沉淀核酸。
- 异丙醇沉淀和乙醇洗
- 离心反应管30分钟,在大约16,000 xg离心和4℃。小心使用移液管除去上清液,留下沉淀完好无损。
- 加入500μl100%乙醇。打跑与温和的震荡或吹打颗粒而不触及沉淀。离心16,000 xg离心5分钟,4℃。
- 小心地弃上清乙醇。使用P10移液管以除去尽可能多的乙醇尽可能不干扰沉淀。
- 空气干燥在RT颗粒15分钟。
- 悬浮颗粒20微升的ULTRA纯0.1X的Tris-EDTA缓冲液。让样品在反复冰浴10分钟和吸管,以确保充分的再悬浮。如果沉淀不溶解,管转移到40℃的热块最多需要10分钟,以帮助溶解。
- 测量使用分光光度计19的核酸浓度。
- 如果需要的话,使用柱清理试剂盒,按照制造商的协议20由RNA分离的DNA。
- 存储核酸在-80℃或继续。
2. 16S rRNA基因V4高变区PCR扩增
注意:请在套12个样品或更少的PCR扩增,以减少污染和人为错误的风险。如果执行多轮扩增,串行编号扩增批次, 表1记录每个样品的扩增批号。
- Ø准备F中的试剂和PCR罩
- 添加PCR扩增设置信息表1,将作为在序列分析阶段的映射文件的基础。
- 从PCR罩卸下所有材料和用漂白粉其次是净化剂,去除RNA酶,DNA酶和DNA彻底清洁内表面。请务必将其放置在引擎盖前去污每试剂和设备部件(如移液管)的。穿与核酸去污剂在引擎盖工作之前清洁新鲜手套。
- 如果需要的话,稀释核酸模板50 - 100毫微克/微升使用DNA和无核酸酶的水。
- 的5倍高保真(HF)缓冲液,dNTPs,并在干净的PCR罩引物的解冻等分试样。轻轻涡旋离心解冻后所有的解决方案。为了最大限度地减少冻融和股票污染的风险,准备5×HF缓冲液,dNTPs和引物的等分试样。
- 一个地方对于微量离心管和PCR板式冷却器进油烟机清洁台式凉架上。
- 对于PCR反应,通过结合15.5微升超纯水,5微升5×HF缓冲液,0.5微升的dNTP,0.5μl的515F正向引物,0.75微升3%的DMSO和0.25微升聚合酶的为制备主混合物每个反应。装配的所有反应成分在冷却器并添加最后聚合酶。吹打调匀。添加两个额外的样品反应计数准备主结构的时候,考虑到移液错误。
- PCR反应设置:
注意:一式三份进行扩增,这意味着每个样品在三个独立的25微升反应扩增。运行与每对引物无模板水控制。很快,但工作认真细致,避免引入任何污染。- 标记与个别帽和地点的8孔条成的PCR冷却器。
- 吸取90μl反应混合液进入第一好。
- 加入2微升反向引物( 参考文件1)的。一定要仔细注意与表1中每个样品所用的反向引物的条形码。
- 拌匀,并转移23μl反应混合液至第四孔(无模板对照)。
- 加入2微升水至第四阱。
- 添加6微升适当样品到第一井。拌匀和25微升转移到第二井。变化的提示,并从第一井转移另一个25μl到第三阱。坚决帽每口井,确保不要触碰井的过程中内部或盖。
- 重复每个样品。
- 进行PCR扩增
- 传送条管,以热循环仪,并运行下列程序:在98℃在57℃在98℃30秒,然后用10秒的30个循环,30秒和72℃12秒,接着是10分钟保持在72℃,并最终保持一ŧ4℃。
- 执行干净实验台上以下步骤。快速旋转管从墙壁收集液体。从每个样品中结合一式三份的PCR反应,以75微升的总体积,到无菌的标记管。同时传送25微升每个无模板控制到一个单独的无菌管。请勿将来自不同样品扩增子呢。
- 通过凝胶电泳样品的成功的PCR扩增的验证。
- 制备1.5%琼脂糖凝胶(1.5克琼脂糖粉末在100毫升1×TAE缓冲液的)具有足够的孔,以容纳每个扩增子,控水,和阶梯21。
- 而凝胶硬化(约30分钟),对于电泳制备样品:加入1μl6×上样染料于一个新的,标记的管中。该管中,加入5微升扩增子,并通过吹打混合。
- 当凝胶已经设置,去掉梳,放置在电泳槽的凝胶和填充用1×TAE缓冲液的罐。
- 到了第一口井,加入5微升DNA阶梯。
- 负载5微升样品扩增子至另一个的好。负载5微升无模板的扩增子的一个单独的井。根据需要,对于每个样品继续。
- 当所有样品已经被加载,滑入到位水箱盖,并打开电源以120 V.让胶30运行 - 60分钟。
- 查看在紫外光下凝胶。
- 周围380基点注意到一个强带验证每一个样品成功扩增。如果有一个双带,再扩增具有不同的反向条形码(步骤2.3)的样品。如果没有带在所有的,即使用相同的反向条形码或新的反向条形码(步骤2.3)重新扩增样品。如果再放大不成功,PCR抑制剂可以存在于样品,在这种情况下,执行基于列的DNA的清理以去除PCR抑制剂英寸
注:成功扩增可能不是可能的,如果在细菌DNA的浓度或iginal样本不足(<5纳克/微升)。 - 通过注意在无模板对照没有的频带的验证缺乏试剂的污染。
- 周围380基点注意到一个强带验证每一个样品成功扩增。如果有一个双带,再扩增具有不同的反向条形码(步骤2.3)的样品。如果没有带在所有的,即使用相同的反向条形码或新的反向条形码(步骤2.3)重新扩增样品。如果再放大不成功,PCR抑制剂可以存在于样品,在这种情况下,执行基于列的DNA的清理以去除PCR抑制剂英寸
- 在-20℃保存剩余的70微升扩增的。丢弃剩余的20微升无模板对照的,假定它没有产生的频带。
3,库池和高通量测序
- 每个扩增子 - (5微升2)集成到一个无菌试管由等量的结合创建扩增子池。如果从样品乐队显得格外疲软,成交量相对两次添加到样品的其余部分。
- 从使用PCR清理试剂盒中的扩增子池中移除PCR引物,按照生产商的说明22。如果扩增子池体积为100微升进行清理与多个列。注意:每列都有一个100微升的能力。
- 在-20&#储存库176℃或进行下一步骤。
- 如果适用,结合无引物扩增子池,以创建最终库。确定使用分光光度计或荧光系统23库的DNA浓度。 1.8之间的260/280比率 - 2.0表明纯净的DNA。
- 稀释库20纳米。通过使用电泳器械周围400bp的可视化的单一带确认的库的质量。确认使用荧光系统23库的浓度。
- 在水中进行最后的1:10稀释到库稀释至2纳米。然后,存储库在20℃下去。
- 送的最终文库的等分试样与三个必需测序引物(读取1,读2,并编制索引;见材料/设备的表)上的Illumina序器来进行测序。如果少于300个样本已被多路复用测序,采用单端300 bp的运行,并有12bp的指数读Ø呐MiSeq,与下午5点终浓度图书馆和10%的变性PhiX秒杀项。见Caporaso 等 ISMEĴ,2012 10的详细测序说明补充材料。
4.序列分析
注:此处轮廓是使用QIIME 1.8.0软件包序列分析碱性管道。为简单起见,所提供的命令假定映射文件被称为的mapping.txt,12 bp的索引读取文件称为index.fastq和300bp的序列读文件被称为sequences.fastq。安装QIIME或MacQIIME 16和熟悉UNIX的基本知识,以执行这些命令。阅读完整的指南QIIME:
- 完成实验( 表1)的映射文件。包括尽可能多的元数据成为可能。注意哪些样本已提取或在同一批次放大,以确定是否有间歇的效果。 保存映射文件为文本文件, 例如,的mapping.txt。 validate_mapping_file.py -m的mapping.txt -o mapping_output:通过执行以下命令,验证映射文件的格式
- 查询的测序质量读取使用高通量序列数据质量检查程序,如FastQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)。 图5显示了每碱基序列的质量可从成功运行可以预期的。
注意:定序分配每个核苷酸碱基的PHRED质量得分,其对应于该基地已误称为概率。一个PHRED质量分数为10表示有10%的机会核苷酸被错误地分配ED,20表示1%的机会,30表示0.1%的几率,和40(最高分)表示0.01%的几率24。 - 使用映射文件作为密钥,解复用,质量过滤测序数据,并通过执行该命令25的结果保存到文件夹(在这种情况下,被称为"sl_out"):split_libraries_fastq.py --rev_comp_mapping_barcodes -i sequences.fastq -o sl_out / -b index.fastq -m的mapping.txt -q 29
注:Q标志表示最大不可接受PHRED质量得分, 例如,"-q 29"过滤出低于30分PHRED任何序列,保证了基地的呼叫99.9%的准确率。 - 使用Greengenes 16S操作分类单元(OTU)参考数据库26(http://qiime.org/home_static/dataFiles.html),进行开放式参考OTU采摘通过执行以下命令27:pick_open_reference_otus.py -i sl_out / seqs。 FNA -r 97_otus.fasta -o ucrss / -s 0.1
注:-s标志表示未能对准,将被包括在从头聚类参考数据库序列的分数。 "-s 0.1"包括在从头聚类失败序列的10%。使用-a标志并行的OTU采摘过程,从数天缩短处理时间,以小时,如果多内核可用。 - summarize_taxa.py -i ucress / otu_table_mc2.biom -o summarized_otuSpecies / -L 7:通过执行该命令28日在种的水平合并OTU单板创建一个用户友好的分类表丰
注:结果表可以在任何电子表格软件方便地查看。需要注意的是的16S rRNA测序不能可靠地提供物种水平分辨率。 - 通过计算几个字母多样性指标与QIIME脚本alpha_diversity.py确定每个样品中的生态多样性。然后,确定对使用QIIME脚本beta_diversity.py样品之间的差异。
- 可见ualize的数据, 例如 ,通过使用一个皇帝29主坐标图或热图。
- 执行映射文件类别, 如正式的统计比较,以QIIME的compare_catagories.py脚本30。
注:此命令使用内置的"validate_mapping_file.py"QIIME脚本,使一个新的文件夹,名为"mapping_output",包含一个.html文件指示映射文件中的错误,如果有的话。
结果
的协议,该协议允许使用16S rRNA基因测序拭子相对细菌丰度的测定的一般概述,在图中示出1 .The协议已为人类阴道拭子进行了优化,但可以很容易地适于大多数粘膜采样点和其他主机。 图2展示了高品质的DNA和RNA,可以使用珠打浆协议进行分离。 图3示出的12个样本的成功的PCR扩增,其中与样品每个扩增,得到正确的单个强带大小和各水控制没有取得的频带; 图4示出了测序前的最终文库池的量化; 图5示出了单端300bp的MiSeq运行后的典型序列的质量信息。
"SRC ="/文件/ ftp_upload / 53939 / 53939fig1.jpg"/>
图1.议定书示意图概述。首先,核酸是从拭子由珠打浆在含有苯酚,氯仿和异戊醇的缓冲溶液萃取。 16S rRNA基因的可变区4,然后从所得的核酸使用PCR扩增。然后从多达数百个样品的PCR扩增子被组合并测序上一次运行。所得序列被匹配到参考数据库,以确定相对的细菌丰度。整个协议可在大约三天进行。 请点击此处查看该图的放大版本。
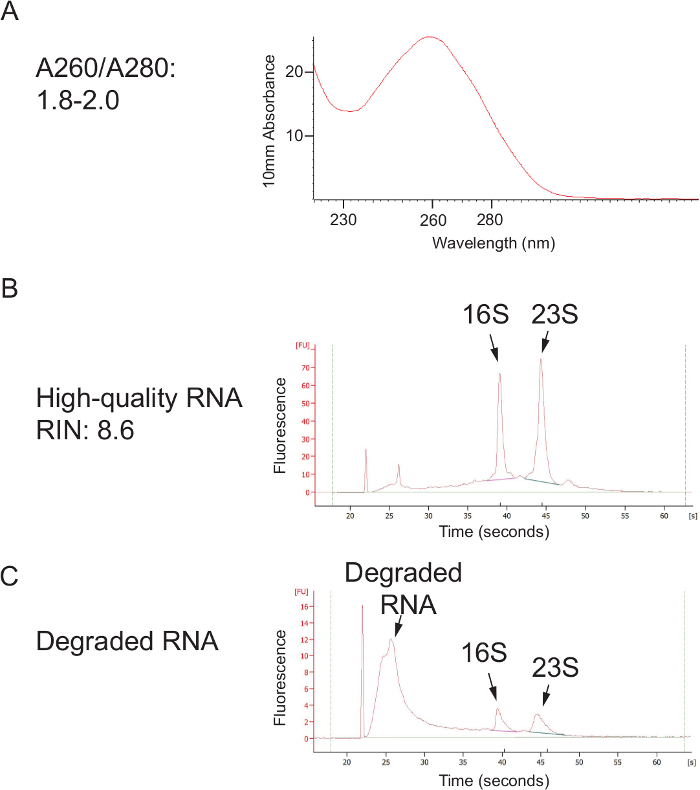
图2.高品质的核酸提取使用苯酚:氯仿珠磨中号ethod。 (A)中的DNA的质量,使用分光光度计来评估。 1.8和2.0之间的A260 / A280比值表示纯核酸不沾苯酚或蛋白质。(B)一列清理后,该协议可以产生高质量的RNA,通过强大的16S和23S rRNA的峰表示。如果采集后(运输和储存期间)的样品不保持冷可能发生(C)RNA降解或RNA酶,如果是加工时在座。 请点击此处查看该图的放大版本。
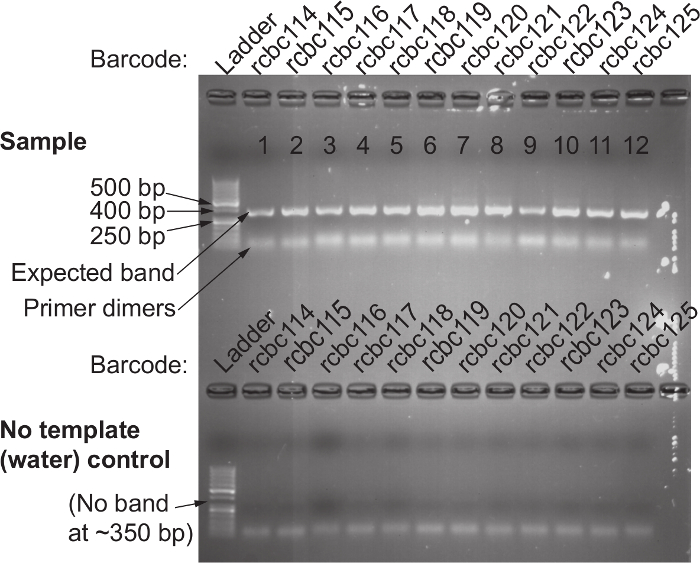
图3.成功的16S rRNA基因扩增的确认使用515F和条形码的806R引物组合。顶部 )凝胶电泳用于确认的单一条带的周围380个碱基对的存在下我ñ每一个与模板,扩增样本。没有的频带的指示不成功扩增;这通常是由于人为错误,并从该样品应重复PCR反应。 底 )否模板(水)控制运行在具有相同的引物对平行应该不会有本带。在水控制的带的存在表明被污染的试剂;丢弃可能被污染,试剂重新做既为引物对模板和水控制的PCR扩增。 请点击此处查看该图的放大版本。
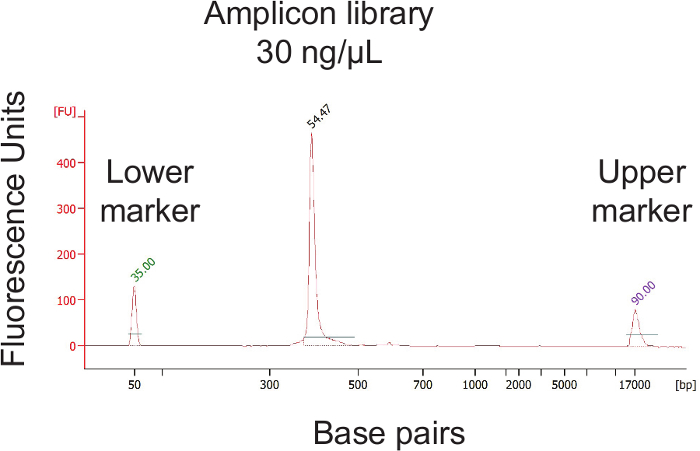
图4决赛图书馆池浓度定量和库的大小验证。汇集个别样品扩增后,日最终库池电子浓度必须确定。库池必须待进一步稀释达到2纳米浓度。 请点击此处查看该图的放大版本。
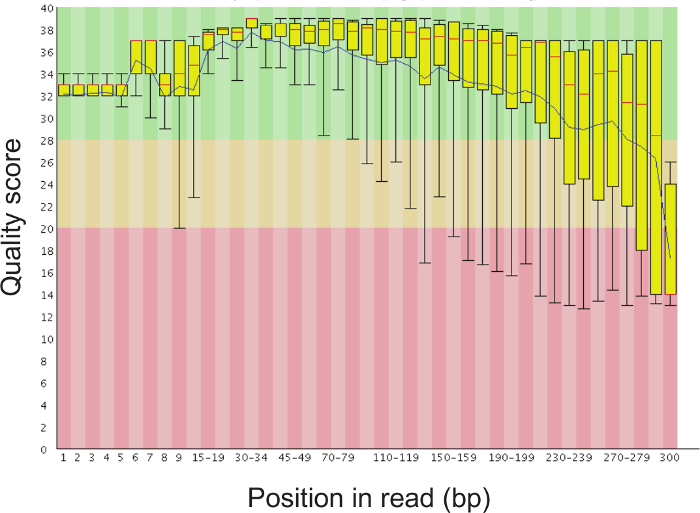
图5.代表律师在读每个位置的序列质量得分的情节,这是正常的序列质量后200个碱基对下降,但平均质量得分应该保持在高于30 请点击这里查看大图这一数字。
| #SampleID | 条形码 序 | LinkerPrimer 序列 | rcbcPrimer | SampleType | 萃取 批量 | 放大 盘子 | 描述 | |||
| http://qiime.org/_static/Examples/File_:#An例如映射文件可以在这里找到 格式/ 例_ Mapping_ FILE.TXT | ||||||||||
| AG2350 | TCCCTTGTCTCC | CCGGACTACHVGGGTWTCTAAT | rcbc000 | 颈椎 拖把 | 1 | 一个 | ||||
表1. 映射文件模板。克雷亚婷准确和全面的映射文件是成功执行该协议的关键。映射文件不仅需要执行QIIME,但它也使研究者能够保持样品条形码和元数据之间的联系,来分析任何系统偏差的数据( 例如,批次到批次变化),并确定元数据和细菌种群之间有趣的相互关系。一个最基本的映射文件提供,但鼓励用户添加元数据包含尽可能多的列。对于阴道拭子其他元数据的例子包括参与者的年龄,日期/拭子采集,激素避孕类型(如适用),性传播感染的检测结果等的时间
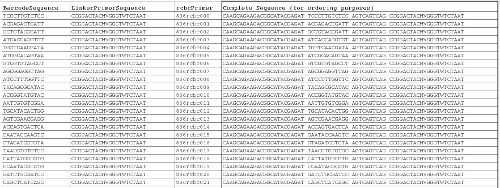
补充文件1.条形码的反向引物序列 10 的名单 。前三列可以被用来完成映射文件,并在最后一栏提供订购的目的,整个引物序列, 请点击此处下载此文件。
讨论
在这里,我们描述了一个人的阴道拭子中的鉴定和相对丰度细菌表征的协议。该协议可以很容易地适用于其他类型的样品,如粪便和其他身体部位拭子,和用于从各种各样的源收集样品。核酸的通过珠打浆在苯酚和氯仿的缓冲溶液中提取允许DNA和RNA,以通过临床研究收集珍贵样品工作时,这是特别重要的隔离。分离的细菌DNA是极好的细菌分类鉴定基因组和组装,而RNA同时采集提供了机会,通过RNA-seq中确定细菌的功能,主机和病毒的贡献。所描述的协议使用验证的一步法引物组已成功地部署在广泛范围的样品类型,包括人,犬的,和环境样品10 。数以千计的条形码引物的可用性使测序成本的样品和巨大的储蓄复用。完全成本(包括所有的试剂,单个测序运行,和引物但不包括设备)约为20 $每个样品时,200个样品进行复用。此外,还有当从同一样品网站的多个拭子穿过整个管道独立处理的非常高的再现性。总体而言,该协议是成本高效,灵活,可靠和可重复的。
这个协议的核酸提取部分是通过用酚和氯仿工作时所需要的安全预防措施,和自动化的管道高通量,96孔板格式的挑战限制。此外,所用的蓬勃珠磨机械剪板机裂解细菌DNA约6千碱基片段;如果较长的DNA片段所必需的下游应用,珠击S的持续时间HOULD被缩短。这个协议的细菌鉴定部的局限性是固有的,它依赖于16S rRNA基因测序的任何方法。 16S rRNA基因测序是理想的细菌鉴定属,甚至种的水平,但很少提供应变水平鉴定。而16S rRNA基因的V4可变区提供之中大多数细菌物种11健壮歧视,附加的计算方法如Oligotyping 31可能需要用来精确识别某些物种,如卷曲乳杆菌 。最后,关于特定样品内的精确细菌功能能力信息无法通过16S rRNA基因测序单独虽然这个协议使全基因组DNA和RNA,可以实现这一目的可使用的萃取而测定。
为保证这一协议成功的最关键步骤是采取非常谨慎,以防止造成污染DURING样品收集,核酸提取和PCR扩增。戴上干净的手套,并用消毒棉签,管,剪刀确保样本采集的时间不育。为了评估集合材料的污染,通过直接放置附加的未使用的拭子插入输送管在采样时收集阴性对照拭子。在实验室中,执行只含有消毒用品和只使用分子级,无DNA试剂消毒罩所有预扩增步骤。在核酸提取,防止交叉污染,通过使用新的无菌镊子和新鲜的手套每个样本,并保持除非使用全封闭的管。在并行处理中未使用的棉签确保样品收集和核酸提取两个不育;未使用拭子不应该产生异丙醇沉淀和乙醇洗涤后的颗粒。如果粒料确实出现,则执行16S rRNA基因的扩增,以确定的可能来源的污染( 例如, 链球菌属或葡萄球菌的存在将指示皮肤污染)。另外,进行PCR扩增以并联无模板的对照反应,以确保PCR试剂和反应都没有被污染。如果一个乐队出现在一个无模板对照,弃试剂和用新鲜试剂重复放大。采取这些预防措施,将确保目标细菌的测序成功。
PCR扩增步骤往往需要最故障排除。在套十二样品扩增提供效率和一致性之间的平衡。完全没有在给定的放大集所有样品的乐队表示系统故障, 如忘记加试剂或不正确编程热循环。没有来自几个样品的频带通常是由于人为错误,并且扩增应重新-R未与样品和反向引物的相同的配对。在持续不存在的频带的情况下,可将样品使用的反向引物具有不同的条形码再扩增。具有多个反向引重复扩增失败可能表示存在于样品中的抑制剂。在这种情况下,清洗该DNA与一列将经常除去抑制剂,而不显著改变相对细菌丰度。如果放大后多频段造成的,重新放大具有不同的反向引物的条形码样本。
除了防止环境污染和保证单个特定产物的扩增,成功测序制备文库池时依赖于治疗。我们的目标是等摩尔量的每种样品的扩增子的结合,以确保每个样品大约相同数目的测序读操作。如果之前扩增核酸浓度具有可比性,简单地增加每个SA等体积创建库池时mple的扩增子就足够了。然而,如果核酸浓度有很大的不同,并且在等体积增加,与低核酸浓度的样品将很差具有低数目的读取表示。在这种情况下,有可能从基于凝胶带的相对强度的低浓度样品添加量较高的扩增子。可替代地,能够更严格地从各扩增子除去引物,使用荧光的dsDNA定量试剂盒量化个体样品的扩增子浓度,并精确地结合等摩尔量各样品。
一旦生成一个均衡的扩增子池,它成为至关重要的仔细测量池的浓度。随后小心稀释和穗与PhiX增加了复杂性阅读是实现最佳的测序结果的关键。使用sequen高通量测序仪通过合成CING是在流动池的群密度非常敏感。加载一个过于集中将导致overclustering,质量得分较低,较低的数据输出,和不准确的解复用32的库池。加载库池太稀也会导致低的数据输出。仔细量化库池测序之前将保证最佳的效果。
16S rRNA基因测序提供了一个给定的样本中存在的细菌进行了全面评估,并在产生假设一个绝对关键的第一步。一组丰富的元数据进一步的存在使得研究者来测试特定细菌种类和重要的生物因子之间的关联。此外,相同的16S信息可以用来推断与工具如PICRUSt 33使用细菌的功能。最终的目标是使用16S表征,以确定新的关联性,可以是进一步的测试和模型系统验证,加入到我们的细菌微生物对人类健康和疾病的影响,越来越多的了解。
披露声明
The authors have nothing to disclose.
致谢
我们要感谢伊丽莎白·伯恩,大卫Gootenberg,和克里斯蒂娜Gosmann有关该协议的关键反馈;梅根·鲍德里奇,斯科特·汉德利,辛迪摩纳哥,和Jason诺曼样品制备指导和示范;温迪·加勒特,柯蒂斯Huttenhower,跳过处女,和布鲁斯·沃克协议的建议和富有成果的讨论;和杰西卡的Hoisington洛佩兹测序的支持。这项工作是由比尔和梅琳达·盖茨基金会和NIAID(1R01AI111918)的支持。 DSK从宝来惠康基金获得更多的支持。 MNA由奖号T32GM007753从NIGMS,而保罗和黛西索罗斯奖学金支持。内容完全是作者的责任,并不一定代表NIGMS或美国国立卫生研究院的官方意见。
材料
| Name | Company | Catalog Number | Comments |
| Equipment: | |||
| Mini-Beadbeater-16 | BioSpec | 607 | |
| PCR workstation | Any PCR hood can be used, e.g., the AirClean 600. | ||
| Thermocycler | Any thermal cycler with a heated lid can be used, e.g., MJ Research PTC-200. | ||
| Electrophoresis system | Any electrophoresis system can be used, e.g. the Thermo Scientific Owl EasyCast B1 Mini Gel Electrophoresis system. | ||
| Nanodrop | Thermo Scientific | 2000C | Any other DNA quantification method will be sufficient |
| Bioanalyzer | Agilent | 2100 | An alternative is the Agilent 2200 TapeStation Instrument. Not absolutely necessary but very helpful. |
| MiSeq or HiSeq | Illumina | ||
| Name | Company | Catalog Number | Comments |
| Materials: | |||
| Catch-All Sample Collection swab | Epibio | QEC89100 | Other swabs can be used but the Catch-All swab is recommended by the Human Microbiome Project. |
| ELIMINase | Fisher | 04-355-31 | |
| SteriFlip 50 ml filtration device (0.22 µm) | EMD Millipore | SCGP00525 | |
| 0.1 mm glass beads | BioSpec | 11079101 | |
| 2 ml screw-cap tubes | Sarstedt | 72.694.006 | For bead beating |
| UltraPure 5M NaCl | Life Technologies | 24740-011 | Molecular Biology Grade |
| 1 M Tris-HCl | Ambion (Invitrogen) | AM9856 | Molecular Biology Grade |
| 0.5 M EDTA | Ambion (Invitrogen) | AM9260G | Molecular Biology Grade |
| Sodium Dodecyl Sulfate, 20% Solution | Fisher | BP1311-200 | Molecular Biology Grade |
| UltraPure DNase/RNase-free distilled water | Ambion | 10977-015 | Molecular Biology Grade, for buffer preparation |
| 2-Propanol BioReagent, for molecular biology, ≥99.5% | Sigma | I9516-500ML | Molecular Biology Grade |
| Phenol:Chloroform:IAA, 25:24:1 | Invitrogen | AM9730 | Warning: Toxic |
| 3 M Sodium Acetate, pH 5.5 | Life Technologies | AM9740 | Molecular Biology Grade |
| Disposable sterile polystyrene forceps, PS | Cole Parmer | EW-06443-20 | |
| 1.5 ml, clear, PCR clean tubes | Eppendorf | 22364120 | |
| PCR grade water | MoBio | 17000-11 | For PCR |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| dNTP mix | Sigma | D7295-0.5mL | |
| 0.2 ml PCR 8-tube with attached clear flat caps, natural | USA Scientific | 1492-3900 | Any 8-tube strips that are DNase, RNase, DNA, and PCR inhibitor free will work |
| Agarose | BioExpress | E-3121-25 | |
| 50x TAE buffer | Lonza | 51216 | |
| DNA gel stain | Invitrogen | S33102 | |
| 6x DNA Loading Dye | Thermo (Fisher) | R0611 | |
| 50 bp GeneRuler Ladder | Thermo (Fisher) | SM0373 | |
| AllPrep DNA/RNA kit | Qiagen | 80284 | |
| UltraClean PCR Clean-up Kit | MoBio | 12500-100 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Thermo Fisher Scientific | P11496 | An alternative is Qubit Fluorometric Quantification (Life Technologies) |
| Name | Company | Catalog Number | Comments |
| Primers: | |||
| 515F (forward primer) 5'-AATGATACGGCGACCACCGAG ATCTACACTATGGTAATTGT GTGCCAGCMGCCGCGGTAA-3' | Order at 100 nmole; Purification: Standard Desalting. Resuspend at 100 µM. **Critical: primers must be resuspended with MoBio PCR Grade Water (see above) in a hood to avoid contamination.** | ||
| Reverse primers, see the Supplemental Code File and: ftp://ftp.metagenomics.anl.gov/data/misc/EMP/SupplementaryFile1_barcoded _primers_515F_806R.txt | IDT is recommended | If ordering large sets of primers, order as a 96-well plate at the 100 nmole scale. Resuspend at 100 μM. Full directions for primer ordering and resuspension at http://www.earthmicrobiome.org/files/2013/04/EMP_primer_ordering_and _resuspension.doc. **Critical: primers must be resuspended with MoBio PCR Grade Water (see above) in a hood to avoid contamination.** | |
| Read 1 Sequencing Primer 5'-TAT GGT AAT TGT GTG CCA GCM GCC GCG GTA A-3' | 25 nmole; Purification: Standard Desalting. Resuspend at 100 µM. | ||
| Read 2 Sequencing Primer 5'-AGT CAG TCA GCC GGA CTA CHV GGG TWT CTA AT-3' | 26 nmole; Purification: Standard Desalting. Resuspend at 100 µM. | ||
| Index Sequencing Primer 5'-ATT AGA WAC CCB DGT AGT CCG GCT GAC TGA CT-3' | 27 nmole; Purification: Standard Desalting. Resuspend at 100 µM. | ||
| PhiX Control v3 | Illumina | FC-110-3001 | Required if performing the sequencing in-house. If the sequencing will be performed by a third-party sequencing center, they will already have PhiX. |
参考文献
- Huttenhower, C. Structure, function and diversity of the healthy human microbiome. Nature. 486, 207-214 (2012).
- O'Hanlon, D. E., Moench, T. R., Cone, R. A. Vaginal pH and microbicidal lactic acid when lactobacilli dominate the microbiota. PloS one. 8, e80074(2013).
- Aldunate, M. Vaginal concentrations of lactic acid potently inactivate HIV. The Journal of antimicrobial chemotherapy. 68, 2015-2025 (2013).
- Anahtar, M. N., et al. Cervicovaginal bacteria are a major modulator of host inflammatory responses in the female genital tract. Immunity. 42, 965-976 (2015).
- Reyes, A., Wu, M., McNulty, N. P., Rohwer, F. L., Gordon, J. I. Gnotobiotic mouse model of phage-bacterial host dynamics in the human gut. Proceedings of the National Academy of Sciences of the United States of America. 110, 20236-20241 (2013).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical biochemistry. 162, 156-159 (1987).
- Srinivasan, S., et al. Temporal variability of human vaginal bacteria and relationship with bacterial vaginosis. PloS one. 5, e10197(2010).
- Dols, J. A., et al. Microarray-based identification of clinically relevant vaginal bacteria in relation to bacterial vaginosis. American journal of obstetrics and gynecology. 204, 301-307 (2011).
- Segata, N., et al. Metagenomic microbial community profiling using unique clade-specific marker genes. Nature methods. 9, 811-814 (2012).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. The ISME journal. 6, 1621-1624 (2012).
- Caporaso, J. G., et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America. 108, Suppl 1 4516-4522 (2011).
- 16S rRNA Amplification Protocol. Earthmicrobiome Project. , Available from: www.earthmicrobiome.org (2015).
- Ravel, J., et al. Vaginal microbiome of reproductive-age women. Proceedings of the National Academy of Sciences of the United States of America. 108, Suppl 1 4680-4687 (2011).
- Srinivasan, S., et al. Bacterial communities in women with bacterial vaginosis: high resolution phylogenetic analyses reveal relationships of microbiota to clinical criteria. PloS one. 7, e37818(2012).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature methods. 7, 335-336 (2010).
- Werner, J. MacQIIME. , Available from: http://www.wernerlab.org/software/macqiime (2015).
- Schloss, P. D., et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and environmental microbiology. 75, 7537-7541 (2009).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature. 10, 996-998 (2013).
- NanoDrop 2000/2000c Spectrophotometer, V1.0 User Manual. Thermo Fisher Scientific. , Available at: http://www.thermoscientific.com/content/dam/tfs/ATG/CAD/CAD Documents/Product Manuals & Specifications/Molecular Spectroscopy/UV Visible Spectrophotometers/Spectrophotometer Systems/NanoDrop/NanoDrop-2000-User-Manual-EN.pdf (2009).
- AllPrep DNA/RNA Mini Kit. Qiagen. , Available from: https://http://www.qiagen.com/us/shop/sample-technologies/rna-sample-technologies/dna-rna-protein/allprep-dnarna-mini-kit/ - orderinginformation (2015).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of visualized experiments. , (2012).
- UltraClean PCR Clean-Up Kit Instruction Manual. MoBio. , Available from: http://www.mobio.com/images/custom/file/12500(1).pdf (2013).
- Quant-iT PicoGreen dsDNA Reagent. Invitrogen. , Available at: https://tools.thermofisher.com/content/sfs/manuals/mp07581.pdf (2008).
- Quality Scores for Next-Generation Sequencing. Illumina. , Available from: http://www.illumina.com/documents/products/technotes/technote_Q-Scores.pdf (2011).
- split_libraries_fastq.py. QIIME. , Available from: http://qiime.org/scripts/split_libraries_fastq.html (2015).
- DeSantis, T. Z., et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Applied and environmental microbiology. 72, 5069-5072 (2006).
- pick_open_reference_otus.py. QIIME. , Available from: http://qiime.org/scripts/pick_open_reference_otus.html (2015).
- summarize_taxa_otus.py. QIIME. , Available from: http://qiime.org/scripts/summarize_taxa.html (2015).
- Vazquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: a tool for visualizing high-throughput microbial community data. GigaScience. 2, 16(2013).
- Comparing categories. QIIME. , Available from: http://qiime.org/tutorials/category_comparison.html (2015).
- Eren, A. M., et al. Exploring the diversity of Gardnerella vaginalis in the genitourinary tract microbiota of monogamous couples through subtle nucleotide variation. PloS one. 6, e26732(2011).
- Diagnosing and preventing flow cell overclustering on the MiSeq system. Illumina. , Available from: http://support.illumina.com/content/dam/illumina-marketing/documents/products/other/miseq-overclustering-primer-770-2014-038.pdf (2015).
- Langille, M. G., et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature biotechnology. 31, 814-821 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。