A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
رسم الخرائط الوراثية للاختلافات التسامح الحراري بين أنواع الخميرة Saccharomyces عن طريق تحليل الهيميزيغوستي على نطاق الجينوم المتبادل
In This Article
Summary
الهيميزيغوستي المتبادلة عن طريق التسلسل (RH-seq) هو طريقة جديدة قوية لرسم الأساس الوراثي للفرق سمة بين الأنواع. يتم إنشاء برك من hemizygotes عن طريق الطفرات transposon ويتم تتبع لياقتهم من خلال النمو التنافسي باستخدام تسلسل عالية في جميع أنحاء. تحليل البيانات الناتجة يحدد الجينات الكامنة وراء السمة.
Abstract
الهدف الرئيسي لعلم الوراثة الحديثة هو فهم كيف ولماذا تختلف الكائنات الحية في البرية في النمط الظاهري. وحتى الآن، تقدم الحقل إلى حد كبير على أساس قوة أساليب رسم خرائط الروابط والروابط، التي تتبع العلاقة بين متغيرات تسلسل الحمض النووي والنمط الظاهري عبر الذرية المؤتلفة من التزاوج بين أفراد من نوع ما. وهذه النهج، وإن كانت قوية، ليست ملائمة تماما للاختلافات بين الأنواع المعزولة من الناحية الإنجابية. هنا نقوم بوصف طريقة جديدة للتشريح على نطاق الجينوم من الاختلاف الصفات الطبيعية التي يمكن تطبيقها بسهولة على الأنواع غير المتوافقة. استراتيجيتنا، RH-seq، هو تنفيذ على نطاق الجينوم لاختبار hemizygote المتبادلة. نحن تسخيرها لتحديد الجينات المسؤولة عن ارتفاع في درجة الحرارة ضرب نمو الخميرة Saccharomyces سيريفيسياي نسبة إلى الأنواع الشقيقة S. paradoxus. يستخدم RH-seq الطفرات المتحولة لإنشاء مجموعة من hemizygotes المتبادلة، والتي يتم تعقبها بعد ذلك من خلال المنافسة في درجات الحرارة العالية عن طريق تسلسل الإنتاجية العالية. يوفر سير عمل RH-seq لدينا كما هو منصوص عليه هنا طريقة صارمة وغير متحيزة لتشريح الصفات القديمة والمعقدة في الخميرة الناشئة، مع التحذير من أن التسلسل العميق كثيفة الموارد ضروري لضمان التغطية الجينية لرسم الخرائط الجينية. ومع انخفاض تكاليف التسلسل، فإن هذا النهج يبشر بالخير الكبير للاستخدام في المستقبل عبر eukaryotes.
Introduction
منذ فجر الحقل، كان من الأهداف الرئيسية في علم الوراثة فهم الأساس الآلي للتباين عبر الأفراد البرية. وبينما نرسم خريطة للمواقع الكامنة وراء سمة من سمات الاهتمام، يمكن أن تكون الجينات الناشئة ذات فائدة فورية كأهداف للتشخيص والمخدرات، ويمكن أن تلقي الضوء على مبادئ التطور. معيار الصناعة نحو هذه الغاية هو اختبار العلاقة بين النمط الجيني والنمط الظاهري عبر السكان عن طريق الربط أو الارتباط1. وعلى الرغم من قوة هذه المناهج، فإن لها قيدًا رئيسيًا واحدًا - فهي تعتمد على لوحات كبيرة من الذرية المؤتلفة من الصلبان بين الأفراد المتداخلين. فهي لا فائدة لها في دراسة الأنواع التي لا يمكن أن تتزاوج لتشكيل ذرية في المقام الأول. وعلى هذا النحو، لم يكن لدى الحقل سوى قدرة ضئيلة على تشريح غير متحيز للاختلافات بين الأنواع المعزولة من الناحية الإنجابية2.
في هذا العمل نبلغ عن الأسس التقنية لطريقة جديدة، RH-seq3،للدراسات الاستقصائية على نطاق الجينوم من الأساس الوراثي للاختلاف سمة بين الأنواع. هذا النهج هو نسخة موازية على نطاق واسع من اختبار hemizygote المتبادلة4،5، الذي تم تصوره لأول مرة كوسيلة لتقييم الآثار الفينوتية للاختلافات الاليلية بين اثنين من الخلفيات المتميزة وراثيا في مكان معين(الشكل 1A). في هذا المخطط، يتم تزاوج الفردين المتباينة أولا لتشكيل هجين، نصف الجينوم الذي يأتي من كل من الآباء والأمهات. في هذه الخلفية، يتم إنشاء سلالات متعددة، تحتوي كل منها على نسخة تمت مقاطعتها أو حذفها من أليل كل أحد الوالدين من موضع المركز. هذه السلالات هي hemizygous لأنها لا تزال ثنائية في كل مكان في الجينوم إلا في موضع الاهتمام، حيث أنها تعتبر haploid، ويشار إليها على أنها متبادلة منذ كل يفتقر إلى أليل أحد الوالدين فقط، مع الأليل المتبقية المستمدة من والد آخر. ب يقارن ال [فينوموالأنواع] من هذا تبادليّة [هميزجوت] سلالات, واحدة يستطيع استنتاج تّماإذا [دنا] تسلسل [فرينت] في ال يتلاعب مكان يسهم إلى الصفة الفائدة, بما أنّ [فرينت] في الموقع يكون الفرق وحيد وراثيّة بين التبادليّة سلالات hemizygote. وبهذه الطريقة، من الممكن ربط الاختلافات الجينية بين الأنواع بالفرق الفينوتي بينها في إعداد تجريبي جيد التحكم فيه. وحتى الآن كانت تطبيقات هذا الاختبار في إطار المرشح - الجينات - أي الحالات التي تكون فيها الفرضية بالفعل في متناول اليد أن الاختلاف الطبيعي في موقع مرشح قد يؤثر على سمة.
في ما يلي، وضعنا البروتوكول لشاشة hemizygosity على نطاق الجينوم المتبادلة، وذلك باستخدام الخميرة كنظام نموذجي. لدينا طريقة يخلق تكملة الجينوم من المسوخ hemizygote، من خلال توليد قابلة للحياة، الهجينة F1 العقيمة بين الأنواع وإخضاعها لتحول المتحولين. نحن نجمع الهيميزيغوت، ونقيس الأنماط الظاهرية في الاختبارات المستندة إلى التسلسل، ونختبر الاختلافات في التردد بين استنساخ المسبح الذي يحمل أليلات الوالدين لجين معين. والنتيجة هي كتالوج من loci التي المتغيرات بين الأنواع تؤثر على سمة الفائدة. نقوم بتنفيذ سير عمل RH-seq لتوضيح الأساس الوراثي للاختلافات التسامح الحراري بين نوعين من الخميرة الناشئة، Saccharomyces cerevisiae و S. paradoxus، والتي تباينت ~ 5 مليون سنة مضت6.
Protocol
1. إعداد بلازميد يحتوي على piggyBac للتحول
- خط إلى مستعمرات واحدة سلالة القولونية إيواء بلازميد pJR487 على LB + carbenicillin لوحة أجار. حضانة لمدة ليلة واحدة عند 37 درجة مئوية أو حتى تظهر مستعمرات واحدة.
ملاحظة: يمكن العثور على وصف لكيفية استنساخ pJR487 بلازميد في عملنا السابق3. - تلقيح 1 لتر من LB + carbenicillin في 100 ميكروغرام / مل مع مستعمرة واحدة من القولونية E. تحتوي على pJR487 في قارورة زجاجية 2 L. تنمو بين عشية وضحاها في 37 درجة مئوية مع الهز في 200 دورة في الدقيقة حتى المشبعة (OD600 ≥ 1.0).
- تنقية الحمض النووي بلازميد من الثقافة باستخدام مجموعة إعداد بلازميد على نطاق واسع كما هو موضح في البروتوكول المنشور للشركة المصنعة (انظر جدول المواد للحصول على التفاصيل). Elute الحمض النووي بعد 10 دقيقة حضانة مع 5 مل من العازلة elution ارتفعت درجة حرارة إلى 37 درجة مئوية.
- قياس كمية ونوعية الحمض النووي بلازميد مع مقياس الطيف (انظر جدول المواد للحصول على التفاصيل).
- كرر الخطوات 1.2 – 1.4 حتى يتم عزل ما لا يقل عن 11 ملغ من الحمض النووي بلازميد بمعدلA 260:A280 من 1.8 على الأقل. قد يستغرق هذا عدد قليل من الإعدادية، اعتمادا على الكفاءة.
- خلط جميع البلازميد الإعدادية معا في أنبوب واحد وجلب حجم يصل إلى 20 مل مع العازلة elution أو الماء. قياس الكمية النهائية والجودة مرة أخرى مع مقياس الطيف الضوئي. يجب أن يكون تركيز البلازميد 538 نانوغرام/ميكرولتر على الأقل في هذا الحجم النهائي 20 مل. وإذا كان التركيز أعلى من 538 نانوغرام/ميكرولتر، فإن تخفيف البلازميد بعازل أو ماء إلى 538 نانوغرام/ميكرولتر. يمكن تخزين البلازميد عند 4 درجات مئوية حتى بضعة أسابيع حتى الاستخدام.
2- إنشاء مجموعة من الهيميزيغوتات المتبادلة غير المستهدفة على نطاق الجينوم
- إعداد خلايا الخميرة الهجينة للتحول
- خط من JR507 من سلالة الفريزر -80 درجة مئوية إلى مستعمرات واحدة على لوحة أجار YPD. حضانة في 26 درجة مئوية لمدة 2 أيام أو حتى تظهر المستعمرات.
ملاحظة: JR507 هو سلالة هجينة مصنوعة من خلال التزاوج خلية واحدة من الجراثيم الهابلويد من S. سيريفيسياي DBVPG1373 و S. paradoxus Z1 (باستخدام مجهر تشريح رباعي)3. - تلقيح 100 مل من السائل YPD في قارورة زجاجية 250 مل مع مستعمرة واحدة من JR507 ويهز في 28 درجة مئوية، 200 دورة في الدقيقة لمدة 24 ساعة، أو حتى يتم الوصول إلى مرحلة ثابتة.
- في اليوم التالي، وقياس الكثافة البصرية في 600 نانومتر (OD600)من الثقافة بين عشية وضحاها. خلق ثقافة جديدة من خلال تخفيف بعض من الثقافة بين عشية وضحاها مع السائل الطازج YPD في قارورة زجاجية جديدة 1 L إلى OD600 من 0.2 وحجم 500 مل.
ملاحظة: حساب مثال للتخفيف الخلفي إذا كانت الثقافة بين عشية وضحاها يحتوي على OD600 من 5.0 حيث C الكثافة البصرية و V هو حجم: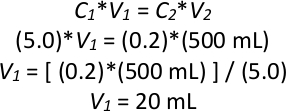
وهكذا، سيتم إضافة 20 مل من الثقافة المشبعة بين عشية وضحاها إلى 480 مل من YPD السائل لجعل ما مجموعه 500 مل من الثقافة في OD600 من 0.2. - كرر الخطوة 2.1.3 ثلاث مرات أخرى لجعل ما مجموعه أربع ثقافات 500 مل في OD600 من 0.2 في أربعة قوارير زجاجية 1 L، وذلك باستخدام نفس الثقافة بين عشية وضحاها لجميع الثقافات الأربع الجديدة. احتضان كل منهم في 28 درجة مئوية لمدة 6 ساعات (2-3 أجيال) تهتز في 200 دورة في الدقيقة.
- الجمع بين اثنين من الثقافات 500 مل لخلق ثقافة 1 L. الجمع بين الثقافات 500 مل المتبقية لخلق ثقافة أخرى 1 L. عند هذه النقطة، هناك ثقافتان L 1. كل من هذه الثقافات L 1 سوف تخضع للتحول مع pJR487 في الخطوات التالية.
- خط من JR507 من سلالة الفريزر -80 درجة مئوية إلى مستعمرات واحدة على لوحة أجار YPD. حضانة في 26 درجة مئوية لمدة 2 أيام أو حتى تظهر المستعمرات.
- تحويل pJR487 إلى خلايا الخميرة الهجينة
- تقسيم كل من الثقافات L 1 إلى عشرين aliquots 50 مل في 20 أنابيب مخروطية بلاستيكية لما مجموعه 40 أنابيب. وضع جانبا 20 أنابيب وتنفيذ الخطوات التالية على 20 أنابيب في وقت واحد.
- الطرد المركزي كل من أنابيب العشرين لمدة 3 دقائق في 1000 × ز لبيليه خلايا الخميرة. تجاهل الـ supernatant.
- إعادة تعليق كل بيليه مع 25 مل من H2O معقمة عن طريق الدوامة. الطرد المركزي لمدة 3 دقائق في 1000 × ز. تجاهل الـ supernatant.
- إعادة تعليق كل بيليه مع 5 مل من 1X TE، 0.1 M LiOAc العازلة عن طريق الدوامة. الطرد المركزي لمدة 3 دقائق في 1000 × ز. تجاهل الـ supernatant.
- كرر الخطوة 2.2.4. في حين أن الخلايا هي الطرد المركزي، وإعداد ما لا يقل عن 120 مل من حل 39.52٪ البولي ايثيلين غليكول، 0.12 M LiOAc و 1.2X Tris-EDTA العازلة (12 mM Tris-HCl و 1.2 mM EDTA). تخزين على الجليد.
- لإعداد الحمض النووي بلازميد للتحول، يغلي أولا 4 مل من الحمض النووي الحيوانات المنوية السلمون في 100 درجة مئوية لمدة 5 دقائق وتبريده على الفور على الجليد لمدة 5 دقائق. ثم، مزيج 20 مل من pJR487 (التي تم الحصول عليها في القسم 1) في تركيز 538 نانوغرام / ميكرولتر مع 4 مل من الحمض النووي الحيوانات المنوية السلمون المبردة لحجم إجمالي قدره 24 مل. يُحفظ على الجليد حتى يُستعمل.
- إضافة 600 ميكرولتر من الحمض النووي بلازميد مختلطة مع الحمض النووي الحيوانات المنوية السلمون على رأس كل بيليه الخلية. لا تقم بإعادة الإيقاف بعد.
- إضافة 3 مل من PEG-LiOAc-TE الحل المحرز في الخطوة 2.2.5 إلى كل بيليه. إعادة تعليق بيليه عن طريق الأنابيب صعودا وهبوطا ودوامة.
- حضانة كل أنبوب لمدة 10 دقائق في درجة حرارة الغرفة.
- صدمة الحرارة كل أنبوب لمدة 26 دقيقة في حمام مائي تعيين إلى 39 درجة مئوية.
ملاحظة: كل بضع دقائق، عكس كل أنبوب لمنع الخلايا من الاستقرار في الجزء السفلي من الأنبوب. - الطرد المركزي كل أنبوب لمدة 3 دقائق في 1000 × ز. تجاهل supernatant وإعادة تعليق كل بيليه في 10 مل من YPD عن طريق الدوامة. الجمع بين جميع الأنابيب العشرين في قارورة زجاجية جديدة. يجب أن يكون الحجم الإجمالي للخلايا ~ 200 مل.
- نقل 66.6 مل من الخلايا إلى قارورة زجاجية جديدة 1 لتر وجلب ما يصل إلى حجم 500 مل مع YPD السائل. كرر مرتين إضافيتين لاستخدام 200 مل بالكامل من الخلايا المحولة. قياس OD600 من كل ثقافة 500 مل جديدة (نتوقع OD600 من ~ 0.35-4).
- هز جميع القوارير الثلاثة عند درجة حرارة 28 درجة مئوية لمدة ساعتين للتعافي (<1 جيل) عند 200 دورة في الدقيقة.
- إضافة 0.5 مل من 300 ملغ / مل G418 إلى كل من القوارير الثلاثة، إلى تركيز نهائي من 300 ميكروغرام / مل G418 ووضعها مرة أخرى ليهز في 28 درجة مئوية، 200 دورة في الدقيقة.
ملاحظة: قبل هذه الخطوة، الخلايا المختلطة المحولة تم استرداد من التحويل. على الإضافة من [غ418], انتقيت وجود من ال [بلازميد] [بجر487] ل. أي الخلايا التي لم تأخذ بلازميد أثناء التحول سوف تبدأ في الموت. - كرر الخطوات 2.2.2 – 2.2.14 مع الأنابيب المخروطية الـ 20 المتبقية من الخلايا. عند هذه النقطة يجب أن يكون هناك ستة 1 L قوارير الزجاج، مع كل 500 مل من الخلايا مع G418 وأضاف.
- احتضان جميع قوارير الخلايا الستة عند 28 درجة مئوية، تهتز عند 200 دورة في الدقيقة، لمدة يومين تقريباً أو حتى يتم الوصول إلى OD600 من حوالي 2.3 في كل قارورة. الجمع بين جميع القوارير الستة معا لخلق ثقافة واحدة.
ملاحظة: على الرغم من أنه لن يتم استخدام كافة الخلايا في هذه الثقافة في خطوات المصب، كان الهدف من استخدام مثل هذه وحدات التخزين الكبيرة إنشاء أكبر عدد ممكن من أحداث التحويل الفريدة وتطبيع أي ة تحيز عبر تحويل واحد عن طريق تجميع كل منهم معا. - استخدام الثقافة التي تم إنشاؤها في 2.2.16 لتلقيح اثنين من القوارير 1 لتر جديدة مع 500 مل من YPD + G418 (300 ميكروغرام / مل) إلى OD600 من 0.2. سيكون هناك ثقافة بقايا التي يمكن تجاهلها.
- احتضان كل من 1 لتر قوارير في 28 درجة مئوية بين عشية وضحاها، مع هز في 200 دورة في الدقيقة، حتى يصل كل OD600 من ~ 2.2 (~ 3.5 أجيال). الجمع بين الثقافتين في ثقافة واحدة وقياس OD600 من الثقافة مجتمعة مرة أخرى.
ملاحظة: عند هذه النقطة، يجب أن تكون الثقافة تتألف بالكامل تقريبا من الخلايا التي تأوي pJR487 بلازميد. في جزء من السكان من الخلايا، فإن transposon PiggyBac قد تم نقلها من بلازميد في الجينوم من قبل transposase أعرب قبالة بلازميد. ومع ذلك، يمكن أن يؤدي التعبير المستمر عن الترانسبوساز إلى النقل أثناء عملية الاختيار، الأمر الذي من شأنه أن يحجب العلاقة بين النمط الجيني والنمط الظاهري. والهدف من الخطوات العديدة التالية هو إجراء اختيار مضاد ضد وجود بلازميد، لضمان عدم وجود مزيد من التعبير عن transposase. التجمع الناتج هو مزيج من الخلايا مع أو بدون transposon دمجها في الجينوم، ولكن يتم الكشف فقط الخلايا التي تحتوي على transposon أثناء خطوات رسم الخرائط اللاحقة. الوقت في التحول الذي يتم خلاله التعبير عن transposase، قبل فقدان ترميز بلازميد، قد يحكم فرصة أن استنساخ معين بعد الطفرات يأوي أكثر من إدراج transposon واحد. ويمكن تقدير تواتر هذه، التي تظهر على أنها طفرات "ثانوية" في تحليل أي جين واحد في وقت واحد، عن طريق تجميع عدد محدد من المستعمرات بعد الطفرات، ثم الجمع بين الحمض النووي وتسلسل تأكيد عدد الإدراج المستقل مواقع في حمام السباحة. - الطرد المركزي 25 مل من هذه الثقافة لمدة 3 دقائق في 1000 × ز. حساب عدد إجمالي وحدات OD600 من الخلايا الموجودة في 25 مل (انظر حساب المثال أدناه). تجاهل supernatant وإعادة تعليق في ما يكفي H2O لإنشاء تعليق خلية من 1.85 OD600/mL عن طريق الدوامة.
ملاحظة: حساب مثال لإعادة تعليق الخلايا في الماء إذا كان OD600 من الثقافة مجتمعة 2.2: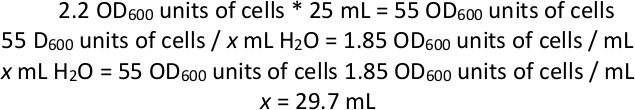
لذلك، بعد الغزل 25 مل من ثقافة الخلية وتجاهل supernatant، إضافة ما يكفي H2O إلى الخلايا لجعل الحجم الإجمالي للخلايا والماء تصل إلى ~ 29.7 مل (منذ بيليه الخلية سيكون لها أيضا حجم، إضافة أقل من 29.7 مل من H2O). - باستخدام الخرز الزجاجي، لوحة 1 مل من الخلايا المعاد تعليقها في الماء على كل من 12 لوحات أجار الاصطناعية مربع كبير كاملة مع 5-FOA. حضانة كل لوحة في 28 درجة مئوية لمدة 1-2 أيام أو حتى تشكل العشب على لوحة.
- باستخدام صرير معقم صغير، كشط الخلايا من كل من 6 لوحات وإلى أنبوب مع 35 مل من الماء المعقم. كرر مع لوحات 6 أخرى لما مجموعه اثنين من أنابيب الخلايا والماء. الجمع بين جميع تعليق الخلية في أنبوب واحد. قياس OD600 من هذا التعليق، وذلك باستخدام الماء كفراغ. جلب تركيز OD600/ مل من الخلايا إلى 44.4 OD600 وحدة / مل مع الماء. في تجربتنا، كفاءة تبديل (نسبة الخلايا KAN + التي هي URA-) في المتوسط 50٪.
- تحديد عدد مخزونات الفريزر -80 درجة مئوية من الخلايا لتخزينها. يمكن استخدام كل aliquot في المستقبل لتجربة واحدة.
ملاحظة: نظراً لكيفية استهلاك الوقت إنشاء التجمع تخزين قارورة متعددة في حالة إساءة استخدام عرضي أو لإجراء تجارب النسخ المتماثل. 20-30 الأسهم هي عدد معقول. - وسوف تحتوي على كل مخزون الفريزر 40 OD600 وحدة من الخلايا في 1 مل من 10٪ DMSO. إضافة 900 درجة مئوية من الخلايا إلى 100 درجة مئوية من DMSO. كرر للعدد الإجمالي لمخزون الفريزر الذي تم إنشاؤه. تخزين كل في -80 درجة مئوية للاستخدام في المستقبل.
3- اختيار الهيميزيغوتات المتبادلة في شكل مجمع
- قم بالذوبان من الفريزر -80 درجة مئوية على قطعة واحدة من الهيميزيغوتات المتبادلة المجمعة من القسم 2 في درجة حرارة الغرفة.
ملاحظة: لا تدع aliquot الجلوس لفترة طويلة في درجة حرارة الغرفة بمجرد ذوبان، واستخدامها على الفور. - استخدام aliquot 1 مل كامل لتلقيح 150 مل من السائل YPD في قارورة زجاجية 250 مل. قياس OD600 من هذه الثقافة، ومن ثم احتضان في 28 درجة مئوية، ويهز في 200 دورة في الدقيقة، لمدة 7 ساعات تقريبا، أو حتى الثقافة قد مرت 2-3 تضاعف السكان. في هذه المرحلة، والثقافة على استعداد لاستخدامها لتلقيح الثقافات التي تمر الاختيار.
ملاحظة: حساب المثال: إذا كان OD600 من قارورة الأصلي يقيس 0.25، احتضان الثقافة حتى تصل إلىOD 600 من 1.0 على الأقل. إذا كانت هناك أي نقاط عينة مرغوب فيها في "الوقت صفر" (T-0)، كوسيلة للتحقيق في السكان hemizygote قبل الاختيار، يمكن أن تؤخذ الكريات الخلية الآن عن طريق الطرد المركزي 5-10 مل من الثقافة لكل بيليه في 1000 × ز لمدة 3 دقائق، والتخلص من supernatant و تجميد عند -80 درجة مئوية. - استخدم بركة hemizygote المزروعة لتلقيح الثقافات للاختيار في مخطط تكرار مناسب، في كل من درجة الحرارة العالية (39 درجة مئوية) ودرجة الحرارة المتساهلة (28 درجة مئوية). على الأقل، إنشاء ثلاث ثقافات اختيار تكرار البيولوجية في كل درجة حرارة، لما مجموعه ست ثقافات الاختيار.
- إنشاء كل ثقافة الاختيار مع مجموع 500 مل في قارورة زجاجية 2 L مع YPD السائل وتلقيح إلى OD600 من 0.02. هز كل ثقافة اختيار عند 100 دورة في الدقيقة عند 28 درجة مئوية أو 39 درجة مئوية حتى تضاعف عدد السكان 6-7 (المقابلة لOD600 من ~ 1.28-2.56). في محاولة لمطابقة أقرب ما يمكن ODالنهائي 600 من جميع الثقافات الاختيار.
ملاحظة: سوف تنمو ثقافات التحديد عند 28 درجة مئوية بشكل أسرع من ثقافات التحديد عند 39 درجة مئوية. وبالتالي، فإن ثقافات الاختيار في 39 درجة مئوية سوف تنفق فترة أطول من الوقت في الحاضنة. المضي قدما في الخطوات التالية مع كل قارورة كما يصبح جاهزا، بغض النظر عن العدد الإجمالي للساعات التي يقضيها في الحاضنة. في تجربتنا، استغرق الثقافات في 28 درجة مئوية أو 39 درجة مئوية ~ 12 أو ~ 18 ساعة، على التوالي، للوصول إلى OD من ~ 2.0. اختيارات طويلة يمكن أن يكون لها ميزة تضخيم آثار اللياقة البدنية الصغيرة، ولكن أيضا السماح دي نوفو الطفرات الخلفية أن تنشأ، والتي من شأنها أن تدخل الضوضاء في التوزيع النهائي للياقة البدنية عبر المتحولين عبر المتحولين في أي جين واحد / آليل. وعلى هذا النحو، من المهم الحد من وقت التحديد في تجربة RH-seq.
- إنشاء كل ثقافة الاختيار مع مجموع 500 مل في قارورة زجاجية 2 L مع YPD السائل وتلقيح إلى OD600 من 0.02. هز كل ثقافة اختيار عند 100 دورة في الدقيقة عند 28 درجة مئوية أو 39 درجة مئوية حتى تضاعف عدد السكان 6-7 (المقابلة لOD600 من ~ 1.28-2.56). في محاولة لمطابقة أقرب ما يمكن ODالنهائي 600 من جميع الثقافات الاختيار.
- حصاد الكريات الخلية من كل ثقافة الاختيار. حساب الحجم المطلوب للحصول على 7 وحدات من الخلايا والطرد المركزي بـ 7 وحدة من وحدات OD600 في 1000 × ز لمدة 3 دقائق على الأقل أربع كريات من هذا الحجم من كل ثقافة اختيار كنسخ تقنية لإعداد المكتبة وتسلسلها (انظر القسمين 4 و5 ، أدناه). تجاهل supernatant وتخزينها في -80 درجة مئوية.
ملاحظة: مثال إذا قارورة تحديد يحتوي على OD600 النهائي من 2.0: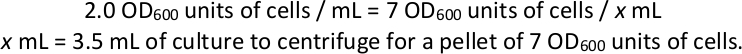
4. Tn-seq بناء المكتبة وتسلسل Illumina لتحديد وفرة من الهميزيغوتات المتحولة
- إذابة على الجليد كل بيليه الخلية من القسم 3 التي سيتم تسلسلها.
- عزل مجموع الحمض النووي الجينومي (gDNA) من كل بيليه الخلية باستخدام مجموعة تنقية gDNA الخميرة بناء على تعليمات الشركة المصنعة. إعادة تعليق الحمض النووي في 50 درجة مئوية من العازلة elution يسخن إلى 65 درجة مئوية.
- قياس كمية gDNA من كل بيليه باستخدام مقياس الفلورة. الحد الأدنى من الكمية الإجمالية من gDNA المطلوبة لكل بيليه الخلية لإنشاء مكتبة تسلسل الجيل التالي (NGS) لTn-seq باستخدام الإجراء التالي هو 1 ميكروغرام.
ملاحظة: يمكن استخدام أقل من 1 ميكروغرام من gDNA لإنشاء مكتبة، ولكن الكمية النهائية للمكتبة ونوعيتها ستعاني. - اتبع بروتوكول ًا ثابتًا لإنشاء مكتبات Tn-seq7. لاحظ المعلومات التالية ذات الصلة الفريدة لهذا البروتوكول:
- بعد القص gDNA، إصلاح نهاية والربط محول، تضخيم gDNA التي تحتوي على transposon عن طريق PCR. لهذا PCR، استخدم التمهيدي الأمامي والعكسي التالي، والتي هي محددة لمحولات transposon PiggyBac وNGS، على التوالي:
إلى الأمام (N – النيوكليوتيد العشوائي)
5' ATGATACGGACCACCTACACTACTACTTTTTTACTACGACG
CTCTTCCGATNNNNCAAATATATCAGTGTGTCAAT 3'
عكس (تمتد من Ns يمثل فهرس 6-bp فريدة من نوعها المستخدمة لتعدد الإرسال. انظر أدناه للحصول على مزيد من المعلومات عن المؤشرات)
5' CAAGCAGAGACGGCATACGAGATNNNNNNGTGGTGTGTGT
ACGTGTGCTCTTCCGATCT 3' - استخدم خطوات التنظيف المضمنة مع الخرز الانتقائي الحجم لتقليل نسبة الشظايا المستنسخة في المكتبة النهائية التي تكون قصيرة جداً بحيث لا يمكن تضمين تسلسل جيني قابل للتعيين.
ملاحظة: بعد اتباع الحد الأدنى من متطلبات النسخ المتماثل حتى الآن لثقافات التحديد، سيكون هناك 24 عينة gDNA فردية للتسلسل. وبالنظر إلى التكلفة الحالية للتسلسل، فمن غير المحتمل أن يتم تشغيل كل عينة بمفردها. لدمج العينات على نفس المسار، قم بإنشاء العديد من التمهيديات العكسية، ولكل منها فهرس زوج فريد من نوعه مكون من 6 قاعدة. يمكن دمج العينات ذات الفهارس المختلفة في نفس مسار التسلسل وفصلها حسابيًا بعد ذلك.
- بعد القص gDNA، إصلاح نهاية والربط محول، تضخيم gDNA التي تحتوي على transposon عن طريق PCR. لهذا PCR، استخدم التمهيدي الأمامي والعكسي التالي، والتي هي محددة لمحولات transposon PiggyBac وNGS، على التوالي:
- تسلسل نهاية واحدة 150 bp يقرأ من كل مكتبة باستخدام تقنيات NGS عبر ثمانية ممرات.
ملاحظة: يعتمد مقدار قراءات التسلسل المطلوبة بشكل كبير على جودة المكتبات التي تم إعدادها في الخطوة السابقة (أي نسبة الحمض النووي في المكتبة التي تحتوي بالفعل على الحمض النووي عبر الترانسبوسون، الذي يمثل الحمض النووي القادم من hemizygotes المتبادلة). وهناك عاملان رئيسيان يسهمان في ذلك. أولاً، بما أن الخلايا التي لا تحتوي على transposon متكاملة لا يتم تحديدها مقابل أثناء إنشاء تجمع، فإن كل ثقافة ستكون مزيجاً من الخلايا مع وبدون transposon. ثانيا، حتى داخل الجينوم من الهيميزيغوتات التي تحتوي على transposon المتبادلة، فإن معظم الجينوم لا transposon تحتوي على تسلسل، وهذا gDNA سيكون لا مفر منه جزءا من إعداد المكتبة. والهدف من تضخيم PCR النهائي للحمض النووي الذي يحتوي على transposon هو زيادة نسبة الحمض النووي المحتوي على transposon إلى هذين المصدرين من gDNA الخلفية. وكلما زاد كفاءة هذا التضخيم، ستكون نسبة القراءة الأعلى قادرة على استخدامها في التحليل المصب. كلما كانت جودة المكتبات أقل، كلما كان من الضروري إجراء المزيد من التسلسل، حيث أن نسبة متزايدة من القراءات لن تحتوي على الحمض النووي ترانسبوسون ولن تكون مفيدة. وبالنظر إلى القيود المذكورة أعلاه، كانت ثمانية مسارات للتسلسل قادرة على تتبع وفرة الهيميزيغوت المتبادلة إلى درجة معقولة. ومن شأن زيادة التسلسل أن تسمح بإجراء تحليل أعمق.
5. رسم خرائط لمواقع عمليات الإدراج عبر الtransposon وتحليل RH-seq
ملاحظة: تم إنجاز تحليل البيانات التالية مع البرامج النصية Python المخصصة (وجدت على الإنترنت في https://github.com/weiss19/rh-seq) ولكن يمكن إعادة تحرير باستخدام لغات البرمجة النصية الأخرى. ويرد أدناه موجز للخطوات الرئيسية في هذه العملية. تنفيذ الخطوات التالية على كل ملف قراءة النسخ المتماثل الفردية إلا إذا تمت الإشارة إلى ضمها.
- تقليم تسلسل محول خارج من القراءة وفصل كل قراءة النسخ المتماثل وفقا للفهرس.
- البحث عن قراءات تحتوي على تقاطعات الجينوم عبر بوسون. لتحقيق ذلك، البحث داخل كل قراءة لآخر 20 أزواج قاعدة من transposon، CAGACTATTTCTAGGGTtaA. تجاهل كافة القراءات التي لا تحتوي على هذا التسلسل.
ملاحظة: في تجربتنا، نسبة قراءة تعيين إلى نهاية transposon 83-95%. - تقليم ما تبقى، transposon التي تحتوي على يقرأ لاحتواء فقط تسلسل المصب من نهاية 3 'من transposon. عن طريق رسم خريطة هذا التسلسل إلى جينوم الخميرة، وتحديد السياق الجيني للإدراج transposon لكل قراءة (الخطوة 5.4 أدناه).
- استخدم BLAT أو أداة رسم خرائط مكافئة لتعيين تسلسل المصب للtransposon إلى S. cerevisiae DBVPG1373 x S. paradoxus Z1 المجين الهجين (اسم البرنامج النصي: map_and_pool_BLAT.py).
- تجاهل أي قراءات التي يوجد أقل من 50 أزواج أساسية من تسلسل قابل للاستخدام المصب من نهاية 3 ' من transposon. تسلسل قصيرة من الصعب تعيين بشكل فريد.
- في حالة استخدام BLAT، استخدم المعلمات التالية: الهوية = 95، حجم التجانب = 12.
- إنشاء جينوم هجين أساسي لاستخدامه في رسم الخرائط عن طريق ربط أحدث إصدارات الجينوم المرجعي من S. سيريفيسياي S288c و S. paradoxus CBS432.
ملاحظة: يمكن العثور على ملف تعليق توضيحي أساسي يصف الحدود الجينية للجينات الفردية عبر الجينوم الهجين في مستودع Github المذكور أعلاه (اسم الملف: YS2+CBS432+plasmid_clean). فقط استخدام يقرأ أي خريطة إلى موقع واحد في الجينوم الهجين (أي هي فريدة من نوعها إما S. سيريفيسياي أو S. مفارقة). ومن المتوقع حدوث تواتر موحد لأحداث الإدراج عبر الجينوم؛ يتم الإبلاغ عن توزيع مواقع الإدراج عبر الجينوم في مكان آخر3.
- حساب العدد الإجمالي للقراءة تعيين إلى كل موقع إدراج transposon فريدة من نوعها، والتي نستنتج كل نشأت من خلايا استنساخ متحولة إدراج transposon واحد. يُشار إلى مجموع كافة هذه القيم من مكتبة واحدة باسم العدد الإجمالي للقراءات المعينة لتلك المكتبة.
- في الحالات حيث يوجد تعيين عمليات إدراج متعددة ضمن 3 أزواج أساسية من بعضها البعض، ضمكل منهم إلى نقطة إدراج واحدة، تعيين كافة القراءات إلى موقع واحد مع أعلى عدد للقراءة. هذه القيمة، نإدراج، يمثل وفرة من استنساخ الإدراج في بيليه الخلية التي تم تسلسل gDNA. عند هذه النقطة، سيكون هناك قوائم من نإدراج، كل وفرة من إدراج transposon تعيين فريدة من نوعها، قائمة واحدة لكل خلية بيليه تسلسل.
ملاحظة: إدراج transposon PiggyBac في تسلسل TTAA في الجينوم، تسلسل زوج قاعدة 4. وهكذا، فإننا نستنتج أن الإدراج رسم الخرائط داخل 3 أزواج قاعدة من بعضها البعض يجب أن تكون نشأت من نفس موقع TTAA. - بما أنه سيكون هناك عدد مختلف قليلاً من إجمالي القراءات القادمة من كل مكتبة متسلسلة، قم بتطبيع قيمإدراج n عبر كافة الملفات إذا كان سيتم مقارنتها. القيام بذلك عن طريق جدولة العدد الإجمالي للقراءات المعينة من كل مكتبة فردية، نبيليه،واتخاذ متوسط جميع نبيليه عبر جميع المكتبات، بيليه>. ضرب كل نإدراج في بيانات مكتبة فردية بنسبة بيليه> / نبيليه لحسابإدراج،وفرة تطبيع استنساخ إدراج transposon معينة.
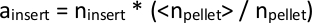
بدلاً من ذلك، يمكن تقدير حجم المكتبة باستخدام الأدوات المتوفرة مثل DESeq28 (اسم البرنامج النصي: total_reads_and_normalize.py). - جدولة مجموعة كافة عمليات الإدراج التي تم تعيينها عبر كافة المكتبات. بالنسبة للإدراجات الموجودة في بعض المكتبات ولكن ليس في مكتبات أخرى، قم بتعيينإدراج = 1 لحسابات المتلقين للمعلومات.
- تصفية القراءة للعثور على تلك الإدراجات التي تقع ضمن الجينات وفقا لملف التعليق التوضيحي (اسم البرنامج النصي: remove_NC_and_plasmid_inserts.py).
- لكل إدراج فريد، حساب متوسط الوفرة عبر النسخ المتماثل التقنية لكل اختيار (كل ثقافة إما في28 درجة مئوية أو 39 درجة مئوية)، <إدراج >التقنية (اسم البرنامج النصي: combine_tech_reps_V2.py).
- لكل إدراج فريد، حساب متوسط الوفرة عبر عمليات التكرارالبيولوجية لكل درجة حرارة، <إدراج >إجمالي، عن طريق أخذ متوسط جميع <إدراج >التقنية في كل درجة حرارة. في نفس الوقت، حساب معامل التباين لكل إدراج، إدراج السيرة الذاتية،إجمالي عبر <إدراج >التقنية (اسم البرنامج النصي: combine_bio_reps.py).
ملاحظة: في هذه المرحلة، لكل درجة حرارة، 28 درجة مئوية و 39 درجة مئوية، هناك قائمة من عمليات الإدراج transposon فريدة من نوعها، ومتوسط وفرة ومعامل الاختلاف بين النسخ المتماثل البيولوجيلكل. يتم الإبلاغ عن هذه البيانات لتجربتنا في مكان آخر3. - قم بتصفية قائمة كافة عمليات الإدراج لأولئك الذين لديهم، إما 28 درجةمئوية أو 39 درجة مئوية، <إدراج >إجمالي > 1.1، وإدراجالسيرةالذاتية،إجمالي ≤ 1.5 (اسم البرنامج النصي: filter_inserts.py).
- لكل إدراج فريد، حساب السجل2(<إدراج >إجمالي،28 درجة مئوية / insert>المجموع، 39 درجةمئوية). تمثل هذه القيمة "التسامح الحراري" لاستنساخ متحولة إدراج transposon معينة (اسم البرنامج النصي: fitness_ratios.py).
- فرز جميع عمليات الإدراج فريدة من نوعها حسب الجينات وآليل(S. سيريفيسياي أو S. مفارقة)،وجدولة عدد من الإدراج في كل الألايل. تصفية الجينات بحيث يتم تحليل الجينات التي تحتوي على ما لا يقل عن 5 إدراجات في كل أليل (اسم البرنامج النصي: organize_and_filter_genes.py).
ملاحظة: عمليات إدراج فريدة متعددة عبر كل أليل تسمح بقياس أكثر دقة من أن التسامح الحراري hemizygote المتبادلة. خفض عدد عمليات الإدراج المطلوبة لكل الأليل أمر ممكن ولكن سوف يعرض للخطر دقة هذا التدبير وزيادة عبء الاختبار المتعدد من خلال السماح باختبار المزيد من الجينات. بالإضافة إلى ذلك، فإن تصفية الجينات مع عدد قليل جدا من عمليات الإدراج في الأليل سيساعد على الحد من تأثير على نتائج الاختبار من أي استنساخ hemizygote الفردية التي تأوي طفرة موقع ثانوي أن يضفي على النمط الظاهري متباينة جدا. - لكل جين المتبقية في مجموعة البيانات بعد التصفية المذكورة أعلاه، قارن التحمل الحراري (سجل2 النسب) من جميع عمليات الإدراج في S. سيريفيسياي أليل لتلك الموجودة في S. مفارقة أليل باستخدام اختبار مان ويتني U. بدلاً من ذلك، يمكن تنفيذ نموذج الانحدار، تكييفها من DESeq28 (اسم البرنامج النصي: mann_whitney_u.py).
- تصحيح القيم pلاختبار متعددة باستخدام الأسلوب Benjamini-Hochberg.
- الجينات ذات القيم pالهامة (مثلا، ≤ 0.01) هي مرشحة للجينات الهامة للاختلافات في التسامح الحراري بين النوعين.
النتائج
لقد تزاوجنا مع S. cerevisiae و S. paradoxus لتشكيل هجين معقم، والتي تعرضنا لها لتحول المتحولين. وكان كل استنساخ المتحولة hemizygote، هجين ثنائي الذي يتم تعطيل أليل واحد من جين واحد (الشكل 1A، الشكل 2). تنافسنا hemizygotes ضد بعضها البعض من خلال النمو في 39 درجة مئو...
Discussion
مزايا RH-seq على الأساليب الإحصائية الوراثية السابقة هي عدة أضعاف. وعلى النقيض من تحليل الروابط والروابط، فإن RH-seq يتيح دقة رسم خرائط الجينات الواحدة؛ على هذا النحو، فإنه من المرجح أن تكون ذات فائدة كبيرة حتى في دراسات الاختلاف سمة عبر الأفراد من نوع معين، فضلا عن الاختلافات بين محددة. أيضا، ?...
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
نشكر ج. روب، ر. هاكلي، إ. غريغورييف، أ. أركين و ج. سكركر على إسهاماتهم في الدراسة الأصلية، ف. الزابن، أ. فلوري، ج. جيسلمان، ج. هونغ، ج. كيم، م. ماورير، ول. أولتروج على المساعدة التقنية، د. سافاج على كرمه بالفحص المجهري بلاكمان، س. كارادتي، أ. فلامهولز، ف. غواتشي، د. كوشلااند، س. نيلسون، وأ. ساسيكومار لإجراء مناقشات؛ كما نشكر ج. دوبر (قسم الهندسة الحيوية، جامعة كاليفورنيا بيركلي) على بلازميد PiggyBac. وقد تم دعم هذا العمل من قبل R01 GM120430-A1 ومن قبل مشروع التسلسل المجتمعي 1460 إلى RBB في معهد الجينوم المشترك التابع لوزارة الطاقة الأميركية، وهو مكتب تابع لمكتب المستخدمين العلميين التابع لوزارة الطاقة. وحظي العمل الذي اضطلع به هذا الأخير بدعم من مكتب العلوم التابع لوزارة الطاقة الأمريكية بموجب العقد رقم DE-AC02-05CH11231.
Materials
| Name | Company | Catalog Number | Comments |
| 1-2 plasmid Gigaprep kits | Zymo Research | D4204 | The number of kits required depends on how efficient your preps are in each kit. This kit comes with 5 individual plasmid prep columns. Run 1 L of saturated E. coli culture through each prep column, as using more than 1 L per column can cause clogging of the prep filter, leading to low yield and poor quality DNA. |
| 10X Tris-EDTA (TE) buffer (100 mM Tris-HCl and 10 mM EDTA) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 1M LiOAc | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 300 mg/mL Geneticin (G418) | Gibco | 11811023 | |
| 52% polyethylene glycol (PEG) 3350 | Sigma | 1546547 | Dissolve in water and filter sterilize through a 0.22 μm filter before use. 1X trafo mix: 228 uL 52% PEG, 36 uL 1M LiOAc, 36 uL 10X TE buffer |
| Autoclaved LB liquid broth | BD Difco | 244620 | Make LB liquid broth using your powder from any brand, and milliQ water. Autoclave it before use. |
| Carbenicillin stock in water (100 mg/mL) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| Complete synthetic agar plates (24.1cm x 24.1cm) with 5-fluoroorotic acid (5-FOA) [0.2% drop-out amino acid mix without uracil or yeast nitrogen base (YNB), 0.005% uracil , 2% D-glucose, 0.67% YNB without amino acids, 0.075% 5-FOA] | 5-FOA: Zymo Research, Drop-out mix: US Biological, Uracil: Sigma, D-glucose: Sigm), YNB: Difco | 5-FOA: F9001-5, Drop-out mix: D9535, Uracil: U0750, D-glucose: G8270, YNB: DF0919 | |
| DMSO | Any | N/A | |
| E. coli strain carrying pJR487 (CEN-/ARS+ piggyBac-containing plasmid) | N/A | N/A | Request from Brem lab. |
| Hybrid yeast strain JR507 (S. cerevisiae DBVPG1373 x S. paradoxus Z1, URA-/URA-) | N/A | N/A | Request from Brem lab. |
| Illumina Hiseq 2500 | used for SE-150 reads | ||
| Large shaking incubators with variable temperature settings | Any | N/A | |
| LB + carbenicillin agar plates (100 μg/mL) | Agar: BD Difco | Agar: 214010 | Make LB agar plates as normal and add carbenicillin to 100 μg/mL before drying. |
| Nanodrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit Fluorimeter | Thermo Scientific | Q33240 | |
| Salmon sperm DNA | Invitrogen | 15632011 | |
| Water bath at 39°C | Any | N/A | |
| Yeast fungal gDNA prep kit | Zymo Research | D6005 | |
| Yeast peptone dextrose (YPD) liquid media | BD Difco | Peptone: 211677, Yeast Extract: 212750 | Add filter-sterilized D-glucose to 2% after autoclaving. |
| YPD + G418 agar plates (300 μg/mL) | Agar: BD Difco | Agar: 214010 | Make YPD agar plates as normal and add G418 to 300 μg/mL before drying. |
| YPD agar plates | Agar: BD Difco | Agar: 214010 |
References
- Flint, J., Mott, R. Finding the molecular basis of quantitative traits: successes and pitfalls. Nature Reviews Genetics. 2, 437-445 (2001).
- Allen Orr, H. The genetics of species differences. Trends in Ecology and Evolution. 16, 343-350 (2001).
- Weiss, C. V., et al. Genetic dissection of interspecific differences in yeast thermotolerance. Nature Genetics. 50, 1501-1504 (2018).
- Stern, D. L. Identification of loci that cause phenotypic variation in diverse species with the reciprocal hemizygosity test. Trends in Genetics. 30, 547-554 (2014).
- Steinmetz, L. M., et al. Dissecting the architecture of a quantitative trait locus in yeast. Nature. 416, 326-330 (2002).
- Scannell, D. R., et al. The Awesome Power of Yeast Evolutionary Genetics: New Genome Sequences and Strain Resources for the Saccharomyces sensu stricto Genus. G3 (Bethesda). 1, 11-25 (2011).
- Wetmore, K. M., et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons. MBio. 6, e00306-e00315 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15, 550 (2014).
- Wilkening, S., et al. An evaluation of high-throughput approaches to QTL mapping in Saccharomyces cerevisiae. Genetics. 196, 853-865 (2014).
- Kim, H. S., Huh, J., Riles, L., Reyes, A., Fay, J. C. A noncomplementation screen for quantitative trait alleles in saccharomyces cerevisiae. G3 (Bethesda). 2, 753-760 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved