需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过全基因组互惠性分析,对糖母菌菌种间热耐性差异的遗传图谱
摘要
通过测序(RH-seq)的互惠血性是绘制物种间特征差异遗传基础的有力新方法。血吸血池由转位子诱变产生,其适应性通过高全测序的竞争性增长进行跟踪。对结果数据的分析可确定特征背后的基因。
摘要
现代遗传学的中心目标是了解野生生物在表型上是如何和为什么不同的。迄今为止,该领域已在很大程度上依靠链接和关联映射方法的强度,该方法从物种个体之间的交配中追踪DNA序列变异和表型在重组后代之间的关系。这些方法虽然强大,但并不适合生殖隔离物种之间的特征差异。在这里,我们描述了一种全基因组解剖自然特性变异的新方法,这种变异可以很容易地应用于不相容的物种。我们的战略,RH-seq,是一个全基因组的对内血吸血测试的实施。我们利用它来识别导致酵母糖菌相对于其姐妹物种S.RH-seq利用转轴龙诱变来创建一个互惠血吸体池,然后通过高通量测序通过高温竞争进行跟踪。我们这里介绍的RH-seq工作流程提供了一种严谨、无偏见的方式来解剖萌芽酵母包层中的古老、复杂的特征,并告诫需要资源密集型深度测序,以确保基因图谱的基因组覆盖。随着测序成本的降低,这种方法为真核生物的未来应用带来了巨大的希望。
引言
自该领域诞生以来,了解野生个体变异的机械基础一直是遗传学的首要目标。当我们绘制出一个感兴趣的特征的位点时,新兴基因可以立即用作诊断和药物的目标,并可以揭示进化的原理。为此的行业标准是通过联系或关联1来测试基因型和表型之间的关系。这些方法功能强大,但存在一个关键限制——它们依赖于来自不育个体间交叉的重组后代的大面板。它们对于研究那些不能交配形成后代的物种没有用处。因此,该场几乎没有能力对生殖分离的物种2之间的特征差异进行无偏见的解剖。
在这项工作中,我们报道了一种新的方法RH-seq3的技术基础,用于对物种间特征变异的遗传基础进行基因组尺度调查。这种方法是互惠性异形测试4,5的大规模平行版本,它最初被设想为一种评估两个基因不同背景之间等位基因差异的表型效应的方法。特定位点 (图 1A)在这个方案中,两个不同的个体首先交配形成一个杂交,其中一半的基因组来自各自的父母。在此背景中,将生成多个菌株,每个菌株包含每个父位点的中断或删除副本。这些菌株是双重体,因为它们在基因组中无处不在,除了在兴趣点,它们被认为是单倍体,并被称为互惠,因为每个只缺乏一个父母的等位基因,其剩余的等位基因派生自其他父级。通过比较这些互惠性窒息菌株的表型 , 可以得出结论 , 在纵的位点 DNA 序列变异是否有助于感兴趣的特征 , 因为在位点变异是互惠之间的唯一遗传差异赫米西戈特菌株。通过这种方式,在控制良好的实验设置中,物种之间的遗传差异与物种之间的表型差异是有可能的。迄今为止,该测试的应用一直在候选基因框架中,也就是说,假设已经具备,候选位点的自然变异可能会影响特征。
接下来,我们用酵母作为模型系统,为基因组尺度的互惠性全致度屏幕奠定了协议。我们的方法通过在物种之间产生可行的无菌F1杂交物,并使它们被转位子诱变,从而创造了血小生子突变体的基因组补充。我们汇集出麻囊,在基于测序的检测中测量其表型,并测试池中具有给定基因的两个父母等位基因的克隆之间的频率差异。结果是一个位点目录,物种之间的变异会影响感兴趣的特征。我们实施RH-seq工作流程,以阐明两个萌芽酵母物种,糖母细胞和S.悖论之间的热耐受性差异的遗传基础,这一物种在500万年前就发生了分歧。
研究方案
1. 准备含有猪囊的质粒进行转化
- 将含有质粒pJR487的大肠杆菌菌株分到单菌群,放入LB + 卡比西林琼脂板。在37°C或单个菌落出现之前孵育1晚。
注:关于如何克隆质粒pJR487的描述可以在我们以前的工作3中找到。 - 在 100 μg/mL 时接种 1 L 的 LB + 卡比西林,在 2 L 玻璃瓶中使用单个含有 pJR487 的大肠杆菌。在 37°C 下生长过夜,在 200 rpm 转速下摇动,直到饱和(OD600 = 1.0)。
- 按照制造商公布的协议(详情见材料表)中所示,使用大规模质粒制备试剂盒从培养物中纯化质粒DNA。在10分钟孵育后,用5mL的洗脱缓冲液加热至37°C后,使DNA脱脂。
- 使用分光光度计测量质粒DNA的数量和质量(详情见材料表)。
- 重复步骤1.2= 1.4,直到在A260:A280比率至少1.8的比例下,总共分离出至少11mg质粒DNA。这可能需要一些准备,具体取决于效率。
- 将所有质粒预准备混合到一个管中,通过洗脱缓冲液或水使总体积达到20 mL。再次使用分光光度计测量最终数量和质量。在这最后20 mL体积中,质粒的浓度应至少为538纳克/μL。如果浓度高于538纳克/μL,用洗脱缓冲液或水将质粒稀释至538纳克/μL。疟原虫可储存在4°C下,直到使用前几周。
2. 创建一个无目标全基因组互惠造份子库
- 混合酵母细胞的转化制备
- 从-80°C冷冻库存应变到YPD琼脂板上的单菌落,将JR507划出。在26°C孵育2天或直到菌落出现。
注:JR507是一种通过单细胞交配的单细胞交配的S.cerevisiae DBVPG1373和S.悖论Z1(使用四分分压显微镜)3。 - 在 250 mL 玻璃瓶中接种 100 mL 的液体 YPD,单片位为 JR507,在 28°C、200 rpm 下摇动 24 小时,或直到达到静止相。
- 第二天,测量隔夜培养的600nm(OD600)的光学密度。将一些隔夜文化与新鲜液体 YPD 重新稀释到新的 1 L 玻璃瓶中,以 OD600的 0.2 和 500 mL 的体积来创造新的文化。
注: 如果隔夜培养的 OD600为 5.0,其中 C 为光学密度,V 为体积,则反稀释的示例计算: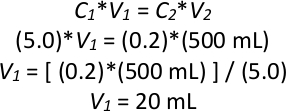
因此,在480 mL的液体YPD中,将20 mL的饱和过夜培养基添加到500 mL的培养基中,在OD600的0.2下。 - 重复步骤 2.1.3 三次,在四个 1 L 玻璃瓶中的 OD600 0.2 中,使总共四个 500 mL 培养物,对所有四种新文化使用相同的隔夜培养。在 28°C 下孵育它们,在 200 rpm 下摇动 6 小时(2-3 代)。
- 结合两个 500 mL 区域性,创建 1 L 区域性。合并剩余的两个 500 mL 区域性,以创建另一个 1 L 区域性。此时,有两个 1 L 区域性。在以下步骤中,这 1 L 区域性中的每一个都将使用 pJR487 进行转换。
- 从-80°C冷冻库存应变到YPD琼脂板上的单菌落,将JR507划出。在26°C孵育2天或直到菌落出现。
- pJR487转化为混合酵母细胞
- 将每个 1 L 培养物分成 20 mL 等分,在 20 个塑料锥形管中,共 40 个管。留出 20 个管,一次在 20 个管上执行以下步骤。
- 在1000 x g下将20个管子中的每一个离心3分钟,以颗粒酵母细胞。丢弃上清液。
- 通过涡旋将每粒颗粒与25 mL的无菌H2O重新悬浮。在1,000 x g下离心3分钟。丢弃上清液。
- 通过涡旋将每粒颗粒与 5 mL 的 1x TE、0.1 M LiOAc 缓冲液重新悬浮。在1,000 x g下离心3分钟。丢弃上清液。
- 重复步骤 2.2.4。当细胞离心时,制备至少120 mL的39.52%聚乙烯乙二醇溶液、0.12 M LiOAc和1.2x Tris-EDTA缓冲液(12 mM Tris-HCl 和 1.2 mM EDTA)。储存在冰上。
- 为了准备质粒DNA进行转化,首先在100°C下煮4mL鲑鱼精子DNA5分钟,并立即在冰上冷却5分钟。然后,将20 mL的pJR487(在第1节中获得)与4 mL冷却鲑鱼精子DNA混合,总体积为24 mL。保持冰上,直到使用。
- 在每个细胞颗粒上加入600 μL的质粒DNA与鲑鱼精子DNA混合。不要重新挂起。
- 将步骤 2.2.5 中制造的 3 mL PEG-LiOAc-TE 溶液添加到每个颗粒中。通过上下移液和涡流来重新悬浮颗粒。
- 在室温下孵育每管10分钟。
- 热休克每管26分钟,在水浴设置为39°C。
注:每隔几分钟,反转每个管以防止细胞沉降在管的底部。 - 在1000 x g下将每根管子离心3分钟。丢弃上清液,通过涡旋将每个颗粒重新悬浮在 10 mL 的 YPD 中。将所有二十根管子放入一个新的玻璃瓶中。细胞的总体积应为±200 mL。
- 将 66.6 mL 的电池转移到新的 1 L 玻璃瓶中,用液体 YPD 将容量达到 500 mL。再重复两次,以使用整个200 mL的转化细胞。测量每个新的 500 mL 培养的 OD600(预期 OD600为 ±0.35-4)。
- 在 28°C 下摇动所有三个烧瓶 2 小时,以 200 rpm 转速恢复(<1 代)。
- 将 0.5 mL 的 300 mg/mL G418 添加到三个烧瓶中,最终浓度为 300 μg/mL G418,并在 28°C、200 rpm 下放回摇摇。
注:在此步骤之前,转换后的混合细胞已从转换中恢复。加入G418后,选择质粒pJR487的存在。任何在转化过程中没有接受质粒的细胞将开始死亡。 - 对剩余的20个圆锥形细胞管重复步骤2.2.2 = 2.2.14。此时应有 6 个 1 L 玻璃瓶,每个烧瓶都添加了 500 mL 的电池,并添加了 G418。
- 在28°C下孵育所有六个细胞瓶,在200rpm下摇动,大约2天,或直到每个烧瓶达到+2.3的OD600。将所有六个烧瓶组合在一起,创建单一文化。
注:尽管此区域性中的所有单元格不会在下游步骤中使用,但使用如此大的容量的目标是创建尽可能多的唯一转换事件,并通过将所有这些转换集中来规范化单个转换中的任何偏差一起。 - 使用 2.2.16 中创建的培养基,用 500 mL 的 YPD = G418 (300 μg/mL) 为 OD600的 0.2 接种两个新的 1 L 烧瓶。将会有可以丢弃的剩文化。
- 在 28°C 下孵育两个 1 L 烧瓶过夜,在 200 rpm 下摇动,直到每个烧瓶达到 OD600的 ±2.2(±3.5 代)。将两种文化合并为一种区域性,并再次测量组合区域性的 OD600。
注:此时,培养体应几乎完全由含有质粒pJR487的细胞组成。在部分细胞种群中,PiggyBac转位子将通过质粒表达的转位酶从质粒转成基因组。然而,转位酶的持续表达可能导致在选择过程中换位,这将掩盖基因型和表型之间的关系。接下来的几个步骤的目标是对质粒的存在进行反选择,以确保转位酶不再表达。生成的池是包含或不集成到基因组中的转位子的细胞的组合,但在后续映射步骤中只检测到包含转位子的细胞。在质粒编码丢失之前,在转位酶表达转化过程中的时间可能控制诱变后给定克隆具有多个转位子插入的机会。这些突变的频率,在分析任何一个基因时,一次都表现为"二次"突变,可以通过在诱变后排列一个定义的菌落数,然后结合它们的DNA和序列确认独立插入的数量来估计池中的位置。 - 在1000 x g下将这种培养的25 mL离心3分钟。计算 25 mL 中的总 OD600个单元的单元数(请参阅下面的示例计算)。丢弃上清液,重新悬浮在足够的H2O中,通过涡旋产生1.85 OD600/mL的细胞悬浮液。
注:如果组合培养的OD600为2.2,则水中细胞重新悬浮的示例计算: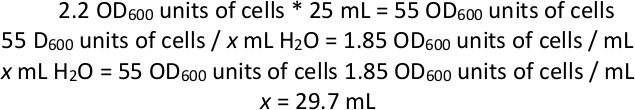
因此,在旋转25 mL的细胞培养物并丢弃上清液后,向细胞中加入足够的H2 O,使细胞和水的总体积达到+29.7 mL(因为细胞颗粒也会有体积,加入小于29.7 mL的H2O)。 - 使用玻璃珠,将水中的再悬浮细胞的1 mL板板到12个大方形完整的合成琼脂板中,每个板带有5-FOA。在28°C下孵育每个盘子1-2天,或直到板上形成草坪。
- 使用小型无菌刮刀,将6个板中的每块细胞刮掉,放入一个装有35 mL无菌水的管子中。用其他6个板重复,总共两管细胞和水。将所有细胞悬浮液组合在一根管中。测量此悬架的 OD600,用水为空白。将细胞的OD600/mL浓度与水一起至44.4 OD600单位/mL。根据我们的经验,转位效率(作为URA的KAN+细胞的比例)平均为50%。
- 确定要储存的 -80 °C 冷冻库数量。将来,每个等分可用于单个实验。
注: 考虑到池的生成费时,在意外误用或执行复制实验时存储多个小瓶。20-30只股票是一个合理的数字。 - 每个冷冻库将包含 40 个 OD600单位的细胞,1 mL 的 10% DMSO。将900μL的细胞添加到100μL的DMSO中。对创建的冷冻库存总数重复上述步骤。将每个储存在-80°C,以供将来使用。
3. 以集合格式选择互惠的希米奇戈特
- 在室温下,从-80°C冷冻器中解冻,从第2节解冻一个与第2节的池对合体。
注意:一旦水温解冻,请勿让等量分长时间坐在室温下,立即使用。 - 使用整个 1 mL 等分在 250 mL 玻璃瓶中接种 150 mL 的液体 YPD。测量此区域性的 OD600,然后在 28°C 孵育,在 200 rpm 下摇动,等待 +7 小时,或直到该培养值经过 2-3 个总体加倍。在这一点上,文化准备用于接种正在选择的文化。
注:示例计算:如果原始烧瓶的 OD600测量值为 0.25,则孵育培养法直到达到至少 1.0 的 OD600。如果任何采样点在"时间-零"(T-0)处需要,作为在选择前调查血吸血种群的方法,现在可以通过在1000 x g下每粒5-10mL的培养物取细胞颗粒3分钟,丢弃上清液和在-80°C冷冻。 - 使用生长的Hemizygote池在高温(39°C)和允许温度(28°C)下,在合适的复制方案中接种培养物。至少,在每个温度下设置三个生物复制选择培养物,总共六个选择培养物。
- 在 2 L 玻璃瓶中,使用液体 YPD 创建每个选择培养基,总和为 500 mL,并接种到 OD600的 0.02。在 28°C 或 39 °C 下以 100 rpm 的转速摇动每个选择培养基,直到出现 6-7 个总体加倍(对应于 ±1.28-2.56 的 OD600)。尽量匹配所有选择区域性的最终 OD600。
注:在 28°C 下选择区域性的生长速度将快于 39°C 的选型培养。因此,在39°C下选择培养物将在培养箱中停留更长时间。无论在培养箱中花费的总小时数如何,在每个烧瓶准备就绪时,继续执行以下步骤。根据我们的经验,在28°C或39°C下培养,分别需要+12或+18小时,达到±2.0的OD。长选择可以有放大小健身效应的优势,但也允许出现新背景突变,这将引入噪声到在任何一个基因/等位基因的转位子突变体的最终分布中。因此,在 RH-seq 实验中限制选择时间非常重要。
- 在 2 L 玻璃瓶中,使用液体 YPD 创建每个选择培养基,总和为 500 mL,并接种到 OD600的 0.02。在 28°C 或 39 °C 下以 100 rpm 的转速摇动每个选择培养基,直到出现 6-7 个总体加倍(对应于 ±1.28-2.56 的 OD600)。尽量匹配所有选择区域性的最终 OD600。
- 从每种选择培养基中收获细胞颗粒。计算从每个选择培养培养中获取 7 个 OD600单位的细胞和离心机所需的体积,在 1,000 x g下至少 3 分钟,至少 4 粒,作为库制备和测序的技术复制(参见第 4 和 5 节,下面)。丢弃上清液并储存在-80°C。
注: 选择烧瓶的最终 OD600为 2.0, 示例: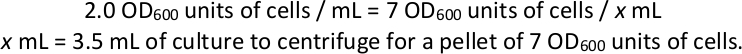
4. Tn-seq图书馆建设和Illumina测序以确定转位子突变体赫米西戈的丰度
- 解冻在冰上每个细胞颗粒从第3节,将被排序。
- 按照制造商的说明,使用酵母 gDNA 纯化试剂盒从每个细胞颗粒中分离出总基因组 DNA (gDNA)。在加热至65°C的50μL洗脱缓冲液中重新悬浮DNA。
- 使用荧光计量化每个颗粒中的 gDNA 数量。使用以下步骤为 Tn-seq 创建下一代测序 (NGS) 库所需的每个细胞颗粒所需的最小 gDNA 总量为 1 μg。
注: 不到 1 μg gDNA 可用于创建库,但库的最终数量和质量将受到影响。 - 遵循用于创建 Tn-seq 库的既定协议7。请注意此协议所特有的以下相关信息:
- gDNA 剪切、端修复和适配器连接后,通过 PCR 放大包含转座子的 gDNA。对于该 PCR,请使用以下正向和反向底漆,它们分别特定于 PiggyBac 转架和 NGS 适配器:
正向(N = 随机核苷酸)
5' ATGATACGACCACCACCACCACTACACACG
CTCTCCTATATATNNNNNNNNNNATATATATATATATATATGCGCATATAT3'
反向(Ns 的延伸表示用于多路复用的唯一 6 bp 索引)。有关指数的更多信息,请参阅下文)
5' 卡卡加加格加特加特纳格格格加特加特格格格格格格格
ACGTGTCTTCTCATCT 3' - 使用包含的清理步骤与大小选择性珠子,以尽量减少克隆片段在最终库中的比例,这将是太短,以包括可映射的基因组序列。
注:在遵循选择培养物的最低复制要求后,将有24个单独的gDNA样本进行测序。考虑到目前的测序成本,每个样本都不太可能自行运行。要在同一车道上组合样本,请创建多个反向引注,每个引基都有唯一的 6 基对索引。具有不同索引的样本可以组合到同一个测序通道中,然后以计算方式分离。
- gDNA 剪切、端修复和适配器连接后,通过 PCR 放大包含转座子的 gDNA。对于该 PCR,请使用以下正向和反向底漆,它们分别特定于 PiggyBac 转架和 NGS 适配器:
- 使用 NGS 技术从每个库中读取每个通道的单端 150 bp。
注:所需的测序读取量在很大程度上取决于上一步准备的库的质量(即库中实际包含转座子DNA的DNA比例,表示来自互惠的DNA)。有两个主要因素促成了这种情况。首先,由于没有集成转位子的细胞在池创建过程中不会反选择,因此每种培养物都是具有转位子和无转位子的细胞的组合。其次,即使在含有转座子的基因组中,大多数基因组也不含有序列的转座,而且这个gDNA将不可避免地成为库制备的一部分。含转座子DNA的最终PCR扩增的目标是提高含转座龙DNA与这两个背景gDNA来源的比例。这种放大效率越高,在下游分析中使用的读取比例就越高。图书馆质量越低,需要执行的测序就越多,因为越来越多的读取将不包含转座核DNA,也不会有用。鉴于上述限制,八条测序通道能够以合理程度跟踪对等的西皮戈特丰度。更多的测序将允许进行更深入的分析。
5. 绘制转波龙插入和RH-seq分析的位置图
注: 以下数据分析是使用自定义 Python 脚本完成的(在 https://github.com/weiss19/rh-seq 上联机找到),但可以使用其他脚本语言重新完成。下面概述了该过程的主要步骤。对每个复制读取文件执行以下步骤,除非已注意到要合并这些文件。
- 将适配器序列从读取中修剪出来,并根据索引分离每个复制的读取。
- 查找包含转位龙-基因组结的读取。为此,请在每次读取中搜索转波龙的最后 20 个碱基对,即 CAGATATATTTCTAGTTTTTTA。放弃所有不包含此序列的读取。
注:根据我们的经验,到转波子末端的读取映射比例为 83-95%。 - 修剪剩余的、包含转位子的读取,以仅包含转位子 3' 端下游的序列。通过将此序列映射到酵母基因组,确定每次读取的转位龙插入的基因组上下文(下文步骤 5.4)。
- 使用 BLAT 或等效的映射工具将转位子下游的序列映射到S. cereviae DBVPG1373 x S. 悖论Z1 混合基因组(脚本名称:map_和_pool_BLAT.py)。
- 放弃转座位点 3' 端下游可用序列少于 50 个基本序列的任何读取。短序列很难唯一映射。
- 如果使用 BLAT,请使用以下参数:标识 = 95,切片大小 = 12。
- 通过连接S.cereviae S288c和S.悖论CBS432的参考基因组的最新版本,创建一个基本的混合基因组,用于映射。
注:描述混合基因组中单个基因的基因组边界的基本注释文件可在上面列出的 Github 存储库中找到(文件名:YS2_CBS432_plasmid_clean)。仅使用读取到混合基因组中单个位置的映射(即 S. cerevisae 或 S. 悖论所独有)。预期整个基因组的插入事件的频率是均匀的;在基因组中插入位置的分布报告在其他地方3。
- 对每个唯一转位子插入位置的读取映射总数进行统计,我们推断所有读取都源自单个转位子插入突变克隆的细胞。单个库中所有此类值的总和称为该库的映射读取总数。
- 如果彼此的 3 个基本对内有多个插入映射,则将它们全部合并到单个插入点,将所有读取分配给读取计数最高的单个位置。此值 n插入表示从 gDNA 测序的细胞颗粒中插入克隆的丰度。在这一点上,将有n插入的列表,每个丰富的一个独特的映射转波龙插入,每个细胞颗粒序列一个列表。
注:PiggyBac转位龙插入基因组中的TTAA序列,4个碱基对序列。因此,我们推断,彼此的 3 个基本对内的插入映射必须来自同一个 TTAA 站点。 - 由于每个排序库的总读取数略有不同,因此如果要比较所有文件,则规范化 n插入值。为此,通过制表每个单个库的映射读取总数,n颗粒,并获取所有库中所有 n颗粒的平均值,颗粒>gt;;将单个库数据中的每个 n插入乘以 颗粒> / n颗粒的比率来计算插入,即给定转位子插入克隆的标准化丰度。
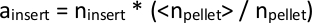
或者,可以使用可用工具(如 DESeq2 8(脚本名称:总计_读取_和_normalize.py)来估计库大小。 - 列出映射在所有库上的所有插入集。对于在某些库中找到的插入,而不是其他库中的插入,请为下游计算设置插入= 1。
- 筛选读取以根据注释文件查找属于基因中的插入(脚本名称:删除_NC_和_plasmid_insert.py)。
- 对于每个唯一插入,计算每个选择(每个区域性在 28 °C 或 39 °C 处)的技术复制之间的平均丰度,插入>技术(脚本名称:合并_tech_reps_V2.py)。
- 对于每个唯一的插入,计算每个温度的生物复制的平均丰度,插入> 总计,通过采用所有 插入的平均值>技术在每个温度下。同时,计算每次插入的变异系数,CV插入,总计跨 插入>技术(脚本名称:合并_bio_reps.py)。
注:此时,对于每个温度为28°C和39°C,有一个独特的转座子插入列表,其平均丰度和每种生物复制之间的变异系数。我们的实验数据在别处报告。 - 筛选在 28 °C 或 39 °C、插入>总计> 1.1 和 CV插入(总计= 1.5(脚本名称: filter_inserts.py) 下的所有插入的列表。
- 对于每个唯一插入,计算日志2(插入>总计,28 °C / insert>总计,39 °C)。此值表示给定转位子插入突变克隆的"热容差"(脚本名称:fitness_yy.py)。
- 按基因和等位基因(S.cerevisae或S.悖论)对所有唯一的插入进行排序,并列出每个等位基因中的插入数。过滤基因,以便只分析每个等位基因中至少有 5 个插入基因(脚本名称:组织_和_filter_genes.py)。
注:每个等位基因的多次唯一插入允许更准确地测量该互惠性热容差。降低每个等位方所需的插入数是可能的,但会损害此测量的准确性,并通过允许测试更多基因来增加多重测试负担。此外,过滤掉每个等位基因插入太少的基因,将有助于减少任何个体血吸子克隆对测试结果的影响,这些克隆具有二次位点突变,从而形成非常不同的表型。 - 对于上述过滤后数据集中的每个基因,使用 Mann-Whitney U 测试将S.cerevisae等位基因中所有插入的热容差(对数2比率)与S. 悖论等位基因进行比较。或者,可以实现回归模型,根据 DESeq28(脚本名称:mann_whitney_u.py)进行调整。
- 使用本杰明-霍奇伯格方法更正多个测试的p值。
- 具有显著p值的基因(例如 = 0.01)是两个物种之间热耐受性差异的重要基因的候选基因。
结果
我们交配S.cerevisae和S.paradoxus形成无菌杂交,我们进行转泊。每个诱变克隆都是血吸虫,一种双倍体杂交,其中一个基因的等位基因被打乱(图1A,图2)。我们在39°C下通过生长来相互竞争,在28°C(图1B)的单独实验中,我们从每种培养物中分离出DNA。为了报告每个半球体的适应性,我们通过批量测序对丰度进行了量化,?...
讨论
与以前的统计遗传方法相比,RH-seq的优点是几倍。与链接和关联分析相比,RH-seq提供单基因映射分辨率;因此,即使在研究特定物种个体之间的特征变异以及特异性差异时,它也可能具有重大效用。此外,以前在全基因组对内造异性分析的尝试使用基因缺失突变体的集合,其中一些含有二次突变,可能导致假阳性结果9,10。RH-seq策略通过依次生成和对每个基因中的?...
披露声明
作者没有什么可透露的。
致谢
我们感谢J.Roop、R.哈克利、I.格里戈里耶夫、阿·阿金和J.斯克尔对原始研究的贡献,F.AlZaben,A.Flury,G.Geiselman,J.Hong,J.Kim,M.Maurer和L.Oltrogge,D.萨维奇慷慨地提供显微镜资源,B.布莱克曼,S.科拉迪蒂,A.弗拉姆霍尔茨,V.瓜奇,D.科什兰,C.纳尔逊和A.萨西库马尔讨论;我们还感谢J.Dueber(加州大学伯克利分校生物工程系)的PiggyBac质粒。这项工作得到了R01 GM120430-A1和美国能源部科学用户设施办公室联合基因组研究所1460至RBB社区测序项目的支持。后者的工作得到美国能源部科学办公室根据第1号合同的支持。去 AC02-05CH11231。
材料
| Name | Company | Catalog Number | Comments |
| 1-2 plasmid Gigaprep kits | Zymo Research | D4204 | The number of kits required depends on how efficient your preps are in each kit. This kit comes with 5 individual plasmid prep columns. Run 1 L of saturated E. coli culture through each prep column, as using more than 1 L per column can cause clogging of the prep filter, leading to low yield and poor quality DNA. |
| 10X Tris-EDTA (TE) buffer (100 mM Tris-HCl and 10 mM EDTA) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 1M LiOAc | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 300 mg/mL Geneticin (G418) | Gibco | 11811023 | |
| 52% polyethylene glycol (PEG) 3350 | Sigma | 1546547 | Dissolve in water and filter sterilize through a 0.22 μm filter before use. 1X trafo mix: 228 uL 52% PEG, 36 uL 1M LiOAc, 36 uL 10X TE buffer |
| Autoclaved LB liquid broth | BD Difco | 244620 | Make LB liquid broth using your powder from any brand, and milliQ water. Autoclave it before use. |
| Carbenicillin stock in water (100 mg/mL) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| Complete synthetic agar plates (24.1cm x 24.1cm) with 5-fluoroorotic acid (5-FOA) [0.2% drop-out amino acid mix without uracil or yeast nitrogen base (YNB), 0.005% uracil , 2% D-glucose, 0.67% YNB without amino acids, 0.075% 5-FOA] | 5-FOA: Zymo Research, Drop-out mix: US Biological, Uracil: Sigma, D-glucose: Sigm), YNB: Difco | 5-FOA: F9001-5, Drop-out mix: D9535, Uracil: U0750, D-glucose: G8270, YNB: DF0919 | |
| DMSO | Any | N/A | |
| E. coli strain carrying pJR487 (CEN-/ARS+ piggyBac-containing plasmid) | N/A | N/A | Request from Brem lab. |
| Hybrid yeast strain JR507 (S. cerevisiae DBVPG1373 x S. paradoxus Z1, URA-/URA-) | N/A | N/A | Request from Brem lab. |
| Illumina Hiseq 2500 | used for SE-150 reads | ||
| Large shaking incubators with variable temperature settings | Any | N/A | |
| LB + carbenicillin agar plates (100 μg/mL) | Agar: BD Difco | Agar: 214010 | Make LB agar plates as normal and add carbenicillin to 100 μg/mL before drying. |
| Nanodrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit Fluorimeter | Thermo Scientific | Q33240 | |
| Salmon sperm DNA | Invitrogen | 15632011 | |
| Water bath at 39°C | Any | N/A | |
| Yeast fungal gDNA prep kit | Zymo Research | D6005 | |
| Yeast peptone dextrose (YPD) liquid media | BD Difco | Peptone: 211677, Yeast Extract: 212750 | Add filter-sterilized D-glucose to 2% after autoclaving. |
| YPD + G418 agar plates (300 μg/mL) | Agar: BD Difco | Agar: 214010 | Make YPD agar plates as normal and add G418 to 300 μg/mL before drying. |
| YPD agar plates | Agar: BD Difco | Agar: 214010 |
参考文献
- Flint, J., Mott, R. Finding the molecular basis of quantitative traits: successes and pitfalls. Nature Reviews Genetics. 2, 437-445 (2001).
- Allen Orr, H. The genetics of species differences. Trends in Ecology and Evolution. 16, 343-350 (2001).
- Weiss, C. V., et al. Genetic dissection of interspecific differences in yeast thermotolerance. Nature Genetics. 50, 1501-1504 (2018).
- Stern, D. L. Identification of loci that cause phenotypic variation in diverse species with the reciprocal hemizygosity test. Trends in Genetics. 30, 547-554 (2014).
- Steinmetz, L. M., et al. Dissecting the architecture of a quantitative trait locus in yeast. Nature. 416, 326-330 (2002).
- Scannell, D. R., et al. The Awesome Power of Yeast Evolutionary Genetics: New Genome Sequences and Strain Resources for the Saccharomyces sensu stricto Genus. G3 (Bethesda). 1, 11-25 (2011).
- Wetmore, K. M., et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons. MBio. 6, e00306-e00315 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15, 550 (2014).
- Wilkening, S., et al. An evaluation of high-throughput approaches to QTL mapping in Saccharomyces cerevisiae. Genetics. 196, 853-865 (2014).
- Kim, H. S., Huh, J., Riles, L., Reyes, A., Fay, J. C. A noncomplementation screen for quantitative trait alleles in saccharomyces cerevisiae. G3 (Bethesda). 2, 753-760 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。