È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Mappatura genetica delle differenze di termotolleranza tra le specie di Saccamici Yeast tramite l'analisi dell'emizigosità reciproca genomica
In questo articolo
Riepilogo
L'emizigozia reciprocata tramite sequenziamento (RH-seq) è un nuovo potente metodo per mappare la base genetica di una differenza tra le specie. I pool di emizigotes sono generati dalla mutagenesi trasposonista e la loro forma fisica è monitorata attraverso la crescita competitiva utilizzando il sequenziamento ad alto tutto. L'analisi dei dati risultanti individua i geni alla base del tratto.
Abstract
Un obiettivo centrale della genetica moderna è quello di capire come e perché gli organismi in natura differiscono nel fenotipo. Ad oggi, il campo è avanzato in gran parte sulla forza del collegamento e sui metodi di mappatura delle associazioni, che tracciano la relazione tra le varianti della sequenza del DNA e il fenotipo attraverso la progenie ricombinante dagli accoppiamenti tra individui di una specie. Questi approcci, anche se potenti, non sono adatti alle differenze tra di esse tra specie riproduttivamente isolate. Qui descriviamo un nuovo metodo per la dissezione dell'intersezione a livello genomico della variazione dei tratti naturali che può essere facilmente applicato a specie incompatibili. La nostra strategia, RH-seq, è un'implementazione a livello di genoma del test reciproco dell'emizigote. L'abbiamo sfruttato per identificare i geni responsabili della sorprendente crescita ad alta temperatura del lievito Saccharomyces cerevisiae rispetto alla sua specie gemella S. paradoxus. RH-seq utilizza la mutagenesi trasposoni per creare un pool di emizigoti reciproci, che vengono poi tracciati attraverso una concorrenza ad alta temperatura tramite il sequenziamento ad alto contenuto di velocità. Il nostro flusso di lavoro RH-seq, come qui riportato, fornisce un modo rigoroso e imparziale per sezionare tratti antichi e complessi nella clade di lievito in erba, con l'avvertenza che è necessario un sequenziamento profondo ad alta intensità di risorse per garantire la copertura genomica per la mappatura genetica. Con il calo dei costi di sequenziamento, questo approccio promette molto per un uso futuro negli eucarioti.
Introduzione
Fin dagli albori del campo, è stato un obiettivo primario nella genetica comprendere la base meccanicistica della variazione tra gli individui selvatici. Mentre tracciamo i loci alla base di un tratto di interesse, i geni emergenti possono essere di uso immediato come obiettivi per la diagnostica e la droga, e possono far luce sui principi dell'evoluzione. Lo standard di settore a tal fine è quello di verificare la necessità di una relazione tra genotipo e fenotipo tra una popolazione tramite collegamento o associazione1. Per quanto potenti siano questi approcci, hanno una limitazione fondamentale: si basano su grandi pannelli di progenie ricombinante provenienti da croci tra individui interfertili. Sono inutile nello studio di specie che non possono accoppiarsi per formare la progenie in primo luogo. Come tale, il campo ha avuto poca capacità di dissezione imparziale delle differenze di tratto tra le specie riproduttivamente isolate2.
In questo lavoro riportiamo le basi tecniche di un nuovo metodo, RH-seq3, per indagini su scala genomica della base genetica della variazione dei tratti tra le specie. Questo approccio è una versione massicciamente parallela del test reciproco dell'emizigote4,5, che è stato inizialmente concepito come un modo per valutare gli effetti fenotipici delle differenze alleliche tra due sfondi geneticamente distinti in un particolare locus (Figura 1A). In questo schema, i due individui divergenti vengono prima accoppiati per formare un ibrido, la metà del cui genoma proviene da ciascuno dei rispettivi genitori. In questo contesto, vengono generati più ceppi, ciascuno contenente una copia interrotta o eliminata dell'allele di ogni genitore del locus. Questi ceppi sono emiziosi in quanto rimangono diploidi ovunque nel genoma tranne che nel luogo di interesse, dove sono considerati aploidi, e sono indicati come reciproci poiché ognuno manca solo all'allele di un genitore, con il suo allele rimanente derivato dal altro genitore. Confrontando i fenotipi di questi ceppi emizigote reciproci, si può concludere se le varianti della sequenza del DNA al locus manipolato contribuiscono al tratto di interesse, poiché le varianti al locus sono l'unica differenza genetica tra il reciproco ceppi emizygote. In questo modo, è possibile collegare le differenze genetiche tra le specie a una differenza fenotipica tra di loro in una configurazione sperimentale ben controllata. Ad oggi le applicazioni di questo test sono state in un quadro candidato-gene, cioè i casi in cui l'ipotesi è già in corso che la variazione naturale in un locus candidato potrebbe avere un impatto su un tratto.
In quello che segue, stabiliamo il protocollo per uno schermo di emizigosità reciproca su scala genomica, usando il lievito come sistema modello. Il nostro metodo crea un complemento genomico di mutanti emizigote, generando ibridi F1 vitali e sterili tra le specie e sottoponendoli a mutagenesi tra trasposoni. Mettiamo insieme gli emizigoti, misuriamo i loro fenotipi nei saggi basati sul sequenziamento e verifichiamo le differenze di frequenza tra i cloni della piscina recanti gli alleli dei due genitori di un determinato gene. Il risultato è un catalogo di loci in cui le varianti tra le specie influenzano il tratto di interesse. Implementiamo il flusso di lavoro RH-seq per chiarire la base genetica delle differenze di termotolleranza tra due specie di lievito in erba, Saccharomyces cerevisiae e S. paradoxus, che ha diversificato 5 milioni di anni fa6.
Protocollo
1. Preparazione del plasmide contenente piggyBac per la trasformazione
- Streak fuori a singole colonie il ceppo E. coli che ospita pJR487 pJR487 su un piatto di agar Carbenicillin LB. Incubare per 1 notte a 37 gradi centigradi o fino a quando non appaiono singole colonie.
NOTA: Una descrizione di come plasmidp è stato clonato può essere trovato nel nostro lavoro precedente3. - Inoculare 1 L l di LB : carbenicillina a 100 g/mL con una singola colonia di E. coli contenente pJR487 in un flacone di vetro da 2 L. Coltivare durante la notte a 37 gradi centigradi con agitazione a 200 rpm fino a saturare (OD600 x 1,0).
- Purificare il DNA plasmide dalla coltura utilizzando un kit di preparazione plasmide su larga scala come indicato nel protocollo pubblicato dal produttore (vedere Tabella dei materiali per i dettagli). Esolute il DNA dopo un'incubazione di 10 minuti con 5 mL di tampone di eluizione riscaldato a 37 gradi centigradi.
- Misurare la quantità e la qualità del DNA plasmide con uno spettrometro (vedere Tabella dei materiali per i dettagli).
- Ripetere i passaggi da 1,2 a 1,4 fino a quando un totale di almeno 11 mg di DNA plasmide a un rapporto A260:A280 di almeno 1,8 sono isolati. Questa operazione può richiedere alcune preparatrici, a seconda dell'efficienza.
- Mescolare tutti i plasmici in un unico tubo e portare il volume totale fino a 20 mL con tampone di eluizione o acqua. Misurare di nuovo la quantità e la qualità finali con uno spettrofotometro. La concentrazione di plasmide deve essere di almeno 538 ng/L in questo volume finale di 20 mL. Se la concentrazione è superiore a 538 ng/L, diluire il plasmide con tampone di eluizione o acqua a 538 ng/L. Plasmide può essere conservato a 4 gradi centigradi fino a poche settimane fino all'uso.
2. Creazione di un pool di emizigoti reciproci a livello di genoma non mirati
- Preparazione di cellule di lievito ibrido per la trasformazione
- Striatura fuori JR507 da un ceppo di stoccaggio congelatore -80 gradi a singole colonie su una piastra di YPD agar. Incubare a 26 gradi centigradi per 2 giorni o fino a quando non appaiono colonie.
NOTA: JR507 è un ceppo ibrido fatto attraverso l'accoppiamento a singola cellula di spore aploidi di S. cerevisiae DBVPG1373 e S. paradoxus n. 1 (utilizzando un microscopio a dissezione tetrad)3. - Inoculare 100 mL di YPD liquido in un flacone di vetro da 250 mL con una singola colonia di JR507 e agitare a 28 gradi centigradi, 200 rpm per 24 ore, o fino a raggiungere la fase stazionaria.
- Il giorno successivo, misurare la densità ottica a 600 nm (OD600) della coltura durante la notte. Creare una nuova cultura diluendo un po'della cultura notturna con YPD liquido fresco in un nuovo fiaschetta di vetro da 1 L a un OD600 di 0,2 e un volume di 500 mL.
NOTA: Esempio di calcolo di una diluizione posteriore se la coltura notturna ha un OD600 di 5.0, dove C è densità ottica e V è volume: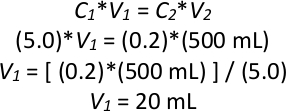
Così, 20 mL di coltura satura durante la notte verrebbero aggiunti a 480 mL di YPD liquido per fare un totale di 500 mL di coltura a un OD600 di 0,2. - Ripetere il passaggio 2.1.3 altre tre volte per fare un totale di quattro colture da 500 mL a un OD600 di 0,2 in quattro flaconi di vetro da 1 L, utilizzando la stessa cultura notturna per tutte e quattro le nuove culture. Incubarli tutti a 28 gradi centigradi per 6 ore (2-3 generazioni) tremando a 200 giri/min.
- Combinare due delle impostazioni cultura da 500 mL per creare una cultura 1 L. Combinare le altre due impostazioni cultura da 500 mL per creare un'altra coltura da 1 L. A questo punto, ci sono due culture 1 L. Ognuna di queste impostazioni cultura da 1 L sarà soggetta a trasformazione con pJR487 nei passaggi seguenti.
- Striatura fuori JR507 da un ceppo di stoccaggio congelatore -80 gradi a singole colonie su una piastra di YPD agar. Incubare a 26 gradi centigradi per 2 giorni o fino a quando non appaiono colonie.
- Trasformazione del pJR487 in cellule ibride di lievito
- Dividere ciascuna delle colture da 1 L in 20 mL in 20 tubi conici in plastica per un totale di 40 tubi. Mettere da parte 20 tubi ed eseguire i seguenti passaggi su 20 tubi alla volta.
- Centrifugare ciascuno dei venti tubi per 3 min a 1.000 x g per pellet le cellule lievito. Scartare il super-attardato.
- Risospendere ogni pellet con 25 mL di sterile H2O mediante vortice. Centrifuga per 3 min a 1.000 x g. Scartare il super-attardato.
- Risospendere ogni pellet con 5 mL di 1x TE, 0,1 M di tampone LiOAc mediante vortice. Centrifuga per 3 min a 1.000 x g. Scartare il super-attardato.
- Ripetere il passaggio 2.2.4. Mentre le cellule sono centrifusi, preparare almeno 120 mL di una soluzione di 39,52% di glicole di polietilene, 0,12 M LiOAc e 1,2x buffer Tris-EDTA (12 mM Tris-HCl e 1,2 mM EDTA). Conservare sul ghiaccio.
- Per preparare il DNA plasmide per la trasformazione, far bollire 4 mL di DNA dello sperma di salmone a 100 gradi centigradi per 5 min e raffreddarlo immediatamente sul ghiaccio per 5 min. Quindi, mescolare 20 mL di pJR487 (ottenuto nella sezione 1) ad una concentrazione di 538 ng/L con i 4 mL di DNA di spermadio di salmone raffreddato per un volume totale di 24 mL. Tenere sul ghiaccio fino all'uso.
- Aggiungete 600 l di DNA plasmico mescolato con dna di sperma di salmone sopra ogni pellet cellulare. Non risospendere ancora.
- Aggiungere 3 mL di soluzione PEG-LiOAc-TE realizzata al passaggio 2.2.5 per ogni pellet. Risospendere il pellet pipetting su e giù e vortice.
- Incubare ogni tubo per 10 min a temperatura ambiente.
- Scossa di calore ogni tubo per 26 min in un bagno d'acqua impostato a 39 gradi centigradi.
NOTA: Ogni pochi minuti, invertire ogni tubo per evitare che le cellule si assestino sul fondo del tubo. - Centrifugare ogni tubo per 3 min a 1.000 x g. Scartare il supernatante e rimettere in sospensione ogni pellet in 10 mL di YPD mediante vortice. Combinare tutti i venti tubi in un nuovo flacone di vetro. Il volume totale delle celle deve essere di 200 mL.
- Trasferire 66,6 mL di cellule in un nuovo flacone di vetro da 1 L e portare fino a un volume di 500 mL con YPD liquido. Ripetere altre due volte per utilizzare l'intero 200 mL di celle trasformate. Misurare l'OD600 di ogni nuova coltura 500 mL (si prevede un OD600 di 0,35-4 usd).
- Agitare tutti e tre i flaconi a 28 gradi centigradi per 2 ore per recuperare (<1 generazione) a 200 giri/min.
- Aggiungete 0,5 mL di 300 mg/mL G418 a ciascuno dei tre flaconi, ad una concentrazione finale di 300 g/mL G418 e rimessi a tremare a 28 gradi centigradi, 200 giri/m.
NOTA: prima di questo passaggio, le celle ibride trasformate si sono ripristinate dalla trasformazione. Dopo l'aggiunta di G418, viene selezionata la presenza del pLAsmide pJR487. Tutte le cellule che non hanno assorbito il plasmide durante la trasformazione inizieranno a morire. - Ripetere i passaggi da 2.2.2 – 2.2.14 con i restanti 20 tubi conici delle celle. A questo punto dovrebbero esserci sei flaconi di vetro da 1 L, ciascuno con 500 mL di cellule con l'aggiunta di G418 G418.
- Incubare tutti e sei i flaconi di cellule a 28 gradi centigradi, agitando a 200 giri/min, per circa 2 giorni o fino a raggiungere un OD600 di 2,3 dollari in ogni fiaschetta. Combina tutti e sei i flaconi per creare un'unica cultura.
NOTA: anche se tutte le celle di questa coltura non verranno utilizzate in passaggi a valle, l'obiettivo dell'utilizzo di volumi così grandi è stato quello di creare il maggior numero possibile di eventi di trasformazione unici e di normalizzare eventuali pregiudizi in una singola trasformazione raggruppandoli tutti insieme. - Utilizzate la cultura creata nella versione 2.2.16 per inoculare due nuovi flaconi da 1 L con 500 mL di YPD ( G418) a un OD600 di 0,2. Ci sarà una cultura rimasta che può essere scartata.
- Incubare entrambi i flaconi da 1 L a 28 gradi durante la notte, con agitazione a 200 giri/min, fino a raggiungere un OD600 di 2,2 dollari (3,5 generazioni). Combina entrambe le culture in un'unica cultura e misura di nuovo l'OD600 della cultura combinata.
NOTA: A questo punto, la coltura dovrebbe essere costituita quasi interamente da celle che ospitano pJR487 pJRplasmid. In parte della popolazione di cellule, il trasposione PiggyBac sarà stato trasposto dal plasmide nel genoma dalla trasposasi espressa al largo del plasmico. Tuttavia, l'espressione continua della trasposasi può portare alla trasposizione nel corso di una selezione, che oscurerebbe la relazione tra genotipo e fenotipo. L'obiettivo dei prossimi passi è quello di eseguire una controselezione contro la presenza del plasmide, per garantire che non ci sia più espressione della trasposasi. Il pool risultante è un mix di cellule con o senza il trasposone integrato nel genoma, ma solo le cellule contenenti il trasposone vengono rilevate durante le fasi di mappatura successive. Il tempo nella trasformazione durante la quale si esprime la trasposasi, prima che la codifica del plasmide vada persa, può governare la possibilità che un dato clone dopo la mutagenesi contturi più di un inserimento di trasposoni. La frequenza di queste, che si manifestano come mutazioni "secondarie" nell'analisi di un gene alla volta, può essere stimata mettendo in evidenza un numero definito di colonie dopo la mutagenesi, combinando il loro DNA e la conferma della sequenza che conferma il numero di inserimenti indipendenti posizioni nella piscina. - Centrifuga 25 mL di questa coltura per 3 min a 1.000 x g. Calcolare il numero totale di oD600 unità di celle che si trovano in 25 mL (vedere il calcolo di esempio di seguito). Scartare il supernatante e riutilizzare in abbastanza H2O per creare una sospensione cellulare di 1.85 OD600/ mL mediante vortice.
NOTA: Esempio di calcolo per la risospensione delle cellule in acqua se OD600 di coltura combinata è stato 2.2: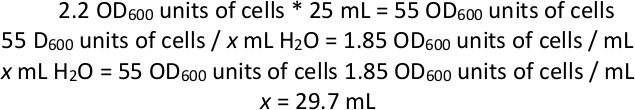
Quindi, dopo aver filato 25 mL di coltura cellulare e scartato il supernatale, aggiungi abbastanza H2O alle cellule per portare il volume totale delle cellule e dell'acqua fino a 29,7 mL (dal momento che il pellet cellulare avrà anche un volume, aggiungi meno di 29,7 mL di H2O). - Utilizzando perline di vetro, piastra 1 mL di celle risospese in acqua su ciascuna delle 12 grandi piastre di agar sintetico quadrate con 5-FOA. Incubare ogni piatto a 28 gradi centigradi per 1-2 giorni o fino a quando non si forma un prato sulla piastra.
- Utilizzando piccoli schizzinosi sterili, raschiare le cellule da ciascuna delle 6 piastre e in un tubo con 35 mL di acqua sterile. Ripetere con le altre 6 piastre per un totale di due tubi di cellule e acqua. Combinare tutte le sospensioni cellulari in un unico tubo. Misurare l'OD600 di questa sospensione, utilizzando l'acqua come uno spazio vuoto. Portare la concentrazione oD600/ mL delle cellule a 44,4 OD600 unità/mL con acqua. Nella nostra esperienza, l'efficienza di trasposizione (la percentuale di cellule KAN- che sono URA-) è in media del 50%.
- Determinare il numero di scorte di celle congelatore a -80 gradi centigradi. Ogni aliquota può essere utilizzata in futuro per un singolo esperimento.
NOTA: dato quanto tempo richiede la generazione del pool, archiviare più fiale in caso di uso improprio accidentale o per l'esecuzione di esperimenti di replica. 20-30 azioni sono un numero ragionevole. - Ogni stock di congelatore conterrà 40 OD600 unità di celle in 1 mL del 10% di DMSO. Aggiungete 900 celle a 100 -L l di DMSO. Ripetere l'operazione per il numero totale di scorte congelatori create. Conservare ciascuno a -80 gradi centigradi per un uso futuro.
3. Selezione degli emizigotes reciproci in un formato raggruppato
- Scongelare dal congelatore -80 gradi centigradi un'unica aliquota di emizigotes reciproci raggruppati dalla sezione 2 a temperatura ambiente.
NOTA: Non lasciare che l'aliquota si sieda a lungo a temperatura ambiente una volta che si scongela, usarlo immediatamente. - Utilizzare l'intero 1 mL aliquot per inoculare 150 mL di YPD liquido in un flacone di vetro da 250 mL. Misurare l'OD600 di questa coltura, e poi incubare a 28 gradi centigradi, agitando a 200 rpm, per 7 ore, o fino a quando la coltura ha attraversato 2-3 raddoppi di popolazione. A questo punto, la cultura è pronta per essere utilizzata per inoculare le culture sottoposte a selezione.
NOTA: Esempio di calcolo: se l'OD600 del pallone originale misura 0,25, incubare la coltura fino a raggiungere un OD600 di almeno 1,0. Se si desiderano punti campione a "Time-zero" (T-0), per studiare la popolazione di emizygote prima della selezione, i pellet cellulari possono essere presi ora centrifundo 5-10 mL di coltura per pellet a 1.000 x g per 3 min, scartando il congelamento a -80 gradi centigradi. - Utilizzare la piscina emizigote coltivata per inoculare colture per la selezione in uno schema di replica adatto, sia ad alta temperatura (39 gradi centigradi) che a temperatura permissiva (28 gradi centigradi). Come minimo, impostare tre colture biologiche di selezione replica ad ogni temperatura, per un totale di sei colture di selezione.
- Creare ogni coltura di selezione con un totale di 500 mL in un flacone di vetro da 2 L con YPD liquido e inoculare a un OD600 di 0,02. Agitare ogni coltura di selezione a 100 giri/min o 39 gradi centigradi fino a 6-7 raddoppi della popolazione (corrispondente a un OD600 di 1,28-2,56 dollari). Cerca di abbinare il più fedelmente possibile l'OD600 finale di tutte le impostazioni cultura di selezione.
NOTA: le colture di selezione a 28 gradi centigradi cresceranno più velocemente delle culture di selezione a 39 gradi centigradi. Di conseguenza, le colture di selezione a 39 gradi centigradi trascorreranno un periodo di tempo più lungo nell'incubatrice. Procedere con i seguenti passaggi con ogni fiaschetta come diventa pronto, indipendentemente dal numero totale di ore trascorse nell'incubatrice. Nell'esperienza della nostra esperienza, le culture a 28 o 39 gradi centigradi hanno impiegato rispettivamente 12 o 18 ore per raggiungere un OD di 2,0 dollari. Le selezioni lunghe potrebbero avere il vantaggio di amplificare piccoli effetti di fitness, ma anche consentire l'insorgere di mutazioni di fondo de novo, che introdurrebbero rumore nella distribuzione finale di fitness tra mutanti trasposoni in un gene/allele. Come tale è importante limitare il tempo di selezione in un esperimento RH-seq.
- Creare ogni coltura di selezione con un totale di 500 mL in un flacone di vetro da 2 L con YPD liquido e inoculare a un OD600 di 0,02. Agitare ogni coltura di selezione a 100 giri/min o 39 gradi centigradi fino a 6-7 raddoppi della popolazione (corrispondente a un OD600 di 1,28-2,56 dollari). Cerca di abbinare il più fedelmente possibile l'OD600 finale di tutte le impostazioni cultura di selezione.
- Raccogli pellet cellulari di ogni coltura di selezione. Calcolare il volume necessario per ottenere 7 OD600 unità di cellule e centrifugare a 1.000 x g per 3 min almeno quattro pellet di questo volume da ogni coltura di selezione come repliche tecniche per la preparazione della libreria e il sequenziamento (vedi sezioni 4 e 5 , di seguito). Scartare il supernatante e conservarlo a -80 gradi centigradi.
NOTA: Esempio se una fiaschetta di selezione ha un OD600 finale di 2.0: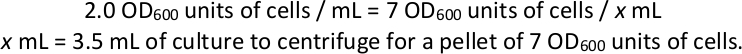
4. Costruzione della biblioteca Tn-seq e sequenziamento Illumina per determinare l'abbondanza di emizigoti mutanti trasposoni
- Scongelare sul ghiaccio ogni pellet cellulare dalla sezione 3 che sta per essere sequenziato.
- Isolare il DNA genomico totale (gDNA) da ogni pellet cellulare utilizzando un kit di purificazione gDNA di lievito seguendo le istruzioni del produttore. Risospendere il DNA in 50 gradi centigradi di tampone di eluizione riscaldato a 65 gradi centigradi.
- Quantificare la quantità di gDNA da ogni pellet utilizzando un fluorimetro. La quantità totale minima di gDNA necessaria per ogni pellet cellulare per creare una libreria di sequenziamento (NGS) di nuova generazione per Tn-seq utilizzando la seguente procedura è 1 g.
NOTA: È possibile utilizzare meno di 1 g di gDNA per creare una libreria, ma la quantità finale e la qualità della libreria ne risentiranno. - Seguire un protocollo stabilito per la creazione di librerie Tn-seq7. Tenere presente le seguenti informazioni rilevanti che sono univoche per questo protocollo:
- Dopo la tosatura gDNA, la riparazione finale e la legatura dell'adattatore, amplificare il gDNA contenente il trasposone tramite PCR. Per tale PCR, utilizzare il seguente primer in avanti e indietro, che sono specifici per il trasposone PiggyBac e gli adattatori NGS, rispettivamente:
Avanti (N – nucleotide casuale)
5' ATGATACGGCACCACCGAATCTCTCTCTCTCTCTCCTACACGAG
CTCTTCCGATCTNNNNNNAGANANANATTCAAGAATGCATGCGTCAAT 3'
Retro (l'estensione di Ns rappresenta un indice univoco di 6 bp utilizzato per il multiplexing. Vedi sotto per ulteriori informazioni sugli indici)
5' CAAGCAGAGaACGGCATACGAGAATNNNNGGTGGAGTTCAG
ACGTGTGCTCTCTGATCTCT 3' - Utilizzare le istruzioni di pulizia incluse con perline selettive per ridurre al minimo la proporzione di frammenti clonati nella libreria finale che sarebbe troppo breve per includere la sequenza genomica mappabile.
NOTA: dopo aver seguito i requisiti minimi di replica fino ad ora per le impostazioni cultura di selezione, ci saranno 24 campioni gDNA individuali per il sequenziamento. Dato il costo corrente per la sequenza, è improbabile che ogni campione venga eseguito da solo. Per combinare i campioni sulla stessa corsia, creare più primer invertiti, ciascuno con un indice di coppia univoco a 6 basi. I campioni con indici diversi possono essere combinati nella stessa corsia di sequenziamento e separati computazionalmente in seguito.
- Dopo la tosatura gDNA, la riparazione finale e la legatura dell'adattatore, amplificare il gDNA contenente il trasposone tramite PCR. Per tale PCR, utilizzare il seguente primer in avanti e indietro, che sono specifici per il trasposone PiggyBac e gli adattatori NGS, rispettivamente:
- Sequenza single-end 150 bp legge da ogni libreria utilizzando le tecnologie NGS su otto corsie.
NOTA: la quantità di letture di sequenziamento richieste dipende in larga misura dalla qualità delle librerie preparate nel passaggio precedente (cioè la percentuale di DNA nella libreria che contiene effettivamente il DNA trasposizione, che rappresenta il DNA proveniente da emizigote scambiosi). Ci sono due fattori principali che contribuiscono a questo. In primo luogo, poiché le cellule senza un trasposone integrato non sono controdeselezionate durante la creazione della piscina, ogni coltura sarà un mix di cellule con e senza trasposon. In secondo luogo, anche all'interno dei genomi degli emizigoti reciproci contenenti trasposoni, la maggior parte del genoma non è trasposone contenente sequenza, e questo gDNA sarà inevitabilmente parte della preparazione della libreria. L'obiettivo dell'amplificazione PCR finale del DNA contenente trasposoni è quello di aumentare il rapporto tra il DNA contenente trasposoni a queste due fonti di gDNA di fondo. Più efficiente è questa amplificazione, maggiore sarà la percentuale di letture che sarà in grado di essere utilizzata nell'analisi a valle. Più qualità le biblioteche sono inferiori, più sequenziamento dovrà essere fatto, poiché una percentuale crescente di letture non conterrà DNA trasposizione e non sarà utile. Considerati i vincoli di cui sopra, otto corsie di sequenziamento erano in grado di tracciare l'abbondanza di emizigote reciproche in misura ragionevole. Un maggiore sequenziamento consentirebbe un'analisi più approfondita.
5. Mappatura delle posizioni degli inserimenti di trasposoni e analisi RH-seq
NOTA: la seguente analisi dei dati è stata eseguita con script Python personalizzati (disponibili online all'https://github.com/weiss19/rh-seq), ma potrebbe essere rifatta utilizzando altri linguaggi di scripting. Di seguito sono descritti i passaggi principali del processo. Eseguire la procedura seguente su ogni singolo file di lettura replica, a meno che non venga indicato per combinarli.
- Tagliare le sequenze dell'adattatore fuori dalle letture e separare le letture di ogni replica in base all'indice.
- Trova letture contenenti giunzioni trasposizione-genoma. A tale scopo, cercare all'interno di ogni lettura le ultime 20 coppie di base del trasposone, CAGACTATCTCTTTCTAGGTAAA. Elimina tutte le letture non contenenti questa sequenza.
NOTA: Nella nostra esperienza, la percentuale di letture mappatura alla fine del trasposone è 83-95%. - Tagliare le letture rimanenti contenenti trasposoni per contenere solo la sequenza a valle della fine del trasposone del 3'. Mappando questa sequenza al genoma del lievito, determinare il contesto genomico dell'inserimento del trasposone per ogni lettura (passaggio 5.4 più avanti).
- Utilizzare BLAT o uno strumento di mappatura equivalente per mappare la sequenza a valle del trasposone al S. cerevisiae DBVPG1373 x S. paradossale 1 genoma ibrido (nome dello script: map_and_pool_BLAT.py).
- Eliminare tutte le letture per le quali sono presenti meno di 50 coppie di basi di sequenza utilizzabile a valle di 3' fine del trasposone. Le sequenze brevi sono difficili da mappare in modo univoco.
- Se si utilizza BLAT, utilizzare i seguenti parametri: identità : 95, dimensione del riquadro : 12.
- Creare un genoma ibrido di base da utilizzare per la mappatura concatenando le versioni più recenti dei genomi di riferimento di S. cerevisiae S288c e S. paradoxus CBS432.
NOTA: un file di annotazione di base che descrive i confini genomici dei singoli geni attraverso il genoma ibrido è disponibile nel repository Github elencato in precedenza (Nome file: YS2-CBS432-plasmid_clean). Utilizzare solo letture che mappano in una singola posizione nel genoma ibrido (cioè sono uniche per S. cerevisiae o S. paradoxus). Si prevede una frequenza uniforme di eventi di inserimento in tutto il genoma; la distribuzione delle posizioni di inserimento tra i genomi è riportata altrove3.
- Tally il numero totale di letture mappatura a ogni posizione di inserimento trasposone unico, che deduciamo tutti ha avuto origine da cellule di un singolo clone mutante di inserimento trasposon. La somma di tutti questi valori da una singola libreria viene definita numero totale di letture mappate per tale libreria.
- Nei casi in cui sono presenti più inserimenti mapping all'interno di 3 coppie di base tra loro, combinarli tutti in un singolo punto di inserimento, assegnando tutte le letture alla singola posizione con il numero di letture più alto. Questo valore, ninsert, rappresenta l'abbondanza di tale clone di inserimento nella cella pellet da cui gDNA è stato sequenziato. A questo punto, ci saranno liste di ninsert, ciascuna l'abbondanza di un unico inserimento transposon mappato, un elenco per ogni pellet cella sequenziato.
NOTA: il trasposo PiggyBac inserisce le sequenze TTAA nel genoma, una sequenza di 4 coppie di basi. Pertanto, deduciamo che gli inserimenti che si mappano all'interno di 3 coppie di base l'uno dell'altro devono aver avuto origine dallo stesso sito TTAA. - Poiché ci sarà un numero leggermente diverso di letture totali provenienti da ogni libreria in sequenza, normalizzare i valori diinserimento n in tutti i file se devono essere confrontati. A tale scopo, è possibile che il numero totale di letture mappate di ogni singola libreria, npellet,e prenda la media di tutti i npellet in tutte le librerie, pellet>. Moltiplicare ogni inserto n nei dati di una singola libreria per il rapporto di pellet> / npellet per calcolare un inserto, l'abbondanza normalizzata di un determinato clone di inserimento trasposoni.
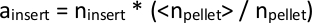
In alternativa, è possibile stimare le dimensioni della libreria utilizzando strumenti disponibili come DESeq28 (nome dello script: total_reads_and_normalize.py). - Anteceria l'insieme di tutti gli inserimenti mappati in tutte le librerie. Per gli inserimenti trovati in alcune librerie, ma non in altre, impostare un inserto 1 per i calcoli a valle.
- Filtrare le letture per trovare gli inserimenti che rientrano nei geni in base al file di annotazione (Nome script: remove_NC_and_plasmid_inserts.py).
- Per ogni inserimento univoco, calcolare l'abbondanza media tra le repliche tecniche di ogni selezione (ogni lingua a 28 o 39 gradi centigradi), insert>technical (nome dello script: combine_tech_reps_V2.py).
- Per ogni inserimento univoco, calcolare l'abbondanza media tra repliche biologiche di ogni temperatura, insert>total, prendendo la media di tutto insert>tecnico ad ogni temperatura. Allo stesso tempo, calcolare il coefficiente di variazione per ogni inserimento,CV insert,total across the insert>technical (Nome script: combine_bio_reps.py).
NOTA: A questo punto, per ogni temperatura, 28 e 39 gradi centigradi, c'è un elenco di inserimenti di trasposoni unici, la loro abbondanza media e il coefficiente di variazione tra repliche biologiche per ciascuno. Questi dati per il nostro esperimento sono riportati altrove3. - Filtrare l'elenco di tutti gli inserimenti per quelli che hanno, a 28 o 39 gradi centigradi, insert>total > 1.1 e CVinsert,totale : 1,5 (nome dello script: filter_inserts.py).
- Per ogni inserimento univoco, calcolare il log2(insert>total,28 c / insert>total,39)C . Questo valore rappresenta la "termotolleranza" di un dato clone mutante di inserimento trasposone (Nome dello script: fitness_ratios.py).
- Ordinare tutti gli inserimenti unici per gene e per allele (S. cerevisiaee o S. paradoxus) e tabulare il numero di inserimenti in ogni allele. Filtrare i geni in modo che vengano analizzati solo i geni che hanno almeno 5 inserimenti in ogni allele (Nome dello script: organize_and_filter_genes.py).
NOTA: Più inserimenti unici in ogni allele consentono una misura più accurata della termotolleranza di quell'emizigote reciproco. È possibile ridurre il numero di inserimenti necessari per allele, ma comprometterel'accuratezza di questa misura e aumentare il carico di test multiplo consentendo il test di più geni. Inoltre, filtrare i geni con troppo pochi inserimenti per allele contribuirà a ridurre l'impatto sui risultati dei test di ogni singolo clone di emizigote che ospita una mutazione del sito secondario che conferisce un fenotipo molto disparato. - Per ogni gene rimanente nel set di dati dopo il filtraggio di cui sopra, confrontare le termotolleranze (rapporto di log 2) di tutti gli inserimenti nell'allele S. cerevisiae con quelle nell'allele S. paradoxus utilizzando un test Mann-Whitney U. In alternativa, è possibile implementare un modello di regressione, adattato da DESeq28 (nome dello script: mann_whitney_u.py).
- Valori pcorretti per più test con il metodo Benjamini-Hochberg.
- I geni con valori psignificativi (ad esempio, 0,01) sono candidati per geni importanti per le differenze nella termotolleranza tra le due specie.
Risultati
Abbiamo accoppiato S. cerevisiae e S. paradoxus per formare un ibrido sterile, che abbiamo sottoposto alla mutagenesi della trasposonia. Ogni clone mutagenizzato era un emizygote, un ibrido diploide in cui un allele di un gene viene interrotto (Figura 1A, Figura 2). Abbiamo gareggiato gli emizigotes l'uno contro l'altro per crescita a 39 gradi centigradi e, in un esperimento separato come controllo, a 28 gradi centigradi (
Discussione
I vantaggi di RH-seq rispetto ai precedenti metodi statistico-genetici sono molteplici. A differenza del collegamento e dell'analisi delle associazioni, RH-seq offre la risoluzione della mappatura di un singolo gene; come tale, sarà probabilmente di notevole utilità anche negli studi di variazione dei tratti tra gli individui di una determinata specie, così come le differenze interspecifiche. Inoltre, i precedenti tentativi di analisi dell'emizigosità reciproca a livello di genoma hanno utilizzato raccolte di mutanti...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo J. Roop, R. Hackley, I. Grigoriev, A. Arkin e J. Skerker per il loro contributo allo studio originale, F. Al'aben, A. Flury, G. Geiselman, J. Hong, J. Kim, M. Maurer, e L. Oltrogge per l'assistenza tecnica, D. Savage per la sua generosità con la microscopia risorse, e B. Blackman, S. Coradetti, A. Flamholz, V. Guacci, D. Koshland, C. Nelson, e A. Sasikumar per le discussioni; ringraziamo anche J. Dueber (Dipartimento di Bioingegneria, UC Berkeley) per il plasmide PiggyBac. Questo lavoro è stato supportato da R01 GM120430-A1 e dal Community Sequencing Project 1460 a RBB presso il Department of Energy Joint Genome Institute degli Stati Uniti, un DOE Office of Science User Facility. Il lavoro condotto da quest'ultimo è stato sostenuto dall'Ufficio di Scienza del Dipartimento dell'Energia degli Stati Uniti sotto contratto n. DE-AC02-05CH11231.
Materiali
| Name | Company | Catalog Number | Comments |
| 1-2 plasmid Gigaprep kits | Zymo Research | D4204 | The number of kits required depends on how efficient your preps are in each kit. This kit comes with 5 individual plasmid prep columns. Run 1 L of saturated E. coli culture through each prep column, as using more than 1 L per column can cause clogging of the prep filter, leading to low yield and poor quality DNA. |
| 10X Tris-EDTA (TE) buffer (100 mM Tris-HCl and 10 mM EDTA) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 1M LiOAc | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 300 mg/mL Geneticin (G418) | Gibco | 11811023 | |
| 52% polyethylene glycol (PEG) 3350 | Sigma | 1546547 | Dissolve in water and filter sterilize through a 0.22 μm filter before use. 1X trafo mix: 228 uL 52% PEG, 36 uL 1M LiOAc, 36 uL 10X TE buffer |
| Autoclaved LB liquid broth | BD Difco | 244620 | Make LB liquid broth using your powder from any brand, and milliQ water. Autoclave it before use. |
| Carbenicillin stock in water (100 mg/mL) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| Complete synthetic agar plates (24.1cm x 24.1cm) with 5-fluoroorotic acid (5-FOA) [0.2% drop-out amino acid mix without uracil or yeast nitrogen base (YNB), 0.005% uracil , 2% D-glucose, 0.67% YNB without amino acids, 0.075% 5-FOA] | 5-FOA: Zymo Research, Drop-out mix: US Biological, Uracil: Sigma, D-glucose: Sigm), YNB: Difco | 5-FOA: F9001-5, Drop-out mix: D9535, Uracil: U0750, D-glucose: G8270, YNB: DF0919 | |
| DMSO | Any | N/A | |
| E. coli strain carrying pJR487 (CEN-/ARS+ piggyBac-containing plasmid) | N/A | N/A | Request from Brem lab. |
| Hybrid yeast strain JR507 (S. cerevisiae DBVPG1373 x S. paradoxus Z1, URA-/URA-) | N/A | N/A | Request from Brem lab. |
| Illumina Hiseq 2500 | used for SE-150 reads | ||
| Large shaking incubators with variable temperature settings | Any | N/A | |
| LB + carbenicillin agar plates (100 μg/mL) | Agar: BD Difco | Agar: 214010 | Make LB agar plates as normal and add carbenicillin to 100 μg/mL before drying. |
| Nanodrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit Fluorimeter | Thermo Scientific | Q33240 | |
| Salmon sperm DNA | Invitrogen | 15632011 | |
| Water bath at 39°C | Any | N/A | |
| Yeast fungal gDNA prep kit | Zymo Research | D6005 | |
| Yeast peptone dextrose (YPD) liquid media | BD Difco | Peptone: 211677, Yeast Extract: 212750 | Add filter-sterilized D-glucose to 2% after autoclaving. |
| YPD + G418 agar plates (300 μg/mL) | Agar: BD Difco | Agar: 214010 | Make YPD agar plates as normal and add G418 to 300 μg/mL before drying. |
| YPD agar plates | Agar: BD Difco | Agar: 214010 |
Riferimenti
- Flint, J., Mott, R. Finding the molecular basis of quantitative traits: successes and pitfalls. Nature Reviews Genetics. 2, 437-445 (2001).
- Allen Orr, H. The genetics of species differences. Trends in Ecology and Evolution. 16, 343-350 (2001).
- Weiss, C. V., et al. Genetic dissection of interspecific differences in yeast thermotolerance. Nature Genetics. 50, 1501-1504 (2018).
- Stern, D. L. Identification of loci that cause phenotypic variation in diverse species with the reciprocal hemizygosity test. Trends in Genetics. 30, 547-554 (2014).
- Steinmetz, L. M., et al. Dissecting the architecture of a quantitative trait locus in yeast. Nature. 416, 326-330 (2002).
- Scannell, D. R., et al. The Awesome Power of Yeast Evolutionary Genetics: New Genome Sequences and Strain Resources for the Saccharomyces sensu stricto Genus. G3 (Bethesda). 1, 11-25 (2011).
- Wetmore, K. M., et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons. MBio. 6, e00306-e00315 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15, 550 (2014).
- Wilkening, S., et al. An evaluation of high-throughput approaches to QTL mapping in Saccharomyces cerevisiae. Genetics. 196, 853-865 (2014).
- Kim, H. S., Huh, J., Riles, L., Reyes, A., Fay, J. C. A noncomplementation screen for quantitative trait alleles in saccharomyces cerevisiae. G3 (Bethesda). 2, 753-760 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon