Method Article
ثنائي الفينيل متعدد الكلور الرقمي القطيرات للكشف عن طفرات Indels في مجموعات البعوض Anopheline المعدلة وراثيا
In This Article
Summary
يوفر هذا البروتوكول الخطوات من استخراج الحمض النووي إلى الإعداد التجريبي ل PCR القطيرات الرقمية (ddPCR) ، بما في ذلك تحليل لتحديد وتحديد كمي لأحداث الانضمام النهائي غير المتجانسة (NHEJ) في المواقع المستهدفة بعد انشقاق Cas9 الناجم عن الحمض النووي الريبي وإصلاح الحمض النووي. وتشمل الاستخدامات الأخرى لهذه الطريقة تطبيقات مثل الكشف عن تعدد الأشكال والتحقق من متغير تحرير الجينات.
Abstract
وقد عززت التطورات الأخيرة في علم جينوم البعوض وتكنولوجيات الهندسة الوراثية الحاجة إلى أساليب سريعة وفعالة للكشف عن اختلاف تسلسل الحمض النووي المستهدف على نطاق واسع. على وجه التحديد ، والكشف عن عمليات الإدراج والحذف (indels) في المواقع التي تم تحريرها الجينات التي تم إنشاؤها بواسطة دليل CRISPR الجيش الملكي النيبالي (gRNA) / Cas9 بوساطة غير متجانسة نهاية الانضمام (NHEJ) مهم لتقييم إخلاص تولد وتواتر التغيرات غير المقصودة. نحن نصف هنا بروتوكولا ل PCR الرقمية قطرة (ddPCR) التي هي مناسبة تماما لتحليل NHEJ عالية الإنتاجية. وفي حين أن هذه الطريقة لا تنتج بيانات تحدد تباين التسلسل الفردي، فإنها توفر تقديرا كميا لتباين التسلسل داخل مجموعة سكانية. بالإضافة إلى ذلك، يمكن تنفيذ هذا البروتوكول، بموارد مناسبة، في موقع ميداني في المختبر بسهولة أكبر من الجيل التالي أو تسلسل سانجر. كما أن DDPCR لديها وقت أسرع للنتائج من أي من هاتين الطريقتين ، مما يسمح بإجراء تحليل أسرع وكامل للتباين الجيني في المجموعات البرية أثناء التجارب الميدانية للكائنات المعدلة وراثيا.
Introduction
محركات الجينات لديها إمكانات هائلة للسيطرة على مجموعات الحشرات ذات الصلة الطبية والزراعية1،2،3،4،5. على سبيل المثال، يمكن استخدام أنظمة الدفع الجيني المستندة إلى نواة CRISPR Cas وتوجيه الرناسات (gRNAs) لتعديل مجموعات البعوض الناقل عن طريق إدخال الصفات التي تمنح الانكسار لطفيليات الملاريا مما يؤدي إلى انخفاض انتقال العدوى وتقليل المرض1و4و5. ينسخ نظام محرك الجينات نفسه والسمة المرتبطة به من كروموسوم متجانس إلى آخر في الخلايا الجرثومية قبل الميوتيكية ، وهذا يضمن أن غالبية النسل يرثون الدافع ويخلقون إمكانية تعديل السكان على المدى الطويل والمستدام في هذا المجال. ومع ذلك ، فإن أحد عيوب الأساليب المستندة إلى Cas / gRNA هو إمكانية توليد طفرات الإدراج والحذف (indel) من خلال إصلاح الحمض النووي غير المتجانس في نهاية الصلة (NHEJ) ، مما يؤدي إلى توليد أليليس مقاومة للقيادة ، والتي عندما تتراكم إلى تردد عال بما فيه الكفاية في السكان ، يمكن أن توقف نظام القيادة من الانتشار1و2و 3و4 . يفصل هذا البروتوكول طريقة عالية الإنتاجية وموثوقة يمكنها تحديد انتشار وكمية نسبية من طفرات indel ، على مستوى السكان والأفراد على حد سواء ، أثناء محرك الجينات القائم على Cas / gRNA.
توفر أساليب تسلسل الجيل التالي (NGS) دقة تسلسل لا مثيل لها. ومع ذلك، فإن التكلفة والمتطلبات التقنية المرتبطة NGS تحظر الاختبار الروتيني والحد من استخدامه كوسيلة عالية الإنتاجية لتقييم indels6،7،8. وقد استخدمت منذ فترة طويلة أساليب القياس الكمي التقليدية ل PCR كإجراء تقييم قياسي ل indels الجينوم؛ ومع ذلك، هذه الطرق كثيفة العمالة، تستغرق وقتا طويلا لشراء البيانات، ولها درجة عالية من التباين. وقد ثبت الرقمية قطرة PCR (ddPCR) لتكون أكثر حساسية في الكشف عن الطفرات من تسلسل سانجر في بعض التطبيقات ولها حد الكشف أقل من NGS في الآخرين6،7،8،9. وعلاوة على ذلك، فإن تكلفة تقييم مجموعة عينة ووقت الدوران للحصول على النتائج أقل تكلفة وأسرع، على التوالي، بالنسبة ل ddPCR من تسلسل سانجر أو NGS9. باستخدام نظام التحقيق المزدوج، يحدد المقايسة المنسدلة أليلات NHEJ استنادا إلى عدم وجود تسلسل من النوع البري (WT) في موقع قطع Cas9 المستهدف الموجه من GRNA. في هذا المقايسة، يتم تضخيم أمبليكون قصيرة بما في ذلك موقع القطع المتوقع للنظام القائم على Cas/gRNA مع زوج التمهيدي محددة. تم تصميم مسبار فلوري واحد لربط منطقة محفوظة من أمبيرليكون ومسبار فلوري آخر يتعرف على تسلسل WT لموقع القطع. في وجود أليل NHEJ ، فإن هذا الأخير لن يرتبط بالتضخيم.
يوفر استخدام ddPCR القدرة على تصميم التمهيديات لاستهداف عمليات الحذف والاختلافات الفردية بين الزوج الأساسي والإدراج ، مما سيسمح بتصنيف NHEJ في تحليلات مجموعات البعوض9. وبالنظر إلى هذه الميزات الجذابة ، أنشأنا بروتوكولا ل ddPCR للكشف عن الإنتاجية العالية للإندلات المتولدة من نظام محرك الجينات القائم على Cas / gRNA في البعوض.
Protocol
1. استخراج الحمض النووي
- إعداد EDTA/ Nuclei Lysis Buffer (EDTA/NLS) بنسبة 500 ميكرولتر من NLS و 120 ميكرولتر من EDTA لكل عينة. توسيع نطاق لعينات متعددة. يبرد الخليط على الثلج.
ملاحظة: سوف يتحول الحل غائما في 2-5 دقيقة عند تبريده اعتمادا على مستوى الصوت. - تجانس عينة البعوض باستخدام متجانس ميكانيكي لمدة 10-15 ثانية في أنبوب طرد مركزي صغير سعة 1.5 مل مليء ب 600 ميكرولتر من EDTA/NLS المبردة؛ مزيج تماما.
- إضافة 17.5 ميكرولتر من 20 ملغم / مل من بروتيناز K إلى الأنبوب وتخلط جيدا.
- حضانة بين عشية وضحاها في 55 °C. بالتناوب، احتضان العينة في 55 درجة مئوية لمدة 3 ساعة مع اهتزاز ودوامة العينة كل 1 ساعة.
- إضافة 200 ميكرولتر من البروتين هطول الأمطار الحل لعينة درجة حرارة الغرفة ودوامة بقوة لمدة 20 ثانية.
- قم بتبريد العينة لمدة 5 دقائق على الجليد.
- طرد مركزي العينة إلى البروتينات بيليه في 15,890 RCF لمدة 4 دقائق.
- يستنشق بعناية الفائقة التي تحتوي على الحمض النووي ونقله إلى أنبوب طرد دقيق نظيف 1.5 مل تحتوي على 600 ميكرولتر من ايزوبروبانول.
- مزيج بلطف الحل عن طريق عكس أنبوب 5-10 مرات. جهاز طرد مركزي لمدة دقيقة واحدة عند 15,890 RCF. decant بعناية supernatant مع الحفاظ على بيليه الحمض النووي.
- إضافة 600 ميكرولتر من درجة حرارة الغرفة 70٪ الإيثانول. غسل الحمض النووي بيليه عن طريق عكس بلطف الأنبوب.
- جهاز طرد مركزي لمدة دقيقة واحدة عند 15,890 RCF. إزالة بعناية supernatant عن طريق الطموح باستخدام طرف ماصة الزجاج.
- عكس الأنبوب على ورق ماصة نظيفة والهواء الجاف بيليه لمدة 10-15 دقيقة.
- إعادة إنفاق الحمض النووي مع الماء PCR الصف. استخدم 20 ميكرولتر لكل عينة بعوضة فردية أو 100 ميكرولتر ل 10 بعوضة مجمعة.
ملاحظة: يتم تكييف طرق استخراج الحمض النووي لعينات البعوض باستخدام مجموعة متاحة تجاريا (انظر جدول المواد)من الحمض النووي الجينومي المعزول للشركة المصنعة من خلايا زراعة الأنسجة وبروتوكول الأنسجة الحيوانية.
2. ردود فعل ddPCR وإعداد جيل القطيرات
- قياس الحمض النووي باستخدام مقياس الفلور.
ملاحظة: بالنسبة إلى المقايسة المنسدلة، يوصى باستخدام نطاق يتراوح بين 3000 و30000 نسخة من الجينوم haploid لكل تفاعل، والذي تم تصميمه للكشف عن أحداث NHEJ باستخدام مسبار وضع العلامات HEX الذي يرتبط بتسلسل WT لموقع القطع المستهدف ولن يكون معرفا (إسقاط) إذا كان هناك حذف أو إدراج في الموقع المستهدف، تشير إلى وجود متغير NHEJ. - حساب رقم النسخ باستخدام وزن الجينوم haploid وتركيز الحمض النووي في استخراج. ويتم ذلك عن طريق ضرب تركيز الحمض النووي المستخرج بالحجم المستخدم للحصول على كتلة الحمض النووي الإجمالية، ثم تقسيمه على وزن الجينوم haploid. تأكد من أن حجم المضافة بين 1-10 ميكرولتر. تمييع حسب الضرورة لتكون في نطاق نسخ الجينوم haploid الموصى بها.
ملاحظة: يقدر واحد Anopheles غامبيا haploid الجينوم أن يكون 0.27 pg لكل بعوضة الكبار10. - تصميم التمهيديات والتحقيقات. تصميم إلى الأمام وعكس الأوليغونوكليوتيد التمهيدي مع درجة حرارة ذوبان التمهيدي (TM) في نطاق 55-60 درجة مئوية التي تحيط 5'- و 3'ends من موقع الهدف GRNA إنتاج أمبيرليكون من 150-400 نقطة أساس.
- HEX (هيكساكلورو-فلوريسين) المسمى التحقيق للكشف NHEJ: تصميم أوليغونوكليوتيد من ~ 15-20 نقطة أساس في طول مكمل للموقع المستهدف وإضافة HEX-التحقيق إلى 5'end وBHQ1 (BLack هول كوينشر 1) إلى نهاية 3. يجب أن يكون TM من مسبار التحلل المائي أعلى من TM من التمهيديات ب 3-10 درجة مئوية.
- FAM (6-carboxyfluorescein) المسمى التحقيق للإشارة WT: تصميم oligonucleotide ~ 15-20 نقطة أساس في طول مكملة لموقع الجينوم المحفوظة البعيدة (حوالي 25 نقطة أساس) من الموقع المستهدف وإضافة FAM-التحقيق إلى 5'end وBHQ1 إلى نهاية 3. يجب أن يكون TM من مسبار التحلل المائي أعلى من TM من التمهيديات ب 3-10 درجة مئوية.
3. PCR إعداد رد فعل
- إعداد 25 ميكرولتر من مزيج عينات ddPCR مع المكونات التالية: ddPCR supermix للمسابير (لا UTP): 12.5 ميكرولتر، التمهيديات الأمامية والعكسية (10 ميكرومتر): 1 ميكرولتر لكل منهما، مسابير HEX/FAM (10 ميكرومتر): 0.625 ميكرولتر لكل منها، الحمض النووي: 1-5 ميكرولتر (3750-37500 نسخة جينوم هابلويد) والماء: ما يصل إلى 25 ميكرولتر.
- مزيج تماما ردود الفعل عن طريق دوامة أو ارتجاع الأنابيب (صعودا وهبوطا) (20x).
ملاحظة: إذا كانت ردود الفعل في لوحة 96-جيدا، ماصة وحدة التخزين بأكملها صعودا وهبوطا 20 مرة بدلا من دوامة لتجنب تشكيل فقاعة. - طرد مركزي لفترة وجيزة العينات لتسوية الخليط في الجزء السفلي من الأنبوب أو جيدا.
ملاحظة: تأكد من أن ردود الفعل في درجة حرارة الغرفة لتوليد القطيرات. إعداد 1x من مزيج ddPCR للنواب الإضافية / غير المستخدمة في كل خرطوشة (تحتوي كل خرطوشة على 8 آبار).
4. جيل القطيرات
- باستخدام ماصة متعددة القنوات 50 ميكرولتر، قم بتحميل 20 ميكرولتر من مزيج عينة ddPCR في الصف الأوسط من الخرطوشة(الشكل 1A، أعلى).
- تحميل 70 ميكرولتر من النفط في الصف السفلي. تحميل 20 ميكرولتر من 1x ddPCR supermix في الآبار غير المستخدمة.
ملاحظة: لا تقدم فقاعات. - ضع طوقا لمس الحواف فقط، وتجنب منطقة مركز مقعر(الشكل 1B).
- ضع اللوحة بشكل آمن في مولد القطيرات واغلق الغطاء لبدء التشغيل.
- باستخدام ماصة متعددة القنوات، نقل 40 ميكرولتر من مزيج emersion من الصف العلوي من خرطوشة(الشكل 1A،أسفل) إلى لوحة 96 جيدا.
- رسم عينة السائل لمدة 3-5 ق في زاوية 45-30 درجة. طرد الخليط ببطء لأكثر من 3 ق في زاوية 45 درجة في جانب البئر، مما يسمح لها بالتنقيط أسفل الجانب. لا بأس أن تذهب إلى المحطة الثانية (الطرد الكامل) من ماصة لطرد جميع السائل.
- باستخدام الأختام الحرارة احباط، ختم لوحات لمدة 5 ق في 180 درجة مئوية.
5. PCR
- ضع اللوحة المختومة في الترموسيكلر(الشكل 1C)وحدد شروط PCR الموصى بها إذا اتبعت إرشادات NHEJ المنسدلة على النحو التالي:
- التشبع الأولي عند 95 درجة مئوية لمدة 10 دقائق.
- تعيين 40 دورات من 94 درجة مئوية لمدة 30 ق إلى denature، 55 درجة مئوية لمدة 1 دقيقة إلى آنال، و 60 درجة مئوية لمدة 2 دقيقة لتمديد.
- اضغط على 98 درجة مئوية لمدة 10 دقائق.
- عقد في 4 °C.
ملاحظة: قد يتم تحسين درجة حرارة التلين للتمهيدي محددة ومجموعات التحقيق باستخدام التدرج الحراري. استخدم معدل منحدر قدره درجتين مئويتين/ثانية لجميع الخطوات. وينبغي تعديل شروط PCR اعتمادا على كل تصميم تجريبي وإعداد.
6. قراءة القطيرات
- ضع اللوحة بشكل آمن في قارئ القطرات مع A-1 جيدا في أعلى اليسار (الزاوية الممهدة ، والثلاثة الآخرون محافة)(الشكل 1D).
- إعداد لوحة في البرنامج: تعيين FAM كقناة مرجعية معروفة و HEX باعتبارها واحدة غير معروفة(الشكل 2A).
- تشغيل تجربة القراءة قطرة كما القياس الكمي المباشر. بعد انتهاء التشغيل، قم بتغيير نوع التجربة إلى Drop-Off (DOF) للتحليل (الشكل 2A).
7. تحليل
- تعيين المعلمات التجريبية الصحيحة(الشكل 2A): معلومات العينة، SuperMix، اسم الهدف (WT أو NHEJ)، نوع الهدف (المرجع أو غير معروف)، إشارة Ch1 (HEX أو FAM)، إشارة Ch2 (HEX أو FAM)، وتعيين يدويا عتبة لعدد قطرات (يوصي أعلاه 10،000 للحصول على نتائج موثوق بها). سيقوم البرنامج بإجراء معظم التحليل باستخدام المعلمة المعينة.
- تحقق من عدد القطرات في علامة التبويب قطرة؛ التأكد من أن جميع فوق 10،000 (الشكل 2B).
- تحقق من سعة 1 D لفصل الإشارات الفعال عن السلبيات.
- في محرر لوحة، تسليط الضوء على لوحة كاملة وتعيين نوع التجربة لإسقاط.
- تعيين هدف WT كمرجع، تعيين قناة واحدة ل FAM والقناة الثانية ل HEX.
- تعيين NHEJ الهدف كما غير معروف، تعيين القناة الأولى لFAM والقناة الثانية كما لا شيء.
- في علامة التبويب السعة 2D تعيين "عتبات الكتلة" باستخدام أدوات الرسم البياني لكل عينة. النظر في الذيل المقترنة الكتلة WT; وهذا أمر طبيعي لNHEJ المقايسات (الشكل 3A).
ملاحظة: ضمن علامة التبويب نسبة، انقر فوق رمز الترس من أعلى يمين الرسم البياني. حدد وفرة الكسور. الرسم البياني الآن رسم نقطة المقابلة للنسبة المئوية للأحداث NHEJ (الشكل 3B).
النتائج
ويظهر تطبيق هذا الإجراء في كاربالار - ليخارازووآخرون. يستخدم المقايسة المنسدلة ddPCR مسبارين فلوريين لتمييز تسلسل WT و indel: يرتبط مسبار FAM بتسلسل محفوظ داخل amplicon ، في حين يستهدف مسبار HEX تسلسل WT للموقع المستهدف(الشكل 4A). في وجود indel ، فإن التحقيق HEX لا ربط. ويمكن الاطلاع على النتائج التمثيلية في الشكل 2والجدول 1 والجدول 2 من كاربالار - ليخارازو وآخرون. باستخدام هذا البروتوكول، وقد ثبت ddPCR للكشف عن مجموعة واسعة من الأحداث NHEJ CRISPR-Cas9 المستحثة وتحديد تردد NHEJ في عينة فردية أو مجمعة. 15 عينة مجمعة مختلفة من 10 البعوض كل تحتوي على مختلف الأليل NHEJ (الجدول 2 من كاربالار-Lejarazúوآخرون. تم تحليل هذه مع ddPCR باستخدام البروتوكول والمعلمات المعروضة هنا. وتبين نتائج الجدول 19 أن جميع العينات ال 15 حملت أليلات إنديل بنسبة 100 في المائة على النحو المحدد في المقايسة المنسدلة(الشكل 4ب). وفي تجربة أخرى، تم فحص 11 عينة مجمعة من بعوض WT وبعوض NHEJ بنسب مئوية مختلفة من NHEJ (0٪، 10٪، 20٪، 30٪، 40٪، 50٪، 60٪، 70٪، 80٪، 90٪، و 100٪) مع هذا البروتوكول ddPCR، والنتائج(الشكل 2؛ Carballar- Lejarazú وآخرون9) أظهرت أن النسبة المئوية المحددة قريبة من تقنية مقارنة Indel Detection بواسطة تحليل Amplicon(الشكل 4C).

الشكل 1:الإعداد التجريبي والإجراءات. (أ) إعداد خرطوشة لتوليد القطيرات. (أعلى) يتم تعبئة العينات في الصف الأوسط من الخرطوشة، بينما يتم تعبئة الزيت في الصف السفلي. (أسفل) الصف العلوي مليئة قطرات مستحلب بعد جيل قطرة. (ب) مولد قطرة مع خرطوشة مليئة عينة وتغطيها طوقا في مكان. (ج) لوحة 96 جيدا مغطاة ختم احباط في دورة الحرارية. (D) قارئ قطرات مع 96-جيدا لوحة في مكان مع غطاء معدني ملتصق على لوحة لتأمينه. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
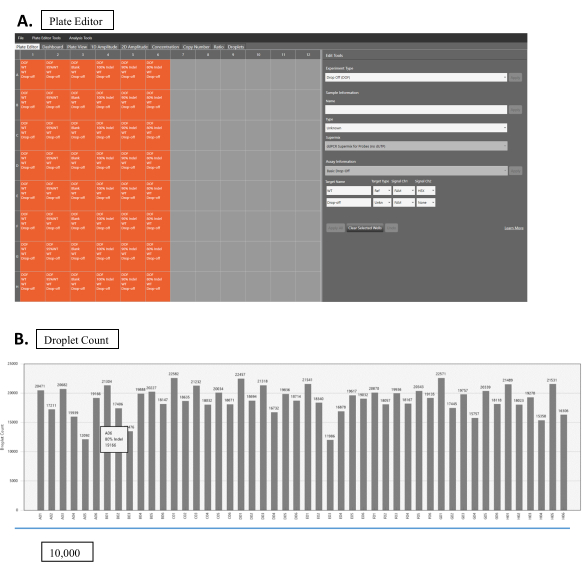
الشكل 2: قراءة القطيرات. (أ) واجهة البرمجيات لقراءة القطيرات. تظهر الصناديق البرتقالية آبارا مع عينات. الصناديق الرمادية هي آبار فارغة. يتم إعداد المعلمات التجريبية في لوحة أدوات التحرير (الجانب الأيمن). يمكن تحرير كل عينة من خلال النقر على مربع العينة المعني. حدد إسقاط (DOF) لنوع تجريبي. في نموذج المعلومات، قم بتعبئة المعلومات المناسبة الخاصة باسم العينة ونوعها،بالإضافة إلى SuperMix. اختر الإفلات الأساسي لمعلومات الفحص. لعينة WT، اختر WT للاسم الهدف، المرجع لنوع الهدف، و FAM و HEX لكل من الإشارة Ch1 و Ch2، على التوالي. للحصول على عينات NHEJ، قم بتعبئة الاسم المناسب للاسم الهدف، واختر غير معروف للنوع الهدف، واختر FAM للإشارة Ch1. اترك الإشارة Ch2 في لا شيء. (B) نتائج العد القطيرات لعينات متعددة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
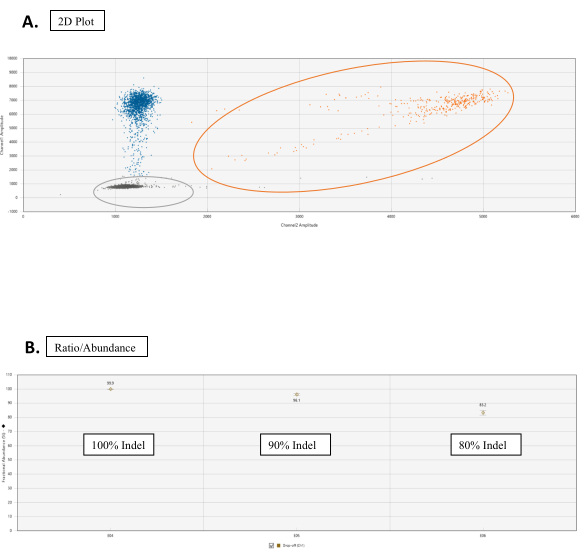
الشكل 3:تحليل المقايسة المنسدلة. (أ) مؤامرة 2D الكتلة لعدد قطرات من الأليل WT وNHEJ. في علامة التبويب السعة 2D، يتم إلغاء تصنيف جميع القطرات بشكل افتراضي. في هذا الشكل، يتم تعيين الألوان يدويا للتمييز. الكتلة النقاط البرتقالية هي WT allele التهم التي تم الحصول عليها عن طريق ربط كل من FAM و HEX تحقيقات في تسلسل المرجع وتسلسل الموقع الهدف، على التوالي. تمثل النقاط الزرقاء عشرات القطرات التي لها ربط FAM بالتسلسل المرجعي ولكن لا يوجد ربط HEX في تسلسل الموقع المستهدف (وبالتالي إسقاط HEX). النقاط الرمادية هي قطرات فارغة لا تحتوي على ربط FAM أو HEX. (ب) الرسوم البيانية النسبة / وفرة من الأحداث NHEJ. ضمن علامة التبويب نسبة، حدد وفرة الكسور لرسم بياني مع النسبة المئوية لمراسل أحداث NHEJ. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
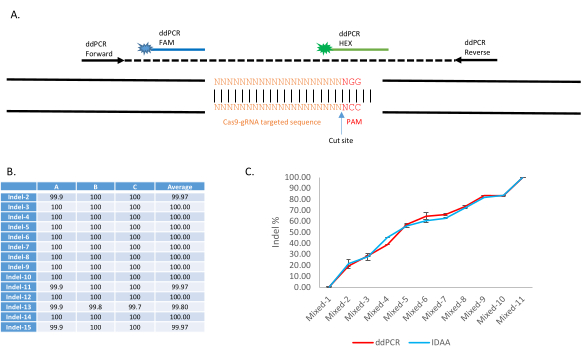
الشكل 4:تطبيق المقايسة المنسدلة مع ddPCR لتحديد الهوية غير المتجانسة في نهاية الصلة وتحديد كمي في خط Anopheles stephensi المعدلة وراثيا، AsMCRkh1. (أ) عرض تخطيطي لمقايسة الإنزال ddPCR للكشف عن الطفرات في موقع الحمض النووي المستهدف مع نظام مزدوج التحقيق. تضخيم amplicon من 150-400 نقطة أساس مع التمهيديات إلى الأمام وعكس. تم تصميم مسبار يحمل علامة FAM لربط تسلسل محفوظ من amplicon ، في حين تم تصميم مسبار يحمل علامة HEX لربط الموقع المستهدف ب WT gRNA. (ب) الكشف عن أنواع مختلفة من indels مع ddPCR. تم تحليل 15 بركة من 10 بعوض AsMCRkh1 تحتوي كل منها على أنواع مختلفة من ال indel ، بما في ذلك الإدراج والحذف والتبديل ، مع فحص قطرة ddPCR. ويمكن الاطلاع على تفاصيل الطفرات والتسلسلات في الجدول 2 والجدول S3 من كاربالار وآخرون. 9.(ج) تحديد كمي من NHEJ في عينات مختلطة من البعوض AsMCRkh1 وWT مع نسب مختلفة (10:0، 9:1، 8:2، 7:3، 6:4، 5:5، 4:6، 3:7، 2:8، 1:9، و 0:10) باستخدام ddPCR وتقنية مقارنة من الكشف عن Indel بواسطة تحليل أمبليكون9. صور مقتبسة من كاربالار ليجارازو وآخرون Biotechniques. 68(4):172-179 (2020)9. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
| التمهيدي / التحقيق | تسلسل (5' » 3') |
| ddPCR التمهيدي الأمامي | أتغاتكاتجتكاكاتك |
| ddPCR التمهيدي العكسي | أكجتاكتغغاكا |
| ddPCR هيكس التحقيق (BHQ1) | [HEX]-TTCTACGGGCAGGGC-[BHQ1] |
| ddPCR FAM التحقيق (BHQ1) | [6FAM]-CCACGTGGATCGAAGG-[BHQ1] |
| HEX: هيكساكلورو-فلوريسين، فام: 6-كاربوكسيفلورسين، BHQ: الثقب الأسود كوينشر | |
الجدول 1: تسلسلات التهيئة والتحقيقات.
Discussion
الرقمية قطرة PCR هو وسيلة فعالة لتحديد وجود أليليس indel الناتجة عن الأحداث NHEJ في نظام محرك الجينات المستندة إلى Cas / gRNA ويسمح للقياس الكمي لتواتر هذه الأليل في الأفراد أو السكان. وينبغي اتباع بعض الخطوات الواردة في البروتوكول بعناية خاصة لتحقيق نتائج موثوقة. أولا، يجب إجراء استخراج الحمض النووي الجينومي بعناية لضمان جودة عالية وكمية كافية. واستخراج جيدة تسمح بتحديد دقيق لنسخ الجينوم haploid لكل رد فعل. في تجربتنا، قدمت مجموعة متاحة تجاريا (انظر جدول المواد)باستمرار استخراج الحمض النووي عالية الجودة. ومع ذلك ، يمكن أن يثبت استخراج البعوض الفردي أنه صعب بشكل خاص حيث يصبح من الصعب تصور بيليه الحمض النووي ويمكن امتصاصه بسهولة مع البعوض الخارق إن لم يكن حذرا. ثانيا، يجب تصميم التهيئة والتحقيقات بعناية. قبل الانتهاء من تجربة ddPCR ، تأكد من أن التمهيديات المصممة تؤدي إلى منتج PCR واحد من خلال إجراء أول PCR التقليدية وتصور منتج واحد عن طريق الكهربائي هلام. يجب أيضا تصميم مسبار FAM المرجعي بحيث يكون مكملا لتسلسل محفوظ للغاية. وهذا من شأنه أن يضمن الكشف الدقيق عن أليليس WT في جميع أنحاء مجموعة متنوعة من السكان. سيكون لتركيبات التمهيدي/المسبار لكل تجربة فريدة ظروف مختلفة لدراجة حرارية، ويوصى بتحسين هذه الظروف باستخدام تدرج حراري.
توجد طرق أخرى لتحديد indels، مثل تسلسل سانجر أو NGS. تسلسل سانجر محدود لأنه يحتوي على حد أدنى للكشف وقوة اكتشاف منخفضة لتحديد المتغيرات الجديدة. تسلسل سانجر هو أيضا كثيفة العمالة وليس عالية الإنتاجية. بالمقارنة مع تسلسل سانجر، لا تملك NGS نفس القيود المفروضة على الحساسية المنخفضة، وقوة الاكتشاف، والإنتاجية. فائدة أخرى من NGS هو قدرته على الكشف عن مجموعة متنوعة من الطفرات من تعدد الأشكال النيوكليوتيدية واحدة (SNPs) لإعادة ترتيب. ومع ذلك، فإن نظام المعلومات غير الحكومية هو طريقة أكثر تكلفة وتستغرق وقتا طويلا في تطبيق تحديد ال indels المرتبطة ب Cas9/gRNA لأنه لا يوجد سوى منطقة مستهدفة واحدة ذات أهمية، وهي الأنسب للتحليلات الأكبر على نطاق الجينوم. بالمقارنة مع الأساليب المذكورة أعلاه ، ddPCR هو الإنتاجية العالية ولها وقت سريع بدوره حولها. إذا كانت مواد وأدوات ddPCR متوفرة داخليا ، يمكن معالجة 96 عينة في غضون 1-2 يوم ، مما يجعلها مناسبة تماما للتحليل السريع للتجارب الكبيرة للكائنات المعدلة Cas9 /gRNA.
في حين أن العديد من الفوائد موجودة لddPCR هناك أيضا قيود. أولا، معدات ddPCR غير متوفرة في كثير من الأحيان في بيئة مختبرية مستقلة. قد تكون معدات ddPCR متاحة بشكل جماعي في مؤسسات البحث الأكبر ، ولكن هذا لا يسمح بسهولة توليد البيانات وتحليلها خارج المؤسسة. ثانيا، على عكس البدائل، لا يوفر ddPCR التسلسلات الفريدة الفردية لطفرات indel المحددة. وسيوفر ثنائي الفينيل متعدد الكلور الرقمي القطرة تكرار طفرات إنديل داخل السكان، ولكن بدون التسلسل، لا يمكن للمرء أن يحدد ما إذا كان من المرجح أن تحافظ الهندباء الموجودة على وظيفة الجين ذات الاهتمام أو تمنعها. ولعل طريقة ddPCR هي الأنسب لتحليل المجموعات البرية بعد تجربة إطلاق حقل لكائن محرك أقراص يستند إلى Cas9/gRNA لأنه يمكن أن يحدد بكفاءة وتيرة إدخال المتحول إلى السكان الأصليين وتوليد indels داخل السكان في وقت قريب من الوقت الحقيقي. ونظرا لسرعة وقت التحول في تقرير التنمية المستدامة، سيكون من الممكن إجراء أخذ العينات وتحليل السكان في منطقة التجارب الميدانية أسبوعيا إذا كانت المواد متاحة محليا. وستكون تكاليف بدء شراء واستيراد وإعداد معدات DDPCR مرتفعة في المختبرات النائية ولكن فوائد القدرة على تقييم السكان البريين بدقة لأنها تخضع للتعديل من نظام القيادة تبرر التكاليف.
Disclosures
ولا يوجد لدى صاحبي البلاغ أي إفصاحات.
Acknowledgements
وقدمت مبادرة جامعة كاليفورنيا إيرفين لمكافحة الملاريا التمويل. AAJ هو أستاذ دونالد برين في جامعة كاليفورنيا، إيرفين.
Materials
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| ddPCR Super Mix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DNA extraction reagent (e.g. Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| EDTA (pH 8.0) | Invitrogen | AM9260G | |
| Ethanol, 200 Proof | Thermo Fisher Scientific | A4094 | |
| Isopropanol (Certified ACS) | Thermo Fisher Scientific | A416-500 | |
| Nuclei Lysis Solution (NLS) (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| PCR-grade Water | Any certified PCR-grade water can be used | ||
| Protein Precipitation Solution (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| Proteinase K 20 mg/mL | Thermo Fisher Scientific | AM2546 | |
| Materials | |||
| ddPCR 96-Well Plate | Bio-Rad | 12001925 | |
| Droplet Generator DG8 Cartridge and Gaskets | Bio-Rad | 1864007 | |
| Droplet Generation Oil for probes | Bio-Rad | 1863005 | |
| Fluorescent probes (e.g. FAM/HEX probes) | Sigma-Aldrich | N/A | Probes are experiment specific and can be purchased from any certified seller available. |
| Forward and Reverse oligonucleotide primers | Sigma-Aldrich | N/A | Primers are experiment specific and can be purchased from any certified seller available. |
| Equipment | |||
| C1000 Touch Thermal Cycler | Bio-Rad | 1851148 | Can use other Thermo cycler with gradient function and deep well |
References
- Gantz, V. M., et al. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. Proceedings of the National Academy of Sciences of the United States of America. 112 (49), 6736-6743 (2015).
- Hammond, A., et al. A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae. Nature Biotechnology. 34, 78-83 (2016).
- Courtier-Orgogozo, V., Morizot, B., Boëte, C. Agricultural pest control with CRISPR-based gene drive: time for public debate: Should we use gene drive for pest control. EMBO Reports. 18 (6), 878-880 (2017).
- Carballar-Lejarazú, R., et al. Next-generation gene drive for population modification of the malaria vector mosquito, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 117 (37), 22805-22814 (2020).
- Adolfi, A., et al. Efficient population modification gene-drive rescue system in the malaria mosquito Anopheles stephensi. Nature Communications. 11, 5553 (2020).
- Wang, Z., et al. Comparison of droplet digital PCR and direct Sanger sequencing for the detection of the BRAFV600E mutation in papillary thyroid carcinoma. Journal of Clinical Laboratory Analysis. 33 (6), 22902 (2019).
- Dong, L., Wang, S., Fu, B., Wang, J. Evaluation of droplet digital PCR and next generation sequencing for characterizing DNA reference material for KRAS mutation detection. Scientific Reports. 8 (1), 9650 (2018).
- Bell, C. C., Magor, G. W., Gillinder, K. R., Perkins, A. C. A high-throughput screening strategy for detecting CRISPR_Cas9 induced mutations using next-generation sequencing. BMC Genomics. 15 (1), 1002 (2014).
- Carballar-Lejarazú, R., Kelsey, A., Pham, T. B., Bennett, E. P., James, A. A. Digital droplet PCR and IDAA for the detection of CRISPR indel edits in the malaria species Anopheles stephensi. Biotechniques. 68 (4), 172-179 (2020).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298 (5591), 129-149 (2002).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved