Method Article
Цифровая капельная ПЦР для выявления мутаций Indels в генетически модифицированных популяциях анофелиновых комаров
В этой статье
Резюме
Этот протокол обеспечивает этапы от экстракции ДНК до экспериментальной установки для цифровой капельной ПЦР (ddPCR), включая анализ для идентификации и количественной оценки негомологичных событий конечного соединения (NHEJ) на целевых участках после гРНК-индуцированного расщепления Cas9 и репарации ДНК. Другие применения этого метода включают такие приложения, как обнаружение полиморфизма и проверка вариантов редактирования генов.
Аннотация
Недавние достижения в области геномики комаров и технологий генной инженерии способствовали необходимости в быстрых и эффективных методах обнаружения целевых вариаций последовательности ДНК в больших масштабах. В частности, обнаружение вставок и делеций (indels) в генетически отредактированных сайтах, генерируемых направляющей РНК (гРНК) CRISPR / Cas9-опосредованной негомологичным конечным соединением (NHEJ), важно для оценки точности мутагенеза и частоты непреднамеренных изменений. Здесь мы описываем протокол для цифровой капельной ПЦР (ddPCR), который хорошо подходит для высокопроизводительного анализа NHEJ. Хотя этот метод не дает данных, которые идентифицируют отдельные вариации последовательности, он обеспечивает количественную оценку вариации последовательности в популяции. Кроме того, при наличии соответствующих ресурсов этот протокол может быть реализован в лабораторных условиях на полевой площадке легче, чем секвенирование следующего поколения или секвенирование Сэнгера. ddPCR также имеет более быстрое время обработки результатов, чем любой из этих методов, что позволяет более быстро и полно анализировать генетические вариации в диких популяциях во время полевых испытаний генетически модифицированных организмов.
Введение
Генные драйвы обладают огромным потенциалом для контроля популяций насекомых медицинского и сельскохозяйственного значения1,2,3,4,5. Например, системы генного драйва на основе нуклеаз CRISPR Cas и направляющих РНК (гРНК) могут быть использованы для модификации популяций комаров-переносчиков путем введения признаков, которые придают огнеупорность малярийным паразитам, что приводит к снижению передачи и снижению заболеваемости1,4,5. Система генного драйва копирует себя и связанный с ней признак из одной гомологичной хромосомы в другую в премейотических половых клетках, и это гарантирует, что большинство потомства наследуют драйв и создают потенциал для длительной и устойчивой модификации популяции в полевых условиях. Однако одним из недостатков методов на основе Cas/gRNA является возможность генерации мутаций вставки и делеции (indel) посредством негомологичного восстановления ДНК с конечным соединением (NHEJ), что приводит к генерации резистентных к приводу аллелей, которые при накоплении достаточно высокой частоты в популяции могут остановить распространение системы привода1,2,3,4. . Этот протокол детализирует высокопроизводительный и надежный метод, который может определить распространенность и относительное количество мутаций indel, как на популяционном, так и на индивидуальном уровне, во время генного драйва на основе Cas / gRNA.
Методы секвенирования следующего поколения (NGS) обеспечивают беспрецедентное разрешение секвенирования. Однако стоимость и технические требования, связанные с NGS, запрещают рутинные испытания и ограничивают его использование в качестве высокопроизводительного метода оценки инделей6,7,8. Традиционные методы количественной оценки ПЦР уже давно используются в качестве стандартной процедуры оценки инделей генома; однако эти методы являются трудоемкими, требуют длительного времени для получения данных и имеют высокую степень изменчивости. Было доказано, что цифровая капельная ПЦР (ddPCR) более чувствительна при обнаружении мутаций, чем секвенирование Сэнгера в некоторых приложениях, и имеет более низкий предел обнаружения, чем NGS в других6,7,8,9. Кроме того, затраты на оценку набора образцов и время обработки для получения результатов дешевле и быстрее, соответственно, для ddPCR, чем секвенирование Сэнгера или NGS9. Используя систему с двумя зондами, анализ Drop-Off идентифицирует аллели NHEJ на основе отсутствия последовательности дикого типа (WT) в месте разреза мишени Cas9, направленной на гРНК. В этом анализе короткий ампликон, включающий предсказанный участок разреза системы на основе Cas/gRNA, усиливается определенной парой праймеров. Один флуоресцентный зонд предназначен для связывания с сохраненной областью ампликона, а другой флуоресцентный зонд распознает последовательность WT участка разреза. При наличии аллеля NHEJ последний не будет связываться с ампликоном.
Использование ddPCR обеспечивает возможность проектирования праймеров для целевых удалений, различий между одной парой оснований и вставок, что позволит профилировать NHEJ в анализах популяции комаров9. Учитывая эти привлекательные особенности, мы создали протокол для ddPCR для высокопроизводительного обнаружения инделей, генерируемых системой генного драйва на основе Cas/gRNA у комаров.
протокол
1. Извлечение ДНК
- Получают буфер лизиса ЭДТА/ядер (ЭДТА/НЛС) с соотношением 500 мкл NLS и 120 мкл ЭДТА на образец. Масштабирование для нескольких образцов. Охладить смесь на льду.
ПРИМЕЧАНИЕ: Раствор помутнеет через 2-5 минут при охлаждении в зависимости от объема. - Гомогенизировать образец комара с помощью механического гомогенизатора в течение 10-15 с в микроцентрифужной трубке объемом 1,5 мл, заполненной 600 мкл охлажденного ЭДТА/НЛС; тщательно перемешать.
- Добавьте в пробирку 17,5 мкл 20 мг/мл протеиназы К и тщательно перемешайте.
- Инкубировать в течение ночи при 55 °C. Поочередно инкубируют образец при 55°С в течение 3 ч с встряхиванием и вихрем образца каждые 1 ч.
- Добавьте 200 мкл раствора для осаждения белка в образец комнатной температуры и энергично вихрьте в течение 20 с.
- Охладите образец в течение 5 минут на льду.
- Центрифугируйте образец до белков гранул при 15 890 RCF в течение 4 мин.
- Осторожно аспирируйте супернатант, который содержит ДНК, и перенесите его в чистую микроцентрифужную трубку объемом 1,5 мл, содержащую 600 мкл изопропанола.
- Аккуратно перемешайте раствор, перевернув трубку 5-10 раз. Центрифуга в течение 1 мин при 15 890 RCF. Тщательно декантируйте супернатант, сохраняя гранулу ДНК.
- Добавьте 600 мкл 70% этанола комнатной температуры. Промыть гранулированную ДНК, аккуратно перевернув трубку.
- Центрифуга в течение 1 мин при 15 890 RCF. Осторожно удалите супернатант аспирацией с помощью стеклянного наконечника пипетки.
- Переверните трубку на чистую абсорбирующую бумагу и высушите гранулы на воздухе в течение 10-15 минут.
- Повторное суспендирование ДНК водой класса ПЦР. Используйте 20 мкл на отдельный образец комара или 100 мкл для 10 комаров.
ПРИМЕЧАНИЕ: Методы экстракции ДНК для образцов комаров с использованием коммерчески доступного набора (см. Таблицу материалов)адаптированы из протокола производителя «Выделение геномной ДНК из клеток культуры тканей и тканей животных».
2. реакции ddPCR и подготовка к генерации капель
- Количественно оцените ДНК с помощью флуорометра.
ПРИМЕЧАНИЕ: Для анализа сброса рекомендуется использовать диапазон 3000-30 000 гаплоидных копий генома на реакцию, который предназначен для обнаружения событий NHEJ с помощью зонда, маркирующего HEX, который связывается с последовательностью WT целевого участка разреза и не будет отжигаться (выпадать), если есть делеция или вставка на целевом участке, указание на наличие варианта NHEJ. - Рассчитайте число копий, используя вес гаплоидного генома и концентрацию ДНК в экстракте. Это делается путем умножения концентрации извлеченной ДНК на объем, используемый для получения общей массы ДНК, а затем деления ее на гаплоидный вес генома. Убедитесь, что добавленный объем составляет от 1 до 10 мкл. Разбавляйте по мере необходимости, чтобы быть в рекомендуемом диапазоне гаплоидных копий генома.
ПРИМЕЧАНИЕ: Один геном Гаплоида Anopheles gambiae оценивается в 0,27 пг на взрослого комара10. - Проектирование грунтовок и зондов. Проектирование прямых и обратных олигонуклеотидных праймеров с температурой плавления грунтовки (Tm) в диапазоне 55-60 °C, которые обрамляют 5' и 3'-концы целевого сайта гРНК, производя ампликон 150-400 bp.
- Зонд с маркировкой HEX (гексахлорфлуоресцеин) для обнаружения NHEJ: Спроектируйте олигонуклеотид длиной ~ 15-20 bp, комплементарный целевому участку, и добавьте HEX-зонд к 5'-концу и BHQ1 (BLack Hole Quencher 1) к 3'-концу. Tm гидролизного зонда должен быть на 3-10 °C выше, чем Tm грунтовок.
- Зонд, меченый FAM (6-карбоксифлуоресцеин), для эталонного WT: Спроектируйте олигонуклеотид ~ 15-20 bp в длину, дополняющий законсервированный участок генома, удаленный (около 25 bp) от целевого сайта, и добавьте FAM-зонд к 5'-концу и BHQ1 к 3'-концу. Tm гидролизного зонда должен быть на 3-10 °C выше, чем Tm грунтовок.
3. Подготовка реакции ПЦР
- Подготовьте 25 мкл смеси образцов ddPCR со следующими компонентами: супермикс ddPCR для зондов (без UTP): 12,5 мкл, прямые и обратные праймеры (10 мкМ): по 1 мкл каждый, зонды HEX/FAM (10 мкМ): 0,625 мкл каждый, ДНК: 1-5 мкл (3 750-37 500 гаплоидных копий генома) и вода: до 25 мкл.
- Тщательно перемешайте реакции путем вихревого или рефлюксного пипетирования (вверх и вниз) (20x).
ПРИМЕЧАНИЕ: Если реакции находятся в 96-луночной пластине, пипеткой весь объем вверх и вниз в 20 раз, а не вихрем, чтобы избежать образования пузырьков. - Кратковременно центрифугируйте образцы, чтобы поселить смесь на дне трубки или колодца.
ПРИМЕЧАНИЕ: Убедитесь, что реакции находятся при комнатной температуре для образования капель. Подготовьте 1x смеси ddPCR для дополнительных/неиспользуемых скважин в каждом картридже (каждый картридж имеет 8 лунок).
4. Генерация капель
- Используя многоканальную пипетку объемом 50 мкл, загрузите 20 мкл смеси образцов ddPCR в средний ряд картриджа(рисунок 1A,сверху).
- Загрузите 70 мкл масла в нижний ряд. Загрузка 20 мкл 1x супермикса ddPCR в неиспользуемые скважины.
ПРИМЕЧАНИЕ: Не вводите пузырьки. - Поместите прокладку, касаясь только краев, избегая центральной вогнутой области(рисунок 1B).
- Надежно поместите пластину в генератор капель и закройте крышку, чтобы начать запуск.
- Используя многоканальную пипетку, переведите 40 мкл намывающей смеси из верхнего ряда картриджа(рисунок 1А,снизу) в 96-луночную пластину.
- Вытягивайте жидкий образец в течение 3-5 с под углом 45-30°. Медленно выталкивайте смесь в течение более 3 с под углом 45° в сторону колодца, позволяя ей капать вниз по бокам. Можно идти на вторую остановку (полное изгнание) пипетки, чтобы выгнать всю жидкость.
- Используя термоуплотнители из фольги, запечатайте пластины на 5 с при 180 °C.
5. ПЦР
- Поместите герметичную пластину в термоциклер(рисунок 1C)и установите рекомендуемые условия ПЦР, если они следуют рекомендациям NHEJ Drop-Off следующим образом:
- Начальная денатурация при 95 °C в течение 10 мин.
- Установите 40 циклов при 94 °C в течение 30 с до денатуры, 55 °C в течение 1 мин до отжига и 60 °C в течение 2 мин для продления.
- Подержать при 98 °C в течение 10 мин.
- Удерживать при температуре 4 °C.
ПРИМЕЧАНИЕ: Температура отжига для конкретных грунтовок и наборов зондов может быть оптимизирована с использованием теплового градиента. Используйте скорость рампы 2 ° C / с для всех этапов. Условия ПЦР должны корректироваться в зависимости от каждого экспериментального проекта и установки.
6. Считывание капель
- Надежно поместите пластину в капельный считыватель с колодцем А-1 в левом верхнем углу (сглаженный угол, остальные три обрезаны)(рисунок 1D).
- Настройка пластины в программе: Обозначение FAM как известного опорного канала и HEX как неизвестного(рисунок 2A).
- Запустите эксперимент по чтению капель в качестве прямой количественной оценки. После завершения выполнения измените тип эксперимента на Drop-Off (DOF) для анализа(рисунок 2A).
7. Анализ
- Обозначьте правильные экспериментальные параметры(рисунок 2A): Информация о образце, SuperMix, Имя цели (WT или NHEJ), Тип цели (Ref или Unknown), Сигнал Ch1 (HEX или FAM), Сигнал Ch2 (HEX или FAM) и вручную установите порог для количества капель (рекомендуется выше 10 000 для надежных результатов). Программное обеспечение выполнит большую часть анализа с заданным параметром.
- Проверьте количество капель на вкладке Дроплет; убедитесь, что все они выше 10 000(рисунок 2B).
- Проверьте амплитуду 1 D для эффективного отделения сигнала от негативов.
- В редакторе пластинвыделите всю пластину и установите для параметра Тип эксперимента значение Drop off.
- Установите целевой показатель WT в качестве эталонного,обозначив первый канал для FAM и второй канал для HEX.
- Установите цель NHEJ как неизвестную, обозначив первый канал для FAM, а второй канал как нулевой.
- На вкладке Амплитуда 2D задайте пороговые значения кластера с помощью инструментов графика для каждого образца. Рассмотрим хвост, связанный с кластером WT; это нормально для анализов NHEJ(рисунок 3A).
ПРИМЕЧАНИЕ: На вкладке «Передаточное отношение» нажмите на значок шестеренки в правом верхнем углу графика. Выберите Дробное изобилие. Теперь график будет отображать точку, соответствующую проценту событий NHEJ(рисунок 3B).
Результаты
Применение этой процедуры приводится в carballar-Lejarazú et al.9. В анализе ddPCR Drop-off используются два флуоресцентных зонда для распознавания последовательностей WT и indel: зонд FAM связывается с сохраненной последовательностью внутри ампликона, тогда как зонд HEX нацелен на последовательность WT целевого сайта(рисунок 4A). При наличии инделя зонд HEX не будет связываться. Репрезентативные результаты можно найти на рисунке 2,таблица 1, и таблица 2 Карбаллара-Лехарасу и др.9. Было доказано, что с помощью этого протокола ddPCR обнаруживает широкий спектр событий NHEJ, индуцированных CRISPR-Cas9, и количественно оценивает частоту NHEJ в отдельном или объединенном образце. Пятнадцать различных объединенных образцов по 10 комаров каждый содержал различные аллели NHEJ (таблица 2 Carballar-Lejarazú et al.9). Они были проанализированы с помощью ddPCR с использованием протокола и параметров, представленных здесь. Результаты из таблицы1 9 показывают, что все 15 образцов несли 100% аллели инделя, идентифицированные с помощью анализа Drop-off(рисунок 4B). В другом эксперименте 11 объединенных образцов комаров WT и комаров NHEJ с различными процентами NHEJ (0%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% и 100%) были исследованы с помощью этого протокола ddPCR и результатов(рисунок 2; Carballar- Lejarazú et al.9) показали, что идентифицированный процент близок к сравниваемому методу обнаружения Indel с помощью amplicon Analysis(рисунок 4C).

Рисунок 1:Экспериментальная настройка и процедура. (A) Подготовка картриджа для генерации капель. (Вверху) Образцы заполняются в среднем ряду картриджа, в то время как масло заполняется в нижнем ряду. (Внизу) Верхний ряд заполняется эмульгированными каплями после образования капель. (B)Генератор капель с картриджем, заполненным образцом и закрытым прокладкой на месте. (C)96-луночная пластина, покрытая фольгированным уплотнением в термоциклере. (D)Капельный считыватель с 96-луночной пластиной на месте с металлической крышкой, защелкивающейся над пластиной для ее закрепления. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
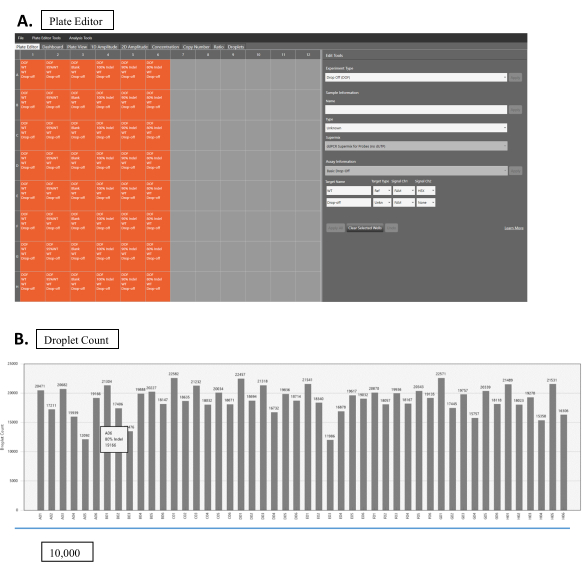
Рисунок 2:Считывание капель. (A) Программный интерфейс для считывания капель. Оранжевые коробки показывают колодцы с образцами. Серые ящики – это пустые колодцы. Экспериментальные параметры задаются на панели «Инструменты редактирования» (справа). Каждый образец можно отредактировать, щелкнув соответствующее поле образца. Выберите Drop Off (DOF) для экспериментального типа. В сведениях о образце заполните соответствующие сведения для имени и типаобразца, а также SuperMix. Выберите Базовый вариант для информации об анализе. Для образца WT выберите WT для целевого имени,Ref для целевого типаи FAM и HEX для signal Ch1 и Ch2соответственно. Для образцов NHEJ введите соответствующее имя для целевого имени,выберите Неизвестно для целевого типаи выберите FAM для Signal Ch1. Оставьте значение Signal Ch2 в значение None. (B) Результаты подсчета капель для нескольких образцов. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
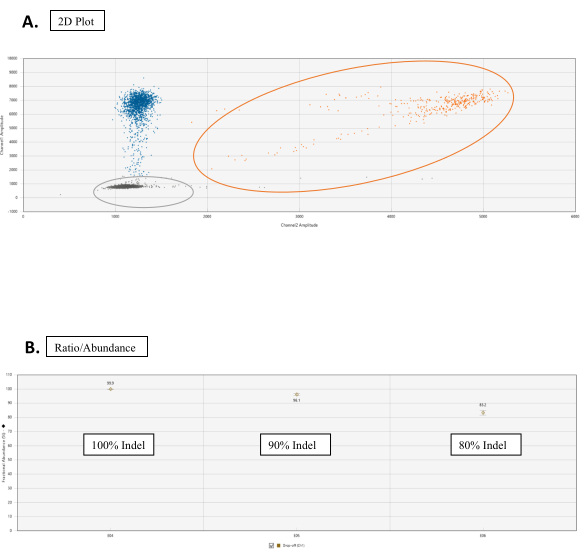
Рисунок 3:Анализ сбрасывания. (A)Кластерный 2D-график для подсчета капель аллелей WT и NHEJ. На вкладке «Амплитуда 2D» все капли по умолчанию не классифицируются. На этом рисунке цвета вручную назначаются для различения. Кластер оранжевых точек представляет собой подсчет аллелей WT, полученный путем связывания зондов FAM и HEX в эталонной последовательности и последовательности целевого сайта соответственно. Синие точки представляют собой десятки капель, которые имеют привязку FAM к эталонной последовательности, но не связывают HEX в последовательности целевого сайта (следовательно, падение HEX). Серые точки — это пустые капли, которые не имеют привязки FAM или HEX. (B)Графики соотношения/численности событий NHEJ. На вкладке Соотношение выберите Дробное изобилие для графика с соответствующим процентом событий NHEJ. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
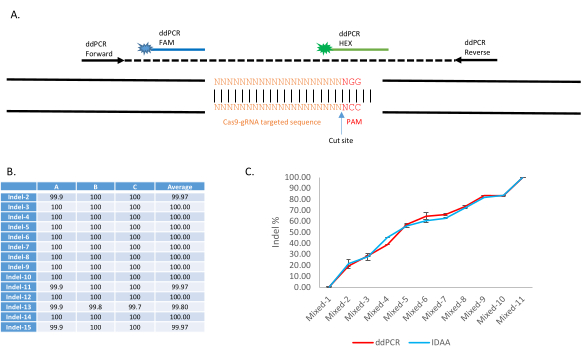
Рисунок 4:Применение анализа Drop-Off с ddPCR для негомологичной идентификации и количественной оценки конечного соединения в трансгенной линии Anopheles stephensi, AsMCRkh1. (A)Схематическое представление анализа ddPCR Drop-Off для обнаружения мутаций на целевом участке ДНК с помощью системы с двумя зондами. Ампликон 150-400 bp усиливается прямым и обратным праймерами. Зонд, меченый FAM, предназначен для связывания с сохраненной последовательностью ампликона, тогда как зонд, меченый HEX, предназначен для связывания с целевым сайтом гРНК WT. (B) Обнаружение различных типов инделов с помощью ddPCR. Пятнадцать пулов из 10 комаров AsMCRkh1, каждый из которых содержит различные типы инделя, включая введение, удаление и замену, были проанализированы с помощью анализа ddPCR Drop-Off. Подробную информацию о мутациях и последовательностях можно найти в Таблице 2 и Таблице S3 Карбаллара и др. 9.(C)Количественная оценка NHEJ в смешанных образцах комаров AsMCRkh1 и WT с различным соотношением (10:0, 9:1, 8:2, 7:3, 6:4, 5:5, 4:6, 3:7, 2:8, 1:9 и 0:10) с использованием ddPCR и сравниваемой методики indel Detection by Amplicon Analysis9. Изображения, адаптированные из Carballar-Lejarazú et al. Biotechniques. 68(4):172-179 (2020)9. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Грунтовка/Зонд | Последовательность (5' » 3') |
| ddPCR Форвард Праймер | ATGATCAAATGTCGACCG |
| ddPCR Обратная грунтовка | ACCGTACTGGTTGAACA |
| ddPCR HEX зонд (BHQ1) | [HEX]-TTCTACGGGCAGGGC-[BHQ1] |
| ddPCR FAM зонд (BHQ1) | [6FAM]-CCACGTGGGATCGAAGG-[BHQ1] |
| HEX: гексахлорфлуоресцеин, FAM: 6-карбоксифлуоресцеин, BHQ: Чёрная дыра Quencher | |
Таблица 1: Последовательности праймеров и зондов.
Обсуждение
Цифровая капельная ПЦР является эффективным методом определения присутствия аллелей инделя, возникающих в результате событий NHEJ в системе генного драйва на основе Cas/gRNA, и позволяет количественно оценить частоту этих аллелей у отдельных лиц или популяций. Некоторые шаги протокола необходимо соблюдать с особой осторожностью для достижения надежных результатов. Во-первых, геномная экстракция ДНК должна быть выполнена тщательно, чтобы обеспечить высокое качество и достаточное количество. Хорошая экстракция позволит точно определить гаплоидные копии генома за реакцию. По нашему опыту, коммерчески доступный набор (см. Таблицу материалов)обеспечивал неизменно высокое качество извлечения ДНК. Тем не менее, извлечение отдельных комаров может оказаться особенно сложным, поскольку гранулу ДНК становится трудно визуализировать и ее можно легко всасывать с супернатантом, если не осторожно. Во-вторых, грунтовки и зонды должны быть тщательно спроектированы. Перед завершением эксперимента ddPCR убедитесь, что разработанные праймеры приводят к одному продукту ПЦР, сначала выполняя традиционную ПЦР и визуализируя один продукт с помощью гелевого электрофореза. Эталонный зонд FAM также должен быть спроектирован таким образом, чтобы он дополнял высококонсервативную последовательность. Это обеспечит точное обнаружение аллелей WT в разнообразной популяции. Комбинации грунтовки и зонда для каждого уникального эксперимента будут иметь различные условия термоциклера, и рекомендуется оптимизировать эти условия с использованием теплового градиента.
Существуют и другие методы идентификации инделей, такие как секвенирование Сэнгера или NGS. Секвенирование Сэнгера ограничено, потому что оно имеет нижний предел обнаружения и низкую способность обнаружения для идентификации новых вариантов. Секвенирование Сэнгера также является трудоемким и не является высокопроизводительным. По сравнению с секвенированием Сэнгера, NGS не имеет таких же ограничений низкой чувствительности, мощности обнаружения и пропускной способности. Еще одним преимуществом NGS является его способность обнаруживать различные мутации от однонуклеотидного полиморфизма (SNP) до перегруппировок. Тем не менее, NGS является более дорогостоящим и трудоемким методом в применении определения Cas9 / gRNA-ассоциированных инделей, поскольку существует только одна целевая область, представляющая интерес, и она лучше всего подходит для более крупных анализов генома. По сравнению с вышеупомянутыми методами, ddPCR обладает высокой пропускной способностью и быстрым временем обработки. Если материалы и инструменты ddPCR доступны собственными силами, 96 образцов могут быть обработаны в течение 1-2 дней, что делает его хорошо подходящим для быстрого анализа крупных испытаний Cas9/gRNA-модифицированных организмов.
Хотя для ddPCR существует много преимуществ, существуют и ограничения. Во-первых, оборудование ddPCR не часто доступно в независимой лабораторной среде. Оборудование ddPCR может быть доступно совместно в более крупных научно-исследовательских учреждениях, но это не позволяет легко генерировать и анализировать данные за пределами учреждения. Во-вторых, в отличие от альтернатив, ddPCR не предоставляет отдельных уникальных последовательностей идентифицированных индел-мутаций. Цифровая капельная ПЦР обеспечит частоту мутаций инделя в популяции, но без последовательности невозможно определить, являются ли присутствующие индели более склонными сохранять или ингибировать функцию интересующего гена. Метод ddPCR, возможно, лучше всего подходит для анализа диких популяций после полевого испытания приводного организма на основе Cas9 / gRNA, поскольку он может эффективно определять частоту внедрения трансгена в местную популяцию и генерации инделов в популяции в режиме, близком к реальному времени. Благодаря быстрому времени обработки ddPCR было бы целесообразно еженедельно проводить отбор проб и анализ популяции в регионе полевых испытаний, если бы материалы были доступны на местном уровне. Начальные затраты на покупку, импорт и установку оборудования ddPCR будут высокими в удаленных лабораториях, но преимущества возможности строгой оценки дикой популяции, поскольку она подвергается модификации из системы привода, оправдают затраты.
Раскрытие информации
Авторы не раскрывают информацию.
Благодарности
Финансирование было предоставлено Инициативой по борьбе с малярией в Ирвине Калифорнийского университета. AAJ является профессором Дональда Брена в Калифорнийском университете в Ирвине.
Материалы
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| ddPCR Super Mix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DNA extraction reagent (e.g. Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| EDTA (pH 8.0) | Invitrogen | AM9260G | |
| Ethanol, 200 Proof | Thermo Fisher Scientific | A4094 | |
| Isopropanol (Certified ACS) | Thermo Fisher Scientific | A416-500 | |
| Nuclei Lysis Solution (NLS) (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| PCR-grade Water | Any certified PCR-grade water can be used | ||
| Protein Precipitation Solution (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| Proteinase K 20 mg/mL | Thermo Fisher Scientific | AM2546 | |
| Materials | |||
| ddPCR 96-Well Plate | Bio-Rad | 12001925 | |
| Droplet Generator DG8 Cartridge and Gaskets | Bio-Rad | 1864007 | |
| Droplet Generation Oil for probes | Bio-Rad | 1863005 | |
| Fluorescent probes (e.g. FAM/HEX probes) | Sigma-Aldrich | N/A | Probes are experiment specific and can be purchased from any certified seller available. |
| Forward and Reverse oligonucleotide primers | Sigma-Aldrich | N/A | Primers are experiment specific and can be purchased from any certified seller available. |
| Equipment | |||
| C1000 Touch Thermal Cycler | Bio-Rad | 1851148 | Can use other Thermo cycler with gradient function and deep well |
Ссылки
- Gantz, V. M., et al. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. Proceedings of the National Academy of Sciences of the United States of America. 112 (49), 6736-6743 (2015).
- Hammond, A., et al. A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae. Nature Biotechnology. 34, 78-83 (2016).
- Courtier-Orgogozo, V., Morizot, B., Boëte, C. Agricultural pest control with CRISPR-based gene drive: time for public debate: Should we use gene drive for pest control. EMBO Reports. 18 (6), 878-880 (2017).
- Carballar-Lejarazú, R., et al. Next-generation gene drive for population modification of the malaria vector mosquito, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 117 (37), 22805-22814 (2020).
- Adolfi, A., et al. Efficient population modification gene-drive rescue system in the malaria mosquito Anopheles stephensi. Nature Communications. 11, 5553 (2020).
- Wang, Z., et al. Comparison of droplet digital PCR and direct Sanger sequencing for the detection of the BRAFV600E mutation in papillary thyroid carcinoma. Journal of Clinical Laboratory Analysis. 33 (6), 22902 (2019).
- Dong, L., Wang, S., Fu, B., Wang, J. Evaluation of droplet digital PCR and next generation sequencing for characterizing DNA reference material for KRAS mutation detection. Scientific Reports. 8 (1), 9650 (2018).
- Bell, C. C., Magor, G. W., Gillinder, K. R., Perkins, A. C. A high-throughput screening strategy for detecting CRISPR_Cas9 induced mutations using next-generation sequencing. BMC Genomics. 15 (1), 1002 (2014).
- Carballar-Lejarazú, R., Kelsey, A., Pham, T. B., Bennett, E. P., James, A. A. Digital droplet PCR and IDAA for the detection of CRISPR indel edits in the malaria species Anopheles stephensi. Biotechniques. 68 (4), 172-179 (2020).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298 (5591), 129-149 (2002).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены