Method Article
Digital-Droplet PCR zum Nachweis von Indel-Mutationen in gentechnisch veränderten Anopheline-Mückenpopulationen
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet die Schritte von der DNA-Extraktion bis zum Versuchsaufbau für die digitale Tröpfchen-PCR (ddPCR), einschließlich der Analyse zur Identifizierung und Quantifizierung von nicht-homologen End-Joining-Ereignissen (NHEJ) an Zielorten nach gRNA-induzierter Cas9-Spaltung und DNA-Reparatur. Andere Anwendungen dieser Methode umfassen Anwendungen wie den Polymorphismus-Nachweis und die Verifizierung von Gen-Editing-Varianten.
Zusammenfassung
Jüngste Fortschritte in der Moskitogenomik und den Gentechniktechnologien haben den Bedarf an schnellen und effizienten Methoden zum Nachweis gezielter DNA-Sequenzvariationen in großem Maßstab gefördert. Insbesondere der Nachweis von Insertionen und Deletionen (Indels) an geneditierten Stellen, die durch CRISPR-Guide-RNA (gRNA)/Cas9-vermittelte nicht-homologe Endverbindung (NHEJ) erzeugt werden, ist wichtig, um die Genauigkeit der Mutagenese und die Häufigkeit unbeabsichtigter Veränderungen zu beurteilen. Wir beschreiben hier ein Protokoll für die digitale Tröpfchen-PCR (ddPCR), das sich gut für die NHEJ-Analyse mit hohem Durchsatz eignet. Während diese Methode keine Daten liefert, die individuelle Sequenzvariationen identifizieren, liefert sie eine quantitative Schätzung der Sequenzvariation innerhalb einer Population. Darüber hinaus kann dieses Protokoll mit geeigneten Ressourcen in einem Labor vor Ort einfacher implementiert werden als die Sequenzierung der nächsten Generation oder der Sanger-Sequenzierung. ddPCR hat auch eine schnellere Bearbeitungszeit für Ergebnisse als jede dieser Methoden, was eine schnellere und vollständigere Analyse der genetischen Variation in Wildpopulationen während Feldversuchen mit gentechnisch veränderten Organismen ermöglicht.
Einleitung
Gene Drives haben ein immenses Potenzial, Insektenpopulationen von medizinischer und landwirtschaftlicher Relevanz zu kontrollieren1,2,3,4,5. Zum Beispiel können Gene-Drive-Systeme, die auf CRISPR-Cas-Nukleasen und Leit-RNAs (gRNAs) basieren, verwendet werden, um Vektormückenpopulationen zu modifizieren, indem Merkmale eingeführt werden, die Malariaparasiten Refraktärität verleihen, was zu einer verminderten Übertragung und weniger Krankheit führt1,4,5. Das Gene-Drive-System kopiert sich selbst und das damit verbundene Merkmal in den prämeiotischen Keimzellen von einem homologen Chromosom zum anderen, und dies stellt sicher, dass die Mehrheit der Nachkommen den Antrieb erbt und das Potenzial für eine lang anhaltende und nachhaltige Populationsmodifikation im Feld schafft. Ein Nachteil der Cas/gRNA-basierten Methoden ist jedoch die Möglichkeit, Insertions- und Deletionsmutationen (Indel) durch nicht-homologe Endverbindungs-DNA-Reparatur (NHEJ) zu erzeugen, was zur Erzeugung von antriebsresistenten Allelen führt, die, wenn sie sich in der Population zu einer ausreichend hohen Frequenz ansammeln, das Antriebssystem daran hindern können, sich auszubreiten1,2,3,4 . Dieses Protokoll beschreibt eine zuverlässige Methode mit hohem Durchsatz, die die Prävalenz und relative Menge von Indelmutationen sowohl auf Populations- als auch auf individueller Ebene während des Cas/gRNA-basierten Gene Drive bestimmen kann.
Next-Generation-Sequencing-Methoden (NGS) bieten eine beispiellose Sequenzierungsauflösung. Die mit NGS verbundenen Kosten und technischen Anforderungen verbieten jedoch Routineprüfungen und schränken seine Verwendung als Hochdurchsatzmethode zur Bewertung der Indel6,7,8 ein. Traditionelle PCR-Quantifizierungsmethoden werden seit langem als Standardbewertungsverfahren für Genom-Indel verwendet; Diese Methoden sind jedoch arbeitsintensiv, benötigen viel Zeit, um Daten zu beschaffen, und weisen ein hohes Maß an Variabilität auf. Die Digital-Droplet-PCR (ddPCR) hat sich in einigen Anwendungen als empfindlicher beim Nachweis von Mutationen erwiesen als die Sanger-Sequenzierung und hat in anderen Anwendungen eine niedrigere Nachweisgrenze als NGS6,7,8,9. Darüber hinaus sind die Kosten für die Bewertung eines Probensatzes und die Bearbeitungszeit für die Erzielung von Ergebnissen für die ddPCR kostengünstiger bzw. schneller als für die Sanger-Sequenzierung oder NGS9. Mit einem Dual-Probe-System identifiziert der Drop-Off-Assay NHEJ-Allele basierend auf dem Fehlen einer Wildtypsequenz (WT) an der gRNA-gerichteten Ziel-Cas9-Cut-Site. In diesem Assay wird ein kurzes Amplikon einschließlich der vorhergesagten Schnittstelle des Cas/gRNA-basierten Systems mit einem spezifischen Primerpaar amplifiziert. Eine Fluoreszenzsonde ist so konzipiert, dass sie an einen konservierten Bereich des Amplikons bindet, und eine andere Fluoreszenzsonde erkennt die WT-Sequenz der Schnittstelle. In Gegenwart eines NHEJ-Allels bindet letzteres nicht an das Amplikon.
Die Verwendung von ddPCR bietet die Möglichkeit, Primer zu entwerfen, um Deletionen, einzelne Basenpaardifferenzen und Insertionen zu erreichen, was eine NHEJ-Profilierung in Mückenpopulationsanalysen ermöglicht9. Angesichts dieser attraktiven Eigenschaften haben wir ein Protokoll für ddPCR für den Hochdurchsatznachweis von Indeln entwickelt, die aus einem Cas/gRNA-basierten Gene-Drive-System in Moskitos erzeugt werden.
Protokoll
1. DNA-Extraktion
- Bereiten Sie EDTA/Nuclei Lysis Buffer (EDTA/NLS) mit dem Verhältnis von 500 μL NLS und 120 μL EDTA pro Probe vor. Scale-up für mehrere Samples. Kühlen Sie die Mischung auf Eis.
HINWEIS: Die Lösung wird je nach Volumen in 2-5 Minuten trüb, wenn sie gekühlt wird. - Homogenisieren Sie die Mückenprobe mit einem mechanischen Homogenisator für 10-15 s in einem 1,5 ml Mikrozentrifugenröhrchen, das mit 600 μL gekühltem EDTA /NLS gefüllt ist. gründlich mischen.
- 17,5 μL 20 mg/ml Proteinase K in das Röhrchen geben und gründlich mischen.
- Über Nacht bei 55 °C inkubieren. Abwechselnd die Probe bei 55 °C für 3 h mit Schütteln inkubieren und die Probe alle 1 h vorwirbeln.
- 200 μL Proteinfällungslösung in die Raumtemperaturprobe geben und 20 s lang kräftig wirbeln.
- Kühlen Sie die Probe für 5 min auf Eis.
- Zentrifugieren Sie die Probe zu Pelletproteinen bei 15.890 RCF für 4 min.
- Aspirieren Sie vorsichtig den Überstand, der die DNA enthält, und übertragen Sie ihn in ein sauberes 1,5 ml Mikrozentrifugenröhrchen mit 600 μL Isopropanol.
- Mischen Sie die Lösung vorsichtig, indem Sie das Röhrchen 5-10 Mal umkehren. Zentrifuge für 1 min bei 15.890 RCF. Den Überstand vorsichtig dekantieren, während das DNA-Pellet erhalten bleibt.
- Fügen Sie 600 μL 70% Ethanol bei Raumtemperatur hinzu. Waschen Sie die pelletierte DNA, indem Sie das Röhrchen vorsichtig umkehren.
- Zentrifuge für 1 min bei 15.890 RCF. Entfernen Sie den Überstand vorsichtig durch Absaugen mit einer Glaspipettenspitze.
- Drehen Sie das Röhrchen auf sauberes saugfähiges Papier und trocknen Sie das Pellet für 10-15 min an der Luft.
- Resuspendieren Sie die DNA mit PCR-Wasser. Verwenden Sie 20 μL pro einzelner Mückenprobe oder 100 μL für 10 gepoolte Moskitos.
HINWEIS: DNA-Extraktionsmethoden für Mückenproben unter Verwendung eines handelsüblichen Kits (siehe Materialtabelle)basieren auf dem Protokoll Isolating Genomic DNA from Tissue Culture Cells and Animal Tissue des Herstellers.
2. ddPCR-Reaktionen und Tröpfchenerzeugungsvorbereitung
- Quantifizieren Sie DNA mit einem Fluorometer.
HINWEIS: Für den Drop-off-Assay wird empfohlen, einen Bereich von 3.000-30.000 haploiden Genomkopien pro Reaktion zu verwenden, der zum Nachweis von NHEJ-Ereignissen mit einer HEX-Markierungssonde entwickelt wurde, die an eine WT-Sequenz der Zielschnittstelle bindet und nicht glüht (abfällt), wenn es an der Zielstelle zu einer Deletion oder Insertion kommt. zeigt das Vorhandensein einer NHEJ-Variante an. - Berechnen Sie die Kopienzahl anhand des haploiden Genomgewichts und der DNA-Konzentration im Extrakt. Dies geschieht, indem die Konzentration der extrahierten DNA mit dem Volumen multipliziert wird, das zur Erzielung der gesamten DNA-Masse verwendet wird, und sie dann durch das haploide Genomgewicht dividiert. Stellen Sie sicher, dass das zugegebene Volumen zwischen 1-10 μL liegt. Verdünnen Sie es nach Bedarf, um im empfohlenen Bereich der haplooiden Genomkopie zu sein.
HINWEIS: Ein haploides Genom von Anopheles gambiae wird auf 0,27 pg pro erwachsener Mücke geschätzt10. - Entwerfen Sie Primer und Sonden. Entwerfen Sie Vorwärts- und Rückwärts-Oligonukleotid-Primer mit einer Primer-Schmelztemperatur (Tm) im Bereich von 55-60 °C, die die 5'- und 3'-Enden der gRNA-Zielstelle flankieren und ein Amplikon von 150-400 bp erzeugen.
- HEX (Hexachloro-Fluorescein)-markierte Sonde für den NHEJ-Nachweis: Entwerfen Sie ein Oligonukleotid von ~ 15-20 bp Länge komplementär zur Zielstelle und fügen Sie die HEX-Sonde am 5'-Ende und BHQ1 (BLack Hole Quencher 1) am 3'-Ende hinzu. Der Tm der Hydrolysesonde sollte 3-10 °C höher sein als der Tm der Primer.
- FAM (6-Carboxyfluorescein)-markierte Sonde für referenz WT: Entwerfen Sie ein Oligonukleotid ~ 15-20 bp Länge komplementär zu einer konservierten Genomstelle, die (etwa 25 bp) von der Zielstelle entfernt ist, und fügen Sie die FAM-Sonde zum 5'-Ende und BHQ1 zum 3'-Ende hinzu. Der Tm der Hydrolysesonde sollte 3-10 °C höher sein als der Tm der Primer.
3. PCR-Reaktionsvorbereitung
- Bereiten Sie 25 μL des ddPCR Sample Mix mit den folgenden Komponenten vor: ddPCR Supermix für Sonden (kein UTP): 12,5 μL, Forward und Reverse Primer (10 μM): je 1 μL, HEX/FAM-Sonden (10 μM): je 0,625 μL, DNA: 1-5 μL (3.750-37.500 haploide Genomkopien) und Wasser: bis zu 25 μL.
- Mischen Sie die Reaktionen gründlich durch Wirbeln oder Refluxpipettieren (auf und ab) (20x).
HINWEIS: Wenn die Reaktionen in einer 96-Well-Platte stattfinden, pipettieren Sie das gesamte Volumen 20 Mal nach oben und unten, anstatt sich zu verwirbeln, um die Bildung von Blasen zu vermeiden. - Zentrifugieren Sie die Proben kurz, um die Mischung am Boden des Röhrchens oder der Vertiefung abzusetzen.
HINWEIS: Stellen Sie sicher, dass die Reaktionen bei Raumtemperatur für die Tröpfchenerzeugung sind. Bereiten Sie 1x ddPCR-Mix für zusätzliche/unbenutzte Vertiefungen in jeder Patrone vor (jede Patrone hat 8 Vertiefungen).
4. Tröpfchenerzeugung
- Mit einer 50-μL-Mehrkanalpipette werden 20 μL der ddPCR-Probenmischung in die mittlere Reihe der Kartusche geladen(Abbildung 1A,oben).
- Laden Sie 70 μL des Öls in die untere Reihe. Laden Sie 20 μL 1x ddPCR Supermix in ungenutzte Vertiefungen.
HINWEIS: Führen Sie keine Blasen ein. - Platzieren Sie die Dichtung, die nur die Kanten berührt, und vermeiden Sie den mittleren konkavierten Bereich (Abbildung 1B).
- Legen Sie die Platte sicher in den Tröpfchengenerator und schließen Sie die Abdeckung, um den Lauf zu starten.
- Übertragen Sie mit der Mehrkanalpipette 40 μL der Emulsionsmischung aus der oberen Reihe der Kartusche(Abbildung 1A,unten) in die 96-Well-Platte.
- Ziehen Sie eine flüssige Probe für 3-5 s in einem Winkel von 45-30°. Stoßen Sie die Mischung langsam für mehr als 3 s in einem Winkel von 45 ° in die Seite des Brunnens aus, so dass sie an der Seite heruntertropfen kann. Es ist in Ordnung, zur zweiten Haltestelle (vollständige Austreibung) der Pipette zu gehen, um die gesamte Flüssigkeit auszustoßen.
- Versiegeln Sie die Platten mit Folien-Heißsiegeln für 5 s bei 180 °C.
5. PCR
- Legen Sie die versiegelte Platte in den Thermocycler (Abbildung 1C) und stellen Sie die empfohlenen PCR-Bedingungen ein, wenn Sie die NHEJ Drop-Off-Richtlinien wie folgt befolgen:
- Anfängliche Denaturierung bei 95 °C für 10 min.
- Stellen Sie 40 Zyklen von 94 °C für 30 s zum Denaturieren, 55 °C für 1 min zum Glühen und 60 °C für 2 min zum Verlängern ein.
- Bei 98 °C für 10 min halten.
- Bei 4 °C halten.
HINWEIS: Die Glühtemperatur für bestimmte Primer und Sondensätze kann durch einen thermischen Gradienten optimiert werden. Verwenden Sie für alle Schritte eine Rampenrate von 2 °C/s. Die PCR-Bedingungen sollten in Abhängigkeit von jedem Versuchsdesign und -aufbau angepasst werden.
6. Tropfen lesen
- Legen Sie die Platte sicher in den Tröpfchenleser mit der Vertiefung A-1 oben links (geglättete Ecke, die anderen drei sind umrandet) (Abbildung 1D).
- Richten Sie die Platte im Programm ein: FAM als bekannten Referenzkanal und HEX als den unbekannten kanalisieren (Abbildung 2A).
- Führen Sie das Droplet-Leseexperiment als direkte Quantifizierung aus. Ändern Sie nach Abschluss des Laufs den Experimenttyp für die Analyse in Drop-Off (DOF) (Abbildung 2A).
7. Würdigung
- Legen Sie die richtigen experimentellen Parameter fest (Abbildung 2A): Probeninformationen, SuperMix, Zielname (WT oder NHEJ), Zieltyp (Ref oder unbekannt), Signal Ch1 (HEX oder FAM), Signal Ch2 (HEX oder FAM), und legen Sie den Schwellenwert für die Tröpfchenzahl manuell fest (empfohlen über 10.000 für zuverlässige Ergebnisse). Die Software führt den größten Teil der Analyse mit dem angegebenen Parameter durch.
- Überprüfen Sie die Anzahl der Tropfen auf der Registerkarte Droplet. Stellen Sie sicher, dass alle über 10.000 liegen (Abbildung 2B).
- Überprüfen Sie die Amplitude von 1 D auf eine effiziente Signaltrennung von Negativen.
- Markieren Sie im Platteneditordie gesamte Platte und legen Sie den Experimenttyp auf Drop off fest.
- Legen Sie das WT-Ziel als Referenzfest und legen Sie Kanal eins für FAM und Kanal zwei für HEX fest.
- Legen Sie das NHEJ-Ziel als unbekannt fest, und legen Sie Kanal eins für FAM und Kanal zwei als keinen fest.
- Legen Sie auf der Registerkarte 2D-Amplitude die Clusterschwellenwerte mit den Diagrammwerkzeugen für jede Probe fest. Betrachten Sie den Schwanz, der dem WT-Cluster zugeordnet ist. Dies ist normal für NHEJ-Assays (Abbildung 3A).
HINWEIS: Klicken Sie auf der Registerkarte Übersetzung oben rechts im Diagramm auf das Zahnradsymbol. Wählen Sie die Bruchhäufigkeit aus. Das Diagramm zeigt nun einen Punkt, der dem Prozentsatz der NHEJ-Ereignisse entspricht (Abbildung 3B).
Ergebnisse
Eine Anwendung dieses Verfahrens erscheint in Carballar-Lejarazú et al.9. Der ddPCR Drop-off-Assay verwendet zwei fluoreszierende Sonden, um WT- und Indelsequenzen zu erkennen: Eine FAM-Sonde bindet an eine konservierte Sequenz innerhalb des Amplikons, während die HEX-Sonde auf die WT-Sequenz der Zielstelle abzielt (Abbildung 4A). In Gegenwart eines Indels bindet die HEX-Sonde nicht. Repräsentative Ergebnisse finden sich in Abbildung 2, Tabelle 1 und Tabelle 2 von Carballar-Lejarazú et al.9. Mit diesem Protokoll wurde nachgewiesen, dass ddPCR eine Vielzahl von CRISPR-Cas9-induzierten NHEJ-Ereignissen erkennt und die NHEJ-Frequenz in einer einzelnen oder gepoolten Probe quantifiziert. Fünfzehn verschiedene gepoolte Proben von jeweils 10 Moskitos enthielten verschiedene NHEJ-Allele (Tabelle 2 von Carballar-Lejarazú et al.9). Diese wurden mit ddPCR unter Verwendung des hier vorgestellten Protokolls und der Parameter analysiert. Die Ergebnisse aus Tabelle 19 zeigen, dass alle 15 Proben 100% Indel-Allele enthielten, wie sie durch den Drop-off-Assay identifiziert wurden (Abbildung 4B). In einem anderen Experiment wurden 11 gepoolte Proben von WT-Mücken und NHEJ-Mücken mit unterschiedlichen NHEJ-Prozentsätzen (0%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% und 100%) mit diesem ddPCR-Protokoll untersucht, und die Ergebnisse (Abbildung 2; Carballar-Lejarazú et al.9) zeigten, dass der identifizierte Prozentsatz der verglichenen Technik des Indel-Nachweises durch Amplicon-Analyse nahe kommt (Abbildung 4C).

Abbildung 1: Versuchsaufbau und -ablauf. (A) Kartuschenvorbereitung für die Tröpfchenerzeugung. (Oben) Die Proben werden in der mittleren Reihe der Kartusche gefüllt, während öl in der unteren Reihe gefüllt wird. (Unten) Obere Reihe gefüllt mit emulgierten Tröpfchen nach Tröpfchenerzeugung. (B) Tröpfchengenerator mit einer mit Probe gefüllten Kartusche, die mit einer Dichtung bedeckt ist. (C) 96-Well-Platte mit Foliendichtung in einem Thermo-Cycler bedeckt. (D) Tröpfchenleser mit 96-Well-Platte an Ort und Stelle mit einer Metallabdeckung, die über die Platte eingerastet ist, um sie zu sichern. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
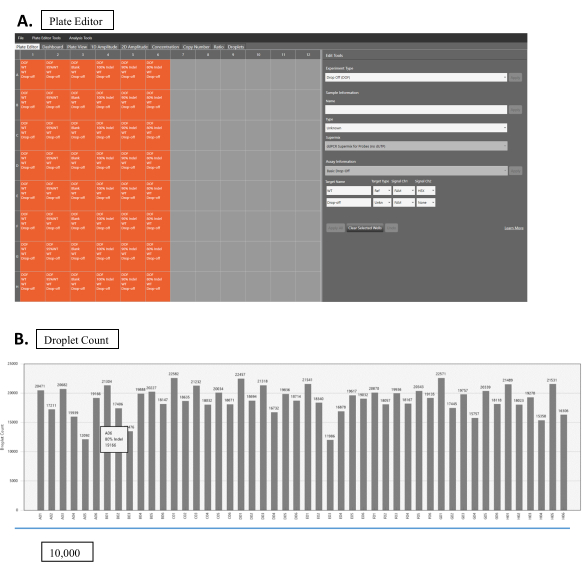
Abbildung 2: Tröpfchenablesung. (A) Softwareschnittstelle zum Ablesen von Tröpfchen. Orangefarbene Boxen zeigen Vertiefungen mit Proben. Graue Kästchen sind leere Brunnen. Experimentelle Parameter werden im Bedienfeld "Bearbeitungswerkzeuge" (rechts) festgelegt. Jedes Sample kann durch Anklicken der jeweiligen Samplebox bearbeitet werden. Wählen Sie Drop Off (DOF) für Experimenteller Typaus. Geben Sie in den Probeninformationen die entsprechenden Informationen für den Namen und den Typder Probe sowie für SuperMix ein. Wählen Sie den Basic Drop-Off für die Assay-Informationen. Wählen Sie für das WT-Beispiel WT als Zielname, Ref für Zieltypund FAM und HEX für Signal Ch1 bzw. Ch2aus. Geben Sie für NHEJ-Beispiele den entsprechenden Namen für den Zielnamen ein,wählen Sie Unbekannt für den Zieltypund fam für Signal Ch1aus. Lassen Sie Signal Ch2 bei None. (B) Ergebnisse der Tröpfchenzahl für mehrere Proben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
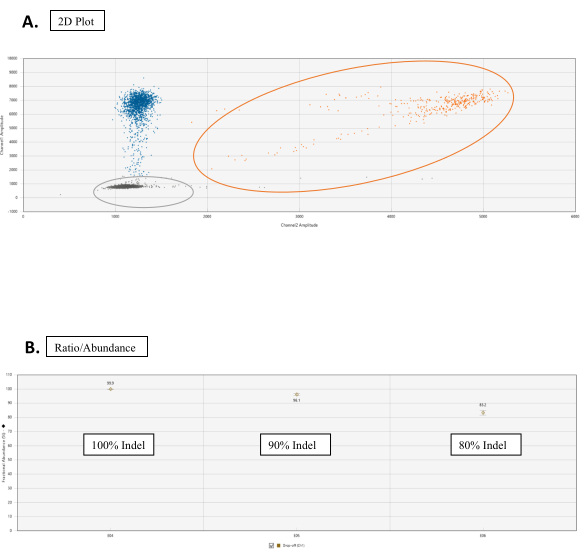
Abbildung 3: Drop-Off-Assay-Analyse. (A) Cluster-2D-Diagramm für die Tröpfchenzahl der WT- und NHEJ-Allele. Auf der Registerkarte "2D-Amplitude" sind alle Tröpfchen standardmäßig nicht klassifiziert. In dieser Abbildung werden Farben zur Unterscheidung manuell zugewiesen. Der orangefarbene Punktcluster sind WT-Allelzahlen, die durch Bindung von FAM- und HEX-Sonden an der Referenzsequenz bzw. der Zielortsequenz erhalten werden. Blaue Punkte stellen Scores von Tröpfchen dar, bei denen FAM an die Referenzsequenz bindet, aber keine HEX-Bindung an der Zielortsequenz (daher Abfall von HEX). Graue Punkte sind leere Tröpfchen, die weder FAM- noch HEX-Bindung haben. (B) Verhältnis/Häufigkeitsgraphen von NHEJ-Ereignissen. Wählen Sie auf der Registerkarte Verhältnis die Option Fractional Abundance für ein Diagramm mit dem entsprechenden Prozentsatz der NHEJ-Ereignisse aus. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
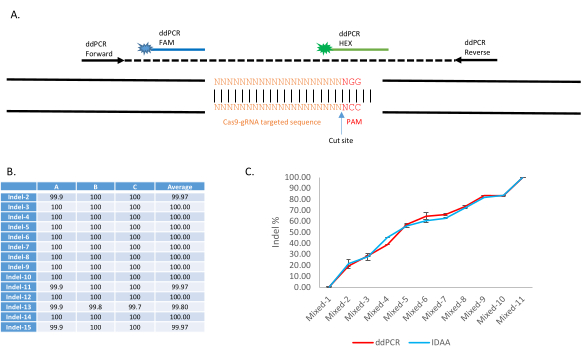
Abbildung 4: Anwendung des Drop-Off-Assays mit ddPCR zur nicht-homologen Endverbindungsidentifizierung und -quantifizierung in der transgenen Anopheles stephensi-Linie, AsMCRkh1. (A) Schematische Darstellung des ddPCR Drop-Off Assays zum Nachweis von Mutationen an einer Ziel-DNA-Stelle mit einem Dual-Probe-System. Ein Amplikon von 150-400 bp wird mit den Vorwärts- und Rückwärtsprimern verstärkt. Eine FAM-markierte Sonde ist so konzipiert, dass sie an eine konservierte Sequenz des Amplikons bindet, während eine HEX-markierte Sonde so konzipiert ist, dass sie an die WT gRNA-Zielstelle bindet. (B) Nachweis verschiedener Arten von Indeln mit ddPCR. Fünfzehn Pools von 10 AsMCRkh1-Moskitos, die jeweils verschiedene Arten von Indel enthielten, einschließlich Insertion, Deletion und Substitution, wurden mit dem ddPCR Drop-Off-Assay analysiert. Details zu Mutationen und Sequenzen finden sich in Tabelle 2 und Tabelle S3 von Carballar et al. 9. (C) Quantifizierung von NHEJ in gemischten Proben von AsMCRkh1- und WT-Mücken mit verschiedenen Verhältnissen (10:0, 9:1, 8:2, 7:3, 6:4, 5:5, 4:6, 3:7, 2:8, 1:9 und 0:10) unter Verwendung von ddPCR und einer verglichenen Technik der Indel-Detektion durch Amplicon-Analyse9. Bilder adaptiert von Carballar-Lejarazú et al. Biotechniques. 68(4):172-179 (2020)9. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Primer/Sonde | Sequenz (5' » 3') |
| ddPCR Forward Primer | ATGATCAAATGTCGACCG |
| ddPCR Reverse Primer | ACCGTACTGGTTGAACA |
| ddPCR HEX-Sonde (BHQ1) | [HEX]-TTCTACGGGCAGGGC-[BHQ1] |
| ddPCR FAM Sonde (BHQ1) | [6FAM]-CCACGTGGGATCGAAGG-[BHQ1] |
| HEX: Hexachlor-Fluorescein, FAM: 6-Carboxyfluorescein, BHQ: Black Hole Quencher | |
Tabelle 1: Sequenzen von Primern und Sonden.
Diskussion
Die digitale Tröpfchen-PCR ist eine effiziente Methode zur Bestimmung des Vorhandenseins von Indel-Allelen, die aus NHEJ-Ereignissen resultieren, in einem Cas/gRNA-basierten Gene-Drive-System und ermöglicht die Quantifizierung der Häufigkeit dieser Allele bei Individuen oder Populationen. Einige Schritte des Protokolls müssen mit besonderer Sorgfalt befolgt werden, um zuverlässige Ergebnisse zu erzielen. Erstens muss die genomische DNA-Extraktion sorgfältig durchgeführt werden, um eine hohe Qualität und ausreichende Quantität zu gewährleisten. Eine gute Extraktion ermöglicht eine genaue Bestimmung der haploiden Genomkopien pro Reaktion. Nach unserer Erfahrung lieferte ein kommerziell erhältliches Kit (siehe Materialtabelle)gleichbleibend hochwertige DNA-Extraktionen. Extraktionen einzelner Moskitos können sich jedoch als besonders schwierig erweisen, da das DNA-Pellet schwer zu visualisieren ist und leicht mit dem Überstand aufgesaugt werden kann, wenn es nicht vorsichtig ist. Zweitens müssen Primer und Sonden sorgfältig entworfen werden. Stellen Sie vor Abschluss des ddPCR-Experiments sicher, dass die entworfenen Primer zu einem einzelnen PCR-Produkt führen, indem Sie zunächst eine herkömmliche PCR durchführen und ein einzelnes Produkt über Gelelektrophorese visualisieren. Die Referenz-FAM-Sonde muss ebenfalls so ausgelegt sein, dass sie eine hochkonservierte Sequenz ergänzt. Dies wird einen genauen Nachweis von WT-Allelen in einer vielfältigen Population gewährleisten. Die Primer/Sonden-Kombinationen für jedes einzelne Experiment haben unterschiedliche Thermocycler-Bedingungen, und es wird empfohlen, diese Bedingungen mit einem thermischen Gradienten zu optimieren.
Es gibt andere Methoden zur Identifizierung von Indeln, wie z.B. Sanger-Sequenzierung oder NGS. Die Sanger-Sequenzierung ist begrenzt, da sie eine niedrigere Nachweisgrenze und eine geringe Entdeckungskraft hat, um neue Varianten zu identifizieren. Die Sanger-Sequenzierung ist auch arbeitsintensiv und hat keinen hohen Durchsatz. Im Vergleich zur Sanger-Sequenzierung weist NGS nicht die gleichen Einschränkungen in Bezug auf geringe Empfindlichkeit, Erkennungsleistung und Durchsatz auf. Ein weiterer Vorteil von NGS ist seine Fähigkeit, eine Vielzahl von Mutationen vom Einzelnukleotid-Polymorphismus (SNPs) bis hin zu Umlagerungen nachzuweisen. NGS ist jedoch eine kostspieligere und zeitaufwendigere Methode bei der Anwendung der Bestimmung von Cas9/gRNA-assoziierten Indeln, da es nur eine Zielregion von Interesse gibt und sich am besten für größere genomweite Analysen eignet. Im Vergleich zu den oben genannten Methoden ist ddPCR ein hoher Durchsatz und hat eine schnelle Durchlaufzeit. Wenn die ddPCR-Materialien und -Instrumente im eigenen Haus verfügbar sind, können innerhalb von 1-2 Tagen 96 Proben verarbeitet werden, was sie für die schnelle Analyse großer Studien mit Cas9/gRNA-modifizierten Organismen gut geeignet macht.
Während viele Vorteile für ddPCR bestehen, gibt es auch Einschränkungen. Erstens ist die ddPCR-Ausrüstung nicht häufig in einer unabhängigen Laborumgebung verfügbar. ddPCR-Geräte können in größeren Forschungseinrichtungen kommunal verfügbar sein, aber dies ermöglicht keine einfache Datengenerierung und -analyse außerhalb der Institution. Zweitens liefert ddPCR im Gegensatz zu den Alternativen nicht die einzelnen eindeutigen Sequenzen identifizierter Indelmutationen. Die digitale Tröpfchen-PCR liefert die Häufigkeit von Indelmutationen innerhalb einer Population, aber ohne die Sequenz kann man nicht feststellen, ob die vorhandenen Indels eher die Funktion des interessierenden Gens erhalten oder hemmen. Die ddPCR-Methode eignet sich vielleicht am besten, um Wildpopulationen nach einem Feldfreisetzungsversuch eines Cas9/gRNA-basierten Antriebsorganismus zu analysieren, da sie die Häufigkeit der Einführung des Transgens in die einheimische Population und die Erzeugung von Indeln innerhalb der Population nahezu in Echtzeit effizient bestimmen kann. Aufgrund der schnellen Durchlaufzeit der ddPCR wäre es möglich, wöchentlich Probenahmen durchzuführen und die Population in einer Feldversuchsregion zu analysieren, wenn Materialien vor Ort verfügbar wären. Die Anlaufkosten für den Kauf, den Import und die Einrichtung der ddPCR-Ausrüstung wären in abgelegenen Labors hoch, aber die Vorteile, eine wild lebende Population streng bewerten zu können, während sie von einem Antriebssystem modifiziert wird, würden die Kosten rechtfertigen.
Offenlegungen
Die Autoren haben keine Offenlegungen.
Danksagungen
Die Finanzierung erfolgte durch die Irvine Malaria Initiative der University of California. AAJ ist Donald Bren Professor an der University of California, Irvine.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| ddPCR Super Mix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DNA extraction reagent (e.g. Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| EDTA (pH 8.0) | Invitrogen | AM9260G | |
| Ethanol, 200 Proof | Thermo Fisher Scientific | A4094 | |
| Isopropanol (Certified ACS) | Thermo Fisher Scientific | A416-500 | |
| Nuclei Lysis Solution (NLS) (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| PCR-grade Water | Any certified PCR-grade water can be used | ||
| Protein Precipitation Solution (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| Proteinase K 20 mg/mL | Thermo Fisher Scientific | AM2546 | |
| Materials | |||
| ddPCR 96-Well Plate | Bio-Rad | 12001925 | |
| Droplet Generator DG8 Cartridge and Gaskets | Bio-Rad | 1864007 | |
| Droplet Generation Oil for probes | Bio-Rad | 1863005 | |
| Fluorescent probes (e.g. FAM/HEX probes) | Sigma-Aldrich | N/A | Probes are experiment specific and can be purchased from any certified seller available. |
| Forward and Reverse oligonucleotide primers | Sigma-Aldrich | N/A | Primers are experiment specific and can be purchased from any certified seller available. |
| Equipment | |||
| C1000 Touch Thermal Cycler | Bio-Rad | 1851148 | Can use other Thermo cycler with gradient function and deep well |
Referenzen
- Gantz, V. M., et al. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. Proceedings of the National Academy of Sciences of the United States of America. 112 (49), 6736-6743 (2015).
- Hammond, A., et al. A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae. Nature Biotechnology. 34, 78-83 (2016).
- Courtier-Orgogozo, V., Morizot, B., Boëte, C. Agricultural pest control with CRISPR-based gene drive: time for public debate: Should we use gene drive for pest control. EMBO Reports. 18 (6), 878-880 (2017).
- Carballar-Lejarazú, R., et al. Next-generation gene drive for population modification of the malaria vector mosquito, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 117 (37), 22805-22814 (2020).
- Adolfi, A., et al. Efficient population modification gene-drive rescue system in the malaria mosquito Anopheles stephensi. Nature Communications. 11, 5553 (2020).
- Wang, Z., et al. Comparison of droplet digital PCR and direct Sanger sequencing for the detection of the BRAFV600E mutation in papillary thyroid carcinoma. Journal of Clinical Laboratory Analysis. 33 (6), 22902 (2019).
- Dong, L., Wang, S., Fu, B., Wang, J. Evaluation of droplet digital PCR and next generation sequencing for characterizing DNA reference material for KRAS mutation detection. Scientific Reports. 8 (1), 9650 (2018).
- Bell, C. C., Magor, G. W., Gillinder, K. R., Perkins, A. C. A high-throughput screening strategy for detecting CRISPR_Cas9 induced mutations using next-generation sequencing. BMC Genomics. 15 (1), 1002 (2014).
- Carballar-Lejarazú, R., Kelsey, A., Pham, T. B., Bennett, E. P., James, A. A. Digital droplet PCR and IDAA for the detection of CRISPR indel edits in the malaria species Anopheles stephensi. Biotechniques. 68 (4), 172-179 (2020).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298 (5591), 129-149 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten