Method Article
أتمتة القياس الكمي الكلي في Caenorhabditis elegans
In This Article
Summary
يصف البروتوكول التالي تطوير وتحسين سير عمل عالي الإنتاجية لزراعة الديدان والتصوير الفلوري ومعالجة الصور الآلية لتحديد كمام البولي جلوتامين كميا كتقييم للتغيرات في البروتيوستاسيس.
Abstract
وقد عزز ارتفاع معدل انتشار الأمراض التشكيلية للبروتين العصبي التنكسي (PCDs) اهتماما كبيرا بهذا الموضوع على مر السنين. وقد دعا هذا الاهتمام المتزايد إلى تنويع وتحسين النماذج الحيوانية القادرة على إعادة إنتاج الأنماط الظاهرية للأمراض التي لوحظت في البشر المصابين ب PCDs. على الرغم من أن نماذج الفئران أثبتت أنها لا تقدر بثمن ، إلا أنها باهظة الثمن وترتبط بطرق شاقة ومنخفضة الإنتاجية. تم تبرير استخدام نموذج Caenorhabditis elegans nematode لدراسة PCDs من خلال سهولة الصيانة النسبية ، والتكلفة المنخفضة ، ووقت التوليد السريع ، مما يسمح بالتطبيقات عالية الإنتاجية. بالإضافة إلى ذلك ، فإن الحفظ العالي بين C. elegans والجينوم البشري يجعل هذا النموذج أداة اكتشاف لا تقدر بثمن. تظهر الديدان الخيطية التي تعبر عن مسالك البولي جلوتامين (polyQ) الموسومة بالفلورسنت على الأنسجة (polyQ) تجميعا يعتمد على العمر وطول polyQ يتميز ببؤر الفلورسنت. غالبا ما يتم توظيف هؤلاء المراسلين كوكلاء لمراقبة التغيرات في البروتيوستاسيس عبر الأنسجة. يستغرق القياس الكمي اليدوي للتجميع وقتا طويلا ، مما يحد من الإنتاجية التجريبية. علاوة على ذلك ، يمكن أن يؤدي التحديد الكمي اليدوي إلى التحيز ، حيث يمكن أن يكون التحديد الكلي ذاتيا للغاية. هنا ، تم توحيد بروتوكول يتكون من زراعة الديدان ، والحصول على الصور ، ومعالجة البيانات لدعم القياس الكمي الكلي عالي الإنتاجية باستخدام C. elegans التي تعبر عن polyQ الخاص بالأمعاء. من خلال تنفيذ خط أنابيب معالجة الصور القائم على C. elegans باستخدام CellProfiler ، وهو برنامج لتحليل الصور ، تم تحسين هذه الطريقة لفصل وتحديد الديدان الفردية وتعداد المجاميع الخاصة بكل منها. على الرغم من أن مفهوم الأتمتة ليس فريدا تماما ، إلا أن الحاجة إلى توحيد مثل هذه الإجراءات من أجل التكرار ، والقضاء على التحيز من العد اليدوي ، وزيادة الإنتاجية مرتفعة. من المتوقع أن هذه الطرق يمكن أن تبسط بشكل كبير عملية فحص المكتبات البكتيرية أو الجينومية أو الدوائية الكبيرة باستخدام نموذج C. elegans.
Introduction
تتميز الأمراض التوافقية للبروتين العصبي التنكسي المعتمد على العمر (PCDs) مثل مرض الزهايمر وباركنسون وهانتنغتون ، أو التصلب الجانبي الضموري ، باختلال البروتين الذي يؤدي إلى التجميع وموت الخلايا وتنكس الأنسجة1. في حين يتم التعرف على سوء طي البروتين على أنه الجاني ، فإن مسببات هذه الأمراض ليست واضحة. على هذا النحو ، تم إعاقة تطوير علاجات فعالة بسبب نقص المعرفة فيما يتعلق بالعوامل والظروف التي تسهم في ظهور المرض وتقدمه. تشير الدراسات الحديثة إلى أن التغيرات في الميكروبيوم تؤثر على بداية وتطور وشدة PCDs2،3،4. ومع ذلك ، فإن تعقيد الميكروبيوم البشري ، أو حتى الفئران ، يجعل من الصعب إجراء دراسات من شأنها أن تكشف عن التأثير الدقيق للميكروبات على مضيفها. لذلك ، غالبا ما تستخدم الكائنات الحية الأبسط ، مثل Caenorhabditis elegans ، كأداة اكتشاف 5,6,7,8. استخدمت الدراسات الحديثة C. elegans للتحقيق في تأثير البكتيريا على البروتينات المضيفة والتسبب في المرض 9,10. الاستعمار البكتيري ، hormesis ، والتغيرات الجينومية هي من بين الحالات النموذجية التي تؤثر على تجميع المسالك polyglutamine (polyQ)9،11،12. بالإضافة إلى ذلك ، تظهر مجموعات البروتين غير المطوية هذه تراكمات تعتمد على طول polyQ والعمر داخل المضيف وترتبط بضعف الحركة 9,13. يمكن للنهج البسيط نسبيا لتحديد كمية البونكتا المصنفة بالفلورسنت أن يولد بيانات مهمة عن الظروف أو العوامل أو الأدوية التي تؤثر على طي البروتين وتجميعه.
على الرغم من أن التحديد الكمي للبوصلة الفلورية قد أثبت أنه إجراء موثوق به وبسيط نسبيا ، إلا أن التحدي لا يزال يتمثل في تطوير بروتوكول من شأنه تسهيل الفحص واسع النطاق للمركبات أو البكتيريا أو الظروف التي تؤثر على تراكم البروتين. إن مفهوم المعالجة الآلية لصور C. elegans وتحديد كمياتها ليس جديدا تماما ، حيث تم تطوير عدد من أدوات الدعم العملية14,15. ومع ذلك ، فإن دمج الثقافة واكتساب الصور وخط أنابيب المعالجة أمر ضروري للقضاء على التباين في النتائج والسماح بشاشات أعلى إنتاجية.
على هذا النحو ، فإن الغرض من هذه المخطوطة هو توحيد الإجراء المستخدم لقياس تجميع polyQ في C. elegans كوكيل للكشف عن التغيرات في البروتيوستاسيس. تم إنجاز هذه المهمة من خلال استخدام CellProfiler ، وهو برنامج مفتوح المصدر لتحليل الصور16 قادر على تحديد الدودة وتجميعها آليا ، ويتم دمجه في بروتوكول أكبر لزراعة الديدان والحصول على الصور ومعالجة البيانات.
Protocol
اتبعت جميع الإجراءات المبادئ التوجيهية للسلامة التي تم استعراضها والموافقة عليها من قبل اللجنة المؤسسية للسلامة الأحيائية بجامعة فلوريدا. واتخذت تدابير مناسبة للسلامة الأحيائية للتخفيف من خطر التعرض لبكتيريا السلامة البيولوجية من المستوى 2.
ملاحظة: بالنسبة لجميع التجارب، يجب نشر C. elegans والحفاظ عليها على ألواح وسائط نمو الديدان الخيطية (NGM) المزروعة بالإشريكية القولونية OP50.
1. إعداد لوحات NGM 10 سم
- امزج 3 جم من كلوريد الصوديوم و 2.5 جم من التربتيكاس-الببتون و 17 جم من الأجار في قارورة سعة 2 لتر ، واملأها إلى 1 لتر بالماء المقطر المزدوج (ddH2O). أضف شريط التحريك المغناطيسي قبل التعقيم.
- قم بتعقيم الخليط لمدة 45 دقيقة عند 121 درجة مئوية وضغط 21 رطل لكل بوصة مربعة. دع الخليط يبرد إلى 50 درجة مئوية في حمام مائي.
- باستخدام تقنيات التعقيم ، أضف المحاليل المعقمة التالية: 1 مل من 1 M CaCl2 · 2H2O، 1 مل من 1 M MgSO4· 7H2O ، 1 مل من الكوليسترول 5 ملغ / مل المذاب في الإيثانول 100 ٪ (يسخن إلى درجة حرارة الغرفة) ، و 25 مل من 1 M KH2PO4 (الرقم الهيدروجيني = 6.0). اخلطي باستخدام لوحة تحريك مغناطيسية. يمكن إجراء الخلط لمدة 1 دقيقة عند 700 دورة في الدقيقة.
- يسكب حتى يملأ الخليط طبق 10 سم بأكمله. بدلا من ذلك ، استخدم ماصة مصلية متدرجة لإضافة ما يقرب من 20 مل من الخليط لكل لوحة.
- اترك الألواح لتجف لمدة 24 ساعة في درجة حرارة الغرفة قبل البذر بالبكتيريا أو قم بتخزين الألواح العادية عند 4 درجات مئوية بعد التجفيف.
ملاحظة: تتم معالجة جميع مكونات الوسائط باستخدام تقنيات التعقيم. يجب تنفيذ الخطوات 1.3-1.4 في غطاء تدفق صفائحي.
2. إعداد أجار NGM مع FUDR في لوحات 24 بئر
- اتبع الخطوات من 1.1 إلى 1.3.
- تكملة NGM مع 5-فلورو-2′-ديوكسي يوريدين (FUDR) ومزيج لتحقيق تركيز نهائي من 100 ميكروغرام / مل.
ملاحظة: يمنع FUDR تكرار الحمض النووي ، ونتيجة لذلك ، يمنع تكاثر C. elegans عن طريق استهداف الخط الجرثومي وتكوين الأجنة ، مما يؤثر في النهاية على العمر. لذلك ، من المهم السماح للديدان بالتطور الكامل إلى شباب بالغين قبل الانتقال إلى لوحات تحتوي على FUDR.
تنبيه: FUDR سام ويجب التعامل معه وفقا لورقة بيانات السلامة الخاصة بالشركة المصنعة. - باستخدام مسدس ماصة ، قم بتوزيع 1 مل من NGM-FUDR في كل بئر.
ملاحظة: يمكن تسهيل هذه العملية باستخدام نظام آلي لصب اللوحات. - اترك اللوحة تجف لمدة 24 ساعة في درجة حرارة الغرفة قبل البذر بالبكتيريا أو تخزين الألواح العادية عند 4 درجات مئوية.
3. بذر لوحات: OP50 وبكتيريا اختبار إضافية
- لتحضير ثقافة E. coli OP50 بين عشية وضحاها، أضف 200 ميكرولتر من أليكوت بكتيري من مرق مجمد إلى قارورة Erlenmeyer سعة 500 مل تحتوي على 250 مل من مرق لوريا الطازج والمعقم (LB).
ملاحظة: يعتمد حجم الوسائط على عدد اللوحات التي تحتاج إلى بذر. لإعداد مزارع بكتيرية أخرى ، قم بتلقيح أنبوب زراعة 16 مل يحتوي على 5 مل من وسط النمو بالبكتيريا من المخزون المجمد باستخدام طرف ماصة دقيقة معقم. - احتضان بين عشية وضحاها في حاضنة 37 درجة مئوية ، تهتز عند 220 دورة في الدقيقة (دورات في الدقيقة).
ملاحظة: استخدم قوارير معقمة مع ما لا يقل عن ضعف حجم عمل الوسائط وختمها بورق الألومنيوم المعقم. تنفيذ خطوة التلقيح والاستغناء البكتيري باستخدام تقنيات التعقيم. - قم بتوزيع 1-2 مل من ثقافة E. coli OP50 بين عشية وضحاها على وسط كل لوحة NGM مقاس 10 سم. لا تحتاج هذه الثقافة إلى الانتشار حول لوحة NGM.
- اترك الألواح لتجف في درجة حرارة الغرفة قبل الاستخدام / التخزين.
ملاحظة: يمكن وضع الألواح المزروعة مع أغطية في غطاء محرك السيارة مع تدفق الهواء لتسهيل التجفيف.
4. زراعة وبذر الأطباق: 24 لوحة بئر
- قم بإعداد ثقافة بين عشية وضحاها من السلالات البكتيرية المطلوبة ، مع الالتزام بتعليمات الزراعة الموجودة في الخطوات 3.1-3.2.
- انقل 200 ميكرولتر من كل مزرعة بكتيرية إلى كل بئر من صفيحة من 24 بئرا تحتوي على أجار NGM. سيغطي حجم البكتيريا الذي تبلغ سعته 200 ميكرولتر منطقة الأجار بأكملها ، مما يزيد من كمية الطعام لضمان عدم تجنب الديدان للعشب البكتيري.
- اترك الألواح متشققة مفتوحة في خزانة السلامة البيولوجية (BSC) لتسهيل التجفيف. تحقق من الألواح بشكل دوري لمنع الجفاف المفرط وتغيير اتجاه اللوحة لتعزيز تدفق الهواء والتجفيف بشكل متساو. يجب أن تجف الألواح في غضون 5 ساعات.
ملاحظة: يجب تنفيذ أي عمل مع بكتيريا السلامة البيولوجية من المستوى 2 في BSCs معتمدة ومعتمدة من قبل لجنة السلامة الأحيائية المؤسسية.
5. تزامن العمر
ملاحظة: يجب تنفيذ جميع الخطوات باستخدام تقنيات التعقيم المناسبة (أي العمل بالقرب من اللهب أو داخل BSC).
- اغسل الخنثى الجرافيدي من ألواح OP50 مقاس 10 سم باستخدام محلول M9 المعقم بالفلتر (5.8 جم من Na2HPO4· 7H2O ، 3.0 g من KH 2 PO 4 ، 5 g من كلوريد الصوديوم ، 0.25 g من MgSO4 · 7H 2 O ، في 1 لتر من ddH2O).
- محلول ماصة M9 على اللوحة عدة مرات باستخدام ماصة مصلية من الزجاج المعقم أو البلاستيك لرفع الديدان من العشب البكتيري.
- جمع تعليق الدودة ونقل الحل إلى أنبوب مخروطي البوليسترين 15 مل.
- جهاز طرد مركزي عند 270 × جم ، درجة حرارة الغرفة (RT ، ~ 23 درجة مئوية) ، لمدة دقيقتين.
- استنشق باستخدام قارورة فخ فراغ وتخلص من supernatant ، تاركا حبيبات الدودة دون إزعاج.
- أعد تعليق الكريات في 5-10 مل من M9 لغسل الديدان وكرر الخطوات 5.2-5.3 مرتين.
- أضف 5 مل من محلول التبييض بنسبة 20٪ (8.25 مل من DDH2O ، 3.75 مل من 1M NaOH ، 3.0 مل من المبيض غير المبيد للجراثيم) إلى الأنبوب وقلبه باستمرار لإذابة الديدان. الديدان جاهزة للطرد المركزي بمجرد أن تذوب بالكامل تقريبا.
ملاحظة: تعتمد أوقات التبييض وحجم محلول التبييض على حجم العينة. يعد التبييض الزائد والأقل من الأخطاء الشائعة. على هذا النحو ، تتطلب هذه العملية عموما التحسين لتحديد متى تكون العينة جاهزة للطرد المركزي. - جهاز طرد مركزي لمدة 2 دقيقة عند 423 × g والتخلص من supernatant.
- أضف 10 مل من M9 المعقم لإعادة تعليق حبيبات البيض.
- جهاز طرد مركزي للأنبوب لمدة 2 دقيقة عند 423 × جم لتكوير البيض. قم بإزالة السوبرناتانت باستخدام قارورة شفطاء.
- كرر الخطوات 5.6-5.6.1.
- أعد تعليق حبيبات البيض في 5 مل من M9 المعقم وضعها على مكسرات طوال الليل في درجة الحرارة المطلوبة.
ملاحظة: ستكون يرقات L1 المتزامنة مع العمر جاهزة للنقل إلى اللوحات في اليوم التالي.
6. إعداد دودة بعد تزامن العمر
- الطرد المركزي للديدان المتزامنة مع العمر عند 270 × g لمدة 3 دقائق عند RT (~ 23 درجة مئوية).
- قم بشفط السوبرناتانت في بيئة نظيفة ، مثل غطاء محرك التدفق أو بجوار موقد بنسن. اترك ما يقرب من 200 ميكرولتر من supernatant وأعد تعليق الديدان.
- باستخدام ماصة دقيقة ، انقل تعليق الدودة المركزة إلى ألواح NGM مقاس 10 سم التي تم زرعها مسبقا باستخدام OP50.
ملاحظة: يمكن لكل طبق أن يدعم 1500 دودة دون نفاد الطعام. ومع ذلك ، قد يتطلب هذا التركيز تعديلا اعتمادا على كثافة العشب البكتيري ودرجة حرارة النمو. يوصى باستخدام لوحات متعددة لمنع الديدان من الجوع. من المهم ملاحظة أن هذه اللوحات يجب ألا تحتوي على FUDR. - اترك الألواح لتجف ؛ ثم عكس وتخزينها في 25 درجة مئوية لمدة 48 ساعة.
ملاحظة: اعتمادا على حالة الشاشة، يمكن وضع الديدان من الخطوة 6.1 مباشرة على لوحات الاختبار (التي تحتوي على بكتيريا أو أدوية أو مركبات اختبار). إذا كانت حالة الاختبار المطلوبة تؤثر على النمو ، فيجب زراعة الديدان على NGM التي تحتوي على OP50 حتى الشباب (~ 48 ساعة) قبل تعريضهم لظروف الاختبار. - بعد حضانة 48 ساعة ، اغسل الديدان من الألواح بمحلول M9 معقم وضعها في أنابيب مخروطية.
ملاحظة: سوف تغرق الديدان البالغة في قاع الأنبوب. سيختلف الوقت الدقيق وفقا لعدد الديدان المستردة من ألواح 10 سم. في ظل هذه الظروف ، تستقر الديدان في غضون 10 دقائق. - قم بإجراء فحص بصري لتحديد مدة وقت الاستقرار بحيث تتم إزالة أي بيض متبقي أو يرقات فقس.
- أضف 10 مل إضافية من M9 لشطف البكتيريا المتبقية من أجسام الديدان.
- الطرد المركزي للديدان لمدة 2-3 دقائق عند 270 × غرام عند 23 درجة مئوية.
- قم بتنفيذ خطوة الغسيل 3 مرات إضافية. للحصول على أفضل النتائج ، اترك حوالي 1-1.5 مل من محلول M9 في الأنبوب بعد الغسيل النهائي.
- انقل 10 ميكرولتر من تعليق الدودة إلى شريحة زجاجية واحسب عدد الديدان.
- اضبط كثافة الدودة على حوالي 150 دودة لكل 10 ميكرولتر من M9. يمكن ضبط تركيز الديدان في التعليق إما عن طريق إزالة أو إضافة محلول M9 بعد الطرد المركزي.
- تأكد من أن التركيز المطلوب قد تم تحديده عن طريق حساب الأعداد من عدة قطرات مختلفة. يوصى بمتوسط الأعداد من ثلاث قطرات على الأقل.
- باستخدام تقنيات التعقيم ، قم بنقل 10 ميكرولتر من تعليق الدودة الذي يحتوي على حوالي 150 دودة إلى كل بئر من لوحة الاختبار.
- افحص الآبار تحت المجهر للتأكد من أن كل منها يحتوي على عدد كاف من الديدان. يمكن إضافة ديدان إضافية قبل الحضانة.
- اترك الألواح لتجف لمدة 10 دقائق تقريبا ؛ ثم عكس ونقل إلى حاضنة 25 درجة مئوية لمدة 72 ساعة.
ملاحظة: يمكن تعديل فترة الحضانة النهائية لاستيعاب احتياجات التجربة. وقت الحضانة البالغ 72 ساعة كاف لدعم نمو 150 حيوانا تتغذى على بكتيريا 200 ميكرولتر في صفيحة من 24 بئرا عند 25 درجة مئوية.
7. تحضير الديدان للتصوير
- لتسهيل تسوية أكثر فعالية وتقليل فقدان العينات، شل حركة الديدان قبل الغسيل لمنع السباحة.
ملاحظة: إذا كنت تعمل مع عدد قليل من العينات، يمكن تحقيق ذلك من خلال التعرض لليفاميزول (100 ميكرومتر). ومع ذلك ، إذا كنت تعمل مع عدد كبير من العينات ، يمكن شل حركة الديدان عن طريق التجميد. بالإضافة إلى ذلك ، فإن التجميد الممتد (18-24 ساعة) سيمنع زيادة تطوير مجاميع polyQ أثناء التحضير.- ضع لوحات متعددة الآبار عند -20 درجة مئوية لمدة 15-20 دقيقة أو حتى تتوقف الديدان عن الحركة.
- قم بإزالة العينات من الفريزر واتركها تجلس لمدة 5 دقائق.
- باستخدام ماصة دقيقة ، أضف 1 مل من M9 ، مبردة إلى 4 درجات مئوية ، إلى بئر مثير للاهتمام ، اضغط مرارا وتكرارا على المكبس وخفضه 4-6 مرات لغسل الديدان في كل بئر.
ملاحظة: في بعض الأحيان ، يمكن للديدان التمسك بنصائح الماصة الدقيقة. على هذا النحو ، يجب استخدام نصائح مختلفة بين كل بئر لمنع اختلاط الديدان. - انقل تعليق الدودة إلى أنبوب جهاز طرد مركزي دقيق واسمح للديدان بالغرق في القاع.
- شفط والتخلص من supernatant. اغسل العينة ثلاث مرات.
- بعد أن تستقر الديدان في القاع أثناء الغسيل النهائي ، تستنشق 500 ميكرولتر من supernatant ، تاركة 500 ميكرولتر لإعادة تعليق الديدان.
- انقل تعليق الدودة المتبقي إلى صفيحة جديدة مسطحة القاع من 24 بئرا وضعها في ثلاجة -20 درجة مئوية لمدة 48 ساعة.
ملاحظة: يقلل تجميد الديدان من التألق في الخلفية ويسمح بتصور أفضل للمجاميع.
8. التصوير
- قم بإزالة الألواح من الفريزر واتركها تذوب ، وامسح التكثيف الزائد ، وأزل الغطاء قبل التصوير.
ملاحظة: تختلف تفاصيل التقاط الصور وفقا للمعدات والبرامج المستخدمة. وينبغي ألا تكون بروتوكولات هذا القسم إلا كدليل، ومن المتوقع إجراء تعديلات. مطلوب أيضا لالتقاط الصور بتنسيق ملف tiff. - أثناء التقاط الصورة ، استخدم إعدادات المجهر التالية: وقت التعرض ، 500 مللي ثانية ؛ تكبير 40x مع محول كاميرا 0.63x (25.2x) ، تم ضبط كثافة GFP على 100٪.
ملاحظة: يمكن لتكوينات وأنظمة المجهر المختلفة الحصول على صور مختلفة عن تلك المقدمة في قسم النتائج. لتحقيق وصف أكثر عالمية للصور المطلوبة ، يتم توفير المزيد من التفاصيل الموضوعية المتعلقة بالصور. يجب أن يتراوح قطر الركام بين 1.0-10.0 بكسل مع كثافة فلورسنت تتراوح بين 0.10-1.0 (وحدات عشوائية ، مقياس 0-1) ليتم تحديدها بشكل صحيح بواسطة CellProfiler. الخلفية الفلورية لهذه الصور المجمعة أقل عموما من عتبة 0.10. بالنسبة للصور الساطعة، تتراوح شدة الدودة من 0.7-1.0 مع كثافة خلفية تتراوح من 0.1-0.2. يتراوح متوسط طول الديدان في صور برايتفيلد من 250 بكسل إلى 350 بكسل في الطول من الرأس إلى الذيل. - اضبط عناصر التحكم في الضوء المنقول حتى تظهر الديدان مضاءة بشكل ساطع مقارنة بالخلفية المظلمة. تجنب التعرض المفرط; سوف يزيد من حجم الدودة.
- قد يكون من الضروري تغيير مواقع الديدان داخل البئر لمنع التكتل المفرط. تفريق الديدان المتكتلة باستخدام طرف ماصة.
- اضبط القناة على GFP لإنشاء مستوى بؤري لكلتا الصورتين.
ملاحظة: من الضروري تحديد المستوى البؤري في قناة GFP. سيؤدي أي تغيير في التركيز البؤري يتم إجراؤه أثناء التقاط صور ساطعة إلى اختلال محاذاة المجاميع والديدان أثناء تحليل الصورة. - التقط صورة ساطعة والتقط على الفور صورة الفلورسنت المقابلة لها دون إزعاج اللوحة.
- قم بتشغيل صور الاختبار عبر خط الأنابيب لتحديد قيم الكثافة والحجم للكائنات الموجودة في الصورة وفقا ل "NOTE" بعد الخطوة 8.2.
ملاحظة: يمكن ل CellProfiler توفير كافة المعلومات الضرورية المتعلقة بكثافة وطول الكائنات قبل التحليل. - لتقييم الصور ، أولا ، قم بتنزيل CellProfiler17. افتح البرنامج واسحب الصور ذات الأهمية وأفلتها في مربع الصور.
- انقر على الصور في قائمة الملفات لفتحها.
- في الزاوية العلوية اليسرى من الشاشة توجد العديد من الرموز. استخدمها لقياس حجم الكائنات أو تكبير منطقة معينة. حدد زجاج المكبر لتسليط الضوء على منطقة مثيرة للاهتمام.
- حدد أيقونة السهم لقياس طول كل من الديدان والمجاميع.
- مرر مؤشر الماوس فوق الكائن المطلوب لتحديد قيمة شدته التي يمكن رؤيتها في الجزء السفلي من الشاشة.
ملاحظة: نظرا لأن جميع الصور مأخوذة بتدرج رمادي، ستكون جميع قيم البكسل الأحمر والأزرق والأخضر متطابقة.
9. تحليل الصور
- لاستخدام خط أنابيب تحليل صورة CellProfiler، قم بتسمية أزواج الصور بشكل صحيح. استخدم التنسيق التالي: P1_A01_S1_C1، حيث يشير P1 إلى اللوحة المحددة والتسمية الرقمية الخاصة بها؛ "A" ، يشير إلى الصف و "01" إلى العمود ؛ يشير "S" إلى زوج صور محدد لبئر واحد ؛ يشير "C" إلى القناة ، حيث يتم استخدام "1" للدلالة على صور brightfield و "2" للصور الفلورية.
- قم بتنزيل CellProfiler (الإصدار 4.1.3 أو أعلى) من الموقع الرسمي17.
- قم بتنزيل خط الأنابيب (الملف التكميلي 1). قم بتحميل خط الأنابيب (خط الأنابيب) إلى CellProfiler عن طريق تحديد ملف > استيراد > خط أنابيب من ملف.
ملاحظة: تم تعطيل الوحدتين 2 و3 لأنهما اعتبرتا غير ضروريتين. ويمكن استعادة وظيفتها إذا لم يسفر تحليل الصور عن فصل الديدان وتحديد هويتها بصورة مرضية؛ ومع ذلك ، يلزم تعديل خط الأنابيب. - لدمج هذه الوحدات، حدد المربعات الموجودة على يسار كل وحدة. إعادة تسمية الصورة الثنائية الإدخال للوحدة النمطية "UntangleWorms" إلى صورة الإخراج من الوحدة النمطية "convertobjectstoimage".
- قد تظهر رسالة خطأ أثناء الاستخدام الأولي المتعلق بالوحدة النمطية 2. في حالة حدوث ذلك ، تابع التحليل.
- قم بتحميل مجموعة تدريب تستخدم لتحديد الديدان إلى وحدة "UntangleWorms". يمكن العثور على مجموعة التدريب هذه في الملف التكميلي 2.
- حدد الوحدة النمطية UntangleWorms لفتح إعداداتها.
- حدد اسم ملف مجموعة التدريب وحدد رمز تحميل الملف.
- تحميل الملف التكميلي 2 (مجموعة التدريب).
- قم بتحميل الصور عن طريق تحديد وحدة الصور في الزاوية العلوية اليمنى. اسحب الصور المسماة بشكل صحيح وأفلتها كما هو موضح في الخطوة 9.1.
- قبل تحليل الصور، حدد مجلد الإخراج المطلوب الذي سيتم استخدامه لتخزين النتائج.
- انقر فوق الزر إعدادات الإخراج الموجود بالقرب من الزاوية السفلية اليسرى من البرنامج.
- حدد رمز المجلد الموجود على يمين الإخراج الافتراضي لاختيار موقع الإخراج المطلوب.
- حدد أيقونة تحليل الصور لبدء تحليل الصور .
- إذا استغرق التحليل وقتا طويلا لإكماله وتوقف في معالجة صورة واحدة ، فمن المحتمل أن يكون ذلك بسبب مشكلات في الحصول على الصورة. في حالة حدوث ذلك، قم بإجهاض التشغيل وتابع لتحديد الصورة (الصور) غير المعالجة عن طريق الفرز عبر مجلد الإخراج وملاحظة أسماء الصور التي لم يتم العثور عليها.
ملاحظة: قد تتطلب الصور معالجة إضافية لإزالة القطع الأثرية الكبيرة أو المضاءة بشكل مكثف والتي لا يمكن معالجتها بواسطة خط الأنابيب. وفي بعض الحالات، قد يلزم استبعاد هذه الصور من التحليل. - بعد التحليل الكامل ، سيقوم البرنامج بتنظيم النتائج في جدول بيانات Excel يحتوي على ديدان فردية (العمود N) وعدد المجاميع الخاصة بها (العمود K).
ملاحظة: سيؤدي التشغيل الناجح إلى إنتاج جدول بيانات Excel يحتوي على عدد المجاميع لكل دودة محددة لكل بئر. يمكن التلاعب بهذه البيانات بطريقة يحددها المجرب. - قم بتنزيل منظم بيانات التعريف (واجهة المستخدم الرسومية) من الملف التكميلي 3 (Windows) أو الملف التكميلي 4 (Mac) لتنظيم البيانات بسهولة من ملف CSV الناتج من CellProfiler.
ملاحظة: لا يحتوي هذا البرنامج على ترخيص رسمي ولن يتم فتحه تلقائيا بعد التنزيل. - اتبع الخطوات 9.14-9.17 في حالة استخدام نظام التشغيل Windows OS 64 بت. لن يعمل البرنامج على نظام التشغيل Windows 32 بت. اتبع الخطوات 9.18-9.20 في حالة استخدام نظام التشغيل Mac OS. الخطوات 9.21-9.22 هي نفسها لكلا نظامي التشغيل.
- بالنسبة لنظام التشغيل Windows ، حدد موقع الملف الذي تم تنزيله واستخراجه إلى الموقع المطلوب.
- حدد موقع المجلد المستخرج وفتحه المسمى gui_windowsOS_64x وقم بتشغيل التطبيق بالنقر فوق رمز التطبيق "gui".
- قد يتم فتح مطالبة تطلب إذنا بالتشغيل. حدد مزيد من المعلومات ثم انقر فوق الثقة على أي حال.
- منظم بيانات التعريف جاهز لسحب وإسقاط ملفات CSV الناتجة عن CellProfiler. تابع إلى الخطوة 9.21.
- بالنسبة لنظام التشغيل Mac OS، حدد موقع الملف الذي تم تنزيله وافتح gui_macOS_64x.zip. يجب أن تقوم هذه الخطوة تلقائيا باستخراج كافة الملفات.
- افتح المجلد المستخرج الموجود في "التنزيلات".
- انقر بزر الماوس الأيمن على تطبيق "gui" وحدد فتح. ستظهر مطالبة تطلب الإذن بالفتح بسبب عدم وجود ترخيص رسمي. حدد فتح وتابع إلى الخطوة 9.21.
- انقر فوق تحميل ملفاتك هنا أو اسحب وأفلت ملفات CellProfiler CSV المطلوبة.
- انقر فوق الزر " تنظيم" ، والذي سيجلب المستخدم إلى شاشة جديدة باستخدام زر تنزيل الملفات . انقر فوق الزر وحدد الموقع المطلوب لحفظ ملف الإخراج. سيظهر ملف الإخراج كاسم الملف الأصلي مع إضافة امتداد "_organized" إلى اسم الملف.
النتائج
الموصوف هنا هو سير عمل C. elegans الذي يتضمن بروتوكولات الاستزراع والحصول على الصور والمعالجة التي تسمح بتقييم تجميع polyQ في وجود بكتيريا مختلفة باستخدام تنسيق لوحة 24 بئرا كمنصة للزراعة والتصوير (الشكل 1). يمكن تعديل هذا البروتوكول لدراسة تأثير البكتيريا أو الظروف المحددة أو الجزيئات الصغيرة أو الأدوية أو التلاعب الجينومي على البروتيوستاسيس المضيف. تم تحسين الطريقة الموصوفة باستخدام الديدان التي تعبر بشكل أساسي عن polyQ المعوي المنصهر في بروتين فلورسنت أصفر (vha6p::p olyQ44::YFP) ؛ ومع ذلك ، يمكن أيضا استخدام النماذج الأخرى التي تبلغ عن البروتيوستاسيس في العضلات أو الخلايا العصبية مع مزيد من التحسين. على سبيل المثال ، توضح التجارب الأولية تطبيق هذه الطرق في تحديد كمام البروتين في الأنسجة الأخرى مثل polyQ العضلي (الشكل التكميلي 1). ومع ذلك ، ستكون هناك حاجة إلى تعديل خط الأنابيب لضبط الحجم الكلي والسطوع بشكل صحيح ، كما هو مذكور في القسم 8 ملاحظة.
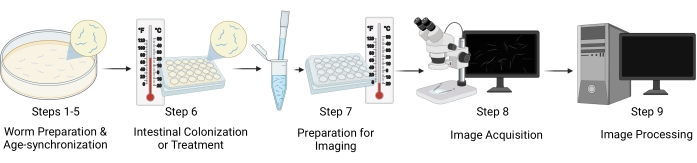
الشكل 1: التمثيل المرئي لسير العمل. وتشمل الخطوات الرئيسية للبروتوكول خمس مراحل متميزة: إعداد الدودة وتزامن العمر (الخطوات 1-5)، والاستعمار المعوي/علاج الدودة (الخطوة 6)، وإعداد العينات للتصوير (الخطوة 7)، والحصول على الصور (الخطوة 8)، ومعالجة الصور (الخطوة 9). يشار إلى "أقسام" البروتوكول باسم "الخطوات" في الشكل. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
كشفت تجارب التحسين الأولية عن صعوبات مختلفة مرتبطة بالاكتظاظ بسبب عدد كبير من النسل ، مما أدى إلى نضوب الطعام بشكل أسرع. وقد أدى تكميل FUDR في ألواح NGM الموصوفة في القسم 2 إلى حل هذه المشكلة (الشكل 2). بالإضافة إلى ذلك ، في وجود FODR ، كان للديدان التي تم تغذيتها ببكتيريا مختلفة حجم جسم أكثر اتساقا ، مما سمح باكتشاف أكثر اتساقا ودقة للديدان.
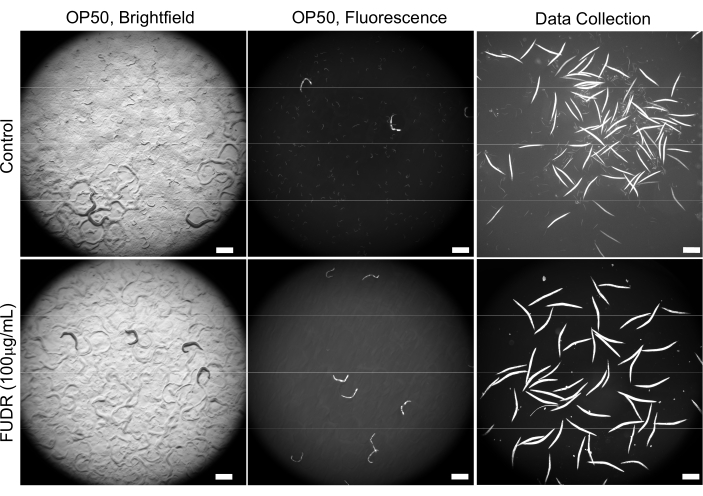
الشكل 2: استخدام FUDR يحسن جودة الصورة عن طريق تقليل النسل. تقضي الألواح المكملة ب FODR على ذرية C. elegans مقارنة بالديدان المزروعة على ألواح NGM غير الخاضعة لسيطرة FUDR المبذرة بالإشريكية القولونية OP50. تم الحصول على الصور بتكبير 25.2x (تكبير 40x مع محول كاميرا 0.63x). أشرطة المقياس = 500 ميكرومتر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
ساهم التألق في الخلفية في الكشف الإيجابي الخاطئ عن مجاميع polyQ. للحد من هذه الإشارة الفلورية في الأمعاء وتحسين الكشف الآلي عن المجاميع ، كان من الضروري تجميد الديدان قبل التصوير. أدى تجميد الديدان عند -20 درجة مئوية لمدة 18-48 ساعة إلى تحسين كبير في الكشف عن ركام polyQ عن طريق القضاء على التألق في الخلفية (الشكل 3A). العين البشرية قادرة على التمييز بين المجاميع والتألق في الخلفية. وبالتالي فإن العد اليدوي قبل التجميد وبعده هو نفسه (الشكل 3B). ومع ذلك، فإن العد الآلي ليس دقيقا، ولكن تجميد العد الآلي حسن بشكل كبير بدقة مماثلة للعد اليدوي (الشكل 3 ب).

الشكل 3: التجميد يحسن الكشف الكلي. (أ) صور الفلورسنت ل C. elegans التي تعبر عن polyQ44::YFP المعوية قبل وبعد التجميد. تمثل الإدراجات صورا مقربة للمنطقة المحددة. أشرطة المقياس = 500 ميكرومتر (B) متوسط عدد المجاميع لكل أمعاء في الديدان المستعمرة باستخدام P. aeruginosa MPAO1 قبل وبعد التجميد باستخدام القياس الكمي اليدوي أو الآلي (خط الأنابيب). تمثل البيانات نسختين بيولوجيتين متماثلتين (n = 60-109). تم حساب الدلالة الإحصائية باستخدام اختبار t للطالب (**** p < 0.0001). تمثل أشرطة الخطأ الخطأ القياسي للمتوسط (SEM). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
تم استخدام إضاءة الحقل الساطع المقلوبة للكشف عن كامل C. elegans (الشكل 4A) وقناة GFP لتصوير مجاميع polyQ44::YFP (الشكل 4B). تم الكشف عن الدودة وفك تشابكها وتكميمها التجميعي لكل دودة من خلال تطبيق خط أنابيب معالجة صور CellProfiler محسن (الملف التكميلي 1) ، والذي يسمح بالحصول على عدد المجاميع لكل دودة فردية (الشكل 4C-D). لاختبار جدوى هذا النهج ودقة الكشف الآلي عن الركام وتحديد كميته ، تم استزراع الديدان التي تعبر عن polyQ44::YFP الخاص بالأمعاء وإعدادها للتصوير وفقا للبروتوكولات المعمول بها (الأقسام 1-7). تم تقييم عدد المجاميع لكل دودة إما باستخدام خط الأنابيب الآلي (الأقسام 8-9) أو العد اليدوي. أجريت كل تجربة في ثلاث تجارب مستقلة باستخدام 90-571 ديدان لكل حالة. في حين أن متوسط عدد المجاميع لكل أمعاء تم الحصول عليها من خلال تجربتين لم يكن له فرق كبير ، فإن الديدان في التجربة الثالثة كان لديها مجاميع أقل بكثير عند تحديدها كميا باستخدام النهج الآلي (الشكل 5A). وأدى متوسط عدد المجاميع من التجارب الثلاث إلى مجاميع طفيفة، ولكن أقل بكثير عندما تم إجراء القياس الكمي باستخدام خط أنابيب CellProfiler (الأقسام 8-9) (الشكل 5B). ومع ذلك ، كان الفرق بين النهجين ضئيلا ، مما يشير إلى أنه يمكن تطبيق الطريقة الآلية على الشاشات واسعة النطاق.
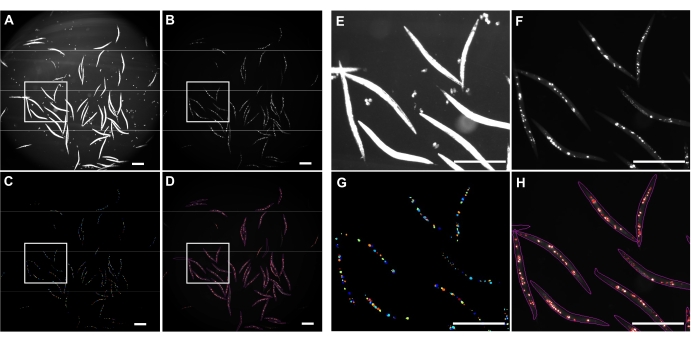
الشكل 4: الكشف الكلي باستخدام CellProfiler. (A) صورة Brightfield المستخدمة لتحديد أجسام الدودة. (ب) صورة الفلورسنت الأصلية التي تم الحصول عليها باستخدام قناة GFP واستخدامها لتحديد وقياس العدد الإجمالي لمجاميع polyQ44::YFP المعوية. (ج) المجاميع المحددة باستخدام CellProfiler. (د) العدد الإجمالي للمجاميع المحددة المتراكبة فوق الصورة الفلورية الأصلية مع الخطوط العريضة للدودة والتجميع. تم التقاط الصور ومعالجتها باستخدام الإعدادات الموضحة في الأقسام 8-9. تمثل اللوحات E-H صورا مقربة للمناطق المحددة المقابلة في الصور A-D. تم الحصول على الصور بتكبير 25.2x (تكبير 40x مع محول كاميرا 0.63x). أشرطة المقياس = 500 ميكرومتر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
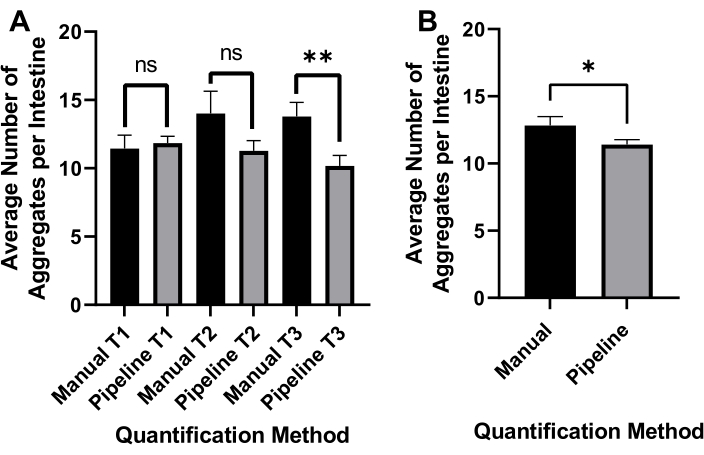
الشكل 5: فعالية القياس الكمي الركامي الآلي. (أ) متوسط العدد الإجمالي لكل أمعاء في الديدان المستعمرة بالتحكم في الإشريكية القولونية OP50 باستخدام العد اليدوي (اليدوي) والقياس الكمي الآلي القائم على CellProfiler (خط الأنابيب). تمثل النتائج البيانات التي تم تحليلها في ثلاث تجارب منفصلة (T1-T3) (n = 90-571). (ب) تم الحصول على متوسط عدد الركام لكل أمعاء باستخدام القياس الكمي اليدوي أو الآلي (خط الأنابيب). تم حساب الدلالة الإحصائية باستخدام اختبار t للطالب (* p < 0.05 ، ** p < 0.01). تمثل أشرطة الخطأ SEM. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
لتقييم قابلية تكرار النتائج بين المجربين المختلفين ، تم الحصول على صور من صفيحة تحتوي على ستة آبار من الديدان التي تم تغذيتها إما Pseudomonas aeruginosa MPAO1 أو Escherichia coli OP50 ، من قبل ثلاثة أفراد ، اثنان منهم لم يكن لديهم خبرة سابقة في تصوير الديدان باستخدام هذه البروتوكولات. الصور التي تم جمعها من كل بئر تحتوي على ما بين 30-115 من الديدان المكتشفة. تم الكشف عن اختلاف غير كبير في التجميع في الديدان من نفس الآبار التي تم تصويرها من قبل المجربين الثلاثة. في حين ظل متوسط عدد المجاميع لكل أمعاء ثابتا جدا بين المجربين الثلاثة للديدان التي تتغذى على MPAO1 و OP50 ، كانت هناك بعض الاختلافات ذات الدلالة الإحصائية في متوسط عدد المجاميع ولكن فقط في الديدان التي استعمرها MPAO1 (الشكل التكميلي 2). تسلط هذه النتائج الضوء على قابلية تكرار النتائج حتى بين المجربين عديمي الخبرة.
لضمان عدم تأثر قابلية تكرار القياس الكمي الكلي بشكل كبير بموضع الدودة ، تم اختيار مجموعة من 15 دودة وتصويرها 15 مرة منفصلة بعد الإثارة بين كل التقاط صورة باستخدام طرف ماصة. تم جمع وتحليل صور الركام في الديدان التي تتغذى على E. coli OP50 و P. aeruginosa MPAO1 باستخدام CellProfiler. وكان متوسط عدد المجاميع من كل مجموعة من هذه المجموعات المختلفة من الصور مختلفا اختلافا طفيفا ولكن غير كبير، مما يدعم أيضا إمكانية تكرار هذا النهج (الشكل التكميلي 3).
وقد ثبت أن استعمار أمعاء C. elegans مع مسببات الأمراض المعوية سالبة الجرام يعطل البروتيوستاسيس عبر الأنسجة ، مع كون P. aeruginosa من بين أقوى محفزات تراكم polyQ9. لتحديد ما إذا كانت هذه البروتوكولات المحسنة ستنجح في اكتشاف وقياس تعزيز التجميع بوساطة P. aeruginosa ، تم استعمار الديدان التي تعبر عن polyQ المعوي باستخدام E. coli OP50 (بكتيريا التحكم) ، و P. aeruginosa MPAO1 ، تم إجراء الأقسام 1-8. تم تحليل الصور التي تم الحصول عليها باستخدام CellProfiler (القسم 9 ، الملف التكميلي 1). تظهر نتائج القياس الكمي الآلي زيادة كبيرة في عدد المجاميع التي تسببها P. aeruginosa MPAO1 ، مما يؤدي باستمرار إلى تعزيز مزدوج مقارنة بالديدان التي تتغذى على التحكم E. coli OP50 (الشكل 6).

الشكل 6: متوسط عدد المجاميع لكل أمعاء في الديدان المستعمرة بالسيطرة على الإشريكية القولونية OP50 و P. aeruginosa MPAO1. تم تقييم عدد المجاميع لكل أمعاء باستخدام CellProfiler (الأقسام 8-9). يتم تمثيل البيانات على أنها متوسط عدد المجاميع لكل أمعاء في الديدان المستعمرة باستخدام OP50 (n = 1068) و MPAO1 (n = 1557). تم حساب الدلالة الإحصائية باستخدام اختبار t للطالب (**** p < 0.0001). تمثل أشرطة الخطأ SEM. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
تم تصميم خط الأنابيب المحسن لدعم الشاشات واسعة النطاق للظروف التي تؤثر على البروتيوستاسيس. لاختبار جدوى هذا النهج في فحص المكتبات الكبيرة من البكتيريا لتأثيرها على البروتيوستاسيس المضيف ، تم استخدام خط الأنابيب الموصوف هنا (الأقسام 1-9) لاختبار تأثير 90 P. aeruginosa السلالات المتحولة الجينية غير الأساسية على تجميع polyQ18. هذه الشاشة التجريبية هي جزء من مشروع أكبر مصمم لفحص جميع سلالات P. aeruginosa المتحولة غير الأساسية لقدرتها على التأثير على البروتيوستاسيس المضيف. من بين 90 سلالة بكتيرية تم اختبارها ، أظهر استعمار أمعاء C. elegans مع مرشح واحد انخفاضا كبيرا في عدد المجاميع (الشكل 7). تم إجراء تجارب المتابعة لتقييم حساسية هذا الفحص عن طريق العد الكلي اليدوي من اختيار عشوائي لستة طفرات P. aeruginosa التي تختلف اختلافا غير كبير عن عنصر التحكم MPAO1. تم إجراء هذه التجارب باستخدام لوحات NGM الأكثر تقليدية 6 سم ، ونقل الديدان إلى سلالات الاختبار مثل L1 لتلخيص الطرق المحددة مسبقا9. كشفت تجارب التأكيد عن طريق العد اليدوي أن أيا من الطفرات ، بما في ذلك تلك التي خفضت بشكل كبير عدد المجاميع (الشكل 7) ، لم تؤثر على تجميع polyQ (الشكل التكميلي 4). وبالإضافة إلى ذلك، لم تكتشف التغيرات الطفيفة في التجميع التي لوحظت في شاشة السلالات المتحولة البالغ عددها 90 سلالة بين العد اليدوي للمرشحين المختارين، مما يشير إلى أن هذه التغييرات يمكن أن تنشأ بسبب التباين البيولوجي والتجريبي، مثل انخفاض قيمة n. بشكل جماعي ، تشير النتائج إلى أنه في حين أن طريقتنا يمكن أن تلتقط تغييرات كبيرة بشكل موثوق ، فمن المحتمل أن يتم تفويت التغييرات الدقيقة ، ويجب تأكيد جميع المرشحين المحتملين بشكل فردي.
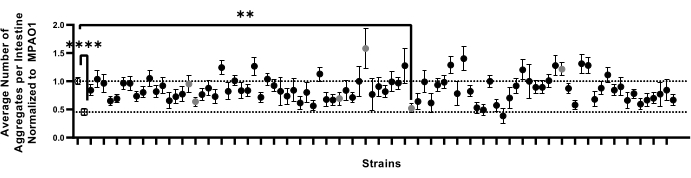
الشكل 7: عدد المجاميع لكل أمعاء في مجموعة عينة تمثيلية من الديدان المستعمرة بواسطة 92 سلالة بكتيرية. يتم تمثيل البيانات على أنها متوسط عدد المجاميع لكل أمعاء طبيعية إلى تلك الموجودة في الديدان المستعمرة باستخدام MPAO1. تمثل الخطوط المنقطة متوسط عدد المجاميع في الديدان المستعمرة باستخدام MPAO1 (أعلى ، دائرة مفتوحة) وتحكم OP50 (أسفل ، مربع مفتوح). تمثل الرموز الصلبة 90 سلالة متحولة مميزة من P. aeruginosa MPAO1. كان متوسط عدد المجاميع لكل دودة بين الديدان المستعمرة باستخدام MPAO1 ومتحور واحد ذا دلالة إحصائية. تمثل الدوائر الرمادية العينات التي تم تأكيدها يدويا (الشكل التكميلي 4). تم حساب الدلالة الإحصائية باستخدام تحليل أحادي الاتجاه للتباين (ANOVA) متبوعا بمقارنة متعددة اختبار دونيت اللاحق المخصص (** p < 0.01 ، **** p < 0.0001). تمثل أشرطة الخطأ SEM. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
لإدارة الكم الكبير من البيانات التي تم إنشاؤها بواسطة CellProfiler ، تم تطوير واجهة مستخدم رسومية (GUI) لأتمتة معالجة البيانات وتنظيمها (الشكل 8). تم تطوير واجهة المستخدم الرسومية باستخدام Tkinter ، وهي مجموعة أدوات أداة Python مفتوحة المصدر عبر الأنظمة الأساسية. من البيانات الوصفية المعطاة ، يستخرج التطبيق عدد المجاميع (العمود K) من كل بئر (العمود J) الموجود في لوحة. تم استخدام مكتبة معالجة بيانات بايثون تسمى "Pandas" لتنفيذ العملية المذكورة أعلاه. يوفر تطبيق واجهة المستخدم الرسومية دعم السحب والإفلات للمستخدمين لتحميل ملفات البيانات. يتم تخزين البيانات في كل ملف في شكل بنية جدولية ثنائية الأبعاد تسمى إطار البيانات. تتم تهيئة زوج قاموس فارغ لكل بئر فريد موجود داخل إطار البيانات. بعد ذلك ، يتم حساب المجاميع المميزة الموجودة في كل بئر وإلحاقها بأزواج القواميس الخاصة بها. العمود الذي يحتوي على بيانات أقل مبطن بسلاسل قيمة فارغة لضمان أن كل عمود متساو في الحجم. أخيرا ، يتم تحويل البنية إلى إطار بيانات يتم تصديره في شكل جدول بيانات إلى الدليل المحدد من قبل المستخدم.

الشكل 8: واجهة المستخدم الرسومية. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل التكميلي 1: الكشف عن مجاميع polyQ الخاصة بالعضلات. تم طلاء الديدان التي تعبر عن polyQ35::YFP الخاص بالعضلات على شكل L1s وتم استزراعها على OP50 لمدة 48 ساعة. بمجرد تطور الديدان إلى شباب بالغين ، تم نقلها إلى لوحات NGM 24 جيدا ، واستكملت ب 100 ميكروغرام / مل FUDR وزرعت مع MPAO1 لمدة 72 ساعة إضافية قبل التصوير. (أ) صورة برايتفيلد المستخدمة لتحديد أجسام الدودة. (ب) صورة فلورسنت أصلية تم الحصول عليها باستخدام قناة GFP. (ج) المجاميع المحددة باستخدام CellProfiler. (د) العدد الإجمالي للمجاميع المحددة المتراكبة فوق الصورة الفلورية الأصلية مع الخطوط العريضة للدودة والتجميع. تم التقاط الصور ومعالجتها باستخدام الإعدادات الموضحة في الأقسام 8-9. تمثل اللوحات E-H صورا مقربة للمناطق المحددة المقابلة في الصور A-D. أشرطة المقياس = 500 ميكرومتر.
الشكل التكميلي 2: إمكانية تكرار القياس الكمي الكلي بين مختلف المجربين. متوسط عدد الركام الذي تم تحديده كميا باستخدام CellProfiler لستة آبار من الديدان المستعمرة باستخدام P. aeruginosa MPAO1 (القضبان السوداء) وستة آبار من الديدان المستعمرة بالتحكم E. coli OP50 (القضبان الرمادية). تم تصوير كل بئر من قبل ثلاثة مجربين (AVS، DMC، RDH). يتم تمثيل البيانات على أنها متوسط عدد المجاميع لكل أمعاء (n = 30-115). تم حساب الدلالة الإحصائية باستخدام ANOVA أحادي الاتجاه متبوعا باختبار المقارنات المتعددة ل Tukey (* p < 0.05 ، ** p < 0.01). تمثل أشرطة الخطأ SEM. يرجى النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 3: تأثير موضع الدودة على قابلية تكرار القياس الكمي الكلي. متوسط العدد الإجمالي لكل أمعاء في الديدان المستعمرة مع السيطرة E. coli OP50 (القضبان الرمادية) و P. aeruginosa MPAO1 (القضبان السوداء). تمثل النتائج متوسط عدد المجاميع لكل أمعاء (15≥n≥12) كميا باستخدام CellProfiler. تم تغيير موضع الديدان داخل الآبار عن طريق الإثارة بين كل عملية استحواذ. لم يتم العثور على فروق ذات دلالة إحصائية في أي من المجموعتين. تم حساب الدلالة الإحصائية باستخدام ANOVA أحادي الاتجاه متبوعا باختبار المقارنات المتعددة ل Tukey. تمثل أشرطة الخطأ SEM. يرجى النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 4: تأكيد الشاشة التجريبية مع العد اليدوي. تم تحديد متوسط عدد المجاميع لكل أمعاء يدويا. تمثل البيانات ملفات تعريف تجميع الديدان المستعمرة مع ستة طفرات بالضربة القاضية MPAO1 (الدوائر الرمادية الشكل 7) مقارنة بعناصر التحكم MPAO1 و OP50 من النوع البري (n = 30). تم حساب الدلالة الإحصائية باستخدام ANOVA أحادي الاتجاه متبوعا بمقارنة متعددة لاختبار دونيت اللاحق المخصص (**** p < 0.0001). تمثل أشرطة الخطأ SEM. يرجى النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 5: تأثير FUDR على تراكم polyQ المعوي. يتم تمثيل البيانات على أنها متوسط عدد مجاميع polyQ44::YFP لكل أمعاء (n = 20). تم نقل الديدان إلى اللوحات المحتوية على FUDR (بدون FUDR) أو الصفائح المحتوية على FODR (100 ميكروغرام / مل) بعد 48 ساعة من النمو عند 25 درجة مئوية على E. coli OP50. تم جمع الأعداد اليدوية بعد 48 ساعة إضافية. تم حساب الدلالة الإحصائية باستخدام اختبار t للطالب (ns = غير معنوي). تمثل أشرطة الخطأ SEM. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 1: خط أنابيب Proteostasis. خط أنابيب تحليل الصور القابل للتنزيل للاستخدام في CellProfiler. يمكن العثور على تعليمات التطبيق في القسم 9. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 2: مجموعة التدريب على فك الدودة المتشابكة. الملف المراد تحميله في وحدة "UntangleWorms". مجموعة التدريب الخاصة هذه خاصة بالديدان المستخدمة في النهج الأولي. التغييرات في حجم الدودة وشكلها ستغير دقة وجودة تحديد الهوية. قد يكون من الضروري إنشاء ملف تدريب أكثر تخصيصا. يمكن العثور على تعليمات لإنشاء مجموعة تدريب جديدة على موقع CellProfiler الرسمي17. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 3: واجهة المستخدم الرسومية لنظام التشغيل Windows. gui_windowsOS_64x.zip. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 4: واجهة المستخدم الرسومية لنظام التشغيل Mac. gui_MacOS_64x.zip. يرجى النقر هنا لتنزيل هذا الملف.
Discussion
يحدد البروتوكول الموصوف إجراءات زراعة C. elegans وتصويرها ومعالجة الصور التي تتضمن CellProfiler ، وهو برنامج مفتوح المصدر لتحليل الصور. تظهر النتائج التمثيلية قابلية التكرار والحد من التحيز وقابلية التوسع. سيؤدي هذا الإجراء الموحد إلى تحسين استراتيجيات الفحص المستخدمة في المكتبات البكتيرية أو الجينومية أو الدوائية الكبيرة. في حين توجد طرق C. elegans آلية أخرى للكشف عن الكائنات ، فإن التقنية الموصوفة توفر خط أنابيب موحد أعلى إنتاجية يدمج بين الاستزراع والحصول على الصور والتحليل.
كان لا بد من اختبار العديد من الاختلافات في زراعة الديدان لتحسين البروتوكول الموصوف هنا. في البداية ، تم نقل الديدان إلى عينة من البكتيريا مباشرة بعد تزامن العمر (مرحلة L1). ومع ذلك ، أدى هذا النهج إلى مجموعة من الديدان ذات الأحجام المتغيرة ، حتى بين الديدان داخل نفس البئر. C. elegans معروفة بتجنب مسببات الأمراض19 ، والتي يمكن أن تسهم في التباين الملحوظ في الحجم وتؤثر في نهاية المطاف على الكشف عن دودة التصوير في المصب ، على وجه الخصوص. للقضاء على هذا التباين ، تم تغطية منطقة NGM بأكملها في كل بئر ببكتيريا الاختبار. علاوة على ذلك ، تم تغذية الديدان E. coli OP50 وسمح لها بالتطور الكامل إلى شباب بالغين لمدة 48 ساعة عند 25 درجة مئوية. السماح للديدان بالوصول إلى مرحلة البلوغ على E. coli OP50 قبل نقلها إلى بكتيريا الاختبار أدى إلى حجم جسم أكثر اتساقا. بالإضافة إلى ذلك ، تم القضاء على الاكتظاظ والنضوب السريع للأغذية من قبل النسل عن طريق استكمال أجار NGM مع FUDR. أدى تنفيذ FUDR إلى إزالة النسل وتعزيز التعرف الآلي على الدودة ، والذي تم حجبه عن طريق اختلاط النسل مع السكان الوالدين. ومع ذلك ، من المهم توخي الحذر واستخدام الضوابط المناسبة عند استخدام FODR ، حيث من المعروف أن المركب يؤثر على C. elegans proteostasis وعمر20,21. وفي ظل الظروف الموصوفة في هذا البروتوكول، لم يؤثر FUDR على تجميع polyQ المعوي (الشكل التكميلي 5)؛ لذلك ، كان استخدامه مناسبا ومفيدا للطريقة الموصوفة.
تبين أن تجميد العينات قبل التصوير كان خطوة حاسمة في التوظيف الناجح لخط الأنابيب. وكانت الأعداد الإجمالية قبل التجميد أعلى بكثير من العد اليدوي (الشكل 3 باء). أدى الحفاظ على الديدان عند -20 درجة مئوية لمدة 18-48 ساعة قبل التصوير إلى تقليل التألق في الخلفية وتحسين الكشف الكلي في نهاية المطاف (الشكل 3A). ولم يتم التحقيق في آثار التجميد على الكشف الكلي إلا بالنسبة ل polyQ ولا ينبغي تعميمها على نماذج أخرى دون مزيد من التحقيق في هذه الآثار.
وعلى الرغم من إبقاء جميع الظروف على حالها، لوحظ أن متوسط عدد المجاميع لكل دودة يمكن أن يختلف بين أشواط مختلفة، في حين أن النسبة بين عدد المجاميع في الحيوانات المستعمرة باستخدام OP50 مقابل MPAO1 ظلت ثابتة (الشكل 6، الشكل التكميلي 2، الشكل التكميلي 5). لذلك ، من الضروري دائما تضمين عنصر تحكم E. coli OP50 ، أو أي عناصر تحكم مرجعية إضافية مناسبة ، في كل تشغيل. ويمكن أن يتأثر هذا التباين في الأعداد الإجمالية بين التجارب بالظروف البيئية (درجة الحرارة والرطوبة)22،23 أو الخلفية الجينية8. في الواقع ، لوحظ أنه بعد زراعة طويلة ، انخفض التألق المعوي بشكل كبير أو فقد تماما ، مما تطلب ذوبان سلالة جديدة من المخزون المجمد. يمكن أن يكون الانخفاض الملحوظ في التألق نتيجة للتغيرات الجينية التي تقمع الجينات المحورة السامة ، مثل تلك التي تعبر عن polyQ. ومع ذلك، فإن التكرار الاستثنائي للنتائج التي لوحظت بين مختلف المجربين (الشكل التكميلي 2)، وبين النسخ المتماثلة البيولوجية (الشكل 5)، وداخل نفس العينة (الشكل التكميلي 3) يؤكد على قوة هذا النهج.
وقد استخدمت العديد من التقارير polyQ المعوية لدراسة البروتينات9،11،12،13،24،25. ومع ذلك ، لا يمكن إجراء مقارنة مباشرة بين النتائج بسبب التباين بين النهج التجريبية وطرق القراءة. ومع ذلك ، يتم تلخيص بعض النتائج من البيانات المنشورة سابقا من خلال القياس الكمي الآلي الموصوف هنا ، بما في ذلك الحث البكتيري للتجميع 9,13 وعدد مماثل من المجاميع 11. بشكل جماعي ، يوفر خط الأنابيب الموصوف أداة قيمة لدراسة البروتيوستاسيس.
الطريقة الموصوفة هنا لديها بعض التحديات المتأصلة. على سبيل المثال ، يتطلب وقتا كافيا لإتقان جميع مكونات هذا البروتوكول ، وهو ما ينطبق بشكل خاص على القسم 8 من البروتوكول ، والذي يتطلب الإلمام بالفحص لتحديد ما إذا كانت الصور التي تم الحصول عليها مناسبة لتحليل خط الأنابيب. الانحرافات عن إعدادات الحصول على الصور المستخدمة في هذا البروتوكول ممكنة. ومع ذلك ، من المحتمل أن تكون هناك حاجة إلى تعديل الإعدادات ومجموعة تدريب الدودة. يمكن لخط الأنابيب هذا التمييز بين المجاميع ذات الأحجام المختلفة وتلك التي تلمس ، مما يحد من "مزج" الركام ويزيد في النهاية من حساسية الكشف. ومع ذلك، قد تنشأ مشاكل عند محاولة تحديد المجاميع الكبيرة التي تتجاوز نطاق الحجم المقبول، لأن توسيع عتبة الحجم الأعلى قد يؤدي إلى أخطاء ناجمة عن سوء التحديد، مثل عدم القدرة على التمييز بين المجاميع التي تلمس. يجب إيجاد توازن بين الدقة والحجم والكثافة قبل تحليل الصورة. يمكن زيادة تحسين كفاءة تحديد الهوية الكلية من خلال دمج التعلم الآلي لإنشاء شبكة عصبية قادرة على تعزيز الكشف الكلي. ويجري حاليا استكشاف هذه التحسينات التي ستساعد كثيرا في معالجة القضايا الراهنة مثل الكشف عن المجاميع التي تقع على مستويات بؤرية مختلفة أو التي لها أشكال غير طبيعية.
أحد نقاط الضعف البارزة في الطريقة الموصوفة هو التباين في الأعداد المجمعة الآلية ، حيث لا يتم تلخيصها دائما عن طريق العد اليدوي في الديدان التي تتغذى على سلالات بكتيرية مختلفة. فعلى سبيل المثال، استنادا إلى الأعداد الآلية، كان لدى الديدان التي تتغذى على المتحور P. aeruginosa 53 (M53) مجاميع أقل بكثير مقارنة بالسلالة البرية (MPAO1) (الشكل 7)؛ ومع ذلك ، لم يظهر تأكيد الضربة أي فرق كبير (الشكل التكميلي 4). بشكل عام ، تحتوي شاشات الأدوية عالية الإنتاجية على معدل مرتفع للكشف عن الضربات الإيجابية الكاذبة ، والطريقة الموصوفة ليست استثناء26. وبالتالي ، يعد تأكيد جميع الضربات المحتملة جزءا مهما من البروتوكول.
في حين تم تحسين هذا البروتوكول ليناسب استراتيجية الفحص لتحديد البكتيريا التي تؤثر على البروتيوستاسيس المضيف ، يمكن تعديل كل خطوة بشكل أكبر لاختبار تأثير مكتبات الحمض النووي الريبي الجينومي أو الجزيئات الصغيرة أو غيرها من الحالات. يمكن إجراء تعديلات إضافية في كل خطوة لتناسب متطلبات استراتيجية فحص محددة. علاوة على ذلك ، توفر هذه التقنية مستوى من المرونة يسمح بتحسين كل خطوة لتناسب نموذجا معينا. على سبيل المثال ، يمكن توسيع هذا النهج ليشمل تجميع polyQ في أنسجة أخرى أو استخراج ميزات أخرى تم اكتشافها في الصور مثل مراقبة التعبير الجيني باستخدام مراسلي الفلورسنت المستحثين (على سبيل المثال ، جينات الصدمة الحرارية) ، وتقييم التوطين تحت الخلوي للبروتينات (على سبيل المثال ، التوطين النووي ل DAF-16) ، ودراسة التجميع في نماذج الأمراض الأخرى (Aβ1-42 ، α-synuclein ، TDP-43 ، إلخ) أو تقييم الأنماط الظاهرية الفسيولوجية ، مثل حجم الدودة.
Disclosures
وقد أعلن صاحبا البلاغ أنه لا يوجد تضارب في المصالح.
Acknowledgements
تم دعم هذا العمل من قبل المعاهد الوطنية للصحة (1RO3AG069056-01) وتمويل جمعية الأمراض المعدية الأمريكية ل DMC. لم يكن للممولين أي دور في تصميم الدراسة أو جمع البيانات وتحليلها أو اتخاذ قرار النشر أو إعداد المخطوطة. نشكر أعضاء مختبر Czyz على التدقيق اللغوي للمخطوطة. تم إنشاء شخصيات كرتونية باستخدام ترخيص BioRender المدفوع.
Materials
| Name | Company | Catalog Number | Comments |
| 1.7 mL Microtubes | Olympus Plastics | Cat#24-282 | Microcentrifuge tubes |
| 10 mL Serological Pipettes | GenClone | Cat#12-104 | Plastic pipettes |
| 15 mL Centrifuge Tubes, Racked | Olympus Plastics | Cat#28-101 | Conical tubes |
| 5-Fluoro-2′-deoxyuridine | Fisher Scientific | D2235100MG | FUDR |
| Accu-jet Pro Pipette Controller | Genesee Scientific | 91-600RB | Pipette gun |
| Agar | Fisher Scientific | Cat#BP1423-2 | Granulated agar |
| BioRender | BioRender | Graphical figure generator | |
| Bleach (Regular) | Clorox | Bar# 044600324111, Splash-less Bleach | 4.5% sodium hypochlorite |
| C. elegans: AM140: rmIs132 [unc-54p::q35::yfp] | Morimoto Lab (Northwestern University) | AM140, Q35::YFP | Muscle polyQ |
| C. elegans: AM738: rmIs297[vha-6p::q44::yfp; rol-6(su1006)] | Morimoto Lab (Northwestern University) | AM738, Q44::YFP | Intestinal polyQ |
| CaCl2·2H2O | Fisher Scientific | Cat#C79-500 | Calcium chloride dihydrate |
| CellProfiler | Broad Institute | Image analysis software | |
| Cholesterol | Fisher Scientific | Cat#ICN10138201 | Cholesterol |
| Circulating Water Bath Head | Lauda | 26LE | Lauda E 100 |
| CoolLED pE300lite 365 dir mount STEREO | CoolLED | 8114931 | Fluorescent light control and emitter |
| 16 mL Culture Tubes | Olympus Plastics | 21-129 | Culture tubes, 17 mm x 100 mm |
| Escherichia coli OP50 | Caenorhabditis Genetics Center | OP50 | E. coli control strain |
| Ethanol, 200 Proof | Decon Labs, Inc. | 2701 | |
| Eyepiece 10x/23B, adjustable, 3d gen | Leica Microsystems | 10450910 | Eyepiece set |
| Filter Set ET GFP - MZ10F | Leica Microsystems | 10450588 | Filter cube |
| GraphPad Prism v9.2.0 | GraphPad Software, Inc. | Statistical analysis tool | |
| Heratherm Incubator IMP180 | Thermo | 51031562 | Refrigerated incubator |
| Innova 4000 | New Brunswick Scientific | M1192-0000 | Shaking incubator |
| K5 Camera | Leica Microsystems | 11547112 | Stereomicroscope camera |
| KH2P04 | Fisher Scientific | Cat#P285-3 | Potassium phosphate monobasic |
| LAS X Imaging Software | Leica Microsystems | Microscope imaging software | |
| Leica MZ10 F Optics Carrier | Leica Microsystems | 10450103 | Stereomicroscope |
| Levamisole | Fisher Scientific | Cat#0215522805 | Levamisole hydrochloride |
| Luria Broth (Lennox) | Apex Bioresearch Products | Cat#11-125 | LB |
| Magnetic Stir Plate | Fisher Scientific | 11-100-49S | Stir plate |
| MgSO4·7H2O | Alfa Aesar | A14491 | Magnesium sulfate heptahydrate |
| Microscope Slides | Premiere | 8205 | Single frosted microscope slides |
| Na2HPO4·7H2O | Fisher Scientific | Cat#S373-500 | Sodium phosphate dibasic heptahydrate |
| NaCl | Fisher Scientific | Cat#S671-500 | Sodium chloride |
| NaOH | Fisher Scientific | Cat#S318-3 | Sodium hydroxide pellets |
| Objective Achromat, f = 100 mm | Leica Microsystems | 10411597 | Objective microscope lens |
| Petri Dishes | Genesee Scientific | Cat#32-107G | 100 mm x 15 mm |
| Pseudomonas aeruginosa Mutant Library | Manoil Lab (University of Washington) | P. aeruginosa mutant library | |
| Suspension Culture Plate 24-Well, Flat Bottom | Olympus Plastics | 25-102 | Used for worm growth and imaging |
| Trinocular Tube 100% M-series | Leica Microsystems | 10450043 | |
| Trypticase Peptone | ThermoFisher, Difco | Cat#211921 | |
| TX-400 Rotor | Thermo Scientific | Cat#75003181 | Swing bucket rotor |
| Vacuum Driven Filter System | GenClone | Cat#25-227 | 500 mL, PES Membrane, .22 µm |
| Video Objective with C-Mount | Leica Microsystems | 10447367 | 0.63x camera adapter tube |
References
- Soto, C. Unfolding the role of protein misfolding in neurodegenerative diseases. Nature Reviews Neuroscience. 4 (1), 49-60 (2003).
- Fang, P., Kazmi, S. A., Jameson, K. G., Hsiao, E. Y. The microbiome as a modifier of neurodegenerative disease risk. Cell Host & Microbe. 28 (2), 201-222 (2020).
- Kundu, P., Blacher, E., Elinav, E., Pettersson, S. Our gut microbiome: The evolving inner self. Cell. 171 (7), 1481-1493 (2017).
- Sherwin, E., Dinan, T. G., Cryan, J. F. Recent developments in understanding the role of the gut microbiota in brain health and disease. Annals of the New York Academy of Sciences. 1420 (1), 5-25 (2018).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7 (1), 13023 (2016).
- Guisbert, E., Czyz, D. M., Richter, K., McMullen, P. D., Morimoto, R. I. Identification of a tissue-selective heat shock response regulatory network. PLoS Genetics. 9 (4), 1003466 (2013).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438 (2011).
- Nollen, E. A., et al. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Walker, A. C., et al. Colonization of the Caenorhabditis elegans gut with human enteric bacterial pathogens leads to proteostasis disruption that is rescued by butyrate. PLOS Pathogens. 17 (5), 1009510 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against α-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Kumsta, C., Chang, J. T., Schmalz, J., Hansen, M. Hormetic heat stress and HSF-1 induce autophagy to improve survival and proteostasis in C. elegans. Nature Communications. 8, 14337 (2017).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108, 14204-14209 (2011).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. Journal of Biological Chemistry. 283 (1), 194-201 (2008).
- Hakim, A., et al. WorMachine: machine learning-based phenotypic analysis tool for worms. BMC Biology. 16 (1), 8 (2018).
- Wählby, C., et al. An image analysis toolbox for high-throughput C. elegans assays. Nature Methods. 9 (7), 714-716 (2012).
- Jones, T. R., et al. CellProfiler Analyst: data exploration and analysis software for complex image-based screens. BMC Bioinformatics. 9 (1), 482 (2008).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomona aeruginosa PA01. Journal of Bacteriology. 194 (23), 6387-6389 (2012).
- Schulenburg, H., Ewbank, J. J. The genetics of pathogen avoidance in Caenorhabditis elegans. Molecular Microbiology. 66 (3), 563-570 (2007).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964 (2014).
- Van Raamsdonk, J. M., Hekimi, S. FUdR causes a twofold increase in the lifespan of the mitochondrial mutant gas-1. Mechanisms of Ageing and Development. 132 (10), 519-521 (2011).
- Haldimann, P., Muriset, M., Vígh, L., Goloubinoff, P. The novel hydroxylamine derivative ng-094 suppresses polyglutamine protein toxicity in Caenorhabditis elegans. Journal of Biological Chemistry. 286 (21), 18784-18794 (2011).
- Shinn-Thomas, J. H. Wrapping culture plates with Parafilm M® increases Caenorhabditis elegans growth. BMC Research Notes. 12 (1), (2019).
- Alexander-Floyd, J., et al. Unexpected cell type-dependent effects of autophagy on polyglutamine aggregation revealed by natural genetic variation in C. elegans. BMC Biology. 18 (1), 18 (2020).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Sink, R., Gobec, S., Pecar, S., Zega, A. False positives in the early stages of drug discovery. Current Medicinal Chemistry. 17 (34), 4231-4255 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved