Method Article
Автоматизация количественной оценки агрегатов в Caenorhabditis elegans
В этой статье
Резюме
Следующий протокол описывает разработку и оптимизацию высокопроизводительного рабочего процесса для культивирования червей, флуоресцентной визуализации и автоматизированной обработки изображений для количественной оценки агрегатов полиглутамина в качестве оценки изменений в протеостазе.
Аннотация
Рост распространенности нейродегенеративных белковых конформационных заболеваний (ПХД) на протяжении многих лет способствовал большому интересу к этой теме. Это повышенное внимание потребовало диверсификации и улучшения животных моделей, способных воспроизводить фенотипы заболеваний, наблюдаемые у людей с ПХД. Хотя мышиные модели оказались бесценными, они дороги и связаны с трудоемкими методами с низкой пропускной способностью. Использование модели нематод Caenorhabditis elegans для изучения ПХД было оправдано относительной простотой обслуживания, низкой стоимостью и быстрым временем генерации, что позволяет использовать приложения с высокой пропускной способностью. Кроме того, высокая сохранность между геномами C. elegans и человека делает эту модель бесценным инструментом открытия. Нематоды, которые экспрессируют флуоресцентно помеченные тканеспецифические полиглутаминные (polyQ) тракты, демонстрируют возрастную и полиQ-зависимую агрегацию, характеризующуюся флуоресцентными очагами. Такие репортеры часто используются в качестве прокси для мониторинга изменений протеостаза в тканях. Ручная агрегированная количественная оценка занимает много времени, ограничивая экспериментальную пропускную способность. Кроме того, ручная количественная оценка фокусов может привести к смещению, поскольку агрегированная идентификация может быть очень субъективной. При этом протокол, состоящий из культивирования червей, сбора изображений и обработки данных, был стандартизирован для поддержки высокопроизводительной агрегированной количественной оценки с использованием C. elegans , которые экспрессируют специфический для кишечника polyQ. Благодаря реализации конвейера обработки изображений на основе C. elegans с использованием CellProfiler, программного обеспечения для анализа изображений, этот метод был оптимизирован для разделения и идентификации отдельных червей и перечисления их соответствующих агрегатов. Хотя концепция автоматизации не совсем уникальна, необходимость стандартизации таких процедур для воспроизводимости, устранения смещения от ручного подсчета и увеличения пропускной способности высока. Ожидается, что эти методы могут значительно упростить процесс скрининга больших бактериальных, геномных или лекарственных библиотек с использованием модели C. elegans .
Введение
Возрастные нейродегенеративные белковые конформационные заболевания (PCD), такие как болезни Альцгеймера, Паркинсона и Хантингтона, или боковой амиотрофический склероз, характеризуются неправильным сворачиванием белка, что приводит к агрегации, гибели клеток и дегенерации тканей1. В то время как неправильное сворачивание белка признано виновником, этиология этих заболеваний не ясна. Таким образом, разработка эффективных методов лечения сдерживается отсутствием знаний о факторах и условиях, которые способствуют возникновению и прогрессированию заболевания. Недавние исследования показывают, что изменения в микробиоме влияют на начало, прогрессирование и тяжесть ПХД 2,3,4. Однако сложность человеческого или даже мышиного микробиома затрудняет проведение исследований, которые бы выявили точное влияние микробов на их хозяина. Поэтому более простые организмы, такие как Caenorhabditis elegans, часто используются в качестве инструмента открытия 5,6,7,8. Недавние исследования использовали C. elegans для изучения влияния бактерий на протеостаз хозяина и патогенез заболевания 9,10. Бактериальная колонизация, гормезис и геномные изменения относятся к числу примерных состояний, влияющих на агрегацию полиглутамина (polyQ) трактов 9,11,12. Кроме того, эти неправильно свернутые белковые кластеры демонстрируют накопление polyQ в зависимости от длины и возраста внутри хозяина и связаны с нарушением моторики 9,13. Относительно простой подход к количественной оценке флуоресцентно меченой пункты может генерировать важные данные об условиях, факторах или лекарствах, которые влияют на сворачивание и агрегацию белка.
Хотя количественная оценка флуоресцентной пункты оказалась надежной и относительно простой процедурой, остается задача разработать протокол, который облегчил бы крупномасштабный скрининг соединений, бактерий или состояний, влияющих на агрегацию белка. Концепция автоматизированной обработки изображений C. elegans и количественной оценки puncta не совсем нова, так как ряд практических вспомогательных инструментов был разработан14,15. Тем не менее, интеграция культивирования, получения изображений и конвейера обработки имеет важное значение для устранения изменчивости результатов и обеспечения более высокой пропускной способности экранов.
Таким образом, целью этой рукописи является стандартизация процедуры, используемой для количественной оценки агрегации polyQ в C. elegans в качестве прокси для обнаружения изменений в протеостазе. Эта задача была выполнена за счет использования CellProfiler, программного обеспечения для анализа изображений с открытым исходным кодом16 , способного к автоматизированной идентификации червей и агрегатов, и интегрированного в более крупный протокол для культивирования червей, получения изображений и обработки данных.
протокол
Все процедуры соответствовали руководящим принципам безопасности, которые были рассмотрены и одобрены Институциональным комитетом по биобезопасности Университета Флориды. Были приняты соответствующие меры по обеспечению биобезопасности для снижения риска воздействия бактерий уровня биологической безопасности-2.
ПРИМЕЧАНИЕ: Для всех экспериментов C. elegans должны размножаться и поддерживаться на пластинах среды роста нематод (NGM), засеянных Escherichia coli OP50.
1. Подготовка плит NGM 10 см
- Соединить 3 г NaCl, 2,5 г триптиказы-пептона и 17 г агара в колбу объемом 2 л и залить до 1 л двойной дистиллированной водой (ddH2O). Добавьте магнитный перемешивание перед автоклавированием.
- Автоклав смеси в течение 45 мин при 121 °C и давлении 21 psi. Дайте смеси остыть до 50 °C на водяной бане.
- Используя асептические методы, добавляют следующие стерильные растворы: 1 мл 1 M CaCl2· 2Н2О, 1 мл 1 М МгСО4· 7Н2О, 1 мл 5 мг/мл холестерина, растворенного в 100% этаноле (подогретом до комнатной температуры), и 25 мл 1 М КХ2РО4 (рН = 6,0). Перемешать с помощью магнитной пластины. Перемешивание может быть выполнено в течение 1 мин при 700 об/мин.
- Заливать до тех пор, пока смесь не заполнит всю 10 см тарелку. В качестве альтернативы используйте градуированную серологическую пипетку, чтобы добавить приблизительно 20 мл смеси на пластину.
- Дайте плитам высохнуть в течение 24 ч при комнатной температуре перед посевом бактериями или храните простые пластины при 4 °C после сушки.
ПРИМЕЧАНИЕ: Все компоненты носителей обрабатываются с использованием асептических методов. Этапы 1.3-1.4 должны выполняться в ламинарной вытяжке.
2. Приготовление агара NGM с FUDR в 24-луночных плитах
- Выполните шаги 1.1-1.3.
- Дополнить NGM 5-фтор-2'-дезоксиуридином (FUDR) и смешать для достижения конечной концентрации 100 мкг/мл.
ПРИМЕЧАНИЕ: FUDR ингибирует репликацию ДНК и, как следствие, блокирует размножение C. elegans , нацеливаясь на зародышевую линию и эмбриогенез, в конечном итоге влияя на продолжительность жизни. Поэтому важно позволить червям полностью развиться в молодых людей, прежде чем переходить на FUDR-содержащие пластины.
ВНИМАНИЕ: FUDR токсичен и должен обрабатываться в соответствии с паспортом безопасности производителя. - Используя пипетку, дозируйте 1 мл NGM-FUDR в каждую лунку.
ПРИМЕЧАНИЕ: Этот процесс может быть облегчен за счет использования автоматизированной системы заливки пластин. - Дайте пластине высохнуть в течение 24 ч при комнатной температуре перед посевом бактериями или хранением простых пластин при 4 °C.
3. Посев пластин: OP50 и дополнительные тестовые бактерии
- Чтобы приготовить культуру E. coli OP50 на ночь, добавьте 200 мкл бактериальной аликвоты из замороженного бульона в колбу Эрленмейера объемом 500 мл, содержащую 250 мл свежего, стерильного бульона Лурия (LB).
ПРИМЕЧАНИЕ: Объем носителя зависит от количества пластин, которые необходимо посеять. Для приготовления других бактериальных культур привите 16-мл культуральную трубку, содержащую 5 мл питательной среды, бактериями из замороженного бульона с помощью стерильного наконечника микропипетки. - Инкубировать в течение ночи в инкубаторе при температуре 37 °C, встряхивая при 220 об/мин (вращений в минуту).
ПРИМЕЧАНИЕ: Используйте стерилизованные колбы, по крайней мере, в два раза больший рабочий объем среды и уплотнение автоклавной алюминиевой фольгой. Выполните этап прививки и дозирование бактерий с использованием асептических методов. - Нанесите 1-2 мл ночной культуры E. coli OP50 на центр каждой 10 см пластины NGM. Эту культуру не нужно распространять вокруг плиты NGM.
- Дайте пластинам высохнуть при комнатной температуре перед использованием/хранением.
ПРИМЕЧАНИЕ: Семенные пластины с крышками могут быть помещены в вытяжку с воздушным потоком для облегчения сушки.
4. Культивирование и посев плит: 24-луночные плиты
- Подготовьте на ночь культуру желаемых штаммов бактерий, придерживаясь инструкций по культивированию, приведенных в шагах 3.1-3.2.
- Перенесите 200 мкл каждой бактериальной культуры в каждую лунку 24-луночной пластины, содержащей агар NGM. Объем бактерий в 200 мкл покроет всю площадь агара, максимизируя количество пищи, чтобы гарантировать, что черви не будут избегать бактериального газона.
- Оставьте пластины потрескавшимися открытыми в шкафу биологической безопасности (BSC), чтобы облегчить сушку. Периодически проверяйте пластины, чтобы предотвратить чрезмерное обезвоживание и изменяйте ориентацию пластин, чтобы обеспечить равномерный воздушный поток и сушку. Пластины должны высохнуть в течение 5 ч.
ПРИМЕЧАНИЕ: Любая работа с бактериями уровня биологической безопасности 2 должна выполняться в сертифицированных BSC и одобрена Институциональным комитетом по биобезопасности.
5. Синхронизация возраста
ПРИМЕЧАНИЕ: Все шаги должны быть выполнены с использованием надлежащих асептических методов (т. Е. Работа близко к пламени или внутри BSC).
- Смойте гравидные гермафродиты с пластин OP50 размером 10 см с помощью стерилизованного фильтром раствора M9 (5,8 г Na2HPO4· 7Н2О, 3,0 г KH2ПО4, 5 г NaCl, 0,25 г MgSO4·7H2O, в 1 л ddH2O).
- Пипетку М9 раствором наносят на пластину несколько раз с помощью стерильного стекла или пластиковой серологической пипетки для поднятия червей с бактериального газона.
- Соберите червячную суспензию и переложите раствор в коническую трубку из полистирола объемом 15 мл.
- Центрифуга при 270 х г, комнатная температура (RT, ~23 °C), в течение 2 мин.
- Аспирируйте с помощью вакуумной колбы-ловушки и выбросьте супернатант, оставив гранулу червя нетронутой.
- Повторно суспендируют гранулу в 5-10 мл M9 для промывки червей и повторите шаги 5,2-5,3 дважды.
- Добавьте 5 мл 20% отбеливающего раствора (8,25 мл ddH2O, 3,75 мл 1M NaOH, 3,0 мл неэмицидного отбеливателя) в трубку и непрерывно инвертируйте для растворения червей. Черви готовы к центрифуге, как только они почти полностью растворяются.
ПРИМЕЧАНИЕ: Время отбеливания и объем отбеливающего раствора будут зависеть от размера образца. Чрезмерное и недостаточное отбеливание являются распространенными ошибками. Таким образом, этот процесс обычно требует оптимизации, чтобы определить, когда образец готов к центрифугированию. - Центрифугу в течение 2 мин при 423 х г и выбросьте супернатант.
- Добавьте 10 мл стерильного M9 для повторного суспендирования яичной гранулы.
- Центрифугируйте пробирку в течение 2 мин при 423 х г , чтобы гранулировать яйца. Удалите супернатант с помощью колбы аспиратора.
- Повторите шаги 5.6-5.6.1.
- Повторно суспендируйте яичную гранулу в 5 мл стерильного M9 и поместите ее на нутритор на ночь при нужной температуре.
ПРИМЕЧАНИЕ: Синхронизированные по возрасту личинки L1 будут готовы к переходу на пластины на следующий день.
6. Подготовка червей после возрастной синхронизации
- Центрифугировать синхронизированных по возрасту червей при 270 х г в течение 3 мин при РТ (~23 °C).
- Аспирируйте супернатант в чистой среде, такой как вытяжка или рядом с горелкой Бунзена. Оставьте примерно 200 мкл супернатанта и повторно суспендируйте червей.
- Используя микропипетку, перенесите концентрированную червячную суспензию на пластины NGM размером 10 см, которые ранее были засеяны OP50.
ПРИМЕЧАНИЕ: Каждая тарелка может поддерживать 1 500 червей без нехватки пищи; однако эта концентрация может потребовать корректировки в зависимости от плотности бактериального газона и температуры роста. Рекомендуется использовать несколько пластин, чтобы черви не голодали. Важно отметить, что эти пластины не должны содержать FUDR. - Дайте пластинам высохнуть; затем инвертировать и хранить при 25 °C в течение 48 ч.
ПРИМЕЧАНИЕ: В зависимости от состояния экрана, черви с этапа 6.1 могут быть помещены непосредственно на тестовые пластины (содержащие бактерии, лекарства или тестовые соединения). Если желаемое условие теста влияет на развитие, червей следует культивировать на NGM, содержащих OP50, до молодых людей (~ 48 ч), прежде чем подвергать их условиям тестирования. - После 48-часовой инкубации промыть червей с пластин стерильным раствором М9 и поместить их в конические трубки.
ПРИМЕЧАНИЕ: Взрослые черви будут опускаться на дно трубки. Точное время будет варьироваться в зависимости от количества червей, извлеченных из 10-сантиметровых пластин. В этих условиях глисты оседают в течение 10 мин. - Выполните визуальный осмотр, чтобы определить продолжительность времени оседания, чтобы удалить любые остаточные яйца или вылупившиеся личинки.
- Добавьте еще 10 мл M9, чтобы смыть остаточные бактерии из тел червей.
- Центрифугировать червей в течение 2-3 мин при 270 х г при 23 °С.
- Выполните этап стирки еще 3 раза. Для достижения наилучших результатов оставьте около 1-1,5 мл раствора M9 в пробирке после окончательной промывки.
- Переложите 10 мкл суспензии червя на стеклянную горку и подсчитайте количество червей.
- Отрегулируйте плотность червя примерно до 150 червей на 10 мкл M9. Концентрацию червей во суспензии можно регулировать, удаляя или добавляя раствор М9 после центрифугирования.
- Подтвердите, что желаемая концентрация была установлена путем усреднения подсчетов от нескольких разных капель. Рекомендуется усреднять подсчеты не менее чем от трех капель.
- Используя асептические методы, перенесите 10 мкл червячной суспензии, содержащей приблизительно 150 червей, в каждую лунку испытательной пластины.
- Осмотрите скважины под микроскопом, чтобы убедиться, что в каждой из них достаточное количество червей. Дополнительные черви могут быть добавлены перед инкубацией.
- Дайте тарелкам высохнуть примерно на 10 минут; а затем инвертировать и перевести в инкубатор при температуре 25 °C в течение 72 ч.
ПРИМЕЧАНИЕ: Окончательный инкубационный период может быть скорректирован с учетом потребностей эксперимента. Инкубационного времени 72 ч достаточно для поддержания роста 150 животных, питающихся бактериями 200 мкл в 24-луночной пластине при 25 °C.
7. Подготовка червей к визуализации
- Чтобы облегчить более эффективное оседание и свести к минимуму потерю образцов, обездвиживайте червей перед стиркой, чтобы предотвратить плавание.
ПРИМЕЧАНИЕ: При работе с небольшим количеством образцов это может быть достигнуто путем воздействия левамизола (100 мкМ). Однако при работе с большим количеством образцов червей можно обездвижить путем замораживания. Кроме того, длительное замораживание (18-24 ч) предотвратит дальнейшее развитие заполнителей polyQ во время приготовления.- Поместите многоскважинные плиты при -20 °C в течение 15-20 мин или до тех пор, пока черви больше не будут двигаться.
- Извлеките образцы из морозильной камеры и дайте им постоять в течение 5 минут.
- Используя микропипетку, добавьте 1 мл M9, охлажденного до 4 °C, в интересующую лунку, многократно нажмите и вдавите плунжер 4-6 раз, чтобы промыть червей в каждой лунке.
ПРИМЕЧАНИЕ: Иногда черви могут прилипать к наконечникам микропипетки. Таким образом, между каждой скважиной следует использовать различные наконечники, чтобы предотвратить смешивание червей. - Перенесите суспензию червя в трубку микроцентрифуги и дайте червям опуститься на дно.
- Аспирировать и выбросить супернатант. Вымойте образец в общей сложности три раза.
- После того, как черви оседают на дно во время окончательной промывки, аспирируют 500 мкл супернатанта, оставляя 500 мкл для повторного суспендирования червей.
- Переложите оставшуюся червячную суспензию на новую плоскодонную 24-луночную пластину и поместите ее в морозильную камеру при температуре -20 °C на 48 ч.
ПРИМЕЧАНИЕ: Замораживание червей уменьшает фоновую флуоресценцию и позволяет лучше визуализировать агрегаты.
8. Визуализация
- Извлеките пластины из морозильной камеры и дайте оттепели, вытрите лишнюю конденсацию и снимите крышку перед визуализацией.
ПРИМЕЧАНИЕ: Детали захвата изображения будут варьироваться в зависимости от используемого оборудования и программного обеспечения. Протоколы для этого раздела должны служить лишь руководством, и следует ожидать внесения изменений. Также требуется захватывать изображения в формате файла tiff. - Во время захвата изображения используйте следующие настройки микроскопа: Время экспозиции, 500 мс; 40-кратное увеличение с адаптером камеры 0,63x (25,2x), интенсивность GFP установлена на 100%.
ПРИМЕЧАНИЕ: Различные конфигурации и системы микроскопов могут получать изображения, отличные от тех, которые представлены в разделе результатов. Для достижения более универсального описания требуемых изображений предоставляются более объективные сведения об изображениях. Агрегаты должны иметь диаметр от 1,0 до 10,0 пикселей с интенсивностью флуоресценции 0,10-1,0 (произвольные единицы, масштаб 0-1), чтобы быть правильно идентифицированными CellProfiler. Флуоресцентный фон для этих агрегированных изображений, как правило, ниже порога 0,10. Для ярких полевых изображений интенсивность червя колеблется в пределах 0,7-1,0 с фоновой интенсивностью 0,1-0,2. Средняя длина червей на ярких изображениях колеблется от 250 пикселей до 350 пикселей в длину от головы до хвоста. - Отрегулируйте элементы управления проходящим светом до тех пор, пока черви не станут ярко освещенными по сравнению с темным фоном. Избегайте передержки; это увеличит размер червя.
- Может потребоваться изменить положение червей внутри скважины, чтобы предотвратить чрезмерное слипание. Рассейте слипшихся червей с помощью наконечника пипетки.
- Установите канал на GFP, чтобы установить фокальную плоскость для обоих изображений.
ПРИМЕЧАНИЕ: Важно, чтобы фокальная плоскость определялась в канале GFP. Любое изменение фокуса, сделанное при захвате ярких изображений, приведет к смещению агрегатов и червей во время анализа изображения. - Захватите яркое изображение поля и сразу же сделайте соответствующее флуоресцентное изображение, не нарушая пластину.
- Запустите тестовые изображения через конвейер, чтобы определить значения интенсивности и размера для объектов в изображении в соответствии с «ПРИМЕЧАНИЕМ» после шага 8.2.
ПРИМЕЧАНИЕ: CellProfiler может предоставить всю необходимую информацию относительно интенсивности и длины объектов до анализа. - Чтобы оценить изображения, сначала загрузите CellProfiler17. Откройте программное обеспечение и перетащите интересующие изображения в поле «Изображения».
- Нажмите на изображения в списке файлов, чтобы открыть их.
- В левом верхнем углу экрана расположены несколько иконок; используйте их для измерения размера объектов или увеличения определенной области. Выберите лупу, чтобы выделить интересующую область.
- Щелкните значок стрелки, чтобы измерить длину червей и агрегатов.
- Наведите курсор на нужный объект, чтобы определить его значение интенсивности, которое можно увидеть в нижней части экрана.
ПРИМЕЧАНИЕ: Поскольку все изображения сделаны в оттенках серого, все значения красного, синего и зеленого пикселей будут идентичными.
9. Анализ изображений
- Чтобы использовать конвейер анализа изображений CellProfiler, присвойте парам изображений правильное имя. Используйте следующий формат: P1_A01_S1_C1, где P1 относится к конкретной табличке и ее соответствующему числовому обозначению; "A" относится к строке, а "01" - к столбцу; "S" относится к определенной паре изображений для одной скважины; «C» относится к каналу, где «1» используется для обозначения ярких изображений и «2» для флуоресцентных изображений.
- Скачайте CellProfiler (версия 4.1.3 или выше) с официального сайта17.
- Загрузите конвейер (дополнительный файл 1). Загрузите конвейер (конвейер) в CellProfiler, выбрав Файл > Импортировать > Конвейер из Файла.
ПРИМЕЧАНИЕ: Модули 2 и 3 были отключены, поскольку они были сочтены ненужными. Их функция может быть восстановлена, если анализ изображений не дает удовлетворительного разделения и идентификации червей; однако требуется модификация трубопровода. - Чтобы включить эти модули, выберите поля, расположенные слева от каждого модуля. Переименуйте входное двоичное изображение для модуля "UntangleWorms" в выходное изображение из модуля "convertobjectstoimage".
- Во время первоначального использования, связанного с модулем 2, может появиться сообщение об ошибке. Если это произойдет, приступайте к анализу.
- Загрузите обучающий набор, используемый для идентификации червей, в модуль "UntangleWorms". Этот обучающий набор можно найти в Дополнительном файле 2.
- Выберите модуль UntangleWorms, чтобы открыть его настройки.
- Определите имя файла набора обучения и выберите значок отправки файла.
- Загрузите дополнительный файл 2 (обучающий набор).
- Загрузите изображения, выбрав модуль Изображения в левом верхнем углу. Перетащите правильно названные изображения, как описано в шаге 9.1.
- Перед анализом изображений выберите нужную выходную папку, которая будет использоваться для хранения результатов.
- Нажмите на кнопку Настройки вывода , расположенную в левом нижнем углу программы.
- Щелкните значок папки справа от элемента Вывод по умолчанию , чтобы выбрать желаемое расположение вывода.
- Щелкните значок Анализ изображений , чтобы начать анализ изображений.
- Если анализ занимает слишком много времени и застрял при обработке одного изображения, это, вероятно, связано с проблемами с получением изображения. В этом случае прервите запуск и перейдите к идентификации необработанных изображений, отсортировав выходную папку и отметив, какие имена изображений не найдены.
ПРИМЕЧАНИЕ: Изображения могут потребовать дополнительной обработки для удаления больших или интенсивно освещенных артефактов, которые не могут быть обработаны конвейером. В некоторых случаях такие изображения, возможно, потребуется исключить из анализа. - После полного анализа программное обеспечение организует результаты в электронную таблицу Excel, содержащую отдельных червей (столбец N) и их соответствующее количество агрегатов (столбец K).
ПРИМЕЧАНИЕ: При успешном запуске будет создана электронная таблица Excel, содержащая количество агрегатов на идентифицированного червя для каждой скважины. Этими данными можно манипулировать способом, определенным экспериментатором. - Загрузите организатор метаданных (графический интерфейс пользователя) из дополнительного файла 3 (Windows) или дополнительного файла 4 (Mac), чтобы удобно упорядочить данные из выходного CSV-файла из CellProfiler.
ПРИМЕЧАНИЕ: Это программное обеспечение не имеет официальной лицензии и не будет автоматически открываться после загрузки. - Выполните действия 9.14-9.17 при использовании 64-разрядной версии ОС Windows. Программное обеспечение не будет работать на Windows 32-bit. Выполните действия 9.18-9.20 при использовании Mac OS. Шаги 9.21-9.22 одинаковы для обеих операционных систем.
- Для ОС Windows найдите загруженный файл и извлеките его в нужное место.
- Найдите и откройте извлеченную папку с именем gui_windowsOS_64x и запустите приложение, щелкнув значок приложения «gui».
- Может открыться запрос на получение разрешения на запуск. Выберите Дополнительные сведения и нажмите кнопку Доверять в любом случае.
- Организатор метаданных готов к перетаскиванию выходных CSV-файлов CellProfiler. Перейдите к шагу 9.21.
- Для Mac OS найдите загруженный файл и откройте gui_macOS_64x.zip. Этот шаг должен автоматически извлечь все файлы.
- Откройте извлеченную папку, найденную в разделе "Загрузки".
- Щелкните правой кнопкой мыши на приложении "gui" и выберите Открыть. Появится запрос на разрешение на открытие из-за отсутствия официальной лицензии. Выберите Открыть и перейдите к шагу 9.21.
- Нажмите Загрузить ваши файлы здесь или перетащите нужные CSV-файлы CellProfiler.
- Нажмите кнопку Упорядочить , которая приведет пользователя к новому экрану с кнопкой Загрузить файлы . Нажмите на кнопку и выберите нужное место для сохранения выходного файла. Выходной файл будет отображаться как исходное имя файла с расширением "_organized", добавленным к имени файла.
Результаты
Здесь описан рабочий процесс C. elegans , который включает в себя протоколы культивирования, получения изображений и обработки, которые позволяют оценивать агрегацию polyQ в присутствии различных бактерий с использованием 24-луночного формата пластины в качестве платформы культивирования и визуализации (рисунок 1). Этот протокол может быть скорректирован для изучения влияния бактерий, конкретных условий, малых молекул, лекарств или геномных манипуляций на протеостаз хозяина. Описанный способ был оптимизирован с использованием червей, которые конститутивно экспрессируют кишечный polyQ, слитый с желтым флуоресцентным белком (vha6p::p olyQ44::YFP); однако другие модели, которые сообщают о протеостазе в мышцах или нейронах, также могут быть использованы с дальнейшей оптимизацией. Например, предварительные эксперименты демонстрируют применение этих методов при количественной оценке белковых агрегатов в других тканях, таких как мышечный polyQ (дополнительный рисунок 1). Тем не менее, потребуется модификация трубопровода для правильной настройки совокупного размера и яркости, как указано в разделе 8 ПРИМЕЧАНИЕ.
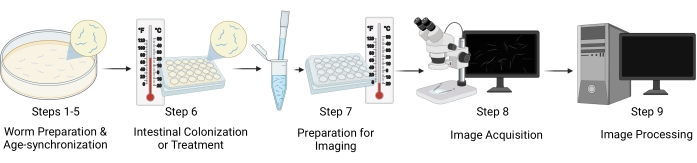
Рисунок 1: Визуальное представление рабочего процесса. Основные этапы протокола включают в себя пять различных этапов: подготовка червей и синхронизация возраста (этапы 1-5), кишечная колонизация/лечение червями (этап 6), подготовка образцов для визуализации (этап 7), получение изображений (этап 8) и обработка изображений (этап 9). «Разделы» протокола упоминаются как «Шаги» на рисунке. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Первоначальные эксперименты по оптимизации выявили различные трудности, связанные с перенаселенностью из-за большого количества потомств, что привело к более быстрому истощению пищи. Добавление FUDR в пластины NGM, описанное в разделе 2, решило эту проблему (рисунок 2). Кроме того, в присутствии FUDR черви, которых кормили различными бактериями, имели более последовательный размер тела, что позволяло более равномерно и точно обнаруживать червей.
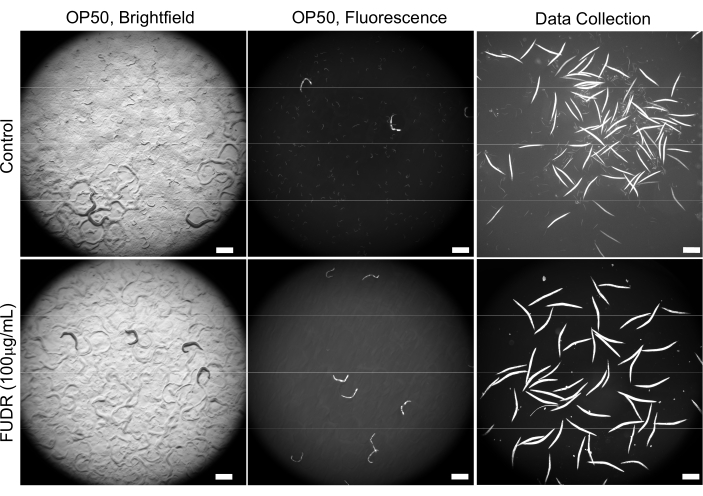
Рисунок 2: Использование FUDR улучшает качество изображения за счет уменьшения потомства. Пластины, дополненные FUDR, устраняют потомство C. elegans по сравнению с червями, выращенными на не-FUDR контрольных пластинах NGM, засеянных E. coli OP50. Изображения были получены с увеличением 25,2x (увеличение 40x с адаптером камеры 0,63x). Шкала = 500 мкм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Фоновая флуоресценция способствовала ложноположительному обнаружению агрегатов polyQ. Чтобы уменьшить такой флуоресцентный сигнал в кишечном тракте и улучшить автоматическое обнаружение агрегатов, необходимо было заморозить червей перед визуализацией. Замораживание червей при -20 °C в течение 18-48 ч значительно улучшало обнаружение агрегатов polyQ за счет устранения фоновой флуоресценции (рисунок 3A). Человеческий глаз способен различать агрегаты и фоновую флуоресценцию; следовательно, ручной подсчет до и после замораживания одинаков (рисунок 3B). Однако автоматизированный подсчет не так точен, но замораживание значительно улучшило автоматические подсчеты с точностью, сопоставимой с ручным подсчетом (рисунок 3B).

Рисунок 3: Замораживание улучшает обнаружение агрегатов. (A) Флуоресцентные изображения C. elegans , экспрессирующих кишечный polyQ44::YFP до и после замораживания. Вставки представляют собой изображения выделенной области крупным планом. Шкала баров = 500 мкм. (B) Среднее количество агрегатов на кишечник у червей, колонизированных P. aeruginosa MPAO1 до и после замораживания с использованием ручной или автоматизированной (трубопроводной) количественной оценки агрегатов. Данные представляют две биологические реплики (n = 60-109). Статистическая значимость рассчитывалась с помощью t-теста Стьюдента (**** p < 0,0001). Полосы погрешностей представляют стандартную погрешность среднего значения (SEM). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Инвертированное яркое освещение использовалось для обнаружения целых C. elegans (рисунок 4A) и канала GFP для изображения агрегатов polyQ44::YFP (рисунок 4B). Обнаружение, распутывание червей и агрегированная количественная оценка для каждого червя были выполнены путем применения оптимизированного конвейера обработки изображений CellProfiler (Дополнительный файл 1), который позволяет получить количество агрегатов на отдельного червя (рисунок 4C-D). Чтобы проверить осуществимость этого подхода и точность автоматизированного обнаружения и количественной оценки агрегатов, черви, экспрессирующие специфический для кишечника polyQ44::YFP, были культивированы и подготовлены к визуализации в соответствии с установленными протоколами (разделы 1-7). Количество агрегатов на червя оценивалось либо с помощью автоматизированного трубопровода (участки 8-9), либо ручного подсчета. Каждый эксперимент проводился в трех независимых испытаниях с использованием 90-571 червя на состояние. В то время как среднее количество агрегатов на кишечник, полученное в двух испытаниях, не имело существенной разницы, черви в третьем испытании имели значительно меньше агрегатов при количественной оценке с использованием автоматизированного подхода (рисунок 5А). Среднее количество агрегатов из трех испытаний привело к незначительному, но значительно меньшему количеству агрегатов, когда количественная оценка проводилась с использованием конвейера CellProfiler (разделы 8-9) (рисунок 5B). Тем не менее, разница между двумя подходами была минимальной, что указывает на то, что автоматизированный метод может быть применен к крупномасштабным экранам.
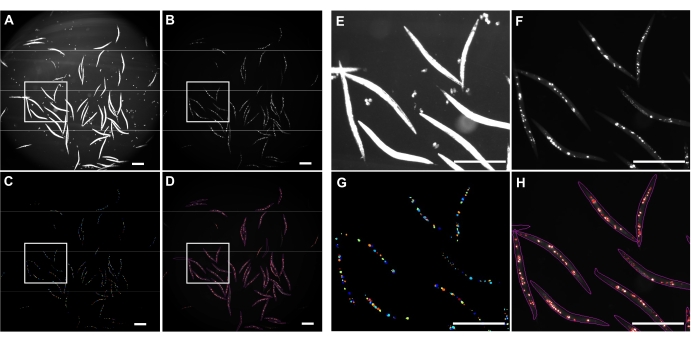
Рисунок 4: Агрегированное обнаружение с помощью CellProfiler. (A) Изображение Brightfield, используемое для идентификации тел червей. (B) Исходное флуоресцентное изображение, полученное с использованием канала GFP и используемое для идентификации и количественной оценки общего числа кишечных агрегатов polyQ44::YFP. (C) Агрегаты, идентифицированные с помощью CellProfiler. (D) Общее число идентифицированных агрегатов, наложенных на исходное флуоресцентное изображение с червячными и агрегированными контурами. Захват и обработка изображений выполнялись с использованием настроек, описанных в разделах 8-9. Панели E-H представляют собой крупные планы изображений соответствующих очерченных областей на изображениях A-D. Изображения были получены с увеличением 25,2x (увеличение 40x с адаптером камеры 0,63x). Шкала = 500 мкм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
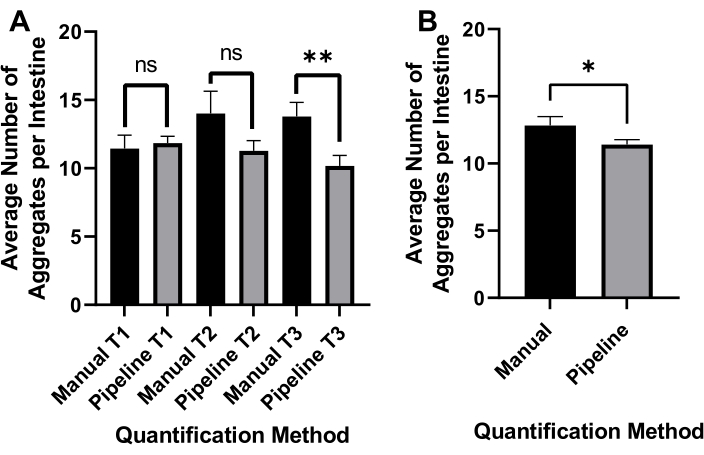
Рисунок 5: Эффективность автоматизированной агрегированной количественной оценки. (A) Среднее совокупное число на кишечник у червей, колонизированных контрольной кишечной палочкой OP50 с использованием ручного подсчета (ручной) и автоматизированной количественной оценки на основе CellProfiler (Pipeline). Результаты представляют собой данные, проанализированные в трех отдельных испытаниях (T1-T3) (n = 90-571). (B) Среднее количество агрегатов на кишечник было получено с использованием ручной или автоматизированной (трубопроводной) количественной оценки агрегатов. Статистическая значимость рассчитывалась с помощью t-теста Стьюдента (* p < 0,05, ** p < 0,01). Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Чтобы оценить воспроизводимость результатов среди различных экспериментаторов, изображения с пластины, содержащей шесть лунок червей, которых кормили либо Pseudomonas aeruginosa MPAO1, либо Escherichia coli OP50, были получены тремя лицами, двое из которых не имели опыта визуализации червей с использованием этих протоколов. Изображения, собранные из каждой скважины, содержали от 30 до 115 обнаруженных червей. Незначительная разница в агрегации была обнаружена у червей из тех же скважин, которые были сфотографированы тремя экспериментаторами. В то время как среднее количество агрегатов на кишечник оставалось очень последовательным между тремя экспериментаторами для червей, получавших MPAO1 и OP50, были некоторые статистически значимые различия в среднем количестве агрегатов, но только у червей, колонизированных MPAO1 (дополнительный рисунок 2). Эти результаты подчеркивают воспроизводимость результатов даже между неопытными экспериментаторами.
Чтобы гарантировать, что воспроизводимость совокупной количественной оценки не оказывает существенного влияния на положение червя, был выбран набор из 15 червей и изображен 15 отдельных раз после перемешивания между каждым захватом изображения с использованием наконечника пипетки. Изображения агрегатов у червей, которых кормили E. coli OP50 и P. aeruginosa MPAO1, были собраны и проанализированы с помощью CellProfiler. Среднее число агрегатов из каждого из этих различных наборов изображений незначительно, но незначительно отличалось, что еще больше подтверждало воспроизводимость этого подхода (дополнительный рисунок 3).
Было показано, что колонизация кишечника C. elegans грамотрицательными кишечными патогенами нарушает протеостаз в тканях, причем P. aeruginosa является одним из наиболее мощных индукторов агрегации polyQ9. Чтобы определить, будут ли эти оптимизированные протоколы успешно обнаруживать и количественно оценивать P. aeruginosa-опосредованное усиление агрегации, черви, экспрессирующие кишечный polyQ, были колонизированы E. coli OP50 (контрольные бактерии) и P. aeruginosa MPAO1, разделы 1-8. Полученные изображения были проанализированы с помощью CellProfiler (раздел 9, Дополнительный файл 1). Результаты автоматизированной количественной оценки показывают значительное увеличение числа агрегатов, индуцированных P. aeruginosa MPAO1, что последовательно приводит к двукратному улучшению по сравнению с червями, получавшими контрольную E. coli OP50 (рисунок 6).

Рисунок 6: Среднее количество агрегатов на кишечник у червей, колонизированных контрольной E. coli OP50 и P. aeruginosa MPAO1. Количество агрегатов на кишечник оценивали с помощью CellProfiler (разделы 8-9). Данные представлены в виде среднего числа агрегатов на кишечник у червей, колонизированных OP50 (n = 1068) и MPAO1 (n = 1557). Статистическая значимость рассчитывалась с помощью t-теста Стьюдента (**** p < 0,0001). Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Оптимизированный конвейер был разработан для поддержки крупномасштабных экранов для условий, влияющих на протеостаз. Чтобы проверить осуществимость этого подхода при скрининге больших библиотек бактерий на предмет их влияния на протеостаз хозяина, был использован описанный в настоящем описании конвейер (разделы 1-9) для проверки влияния несущественных мутантных штаммов 90 P. aeruginosa на агрегацию18 полиQ. Этот пилотный экран является частью более крупного проекта, предназначенного для скрининга всех несущественных мутантных штаммов P. aeruginosa на предмет их способности влиять на протеостаз хозяина. Из 90 протестированных бактериальных штаммов колонизация кишечника C. elegans одним кандидатом показала значительное снижение количества агрегатов (рисунок 7). Последующие эксперименты для оценки чувствительности этого анализа проводились с помощью ручного агрегированного подсчета из случайного отбора шести мутантов P. aeruginosa , которые незначительно отличались от контроля MPAO1. Эти эксперименты проводились с использованием более традиционных пластин NGM размером 6 см, перенося червей на тестовые штаммы как L1 для рекапитуляции ранее установленных методов9. Эксперименты по подтверждению методом ручного подсчета показали, что ни один из мутантов, включая того, который значительно уменьшил количество агрегатов (рисунок 7), не влиял на агрегацию polyQ (дополнительный рисунок 4). Кроме того, тонкие изменения в агрегации, наблюдаемые на экране 90 мутантных штаммов, не были обнаружены среди ручных подсчетов отобранных кандидатов, что указывает на то, что такие изменения могут возникнуть из-за биологической и экспериментальной изменчивости, такой как низкое значение n. В совокупности результаты показывают, что, хотя наш метод может надежно улавливать значительные изменения, тонкие из них, вероятно, будут пропущены, и все потенциальные кандидаты должны быть индивидуально подтверждены.
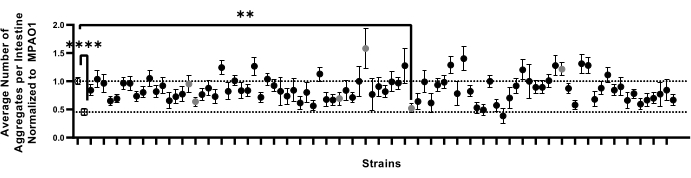
Рисунок 7: Количество агрегатов на кишечник в репрезентативном выборочном наборе червей, колонизированных 92 бактериальными штаммами. Данные представлены в виде среднего количества агрегатов на кишечник, нормализованного до количества червей, колонизированных MPAO1. Пунктирные линии представляют среднее количество агрегатов в червях, колонизированных MPAO1 (сверху, открытый круг) и OP50 (снизу, открытый квадрат). Сплошные символы представляют 90 различных нокаутирующих мутантных штаммов P. aeruginosa MPAO1. Среднее количество агрегатов на червя между червями, колонизированными MPAO1, и одним мутантом было статистически значимым. Серые круги представляют собой образцы, которые были подтверждены вручную (дополнительный рисунок 4). Статистическая значимость была рассчитана с использованием одностороннего дисперсионного анализа (ANOVA) с последующим множественным сравнением пост-хок теста Даннетта (** p < 0,01, **** p < 0,0001). Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Для управления большим объемом данных, генерируемых CellProfiler, был разработан графический интерфейс пользователя (GUI) для автоматизации обработки и организации данных (рисунок 8). Графический интерфейс был разработан с использованием Tkinter, кроссплатформенного инструментария виджетов Python с открытым исходным кодом. Из заданных метаданных приложение извлекает количество агрегатов (столбец K) из каждой скважины (колонка J), присутствующей в пластине. Для выполнения вышеупомянутого процесса использовалась библиотека обработки данных Python под названием «Pandas». Приложение GUI обеспечивает поддержку перетаскивания для пользователей для загрузки файлов данных. Данные в каждом файле хранятся в виде двумерной табличной структуры, называемой фреймом данных. Пустая пара словарей инициализируется для каждой уникальной скважины, найденной в рамке данных. Затем различные агрегаты, найденные в каждой скважине, подсчитываются и добавляются к соответствующим словарным парам. Столбец с меньшими данными заполняется пустыми строками, чтобы гарантировать, что каждый столбец имеет равный размер. Наконец, структура преобразуется в фрейм данных, который экспортируется в виде электронной таблицы в каталог, указанный пользователем.

Рисунок 8: Графический интерфейс пользователя. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный рисунок 1: Обнаружение мышечно-специфических агрегатов polyQ. Черви, экспрессирующие мышечно-специфический полиQ35::YFP, покрывались как L1s и культивировались на OP50 в течение 48 ч. Как только черви превратились в молодых людей, их переносили на 24-луночные пластины NGM, добавляли 100 мкг / мл FUDR и засеивали MPAO1 в течение дополнительных 72 ч перед визуализацией. (A) Изображение Брайтфилда, используемое для идентификации тел червей. (B) Исходное флуоресцентное изображение, полученное с помощью канала GFP. (C) Агрегаты, идентифицированные с помощью CellProfiler. (D) Общее число идентифицированных агрегатов, наложенных на исходное флуоресцентное изображение с червячными и агрегированными контурами. Захват и обработка изображений выполнялись с использованием настроек, описанных в разделах 8-9. Панели E-H представляют собой крупные планы изображений соответствующих очерченных областей на изображениях A-D. Шкала = 500 мкм. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 2: Воспроизводимость совокупной количественной оценки между различными экспериментаторами. Среднее количество агрегатов количественно определено с помощью CellProfiler для шести скважин червей, колонизированных P. aeruginosa MPAO1 (черные полосы) и шести скважин червей, колонизированных контрольной E. coli OP50 (серые полосы). Каждая скважина была сфотографирована тремя экспериментаторами (AVS, DMC, RDH). Данные представлены в виде среднего количества агрегатов на кишечник (n = 30-115). Статистическая значимость была рассчитана с использованием одностороннего ANOVA, за которым последовал тест множественных сравнений Туки (* p < 0,05, ** p < 0,01). Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 3: Влияние положения червя на воспроизводимость совокупной количественной оценки. Среднее совокупное число на кишечник у червей, колонизированных контрольной кишечной палочкой OP50 (серые полосы) и P. aeruginosa MPAO1 (черные полосы). Результаты представляют собой среднее количество агрегатов на кишечник (15≥n≥12), количественное с помощью CellProfiler. Положение червей внутри скважин изменялось волнением между каждым приобретением. Статистически значимых различий не было обнаружено ни в одной из групп. Статистическая значимость была рассчитана с использованием одностороннего ANOVA, за которым последовал тест множественных сравнений Туки. Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 4: Подтверждение пилотного экрана с ручным подсчетом. Среднее количество агрегатов на кишечник было количественно определено вручную. Данные представляют собой агрегационные профили червей, колонизированных шестью нокаутирующими мутантами MPAO1 (серые круги , рисунок 7), по сравнению с контрольными элементами MPAO1 и OP50 дикого типа (n = 30). Статистическая значимость рассчитывалась с использованием одностороннего ANOVA с последующим множественным сравнением пост-хок теста Даннетта (**** p < 0,0001). Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 5: Влияние FUDR на агрегацию полиQ кишечника. Данные представлены в виде среднего числа агрегатов polyQ44::YFP на кишечник (n = 20). Червей переносили на контрольные (без FUDR) или FUDR-содержащие пластины (100 мкг/мл) после 48 ч роста при 25 °C на E. coli OP50. Ручные подсчеты были собраны еще через 48 часов. Статистическая значимость была рассчитана с использованием t-теста Стьюдента (ns = незначителен). Панели ошибок представляют SEM. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 1: Конвейер протеостаза. Загружаемый конвейер анализа изображений для использования в CellProfiler. Инструкцию по применению можно найти в разделе 9. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 2: Тренировочный набор распутывания червя. Файл для загрузки в модуль "UntangleWorms". Этот конкретный обучающий набор специфичен для червей, используемых в первоначальном подходе. Изменения в размере и форме червя изменят точность и качество идентификации. Может потребоваться создать более персонализированный обучающий файл. Инструкцию по созданию нового обучающего набора можно найти на официальном сайте CellProfiler17. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 3: Графический интерфейс пользователя для операционной системы Windows. gui_windowsOS_64x.zip. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 4: Графический интерфейс пользователя для операционной системы Mac. gui_MacOS_64x.zip. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Описанный протокол описывает процедуры культивирования, визуализации и обработки изображений C. elegans , которые включают CellProfiler, программное обеспечение для анализа изображений с открытым исходным кодом. Репрезентативные результаты демонстрируют воспроизводимость, уменьшение предвзятости и масштабируемость. Эта стандартизированная процедура улучшит стратегии скрининга, используемые с большими бактериальными, геномными или лекарственными библиотеками. В то время как существуют другие автоматизированные методы обнаружения объектов C. elegans , описанный метод предлагает стандартизированный конвейер с более высокой пропускной способностью, который объединяет культивирование, получение изображений и анализ.
Несколько вариантов культивирования червей должны были быть протестированы для оптимизации протокола, описанного в настоящем документе. Первоначально черви переносились на образцы бактерий сразу после возрастной синхронизации (стадия L1). Однако такой подход привел к популяции червей с переменными размерами, даже среди червей в пределах одной скважины. C. elegans известны избеганием патогенов19, что могло способствовать наблюдаемой изменчивости в размерах и в конечном итоге повлиять на обнаружение червей вниз по течению, в частности. Чтобы исключить такую изменчивость, вся площадь NGM в каждой скважине была покрыта тестовыми бактериями. Кроме того, червей кормили E. coli OP50 и позволяли полностью развиться в молодых людей в течение 48 ч при 25 °C. Позволив червям достичь совершеннолетия на E. coli OP50 до переноса их на тестируемые бактерии, привело к более стабильному размеру тела. Кроме того, перенаселенность и быстрое истощение продуктов питания потомством были устранены путем добавления агара NGM с FUDR. Внедрение FUDR удалило потомство и улучшило автоматическую идентификацию червей, которая была скрыта смешиванием потомства с родительской популяцией. Тем не менее, важно быть осторожным и использовать соответствующие средства контроля при использовании FUDR, так как соединение, как известно, влияет на протеостаз C. elegans и продолжительность жизни20,21. В условиях, описанных в этом протоколе, FUDR не влиял на агрегацию полиQ кишечника (дополнительный рисунок 5); поэтому его использование было подходящим и выгодным для описанного способа.
Замораживание образцов перед визуализацией оказалось критическим шагом в успешном использовании трубопровода. Совокупные подсчеты до замораживания были значительно выше, чем ручные подсчеты (диаграмма 3В). Сохранение червей при -20 °C в течение 18-48 ч до получения изображения снижало фоновую флуоресценцию и в конечном итоге улучшало обнаружение агрегатов (рисунок 3A). Влияние замораживания на обнаружение агрегатов было исследовано только для polyQ и не должно быть обобщено на другие модели без дальнейшего изучения таких эффектов.
Несмотря на то, что все условия сохраняются одинаковыми, было отмечено, что среднее число агрегатов на червя может варьироваться между различными прогонами, в то время как соотношение между количеством агрегатов у животных, колонизированных OP50, и MPAO1 остается неизменным (рисунок 6, дополнительный рисунок 2, дополнительный рисунок 5). Поэтому важно всегда включать E. coli OP50 control или любые дополнительные подходящие эталонные элементы управления в каждый запуск. На такую изменчивость совокупных подсчетов между экспериментами могут влиять условия окружающей среды (температура, влажность)22,23 или генетический фон8. Фактически было замечено, что после длительной культивирования флуоресценция кишечника резко снижалась или полностью терялась, что требовало размораживания нового штамма из замороженного запаса. Наблюдаемое снижение флуоресценции может быть результатом генетических изменений, которые подавляют токсичные трансгены, такие как экспрессирующие polyQ. Тем не менее, исключительная воспроизводимость результатов, наблюдаемых между различными экспериментаторами (дополнительный рисунок 2), между биологическими репликами (рисунок 5) и в пределах одной и той же выборки (дополнительный рисунок 3), подчеркивает силу этого подхода.
Многочисленные отчеты использовали кишечный polyQ для изучения протеостаза 9,11,12,13,24,25. Однако прямое сравнение результатов не может быть сделано из-за изменчивости экспериментальных подходов и методов считывания. Тем не менее, некоторые результаты из ранее опубликованных данных повторяются автоматизированной количественной оценкой, описанной в настоящем описании, включая бактериальную индукцию агрегации 9,13 и сопоставимое число агрегатов11. В совокупности описанный конвейер предлагает ценный инструмент для изучения протеостаза.
Способ, описанный в настоящем описании, имеет некоторые неотъемлемые проблемы. Например, требуется достаточно времени для освоения всех компонентов этого протокола, что особенно верно для раздела 8 протокола, который требует знакомства с анализом, чтобы определить, подходят ли полученные изображения для анализа конвейера. Возможны отклонения от настроек получения изображения, используемых в этом протоколе; однако, скорее всего, потребуется изменить настройки и набор для обучения червей. Этот конвейер может различать агрегаты различных размеров и те, которые касаются, что ограничивает «смешивание» агрегатов и в конечном итоге повышает чувствительность обнаружения. Однако при попытке идентифицировать крупные агрегаты, которые превышают допустимый диапазон размеров, могут возникнуть проблемы, поскольку расширение верхнего порогового значения размера может привести к ошибкам, вызванным плохой идентификацией, такой как неспособность дифференцировать агрегаты, которые касаются. Баланс между точностью, размером и интенсивностью должен быть найден до анализа изображения. Эффективность агрегированной идентификации может быть дополнительно улучшена путем включения машинного обучения для создания нейронной сети, способной улучшить обнаружение агрегатов. Такие усовершенствования в настоящее время изучаются и будут в значительной степени способствовать решению текущих проблем, таких как обнаружение агрегатов, которые лежат на разных фокальных плоскостях или имеют аномальные формы.
Одним из заметных недостатков описанного метода является изменчивость автоматизированных агрегатных подсчетов, поскольку они не всегда повторяются ручными подсчетами у червей, получавших различные бактериальные штаммы. Например, основываясь на автоматизированных подсчетах, черви, питавшиеся мутантом P. aeruginosa 53 (M53), имели значительно меньше агрегатов по сравнению со штаммом дикого типа (MPAO1) (рисунок 7); однако подтверждение хита не показало существенной разницы (дополнительный рисунок 4). В целом, высокопроизводительные скрининги лекарств имеют высокий уровень обнаружения ложноположительных попаданий, и описанный метод не является исключением26. Таким образом, это критическая часть протокола для подтверждения всех потенциальных попаданий.
Хотя этот протокол был оптимизирован для соответствия стратегии скрининга для идентификации бактерий, которые влияют на протеостаз хозяина, каждый шаг может быть дополнительно модифицирован для проверки эффекта геномных библиотек РНКI, малых молекул или других условий. На каждом этапе могут быть внесены дополнительные изменения в соответствии с требованиями конкретной стратегии скрининга. Кроме того, этот метод обеспечивает уровень гибкости, который позволяет оптимизировать каждый шаг в соответствии с конкретной моделью. Например, этот подход может быть распространен на агрегацию polyQ в других тканях или извлечение других признаков, обнаруженных на изображениях, таких как мониторинг экспрессии генов с использованием индуцируемых флуоресцентных репортеров (например, генов теплового шока), оценка субклеточной локализации белков (например, ядерная локализация DAF-16), изучение агрегации в других моделях заболеваний (Aβ1-42, α-синуклеин, TDP-43 и т. Д.) например, размер червя.
Раскрытие информации
Авторы заявили, что никакого конфликта интересов не существует.
Благодарности
Эта работа была поддержана Национальными институтами здравоохранения (1RO3AG069056-01) и Обществом инфекционных заболеваний Америки, финансирующим dMC. Спонсоры не играли никакой роли в разработке исследования, сборе и анализе данных, принятии решения о публикации или подготовке рукописи. Мы благодарим членов Czyz Lab за корректуру рукописи. Мультяшные фигурки были сгенерированы с использованием платной лицензии BioRender.
Материалы
| Name | Company | Catalog Number | Comments |
| 1.7 mL Microtubes | Olympus Plastics | Cat#24-282 | Microcentrifuge tubes |
| 10 mL Serological Pipettes | GenClone | Cat#12-104 | Plastic pipettes |
| 15 mL Centrifuge Tubes, Racked | Olympus Plastics | Cat#28-101 | Conical tubes |
| 5-Fluoro-2′-deoxyuridine | Fisher Scientific | D2235100MG | FUDR |
| Accu-jet Pro Pipette Controller | Genesee Scientific | 91-600RB | Pipette gun |
| Agar | Fisher Scientific | Cat#BP1423-2 | Granulated agar |
| BioRender | BioRender | Graphical figure generator | |
| Bleach (Regular) | Clorox | Bar# 044600324111, Splash-less Bleach | 4.5% sodium hypochlorite |
| C. elegans: AM140: rmIs132 [unc-54p::q35::yfp] | Morimoto Lab (Northwestern University) | AM140, Q35::YFP | Muscle polyQ |
| C. elegans: AM738: rmIs297[vha-6p::q44::yfp; rol-6(su1006)] | Morimoto Lab (Northwestern University) | AM738, Q44::YFP | Intestinal polyQ |
| CaCl2·2H2O | Fisher Scientific | Cat#C79-500 | Calcium chloride dihydrate |
| CellProfiler | Broad Institute | Image analysis software | |
| Cholesterol | Fisher Scientific | Cat#ICN10138201 | Cholesterol |
| Circulating Water Bath Head | Lauda | 26LE | Lauda E 100 |
| CoolLED pE300lite 365 dir mount STEREO | CoolLED | 8114931 | Fluorescent light control and emitter |
| 16 mL Culture Tubes | Olympus Plastics | 21-129 | Culture tubes, 17 mm x 100 mm |
| Escherichia coli OP50 | Caenorhabditis Genetics Center | OP50 | E. coli control strain |
| Ethanol, 200 Proof | Decon Labs, Inc. | 2701 | |
| Eyepiece 10x/23B, adjustable, 3d gen | Leica Microsystems | 10450910 | Eyepiece set |
| Filter Set ET GFP - MZ10F | Leica Microsystems | 10450588 | Filter cube |
| GraphPad Prism v9.2.0 | GraphPad Software, Inc. | Statistical analysis tool | |
| Heratherm Incubator IMP180 | Thermo | 51031562 | Refrigerated incubator |
| Innova 4000 | New Brunswick Scientific | M1192-0000 | Shaking incubator |
| K5 Camera | Leica Microsystems | 11547112 | Stereomicroscope camera |
| KH2P04 | Fisher Scientific | Cat#P285-3 | Potassium phosphate monobasic |
| LAS X Imaging Software | Leica Microsystems | Microscope imaging software | |
| Leica MZ10 F Optics Carrier | Leica Microsystems | 10450103 | Stereomicroscope |
| Levamisole | Fisher Scientific | Cat#0215522805 | Levamisole hydrochloride |
| Luria Broth (Lennox) | Apex Bioresearch Products | Cat#11-125 | LB |
| Magnetic Stir Plate | Fisher Scientific | 11-100-49S | Stir plate |
| MgSO4·7H2O | Alfa Aesar | A14491 | Magnesium sulfate heptahydrate |
| Microscope Slides | Premiere | 8205 | Single frosted microscope slides |
| Na2HPO4·7H2O | Fisher Scientific | Cat#S373-500 | Sodium phosphate dibasic heptahydrate |
| NaCl | Fisher Scientific | Cat#S671-500 | Sodium chloride |
| NaOH | Fisher Scientific | Cat#S318-3 | Sodium hydroxide pellets |
| Objective Achromat, f = 100 mm | Leica Microsystems | 10411597 | Objective microscope lens |
| Petri Dishes | Genesee Scientific | Cat#32-107G | 100 mm x 15 mm |
| Pseudomonas aeruginosa Mutant Library | Manoil Lab (University of Washington) | P. aeruginosa mutant library | |
| Suspension Culture Plate 24-Well, Flat Bottom | Olympus Plastics | 25-102 | Used for worm growth and imaging |
| Trinocular Tube 100% M-series | Leica Microsystems | 10450043 | |
| Trypticase Peptone | ThermoFisher, Difco | Cat#211921 | |
| TX-400 Rotor | Thermo Scientific | Cat#75003181 | Swing bucket rotor |
| Vacuum Driven Filter System | GenClone | Cat#25-227 | 500 mL, PES Membrane, .22 µm |
| Video Objective with C-Mount | Leica Microsystems | 10447367 | 0.63x camera adapter tube |
Ссылки
- Soto, C. Unfolding the role of protein misfolding in neurodegenerative diseases. Nature Reviews Neuroscience. 4 (1), 49-60 (2003).
- Fang, P., Kazmi, S. A., Jameson, K. G., Hsiao, E. Y. The microbiome as a modifier of neurodegenerative disease risk. Cell Host & Microbe. 28 (2), 201-222 (2020).
- Kundu, P., Blacher, E., Elinav, E., Pettersson, S. Our gut microbiome: The evolving inner self. Cell. 171 (7), 1481-1493 (2017).
- Sherwin, E., Dinan, T. G., Cryan, J. F. Recent developments in understanding the role of the gut microbiota in brain health and disease. Annals of the New York Academy of Sciences. 1420 (1), 5-25 (2018).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7 (1), 13023 (2016).
- Guisbert, E., Czyz, D. M., Richter, K., McMullen, P. D., Morimoto, R. I. Identification of a tissue-selective heat shock response regulatory network. PLoS Genetics. 9 (4), 1003466 (2013).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438 (2011).
- Nollen, E. A., et al. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Walker, A. C., et al. Colonization of the Caenorhabditis elegans gut with human enteric bacterial pathogens leads to proteostasis disruption that is rescued by butyrate. PLOS Pathogens. 17 (5), 1009510 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against α-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Kumsta, C., Chang, J. T., Schmalz, J., Hansen, M. Hormetic heat stress and HSF-1 induce autophagy to improve survival and proteostasis in C. elegans. Nature Communications. 8, 14337 (2017).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108, 14204-14209 (2011).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. Journal of Biological Chemistry. 283 (1), 194-201 (2008).
- Hakim, A., et al. WorMachine: machine learning-based phenotypic analysis tool for worms. BMC Biology. 16 (1), 8 (2018).
- Wählby, C., et al. An image analysis toolbox for high-throughput C. elegans assays. Nature Methods. 9 (7), 714-716 (2012).
- Jones, T. R., et al. CellProfiler Analyst: data exploration and analysis software for complex image-based screens. BMC Bioinformatics. 9 (1), 482 (2008).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomona aeruginosa PA01. Journal of Bacteriology. 194 (23), 6387-6389 (2012).
- Schulenburg, H., Ewbank, J. J. The genetics of pathogen avoidance in Caenorhabditis elegans. Molecular Microbiology. 66 (3), 563-570 (2007).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964 (2014).
- Van Raamsdonk, J. M., Hekimi, S. FUdR causes a twofold increase in the lifespan of the mitochondrial mutant gas-1. Mechanisms of Ageing and Development. 132 (10), 519-521 (2011).
- Haldimann, P., Muriset, M., Vígh, L., Goloubinoff, P. The novel hydroxylamine derivative ng-094 suppresses polyglutamine protein toxicity in Caenorhabditis elegans. Journal of Biological Chemistry. 286 (21), 18784-18794 (2011).
- Shinn-Thomas, J. H. Wrapping culture plates with Parafilm M® increases Caenorhabditis elegans growth. BMC Research Notes. 12 (1), (2019).
- Alexander-Floyd, J., et al. Unexpected cell type-dependent effects of autophagy on polyglutamine aggregation revealed by natural genetic variation in C. elegans. BMC Biology. 18 (1), 18 (2020).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Sink, R., Gobec, S., Pecar, S., Zega, A. False positives in the early stages of drug discovery. Current Medicinal Chemistry. 17 (34), 4231-4255 (2010).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены