需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
监测RNA结构平衡的变动情况“过氧化”和“氧化”羟基自由基足
摘要
本协议描述了如何量化的镁(二)依赖RNA的三级结构的形成由两个羟基自由基足迹方法。
摘要
RNA分子在生物学中发挥的重要作用。除了传递遗传信息,RNA折叠成独特的三级结构,实现特定的生物学作用,作为调节剂,粘结剂或催化剂。有关大专院校的联系形成的信息是必须了解的RNA分子的功能。羟基自由基(•OH)是独特的结构由于其反应活性高,体积小的核酸探针。,作为一个足迹探头使用时,羟基自由基地图的溶剂可及表面的DNA和RNA的磷酸骨干如单核苷酸分辨率罚款。羟基自由基足可以用来识别内分子间的接触面,例如在DNA -蛋白质和RNA -蛋白质复合物的核苷酸。 3平衡和动力学4转换可以由羟基自由基足迹作为一个soluti功能进行变量或时间,分别。的足迹的一个主要特点是有限的接触探头(例如,“单次击中动力学”)中的每个核苷酸的聚合物均匀采样的结果。5
在这个视频文章中,我们使用的四膜虫核酶的小四至小六的域名,说明RNA样品制备和一个镁(II)介导的折叠等温线的决心。我们描述了使用众所周知的羟基自由基足迹协议,要求H 2 O 2(我们称之为“过氧化”协议)和一个有价值的,但并不广为人知,替代使用自然溶解O 2的(我们称之为“氧化“协议)。提交的数据压缩,转换和分析程序概述。
研究方案
1。足迹试剂的制备
- 准备了10倍的反应缓冲液含有钠cacodylate 100毫米,1毫米EDTA和1个M氯化钾。调整pH值至7.4。使用0.2μm的醋酸纤维过滤设备(NALGENE)过滤器的缓冲区。注:不吸管的RNA直接到10倍缓冲区。
- 准备如表1所示,每个反应的滴定反应混合。滴定混合量(1X缓冲和镁(二)所需的浓度)应为90μL,然后加入1X缓冲的RNA加入10μl。
- 准备一个核糖核酸酶T1消化缓冲液6.63M尿素,柠檬酸钠20MM,为1mM EDTA,0.25微克/μL的tRNA,0.025%二甲苯蓝,0.025%溴酚蓝。这个缓冲区可以存储在4℃至6个月。
- Fenton反应需要250毫米的铁(NH 4)2(SO 4)2,250毫米EDTA,和500毫米L -抗坏血酸钠新鲜配制的水溶液。浓度在第1章指出.5是平衡的实验,如钠cacodylate缓冲区,不清除自由基和不同的二价离子浓度的最佳选择。
- 过氧化(1.5.1)和氧化(1.5.2)的足迹反应试剂。
- 过氧化Fenton反应需要100毫米的铁EDTA的解决方案,和50毫米钠L -抗坏血酸和1.5%H 2 O 2,新鲜配制的水溶液。准备用EDTA铁EDTA溶液混合铁(NH 4)2(SO 4)2,有超逾铁EDTA的1.1倍。
- 前发起的氧化裂解反应(3.4.2),创建相结合,2μL的Fe(NH 4)2(SO 4)2(250毫米),EDTA的2.2μL(250毫米),62.5μLFenton反应组合L -抗坏血酸钠(500毫米),22.5μL10X反应缓冲液和135.5μLH 2 O的终浓度达到0(3.4.2)的足迹反应混合物。,分别为10毫米,0.11毫米和6.61毫米。
2。足迹实验RNA的制备
- RNA可以在体外转录DNA模板6标准生产,如果少于50个核苷酸,购买(例如,在集成DNA技术,www.idtdna.com )。实验室按照建议的清洁程序,以保持在足迹实验样品RNA的完整性。
- 8脱磷,在体外转录RNA是必要的前 32 P标签的RNA的5'端。 10 pmol kinasing末端标记的RNA 9解决方案应该是成功的磷酸灭活后的阴天。
- 净化放射性标记的RNA变性凝胶电泳。随后的两个凝胶0.3 M醋酸钠(pH 5.5)中提取的RNA恢复。 RNA沉淀混合水溶液0.5毫升,1毫升乙醇和随后孵育1分钟干冰。在4 ° C,12500 RPM 0.5小时旋转样品。弃上清,用70%乙醇洗涤RNA沉淀。额外的离心去除上清液后,在真空干燥颗粒。
- P标记的 32 RNA样品溶解在330 UL 1X Buffer.Pipet 30μL的反应管中的RNA溶液,沉淀用乙醇和真空干燥RNA生成参考阶梯(步骤3.5),保持此管。变性加热的缓冲的RNA溶液在95 ° C为2分钟。样品冷却到室温为15分钟,快速旋转,使管放回解决方案的盖子上的冷凝液。
3。足迹实验
- 电泳梳,确定每分析凝胶的数据点的最大数量。我们使用30梳。参考阶梯占地2井和uncleaved控制之一。准备27个反应管。最后MG(二)concentrati项应均匀分布的对数遍及各地的滴定中点量级的规模。
- 另外,在50 RNA和镁(二)解决方案° C孵育5分钟。 90(二)镁相应数额在1x缓冲液10μLRNA溶液混合,达到100微升的最终体积。孵育30分钟的解决方案,在50 ° C。
- 让我们的解决方案,在25 ° C为1小时的平衡而发生折叠。
- 过氧化(3.4.1)(3.4.2)或氧化羟基自由基足迹反应。
- 开始放置铁EDTA(100毫米)2μL液滴的过氧化反应,反应的顶部内彼此分开2μLL -抗坏血酸钠(50毫米)和2μlH 2 O 2(1.5% )管内含有RNA的解决方案,并开始严厉混合的足迹反应。停止15秒后加入300μL冷无水乙醇的反应。转动管的3-5倍。沉淀,洗涤和在步骤2.4干燥沉淀。
- 加入5μL新鲜配制的Fenton反应混合物(1.6)开始氧化羟自由基footprintingreaction。孵育30分钟,在25 ° C。添加300μL冷无水乙醇淬火反应和组合转向管的3-5倍。沉淀,清洗和干燥的颗粒为在步骤2.4中表示。
- 根据标准程序10使用在步骤1.3编写的解决方案生成一个核糖核酸酶T1消化参考样本。
- 溶解于8μL凝胶上样染料II(Ambion公司),并确认RNA的RNA颗粒悬浮使用盖革计数器。
- 11。负载样本,包括两个引用和控制的标准协议,准备变性,8%聚丙烯酰胺测序胶。独立的RNA片段在60瓦 - 75 W为2.5小时。干凝胶置于存储荧光粉屏幕一夜之间。扫描成像系统胶片放射自显影,如荧光屏。风暴865(GE Healthcare公司)或台风(GE Healthcare公司)。凝胶图像文件传输到您的计算机。
4。数据分析
- 每个核苷酸的羟基自由基反应,是由每个频带的量化分析凝胶强度。下载,安装和开放SAFA,一个易于使用的开源软件为单波段的装修和量化12,13按照用户手册的说明。简而言之,负载的RNA序列。txt文件的后面。凝胶文件的凝胶图片。调整波段的强度,定义车道,选择一个锚车道,并执行凝胶对齐。频段分配到核苷酸发生在核糖核酸酶T1消化阶梯参考。乐队量化积分和使用的正常化/ colorplot功能正常化和分配潜在不变的残留物。另存为。txt文件的输出。
- SAFA输出电子表格,其中包含代表胶道和行代表在列tegrated波段相应的RNA片段的单个波段的密度。首先,确定保护的网站显示在溶剂可显着的变化,通过比较没有镁(II)与终点毫克(二)浓度在我们的例子(50毫米)的配置文件样本所得的个人资料的保护。该值越低,受保护的碱基对羟基自由基的攻击,反之亦然。
- 创建折叠等温线从个人或群组的核苷酸与镁(Ⅱ)浓度的归波段积分。等温线是单独缩放FI = L +(U - L)的分数饱和度•
 其中,f表示乐队的集成密度(S)分析,L和U代表的上限和下限过渡
其中,f表示乐队的集成密度(S)分析,L和U代表的上限和下限过渡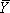 14的数据都适合使用非线性升是小数饱和。东广场分析程序(我们使用或者原产地(OriginLab)或GraphPad(GraphPad软件公司)。)向山式(1)
14的数据都适合使用非线性升是小数饱和。东广场分析程序(我们使用或者原产地(OriginLab)或GraphPad(GraphPad软件公司)。)向山式(1) 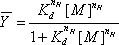
其中K,D是平衡解离常数,[M],对应的变量,可调节的折叠反应,镁(二)在这个例子中的浓度,N H Hill系数。此过程尺度过渡到小数饱和,决定了它的中点,并提供了一个过渡是否是S形的现象学测试。来自测序凝胶(图3A)SAFA(图3B)进行了分析和适合如上所述的等温线如图3C所示。如图3所示的分数饱和等温线所产生的电子表格中的缩放SAFA产生对最适合使用的方程分数的上限和下限(U和 L)值=值 /(U - L) - L /( U - L)。
5。代表性的成果:
图3显示了代表小四至小六的RNA•OH足迹实验的结果。凝胶图像(左)表示,一)背景乳沟是最小的(右线),B)单核苷酸分配是可能的,因为在T1巷(核糖核酸酶T1劈开每个G)定义的乐队,和c)的羟基自由基诱导的RNA碎片以及上述背景。从低到高镁(二)的过渡是隶属于下降表明RNA三级结构形成的乐队的个人和组的集成条带密度。单次或连续核苷酸的裂解变化随之而来被称为一个“保护”的群体。 •OH保护镁(二)特点的小四至小六RNA介导的折叠密切对应溶剂人迹罕至的地区,其晶体结构中观察到的分子。
折叠的RNA分子的彗星一个有非常明显的结构性或动态的现象,但没有与相关的大专接触保护的不同程度(即,如何接近背景带密度下降)。因此,一些RNA分子将显示定义良好的结构转换,如在图3所示,而有些则不会。乐队强度SAFA 12分析(图3B)量化和规范化。输出是一个“热”的,可视情节对•OH的相对程度的保护。颜色过渡介绍后,镁(二)除无障碍变化。白色红色或蓝色显示更容易或多个受保护的核苷酸,分别。每个阴影的程度,是隶属于一个数值,可以被绘制成一个保护曲线(图3C),如山式(1)通过一项具有约束力的模型分析。解离常数与保护153-155附属的平衡是两倍左右,相应的价值,保护163-164。
ontent“>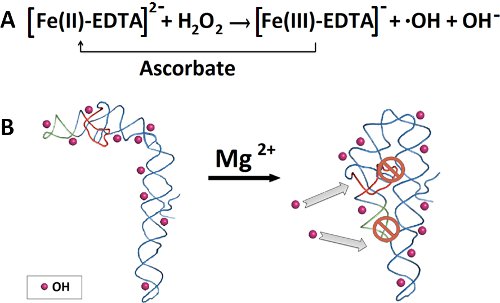
图1。羟自由基的生产和P4 - P6 RNA折叠。 A)铁(II)催化羟自由基的过氧化氢的生成。抗坏血酸亚铁降低铁(Ⅲ)回。 B)在镁(二)的情况下,没有小四至小六的三级结构形成,使羟基自由基渗透和切割所有可访问的骨干岗位。增加镁(二)启动折叠小四至小六,只允许溶剂可及骨干羟基自由基裂解。
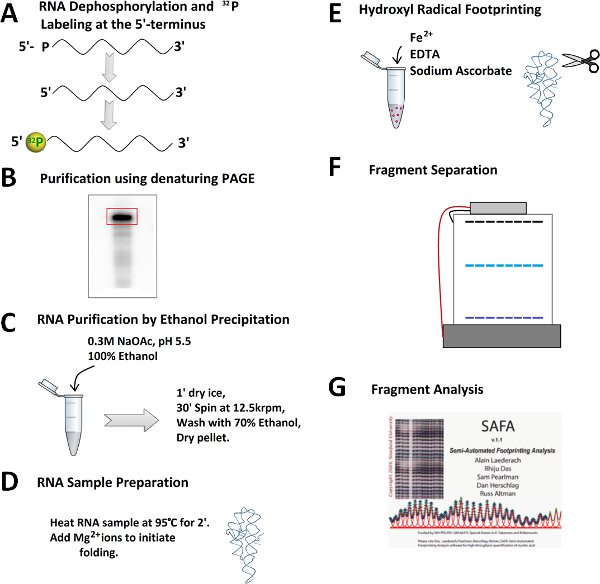
图2。大纲为羟基自由基足迹实验。一)Dephophorylation和32个P 5' -端的RNA标记。二)净化32 P标记在变性聚丙烯酰胺凝胶中的RNA。三)RNA的频段切除,随后的RNA提取,乙醇沉淀。 ð)Prefolding和折叠的RNA。五)新鲜准备的Fenton反应混合物除了生成羟基自由基。 F)的RNA片段,变性聚丙烯酰胺凝胶电泳分离。 G)的定量RNA片段SAFA软件。
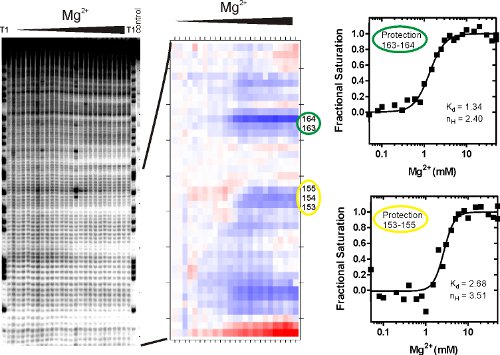
图3过氧化芬顿足迹反应后的RNA片段分析。 (一)的RNA被暴露的羟基自由基和卵裂产品使用变性聚丙烯酰胺凝胶电泳(PAGE)得到解决。图片显示,增加镁的浓度(二)过氧化裂解产物。参考和控制车道标记。此图像文件的SAFA的方案,其中定量每个频段的音量输入。 (二)“热”的情节所产生的SAFA。 (三)有关集成条带密度SAFA核苷酸的数量输出值的电子表格是如山equati绑定模型分析(1)。保护区被选为乐队Mg(II)浓度的功能不可或缺的密度增加。在K D是在被监控的网站,其中一半的RNA折叠的Mg(II)的浓度。 Hill系数,N H,是衡量斜坡曲线上具有约束力的协同信息,并提供了一个在这个特定的站点折叠的镁离子的数量的较低的估计。
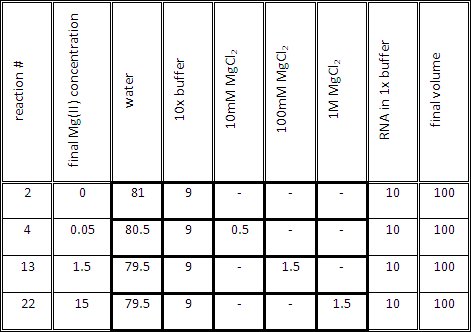
表1卷生成的RNA样品含有不同的Mg(II)浓度的代表。
讨论
羟基自由基足迹是一个有价值的工具,以评估核酸的溶剂可及表面面积。定性和定量的形成三级结构14,可以遵循作为一个功能参数,如离子种类和浓度,pH值,温度,结合蛋白或折叠的共同因素。引人注目的一条直线前进和廉价的协议,以及由此产生的溶剂可及单核苷酸水平上的折叠信息的结合,使得这种方法非常有吸引力的。传统的•OH足迹反应是增加H 2 O 2,我们称为“过氧?...
披露声明
没有利益冲突的声明。
致谢
这项工作是支持的国家卫生RO1 - GM085130研究所和国家科学基金会MCB0929394的资助。我们感谢她的盛情款待,让我们在她的实验室电影马里昂施密特博士。
材料
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
参考文献
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。