A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שיווי משקל ניטור שינויים ב-RNA על ידי מבנה 'Peroxidative' ו 'חמצוני "footprinting רדיקלי הידרוקסיל
In This Article
Summary
פרוטוקול זה מתאר כיצד לכמת את Mg (II) תלויי היווצרות של RNA מבנה שלישוני על ידי שתי שיטות footprinting רדיקלי הידרוקסיל.
Abstract
מולקולות רנ"א תפקיד חיוני בביולוגיה. בנוסף להעברת המידע הגנטי, RNA יכול להתקפל למבנה שלישוני ייחודי למלא תפקיד ביולוגיים ספציפיים כמו קלסר הרגולטור, או זרז. מידע על היווצרות קשר שלישוני הכרחי להבין את הפונקציה של מולקולות RNA. רדיקלים הידרוקסיל (OH •) הן בדיקות ייחודי של המבנה של חומצות גרעין בשל תגובתיות גבוהה שלהם בגודל קטן. 1 כאשר שימש בדיקה footprinting, רדיקלים הידרוקסיל למפות את פני השטח נגיש ממס של עמוד השדרה phosphodiester של ה-DNA ו-RNA 1 עם 2 קנס כמו רזולוציה נוקלאוטיד בודד. Footprinting רדיקלי הידרוקסיל יכול לשמש כדי לזהות את נוקלאוטידים בתוך משטח מגע מולקולאריים, למשל חלבון-DNA ו-RNA-1 חלבון קומפלקסים. שיווי משקל 3 ו 4 מעברים הקינטית ניתן לקבוע על ידי ביצוע footprinting רדיקלי הידרוקסיל כפונקציה של solutiעל משתנים או זמן, בהתאמה. תכונה מרכזית של footprinting היא כי חשיפה מוגבלת בדיקה (למשל, "יחיד פגע קינטיקה ') תוצאות דגימה אחידה של כל נוקלאוטיד של הפולימר. 5
במאמר זה וידאו, אנו משתמשים תחום P4-P6 של ribozyme Tetrahymena כדי להמחיש הכנה RNA מדגם וקביעת (II) בתיווך איזותרמות Mg מתקפלים. אנו מתארים את השימוש בפרוטוקול footprinting הידרוקסיל ידוע קיצוני הדורש H 2 O 2 (שאנו מכנים זה את פרוטוקול "peroxidative") לבין בעל ערך, אך לא ידוע, חלופה המשתמשת O טבעי מומס 2 (אנחנו קוראים לזה ' חמצוני "פרוטוקול). סקירה כללית של צמצום הנתונים, שינוי נהלים ניתוח מוצג.
Protocol
1. הכנה של ריאגנטים footprinting
- הכן חיץ התגובה 10x המכיל נתרן 100 mM cacodylate, 1 mM EDTA, ו 1 M KCl. התאם את ה-pH ל -7.4. סנן החיץ באמצעות אצטט 0.2 מיקרומטר מסנן למכשיר (Nalgene). הערה: לא RNA pipet ישירות לתוך חוצץ 10x.
- מכינים את תערובת התגובה טיטרציה עבור כל תגובה כמפורט בטבלה 1. נפח לערבב טיטרציה (1x חיץ Mg (II) בריכוז הרצוי) צריך להיות 90 μl, לפני הוספת 10μl של רנ"א במאגר 1x.
- הכן חיץ T1 RNAse העיכול המכיל אוריאה 6.63M, ציטרט הנתרן 20mm, 1mm EDTA, 0.25 מיקרוגרם / μl tRNA, cyanol קסילן 0.025%, וכחול bromphenol 0.025%. חיץ זה ניתן לאחסן 4 ° C עד 6 חודשים.
- תגובת פנטון דורשת תמיסות מימיות המוכן טרי של 250 מ"מ Fe (NH 4) 2 (SO 4) 2, 250 mM EDTA, נתרן 500 mM ו-L-ascorbate. ריכוזים ציינו בפרק 1.5 הם אופטימליים עבור ניסויים שיווי משקל חיץ כגון cacodylate נתרן שאינו לחפש רדיקלים ומגוונים ריכוזי יון divalent.
- ריאגנטים עבור התגובה (1.5.2) peroxidative (1.5.1) ו footprinting חמצוני.
- התגובה peroxidative פנטון דורשת תמיסות מימיות של פתרון מוכן טרי 100 Fe-EDTA מ"מ, ו 50 נתרן mM L-ascorbate ו -1.5% H 2 O 2. הפתרון Fe-EDTA הוא מוכן על ידי ערבוב Fe (NH 4) 2 (SO 4) 2 עם EDTA יש פי 1.1 עודף של EDTA על ברזל.
- רגע לפני שיזם את התגובה מחשוף חמצוני (3.4.2), ליצור את התמהיל התגובה פנטון על ידי שילוב של 2 μl Fe (NH 4) 2 (SO 4) 2 (250 מ"מ), 2.2 μl של EDTA (250 מ"מ), 62.5 μl נתרן L-ascorbate (500 מ"מ), 22.5 תגובה μl חיץ 10x ו 135.5 μl של H 2 O להגיע אל ריכוזי הסופי בתערובת footprinting תגובה (3.4.2) של 0.10 מ"מ, 0.11 מ"מ, 6.61 מ"מ, בהתאמה.
2. RNA הכנה לקראת ניסוי footprinting
- RNA יכול להיות מיוצר על ידי תקן שעתוק במבחנה של תבניות DNA 6 או, אם פחות מ 50 נוקלאוטידים, ניתן לרכוש (למשל ב-DNA משולב טכנולוגיות, www.idtdna.com). עקבו מומלץ ניקיון נהלים מעבדה כדי לשמור על שלמות מדגם של רנ"א במהלך הניסויים footprinting 7.
- Dephosphorylation 8 של במבחנה RNA עיבד הכרחי לפני תיוג 32 P הסוף "5 של רנ"א. 10 pmol של רנ"א הם סוף שכותרתו ידי kinasing. 9 הפתרון צריך להיות מעונן לאחר איון מוצלח של phosphatase.
- לטהר RNA מסומן רדיואקטיבית בעזרת ג'ל אלקטרופורזה denaturing. רנ"א הוא התאושש לאחר מכן על ידי שתי עקירות באמצעות ג'ל 0.3 נתרן אצטט M (pH 5.5). RNA הוא זירז ידי ערבוב 0.5 פתרון מ"ל מימית עם אתנול 1 מ"ל ובעקבותהדגירה דקה 1 על קרח יבש. ספין מדגם ב 4 ° C, 12,500 סל"ד עבור 0.5 שעות. בטל supernatant ולשטוף RNA גלולה עם אתנול 70%. הסר supernatant לאחר צנטריפוגה נוספים יבש גלולה בחלל ריק.
- ממיסים את P-32 שכותרתו דגימות רנ"א ul 330 μl של 1x Buffer.Pipet 30 הפתרון RNA בצינור תגובה, המשקע עם אתנול ו-RNA יבש בחלל ריק; לשמור על צינור להפקת הסולם הפניה (שלב 3.5). לפגל הפתרון שנאגרו RNA על ידי חימום על 95 מעלות צלזיוס למשך 2 דקות. מגניב דגימות לטמפרטורת החדר במשך 15 דקות ולתת סיבוב מהיר כדי להפיל את ותרכיזים על העפעפיים של צינורות אל הפתרון.
3. ניסוי footprinting
- מסרק אלקטרופורזה קובע את המספר המרבי של נקודות נתונים לכל ג'ל אנליטית. אנו משתמשים גם מסרק 30. סולם התייחסות תופסת 2 בארות אחד לשלוט uncleaved. הכן 27 מבחנות התגובה. Mg הסופי (II) concentratiתוספות צריכה להיות מחולקת באופן שווה על סולם לוגריתמי פורש סדרי גודל סביב אמצע טיטרציה.
- בנפרד, דגירה את הרנ"א (II) פתרונות Mg ב 50 מעלות צלזיוס למשך 5 דקות. מערבבים 10 μl של פתרון RNA עם μl 90 סכום המקביל Mg (II) במאגר 1x להגיע נפח סופי של 100 μl. דגירה הפתרונות למשך 30 דקות ב 50 ° C.
- בואו הפתרונות לאזן במהירות של 25 מעלות צלזיוס במשך שעה 1 תוך קיפול מתרחשת.
- Peroxidative (3.4.1) או חמצוני (3.4.2) footprinting הידרוקסיל תגובה קיצונית.
- התחל את התגובה peroxidative ידי הצבת טיפות של μl 2 Fe-EDTA (100 מ"מ), 2 נתרן μl L-ascorbate (50 מ"מ) 2 μl H 2 O 2 (1.5%) מופרדים זה מזה על החלק הפנימי העליון של התגובה שפופרת המכילה את הפתרון RNA ולהתחיל את התגובה footprinting ידי ערבוב נמרץ. עצור את התגובה לאחר 15 שניות על ידי הוספת 300 μl אתנול אבסולוטי קר. סובבו את הצינורות 3-5 פעמים. , לזרז לרחוץיבש את הכדור כמו בשלב 2.4.
- הפעל את footprintingreaction חמצוני רדיקלי הידרוקסיל ידי הוספת 5 μl של תמהיל פנטון מוכן טרי התגובה (1.6). דגירה למשך 30 דקות במהירות של 25 ° C. הוסף 300 μl של אתנול אבסולוטי קר להרוות את התגובה ומערבבים על ידי סיבוב צינורות 3-5 פעמים. נמהרת, לשטוף ולייבש גלולה כמצוין בשלב 2.4.
- צור T1 RNase מדגם לעכל התייחסות פי הנהלים תקן 10 באמצעות פתרון מוכן בשלב 1.3.
- ממיסים כדורי רנ"א 8 μl ג'ל טעינת צבע II (AMBION) ולאשר RNA הוא resuspended באמצעות מונה גייגר.
- הכן denaturing, 8% רצף ג'ל polyacrylamide על פי פרוטוקולים סטנדרטיים. 11 דגימות טען כולל שתי הפניות שליטה. הפרד RNA שברים ב 60 וואט - 75 וואט עבור 2.5 שעות. לחשוף ג'ל יבשים למסך אחסון זרחן לילה. סריקה זרחן מסך עם מערכת הדמיה עבור autoradiography filmless, למשל. סטורם 865 (GE Healthcare), או טייפון (GE Healthcare). העברת קובץ התמונה ג'ל למחשב.
4. ניתוח נתונים
- תגובתיות רדיקלי הידרוקסיל של כל נוקלאוטיד נקבעת על ידי כימות עוצמת הלהקה על כל ג'ל האנליטי. הורד, התקן ולפתוח ספא, קל לשימוש בתוכנות קוד פתוח עבור הולם הלהקה כימות יחיד. 12, 13 בצע את ההוראות במדריך למשתמש. בקצרה, לטעון את רצף הרנ"א כמו. קובץ txt ואחריו התמונה ג'ל כקובץ. ג'ל. התאם בעוצמות הלהקה, להגדיר את הנתיבים, לבחור במסלול עוגן, ולבצע יישור ג'ל. הקצאה של להקות כדי נוקליאוטידים מתרחשת התייחסות סולם העיכול T1 RNase. לכמת את אינטגרלים הלהקה להשתמש בתכונת נורמליזציה / colorplot לנרמל ולהקצות שאריות משתנה פוטנציאלי. שמור את הפלט כקובץ txt..
- ספא פלטי גיליון אלקטרוני המכיל עמודות המייצגות נתיבים על הג'ל שורות המייצג את בtegrated צפיפות הלהקה של להקות בודדות המקביל לקטע RNA. ראשית, לזהות את האתרים שבהם הגנה להציג שינוי ניכר בנגישות ממס על ידי השוואת פרופיל הגנה נגזר מדגם לא Mg (II) עם פרופיל של ריכוז נקודת הסיום (II) Mg (50 מ"מ בדוגמא שלנו). הנמך את הערך, המוגן יותר נוקלאוטיד הוא נגד ההתקפה מפני רדיקלים הידרוקסיל ולהיפך.
- צור איזותרמות מתקפל מן אינטגרלים הלהקה מנורמל של יחידים או קבוצות של נוקליאוטידים לעומת ריכוז (II) Mg. איזותרמות הם מדורגים בנפרד הרוויה השבר על ידי fi = L + (U - L) •
 היכן f מציין את צפיפות משולב של הלהקה (ים) להיות מנותח, L ו-U מייצגים את גבולות העליון והתחתון המעבר ו
היכן f מציין את צפיפות משולב של הלהקה (ים) להיות מנותח, L ו-U מייצגים את גבולות העליון והתחתון המעבר ו 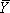 היא רוויה חלקיים. 14 הנתונים הם בכושר באמצעות l שאינו ליניאריממזרח לכיכר תוכנית ניתוח (אנו משתמשים גם מקור (OriginLab) או GraphPad (GraphPad תוכנה, Inc).) למשוואה היל (1),
היא רוויה חלקיים. 14 הנתונים הם בכושר באמצעות l שאינו ליניאריממזרח לכיכר תוכנית ניתוח (אנו משתמשים גם מקור (OriginLab) או GraphPad (GraphPad תוכנה, Inc).) למשוואה היל (1), 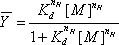
היכן K d הוא קבוע הדיסוציאציה בשיווי משקל, [ז] מתאים לריכוז של המשתנה כי מתווכת התגובה מתקפלים, Mg (II) בדוגמה זו, H n הוא מקדם היל. זה קשקשים הליך המעבר הרוויה השבר, קובע נקודת האמצע שלה מספק מבחן הפנומנולוגית של אם המעבר הוא sigmoidal. איזותרמות נגזר רצף ג'לים (איור 3A) אשר נותחו על ידי סאפא (איור 3B) ובכושר כמתואר לעיל מוצגים באיור 3C. איזותרמות הרוויה השבר שמוצג באיור 3 נוצרו על ידי דרוג בגיליון אלקטרוני הערכים שנוצרו על ידי ספאא נגד גבולות הטוב העליון והתחתון מתאים (U ו-L) באמצעות חלק במשוואה = ערך / (U - L) - L / (U - L).
5. נציג תוצאות:
איור 3 מציג תוצאות נציג P4-P6 RNA • ניסויים OH footprinting. התמונה ג'ל (משמאל) מציין כי המחשוף) הרקע הוא מזערי (נתיב ימין), ב) הקצאה נוקלאוטיד יחיד אפשרי בגלל להקות הגדיר היטב את השביל T1 (RNase cleaves T1 בכל G), ו - ג) רדיקלי הידרוקסיל הפיצול המושרה של RNA הוא הרבה מעל הרקע. המעבר מ נמוך Mg גבוהה (II) המזוהה עם הפחתת צפיפות הלהקה משולבת של בודדים וקבוצות של להקות המעיד על היווצרות של RNA מבנה שלישוני. יחיד או קבוצות של נוקלאוטיד רציף אשר מחשוף שינויים במקביל מכונים 'הגנה'. • הגנות OH האופייניים של Mg (II) בתיווך קיפול של P4-P6 RNA מקרוב מתאימות האזורים נגיש ממס של המולקולה שנצפתה המבנה הגבישי שלה. 15
מולקולות רנ"א מקופל ג יש ובמידות שונות מאוד (כלומר, כמה קרוב רקע הירידה בצפיפות הלהקה) של הגנה קשר שלישוני שלא היה בקורלציה עם תופעה מבנית או דינמי ברור. לכן, כמה מולקולות רנ"א יראה מעברים מבנית מוגדרת היטב, כפי שמוצג באיור 3, וחלק לא. עוצמות בנד הם לכמת מנורמל על ידי ניתוח 12 צפא (איור 3B). הפלט הוא מזימה "תרמית" לדמיין את מידת היחסי של הגנה מפני • OH. המעבר צבע מתאר את השינוי על נגישות בנוסף (II) Mg. הלבן מראה אדום או כחול נוקלאוטידים נגיש יותר או מוגנת יותר, בהתאמה. מידת כל של הצללה המזוהה עם ערך מספרי אשר ניתן להתוות כמו עקומת הגנה (איור 3 ג) ונותחו על ידי מודל מחייב כגון המשוואה היל (1). שיווי משקל קבוע דיסוציאציה המזוהים עם הגנת 153-155 הוא בערך כפול מזה של הערך המקביל להגנת 163-164.
ontent ">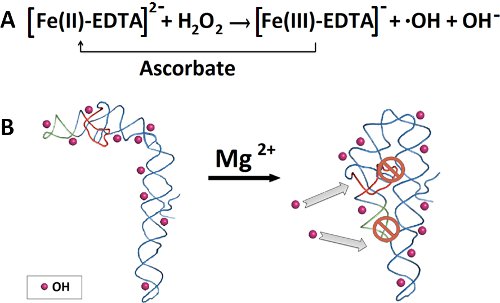
באיור 1. ייצור רדיקלי הידרוקסיל ו P4-P6 קיפול RNA. א) Fe (II) מזרז לייצור של רדיקלים של הידרוקסיל מי חמצן. Ascorbate מפחית Fe (III) חזרה ברזל ברזל. ב) בהעדר Mg (II), מבנה לא P4-P6 שלישוני נוצר, המאפשר רדיקלים הידרוקסיל לחדור לפלח כל הפוזיציות עמוד השדרה נגיש. תוספת של Mg (II) יוזם קיפול של P4-P6, המאפשר רק את עמוד השדרה נגיש ממס להיות ביקע ידי רדיקלים הידרוקסיל.
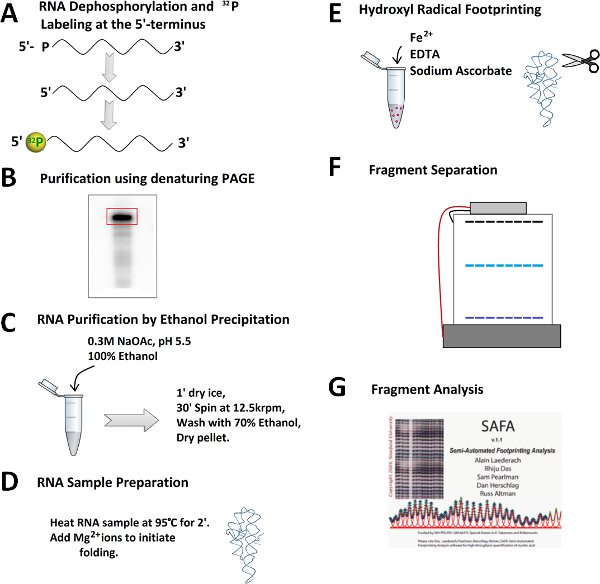
באיור 2. תמציתי לניסוי footprinting הידרוקסיל רדיקל. Dephophorylation) ו - 32 P 5'-end תיוג של רנ"א. ב) טיהור של 32 P-RNA מסומן על ג'ל polyacrylamide denaturing. ג) כריתה של הלהקה RNA, לאחר מיצוי RNA, משקעים אתנול. D) ו Prefoldingקיפול של רנ"א. ה) תוספת של תערובת פנטון טרי שהוכן התגובה ליצירת רדיקלים הידרוקסיל. F) RNA ההפרדה שבר ידי denaturing ג'ל אלקטרופורזה polyacrylamide. G) quantitation של שברי RNA על ידי תוכנה סאפא.
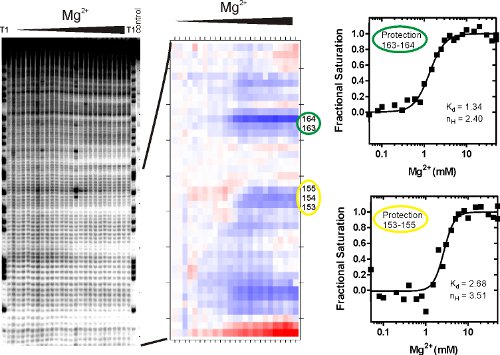
באיור 3. ניתוח של שברי RNA לאחר התגובה פנטון peroxidative footprinting. (א) RNA נחשף רדיקלים הידרוקסיל והמוצרים מחשוף נפתרו באמצעות denaturing ג'ל אלקטרופורזה polyacrylamide (עמוד). התמונה מראה המוצרים מחשוף peroxidative עם ריכוז גדל והולך של Mg (II). נתיבי הפניה ושליטה מסומנים. זה קובץ תמונה הוא קלט לתוכנית ספא אשר quantitates נפח הלהקה כל אחד. (ב) מזימה "תרמית" שנוצר על ידי סאפא. (ג) גיליון אלקטרוני של ערכים הנוגעים צפיפות הלהקה משולבת פלט מספר נוקלאוטידים מתוך ספא מנותח על ידי מודל מחייב כגון equati הילעל (1). אזורי הגנה נבחרו על פי להגדיל את צפיפות הלהקה אינטגרלי כפונקציה של ריכוז (II) Mg. ד K הוא הריכוז (II) Mg שבו מחצית RNA הוא מקופל באתר במעקב. מקדם היל, n H, הוא מדד של השיפוע של העקום אשר נותן מידע על cooperativity מחייב מספק הערכה נמוכה יותר עבור מספר יוני מגנזיום מעורב קיפול של האתר המסוים הזה.
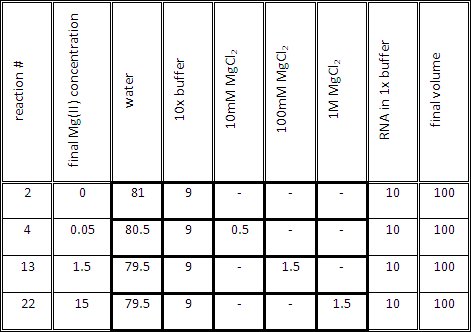
טבלה 1. כרכים נציג ליצירת דגימות RNA המכיל שונים (II) ריכוזים Mg.
Discussion
Footprinting רדיקלי הידרוקסיל היא כלי רב ערך כדי להעריך את אזור ממס משטח נגיש של חומצות גרעין. היווצרות איכותי וכמותי של מבנה שלישוני 14 ניתן בעקבות כפונקציה של פרמטרים כגון סוג הריכוז יונים, pH, טמפרטורה, או קיפול חלבונים מחייב שיתוף גורמים. שילוב מרתק של פרוטוקול קדימ...
Disclosures
אין ניגודי אינטרסים הכריז.
Acknowledgements
עבודה זו נתמכה על ידי מענקים מהמכון הלאומי לבריאות RO1-GM085130 ואת הקרן הלאומית למדע MCB0929394. אנו מודים לד"ר שמידט מריון על האירוח שלה עבור ומאפשר לנו הסרט במעבדה שלה.
Materials
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
References
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved